小麦是我国主要的粮食品种之一,对国家粮食安全具有重要的战略意义。小麦容易发生真菌污染,导致小麦品质下降,并产生真菌毒素。若含有真菌毒素的小麦被人食用,则可能会导致肝炎、肝癌等严重危害人体健康的疾病[1-2]。市场调查结果显示,小麦中真菌毒素的检出率明显高于其他谷物[3],说明小麦的真菌污染的情况比其他谷物严重。小麦真菌污染可能发生在生产、储存、运输、销售等各个阶段,其中小麦仓储过程由于时间周期长、麦粒密度大,极易发生真菌污染的加重和扩散,是决定小麦真菌污染严重程度的关键环节之一。
研究小麦仓储过程中真菌污染的发生规律是降低小麦真菌污染的前提条件和有效途径。目前在此方面的研究主要集中在水分和温度对仓储小麦真菌发生的影响[4-6],发现了抑制真菌发生的最佳湿度和温度,从而为小麦仓储的温湿度条件设置提供了参考。在仓储过程中,小麦中的真菌群落并非一成不变,而是随着时间和空间发生动态变化,因此研究小麦储存过程中真菌群落的时空变化对于小麦仓储真菌污染控制具有指导意义。本研究团队在之前的研究中应用变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)技术探究了玉米储藏过程中的霉菌群落时空分布特征[7],但在小麦领域尚鲜见报道。
研究微生物群落的方法主要有3 类,即培养法、传统分子生物学方法和高通量测序方法[8-9]。培养法通过分离培养研究样品中的微生物群落。由于在培养过程中各种微生物的生长速率不同,导致培养后的微生物群落组成与样品中原始的微生物群落有明显差异。用于研究微生物群落的传统分子生物学方法包括DGGE法[10]、随机扩增多态性DNA技术[11]、限制性片段长度多态性技术[12]等,但这些方法通常只能研究微生物群落中高丰度菌株。高通量测序方法研究微生物多样性具有灵敏度、准确率高等优势,己被广泛应用于各类样品的真菌群落研究中[13-16]。本实验采用高通量测序技术,对河南和黑龙江两地、不同储藏时间、不同储藏位置小麦样品的真菌群落进行研究,为小麦仓储过程中真菌污染的控制提供指导。
1 材料与方法
1.1 材料与试剂
小麦样品采自河南省和黑龙江省,样品编号及相关信息如表1所示。
从河南嵩县储藏粮库采集小麦,粮库中小麦品种主要为西农928、洛旱9号、运旱115、洛旱15。从黑龙江双鸭山粮库中采集储藏小麦,粮库中小麦品种主要为东农121号、垦九9号、辽春14号小麦、小麦新品种东农024。河南小麦采样时间为2016年8月9日,黑龙江小麦取样时间为2016年8月23日。每个样品采样质量约2 kg。粮库南侧、粮库北侧距离墙壁距离大约1~1.5 m。同一个年份的样品是从一个粮仓中取。
表1 小麦样品编号及相关信息
Table 1 Information about wheat samples tested in this study
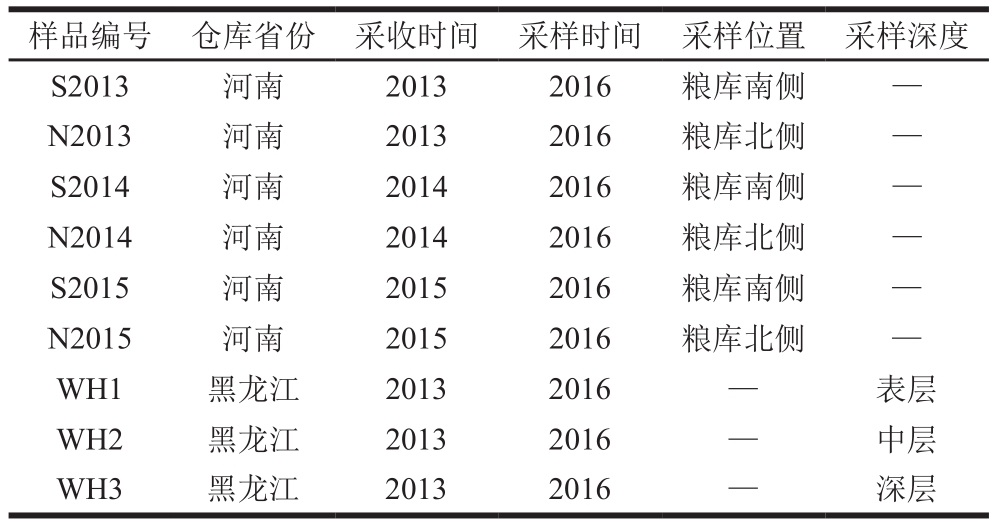
样品编号 仓库省份 采收时间 采样时间 采样位置 采样深度S2013 河南 2013 2016 粮库南侧 —N2013 河南 2013 2016 粮库北侧 —S2014 河南 2014 2016 粮库南侧 —N2014 河南 2014 2016 粮库北侧 —S2015 河南 2015 2016 粮库南侧 —N2015 河南 2015 2016 粮库北侧 —WH1 黑龙江 2013 2016 — 表层WH2 黑龙江 2013 2016 — 中层WH3 黑龙江 2013 2016 — 深层
Phusion超保真DNA聚合酶 美国New England Biolabs公司;高通量测序建库试剂盒 美国Illumina公司。
1.2 仪器与设备
KQ-250B超声波振荡器 中国上海早盈公司;H1850R低温高速离心机 湘仪离心机仪器有限公司;聚合酶链式反应(polymerase chain reaction,PCR)扩增仪 美国Biometra公司;HiSeq 2500 PE250高通量测序仪 美国Illumina公司。
1.3 方法
1.3.1 小麦中真菌基因组DNA提取
采用改良的十六烷基三甲基溴化铵(cetyltrimethyl ammonium bromide,CTAB)法提取小麦的真菌[17-19]。具体参考文献[7]中的玉米中真菌提取方法。
1.3.2 PCR扩增及高通量测序
PCR扩增及高通量测序主要委托北京诺禾致源生物信息科技有限公司完成。具体方法为利用带有标签的ITS1区通用引物(ITS5-1737F和ITS2-2043R)对样品真菌基因组DNA进行扩增。根据PCR产物浓度进行等量混样,充分混匀后使用2%的琼脂糖凝胶进行电泳检测,对目的条带使用Qiagen公司提供的胶回收试剂盒回收产物。使用高通量测序建库试剂盒进行文库构建,构建好的文库经过Qubit和Q-PCR定量,文库合格后,使用HiSeq 2500 PE250进行测序。
1.3.3 生物信息学分析及统计分析
原始数据根据标签序列信息进行拆分,得到样品序列信息。序列经过去除标签序列、去除引物序列、序列拼合、去除低质量序列后,得到有效序列。有效序列利用QIIME软件[20]进行97%相似度的可操作分类单元(operational taxonomic units,OTUs)聚类,代表序列提取,基于GreenGene数据库对代表序列进行注释,得到每个样品中每种OTU的序列数量。利用PAST软件[21]对样品中真菌群落OTU数据进行非度量多维尺度分析(nonmetric multidimensional scaling,NMDS)和Simpson多样性指数分析。利用Draw Venn Diagram在线工具(http://bioinformatics.psb.ugent.be/webtools/Venn/)对样品中真菌群落OTU数据进行韦恩图分析。利用在线分子生态网络分析工具[22]对小麦真菌群落中各物种的关联性进行分析。利用LDA Effect Size(LEfSe)[23]在线工具对不同组之间有显著差异的真菌种类进行分析。利用STEM软件[24]对不同深度小麦中真菌群落的分布做趋势分析。
2 结果与分析
2.1 小麦储藏过程中真菌群落的一般结构特征
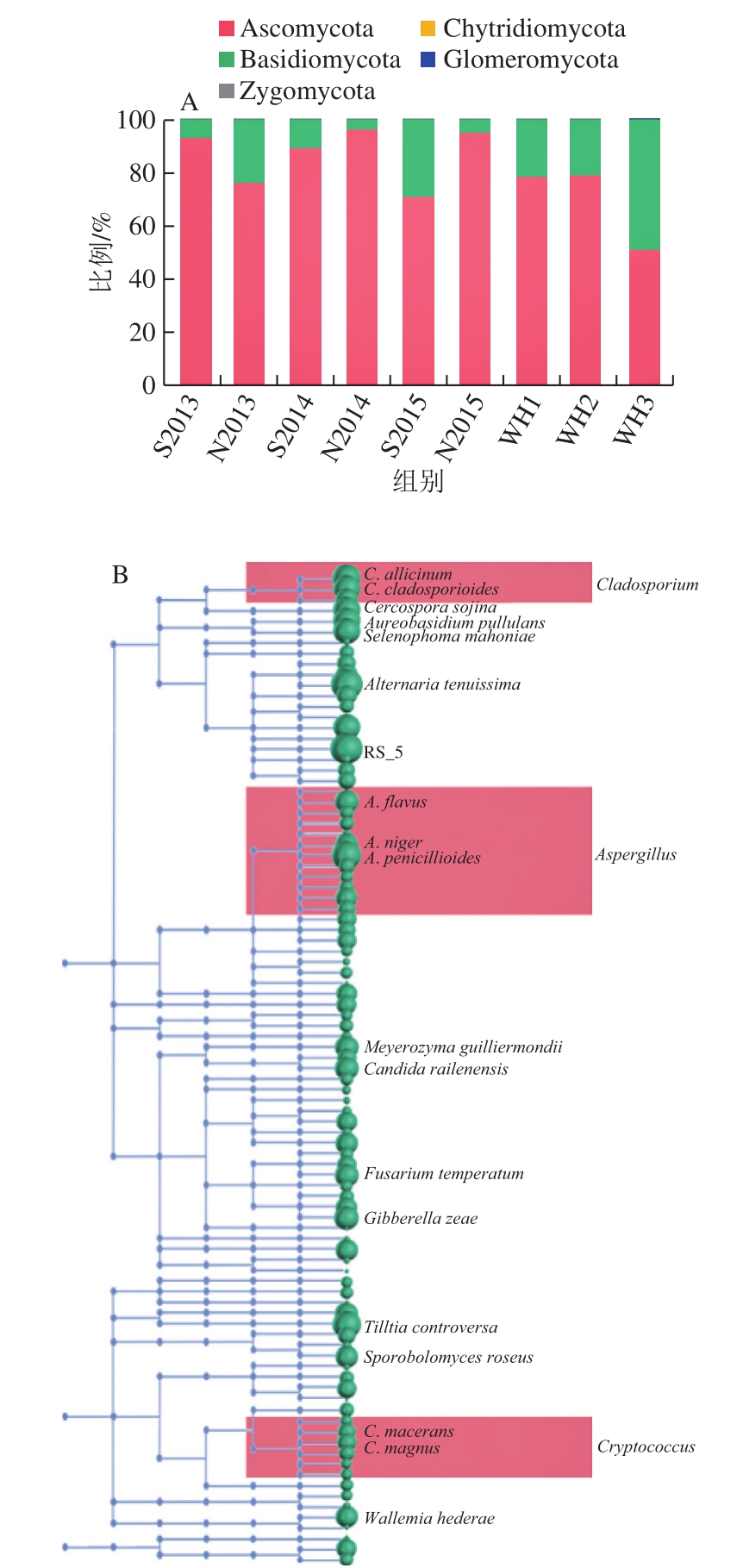
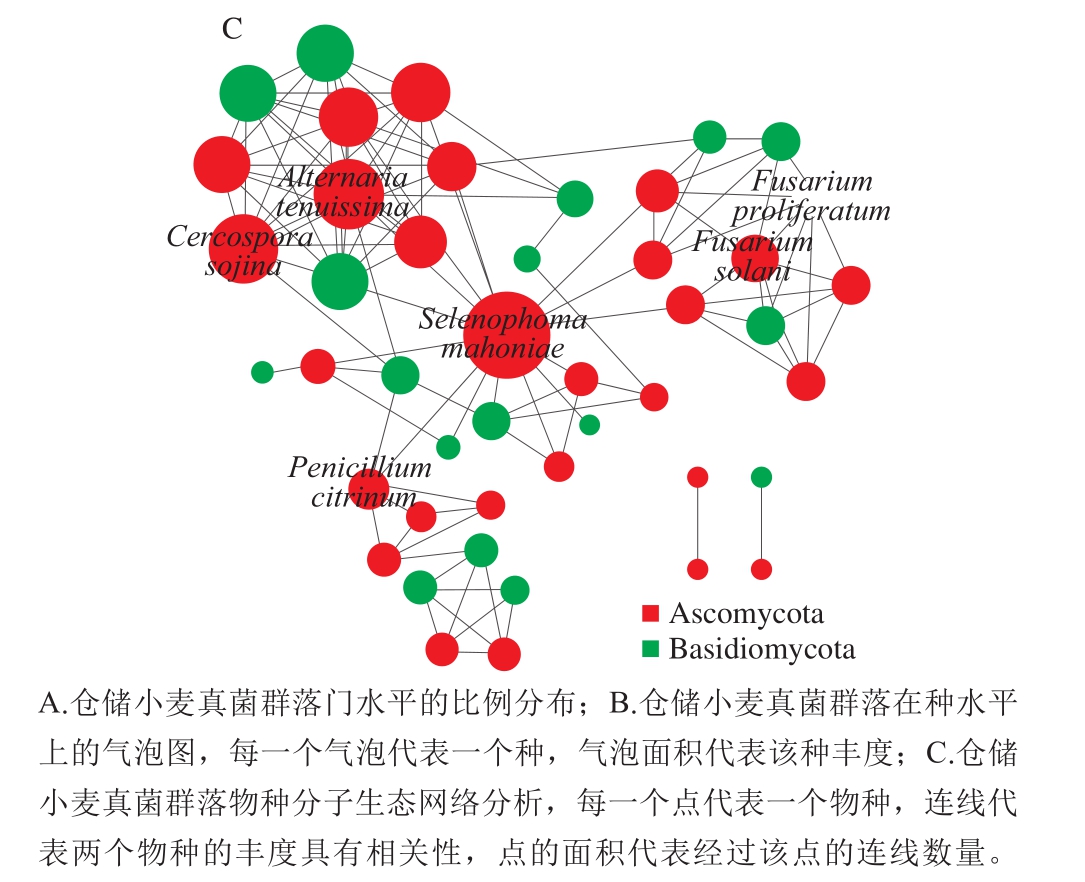
图1 小麦储藏过程中真菌群落的一般结构特征
Fig. 1 General structural characteristics of fungal communities during wheat storage
如图1 A所示,储粮小麦中真菌以子囊菌门(Ascomycota)和担子菌门(Basidiomycota)为主,二者之和占到全部真菌的99.6%以上,其他低丰度微生物属于接合菌门(Zygomycota)、壶菌门(Chytridiomycota)、球囊菌门(Glomeromycota)。不同样品中子囊菌门微生物与担子菌门微生物的比例差异较大,子囊菌门微生物的比例通常大于担子菌门微生物的比例。如图1B所示,在属的分类水平上,物种多样性最高的属为链格孢属(Alternaria)、曲霉属(Aspergillus)和枝孢霉属(Cladosporium)是测序结果中丰度最高的己知菌属,其中曲霉属测得12 个种,包括亮白曲霉(A. candidus)、黄曲霉(A. flavus)、烟曲霉(A. fumigatus)、嗜盐曲霉(A. halophilicus)、蜂蜜曲霉(A. melleus)、黑曲霉(A. niger)、帚状曲霉(A. penicillioides)、局限曲霉(A. restrictus)、曲霉属NRRL 145(Aspergillus sp. NRRL 145)、土曲霉(A. terreus)、小麦曲霉(A. tritici)和杂色曲霉(A. versicolor)。在种的水平上,共发现93 种真菌,平均丰度最高的种为Pleosporaceae sp. RS_5、极细链格孢(A. tenuissima)、帚状曲霉、小麦黑穗菌(Tilletia controversa)、蒜状枝孢菌(C. allicinum)和枝状枝孢菌(C. cladosporioides)。分子生态网络分析可以揭示各种真菌在数量关系上的相关性,结果如图1C所示,Selenophoma mahoniae、极细链格孢(A. tenuissima)和索伊纳尾孢菌(Cercospora sojina)是分子生态网络的中心位置,与其他菌种具有最为丰富的相关性,说明这些菌种可能在仓储小麦真菌群落中起到核心作用。
2.2 小麦储藏时间对其中真菌群落结构的影响
本研究于2016年从河南省粮仓采集入库年份分别为2013年(S2013、N2013)、2014年(S2014、N2014)、2015年(S2015、N2015)的小麦样品,分别储藏3、2、1 a。NMDS图在二维图谱上绘制各个样品的菌群特征,图上两点距离越近,代表菌群特征越相近。由图2A可知,储藏3 a的小麦样品与储藏1 a的小麦样品分别聚类在图谱的不同区域,而储藏2 a的小麦样品中真菌群落介于储藏1 a和储藏3 a的真菌群落之间,可能处于过渡状态。上述结果表明,小麦中真菌群落可能与小麦的储藏时间有关。S2014和N2014在图谱坐标系上的距离较远,这可能是由于过渡状态群落物种组成不稳定,变化较大导致的,其中N2014样品与储藏3 a的样品在图谱上距离较近,说明N2014样品中的真菌群落处于过渡状态的后期,而图谱上S2014样品距离储藏3 a和储藏1 a的样品距离均较远,说明S2014样品中的真菌群落可能处于过渡中期。在多样性方面,小麦真菌Simpson指数随着储藏时间的延长而略微上升,但各组之间均无显著差异。
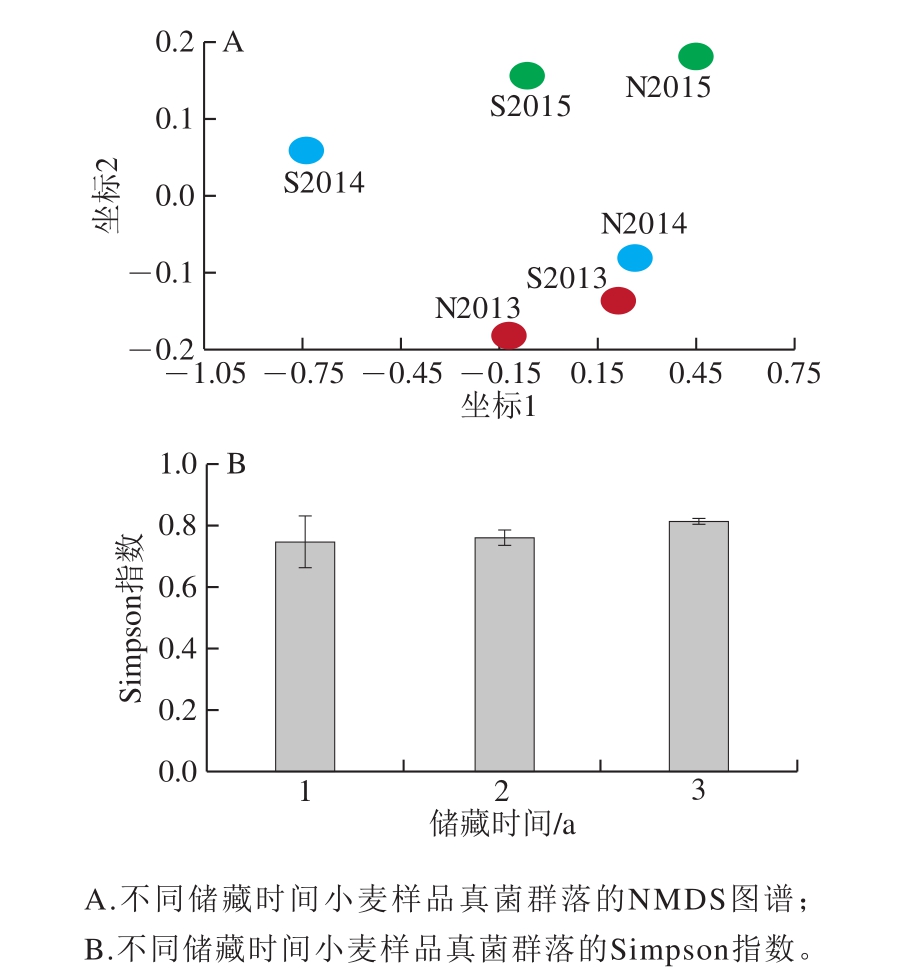
图2 小麦真菌群落与储藏时间之间的关系
Fig. 2 Relationship between fungal communities in wheat and its storage period
储藏时间不同可能导致小麦真菌群落整体结构有所差异,而真菌群落整体结构差异通常是由关键真菌菌种的差异所导致。LEfSe分析(图3)可以得到不同储藏时间真菌群落间的差异菌群。由图3可以看出,储存1 a的小麦样品中,只有Geosmithia属的CCF3652菌株是特征菌群,即该菌株在储存1 a的小麦真菌群落中的比例显著高于该菌株在储存2、3 a的小麦真菌群落中。储存2 a的小麦样品特征真菌包括出芽短梗霉(Aureobasidium pullulans)、S. mahoniae、Candida palmioleophila和Chaetomidium leptoderma,其中S. mahoniae是小麦真菌群落分子生态网络中的核心菌群。储存3 a的小麦样品特征菌群包括亮白曲霉(A. candidus)、C. railenensis、粉红单端孢霉(Trichothecium roseum)、Pleurotus citrinopileatus、少根根霉原变种(Rhizopus arrhizus var. arrhizus)。

图3 利用LEfSe分析不同储藏时间小麦真菌群落中的特征真菌
Fig. 3 LEfSe analysis of characteristic fungi in fungal communities in wheat stored for different years
2.3 小麦在粮库内储藏位置对其中真菌群落结构的影响
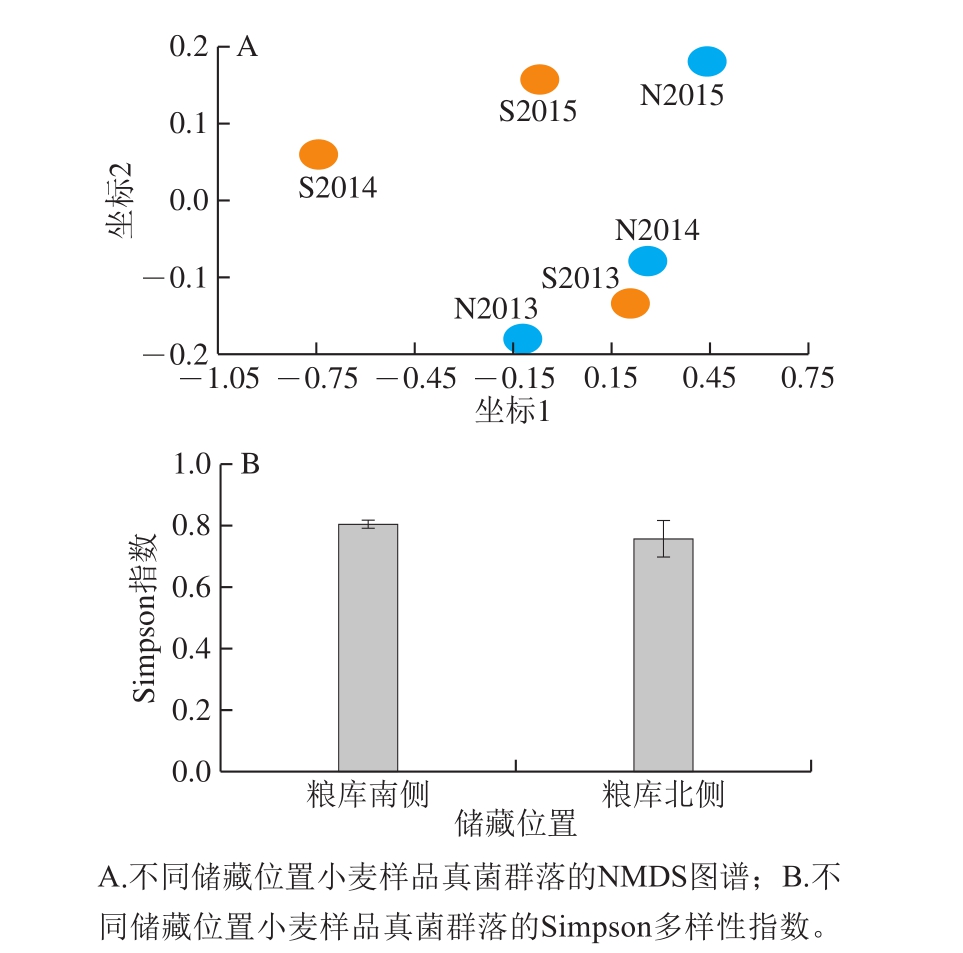
图4 小麦真菌群落与粮库中储存位置的关系。
Fig. 4 Relationship between fungal communities in wheat and its location in warehouse
采集3 个不同仓库南侧和北侧的粮食样品,分析其真菌群落结构,研究粮库内储藏位置对小麦真菌群落结构的影响,结果如图4所示。从粮库南侧和粮库北侧采集的样品真菌群落并未发生分别聚类,而是呈现间隔分布的特点,由此可见储存位置对小麦真菌群落的影响可能没有储藏时间明显。这一现象可能是由于同一粮仓中整体环境比较均一,且粮库中不同位置小麦的真菌孢子可以通过空气进行交流。在多样性方面,从粮库南侧采集的小麦样品中真菌群落的多样性在平均值方面略高于从粮库北侧采集的小麦样品,但差异并不显著(P>0.05)。由于对粮库中真菌群落的空间分布鲜见于前人的报道中,因此本结果需要更深入的研究和更多粮库样品的验证。
2.4 小麦储藏深度对其中真菌群落结构的影响
小麦储藏深度可能影响温度、湿度、氧气浓度等环境条件,从而导致其中真菌群落的组成有所差异。本研究采集了粮仓中表层(WH1)、中层(WH2)、深层(WH3)3 份小麦样品,分析其中真菌群落结构特点。由图5A可以看出,3 份样品中共有真菌种类为87 种,仍然占到所有真菌种类的45.1%,而在各层特有真菌方面,表层小麦样品特有33 种真菌,中层小麦样品特有20 种真菌,深层小麦样品特有8 种真菌。在表层小麦特有的33 种真菌种,粪壳菌纲(Sordariomycetes)的真菌有17 种,占51.5%。进一步分析粪壳菌纲中所有真菌在不同层次小麦样品中分布情况,发现大部分粪壳菌纲真菌由“表层-中层-深层”的分布呈现出减少的趋势(图5B),即粪壳菌纲真菌主要分布在表层或中层的储藏小麦中,而在深层小麦中分布较少。通过粪壳菌纲的例子可以看出,小麦储藏深度对其中的真菌群落有一定程度的影响。
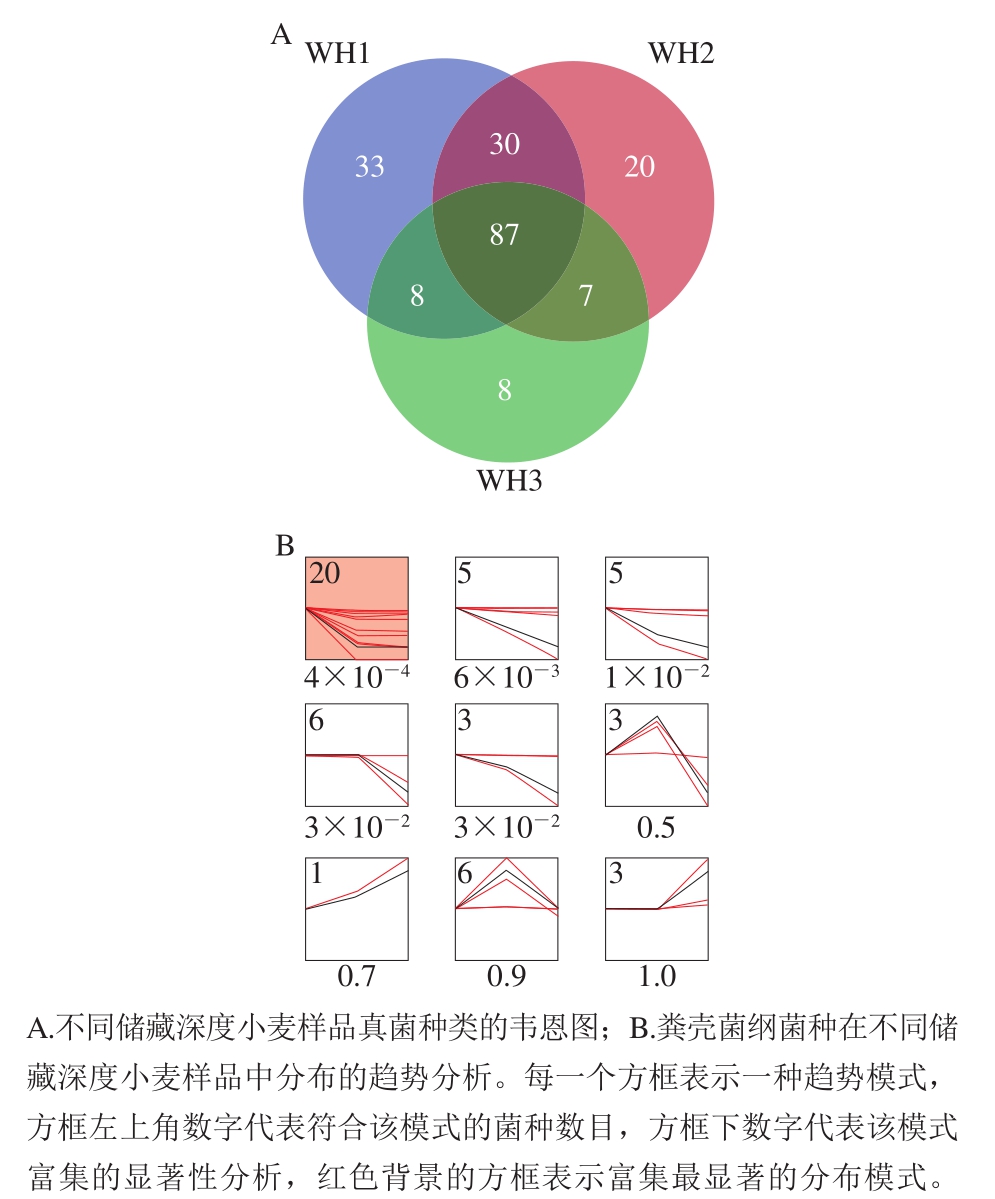
图5 小麦真菌群落与储藏深度之间的关系
Fig. 5 Relationship between fungal communities in wheat and its storage depth in warehouse
2.5 河南和黑龙江两地储藏小麦中真菌群落的比较

图6 河南和黑龙江两地小麦样品中真菌群落的比较。
Fig. 6 Comparison of fungal communities in wheat samples collected from different provinces
整体比较河南省和黑龙江省粮库所采集的小麦样品中的真菌群落,可以发现二者具有很大差异。LefSe分析(图6A)表明,两省样品在种水平上的23 种真菌具有显著性差异,其中既涉及到Pleosporaceae RS_5、帚状曲霉(A. penicillioides)等高丰度菌种,也涉及到S. mahoniae,极细链格孢(A. tenuissima)和索伊纳尾孢菌(C. sojina)等在小麦真菌群落分子生态网络中处于核心位置菌种,由此可见,河南和黑龙江两地储藏小麦种真菌群落结构具有实质性的区别。从图6B可以看出,主要菌群在各个样品中的丰度分布情况。取自同一省份的样品中真菌群落分布较为一致,而河南和黑龙江两地的样品中这些菌种分布差异明显。由于河南省样品属于不同储藏时间,黑龙江省样品采自不同储藏深度,可以推测河南和黑龙江两地对小麦真菌群落的影响可能大于储藏时间和储藏深度对真菌群落的影响。
3 讨 论
在本研究之前,国内外己有利用培养法或传统分子生物学方法研究小麦中真菌群落特征的报道。与前人的报道相比,本研究利用高通量测序技术得到的结果在全面性和灵敏性方面有明显提高。例如,李慧等[25]应用培养法从小麦粉中分离培养出5 株真菌,为链格孢霉、橘灰青霉、黑曲霉各1 株,米曲霉2 株。Tralamazza等[26]利用培养法研究巴西小麦,发现小麦中真菌以链格孢霉、镰刀菌、附球孢菌为主。都立辉等[27]利用PCR-DGGE技术研究储粮小麦中真菌群落,共发现了8 种真菌,主要为帚状曲霉、格孢腔菌、枝孢菌和假丝酵母。本研究利用高通量测序技术,共发现了93 种真菌,其中高丰度真菌种类与培养法和传统的分子生物学方法得到的结果基本一致,但高通量测序方法对低丰度真菌的研究能力是培养法和传统分子生物学方法所不能胜任的。高通量测序方法也存在不足,如培养法分离得到菌株后,可以方便的研究菌株的产毒素能力,而高通量测序方法只能研究真菌物种组成,在真菌毒素方面只能依据物种组成进行推测。
本研究结果表明,链格孢属、曲霉属和枝孢霉属是测序结果中丰度最高的己知菌属。链格孢属和曲霉属都是小麦作物常见的病原菌,而枝孢霉属广泛分布在土壤和空气中,因此这些微生物的孢子附着在仓储小麦上的概率很高。若储藏条件不当,这些真菌的孢子可能萌发,产生真菌毒素,导致仓储小麦的食品安全风险。例如链格孢属微生物可能产生交链孢酚、交链孢酚单甲醚、交链孢烯、交链孢菌酮酸和交链孢毒素等[28],曲霉属微生物可能产生黄曲霉毒素、赭曲霉毒素等[25,29]。利用高通量测序技术分析仓储小麦中真菌种类,有利于改进储藏条件,有针对性的防止相应的真菌毒素产生。
在之前的研究中,本课题组曾提出“依据预测微生物学,构建储粮中微生物生长预测模型,可以快速对储粮中微生物的生长情况进行判断,对储粮中病原微生物和腐败微生物的控制有重要作用”[30]。任何预测模型的建立都必须以实测数据为基础,在实测数据中找到规律,建立模型,之后进行验证。本研究结果对小麦真菌生长预测模型的指导意义包括:首先,建立模型时,应考虑粮库所在省份的差异,河南和黑龙江两地由于地理位置和环境条件的差异,可能需要完全不同的预测模型;其次,储藏时间和储藏深度时可能是影响小麦真菌生长和演替的主要因素,其原因可能是这2 个因素都影响小麦的呼吸状态、营养状态及氧气、温度、湿度等环境条件;再次,由于粮仓中整体环境比较均一,因此储藏位置可能对小麦真菌生长模型的影响较小。本实验可为储粮中微生物生长预测模型建立提供参考,但具体规律仍需更多的样品进行验证。
[1] AHMAD A, YU JAE-HYUK. Occurrence, toxicity, and analysis of major mycotoxins in food[J]. International Journal of Environmental Research and Public Health, 2017, 14(6): 632. DOI:10.3390/ijerph14060632.
[2] FREIRE L, ANDERSON S. Modified mycotoxins: an updated review on their formation, detection, occurrence, and toxic effects[J].Food and Chemical Toxicology, 2018, 111: 189-205. DOI:10.1016/j.fct.2017.11.021.
[3] 许嘉, 林楠, 王志, 等. 北京市市售谷物及制品中真菌毒素污染状况的调查[J]. 中国食物与营养, 2019, 25(3): 28-30.
[4] 陈畅, 丁伟, 唐芳, 等. 小麦储藏水分、温度和真菌生长危害进程预测[J]. 中国粮油学报, 2012, 27(5): 5-9; 26. DOI:10.3969/j.issn.1003-0174.2012.05.002.
[5] 程树峰, 唐芳, 伍松陵. 储粮真菌危害早期检测技术研究(2): 小麦储藏真菌生长规律的研究[J]. 粮油仓储科技通讯, 2008, 24(5): 51-54.
[6] 程树峰, 唐芳, 伍松陵. 小麦储藏危害真菌生长规律的研究[J]. 中国粮油学报, 2009, 24(4): 118-121.
[7] 岳晓禹, 张华, 陈威风, 等. 应用DGGE技术研究玉米储藏过程中的霉菌群落时空分布特征[J]. 食品科学, 2018, 39(4): 307-311.DOI:10.7506/spkx1002-6630-201804046.
[8] 许文涛, 郭星, 罗云波, 等. 微生物菌群多样性分析方法的研究进展[J]. 食品科学, 2009, 30(7): 258-265. DOI:10.3321/j.issn:1002-6630.2009.07.060.
[9] 李东萍, 郭明璋, 许文涛. 16S rRNA测序技术在肠道微生物中的应用研究进展[J]. 生物技术通报, 2015, 31(2): 71-77. DOI:10.13560/j.cnki.biotech.bull.1985.2015.02.010.
[10] AYDIN S, SHAHI A, OZBAYRAM E G, et al. Use of PCR-DGGE based molecular methods to assessment of microbial diversity during anaerobic treatment of antibiotic combinations[J]. Bioresource Technology, 2015, 192: 735-740. DOI:10.1016/j.biortech.2015.05.086.
[11] KERMANI F, SHAMS-GHAHFAROKHI M, GHOLAMI-SHABANI M,et al. Diversity, molecular phylogeny and fingerprint profiles of airborne Aspergillus species using random amplified polymorphic DNA[J]. World Journal of Microbiology & Biotechnology, 2016,32(6): 96. DOI:10.1007/s11274-016-2052-1.
[12] CAMARINHASILVA A, WOSOXLEY M L, JÁUREGUI R, et al.Validating T-RFLP as a sensitive and high-throughput approach to assess bacterial diversity patterns in human anterior nares[J]. Fems Microbiology Ecology, 2015, 79(1): 98-108. DOI:10.1111/j.1574-6941.2011.01197.x.
[13] 白飞荣, 姚粟, 田海霞, 等. 基于高通量测序和可培养方法的勐海发酵普洱茶真菌多样性分析[J]. 食品与发酵工业, 2018, 44(12): 43-51.DOI:10.13995/j.cnki.11-1802/ts.017637.
[14] 胥伟, 姜依何, 吴丹, 等. 高通量测序研究霉变黑毛茶的真菌多样性[J].茶叶科学, 2017, 37(5): 483-492.
[15] 左勇, 王小龙, 张晶, 等. 基于高通量测序技术的宜宾芽菜真菌多样性研究[J]. 中国调味品, 2016, 41(4): 19-22. DOI:10.3969/j.issn.1000-9973.2016.04.004.
[16] 赵红宇, 徐炜桢, 杨国华, 等. 基于高通量测序的郫县豆瓣后发酵期真菌演替变化分析[J]. 食品科学, 2017, 38(16): 51-56. DOI:10.7506/spkx1002-6630-201716008.
[17] MELO S C, PUNGARTNIK C, CASCARDO J C, et al. Rapid and efficient protocol for DNA extraction and molecular identification of the basidiomycete Crinipellis perniciosa[J]. Genetics and Molecular Research, 2006, 5(4): 851-855. DOI:10.1590/S1415-47572006000200033.
[18] TAPIA-TUSSELL R, LAPPE P, ULLOA M, et al. A rapid and simple method for DNA extraction from yeasts and fungi isolated from Agave fourcroydes[J]. Molecular Biotechnology, 2006, 33(1): 67-70.DOI:10.1385/MB:33:1:67.
[19] 吴发红, 黄东益, 黄小龙, 等. 几种真菌DNA提取方法的比较[J].中国农学通报, 2009, 25(8): 62-64.
[20] CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303.
[21] HAMMER Ø, HARPER D A T, RYAN P D. PAST: paleontological statistics software package for education and data analysis[J].Palaeontologia Electronica, 2001, 4(1): 1-9.
[22] DENG Y, JIANG Y H, YANG Y, et al. Molecular ecological network analyses[J]. BMC Bioinformatics, 2012, 13(1): 113.DOI:10.1186/1471-2105-13-113.
[23] SEGATA N, IZARD J, WALDRON L, et al. Metagenomic biomarker discovery and explanation[J]. Genome Biology, 2011, 12(6): R60.
[24] ERNST J, BAR-JOSEPH Z. STEM: a tool for the analysis of short time series gene expression data[J]. BMC Bioinformatics, 2006, 7:191. DOI:10.1186/1471-2105-7-191.
[25] 李慧, 胡梦龙, 蔡军, 等. 小麦粉污染霉菌的分离鉴定及产黄曲霉毒素能力的研究[J]. 食品安全质量检测学报, 2015(9): 3447-3452.
[26] TRALAMAZZA S M, BEMVENUTI R H, ZORZETE P, et al. Fungal diversity and natural occurrence of deoxynivalenol and zearalenone in freshly harvested wheat grains from Brazil[J]. Food Chemistry, 2016,196: 445-450. DOI:10.1016/j.foodchem.2015.09.063.
[27] 都立辉, 和肖营, 张虹, 等. 应用PCR-DGGE技术研究储藏小麦中的真菌多样性[J]. 粮食储藏, 2014(5): 41-45. DOI:10.3969/j.issn.1000-6958.2014.05.009.
[28] 闫璐, 高贵田, 哈益明, 等. 谷物中链格孢毒素的研究进展[J]. 核农学报, 2017, 31(2): 334-341. DOI:10.11869/j.issn.100-8551.2017.02.0334.
[29] 彭春红, 王丽, 安凤平, 等. 制粉和分层研磨对小麦中赭曲霉毒素A的去除效果[J]. 中国粮油学报, 2016, 31(3): 7-11. DOI:10.3969/j.issn.1003-0174.2016.03.002.
[30] 岳晓禹, 张恒业, 辛婷, 等. 储粮预测微生物模型的研究进展[J]. 中国粮油学报, 2012, 27(5): 118-123. DOI:10.3969/j.issn.1003-0174.2012.05.024.