群体感应是细菌间交流的一种方式,通过分泌到胞外的信号分子进行[1]。革兰氏阴性菌间的群体感应是由高丝氨酸内酯类(N-acylhomoserine lactones,AHLs)介导,其中LuxR/I系统是革兰氏阴性菌中较为典型的模式群体感应调控系统。luxI基因编码AHLs合成酶,调控AHLs的合成,当环境中的菌群数量达到一定密度时,合成的AHLs会分泌到胞外,AHLs浓度达到一定的阈值后,会进入胞内,与LuxR蛋白结合,激活细菌的LuxR/I调控系统,进而对下游相关基因控制的性状进行调控[1-2]。研究表明,细菌的群体感应LuxR/I系统对胞外蛋白酶的分泌、生物被膜的形成、泳动性以及生物发光等性状具有调控作用[2]。
蜂房哈夫尼菌(Hafnia alvei)是一种革兰氏阴性、兼性厌氧菌,属于肠杆菌科的条件性致病菌,有鞭毛,能运动,适应生长的温度范围较广(4~42 ℃),因此广泛分布于自然环境中[3]。现阶段对H. alvei的研究,主要围绕其作为食品腐败菌进行。据报道,H. alvei经常分离自低温冷藏的牛肉、鸡肉、原料乳、真空包装、低温贮藏的肉制品以及生鲜海产品中[4-6],并在这些产品的腐败变质中扮演着重要角色。研究表明该菌具有较强的群体感应活性,能够分泌多种AHLs,对其致腐特性具有潜在的调控作用[7]。
生物被膜是细菌为抵抗外界环境的变化而形成,为其本身的一种自我保护机制。它是细菌分泌的胞外多糖,可以茹附到接触物体的表面,进而茹连并包裹自身细胞,并促进细菌的传播和污染[8]。研究表明,H. alvei具有形成强生物被膜的能力[7-8],能够在与食品接触的器械表面形成,进而在加工过程中进入到食品中,导致食品在贮存过程中发生腐败变质。通过研究H. alvei群体感应系统对生物被膜形成的调控作用,以更好地控制H. alvei在食品加工过程中的污染和传播,减少由其造成的食品腐败。
1 材料与方法
1.1 材料与试剂
H. alvei H4野生型菌株分离自腐败即食海参;luxRI基因缺失型H. alvei株∆luxRI,由本实验室构建,详见Zhu Yaolei等[9]方法;紫色杆菌CV026菌株(Chromobacterium violaceum 026)由中国农业科学院周志刚教授惠赠,用于检测AHLs;实验菌株均用LB培养基在30 ℃条件下培养。其中培养∆luxRI菌株时需添加20 μg/mL的氯霉素。
氯霉素 北京索莱宝科技有限公司;无水甲醇、冰醋酸(均为分析纯) 天津市科密欧化学试剂有限公司;结晶紫 生工生物工程(上海)股份有限公司;RNA提取试剂盒(DP430) 天根生化科技有限公司;SYBR Green I Ex Taq酶(RR420)、反转录试剂盒(RR047) 宝日医生物技术有限公司。
1.2 仪器与设备
UV 2102分光光度计 日本岛津公司;M2酶标仪美国分子仪器有限公司;7500荧光定量聚合酶链式反应(polymerase chain reaction,PCR)仪 美国应用生物系统公司;PCR仪 伯乐生命医学产品有限公司;凝胶成像仪 美国UVP公司;场发射扫面电镜 美国FEI公司。
1.3 方法
1.3.1 生长曲线测定
取H. alvei H4和∆luxRI菌株过夜培养的菌液1 mL,分别接种到灭菌的液体100 mL LB培养基中,于160 r/min、30 ℃的恒温摇床中培养,每隔2 h测定其OD600 nm。
1.3.2 AHLs的测定
采用报告平板法测定H. alvei H4和∆luxRI菌株AHLs[10]。
AHLs的提取:取100 mL培养至稳定期的H. alvei H4和∆luxRI菌株的菌液,4 ℃、8 000 r/min离心15 min,取上清液,用旋转蒸发仪将其浓缩至10 mL,加入30 mL 1%甲酸溶液酸化的乙酸乙酯,充分振荡混均匀后静置3 h。取上层溶液蒸干,加入1 mL超纯水充分溶解,取出后置于-20 ℃备用。
AHLs检测:取10 mL过夜培养的紫色杆菌CV026菌液,加入到冷却至60 ℃左右的固体LB培养基中,混匀,倾注到平板中,待其凝固,用1 mL枪头顶端在平板中央打孔,随后加入60 μL AHLs提取液,置于30 ℃恒温培养箱中培养,根据孔周围有无紫色变化,判断AHLs是否存在。
1.3.3 生物被膜的测定
1.3.3.1 结晶紫染色方法测定生物膜形成
将培养至稳定期的H. alvei H4和∆luxRI菌株的菌液用LB溶液按1∶100比例稀释,分别吸取200 μL稀释后的菌液加入到96 孔板中,30 ℃静置培养24 h。培养结束后,测定其OD600 nm,之后弃去孔中菌液,每孔加入250 μL 0.1 mol/L的磷酸盐缓冲液(phosphate buffered saline,PBS)清洗3 遍。随后加入250 μL无水甲醇固定15 min,弃去孔中废液,置于60 ℃烘箱中烘干。取出冷却后,加入250 μL 0.1%结晶紫溶液染色15 min,然后弃去废液,用去离子水清洗3 遍,洗去残余的结晶紫溶液,60 ℃烘干,加入200 μL 33%冰醋酸溶液,在96 孔板振荡器上振荡15 min。最后读取其590 nm波长处吸光度[8]。以OD590 nm/OD600 nm表示生物膜的形成。
1.3.3.2 生物被膜扫描电镜观察
将玻璃盖片(1 cm×1 cm)用洗涤剂清洗干净后,置于HCl溶液中过夜浸泡,去离子水清洗干净,放入平板中灭菌,备用。将处理好的玻璃盖片置于24 孔板中,加入1.3.3.1节稀释好的菌液,培养48 h,从24 孔板中取出玻片,用灭菌的0.1 mol/L PBS充分清洗,洗掉浮游细菌,再用2.5%戊二醛-PBS过夜固定,之后用PBS冲洗数遍,随后用1%锇酸溶液固定1 h,依次用50%、70%、80%、90%乙醇溶液脱水10 min,无水乙醇脱水2 次,每次15 min,再经乙酸异戊酯置换2 次,每次15 min,临界点干燥仪干燥,取出后喷金,扫描电镜进行观察[11-12]。
1.3.4 泳动性的测定
将灭菌好的泳动性培养基倾注到平板中,每个平板20 mL。平板凝固后,用移液器吸取3 μL培养至稳定期的H. alvei H4和∆luxRI菌株的菌液,点到泳动性平板中央,小心移至30 ℃恒温培养箱进行培养[13]。
1.3.5 生物膜相关基因表达的实时聚合酶链式反应(real-time polymerase chain reaction,real-time PCR)测定
1.3.5.1 RNA的提取
取1 mL培养至稳定期的H. alvei H4和∆luxRI菌株的菌液,离心并弃去上清液,用灭菌的0.1 mol/L PBS清洗菌体沉淀2 次,离心弃去上清液。根据天根RNA提取试剂盒说明书进行操作,提取RNA。
1.3.5.2 cDNA的合成
分别取500 ng上述提取的RNA,利用宝生物的反转录试剂盒将其合成cDNA,稀释10 倍作为real-time PCR的模板。
1.3.5.3 real-time PCR条件及体系
采用real-time PCR测定群体感应LuxR/I系统对H. alvei生物膜和泳动性相关基因flgA、flgE、fliA、flhD、flhC表达量的变化。反应体系20 μL:1.6 μL cDNA,0.4 μL上下游引物,7.6 μL DEPC水,10 μL SYBR Green I Ex Taq酶。反应条件:95 ℃预变性5 min;95 ℃变性30 s,59 ℃退火31 s,40 个循环。16S rRNA作为内参基因,采用2-∆∆Ct的方法对结果进行分析[14]。real-time PCR引物信息见表1。
表1 real-time PCR引物序列
Table 1Primers used for real-time PCR

基因名称 编码产物 引物序列(5’-3’)16S rRNA 16S rRNA CTGGACAAAGACTGACGC CGTTTACAGCGTGGACTACC f GGGTTATCACCAATCCTG GGTAATGGGTTGTAAATCG f l gA 鞭毛基体P-环形成蛋白l gE 鞭毛钩蛋白AATCACGACTGCGAAGCT GGAGGTATTGGGCTGTTGC f CACGGTAATTTCGCAGAT GACATAATCCAGCCCAAG fliA 鞭毛合成调控蛋白l hD 鞭毛转录调节因子GCAATGAAATGGCAGATTC AGCAAATGGCTGGAAAGC flhC 鞭毛转录调节因子TTGTGGATAGCGGGATG ACGTGCTGAGCGGTTGAT
2 结果与分析
2.1 H. alvei H4和∆luxRI菌株的生长曲线

图1 H. alvei H4和ΔluxRI菌株的生长曲线
Fig. 1 Growth curves of H. alvei H4 and ΔluxRI
由图1可知,H. alvei H4的群体感应相关基因luxRI敲除后,该菌的生长状况在对数期略低于野生型菌株,在对数后期,2 株菌的生长密度达到一致,进入到稳定期后,二者的生长状况保持相同,无明显差别。
2.2 H. alvei H4和∆luxRI菌株AHLs产生对比
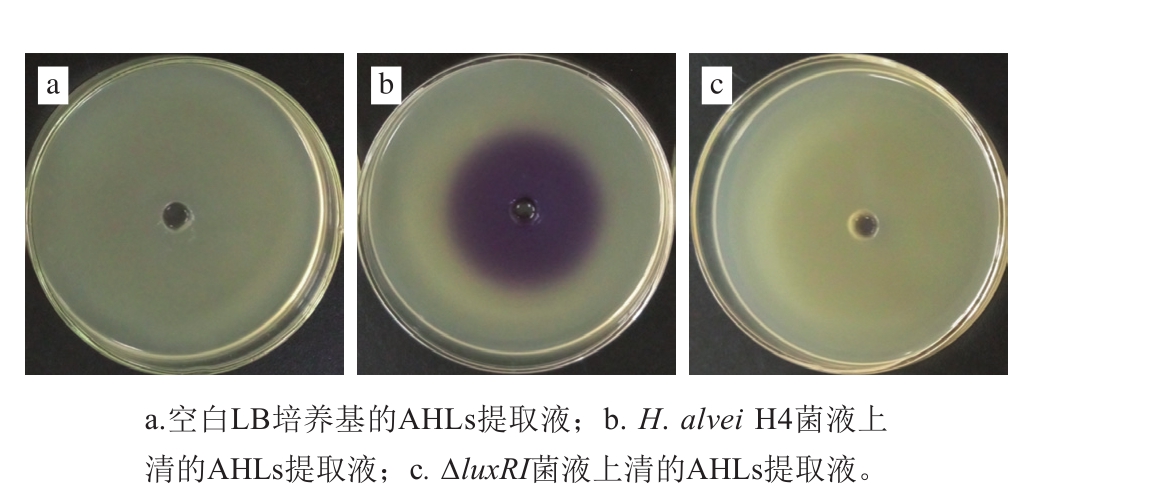
图2 H. alvei H4和ΔluxRI菌株AHLs的产生
Fig. 2 Production of AHLs by H. alvei H4 and ΔluxRI
如图2所示,在添加有H. alvei H4 AHLs提取液的平板中央出现了一个圆形的紫色区域,表明生长在琼脂中的紫色杆菌CV026受到AHLs的诱导作用产生了紫色素,进而说明H. alvei H4能够产生AHLs。而在加有∆luxRI菌株提取液的平板则与对照相同,其中央则没有发现紫色区域,表明∆luxRI菌株不能产生AHLs。
2.3 生物被膜的形成

图3 H. alvei H4和ΔluxRI菌株生物膜的形成
Fig. 3 Biofilm formation of H. alvei H4 and ΔluxRI

图4 H. alvei H4和ΔluxRI菌株生物膜的微观结构
Fig. 4 Biofilm formation of H. alvei H4 and ΔluxRI revealed by SEM
如图3所示,luxRI基因缺失后,∆luxRI菌株形成生物膜的能力显著低于H. alvei H4(P<0.05)。图4A1显示,茹附在玻片表面的细菌数量较多,而图4B1中茹附在玻片上∆luxRI菌株数量明显减少。并且对比图4A2、B2可知,H. alvei H4能够产生大量胞外多糖,茹附在玻片表面形成较厚的网状结构的生物膜,而∆luxRI菌株分泌的胞外多糖相比于H. alvei H4则明显减少,形成的网状结构并不明显。
2.4 泳动性的变化
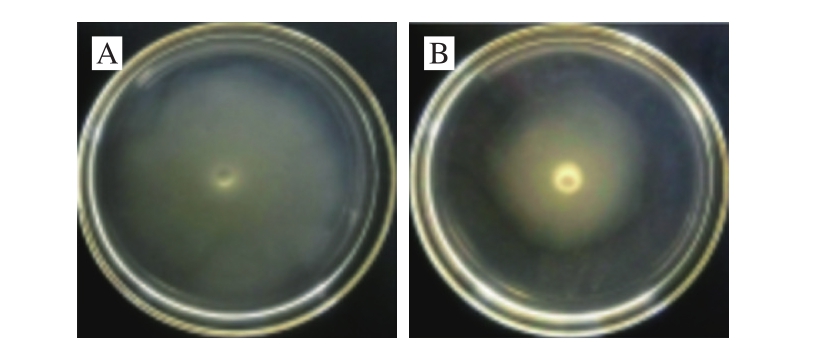
图5 H. alvei H4(A)和ΔluxRI(B)菌株的泳动性
Fig. 5 Swimming motilities of H. alvei H4 (A) and ΔluxRI (B)
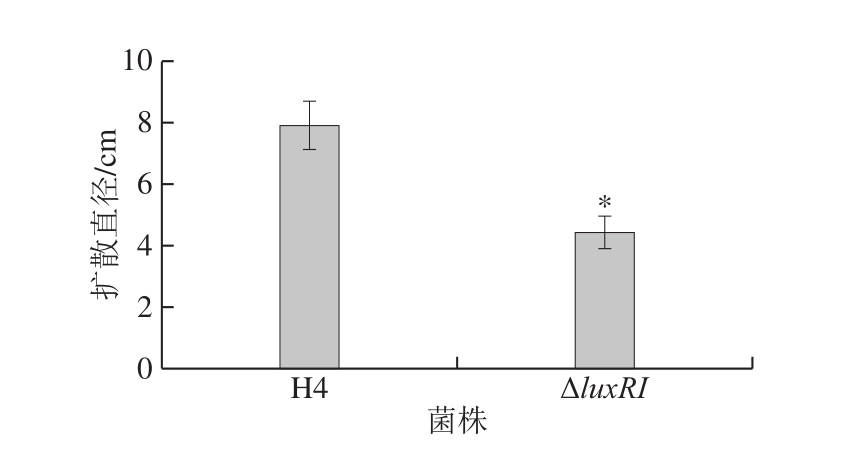
图6 H. alvei H4和ΔluxRI菌株在泳动性平板上的扩散直径
Fig. 6 Diffusion diameters of H. alvei H4 and ΔluxRI strains spread on swimming plates
图5 显示,H. alvei H4在泳动性平板表面培养48 h后,能够向四周扩散,形成圆形的晕圈,表明该菌具有运动性。∆luxRI菌株同样可以在泳动性平板表面扩散,然而其扩散形成圆形晕圈明显小于H. alvei H4。图6表明,在培养相同时间的条件下,∆luxRI菌株的扩散半径明显(P<0.05)小于野生型H. alvei H4,即其泳动性受到显著抑制。
2.5 生物膜和泳动性相关基因的表达
2.5.1 RNA的提取
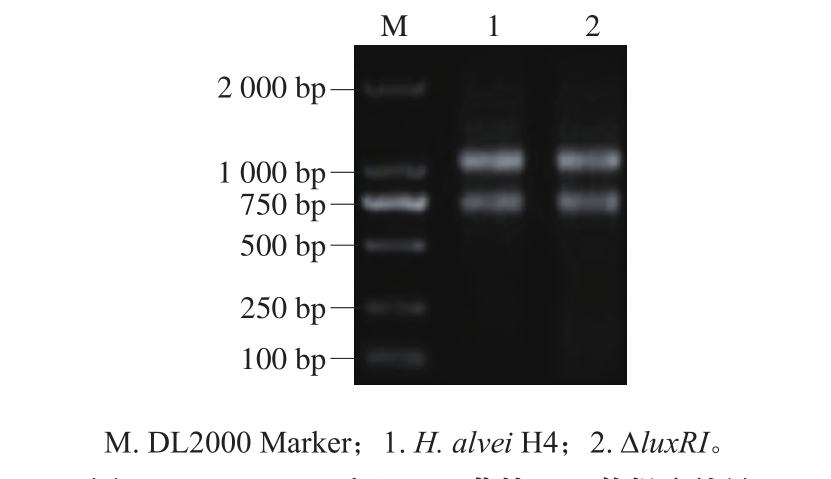
图7 H. alvei H4和ΔluxRI菌株RNA的提取结果
Fig. 7 Electrophoregram of RNA isolated from H. alvei H4 and ΔluxRI
RNA提取结束后,经电泳检测(图7),在泳道1和2均有两条明亮的条带,并且条带清晰、完整,没有发生降解现象,经酶标仪检测,OD260 nm/OD280 nm值在2.0左右,质量浓度均高于200 ng/μL。可用于接下来的cDNA合成。
2.5.2 基因表达的变化

图8 H. alvei H4和ΔluxRI菌株生物膜和泳动性相关基因的表达
Fig. 8 Relative expression levels of biofilm- and motility-related genes in H. alvei H4 and ΔluxRI
如图8所示,H. alvei群体感应相关基因luxRI缺失后,其生物膜和泳动性相关基因flgA、flgE、fliA、flhD和flhC相对表达量均显著下调。其中flgE基因的相对表达量下调了近65%,变化最大。其次是flhC基因,下调了近50%。fliA基因下调了约40%,flgA和flhD基因的相对表达量较f l gE、f l iA和f l hC基因变化较小,下调了近25%。
3 讨 论
研究表明,AHLs是由AHLs合成酶合成的,编码基因为luxI,当luxI缺失后,细菌本身无法产生AHLs,因此细菌间的交流会受到影响,进而导致受群体感应调控性状的变化[15]。当H. alvei H4的luxRI基因敲除后,该菌无法产生AHLs(图2),Liu Li等[16]报道中同样得出相似的结论。生物被膜的形成主要包括5 个阶段,分别为初始茹附、表面附着、形成初期、成熟期和脱落期,其中细菌的泳动性直接影响生物膜形成的第一个阶段,与介质表面的接触[8,17],H. alvei H4在luxRI基因敲除后,其泳动性显著降低(图5、6),从而影响了细菌与介质表面的接触,进一步导致生物膜形成的减少。在Khajanchi等[18]研究中,同样发现Aeromonas hydrophila菌的群体感应AhyRI系统的缺失,会显著抑制该菌生物膜形成和泳动性;同时,Oh等[19]研究发现将Acinetobacter nosocomialis菌株群体感应相关anoR基因敲除后,亦会导致该菌株生物被膜形成的显著性降低。上述学者的研究与本研究结论相同。
细菌的运动性主要依赖于其本身的鞭毛的移动和布朗运动[20],根据Lee[21]、Wang Yao[22]、陈雪凤[23]等报道,fliA、flhD和flhC为鞭毛转录调控因子,这些基因的缺失均会导致细菌生物膜形成和泳动性受到抑制,flgA和flgE为细菌鞭毛合成蛋白的编码基因,直接反映细菌鞭毛的合成状况,影响生物膜形成和泳动性。本研究H. alvei H4群体感应LuxR/I系统的缺失,使得f l gA、f l gE、fliA、flhD和flhC基因的相对表达量被显著抑制(图8),表明上述基因的表达受该菌群体感应的调控,并进一步调控生物膜的形成和泳动性。Atkinson等[24]报道表明,Yersinia pseudotuberculosis菌株泳动性的变化是其群体感应YpsRI和YtbRI系统通过调控flhD/C和fliA基因实现的。在对Sinorhizobium meliloti菌株群体感应系统的研究中[25],发现当其群体感应相关基因sinI缺失后,泳动性基因flaA和flaB的表达受到抑制。在对Pseudomonas fluorescens KM120[26]和Agrobacterium tumefaciens[21]的研究中发现,当其群体感应系统受到抑制而影响生物膜形成时,flgA和flgE基因的表达同样受到抑制作用。同样的,在关于Acidovorax citrulli[27]和Enterococcus faecalis[28]菌株群体感应调控作用的研究中,均表明生物膜、泳动性及相关基因的表达亦受到群体感应的调控作用,研究结果与本实验一致。
在对由细菌导致的食品腐败的研究中,群体感应己逐渐成为一个不可忽略的因素[29],根据研究结果,H. alvei H4群体感应LuxR/I系统对该菌生物被膜的形成和泳动性具有显著的调控作用,因此可以通过作用于群体感应系统,进而控制生物膜的形成以减少腐败细菌在食品加工过程中的传播和污染。近年来,越来越多的研究集中于群体感应抑制剂[30],能够在不抑制细菌生长的条件下,通过影响细菌的群体感应系统,进而抑制细菌生物膜、泳动性等腐败相关特性,达到控制腐败的作用。本研究为控制H. alvei在食品加工过程中生物膜的形成,减少由其造成的食品污染和腐败提供了新的理论支持。
[1] WATERS C M, BASSLER B L. Quorum sensing: cell-tocell communication in bacteria[J]. Annual Review of Cell and Development Biology, 2005, 21: 319-346. DOI:10.1146/annurev.cellbio.21.012704.131001.
[2] BAI A J, RAI V R. Bacterial quorum sensing and food industry[J].Comprehensive Reviews in Food Science and Food Safety, 2011,10(3): 183-193. DOI:10.1111/j.1541-4337.2011.00150.x.
[3] PADILLA D, REMUZGO-MARTÍNEZ S, ACOSTA F, et al. Hafnia alvei and Hafnia paralvei. Taxonomy defined but still far from virulence and pathogenicity[J]. Veterinary Microbiology, 2013,163(1/2): 200-201. DOI:10.1016/j.vetmic.2012.11.041.
[4] BRUHN J B, CHRISTENSEN A B, FLODGAARD L R, et al.Presence of acylated homoserine lactones (AHLs) and AHL-producing bacteria in meat and potential role of AHL in spoilage of meat[J].Applied and Environment Microbiology, 2004, 70(7): 4293-4302.DOI:10.1128/AEM.70.7.4293-4302.2004.
[5] TAN J Y, YIN W F, CHAN K G. Quorum sensing activity of Hafnia alvei isolated from packed food[J]. Sensors, 2014, 14(4): 6788-6796.DOI:10.3390/s140406788.
[6] 李婷婷, 高娜娜, 于海凤, 等. 蜂房哈夫尼亚菌对茹质沙雷氏菌群体感应信号分子产生与生物被膜形成的调控[J]. 食品科学, 2019,40(6): 128-135. DOI:10.7506/spkx1002-6630-20180117-232.
[7] VIANA E S, CAMPOS M E, POCE A R, et al. Biofilm formation and acyl homoserine lactone production in Hafnia alvei isolated from raw milk[J]. Biological Research, 2009, 42: 427-436. DOI:10.4067/S0716-97602009000400004.
[8] SREY S, JAHID I K, HA S D. Biofilm formation in food industries:a food safety concern[J]. Food Control, 2013, 31(2): 572-585.DOI:10.1016/j.foodcont.2012.12.001.
[9] ZHU Y L, HOU H M, ZHANG G L, et al. AHLs regulate biofilm formation and swimming motility of Hafnia alvei H4[J]. Frontiers in Microbiology, 2019, 10: 1330. DOI:10.3389/fmicb.2019.01330.
[10] 孔西曼, 张公亮, 王佳莹, 等. 环境因素对即食海参H. alvei群体感应的影响[J]. 现代食品科技, 2017, 33(1): 87-92. DOI:10.13982/j.mfst.1673-9078.2017.1.014.
[11] 邓旗, 孙力军, 王雅玲, 等. 环境条件对腐败希瓦氏菌生物被膜形成能力的影响[J], 中国食品学报, 2013, 13(10): 43-50. DOI:10.3969/j.issn.1674-7712.2014.17.023.
[12] JAHID I K, MIZAN M F R, HA A J, et al. Effect of salinity and incubation time of planktonic cells on biofilm formation, motility,exoprotease production, and quorum sensing of Aeromonas hydrophila[J]. Food Microbiology, 2015, 49: 142-151. DOI:10.1016/j.fm.2015.01.016.
[13] ABTEU A C, BORGES A, MERGULHAO F, et al. Use of phenyl isothiocyanate for biofilm prevention and control[J]. International Biodeterioration & Biodegradation, 2014, 86: 34-41. DOI:10.1016/j.ibiod.2013.03.024.
[14] SCHMITTGEN T D, LIVAK K J. Analyzing real-time PCR data by the comparative CT method[J]. Nature Protocols, 2008, 3(6): 1101-1108.DOI:10.1038/nprot.2008.73.
[15] AMMOR M S, MICHAELIDIS C, NYCHAS G J E. Insights into the role of quorum sensing in food spoilage[J]. Journal of Food Protection,2008, 71(7): 1510-1525. DOI:10.1089/cmb.2008.0088.
[16] LIU L, YAN Y, FENG L, et al. Quorum sensing asaI mutants affect spoilage phenotypes, motility, and biofilm formation in a marine fish isolate of Aeromonas salmonicida[J]. Food Microbiology, 2018, 76:40-51. DOI:10.1016/j.fm.2018.04.009.
[17] HOU H M, ZHU Y L, WANG J Y, et al. Characteristics of N-acylhomoserine lactones produced by Hafnia alvei H4 isolated from spoiled instant sea cucumber[J]. Sensors, 2017, 17(4): 772.DOI:10.3390/s17040772.
[18] KHAJANCHI B K, SHA J, KOZLOVA E V, et al. N-Acylhomoserine lactones involved in quorum sensing control the type VI secretion system, biofilm formation, protease production, and in vivo virulence in a clinical isolate of Aeromonas hydrophila[J]. Microbiology, 2009,155(11): 3518. DOI:10.1099/mic.0.031575-0.
[19] OH M H, CHOI C H. Role of LuxIR homologue AnoIR in Acinetobacter nosocomialis and the effect of virstatin on the expression of anoR gene[J]. Journal of Microbiology and Biotechnology, 2015,25(8): 1390-1400. DOI:10.4014/jmb.1504.04069.
[20] LI G, TAM L K, TANG J X. Amplified effect of Brownian motion in bacterial near-surface swimming[J]. Proceedings of the National Academy of Sciences, 2008, 105(47): 18355-18359. DOI:10.1073/pnas.0807305105.
[21] LEE J H, KIM Y G, BAEK K H, et al. The multifaceted roles of the interspecies signalling molecule indole in Agrobacterium tumefaciens[J]. Environmental Microbiology, 2015, 17(4): 1234-1244.DOI:10.1111/1462-2920.12560.
[22] WANG Y, DING L S, HU Y B, et al. The flhDC gene affects motility and biofilm formation in Yersinia pseudotuberculosis[J]. Science in China Series C: Life Sciences, 2007, 50(6): 814-821. DOI:10.1007/s11427-007-0101-6.
[23] 陈雪凤, 玉成鹏, 刘琼光. 水稻基腐病菌flhDC和fliA基因的功能[J].中国农业科学, 2016, 49(24): 4726-4734. DOI:10.3864/j.issn.0578-1752.2016.24.005.
[24] ATKINSON S, CHANG C Y, PATRICK H L, et al. Functional interplay between the Yersinia pseudotuberculosis YpsRI and YtbRI quorum sensing systems modulates swimming motility by controlling expression of flhDC and fliA[J]. Molecular Microbiology, 2008, 69(1):137-151. DOI:10.1111/j.1365-2958.2008.06268.x.
[25] GURICH N, GONZALEZ J E. Role of quorum sensing in Sinorhizobium meliloti-alfalfa symbiosis[J]. Journal of Bacteriology,2009, 191(13): 4372-4382. DOI:10.1128/JB.00376-09.
[26] MYSAZA K, SCHMIDT M T, BIALAS W, et al. Role of gallic and p-coumaric acids in the AHL-dependent expression of flgA gene and in the process of biofilm formation in food-associated Pseudomonas fluorescens KM120[J]. Journal of the Science of Food and Agriculture,2016, 96(12): 4037-4047. DOI:10.1002/jsfa.7599.
[27] WANG T, GUAN W, HUANG Q, et al. Quorum-sensing contributes to virulence, twitching motility, seed attachment and biofilm formation in the wild type strain Aac-5 of Acidovorax citrulli[J]. Microbial Pathogenesis,2016, 100: 133-140. DOI:10.1016/j.micpath.2016.08.039.
[28] HE Z, LIANG J, ZHOU W, et al. Effect of the quorum-sensing luxS gene on biofilm formation by Enterococcus faecalis[J]. European Journal of Oral Sciences, 2016, 124(3): 234-240. DOI:10.1111/eos.12269.
[29] ZHU S, WU H, ZENG M, et al. The involvement of bacterial quorum sensing in the spoilage of refrigerated Litopenaeus vannamei[J].International Journal of Food Microbiology, 2015, 192: 26-33.DOI:10.1016/j.ijfoodmicro.2014.09.029.
[30] BRACKMAN G, COENYE T. Quorum sensing inhibitors as antibiofilm agents[J]. Current Pharmaceutical Design, 2015, 21(1): 5-11.DOI:10.2174/1381612820666140905114627.