油橄榄苯丙氨酸解氨酶基因的克隆及其在毕赤酵母中的表达
陈文拴1,黄乾明1,*,陈华萍1,杨泽身2,王安逸2,苏光灿2
(1.四川农业大学理学院,四川 雅安 625014;2.凉山州中泽新技术开发有限责任公司,四川 西昌 615000)
摘 要:采用同源克隆、反转录聚合酶链式反应(reverse transcript ion polymerase chain reaction,RT-PCR)、融合引物嵌套PCR(fusion primer and nested integrated PCR,FPNI-PCR)与3’-cDNA末端快速扩增(rapid amplification of cDNA end,RACE)技术相结合,从 油橄榄(Olea europaea)中克隆得到苯丙氨酸解氨酶(phenylalanine ammonia lyase,PAL)基因全长,命名为OePAL。序列分析表明,OePAL的DNA全长2 970 bp(GenBank登录号KJ511867),含一个内含子(393~1 220 bp);全长cDNA有2 142 bp(GenBank登录号KJ511868),开放阅读框编码713 个氨基酸,与其他植物有较高的同源性。利用该基因构建重组质粒pPICZα A-OePAL, 且在毕赤酵母X33中进行诱导表达。十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate polyacrylamide gelelectrophoresis,SDS-PAGE)检测显示该重组酶的分子质量在77.5 kD左右,纯化后的酶比活力为196.3 U/mg。酶学性质研究表明:该重组酶的最适反应条件为40 ℃,pH 8.8;在供试范围内Cu2+与Na+可以提高该酶活性;以L-苯丙氨酸为底物,在最适条件下该酶的Km为4. 89×10-4mol/L。结果表明成功地克隆到OePAL,构建了表达载体,并进行了功能验证。
关键词:苯丙氨酸解氨酶;基因克隆;表达载体;毕赤酵母X33
油 橄榄(Olea europaea)属于木犀科、木犀榄属,是著名的木本油料树种[1-2],其果实主要用来压榨生产有“液体黄金”之称的橄榄油,橄榄油具有多种药用与保健功效[3-4],随着人们对健康的日益关注,近几十年来油橄榄产品呈现节节攀升的景象。橄榄油之所以能发挥多种药理作用,与其所含的黄酮类物质有很大关系。研究发现黄酮具有抗氧化、抗自由基、改善心血管系统与防癌等多种药理作用[5-7],因此富含黄酮的橄榄油具有极好的药理价值。此外,黄 酮类化合物对植物体本身也具有多种生物学功能,如参与影响植物花朵和果实颜色的形成,在植物的多种逆境胁迫过程中发挥着重要的抗逆作用[8-10]。因此黄酮类化合物一直都是植物次生代谢物研究中的热点。
黄酮类化合物 是苯丙氨酸代谢途径中的次生代谢物,该途径由苯丙氨酸解氨酶(phenylalan ine ammonia lyase,PAL)催化L-苯丙氨酸(L-phenylalanine,L-Phe)脱去氨基生成肉桂酸开始,后经一系列不同酶的催化进入不同的分支[11]。PAL是该途径中的第一个酶,且又是关键酶,其活性与黄酮类物质的合成有着密切的关系[12]。在逆境胁迫条件下的研究发现,植 物PAL活性的变化与其遭受的胁迫因素有紧密的联系,因此PAL可以作为植物抗逆性的一个生理指标[13]。目前已从多种植物中分离出了该酶的基因,研究发现植物PAL属于多基因家族,且可以分成不同的亚族,如在烟草的4 个PAL中,PAL1与PAL2属于同一亚族,而PAL3与PAL4同属另一亚族[14]。植物PAL编码区的长度大多在2 100 bp左右,如水稻PAL的开放阅读框(open reading frame,ORF)长2 151 bp[15],苜蓿PAL1的ORF长2 175 bp[16]。在大肠杆菌中进行的异源表达研究发现,不同来源PAL表达产物的比活力有很大差异,如纯化后的重组膜荚黄芪PAL比活力为6 500 U/mg[17],而重组苦荞PAL则为158.74 U/mg[18],这可能与不同植物PAL之间的差异有密切的关系。在合适的条件下,PAL能逆向催化肉桂酸加氨生成可以作为食品医药工业原料的L-Phe,目前虽已将红酵母(Rhodotorula glutinis)PAL应用于工业生产,但存在活性低、稳定性差等突出问题,限制了L-Phe工业的生产[19]。因此,对不同来源PAL的研究,将会为筛选更合适的工业级PAL酶源提供广阔的空间。鉴于PAL在植物黄酮代谢和逆境胁迫中所发挥的重要作用,以及在工业生产中的应用价值,本研究从油橄榄中克隆出PAL的全长,且实现该基因在毕赤酵母X33中的分泌表达,并进行了相关的酶学性质分析,以期为后续进行的以性状改良为目的的分子辅助育种提供基因资源,为未来该酶的改良以及在生产中的应用提供基础资料。
1 材料与方法
1.1 材料与试剂
油橄榄(Olea europaea)采自西昌凉山州中泽新技术开发有限责任公司北河油橄榄种植基地3 年生配多灵品种的幼嫩叶片。
植物RNA提取试剂盒 天恩泽基因科技有限公司;通用型DNA纯化回收试剂盒、质粒DNA小量提取试剂盒、大肠杆菌DH5α感受态细胞 Tiangen公司;Premix Taq,Prime STAR®Max DNA Polymerase,pMDTM19-T Vector Cloning Kit, Prime ScriptTMRT Reagent Kit,限制性内切酶KpnⅠ、NotⅠ、SacⅠ,Lysis Buffer与T4 DNA连接酶 日本TaKaRa公司;Ni-Agarose His标签蛋白纯化试剂盒 康为世纪公司;实验中所用引物、ZeocinTMSelection Reagent 美国Invitrogen公司;毕赤酵母X33、质粒pPICZα A为本实验室保存;其他化学试剂均为国产或进口分析纯。
LB、YPD、BMGY、BMMY培养基的配制均参照Invitrogen公司 毕赤酵母操作手册。
1.2 仪器与设备
A100/A200型基因扩增仪 郎基科学仪器有限公司;DYY-6B型稳压稳流电泳仪 北京市六一仪器厂;UV-3000型紫外分析仪 上海嘉鹏科技有限公司;Bio-Rad电转仪 美国Bio-Rad公司。
1.3 方 法
1.3.1 油橄榄总DNA与总RNA的提取及cDNA第一链的制备
采用改良的十六烷基三甲基溴化铵(hexadecyltrimethylammonium bromide,CTAB)法提取油橄榄叶片中的总DNA[20]。按照植物RNA提取试剂盒内说明书上的操作步骤提取油橄榄新鲜叶片中的总RNA。利用提取到的总RNA,参照Prime ScriptTMRT Reagent Kit说明书用引物CDSP(实验中使用的引物见表1)合成cDNA第一链[21],产物保存于-20 ℃备用。
表1 扩增PPAALL所用引物Table 1 Primers for cloning PAL used in this studyy

注:“□”表示限制性内切酶序列;“_”表示保护碱基。
引物名称 序列(5’→3’) 用途Tm/℃PU ATGARTAAAGGNACDGATAGTTA 扩增PAL片段53.5 PD GAACACCTTGTCACACTCCTC 53.9 FAD1 GTAATACGACTCACTATAGGGCACGCGTGGTNTCGASTWTSGWGTT扩增PAL 5’端FAD2 GTAATACGACTCACTATAGGGCACGCGTGGTNGTCGASWGANAWGAA FAD3 GTAATACGACTCACTATAGGGCACGCGTGGTWGTGNAGWANCANAGA FAD4 GTAATACGACTCACTATAGGGCACGCGTGGTAGWGNAGWANCAWAGG FSP1 GTAATACGACTCACTATAGGGC 58.2 FSP2 ACTATAGGGCACGCGTGGT 58.9 AP1 AGCTGGATCAAATATTAGATTATGA 55.1 AP2 TTGCGGAAGTTTCACTTTTCATA 59.8 AP3 ACGATTTTGACTCATTTTACCTTACA 59.6 CDSP AAGCAG TGGTATCAACGCAGAGTAC(T)30VN扩增PAL 3’端UPML CTAATACGACTCACTATAGGGCAAG CAGTGGT ATCAACGCAGAGT UPMS CTAATACGACTCACTATAGGGC 58.2 NUP AAGCAGTGGTATCAACGCAGAGT 60.7 ZP1 CAGTCGGTGTTAATGGAGAGCTTCAT 63.2 ZP2 TTGATGATCCCTGCAGCGCAACCTAC 64.8 PS GG GGTACC ATGAGTACTGCTGAGGTCG 扩增PAL全长PX ATAAGAAT GCGGCCGC GCAAATTGGAAGAGGT
1.3.2 油橄榄PAL的克隆
根据同源序列比对与NCBI中收录的油橄榄PAL的cDNA序列片段(GenBank登录号JX266201.1)分别设计扩增PAL的上游兼并引物PU与下游特异引物PD,以总DNA为模板扩增PAL的DNA片段,对扩增到的产物进行回收、连接、转化、挑选阳性克隆提质粒后送往上海英潍捷基公司测序。
根据测序结果设计用于步移PAL 5’端未知序列的3条巢式特异引物AP1、AP2与AP3。以总DNA为模板,用特异引物AP1与另设计的融合有特异引物接头的随机引物FAD 1、FAD2、FAD3和FAD4分别进行第一轮聚合酶链式反应(polymerase chain reaction,PCR),反应参数:94 ℃ 1 min;98 ℃ 1 min;94 ℃ 30 s,55 ℃ 1 m in,72 ℃ 2 min,5 个循环;94 ℃ 30 s;25 ℃ 3 min;72 ℃ 2 min;94℃ 30 s,55 ℃ 1 min,72 ℃ 2 min,2 个循环;94 ℃ 30 s,44 ℃ 1 min,72 ℃ 2 min,15 个循环;72 ℃ 5 min;4 ℃结束[22]。以第一轮PCR产物为模板,用特异引物AP2与FSP1进行第二轮PCR,反应参数:94 ℃ 1 min;94 ℃ 30 s,60 ℃ 15 s,72 ℃ 2 min,30 个循环;72 ℃ 5 min;4 ℃结束[22]。以第二轮PCR产物为模板,用特异引物AP3与FSP2进行第三轮PCR,反应程序同第二轮。对第三轮有明显条带的PCR产物回收连接克隆后进行测序。
根据油橄榄PAL的cDNA序列片段设计扩增其3’端未知序列的两条巢式特异引物ZP1与ZP2。以反转录cDNA第一链为模板,用特异引物ZP1与通用引物UPML 和UPMS(两者物质的量比为1∶5)一起进行第一轮PCR,反应参数:94℃ 2 min; 94℃ 30 s,72 ℃ 2 min,5 个循环;94 ℃ 30 s,66 ℃ 30 s,72 ℃ 2 min,5 个循环;94 ℃ 30 s,63 ℃ 30 s,72 ℃ 2 min,25 个循环;72 ℃ 5 min;4 ℃结束[21]。以第一轮PCR产物为模板,用特异引物ZP2与通用引物NUP进行第二轮PCR,反应参数:94 ℃ 1 min;94 ℃ 30 s,65 ℃ 15 s,72 ℃ 2 min,30 个循环;72 ℃ 5 min;4 ℃结束[21]。对第二轮有明显条带的PCR产物回收连接克隆后进行测序。
将以上扩增到的基因片段进行拼接,确定PAL的5’端与3’端非翻译区(untranslated regions,UTR)。在起始密码子与终止密码子位置分别设计用于扩增PAL全长且加有酶切位点的上下游引物PS(引入KpnⅠ酶切位点)与PX(引入NotⅠ酶切位点),分别以油橄榄总DNA和cDNA第一链为模板扩增PAL的全长DNA和cDNA。
1.3.3 油橄榄PAL的生物信息学分析
比对基因的DNA与cDNA序列,并结合GENSCAN在线确定其外显子、内含子以及ORF序列;用NCBI的BLASTn和BLASTp工具分别对基因完整的ORF与推导的氨基酸序列进行同源比对;采用SignalP 4.1进行信号肽预测;用NetNGlyc 1.0 Server在线进行糖基化位点分析;利用SOPMA在线工具进行多肽链的二级结构分析;通过SWISS-MODEL在线预测蛋白质的三维结构;用SMART在线工具进行结构域分析;利用MEGA 5.0构建系统进化树。
1.3.4 表达载体的构建
用限制性内切酶KpnⅠ与NotⅠ对油橄榄PAL的全长cDNA回收产物和pPICZα A质粒分别进行酶切处理,纯化回收后用T4连接液过夜反应,连接产物转化大肠杆菌DH5α感受态细胞,涂布含25 µg/mL Zeocin的LB平板培养基,用PCR鉴定后挑阳性菌落扩大培养,提质粒后进行酶切鉴定并测序,该正确的表达载体命名为pPICZα A-OePAL。
1.3.5 酵母菌的转化及筛选
将85 µL新鲜的毕赤酵母X33感受态与经SacⅠ线性化的10 µg左右pPICZα A-OePAL DNA混匀后转入0.2 cm的电击杯中预冷5 min,经2 kV、4 ms电击转化后,立即加入1 mL预冷的1 mol/L山梨醇重悬菌体,30 ℃温育1 h左右,全部涂布于含100 µg/mL Zeocin的YPD平板培养基,30 ℃倒置培养2~5 d直至单菌落出现,挑单菌落用Lysis Buffer裂解菌体后PCR鉴定。
1.3.6 重组酵母的诱导表达及表达产物的纯化、检测
挑选重组毕赤酵母X33/pPICZα A-OePAL接种于50 mL的BMGY培养基,30 ℃、220 r/min培养24 h左右;用BMMY培养基重悬菌体,使OD600 nm在1.0左右,30 ℃、220 r/min培养,期间每隔24 h添加至终体积分数为1.0%的甲醇进行诱导表达,同时进行粗酶液活力的测定,待酶活力达到最大时,离心菌液收集上清液,用组氨酸标签试剂盒纯化。另培养毕赤酵母X33/pPICZα A作为阴性对照。表达产物用7.5%分离胶,4%浓缩胶进行十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate polyacrylamide gelelectrophoresis,SDS-PAGE)检测。
1.3.7 重组6×His-OePAL酶活力的测定以及酶学性质分析
将终浓度为100 mmol/L的Tris-HCl(pH 8.5)与10 mmol/L的L-Phe反应混合液3 mL在30 ℃保温5 min,加入30 µL的纯化酶液混匀后测定OD290 nm(对照组以等体积水代替),接着在30 ℃保温1 h后测定OD290 nm,实验中以每小时反应液在290 nm波长处的光密度增加0.01为1 U[19]。采用考马斯亮蓝G-250染色法测定酶液中的蛋白质含量,计算重组6×His-OePAL的比活力。
温度对重组酶活力的影响:p H 8.5,分别在20~60 ℃范围内测定酶活力,并计算相对酶活力(以最高酶活力为100%);将纯化酶液分别在0~80 ℃范围内保温30 min后测定剩余酶活力,以未经保温的酶液作为对照(100%)。
pH值对重组酶活力的影响:在最适反应温度下,分别测定pH 5.0~10.0范围内的酶活力,并计算其相对酶活力(以最高酶活力为100%);将纯化酶液分别在pH 4.0~10.0的不同缓冲液中保温(最适温度)30 min后,于最适条件下测定剩余酶活力,以未经保温的酶液作为对照(100%)。
不同金属离子对重组酶活力的影响:在反应体系中加入终浓度为10 mmol/L的Mn2+、Cu2+、Ag+、Co2+、Ca2+、Na+、Fe3+、Fe2+、Hg2+、Zn2+于最适条件下测定酶活力,以未加金属离子的反应液作为对照(100%)。
重组酶的反应动力学常数Km测定:分别以0.1、0.2、0.4、0.8、1.0 mmol/L的L-Phe为底物在最适条件下测定酶活力,利用Lineweaver-Burk作图法(双倒数作图法)求得米氏常数Km。
2 结果与分析
2.1 油橄榄PAL的克隆结果

图1 油橄榄
PPAALL的PCR扩增图
CR
Fig.1 PCR amplification of PAL from Olea europaea
a. 1为以PU与PD作为引物的PCR产物;b. 1~4分别为以FAD1~FAD4作为随机引物进行的第3轮巢式PCR产物;c. 1为以ZP2与NUP作为引物的PCR产物;d. 1和2分别为以油橄榄总DNA和cDNA第一链为模板且以PS与PX为引物的PCR产物;M1为DNA MarkerⅢ;M2为DNA Marker DL2000。
用引物PU与PD的扩增产物测序有2 595 bp条带(图1a),序列分析表明克隆到了油橄榄PAL片段。用4 种随机引物对PAL 5’端进行染色体步移,最终只有引物FAD1克隆出一条850 bp的明亮条带(图1b),序列分析表明已克隆出包括起始密码子在内的油橄榄PAL 5’端未知序列。3’-cDNA末端快速扩增(rapid amplification of cDNA end,RACE)的测序结果表明,已克隆出包括Poly A在内的PAL 3’端未知序列(图1c)。用引物PS与PX扩增PAL全长DNA与cDNA分别为3 000 bp与2 200 bp左右(图1d)。
2.2 OePAL的生物信 息学分析
2.2.1 OePAL的序列分析

图 22 OOeePPAALL的核苷酸序列及推导的氨基酸序列
Fig.2 Nucleotides and deduced amino acid sequences of OePAL
核苷酸序列中大写字母表示外显子,小写字母表示内含子;下划线“----”表示活性中心特征序列,“ ”表示催化活性位点,“□”表示脱氨位点。
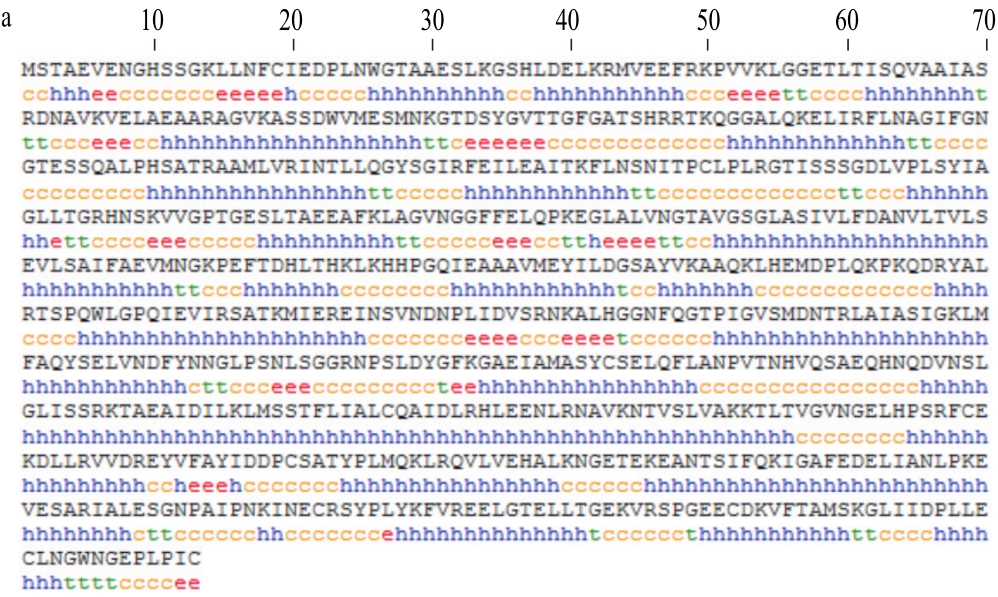
将油橄榄PAL序列在NCBI进行提交,命名为OePAL。OePAL全长DN A有2 970 bp(GenBank登录号KJ511867),包含一段符合典型GT—AG剪切方式的内含子(393~1 220 bp);全长cDNA有2 142 bp (GenBank登录号KJ511868),包含一个完整的ORF,编码一条713 个氨基酸的多肽链(图2)。经NCBI-BLAST发现,OePAL的ORF与GenBank中已收录的茶树(GenBank登录号D26596.1)、马铃薯(GenBank登录号KC631948.1)、烟草(GenBank登录号D17467.1)以及紫苏(GenBank登录号JQ277717.1)的相似度较高,均在80%~81%之间;OePAL的氨基酸序列与丹参(GenBank登录号ABD73282.1)、香蜂草(GenBank登录号CBJ23826.1)和紫苏(GenBank登录号AEZ67457.1)的同源性分别高达88%、89%、90%。通过Clustalw进行多序列比对分析发现,在OePAL的第200~211位氨基酸序列之间存在有典型的PAL活性中心特征序列SGDLVPLSYIAG,此外还有4 个作为脱氨位点的氨基酸残基(L203、V204、L253、A254)和11 个作为催化活性位点的氨基酸残基(N257、G258、N379、D380、N381、H393、H483、N484、Q485、D486、V487),表明成功克隆到了OePAL。
OePAL的氨基酸序列经ProtParam在线分析表明,其分子质量为77.514 4 kD,等电点为5.83,含有71 个带正电荷的氨基酸残基,83 个带负电荷的氨基酸残基,分子式为C3424H5490N944O1052S25,不稳定系数为36.56,属于稳定性蛋白。SignalP 4.1预测OePAL氨基酸序列中不含信号肽序列。糖基化位点分析显示(图3),当阈值为0.5时,该多肽链的第1 40、257、445、609位氨基酸可能被糖基化。

图 33 OOeePAL的糖基化位点分析
Fig.3 Glycosylation site analysis of OePAL protein
2.2.2 OePAL蛋白的结构预测
OePAL蛋白的空间结构分析显示(图4),其中α-螺旋占56.52%,β-转角占5.61%,无规则卷曲占30.86%。结构域分析显示OePAL蛋白中包含一段有496 个(第53~548位)氨基酸残基组成的具有芳香族氨基酸裂解酶活性的功能域,符合PAL家族蛋白特征。

图 44 OOeePAL蛋白的空间结构分析
Fig.4 Spatial structure analysis of OePAL protein
a. OePAL的二级结构预测;b. OePAL的三维结构建模;二级结构图中h为α-螺旋,e为延伸链,t为β-转角,c为无规则卷曲。
2.2.3 系统进化树的构建

图5 不同来源的PPAALL氨基酸序列聚类分析结果
Fig.5 Phylogenetic analysis of amino acid sequences of PAL from different plants
在软件M E G A 5 . 0平台上采用最大可能性法(maximum likelihood methods)构建基于16 种植物PAL氨基酸序列的系统发育树。结果显示(图5),裸子植物明显区别于双子叶植物和单子叶植物单独处于一个分支,属于同一科的植物多处于同一个分支,均符合植物分类学;紫苏、地黄、黄芩和油橄榄同处于一个大的分支,表明油橄榄与它们的亲缘关系要明显近于其他物种。
2.3 表达载体的构建及转化后筛选
重组质粒经酶切检测(图6)及测序,表明成功构建到了pPICZα A-OePAL表达载体。该线性化载体经转化涂板待长出单菌落后,用pPICZα A上的通用引物进行菌落PCR鉴定(图7),结果挑选出了与目标条带分子质量一致的单菌落,表明有OePAL已成功整合到毕赤酵母X33的基因组中。
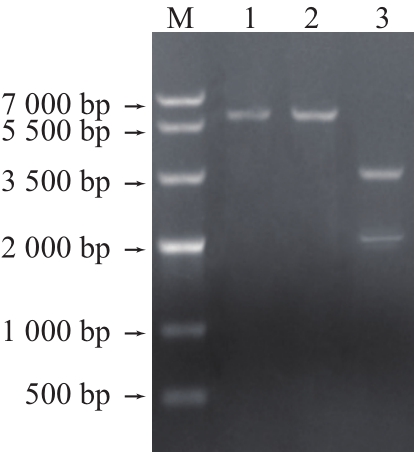
图6 重组质粒pPICZα AA--OOeePPAALL的酶切鉴定
Fig.6 Identification of pPICZα A-OePAL with restriction enzyme digestion
M. DNA Marker Ⅳ;1. KpnⅠ酶切重组质粒pPICZα A-OePAL的结果;2. NotⅠ酶切重组质粒pPICZα A-OePAL的结果;3. KpnⅠ与NotⅠ双酶切重组质粒pPICZα A-OePAL的结果。

图7 重组酵母的基因组PCR鉴定
Fig.7 Identification of PCR amplified products from recombinant yeast genomic DNA
M. DNA Marker Ⅲ;1. 未经转化的酵母单菌落;2. 用线性化的pPICZα A转化后的酵母单菌落;3. 用线性化的pPICZα A-OePAL转化后的酵母单菌落。
2.4 表达产物的SDS-PAGE检测及酶活性分析

图8 表达产物的SDS-PAGE结果
Fig.8 SDS-PAGE analysis of expressed products
M. 蛋白质标准分子质量;1. 重组毕赤酵母X33/pPICZα A诱导表达产物;2. 重组毕赤酵母X33/pPICZα A-OePAL诱导表达产物;3. 纯化后的重组毕赤酵母X33/pPICZα A-OePAL诱导表达产物。
研究温度对6×His-OePAL活力的影响发现(图9a),该酶的最适反应温度为40 ℃,与百合鳞茎和红酵母中PAL的最适温度一致[24-25],在高于50 ℃条件下保温30 min后,其活力下降迅速,与山药、无壳葫芦籽和鹰嘴豆中PAL的特点相似[26-28]。研究pH值对重组酶活力的影响表明(图9b),该酶的最适反应pH值为8.8,处于大多数植物PAL的最适pH 8.0~9. 5范围之内[29],在pH值为7.7~9.2范围内保温30 min后,其相对酶活力仍可保持在80%以上。考察金属离子对重组酶活力的影响发现(图9c),在供试范围内只有Cu2+和Na+对该酶具有激活作用,其
将诱导表达的粗酶液与纯化后的酶液进行SDS-PAGE电泳检测分析,结果见图8,经甲醇诱导的阳性转化子比空载对照组在77.5 kD左右处多出一条明显的条带,经特异纯化的蛋白产物仅在该位置处有一明显条带,表明目的蛋白已经分泌表达。纯化后的重组6×His-OePAL比活力为196.3 U/mg,比纯化前提高了6.4 倍,远高于提取的油橄榄皮瓜尔品种叶片PAL的比活力(约48.81 mU/mg)[23],分析原因可能与提取的油橄榄叶片PAL没有进行后续的纯化,粗提液中含有大量的杂蛋白有直接的关系,此外还可能与油橄榄品种和树龄的差异有一定关系。
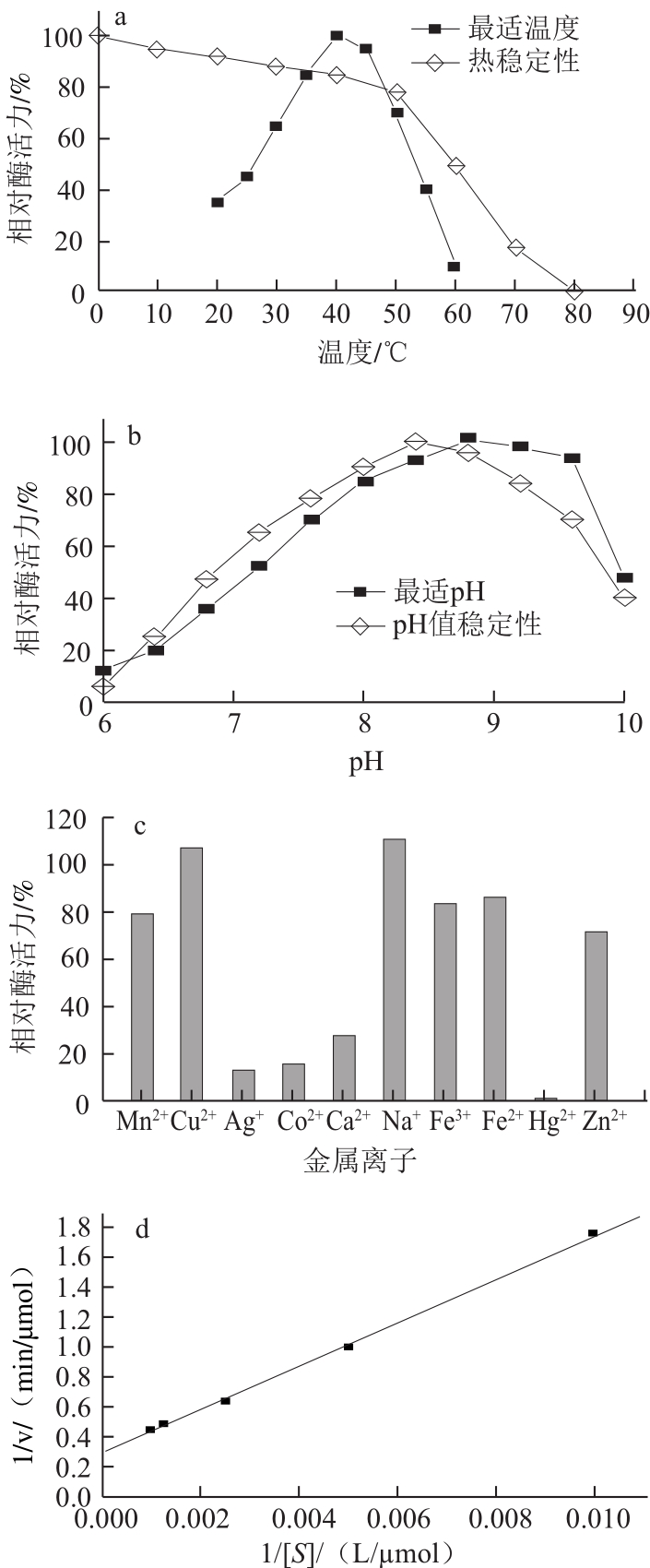
图9 重组酶的酶学性质
Fig.9 Enzymatic properties of recombinant enzyme
2.5 重组酶的酶学性质分析余8 种金属离子均表现抑制作用,其中Hg2+的抑制作用最强,这可能与Cu2+和Na+作为辅基与重组酶结 合后能保持酶蛋白的活性构象从而起到激活作用,而Hg2+与酶分子中的—SH基团结合从而抑制酶的活性有关。金属离子对酶活性的影响不仅与金属离子的浓度有关,而且还与酶的来源差异性有关,如对百合鳞茎PAL的研究发现,随着浓度的升高,Na+可以从抑制作用转变为促进作用,Mg2+可以从促进作用转变为抑制作用[24],在本实验中Cu2+对重组OePAL具有促进作用,而苦荞PAL的原核表达产物在与本实验相同浓度的Cu2+中其催化作用则受到了抑制[18],因此关于金属离子对重组OePAL的影响还有待更深入的研究。利用双倒数作图法求出米氏常数Km为4.89×10-4mol/L(图9d),在大多数PAL的Km(0.3×10-4~1.5×10-2mol/L)范围内偏低[24]。
3 结 论
在本实验中成功地克隆出了OePAL全长,序列分析表明,OePAL的编码序列和翻译的氨基酸序列与其他物种有较高的同源性。酶切连接构建的pPICZα A-OePAL表达载体成功地转入到毕赤酵母X33中,最终实现了分泌表达,表达产物经电泳检测其分子质量与预测的基本一致,经测定该重组酶的比活力为196.3 U/mg。酶学性质研究表明,该重组酶的最适反应条件为40 ℃、pH 8.8,热稳定性与pH值稳定性研究表明该重组酶不耐高温、不耐酸与强碱,Cu2+与Na+对该重组酶的酶促反应有微弱的促进作用,酶促反应动力学研究表明该重组酶与L-Phe的亲和力较低。
虽然目前对植物中苯丙氨酸代谢途径比较清楚,但该代谢过程既受其遗传背景与生长发育程序的调控,也受生长环境中各种诱导因素的刺激,是一个复杂的动态过程。在油橄榄中由苯丙氨酸参与主导次生代谢物合成的关键酶与相关的功能基因和结构基因以及具体的代谢途径尚有待研究,因此,对OePAL的研究不仅为探究与苯丙氨酸相关的次生代谢途径奠定基础,且为采用分子育种技术对油橄榄进行性状改良提供了思路。
参考文献:
[1]LONG H S, TILNEY P M, van WYK B E. The ethnobotany and pharmacognosy of Olea europaea subsp. africana (Oleaceae)[J]. South African Journal of Botany, 2010, 76(2): 324-331.
[2]王晓飞, 李辰, 郑媛媛, 等. 油橄榄叶多酚类成分研究[J]. 中草药, 2011, 42(5): 848-851.
[3]CARAMIA G, GORI A, VALLI E, et al. Virgin olive oil in preventive medicine: from legend to epigenetics[J]. European Journal of Lipid Science and Technology, 2012, 114(4): 375-388.
[4]林远辉, 高蓓, 李玉玉, 等. 橄榄油掺假鉴别技术研究进展[J]. 食品科学, 2013, 34(5): 279-283.
[5]YUAN Shimin, JING Hua. Insights into the monomers and single drugs of Chinese herbal medicine on myocardial preservation[J]. African Journal of Traditional, Complementary and Alternative Medicines, 2011, 8(2): 104-127.
[6]LIU Hail, JIANG Wenb, XIE Mengx. Flavonoids: recent advances as anticancer drugs[J]. Recent Patents on Anti-cancer Drug Discovery, 2010, 5(2): 152-164.
[7]AMMAR R B, BHOURI W, SGHAIER M B, et al. Antioxidant and free radical-scavenging properties of three flavonoids isolated from the leaves of Rhamnus alaternus L.(Rhamnaceae): a structure-activity relationship study[J]. Food Chemistry, 2009, 116(1): 258-264.
[8]SCHIJLEN E G W M, RIC de VOS C H, van TUNEN A J, et al. Modification of flavonoid biosynthesis in crop plants[J]. Phytochemistry, 2004, 65(19): 2631-2648.
[9]NISHIHARA M, NAKATSUKA T. Genetic engineering of flavonoid pigments to modify flower color in floricultural plants[J]. Biotechnology Letters, 2011, 33(3): 433-441.
[10]LIN-WANG K, MICHELETTI D, PALMER J, et al. High temperature reduces apple fruit colour via modulation of the anthocyanin regulatory complex[J]. Plant, Cell and Environment, 2011, 34(7): 1176-1190.
[11]陆娣, 朱宽鹏, 夏晚霞, 等. 植物芪类合成途径相关酶、基因和调控机制研究进展[J]. 现代生物医学进展, 2013, 13(3): 573-577.
[12]郭欣慰, 黄丛林, 吴忠义, 等. 植物类黄酮生物合成的分子调控[J].北方园艺, 2011(4): 204-207.
[13]范丽. 桑树木质素合成基因的生物信息和功能分析[D]. 重庆: 西南大学, 2013: 5.
[14]FUKASAWAAKADA T, WATSON J C, KUNG S D. Phenylalanine ammonia-lyase gene structure, expression, and evolution in nicotiana[J]. Plant Molecular Biology, 1996, 30(4): 711-722.
[15]MINAMI E, OZEKI Y, MATSUOKA M, et al. Structure and some characterization of the gene for phenylalanine ammonia-lyase from rice plants[J]. European Journal of Biochemistry, 1989, 185(1): 19-25.
[16]GOWRI G, PAIVA N L, DIXON R A. Stress responses in alfalfa (Medicago sativa L.) 12. Sequence analysis of phenylalanine ammonia-lyase (PAL) cDNA clones and appearance of PAL transcripts in elicitor-treated cell cultures and developing plants[J]. Plant Molecular Biology, 1991, 17(3): 415-429.
[17]吴松权, 全雪丽, 朴炫春, 等. 膜荚黄芪苯丙氨酸解氨酶在大肠杆菌中的表达和纯化[J]. 延边大学农学学报, 2010, 32(4): 229-232.
[18]薛军, 马双, 邓霞, 等. 苦荞苯丙氨酸解氨酶基因(FtPAL)的原核表达及其逆向催化酶学性质分析[J]. 农业生物技术学报, 2014, 22(1): 64-70.
[19]张健慧, 王首锋. 膜荚黄芪苯丙氨酸解氨酶基因在毕赤酵母中的分泌表达[J]. 浙江大学学报: 农业与生命科学版, 2014, 40(1): 1-8.
[20]GURUDEEBAN S, RAMANATHAN T, SATYAVANI K, et al. Standardization of DNA isolation and PCR protocol for RAPD analysis of Suaeda sp.[J]. Asian Journal of Biotechnology, 2011, 3: 486-492.
[21]李娟. 海湾扇贝抗氧化相关基因的克隆、表达和结构分析[D]. 北京: 中国科学院, 2010: 29-33.
[22]WANG Zhen, YE Shafei, LI Jingjing, et al. Fusion primer and nested integrated PCR (FPNI-PCR): a new high-efficiency strategy for rapid chromosome walking or flanking sequence cloning[J]. BMC Biotechnology, 2011, 11(1): 109-120.
[23]ORTEGA-GARCÍA F, BLANCO S, PEINADO M, et al. Phenylalanine ammonia-lyase and phenolic compounds in leaves and fruits of Olea europaea L. cv. Picual during ripening[J]. Journal of the Science of Food and Agriculture, 2009, 89(3): 398-406.
[24]孙红梅, 赵爽, 王春夏, 等. 百合鳞茎苯丙氨酸解氨酶的分离纯化及酶学性质研究[J]. 园艺学报, 2008, 35(11): 1653-1660.
[25]江柯, 卢涛, 赵德立, 等. 红酵母苯丙氨酸解氨酶的分离纯化及性质研究[J]. 四川大学学报: 自然科学版, 2004, 41(4): 865-868.
[26]江力, 袁怀波, 张世杰, 等. 山药苯丙氨酸解氨酶特性的研究[J]. 食品科学, 2006, 27(10): 36-40.
[27]孟延发. 无壳葫芦籽苯丙氨酸解氨酶的纯化及其基本性质[J]. 兰州大学学报: 自然科学版, 1991, 27(2): 134-139.
[28]孟延发, 辛嘉英. 植物苯丙氨酸解氨酶的研究[J]. 兰州大学学报: 自然科学版, 1990, 26(3): 109-113.
[29]武敏. 盐肤木苯丙氨酸解氨酶基因的克隆及其功能性质研究[D]. 太原: 山西大学, 2012: 48.
Cloning of Phenylalanine Ammonia Lyase Gene from Olea europaea and Its Expression in Pichia pastoris
CHEN Wenshuan1, HUANG Qianming1,*, CHEN Huaping1, YANG Zeshen2, WANG Anyi2, SU Guangcan2
(1. College of Science, Sichuan Agricultural University, Ya’an 625014, China; 2. Liang Shan Zhong Ze New Technology Developme nt Co. Ltd., Xichang 615000, China)
Abstract: The phenylalanine ammonia lyase gene (PAL) was clo ned from Ol ea europaea by homology cloning, RT-PCR, FPNI-PCR (fusion primer and nested integrated PCR) and 3’-RACE, and named as OePAL. Sequencing showed that the full-length DNA of OePAL was 2 970 bp (GenBank, KJ511867) with an intron (393–1 220 bp). Meanwhile, the full-lengt h cDNA of OePAL was 2 142 bp (GenBank, KJ511868). The open reading frame (ORF) of OePAL encoded 713 amino acid residues, and sequence analysis suggested that OePAL had high homology with other botanic PALs. The full-length exon of OePAL expression vector pPICZα A-OePAL was constructed and expressed in Pichia pastoris strain X33. SDS-PAGE analysis showed that the molecular weight of recombinant enzyme was approximately 77.5 kD, and the specific activity of purifi ed enzyme was 196.3 U/mg. The enzymatic properties of recombinant 6×His-OeP AL indicated that the optimum reaction cond itions were 40 ℃ and pH 8.8. Ions such as Cu2+and Na+could enhance the enzyme activity within the tested range. The enzyme had a Kmof 4.89 × 10-4mol/L for L-phenylalanine at the optimum reaction conditions. Results indicated that OePAL was amplified and sub-cloned into the vector of pPICZα A, and its function was validated s uccessfully.
Key words: phenylalanine ammonia lyase (PAL); gene cloning; expression vector; Pichia pastoris strain X33
doi:10.7506/spkx1002-6630-201507023
中图分类号:Q812
文献标志码:A
文章编号:1002-6630(2015)07-0124-07
*通信作者:黄乾明(1964—),男,教授,博士,研究方向为生物化学与分子生物学。E-mail:huangqianming2014@126.com
作者简介:陈文拴(1988—),男,硕士研究生,研究方向为生物化学与分子生物学。E-mail:chenwenshuan@126.com
基金项目:四川省科技厅科技支撑计划项目(12ZC2220)
收稿日期:2014-06-13