表1 MRS培养基上分离乳酸菌的菌落形态及数量和比例
Table 1 Morphological characteristics, proportion and number of lactic acid bacteria on MRS

李 佳1,李 艳1,2,牟德华1,*
(1.河北科技大学生物科学与工程学院,河北 石家庄 050018;2.河北省发酵工程技术研究中心,河北 石家庄 050018)
摘 要:对传统乳制品开菲尔发酵剂通过菌种分离和纯化得到67 株乳酸菌,经过传统形态学分类区分成6 种形态类型。在此基础上进行16S rDNA限制性片段长度多态性聚合酶链反应(polymerase chain reaction-restriction fragment length polymorphism,PCR-RFLP)分析法鉴定为4 种分子类型,分别为:乳明串株菌(L. lactis)、坚强肠球菌(E. durans)、意大利肠球菌(E. italicus)、嗜热链球菌(S. thermophilus)。其中坚强肠球菌菌数超过50%,占所分离菌株的62.7%,为优势菌。
关键词:开菲尔发酵剂;乳酸菌;16S rDNA PCR-RFLP
开菲尔(Kefir)是一种发酵乳,起源于俄罗斯北高加索地区,与普通酸奶不同,它是由数10 种乳酸菌及酵母菌所形成的复合型发酵剂开菲尔粒发酵形成的复合型发酵乳,是复杂微生物的共生体[1-2]。开菲尔能改善消化机能,提高钙的利用率,对肠道疾病、高血压、心脏病、脑血栓、心肌梗塞等有预防和缓解作用[3-4]。开菲尔发酵剂包括开菲尔粒和开菲尔发酵液。农牧民长期把牛奶装在羊皮口袋内,经过自然发酵而形成不规则的乳白色或浅黄色颗粒状物,称为开菲尔粒[5],开菲尔发酵液是利用开菲尔粒在牛奶中生长而获得的发酵液。开菲尔发酵剂中含有多种益生微生物,包括多种乳酸菌和酵母菌。在发酵乳制品的生产过程中,只能使用增殖后的牛奶作为新的发酵剂,这种传统的传代方式使开菲尔发酵剂极不稳定,容易造成杂菌的污染[6],限制了传统开菲尔发酵乳制品的工业化生产,以及新型动物性和植物性发酵乳产品的开发。因此筛选优良性能的乳酸菌,并开发研制具有自主知识产权的发酵剂,对我国发酵乳制品工业的发展有重要意义。
目前,乳酸菌的分子鉴定广泛采用16S rDNA 限制性内切酶片段长度多态性(restriction fragment length polymorphism,RFLP)分析技术[7-8],或与其他鉴定技术结合进行乳酸菌鉴定。16S rDNA具有保守性和高变性,RFLP技术是用不同的限制性内切酶对细胞基因组DNA进行酶切,然后进行电泳分析用于对比不同基因组件的核苷酸差异。16S rDNA聚合酶链式反应(polymerase chain reaction,PCR)技术与RFLP技术结合,可以把乳酸菌鉴定到种水平[9]。本研究从开菲尔粒和开菲尔发酵液中分离筛选乳酸菌,并进行传统的形态分类和分子生物学鉴定,为筛选具有优良性状的乳酸菌,并进一步开发新型动物性和植物性原料发酵乳奠定基础。
1.1 材料与培养基
开菲尔粒和开菲尔发酵液均取自内蒙古自治区牧区的牧民家中,共计414 个样品。
MRS培养基[10]:蛋白胨10 g/L、牛肉膏10 g/L、葡萄糖20 g/L、碳酸钙10 g/L、酵母浸粉5 g/L、柠檬酸氢二胺2 g/L、乙酸钠2 g/L、磷酸氢二钾2 g/L、硫酸镁0.58 g/L、硫酸锰0.25 g/L、吐温-80 1 mL/L、琼脂18 g/L。pH 6.2~6.6,121 ℃灭菌20 min。
1.2 试剂与设备
引物27F(5’-TCC GTA GGT GAA CCT GCG G-3’),1495R(5’-TCC TCC GCT TAT TGA TAT GC-3’)[11]由上海生工生物工程技术服务有限公司合成;HaeⅢ,HinfⅠ,HhaⅠ限制性内切酶、DNA Marker 上海生工生物工程技术服务有限公司。
PCR仪、凝胶成像分析仪 美国Bio-Rad公司;琼脂糖水平电泳仪 北京市六一仪器厂。
1.3 方法
1.3.1 乳酸菌的分离
取1 g开菲尔粒研磨,加入10 mL无菌水溶解,摇匀进行10 倍梯度稀释,选择3 个适宜的稀释度,取0.2 mL稀释液于MRS培养基,37 ℃培养48 h,从平板上挑取乳酸菌,再在相同的培养基上进行划线纯培养取得单菌落[12],保藏。
1.3.2 乳酸菌的形态聚类
将分离得到的乳酸菌进行复筛,接种在MRS培养基上,37 ℃培养4 d,观察乳酸菌的菌落颜色、形态、边缘是否平整、表面的光滑程度等菌落特征。同时进行过氧化氢实验、革兰氏染色和显微镜观察,并记录细胞的显微形态,以此对乳酸菌进行初步的形态聚类和保藏。
1.3.3 乳酸菌的分子鉴定
乳酸菌的分子鉴定采用16S rDNA PCR-RFLP序列分析法。将初步形态聚类的乳酸菌每种形态分别随机选取一株进行分子鉴定,十六烷基三甲基溴化胺(cetyltrimethylammonium bromide,CTAB)法[13-14]提取乳酸菌DNA,PCR扩增采用通用引物,正向引物为27F:5’-TCC GTA GGT GAA CCT GCG G-3’;反向引物为1495R:5’-TCC TCC GCT TAT TGA TAT GC-3’。
PCR反应体系包括:双蒸水34.6 μL,10×Buffer 10 μL,10 mmol/L dNTP 1 μL,10 μmol/L的正向引物2 μL,10 μmol/L的反向引物2 μL,DNA模板2 μL,5 U/μL Taq酶0.4 μL,反应总体积为50 μL。
PCR反应程序[15]:94 ℃ 预变性 5 min,变性 1 min,58 ℃ 退火 1 min,72 ℃ 延伸 2 min,共30 个循环,72 ℃最后延伸10 min。将得到的PCR产物用2%的琼脂糖凝胶进行电泳,电泳条件为:电压90 V,时间50 min,溴化乙锭(ethidium bromide,EB)染色20 min,凝胶成像分析仪进行观察并摄影。
PCR扩增产物进行限制性酶切分析(20 μL):双蒸水7 μL,5×Buffer 2 μL,PCR扩增产物10 μL,分别用0.5 U/μL HhaⅠ、HinfⅠ、HaeⅢ 内切酶[16]1 μL进行酶切。酶切条件均为:37 ℃水浴2.5 h,取出,酶切产物用2%琼脂糖凝胶进行电泳,酶切产物加样量8 μL与1 μL的10×Loading Buffer混匀后加样,加入5 μL的Marker为对照,电泳电压90 V,时间50 min。EB染色20 min,凝胶成像分析仪下进行观察并摄影。
2.1 乳酸菌的分离与形态聚类
开菲尔发酵剂经MRS培养基的分离纯化共得到67 株乳酸菌,均为过氧化氢实验阴性、革兰氏染色阳性的菌株。根据菌落颜色和形态特征,结合显微镜下细胞的形态共分为6 种类型,结果见表1。
表1 MRS培养基上分离乳酸菌的菌落形态及数量和比例
Table 1 Morphological characteristics, proportion and number of lactic acid bacteria on MRS

不同种类的乳酸菌生长在MRS培养基上,其菌落颜色、形态、大小等都有所不同,结合菌落形态和细胞的显微形态可进行乳酸菌的判断和区分。由表1可知,形态2的乳酸菌在开菲尔发酵剂样品中数量最多,占所分离乳酸菌的62.7%,超过50%,为优势菌群,其菌落形态和细胞显微形态如图1所示。
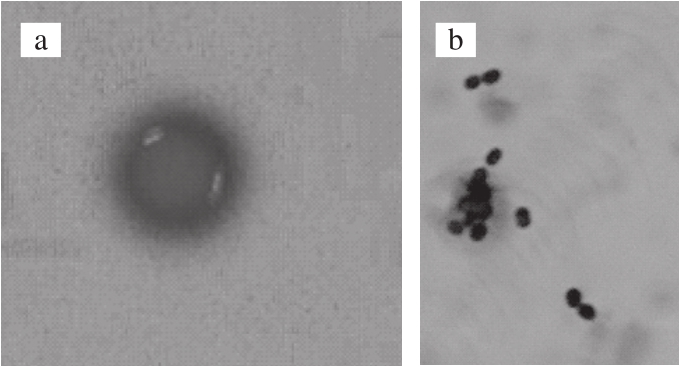
图1 优势乳酸菌的菌落(a)以及细胞显微(b)形态(40×)
Fig.1 Morphology of the predominant lactic acid bacteria colonies (a) and cells (b) (40×)
2.2 乳酸菌的16S rDNA PCR-RFLP序列分析
乳酸菌的16S rRNA约含有1 540 个碱基,结构既具有保守性,又具有高变性,16S rDNA是编码核糖体RNA对应的DNA序列,其原理是从乳酸菌中通过克隆测序或酶切探针杂交获得16S rDNA序列信息,可作为鉴定乳酸菌的分类依据[17-19]。本研究在对分离得到的乳酸菌进行菌落及显微形态区分的基础上,分别从每种形态类型中随机选择1 株乳酸菌进行16S rDNA PCR-RFLP序列分析,6 种形态乳酸菌的PCR扩增产物电泳结果见图2。同时使用HaeⅢ、HinfⅠ和HhaⅠ内切酶对PCR扩增产物进行酶切分析,酶切电泳结果见图3。
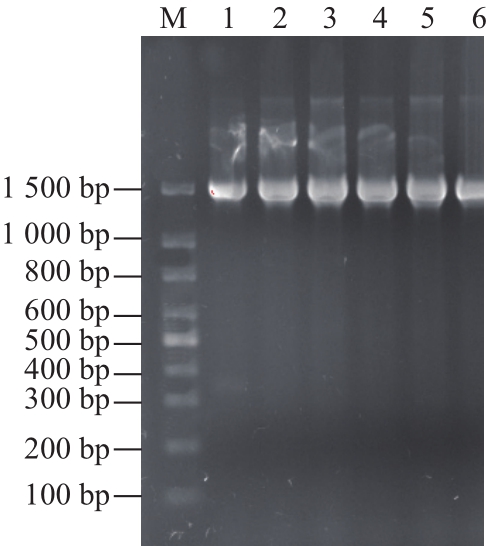
图2 PCR扩增产物电泳图谱
Fig.2 Electrophoretogram of PCR-amplified products of 16S rDNA
M. 100 bp DNA Marker;泳道1~6依次为6 种形态乳酸菌的类型。

图3 PCR产物的3 种限制性内切酶酶切分析图谱
Fig.3 Restriction analysis pattern of PCR-amplified products of 16S rDNA
M. 100 bp DNA Marker;泳道1~6、7~12、13~18依次为内切酶HhaⅠ、HinfⅠ、HaeⅢ的酶切结果。
由图2可知,6 种乳酸菌的PCR扩增片段大小都在1 500 bp左右,经过HaeⅢ、HinfⅠ和HhaⅠ内切酶进行酶切后,可以将其区分为4 种分子类型。Ⅰ类:形态1,PCR产物大小1 492 bp,第1泳道HhaⅠ酶切为4 段:539、409、378、140 bp;第7泳道由HinfⅠ酶切为2 段:1 101、262 bp;第13泳道经HaeⅢ酶切为3 段:870、369、177 bp。Ⅱ类:形态2,PCR产物为1 492 bp,第2泳道经HhaⅠ酶切为4段:541、413、382、222 bp;第8泳道由HinfⅠ酶切为4 段:598、457、312、284 bp;第14泳道经HaeⅢ酶切为3 段:即867、373、170 bp。Ⅲ类:形态3、5,PCR产物1 492 bp,第3和第5泳道经HhaⅠ酶切为4段:535、411、377、219 bp;第9和第11泳道由HinfⅠ酶切为3 段:597、455、269 bp;第15和第17泳道经HaeⅢ酶切为3 段:934、332、166 bp。Ⅳ类:形态4、6,PCR产物1 484 bp,第4和第6泳道经HhaⅠ酶切为4 段:585、530、373、216 bp;第10和第12泳道由HinfⅠ酶切为3段,即595、453和300 bp;第16和第18泳道由HaeⅢ酶切为4 段:473、399、321、162 bp。
根据乳酸菌16S rDNA 扩增产物及3 种限制性内切酶酶切产物电泳图,利用Image Lab软件对PCR以及酶切条带进行解读,不同形态类型的乳酸菌16S rDNA分析结果,即PCR产物分子质量大小和酶切片段分子质量大小见表2。
表2 6种不同类型的乳酸菌16S rDNA PCR-RFLP分析结果
Table 2 results of PFLP analysis of 16S rDNA region for lactic acid bacteria

由表2可知,通过比较不同形态的乳酸菌株PCR扩增产物及酶切片段大小,能够将这6 种不同形态类型的酵母菌区分为4 种分子类型。乳酸菌的PCR产物分子质量大小为1 484~1 492 bp。经限制性内切酶HaeⅢ、HinfⅠ和HhaⅠ酶切分析后得到4 种不同的酶切片段类型。
把4 种分子类型的乳酸菌随机选择1 株进行基因测序,将所得基因序列结果登陆GenBank数据库,进行BLAST(http://www.ncbi.nlm.nih.gov/blast/Blast.Cgi)相似性比对[20-21],以相似度大于99%确定其属种,测序结果见表3。
表3 乳酸菌16S rDNA序列分析
Table 3 results of 16S rDNA sequence Analysis for lactic acid bacteria
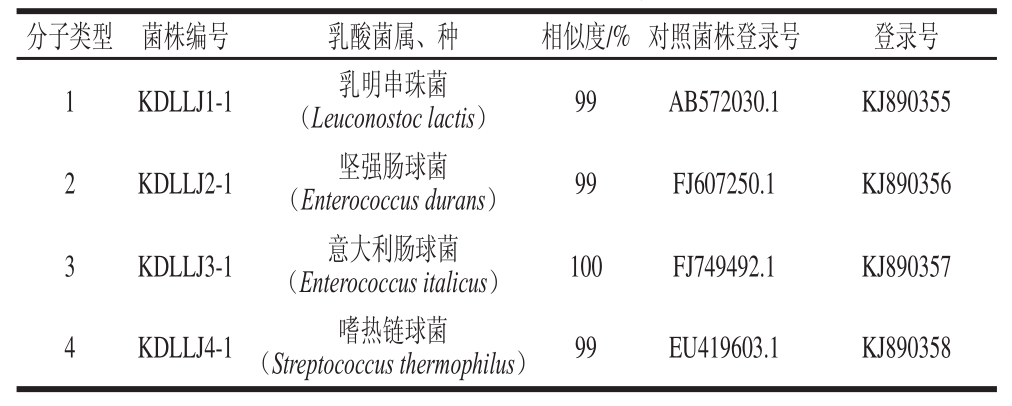
由表3可知,经过形态学区分和分子生物学鉴定(16S rDNA PCR-RFLP分析),可以将6 种形态的乳酸菌归为4 种,分别为乳明串株菌(L. lactis)、坚强肠球菌(E. durans)、意大利肠球菌(E. italicus)、嗜热链球菌(S. thermophilus)。
通过邻接法(Neighbor-Joining)构建的系统发育树判断乳酸菌的同源关系[22],如图4所示。在系统发育树上同一分支的乳酸菌同源关系最近,由图4的聚类结果可以清楚地看出所分离乳酸菌的分类情况及各自的种属名称。
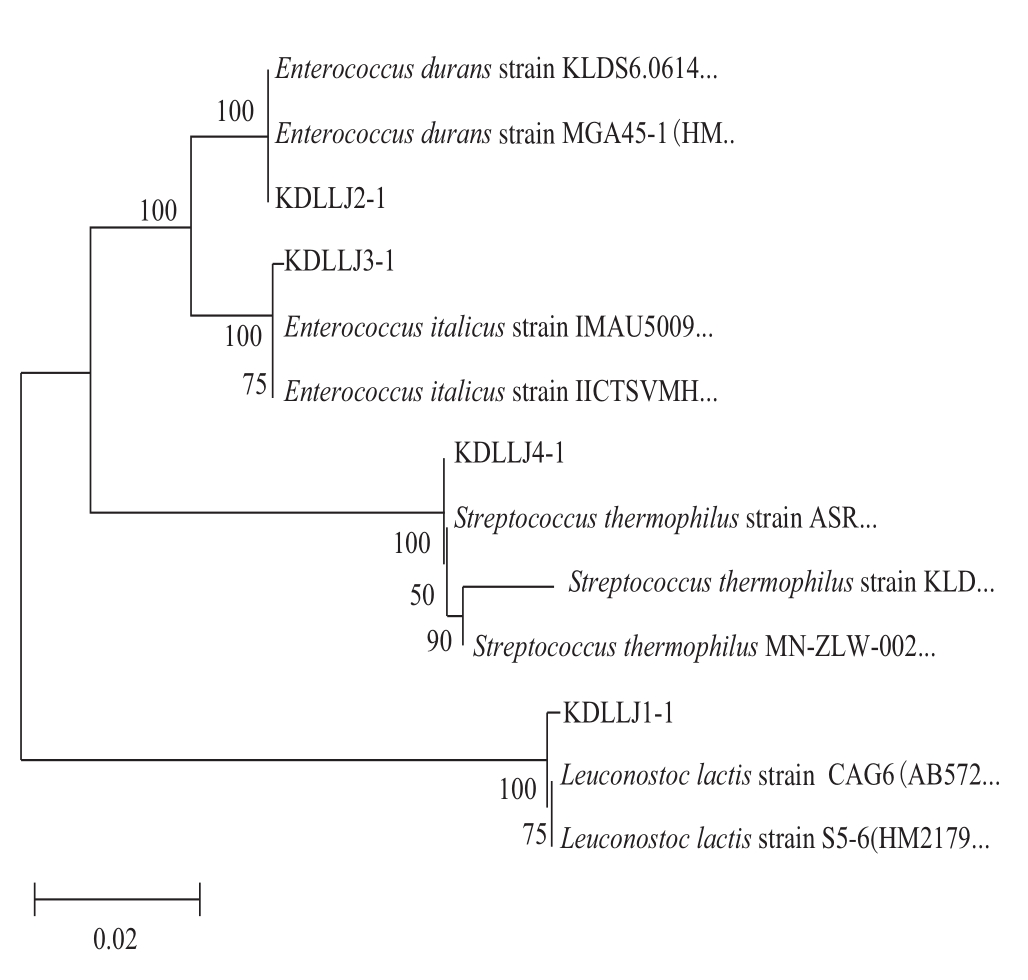
图4 基于16S-rDNA序列和 Neighbor-Joining 法构建的系统发育树
Fig.4 Phylogenetic tree based on the sequences of 16S rDNA region and neighbor-jointing method
本研究对传统乳制品开菲尔发酵剂中分离到的67 株乳酸菌通过形态聚类分为6 种形态类型,16S rDNA PCRRFLP法鉴定为4 种分子类型,其中坚强肠球菌为优势菌群。本研究将传统乳酸菌的形态学分析与现代分子技术相结合,通过基因序列分析使菌种鉴定结果的准确性和可靠性提高。研究结果可以反映传统乳制品中乳酸菌的多样性,为进一步开发研究新型动物性或植物性蛋白乳发酵饮料奠定了理论基础。
参考文献:
[1]张丽霞, 黄开红, 周剑忠. 国外开菲尔粒中乳酸菌的分离纯化与鉴定研究[J]. 江西农业学报, 2008, 20(5): 85-87.
[2]刘慧, 李平兰, 宫品, 等. 开菲尔粒中酵母菌的分离及其发酵性能的研究[J]. 食品科学, 2005, 26(2): 97-100.
[3]宋欢, 韩燕, 张晓敏, 等. 开菲尔发酵奶的研究进展[J]. 中国乳品工业, 2007, 35(4): 51-55.
[4]张慧敏, 李远志. 开菲尔的营养成分、保健功能及其产品研究进展[J].现代食品科技, 2005, 21(4): 118-119.
[5]刘慧, 李平兰, 高秀芝, 等. 开菲尔粒中乳酸菌的分离方法研究[J].食品科学, 2004, 25(6): 56-60.
[6]郭彩华, 蔡慧农, 杨秋明, 等. 开菲尔粒的生长特性及开菲尔发酵剂[J].食品研究与开发, 2013, 34(19): 93-98.
[7]BARBIERI E, SCHIAVANO G F, de SANTI M, et al. Bacterial diversity of traditional Fossa (pit) cheese and its ripening environment[J]. International Dairy Journal, 2012, 23(1): 62-67.
[8]ROBERT H, GABRIEL V, FONTAGNÉ-FAUCHER C. Biodiversity of lactic acid bacteria in French wheat sourdough as determined by molecular characterization using species-specific PCR[J]. International Journal of Food Microbiology, 2009, 135(1): 53-59.
[9]董振玲, 李艳. 乳制品中乳酸菌分子鉴定技术进展[J]. 中国酿造, 2012, 31(6): 9-14.
[10]GEVERS D, HUYS G, SWINGS J. Applicability of rep-PCR fingerprinting for identification of Lactobacillus species[J]. FEMS Microbiology Letters, 2001, 205: 31-36.
[11]艾日登才次克, 于洁, 杜晓华, 等. 16S rDNA RFLP法快速鉴定蒙古国传统乳制品中的乳酸菌[J]. 中国农业科技导报, 2009, 11(2): 63-68.
[12]ZAMFIRA M, VANCANNE YT M, MAKRAS L, et al. Biodiversity of lactic acid bacteria in Romanian dairy products[J]. Systematic and Applied Microbiology, 2006, 29(6): 487-495.
[13]APONTE M, FUSCO V, ANDOLFI R, et al. Lactic acid bacteria occurring during manufacture and ripening of Provolone del Monaco cheese: detection by different analytic al approaches[J]. International Dairy Journal, 2008, 18(4): 403-413.
[14]LIU Wenjun, BAO Qiuhua, JIRIMUTU, et al. Isolation and identification of lactic acid bacteria from Tarag in Eastern Inner Mongolia of China by 16S rRNA sequences and DGGE analysis[J]. Microbiological Research, 2012, 167(2): 110-115.
[15]于洁, 孙志宏, 张家超, 等. 16S rDNA-RFLP技术鉴定西藏地区乳制品中的乳杆菌[J]. 食品与生物技术学报, 2011, 28(6): 804-810.
[16]杜晓华. 内蒙古西部地区酸粥中乳酸菌的分离与鉴定[D]. 呼和浩特: 内蒙古农业大学, 2009.
[17]RANDAZZO C L, RESTUCCI A C, ROMANO A D, et al. Lactobacillus casei, dominant species in naturally fermented Sicilian green olives[J]. International Journal of Food Microbiology, 2004, 90(1): 9-14.
[18]阳大海, 陈卫红, 沈伟达, 等. 开菲尔粒中优势菌的筛选及鉴定[J].安徽农业科学, 2010, 38(6): 3146-3148.
[19]剧柠. 西藏、新疆和云南地区传统发酵乳制品中乳杆菌的生物多样性研究[D]. 呼和浩特: 内蒙古农业大学, 2009.
[20]ZHANG Jiachao, LIU Wenjun, SUN Zhihong, et al. Diversity of lactic acid bacteria and yeasts in traditional sourdoughs collected from western region in Inner Mongolia of China[J]. Food Control, 2011, 22(5): 767-774.
[21]ZHOU Jianzhong, LIU Xiaoli, JIANG Hanhu, et al. Analysis of the microflora in Tibetan kefir grains using denaturing gradient gel electrophoresis[J]. Food Microbiology, 2009, 26(8): 770-775.
[22]包秋华, 吕嫱, 于洁, 等. 四川牦牛奶和曲拉中九株乳酸菌的分子鉴定[J]. 食品与生物技术学报, 2012, 31(4): 369-401.
LI Jia1, LI Yan1,2, MOU Dehua1,*
(1. College of Bioscience and Bioengineering, Hebei University of Science and Technology, Shijiazhuang 050018, China; 2. R&D Center for Fermentation Engineering of Hebei Province, Shijiazhuang 050018, China)
Abstract: Lactic acid bacteria in kefir starter cultures were analyzed and identified to obtain strains with outstanding genetic traits and excellent fermentation characteristics. A total of 67 lactic acid bacteria strains were obtained directly from kefir starter cultures by isolation and streak plating. All of these stains could be divided into 6 different groups by conventional microbiological analysis. Then 4 genotypes were classified by RFLP analysis of the 16S rDNA region, including L. lactis, E. durans, E. italicus and S. thermophilus. E. durans was the dominant species accounting for 62.7% of all the isolates.
Key words: kefir starter cultures; lactic acid bacteria; 16S rDNA PCR-RFLP
doi:10.7506/spkx1002-6630-201507029
中图分类号:TS201.3
文献标志码:A
文章编号:1002-6630(2015)07-0158-04
*通信作者:牟德华(1960—),男,教授,学士,研究方向为农产品加工。E-mail:dh_mou@163.com
作者简介:李佳(1990—),女,硕士研究生,研究方向为农产品加工。E-mail:974705395@qq.com
收稿日期:2014-07-11