
图1 样品总DNA电泳图
Fig.1 Electrophoresis of total DNA extracted from samples
夏小龙 1,彭 珍 1,刘书亮 1,2,*,韩新锋 1,2,周 康 1,2,邹立扣 3,赖海梅 1
(1.四川农业大学食品学院,四川 雅安 625014;2.四川省农产品加工及贮藏工程重点实验室,四川 雅安 625014;3.四川农业大学都江堰校区微生物学实验室,四川 都江堰 611830)
摘 要:基于16S rDNA V 6~V 8可变区的聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reaction-denaturing gradient gel electrophoresis,PCR-DGGE)技术分析肉鸡屠宰加工过程中减菌处理前后胴体或产品细菌多样性。在预冷环节前采用50 ℃、1.5%乳酸溶液对肉鸡胴体冲淋15 s进行减菌处理,采集屠宰加工环节中减菌处理前后的胴体或分割产品表面样品,提取样品中的细菌总DNA,通过16S rDNA V 6~V 8可变区的PCR扩增,变性梯度凝胶电泳,对PCR扩增 片段割胶回收、克隆测序分析减菌前后细菌菌相变化。结果表明,减菌前,胴体清洗环节DGGE条带的数量最多、亮度最 强,细菌污染最严重,其次是分割 环节,而预冷环节细菌种类及数量最少,污 染程度最低;减菌后,各屠宰加工环节细菌种类与数 量较减菌前均有所减少,其中胴体清洗环节与分割环节细菌的种类与数量减少量最多,预冷环节细菌的种类及数量最少,不同屠宰加工环节细菌种类并不完全一致;乳杆菌属细菌在整个肉鸡屠宰加工过程中均有出现,与肠杆菌科和假单胞菌属细菌为肉鸡屠宰加工过程中的优势腐败菌。
关键词:聚合酶链式反应-变性梯度凝胶电泳;肉鸡;屠宰;减菌;细菌菌相
鸡肉是世界上消费量增长速度最快,物美价廉的优质肉类,其安全性愈发受到人们关注。健康鸡肉组织内部是没有腐败微生物与病原微生物污染的,然而肉鸡在商业屠宰过程中,其皮肤、羽毛、消化道等均携带大量的天然菌群,肉鸡胴体及其分割产品表面微生物的污染是不可避免的 [1-2]。但在某些加工环节,如浸烫脱毛、预冷等环节,又能够 有效减少微生物的数量 [3],而有些操作点也会成为交叉污染点。
目前,肉鸡生产过程中微生物污染很多基于传统培养法研究微生物数量以确定污染源 [4-5]。Ampe等 [6]证 明至少25%~50%的微生物菌群不能有效地在外界培养,导致不能全面反映微生物分布的真正情况 [7-8]。变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)可避免传统微生物技术的缺陷,能更全面地认识微生物分布,更加有效地分析微生物污染源 [9-10]。本研究在彭珍等 [11]对肉鸡屠宰加工过程中肉鸡胴体与产品的微生物污染分析及减菌研究基础上,通过聚合酶链式反应(polymerase chain reaction,PCR)-DGGE技术研究在肉鸡商业屠宰加工过程中,预冷环节前使用乳酸结合热水进行减菌处理,分析减菌前后细菌菌相变化情况,旨在探究肉鸡商业屠宰过程中细菌的分布、变化和减菌效果,为实际生产中的微生物污染控制提供理论依据。
1.1 材料与试剂
四川某肉鸡屠宰加工厂生产链上(脱毛、掏膛、胴体清洗、预冷)的肉鸡胴体以及分割、速冻后的肉鸡产品。
TIANDZ柱式细菌DNAOUT 成都康迪生物技术有限公司;Premix Taq、Gold View核酸染料、DNA Marker DL2000、Agarose Gel DNA Purifcation Kit Ver.2.0、X-Gal、IPTG、Amp、pMD19-T载体 宝生物工程(大连)有限公司;0.1 mol/L磷酸盐(pH 7.0)缓冲液、丙烯酰胺、双丙烯酰胺、乙二胺四乙酸二钠(EDTA-Na 2)、去离子甲酰胺、尿素、Tris、过硫酸胺、N,N,N’,N’-四甲基乙二胺、50×TAE缓冲液、6%丙烯酰胺变性胶、固定液、染色液、显色液。
1.2 仪器与设备
DTSK-2401 220变性梯度凝胶电泳、PTC-200 PCR仪(My Cycler TMThermal Cycler)、Gel Doc XR凝胶成像系统 美国Bio-Rad公司;B4i-BR4i冷冻离心机 美国Thermo公司。
1.3 方法
1.3.1 肉鸡胴体及产品表面取样
于肉鸡屠宰加工厂用棉拭子擦拭法分别取脱毛、掏膛、胴体清洗、预冷的肉鸡胴体表面、分割以及速冻环节的肉鸡产品表面污染样品。每支灭菌棉拭子涂2 个点,每点涂5 cm 2,一个样用5 支灭菌棉拭子涂擦,共涂擦50 cm 2(头肛部各5 cm 2、背部10 cm 2、鸡翅和鸡腿各5 cm 2、胸部10 cm 2、腹部10 cm 2) [12]涂后放入装有50 mL磷酸盐的EP管中,于冰盒(4 h内)运至实验室。
1.3.2 减菌处理方法
采用50 ℃、1.5%乳酸溶液在预冷环节前对肉鸡胴体冲淋15 s(以秒表计时)进行减菌处理 [11],每次冲淋12 只肉鸡,分3 个批次冲淋,共冲淋36 只肉鸡,并分别在胴体清洗后、预冷后、分割后及速冻后环节,用棉拭子擦拭法按1.3.1节取样。
1.3.3 样品预处理
将棉拭子在装有磷酸盐的50 mL离心管中,浸提30 min左右后取出,然后将离心管于3 000 r/min离心3 min,去除底部杂质,上清液再于12 000 r/min离心10 min,弃掉上清液,得菌体沉淀。
收集的菌体用5 mL 0.1 mol/L的磷酸盐缓冲液(pH 7.0)悬浮,9 000 r/min离心10 min,重复洗涤3 次,洗净的菌体悬浮在5 mL磷酸盐缓冲液(pH 7.0)中,用移液器吹打后振荡器振荡均匀,平均分装3 份于2 mL 离心管中,-20 ℃冻存。
1.3.4 细菌总DNA提取
按照细菌DNA提取试剂盒使用说明提取细菌总DNA。将提取的细菌总DNA溶于100 μL的Tris-EDTA缓冲液,经1%琼脂糖凝胶电泳检测后,于-20 ℃冰箱保存备用。
1.3.5 PCR反应
用细菌16S rDNA V 6~V 8可变区通用引物,其上游引物为带有GC夹子的U968,下游 引物是L1401 [13]。U968-GC夹子为:5’-CGC CCG GGG CGC GCC CCG GGC GGG GCG GGG-GCA CGG GGG GAA CGC GAA GAA CCT TAC-3’。下游引物L1401为:5’-CGG TGT GTA CAA GAC CC-3’。上述引物均由成都微克天泰技术有限公司合成提供。
PCR扩增反应体系(50 μL):DNA 模板1、0.5 μL的引物(10 μmol)、预混体系25 μL、超纯水23 μL;PCR反应程序:94 ℃预变性5 min,35 个循环(94 ℃变性30 s,64 ℃退火30 s,72 ℃延伸40 s),最终72 ℃延伸7 min,PCR产物再用1%琼脂糖凝胶电泳检测后,将扩增产物放于-20 ℃保存备用。
1.3.6 DGGE分析
1.3.6.1 PCR扩增所得的产物进行电泳分离
参照Muyzer等 [9]的方法,对细菌16S rDNA的V 6~V 8区段的扩增产物进行DGGE分析并作改进。6 g/100 mL丙烯酰胺(质量分数40%丙烯酰胺/双丙烯酰胺),变性剂溶液线性梯度范围为45%~55%,先200 V电压条件下预电泳30 min,再在120 V电压条件下电泳16 h。
1.3.6.2 银染与图谱分析
DGGE电泳结束后,取出胶板,将凝胶取下先用固定液固定20~30 min,去离子水快速清洗2 次,再用AgNO 3溶液避光摇床染色20~30 min,染色结束后,将凝胶从染色液中取出,用去离子水快速清洗2 次,再将凝胶放入已预冷的显影液中摇床显影,条带显色完全后,迅速取出凝胶,将其放于凝胶成像仪上成像。
1.3.6.3 克隆及序列分析
选取图谱中清晰、明亮的优势条带按照Agarose Gel DNA Purifcation Kit Ver.2.0试剂盒说明割胶回收,然后再进行PCR扩增,扩增后再割胶回收,按DP214-03 Universal DNA Purification Kit试剂盒操作回收纯化目的条带,然后再取2 μL不含GC夹子的引物进行16S rDNA的V 6~V 8可变区域扩增,扩增后电泳检测、再将目的条带回收纯化,用1.0%琼脂糖凝胶电泳检验,再连接、转化进行T克隆后,挑白斑菌落活化12~14 h后,将菌液送于上海英俊生物科技有限公司进行测序,将序列登录NCBI,将所得序列与数据库中的已知序列进行相似性比对。将所有序列用ClustalX(1.81)比对后,用NYSYS2软件进行DGGE图谱的聚类分析,最后再用MEGA 5.0软件绘制系统发育树。
2.1 总DNA提取及PCR扩增
用试剂盒提取方法对样品总DNA进行提取,电泳结果如图1所示,可以看出,样品的总DNA提取效果较好,电泳条带清晰可见。

图1 样品总DNA电泳图
Fig.1 Electrophoresis of total DNA extracted from samples
以提取的总DNA为模板 ,采用U968-GC、L1401对各个样品中细菌16S rDNA V 6~V 8可变区进行PCR扩增,PCR产物电泳图如图2所示,扩增产物电泳条带清晰,特异性好,位于500 bp左右,均符合目的条带约450 bp片段长度的要求。
图2 16S rDNA V
6
~V
8
PCR扩增产物电泳图
Fig.2 Electrophoresis of PCR amplification products of the V
6
-V
8
regions of 16S rDNA
2.2 DGGE分析
根据DGGE基本原理,图谱中的每一根条带都对应于微生物群落中一个优势菌群或操作分类单位(operational taxonomic unit,OTU) [14],且图谱中条带数量的多少反映了样品中微生物组成的差 异,条带的亮度反映了样品中微生物的多少。对减菌前后肉鸡屠宰生产链中脱毛、掏膛、胴体清洗、预冷、分割、速冻后的6 个环节的肉鸡胴体或产品表面细菌的DGGE图谱见图3。可以看出,经不同加工环节后的肉鸡胴体或产品表面的细菌DGGE图谱存在一定的差异,且经50 ℃、1.5%乳酸溶液对肉鸡胴体冲淋15 s处理后4 个环节中肉鸡胴体或产品表面细菌的种类和数量均所减少,为进一步分析肉鸡屠宰生产链中细菌的群落结构以及多样性,将图3中所标记的11 条较明显的条带回收纯化、电泳检测、克隆测序结果见表1。
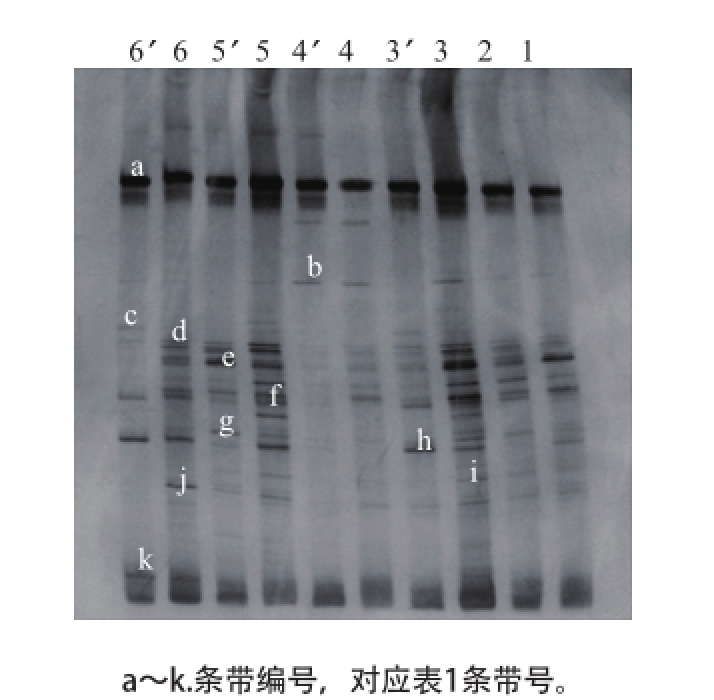
图3 减菌前后肉鸡屠宰生产链细菌DGE图谱
Fig.3 DGE profiles of bacteria in chicken samples collected during slaughtering and processing with and without bacterial reduction
表1 PCR-DGGE图谱中主要条带割胶回收对16S rDNA V
6
~V
8
可变区测序结果
Table1 Microbial species identification after sequencing of the variable V
6
-V
8
of the 16S rDNA gene purified from PCR-DGGE profiles

条带最大相似度/%最大相似菌株最相近的序列接受号a99乳杆菌属(Lactobacillus)FJ542292.1 b99不可培养细菌(uncultured bacterium)CQ899340.1 c99肠杆 菌科细菌(Enterobacteriaceae bacterium)KC560019.1 d97不可培养梭菌(uncultured Clostridiales bacterium)FJ440059.1 e96不可培养细菌(uncultured bacterium)DQ456364.1 f94瘤胃球菌属(Ruminococcus)RN 044265.1 g99 不可培养细菌(uncultured bacterium)GU171208.1 h99假单胞菌属(Pseudomonas sp.)KJ396170.1 i99荧光假单胞菌(Pseudomonas fl uorescens)JQ782495.1 j98不可培养瘤胃球菌属(uncultured Ruminococcus sp.)AY080980.1 k97不可培养假单胞菌属(uncultured Pseudomonas sp.)HM234018.1
图3表明,在1~6肉鸡屠宰加工环节中,不同加工环节之间细菌的种类与数量均有所差异。同一加工环节,不同条带的亮度也有所差异,说明不同种类的细菌数量不同。胴体清洗环节样品(标号3)条带的数量最多、亮度最强,故该环节的细菌种类最为丰富、数量最多,细菌污染最严重;其次是分割环节样品(标号5)中的细菌种类较丰富、数量较多;掏膛环节(标号2)中的细菌种类也较脱毛环节(标号1)丰富,数量较多;速冻环节(标号6)中的条带数量较少,主要为几种优势嗜冷菌;预冷环节(标号4)中的条带数量最少,亮度最弱,该环节细菌的种类与数量是肉鸡屠宰加工环节中最少的,也是细菌污染最轻的环节。
从图3的3-3’、4-4’、5-5’与6-6’对应加工环节减菌处理前后的DGGE图谱可以看出,采用50 ℃ 1.5%乳酸溶液减菌处理后,每个环节条带的数量均有较大程度的减少,条带的亮度也均有减弱,有些条带甚至消失。在胴体清洗环节与分割环节,经减菌处理后条带的数量减少最多,亮度减弱程度最大。而在预冷环节,减菌后几乎所有的条带均消失,仅存的条带亮度也很弱。综上可知,在肉鸡屠宰加工过程中该减菌方法能有效减少胴体及产品表面的细菌种类与数量。图4为所有样品DGGE图谱的聚类分析图,样品3’、5’与6最为相似,首先聚在一起,相似性高达0.88,说明胴体清洗和分割这2 个污染最为严重环节经减菌处理后,其样品中微生物种类的多样性与分割后速冻产品接近,这说明该处理对这2 个环节的污染控制起到了良好的效果。样品1、2相似度为0.88,但样品6’与其他样品相似度最低为0.49,可能是由于减菌处理后,样品表面部分微生物被杀灭,得到有效控制,致使该环节微生物种类最少。从图4的3-3’、4-4’、5-5’与6-6’对应加工环节减菌处理前后PCR-DGGE聚类分析图可知,3-3’、5-5’样品之间,减菌后样品的相似度较减菌前样品的相似度高0.06;4-4’样品之间的相似度均为0.75,减菌前后微生物的种类基本保持一致;6-6’样品之间的相似度差异最大,减菌前样品较减菌后样品的相似度高0.39,这可能是由于速冻环节的低温造成部分种类微生物死亡,导致微生物种类减少。

图4 PCR-DGE聚类分析图
Fig.4 Cluster analysis of PCR-DGE profiles
结合图3和表1可以看出,在不同加工环节的肉鸡胴体表面所取的样品中均存在一条明显的共有条带(标记为a),测序结果表明条带a是乳杆菌属(Lactobacillus),乳杆菌属的最适生长温度范围为30~40 ℃,其广泛分布于环境中,特别是动物、蔬菜及食品中,常寄主于鸟和脊椎动物的消化道以及哺乳动物的尿道,是一种存在于人类体内的益生菌。DGGE图谱显示,肉鸡胴体及分割产品表面主体细菌(条带a)含量较多,其他优势菌种在不同的生产环节中丰度有所差异。胴体清洗环节样品(标号3)条带的数量最多、亮度最强,意味着此环节的细菌种类最为丰富、数量最多。条带b为不可培养细菌(uncultured bacterium),在胴体清洗环节和预冷环节数量较多,减菌处理后,在胴体清洗环节消失,但在预冷环节又生长,很可能是由于预冷池中的水交叉污染所致。条带c为肠杆菌科细菌(Enterobacteriaceae bacterium),由图3可以看出,在肉鸡生产链中调查的6 个环节中,均有肠杆菌科 细菌的存在,但在胴体清洗环节、分割环节和速冻环节存在的数量较多,在其他环节条带c亮度较弱;减菌处理后条带c的亮度明显减弱,说明乳酸结合热水冲淋肉鸡胴体能有效减少肠杆菌科细菌。条带d、f分别是不可培养梭菌(uncultured Clostridiales bacterium)、瘤胃球菌属(Ruminococcus),同样也是在胴体清洗环节条带亮度最强,说明这2种菌的含量较高。条带e、g均为不可培养细菌(uncultured bacterium),在脱毛环节、胴体清洗环节和分割环节污染也较严重,减菌处理后速冻环节两条带的亮度也几乎消失。条带h为假单胞菌属(Pseudomonas sp.),由图3可以看出,在肉鸡屠宰生产链中的胴体清洗环节、分割环节和速冻环节污染较严重,减菌处理后在分割环节假单胞菌属的数量有所减少,但速冻后数量又有所上升,可能是分割环节中由于环境、刀具等的交叉污染所引起的。在脱毛、掏膛、胴体清洗、分割环节还有荧光假单胞菌(Pseudomonas fluorescens)(条带i)的存在,减菌处理后,条带i的亮度明显减弱,说明处理后此种细菌的数量明显减少。条带j为不可培养瘤胃球菌属(uncultured Ruminococcus sp.),处理后,其数量相对于减菌前也明显减少。条带k为不可培养假单胞菌属(uncultured Pseudomonas sp.),在胴体清洗环节和速冻环节污染较严重。

图5 减菌前后肉鸡屠宰生产链中胴体表面样品经DGE分析后所分离纯化细菌微生物系统发育树
Fig.5 Dendrogram of the DGE profiles of bacteria from the surface of chicken carcasses during slaughtering and processing with and without bacterial reduction
图5为减菌前后肉鸡屠宰生产链中胴体表面所取样品经DGGE分析后所分离纯化细菌的微生物系统发育树,不同环节的肉鸡胴体表面的微生物污染存在差异性。菌k和h的亲缘关系比较近,菌k是在胴体清洗环节和速冻环节出现的细菌,菌h在调查的6 个环节中均有出现;菌h和i、k是也是同一属的菌群,均是假单胞菌属,i在胴体清洗和分割环节数量较多。
PCR-DGGE技术可检测出肉鸡屠宰生产链关键环节中的肉鸡胴体表面细菌菌相的多样性,DGGE图谱显示,采用50 ℃、1.5%乳酸溶液在预冷环节前对肉鸡胴体冲淋15 s进行减菌处理,减菌后在对应肉鸡加工环节大多数条带的亮度有所减弱,且条带数目也有所减少,由此可知,肉鸡胴体表面的细菌种类及数量较减菌前均有所减少。预冷环节后,细菌的种类与数量是整个肉鸡屠宰加工环节最低的,说明预冷环节能有效减少细菌的种类和数量,但也应当及时更新预冷池中的水,防止交叉污染。经预冷后,肉鸡进入分割车间,细菌的种类和数量均增加,引起细菌菌相发生变化,在实际生产过程中采取适当的清洗、消毒手段可有效控制交叉污染造成的微生物污染。
本实验中DGGE图谱显示不可培养梭菌(uncultured Clostridiales bacterium)、瘤胃球菌属(Ruminococcus)在除预冷环节外均有发现。这与Gong Jianhua等 [15]研究结果相符,其发现Clostridiales和Ruminococcus在肉鸡盲肠中分别占所有菌群的40%和6%。在肉鸡胴体表面被检测出的原因可能是由于盲肠在掏膛环节被弄破,导致肠道菌群在胴体清洗时被冲洗到肉鸡胴体表面,但在处理后条带亮度明显减弱,且处理后在速冻环节这2 条带几乎消失,说明减菌处理后,这2 种菌的数量明显减少。此外,乳杆菌属、肠杆菌科与假单胞菌属细菌是肉鸡屠宰加工环节中的优势腐败菌。
传统的培养法是应用选择性培养基 筛选特定的某一类微生物,根据菌数来判断优势菌的变化情况 [16-18],工作量大而又繁琐,且鉴定的微生物种类十分有限。PCRDGGE技术也存在固有的局限性 [19],但可以通过测序分析鉴定微生物的种类,包括部分不可培养细菌,对微生物的多样性以及变化情况认识更加清晰、准确。孙彦雨等 [20]为避开传统培养法与PCR-DGGE技术的局限性,采用二者相结合的方法,确定乳酸菌、大肠菌群、肉杆菌、腐败希瓦氏菌以及热杀索丝菌为冰鲜鸡肉中腐败优势菌,该结果与梁荣蓉 [21]以及本研究结果存在一定的差异,这可能是由于不同肉鸡屠宰加工企业生产标准的不同与管理水平的高低造成的。
我国GB 2760—2011《食品添加剂使用标准》中食品工业用加工助剂使用规定,乳酸可作为各类食品加工助剂使用且残留量不需限定。美国食品药物管理局现已批准1.5%~2.5%有机酸如乳酸、柠檬酸等可用于禽类加工企业中 [22]。1992年,美国农业部允许剂量为8%~12%抗菌剂磷酸三钠以喷涂或浸渍的方式用于健康无病的生冷禽类胴体,浸渍时间不超过15 s。Bourassa等 [23]研究也证实,经100 g/L磷酸三钠溶液处理的肉鸡经过45 min预冷后,其沙门氏菌减少率为40%。孙京新等 [24]用不同质量分数的乳酸、不同温度的热水和以不同的喷淋时间研究对猪肉肉色以及去微生物污染的影响,结果发现,乳酸质量分数1.5%、温度45 ℃、时间60 s和乳酸质量分数2.0%、温度35 ℃、时间60 s时对猪肉肉色影响不显著,但对大肠杆菌和沙门氏菌的致死效果均显著。其结果与彭珍等 [11]实验结果相近但又有所差异,可能是由于处理的样品对象不同。国内外学者研究结果普遍认为,多栅栏因子减菌工艺在畜禽屠宰加工生产线上应用时,会显著地减少微生物种类和数量 [25-26]。本研究在肉鸡屠宰加工中预冷环节前使用乳酸结合热水进行减菌处理,采用PCR-DGGE技术分析其减菌处理前后细菌菌相变化规律,较为可靠、清晰地认识了该企业肉鸡屠宰加工过程中细菌菌相的分布、变化情况,同时直观地反映了该减菌方法的减菌效果,为实际生产中微生物污染的防控提供了一定的理论依据。
参考文献:
[1] JSMES C, VINCENT C. The primary chilling of poultry carcasses a review[J]. International Journal of Refrigeration, 2006, 29(6): 847-862.
[2] KOTULA K L, PANDYA Y. Bacterial contamination of broiler chickens before scalding[J]. Journal of Food Protection, 1995, 58(12): 1326-1329.
[3] GEORNARAS I, von HOLY A. Bacterial counts associated with poultry processing at different sampling times[J]. Journal of Basic Microbiology, 2000, 40(5): 343-349.
[4] BLIXT Y, BORCH E. Comparison of shelf life of vacuum packed pork and beef[J]. Meat Science, 2002, 60(4): 371-378.
[5] BORCH E, KANT-MUEMANSH M L, BLIXT Y. Bacterial spoilage of meat products and cured meat[J]. International Journal of Food Microbiology, 1996, 33(1): 103-120.
[6] AMPE F, ben OMAR N, MOIZAN C, et al. Polyphasic study of the spatial distribution of microorganisms in mexican pozol, a fermented maize dough, demonstrates the need for cultivation-independent methods to investigate traditional fermentations[J]. Applied and Environmental Microbiology, 1999, 65(12): 5464-5473.
[7] JAY J M, VLLAI J P, HUGHES M E. Profi le and activity of the bacterial biota of ground beef held from freshness to spoilage at 5-7 ℃[J]. International Journal of Food Microbiology, 2003, 81(2): 105 -111.
[8] GILL C O. Extending the storage life of raw[J]. Meat Science, 1996, 43(1): 99-109.
[9] MUYZER G, WEAL E C, UITTER A. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain raction amplified genes coding for 16S rRNA[J]. Applied and Environmental Microbiology, 1993, 59(3): 695-700.
[10] THEELEN B, SILVESTRI M, GUCHO E, et al. Identification and typing of Malassezia yeasts using amplified fragment length polymorphisms (APLP) , random amplifi ed polymorphic DNA (RAPH) and denaturing gradient gel electrophoresis (DGGE)[J]. FEMS Yeast Research, 2001, 2(1): 79-86.
[11] 彭珍, 刘书亮, 朱冬梅, 等. 肉鸡屠宰加工过程中胴体微生物污染分析及不同冲淋条件对胴体减菌的影响[J]. 食品与发酵工业, 2014, 40(3): 216-221.
[12] GILL C O, MOZA L F, BADONI M, et al. The effects on the microbiological condition of product of carcass dressing, cooling and portioning processes at a poultry packing plant[J]. International Journal of Food Microbiology, 2006, 110(2): 187-193.
[13] NUBEL U, ENGELEN B. Sequence heterogeneities of genes encoding 16S rRNAs in Paenibacillus polymyxa detected by temperature gradient gel electrophoresis[J]. Journal of Bacteriology, 1996, 178(19): 5636-5643.
[14] 刘炜, 马晓军, 侯书贵, 等. 东天山地区庙儿沟雪坑中微生物多样性、群落结构与环境关系研究[J]. 微生物学报, 2007, 47(6): 1019-1026.
[15] GONG Jianhua, SI Weiduo, FORESTER R J, et al. 16S rRNA genebased analysis of mucosa-associated bacterial community and phylogeny in the chicken gastrointestinal tracts: from crops to ceca[J]. FEMS Microbiology Ecology, 2007, 59(1): 147-157.
[16] SOFOS N, SMITH G C. Nonacid meat decontamination technologies: model studies and commercial applications[J]. International Journal of Food Microbiology, 1998, 44(3): 171-188.
[17] BLIXT Y, BORCH E. Comparison of shelf life of vacuum-packed pork and beef[J]. Meat Science, 2002, 60(4): 371-378.
[18] BACON R T, BEIKE K E, SOFOS J N, et al. Microbial populations on animal hides and beef carcasses at different stages of slaughter in plants employing multiple-sequential interventions for decontanmination[J]. Journal of Food Protection, 2000, 63(8): 1080-1086.
[19] 邢德峰, 任南琪, 宋佳秀, 等. 不同16S rDNA靶序列对DGGE分析活性污泥群落的影响[J]. 环境科学, 2006, 27(7): 1424-1428.
[20] 孙彦雨, 周光宏, 徐幸莲. 冰鲜鸡肉贮藏过程中微生物菌相变化分析[J]. 食品科学, 2011, 32(11): 146-151.
[21] 梁荣蓉. 生鲜鸡肉调理制品菌群结构分析和货架期预测模型的研究[D]. 泰安: 山东农业大学, 2010: 40-56.
[22] DINCER A H, BAYSAL T. Decontamination techniques of pathogen bacteria in meat and poultry[J]. Critical Reviews in Microbiology, 2004, 30(3): 197-204.
[23] BOURASSA D V, FLETCHER D L, BUHRR J, et al. Recovery of Salmonellae from trisodium phosphate-treated commercially processed broiler carcasses after chilling and after seven-day storage[J]. Poultry Science, 2004, 83(12): 2079-2082.
[24] 孙京新, 周光宏, 罗欣, 等. 不同工艺参数乳酸处理对猪肉肉色及去微生物污染的影响[J]. 食品科学, 2007, 28(11): 276-278.
[25] BACON R T, BELK K E, SOFOS J N, et al. Microbial populations on animal hides and beef carcasses at different stages of slaughter in plants employing multiple-sequential interventions for decontamination[J]. Journal of Food Protection, 2000, 63(8): 1080-1686.
[26] ELDER R O, KEEN J E, SIRAGUSA G R, et al. Correlation of enterohemorrhagic Escherichia coli O157 prevalence in feces, hides, and carcasses of beef cattle during processing[J]. Proceedings of the National Academy of Sciences of the United States of America, 2000, 97(7): 2999-3003.
Bacterial Flora before and after Bacterial Reduction during Slaughtering and Processing Broiler Chickens
XIA Xiaolong
1, PENG Zhen
1, LIU Shuliang
1,2,*, HAN Xinfeng
1,2, ZHOU Kang
1,2, ZOU Likou
3, LAI Haimei
1
(1. College of Food Science, Sichuan Agricultural University, Ya’an 625014, China; 2. Key Laboratory of Agricultural Products Processing and Preservation Engineering of Sichuan Province, Ya’an 625014, China; 3. Laboratory of Microbiology, Dujiangyan Campus of Sichuan Agricultural University, Dujiangyan 611830, China)
Abstract:The bacterial diversity of carcasses or products of broiler chickens before and after bacterial reduction during slaughter and processing was analyzed by polymerase chain reaction (PCR) amplification and denaturing gradient gel electrophoresis (DGGE) based on the V 6–V 8variable regions of 16S rDNA. Broiler carcasses were rinsed with 1.5% lactic acid solution at 50 ℃ for 15 s for reducing bacteria before the pre-cooling treatment. Bacterial total DNA was extracted from the samples collected from the surface of carcasses or carcass parts before and after bacterial reduction during chicken slaughter and processing and the V 6–V 8regions of 16S rDNA were amplifi ed by PCR using a universal primer. The bacterial community structure was analyzed by DGGE. The results showed that the largest number of bands with the highest brightness in the DGGE profile was observed during carcass cleaning before bacterial reduction, indicating the most serious bacterial contamination, followed in turn by carcass segmentation and pre-cooling where the lowest bacterial count and the smallest number of bacterial species were obtained suggesting minimum bacterial contamination. After bacterial bacteria, both the total bacterial count and the number of bacterial species were reduced during slaughte ring and processing, as compared with those observed before bacterial bacteria. The largest reduction in the two parameters was found during both carcass cleaning and segmentation, and the smallest reduction during the pre-cooling sta ge. The bacterial species were not entirely consistent during the slaughter and processing of broilers, and Lactobacillus was found throughout the entir e process as the predominant spoilage bacteria together with Pseudomonas sp. and Enterobacteriaceae.
Key words:PCR-DGGE; chickens; slaught e r; bacterial reduction; bacterial microfl ora
中图分类号:TS251.8
文献标志码:A
文章编号:
doi:10.7506/spkx1002-6630-201510038
收稿日期:2014-06-09
基金项目:公益性行业(农业)科研专项(200903055)
作者简介:夏小龙(1989—),男,硕士研究生,研究方向为食品微生物。E-mail:xiaolongxia1205@163.com
*通信作者:刘书亮(1968—),男,教授,博士,研究方向为食品微生物与发酵工程。E-mail:lsliang999@163.com