羊羔美酒大曲中乳酸菌多样性及分子鉴定
李 艳
1,2,董振玲
1,3,李 佳
1,牟德华
1,*
(1.河北科技大学生物科学与工程学院,河北 石家庄 050018;2.河北省发酵工程技术研究中心,河北 石家庄 050018;3.河北一然生物科技有限公司,河北 石家庄 050800)
摘 要:目的:分离和鉴定羊羔美酒大曲中的乳酸菌,探寻其菌群多样性,为酿酒工艺条件改进提供参考。方法:将酒曲进行梯度稀释、富集培养、平板画线等分离纯化,获取乳酸菌单菌落。采用菌落特征和个体形态特征结合的方法进行乳酸菌形态鉴定。乳酸菌的分子鉴定采用重复序列聚合酶链式反应(repetitive sequence-based polymerase chain reaction,Rep-PCR)技术和16S rDNA序列分析法。结果:从羊羔美酒大曲中共得到65 株乳酸菌,形态学分为9 类。用Rep-PCR技术在75%的相似性上将其区分为5 类,经基因序列分析,鉴定为分属于4 个属的乳酸菌,分别为:片球菌属(Pediococcus)、明串珠菌属(Leuconostoc)、乳杆菌属(Lactobacillus)、肠球菌属(Enterococcus)。结论:传统微生物分离技术与现代分子鉴定技术相结合,可快速准确鉴定出羊羔美酒大曲中乳酸菌的菌群组成和多样性。
关键词:羊羔美酒大曲;乳酸菌;分子鉴定;重复序列聚合酶链式反应;16S rDNA序列分析
乳酸菌指利用可发酵性糖产生乳酸的一类革兰氏阳性细菌的通称。在分类学上,乳酸菌这个名称是非正式、非规范的
[1]。乳酸菌与中国传统酿造业息息相关。黄酒发酵属糖化和发酵并行的过程,以多品种、高密度酵母菌和乳酸菌协同发酵产生酒精、乳酸和多种风味物质
[2]。乳酸菌对酿酒过程及最终产品的风味形成有突出贡献,发酵初期产生乳酸,使发酵环境的pH值下降,可抑制有害菌繁殖;发酵中后期产生的乳酸可与酵母代谢产生的乙醇作用,形成乳酸乙酯以增强酒的浓醇感和酒香气的柔和性
[3]。酒曲是发酵的动力,酒曲微生物菌群的组成是酿酒的源泉,鉴定酒曲中的乳酸菌对酒曲应用和酿酒工艺改进极有意义。
目前,越来越多的分子生物学方法,如扩增片段长度多态技术、变性梯度凝胶电泳/温度梯度凝胶电泳、16S~23S rDNA间区、16S rDNA序列分析、重复序列聚合酶链式反应(repetitive sequence-based polymerase chain reaction,Rep-PCR)技术等
[4-10]已应用于乳酸菌的分类鉴定。其中Rep-PCR是采用特定引物对细菌基因组DNA的重复序列进行扩增,经电泳技术分离后分析其多态性。该技术可以在亚种和菌株水平上对菌种进行快速而准确的分类和鉴定,并且有操作简便、重复性好、分辨率高等优势。
羊羔美酒是一种北方特色肉酿型黄酒,河北省名酒,利用大曲为糖化剂、发酵剂和生香剂
[11]。本实验检测羊羔酒大曲中的乳酸菌,可以探明羊羔美酒的发酵驱动力,从一个侧面解密酒的风味特征,对进一步优化酿酒工艺,筛选有益于酒类酿造和形成特色风味的乳酸菌奠定基础。
1 材料与方法
1.1 材料与培养基
羊羔美酒大曲由河北味道府酒业有限责任公司提供。
MRS固体培养基
[12](g/L):蛋白胨10.0、牛肉膏10.0、酵母浸粉5.0、柠檬酸氢二铵2.0、葡萄糖20.0、乙酸钠2.0、K
2HPO
42.0、MgSO
40.58、MnSO
40.25、琼脂18.0、CaCO
310,吐温-80 1.0 mL蒸馏水配制,pH 6.0,121 ℃灭菌20 min。
MRS液体培养基:MRS固体培养基中不添加碳酸钙、中性红和琼脂。
1.2 试剂与仪器
引物:27F:5'-AGA GTT TGA TCC TGG CTC AG-3',1495R:5'-CTA CGG CTA CCT TGT TAC GA-3',(GTG)5:5'-GTG GTG GTG GTG GTG-3',BOXAIR:5'-CTA CGG CAA GGC GAC GCT GACG-3';10×PCR Buffer、dNTP、rTaq酶均购自生工生物工程(上海)股份有限公司。
PCR仪、凝胶成像分析仪 美国Bio-Rad公司;琼脂糖水平板电泳仪 北京六一公司。
1.3 方法
1.3.1 酒曲中乳酸菌的分离
样品处理:取成品羊羔美酒大曲,分别用无菌刀在酒曲外表面、中间和内部取样各1.0 g,混合研磨成粉。取1 g加入100 mL无菌水,置于摇床上150 r/min摇30 min,静止,取其上清液进行系列10 倍梯度稀释
[11-13]。选适宜稀释梯度的稀释液0.2 mL倾注法接种于3 个平行MRS培养基中,37 ℃恒温培养48 h。对在MRS培养基上可以产生透明圈并长势良好的菌落进行计数。再选取产生透明圈及在厌氧条件生长的菌落接种于液体MRS培养基(含20%甘油),-20 ℃条件下保藏。
乳酸菌的分离:分别选用大米、糯米、黍米为酒曲活化基质,进行大曲微生物的富集,30 ℃培养5 d。取培养基质1 g,加100 mL无菌水,打浆,取5 mL浆液进行系列10 倍梯度稀释,选3 个合适的稀释度0.2 mL进行3 个平行平板的涂布分离培养。
1.3.2 乳酸菌的初步鉴定和形态聚类
将保存的菌株进行活化,接种于琼脂培养基,划线分离,上层倾注MRS培养基,37 ℃培养48 h,挑取单菌落连续纯化2 次。将已纯化的单菌株进行革兰氏染色和过氧化氢实验,将革兰氏阳性且过氧化氢实验阴性的菌确定为乳酸菌
[14]。将已确定为乳酸菌的菌株采用平板划线法接种于MRS培养基,37 ℃培养5 d后根据其菌落形态及个体形态进行分类。
1.3.3 乳酸菌的分子鉴定
1.3.3.1 基因组DNA的提取
实验菌株接种于MRS液体培养基中,37 ℃培养48 h,获得足够量的菌体。取1.5 mL培养液12 000 r/min离心10 min,弃去上清液。加20 mg/mL的溶菌酶50 μL,37 ℃处理1 h。向每管加入200 μL裂解缓冲液(4 mmol/L Tris-HCl,pH 8.0,20 mmol/L乙酸钠,1 mmol/L乙二胺四乙酸(ethylene diamine tetraacetic acid,EDTA),1%十二烷基硫酸钠(sodium dodecyl sulfate,SDS)),混合均匀。加入10 μL蛋白酶K(10 mg/mL),55 ℃处理30 min,向每管加入66 μL饱和NaCl溶液,充分混合后,12 000 r/min离心10 min,除去蛋白质复合物及细胞壁等残渣。取离心后上清液,加入等体积的Tris饱和酚,14 000 r/min离心5 min,取上清液,用等体积的氯仿异戊醇溶液抽提2 次,取上清液,用预冷1 倍体积的异丙醇沉淀DNA,15 000 r/min离心10 min,弃去上清液。用400 μL 70%的乙醇洗涤沉淀两次。室温干燥后,用50 μL无菌双蒸水溶解DNA
[15-17]。检测DNA的纯度与浓度,将DNA稀释至质量浓度20~50 ng/μL,-20 ℃保存备用
[18]。
1.3.3.2 Rep-PCR分析
(GTG)5 PCR反应体系:10×PCR Buffer 2.5 μL,MgCl
2(25 mmol/L)3 μL,(GTG)5(0.4 μmol/L)1 μL,模板DNA(20~50 ng)1 μL,Taq酶(0.5 U/L)0.2 μL,加灭菌双蒸水补足 25 μL。PCR反应程序:预变性95 ℃ 7 min,94 ℃ 1 min,40 ℃ 1 min,65 ℃ 8 min,35 个循环,最后65 ℃ 20 min
[19-21]。
BOX AIR PCR反应体系:10×PCR Buffer 2.5 μL,MgCl
2(25 mmol/L)3 μL,BOX AIR(0.4 μmol/L)1 μL,模板DNA(20~50 ng)1 μL,Taq酶(0.5 U/L)0.5 μL,加灭菌双蒸水补足25 μL。PCR反应程序:95 ℃预变性7 min;94 ℃ 1 min,53 ℃ 1 min,65 ℃ 8 min,35 个循环;最后65 ℃ 16 min
[22-25]。
PCR产物的检测:吸取8 μL PCR产物,通过1.5%琼脂糖凝胶电泳检测,90 V电压140 min,经过EB染色15 min后,利用凝胶成像系统成像
[26]。
1.3.3.3 16S rDNA序列分析
扩增乳酸菌16S rDNA使用细菌通用引物27F与1495R。PCR反应体系:Buffer 10 μL,Taq酶0.4 μL,dNTP 1 μL,引物2 μL,DNA 2 μL,双蒸水32.6 μL。PCR扩增条件:94 ℃预变性5 min;94 ℃变性1 min,58 ℃退火1 min,72 ℃延伸2 min,30 个循环;72 ℃末端延伸10 min。将PCR产物利用2%琼脂糖凝胶电泳检测片段长度
[27]。
将样品菌株的16S rDNA PCR产物送至生工生物工程(上海)股份有限公司,委托其进行序列测定。
2 结果与分析
2.1 乳酸菌的分离与形态聚类
表1 MRS培养基上(培养5 d)乳酸菌落的形态描述
Table1 Morphological characteristics of lactic acid bacteria on MRS medium (incubation for 5 days)

形态菌落形态显微形态个体形态细胞大小(长×宽)或(直径)/μm 1杆菌白色,湿润圆形,扁平,边缘不整齐,无光泽杆菌,单生或成对3.11×0.97 2乳黄,湿润,圆形,山丘突起,边缘水环状延伸,有光泽杆菌,单生或成链2.32×0.69 3乳白,湿润,圆形,山状突起,边缘水环状延伸,有光泽杆菌,单生或成链1.59×0.57 4乳白,湿润,扁平,边缘不整齐,有光泽,略隆起细胞圆形球菌,菌体多成对生,偶有单生1.04 5白色,湿润,圆形,球状凸起,边缘整齐,有光泽细胞圆形球菌,菌体多成对生,或四联生1.17 6白色,湿润,圆形,棋子状,边缘整齐,有光泽细胞圆形球菌,菌体成对生1.30 7白色,湿润,以棋子状为主,形状变化较大,边缘不整齐细胞圆形球菌,菌体成对或链排列0.91 8乳白色,干燥,圆形,扁平,中心略凸起,边缘整齐细胞圆形球菌,菌体成对或链排列1.00 9菌落乳白色,干燥,圆形,扁平,中心略凸起,边缘整齐 细胞圆形球菌,菌体成对或链排列0.95球菌
从羊羔美酒大曲中共分离得到168 株可以产生透明圈的单菌株,对单菌株进行革兰氏染色和过氧化氢实验后,将65 株过氧化氢实验显示阴性且革兰氏阳性的菌株初步确定为乳酸菌,其中杆菌18 株(27.7%),球菌47 株(72.3%)。根据乳酸菌在MRS培养基上菌落形态和显微镜下细胞形态的不同进行聚类,可将杆菌分为3 类,其中形态2数量最多,共12 株,占乳杆菌数量的66.7%;球菌分为6 类,其中形态4与形态9数量较多,各有13 株和12 株,分别占乳球菌总数量的27.7%与25.5%,形态聚类结果见表1。因此,在羊羔美酒大曲中,优势乳酸菌群为形态2的乳杆菌和形态4与9的乳球菌。
2.2 Rep-PCR技术鉴定乳酸菌
为了选择适合大曲中乳酸菌多样性研究的引物,本研究选取了2 种在乳酸菌Rep-PCR技术报道中使用较多的引 物(GTG)5与BOXAIR。用这两种引物对9 种形态的乳酸菌代表菌株进行基因扩增。由图1可知,用(GTG)5和BOXAIR 分别作为引物的 PCR 产物电泳出现多条带谱,其分子质量大小范围在0.25~0.5 kb 之间,菌株之间的差异能够明显地被区分开来。从(GTG)5扩增结果可知,各菌株均有良好的多态性,扩增条带数目均在9~15 条;而BOXA1R引物扩增结果并不是很理想,泳道5~10的条带均少于8 条,图谱多态性并不理想。因此,(GTG)5在这些乳酸菌基因组中的分布比BOXA1R更为多样,且两者的结果对菌株的分群有差异,暗示这两种短重复序列在基因中的分布也不同
[28]。
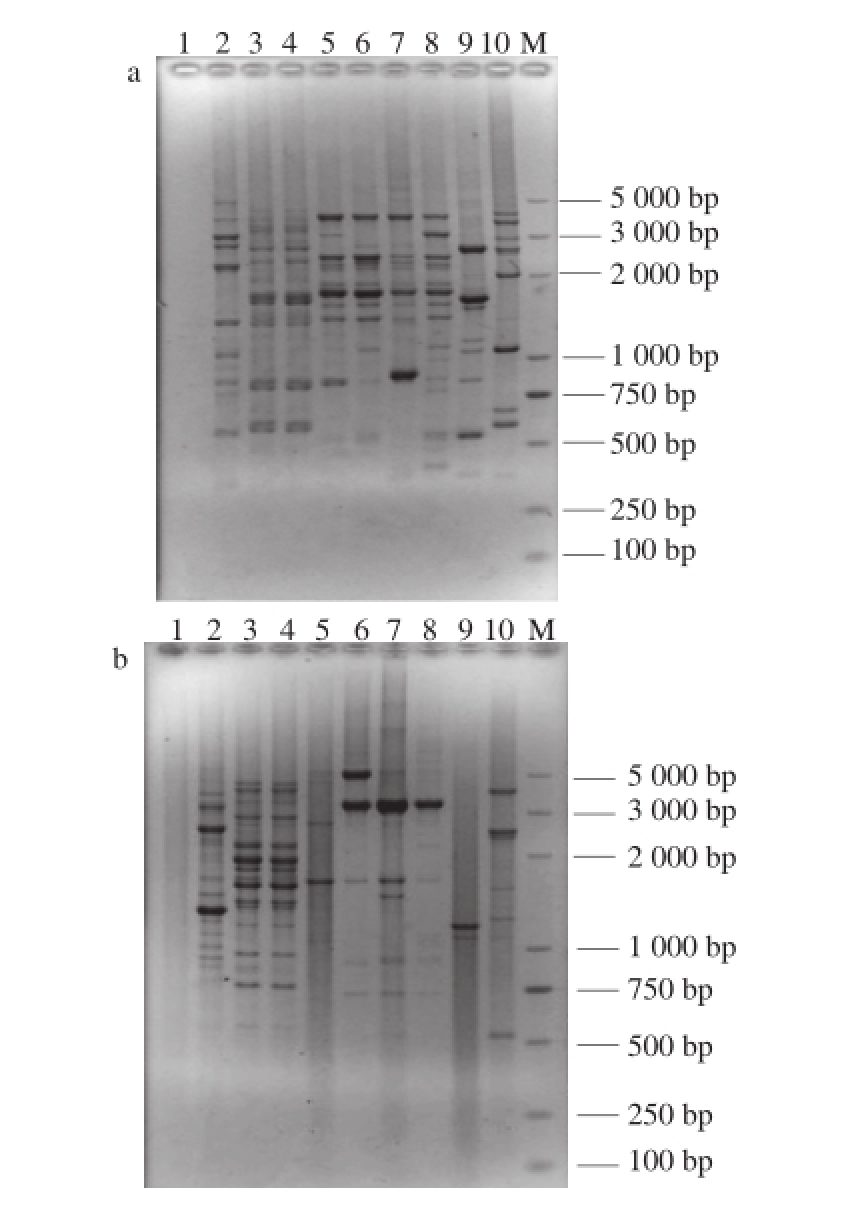
图1 ( GTG)5(a)和BOXA1R(b)扩增条带的比较
Fig.1 Comparison of fingerprints obtained with (GTG) 5 (a) and BOXA1R (b)
M. Marker;泳道1.空白对照;泳道2~10依次为1~9号菌。
以(GTG)5扩增图谱进行多态性分析,使用NTSYSpc2.0软件进行数据处理,采用平均连锁法(unweighted pair-group method with arithmetic means,UPGMA)绘出聚类树状图,并进行菌株的分群,结果见图2。

图2 采用平均连锁法构建的Rep-PCR结果树状图
Fig.2 UPGMA dendrogram generated from Rep-PCR fingerprints
由图2可知,在75%相似性上进行分群,(GTG)5可以将9 种不同类型的乳酸菌区分为5 个群,其中群Ⅱ是最大的一个群,包含了形态4、5、6、7共4 种形态;群Ⅲ包含形态2、3两种形态;其余形态1、形态8、形态9分别属于群Ⅰ、Ⅳ和Ⅴ。使用Rep-PCR技术可以快速地将不同菌株在基因型上进行聚类分析。
2.3 16S rDNA序列分析法鉴定乳酸菌
为了进一步验证Rep-PCR技术的可靠性,将实验菌株进行16S rDNA测序,将所得序列结果登陆GenBank数据库,进行BLAST(http: //www. ncbi. nlm. nih. gov/blast/ Blast. cgi)相似性比对,以相似度大于99%确定其属种,测序结果见表2。
表2 乳酸菌16S rDNA序列分析结果
Table2 16S rDNA sequence analysis of lactic acid bacteria

形态类型菌株编号乳酸菌属、种相似度/%GenBank登录号对照菌株登录号1KDLL1-1 Lactobacillus plantarum subsp.100KC454277AB572042.1 2KDLL2-1Lactobacillus fermentum99KC454278HM218410.1 3KDLL2-2Lactobacillus fermentum99KC454279HM035543.1 4KDLL3-1Pediococcus sp.100KC454280JN798840.1 5KDLL3-2Pediococcus pentosaceus99KC454281AB681648.1 6KDLL3-3Pediococcus pentosaceus99KC454282KJ610832.1 7KDLL3-4Pediococcus pentosaceus99KC454283AB681648.1 8KDLL4-1Enterococcus faecalis100KC454284JF784028.1 9KDLL5-1Leuconostoc fallax100KC454285JF690866.1
在GenBank核酸序列数据库中下载相关菌株16S rDNA序列,与已测序菌株的序列放在一起,使用Clustal X软件进行序列比对;利用MEGA4生物学软件的Neighbor-Joining连接法构建系统发育树,并进行1 000 次Bootstrap检验。构建的系统发育树如图3所示。
结合图2与图3进行分析可知,Rep-PCR结果树状图与16S rDNA序列系统发育树有较好的相关性。因此,(GTG)5-PCR指纹分析技术能够反映出不同菌株的基因组间存在的差异,可以作为鉴别菌种的手段之一
[26-28]。针对羊羔美酒大曲中大量乳酸菌的基因型差异性,在后续研究中,可以建立相关Rep-PCR指纹图谱数据库,结合16S rDNA序列测序技术,便可实时了解羊羔美酒发酵过程中优势乳酸菌的演变规律,为发酵工艺优化与控制奠定基础。本实验为羊羔美酒大曲中乳酸菌的Rep-PCR指纹图谱数据库建立奠定了基础。
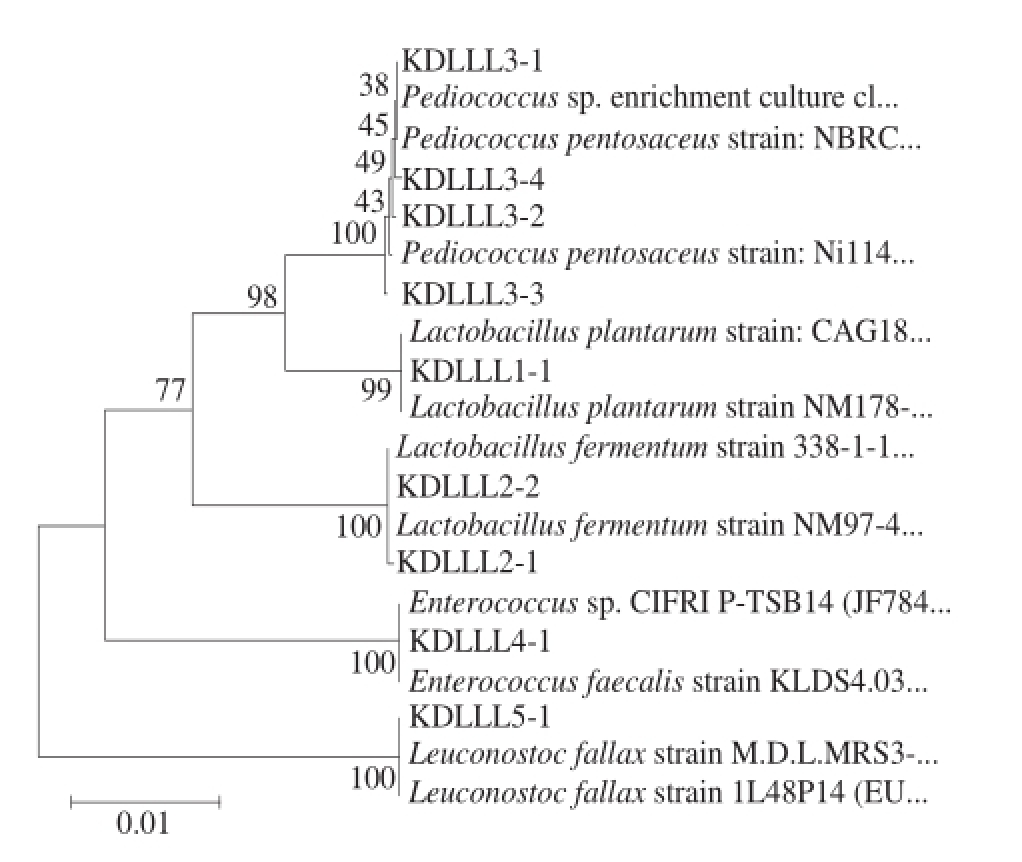
图3 基于16S rDNA序列和Neighbor-Joining法构建的系统发育树
Fig.3 Phylogenetic tree based on 5.8S-ITS region sequences and Neighbor-Jointing method
另外,从以上分子鉴定结果可以看出,羊羔美酒大曲中乳酸菌种类非常丰富,存在着乳杆菌属、片球菌属、明串珠菌属和肠球菌属,其中片球菌属和乳杆菌属所占比重较大。片球菌属、肠球菌属和明串珠菌属都有糖化作用,也可分解蛋白质、产生乳酸、降低pH值。羊羔美酒大曲中丰富的球菌对保证其酿造过程顺利进行起着至关重要的作用。乳杆菌在羊羔酒发酵过程中产生乳酸的同时也会产生不同类型的肽聚糖,而肽聚糖可以成为羊羔酒的风味成分之一。另外,乳酸杆菌也可以利用霉菌等微生物代谢生成的朊、胨和多肽类物质分解成氨基酸,推测这些乳酸杆菌与羊羔美酒独特的香气和富含的22 种氨基酸有着密切联系。实验为后续研究各种乳酸菌单菌株特性、优势菌株的分析、及其在羊羔美酒发酵过程中的作用机制奠定了基础,进而为改进酿酒工艺条件提供参考。
3 结 论
本实验从羊羔美酒大曲中共分离乳酸菌65 株,形态区分为9 类,使用Rep-PCR技术将其区分为5 类,经16S rDNA基因序列分析,将其鉴定为分属于片球菌属、明串珠菌属、乳杆菌属、肠球菌属4 个属的乳酸菌。研究结果反映了羊羔美酒大曲中乳酸菌的多样性,为优化这一我国北方特色地方名酒的酿造工艺、改进传统的配料和研究产品的风味物质特征奠定了基础。
参考文献:
[1] 凌代文, 东秀珠. 乳酸菌的分类鉴定及实验方法[M]. 北京: 中国轻工业出版社, 1999: 1-3.
[2] 毛青钟. 论黄酒发酵过程酵母和乳酸杆菌协同作用关系[J]. 山东食品发酵, 2006(1): 29-31.
[3] 汪建国. 浅谈乳酸菌在黄酒生产中的作用[J]. 生产与技术, 2009,26(3): 37-42.
[4] 剧柠, 靳烨. DNA分子标记技术在乳酸菌多态性研究中的应用[J].微生物学通报, 2008, 35(8): 1297-1301.
[5] 李海星, 曹郁生, 付琳琳. 用于乳酸菌鉴定的几种分子生物学方法[J].江西科学, 2004, 22(5): 393-398.
[6] RANTSIOU K, COMI G, COCOLIN L. The rpoB gene as a target for PCR-DGGE analysis to follow lactic acid bacterial population dynamics during food fermentations[J]. Food Microbiology, 2004,21(4): 481-487.
[7] MUYZER G, de WAAL E C, UITTERLINDEN A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction amplified genes coding for 16S rRNA[J]. Applied and Environmental Microbiology,1993, 59(3): 695-700.
[8] TILSALA-TIMISJ☒RVI A, ALATOSSAVA T. Development of oligonucleotide primers from the 16S-23S rRNA intergenic sequences for identifying different dairy and probiotic lactic acid bacteria by PCR[J]. International Journal of Food Microbiology, 1997, 35(1): 49-56.
[9] BUSSE H J, DENNER E B M, LUBITZ W. Classification and identification of bacteria: current approaches to an old problem. Overview of methods used in bacterial systematics[J]. Journal of Biotechnology, 1996, 47(1): 3-38.
[10] TEMMERMAN R, HUYS G, SWINGS J. Identi☒cation of lactic acid bacteria: culture-dependent and culture-independent methods[J]. Trends in Food Science & Technology, 2004, 15(7/8): 348-359.
[11] 李艳, 董振玲, 牟德华. 羊羔美酒大曲中酵母菌多样性及分子鉴定[J]. 食品科学, 2014, 35(5): 144-149. doi: 10.7506/spkx1002-6630-201405029.
[12] GEVERSA D, HUYS G, SWINGS J. Applicability of rep-PCR fingerprinting for identification of Lactobacillus species[J]. FEMS Microbiology Letters, 2001, 205: 31-36.
[13] SCHEIRLINCK I, van der MEULEN R, van SCHOOR A, et al. In☒uence of geographical origin and flour type on diversity of lactic acid bacteria in traditional belgian sourdoughs[J]. Applied and Environmental Microbiology, 2007, 73(19): 6262-6269.
[14] CAMU N, WINTER T D, VERBRUGGHE K, et al. Dynamics and biodiversity of populations of lactic acid bacteria and acetic acid bacteria involved in spontaneous heap fermentation of cocoa beans in ghana[J]. Applied and Environmental Microbiology, 2007, 73(6):1809-1824.
[15] LEFEBER T, GOBERT W, VRANCKEN G, et al. Dynamics and species diversity of communities of lactic acid bacteria and acetic acid bacteria during spontaneous cocoa bean fermentation in vessels[J]. Food Microbiology, 2011, 28(3): 457-464.
[16] AUDENAERT K, D'HAENE K, MESSENS K, et al. Diversity of lactic acid bacteria from modi☒ed atmosphere packaged sliced cooked meat products at sell-by date assessed by PCR-denaturing gradient gel electrophoresis[J]. Food Microbiology, 2010, 27(1): 12-18.
[17] SINGH A K, RAMESH A. Evaluation of a facile method of template DNA preparation for PCR-based detection and typing of lactic acid bacteria[J]. Food Microbiology, 2009, 26: 504-513.
[18] 于洁, 孙志宏, 张家超, 等. 16S rDNA-RFLP技术鉴定西藏地区乳制品中的乳杆菌[J]. 食品与生物技术学报, 2009, 28(6): 804-809.
[19] HOORDE K V, LEUVEN I V, DIRINCK P, et al. Selection,application and monitoring of Lactobacillus paracasei strains as adjunct cultures in the production of Gouda-type cheeses[J]. International Journal of Food Microbiology, 2010, 144(2): 226-235.
[20] KESMEN Z, YETIMAN A E, GULLUCE A, et al. Combination of culture-dependent and culture-independent molecular methods for the determination of lactic microbiota in sucuk[J]. International Journal of Food Microbiology, 2012, 153: 428-435.
[21] NIKOLIC M, TERZIC-VIDOJEVIC A, JOVCIC B, et al. Characterization of lactic acid bacteria isolated from Bukuljac, a homemade goat's milk cheese[J]. International Journal of Food Microbiology, 2008, 122: 162-170.
[22] MOHAMMED M, El-AZIZ H A, OMRAN N, et al. Rep-PCR characterization and biochemical selection of lactic acid bacteria isolated from the Delta area of Egypt[J]. International Journal of Food Microbiology, 2009, 128(3): 417-423.
[23] 满朝新, 迟涛, 胡博韬, 等. rep-PCR指纹图谱技术在乳酸菌鉴定中的应用[J]. 中国乳品工业, 2010, 38(5): 4-6.
[24] KIM W, HONG Y P, YOO J H, et al. Genetic relationships of Bacillus anthracis and closely related species based on variable-number tandem repeat analysis and BOX-PCR genomic fingerprinting[J]. FEMS Microbiology Letters, 2001, 207(1): 21-27.
[25] TRAN K T M, MAY B K, SMOOKER P M, et al. Distribution and genetic diversity of lactic acid bacteria from traditional fermented sausage[J]. Food Research International, 2011, 44(1): 338-344.
[26] 朱凤玲, 曲凌云, 洪旭光, 等. 两株海洋细菌新种的分离及BOX-PCR鉴定[J]. 海洋环境科学, 2011, 30(5): 622-625.
[27] 马燕, 倪永清, 卢士玲, 等. 新疆特色干酪中乳酸菌的分离鉴定[J].中国酿造, 2011, 30(8): 38-40.
[28] 李俊, 徐玲玫, 樊蕙, 等. 用rep-PCR技术研究中国花生根瘤菌的多样性[J]. 微生物学报, 1999, 39(4): 297-303.
Diversity and Molecular Biological Identification of Lactic Acid Bacteria from Yanggaomeijiu Daqu, a Traditional Chinese Liquor Fermentation Starter
LI Yan
1,2, DONG Zhenling
1,3, LI Jia
1, MOU Dehua
1,*
(1. College of Bioscience and Bioengineering, Hebei University of Science and Technology, Shijiazhuang 050018, China;2. R&D Center for Fermentation Engineering of Hebei Province, Shijiazhuang 050018, China;3. Hebei Inatural Biological Technology Co. Ltd., Shijiazhuang 050800, China)
Abstract:Purpose: The aim of this work was to isolate and identify lactic acid bacteria (LAB) in Yanggaomeijiu Daqu,a fermentation starter for the production of Chinese liquor Yanggaomeijiu, and to explore the diversity of LAB in Yanggaomeijiu Daqu. Methods: Individual bacterial colonies of LAB from Yanggaomeijiu Daqu were obtained through isolation and purification steps including gradient dilution, enrichment, cultivation, and plate streaking. Morphological identification of LAB isolates was performed based on colony and individual morphological characteristics and further molecular identification was carried out by repetitive sequence-based polymerase chain reaction (Rep-PCR) and 16S rDNA sequence analysis. Res ults: A total of 65 LAB isolates were obtained from Yanggaomeijiu Daqu, which could be assigned into 9 different groups by conventional microbiological anal ysis, differentiated into five types by Rep-PCR at a 75% similarity, and belonged to 4 genera including Pediococcus,Leuconostoc, Lactobacillus and Enterococcus by gene sequencing. Conclusion: The diversity and composition of LAB in Yanggaomeijiu Daqu can be identified quickly and accurately by the combination of traditional microbial separation methods and modern molecular biological identification techniques.
Key words:Yanggaomeijiu; lactic acid bacteria; molecular biological identification; repetitive sequence-based polymerase chain reaction (Rep-PCR); 16S rDNA sequence analysis
中图分类号:TS261.1
文献标志码:A
文章编号:1002-6630(2015)13-0167-05
doi:10.7506/spkx1002-6630-201513031
收稿日期:2014-09-19
基金项目:河北省石家庄市科技支撑计划课题(101171271A;10117901A);河北省自然科学基金项目(C2011208028)
作者简介:李艳(1958—),女,教授,本科,研究方向为饮料酒酿造、酿酒微生物。E-mail:lymdh5885@163.com
*通信作者:牟德华(1960—),男,教授,本科,研究方向为农产品加工。E-mail:dh_mou@163.com




