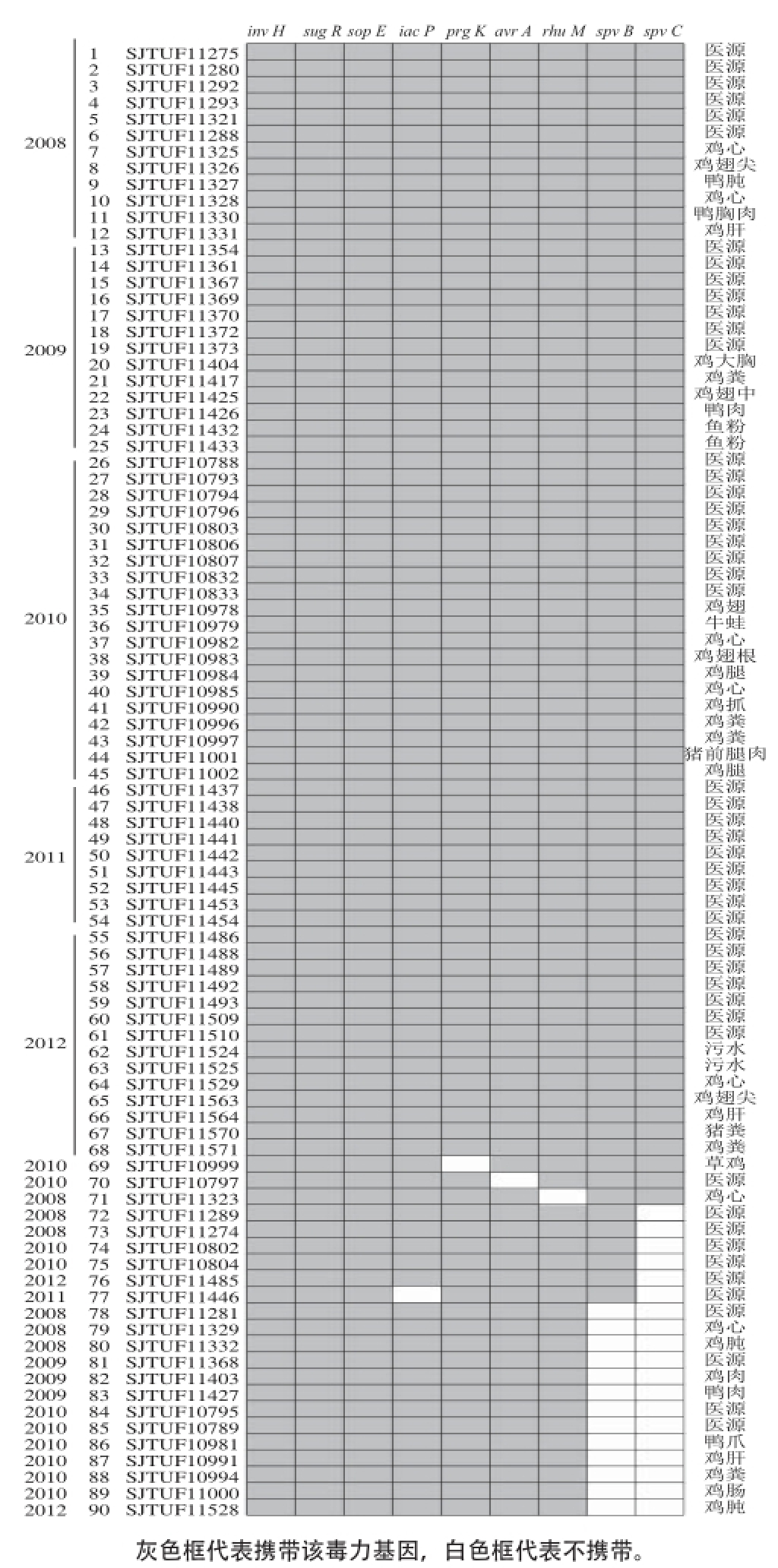
图1 2008—2012年上海地区90 株肠炎沙门氏菌9 种毒力基因分布图
Fig.1 Distribution of 9 virulence genes in 90 Salmonella Enteritidis isolates during 2008 to 2012 in Shanghai
庄孝飞 1,周秀娟 1,许学斌 2,史贤明 1,施春雷 1,*
(1.上海交通大学农业与生物学院,中美食品安全联合研究中心,陆伯勋食品安全研究中心,微生物代谢国家重点实验室,上海 200240;2.上海市疾病预防控制中心,上海 200336)
摘 要:采集上海地区2008—2012年来自临床病人和市售食品的肠炎沙门氏菌分离株90 株;采用聚合酶链式反应(polymerase chain reaction,PCR)方法,对存在于毒力岛SPI和毒力质粒上的9 种常见毒力基因携带情况进行了调查;并采用本实验优化的肠杆菌基因间保守重复序列-聚合酶链式反应(enterobacterial repetitive intergenic consensus-PCR,ERIC-PCR)方法对这些分离株进行亚分型。结果显示:毒力基因invH、sopE、sugR的携带率为100.0%(90/90),iacP、avrA、rhuM、prgK均为98.9%(89/90),而spvB、spvC分别为85.6%(77/90)和78.9%(71/90)。优化的ERIC-PCR体系能够较好地区分8 种主要沙门氏菌血清型,共17 株菌,辛普森指数(D值)为0.970 6。然而,90 株肠炎沙门氏菌分离株却具有一致的ERIC-PCR指纹图谱。在所调查分离株中,毒力基因的携带率较高,尤其是invH、sopE和sugR,对人类健康存在较大威胁;ERIC-PCR虽然可用于区分沙门氏菌不同血清型,但对肠炎沙门氏菌的分型效果不佳。
关键词:肠炎沙门氏菌;毒力基因;ERIC-PCR分型
沙门氏菌是引发食物中毒的主要食源性致病菌之一,目前已确认的血清型有2 600多种,其中肠炎沙门氏菌和鼠伤寒沙门氏菌是全世界范围内最常见的血清型,具有重要的流行病学意义 [1]。在我国,肠炎沙门氏菌SalmonellaEnteritidis成为近年来引发沙门氏菌病大爆发的主要血清型。根据文献报道,2006—2007年上海市非伤寒沙门菌病病例中肠炎沙门氏菌占据首位 [2],80%食源性分离株来自于禽及禽肉制品。同时,食物一旦污染沙门氏菌也会造成巨大经济损失,例如:1996年在我国向欧洲出口的兔肉中发现了肠炎沙门氏菌,经济损失达3 000万 元 [3]。
肠炎沙门氏菌具有很强的致病性,许多毒力因子(如脂多糖、肠毒素、细胞毒素以及毒力蛋白等)的存在与相互作用是导致其致病的罪魁祸首 [4-5],严重威胁人类健康。研究发现,毒力基因主要分布在毒力岛和毒性质粒上 [6],SPI-1~SPI-5是研究的比较多的5 个毒力岛。其中SPI-1全长约40 kb,至少编码29 个毒力基因,如inv、sic、iac、spt、iag、orf、prg、avr等,这些毒力基因编码的蛋白与肠炎沙门氏菌的侵袭力有关 [7]。SPI-3大小约为17 kb,可编码SugR、RhuM等多个毒力因子,与沙门氏菌在吞噬细胞内的存活相关 [8]。小鼠毒力试验亦证明,肠炎沙门氏菌毒力质粒上的毒力基因对引发疾病也具有重要作用 [9]。在毒力质粒上,与致病性相关的spv(Salmonella plasmid virulence)基因有6 个开放阅读框(spvA、spvB、spvC、spvD、orfE、spvR) [10]。不同来源的沙门氏菌对小鼠有不同的致病性,对人类的危害程度也不相同 [11],因此,研究不同来源肠炎沙门氏菌毒力基因携带情况对上海地区肠炎沙门氏菌风险预警和危害控制具有重要意义。
肠杆菌基因间保守重复序列(enterobacterial repetitive intergenic consensus,ERIC)是普遍存在于肠道细菌中,大小约为124~127 bp,在基因组中ERIC的分布和拷贝数具有种属特异性,因而可用于对基因组进行分析研究 [12]。Versalovic等 [13]针对这些序列设计引物进行PCR扩增,将这一技术用于细菌基因组DNA的指纹图谱分析,具有精确、快速、简便等优点,并被广泛地应用于菌种鉴别与病原菌的溯源研究中 [14-15]。Lim等 [16]通过研究随机扩增多态性DNA(random amplified polymorphic DNA, RAPD)、ERIC、单链构象多态性(single-strand conformation polymorphism,SSCP)、核糖体分型4 种分型方法发现,ERIC-PCR对沙门氏菌不同血清型有很好的分型效果。同样方法,陈玲等 [17]把96 株沙门氏菌分离株分为15 个簇,56 种基因型。而Chmielewski等 [18]利用ERIC-PCR将31 株禽源的肠炎沙门氏菌分成3 个基因群。借助分型可以对不同来源和不同血清型甚至不同致病力的沙门氏菌加以区分,对沙门氏菌的风险预警和危害控制同样具有重要意义。
本研究在上海不同地区,采集2008—2012年的食源性和医源性肠炎沙门氏菌分离株,从中随机挑选出90 株用于后续实验。通过对分离株毒力岛(SPI-1、SPI-3)及毒力质粒的毒力基因invH、sopE、iacP、prgK、avrA、 sugR、rhuM、spvB、spvC进行PCR扩增,来调查上海市肠炎沙门氏菌毒力基因的携带情况,并探索了ERIC-PCR分子分型技术对沙门氏菌血清型的分型能力,之后对90株不同年份不同来源的肠炎沙门氏菌进行ERIC-PCR分型,调查2008—2012年肠炎沙门氏菌菌株之间的差异。
1.1 菌株

图1 2008—2012年上海地区90 株肠炎沙门氏菌9 种毒力基因分布图
Fig.1 Distribution of 9 virulence genes in 90 Salmonella Enteritidis isolates during 2008 to 2012 in Shanghai
2008—2012年在上海地区采集市售食品及临床患者样品,并从中分离食源性与医源性肠炎沙门氏菌共90 株,所有研究的肠炎沙门氏菌菌株都是随机挑选的,菌株编号见图1。另外,随机挑选肠炎沙门氏菌、猪霍乱沙门氏菌、鼠伤寒沙门氏菌、婴儿沙门氏菌、乙型副伤寒沙门氏菌、伤寒沙门氏菌、鸭沙门氏菌、鸡白痢沙门氏菌8 种沙门氏菌血清型菌株共17 株用于ERIC-PCR反应体系优化,菌株编号见表1。
表1 8种沙门氏菌血清型菌株
Table1 Strains of eight Salmonella serovars
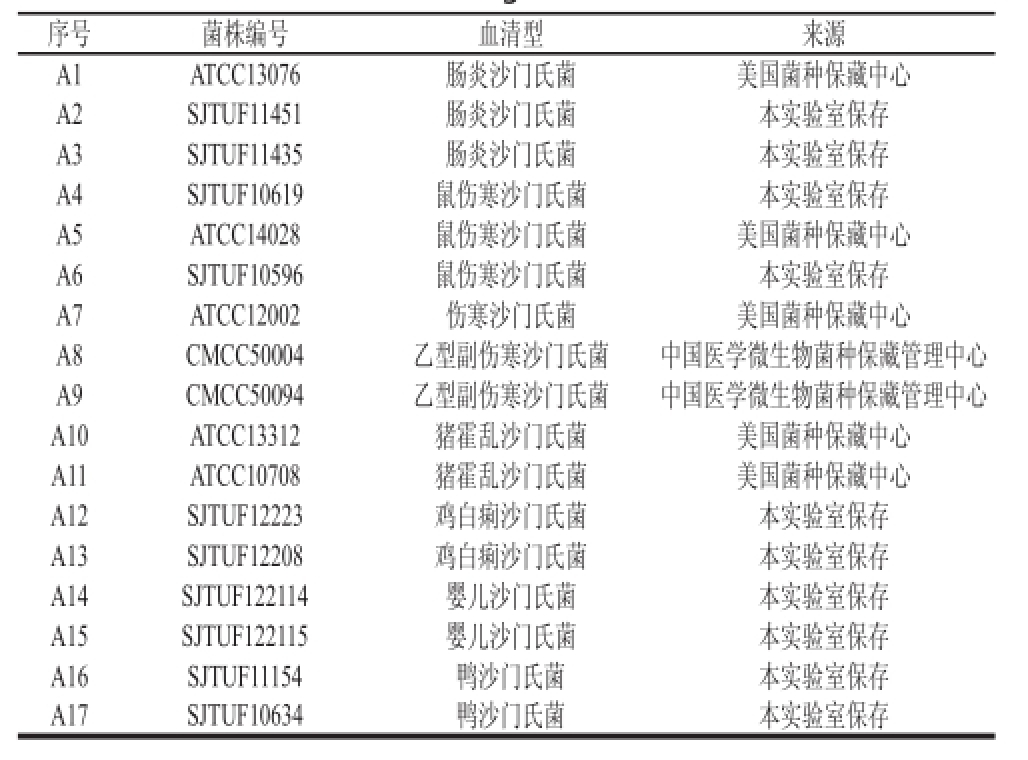
序号菌株编号血清型来源A1ATCC13076肠炎沙门氏菌美国菌种保藏中心A2SJTUF11451肠炎沙门氏菌本实验室保存A3SJTUF11435肠炎沙门氏菌本实验室保存A4SJTUF10619鼠伤寒沙门氏菌本实验室保存A5ATCC14028鼠伤寒沙门氏菌美国菌种保藏中心A6SJTUF10596鼠伤寒沙门氏菌本实验室保存A7ATCC12002伤寒沙门氏菌美国菌种保藏中心A8CMCC50004乙型副伤寒沙门氏菌中国医学微生物菌种保藏管理中心A9CMCC50094乙型副伤寒沙门氏菌中国医学微生物菌种保藏管理中心A10ATCC13312猪霍乱沙门氏菌美国菌种保藏中心A11ATCC10708猪霍乱沙门氏菌美国菌种保藏中心A12SJTUF12223鸡白痢沙门氏菌本实验室保存A13SJTUF12208鸡白痢沙门氏菌本实验室保存A14SJTUF122114婴儿沙门氏菌本实验室保存A15SJTUF122115婴儿沙门氏菌本实验室保存A16SJTUF11154鸭沙门氏菌本实验室保存A17SJTUF10634鸭沙门氏菌本实验室保存
1.2 试剂与仪器
Taq DNA聚合酶 立陶宛Fermentas公司;100/200 bp DNA Ladder分子量标准 天根生化科技(北京)有限公司。
ABI 9700 PCR扩增仪 美国AB公司;5415D台式微量离心机 德国Eppendorf公司;PS2A200核酸电泳仪 美国Hoefer公司;GIS2020凝胶成像仪 上海天能公司;EQR/GL-41超净工作台 新加坡Esco公司。
1.3 方法
1.3.1 肠炎沙门氏菌PCR验证
1.3.1.1 引物序列
根据本研究室已发表的肠炎沙门氏菌特异性靶点SEN1392的引物,引物序列详见表2。
表2 本研究所使用的PCR引物序列列表
Table2 The primer sequences used in this study

基因 位置T m/℃引物序列(5′-3′)大小/bp参考文献SEN13921478314-1480280 58GCCACTGTCTGATGCTCTTG656[19]58GAAAGGCTCCGTGGTTAGT invH SPI-I57AGCAACTGGCCAACGCAAAT314 57TGCAGTCTTTCATGGGCAGCAA sopE SPI-I59ATTGTTGTGGCGTTGGCATCGT356 55AATGCGAGTAAAGATCCGGCCT iacPSPI-I57CACCTCTTGTATTGCCGTTG176 57GGCATATATCCGCAAAGGTC avrA SPI-I55AATGGAAGGCGTTGAATCTG170 59GAGCTGCTTTGGTCCTCAAC prgK SPI-I57TTGAACAGCGACTGGAACAG217 53TCATAATCCACATCGGCAAA[20]
续表2

58CCGGTATACGACTCTGTGATC spvC毒力质粒57ACTCCTTGCACAACCAAATGCGGA571 59TGTCTCTGCATTTCGCCACCATCA ERIC-PCR基因组52ATGTAAGCTCCTGGGGATTCAC不定[13]52AAGTAAGTGACTGGGGTGAGCG 57TCTTACCGTGCTGTGGATGGTTCA rhuMSPI-359CATCGGCTGTACCCGACTAT222 57CAGCACGCTGATGAATGAGT spvB毒力质粒58CGGTTATAGAAGAGCTCCTGT322基因 位置T m/℃引物序列(5′-3′)大小/bp参考文献sugR SPI-359ACTGCTCGCTTGTGGTATCAGGAA331[20][21]
1.3.1.2 PCR反应体系与参数
PCR体系:10×buffer 2.5 μL,Mg 2+(25 mmol/L)1.5 μL,dNTP(2.5 mmol/L)1.5 μL,上下游引物(各10 μmol/L)均为0.5 μL,Taq DNA聚合酶(1 U/μL)1 μL,模板为2.5 μL,总体积为25 μL。
PCR反应参数:95 ℃预变性5 min,94 ℃变性30 s,T m退火30 s,72 ℃延伸30 s,35 次循环后72 ℃延伸10 min,4 ℃保存。取5 μL PCR产物,在电压为170 V条件下,进行凝胶琼脂电泳20 min,其中凝胶为1.5%琼脂糖,最后溴化乙锭(0.5 mg/L)染色后成像观察,所参照的DNA分子质量标准为100 bp DNA Ladder。
1.3.2 肠炎沙门氏菌毒力基因检测
沙门氏菌常见的毒力基因引物序列参见表2,PCR反应体系与参数参考1.3.1.2。
1.3.3 ERIC-PCR分子分型
1.3.3.1 ERIC-PCR引物序列
参见表2。
1.3.3.2 ERIC-PCR反应体系与参数
反应总体积25 μL:10×buffer 2.5 μL,Mg 2+(25 mmol/L)2 μL,dNTP(2.5 mmol/L)2 μL,上下游引物(各10 μmol/L)均为0.5 μL,Taq DNA聚合酶(1 U/μL)1 μL,模板为2.5 μL,其余用ddH 2O补齐。反应条件为:94 ℃预变性5 min;94 ℃变性45 s,52℃退火1 min,72 ℃延伸5 min,共进行35 个循环;72 ℃终延伸10 min。
1.3.3.3 ERIC-PCR产物检测
取5 μL PCR产物与1 μL 10×上样缓冲液混匀后,在1.5%的琼脂糖凝胶上,以140 V电压条件,电泳1 h,最后溴化乙锭(0.5 mg/L)染色后在凝胶成像仪中观察结果并成像。
1.3.3.4 ERIC-PCR指纹图谱分析
确定ERIC-PCR电泳图谱在相同分析区域内的条带数目及相对位置,对不同肠炎沙门氏菌进行ERIC-PCR电泳条带分析。
2.1 肠炎沙门氏菌PCR验证
2008—2012 年采集上海地区不同来源样品的90 株食源性与医源性肠炎沙门氏菌分离株,经PCR验证,菌株均有SEN1392基因扩增条带,结果如图2所示。图2中的条带明亮单一,且大小与Liu Bin等 [19]的结果完全一致,可以确定为肠炎沙门氏菌。

图2 部分肠炎沙门氏菌分离株PCR验证结果
Fig.2 PCR confirmation results for part of Salmonella Enteritidis isolates
2.2 肠炎沙门氏菌毒力基因的检测
对90 株肠炎沙门氏菌毒力岛SPI-1、毒力岛SPI-3及毒力质粒上的9 种毒力基因(invH、sopE、iacP、prgK、avrA、sugR、rhuM、spvB、spvC)进行PCR检测,并计算每种毒力基因的携带率。检出结果(图1)显示:在90 株肠炎沙门氏菌中,均携带有毒力基因invH、sopE与sugR,携带率为100.0%;89 株均含有iacP、avrA、rhuM、prgK的扩增条带,其中2011年医源菌株SJTUF11446无iacP扩增条带;2010年食源菌株SJTUF10999无prgK扩增条带,医源菌株SJTUF10797无avrA条带;2008年食源菌株SJTUF11323无rhuM,表明这4 种毒力基因的携带率为98.9%;与Han等 [22]对采集于医源、鸡肉、农场的54 株肠炎沙门氏菌毒力基因检测结果一致,携带率均在98.0%~100.0%之间,高于Zou Wen等 [20]40.0%~80.0%的结果。携带位于毒力质粒上的毒力基因spvB、spvC的菌株数目分别为77与71,携带率分别为85.6%与78.9%,71 株菌同时携带spvB和spvC两种毒力基因,概率为78.9%。而本研究中spvB 85.6%的携带率介于Han等 [22]100.0%的spvB携带率和杨劲松等 [23]43.3%的携带率之间,78.9%的spvC携带率略高于刘芳萍等 [24]68.2%的spvC携带率,这些差异可能与采集的样品来源及肠炎沙门氏菌区域差异有关。
2.3 沙门氏菌ERIC-PCR分型
本研究利用ERIC-PCR技术对8 种沙门氏菌血清型进行扩增,包括肠炎沙门氏、鼠伤寒沙门氏菌、伤寒沙门氏菌、乙型副伤寒沙门氏菌、猪霍乱沙门氏菌、鸡白痢沙门氏菌、婴儿沙门氏菌和鸭沙门氏菌共17 株。如图3所示,ERIC-PCR指纹图谱扩增条带清晰,每株菌的条带大小都在100~4 000 bp之间,扩增条带数介于11~22之间不等。
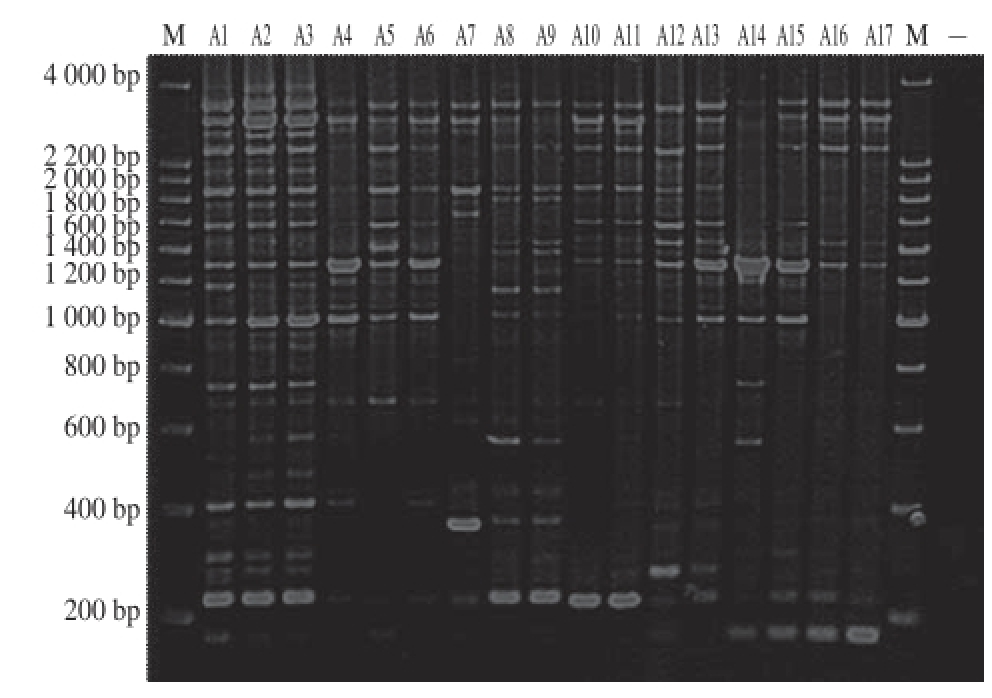
图3 8种沙门氏菌血清型ERIC-PCR指纹图谱
Fig.3 ERIC-PCR profiles for eight Salmonella serovars
M. 200 bp DNA Ladder;A1~A3.肠炎沙门氏菌;A4~A6.鼠伤寒沙门氏菌;A7.伤寒沙门氏菌;A8、A9.乙型副伤寒沙门氏菌;A10、A11.猪霍乱沙门氏菌;A12、A13.鸡白痢沙门氏菌;A14、A15.婴儿沙门氏菌;A16、A17.鸭沙门氏菌;-.阴性对照。
从条带分布上来看,除伤寒沙门氏菌与鸭沙门氏菌外其他沙门氏菌血清型条带分布都比较均匀。通过BioNumerics软件将ERIC-PCR指纹图谱绘制成UPGMA聚类树,并进行遗传进化关系分析,如图4所示。

图4 8种沙门氏菌血清型ERIC-PCR分型聚类分析结果
Fig.4 UPGMA tree of ERIC-PCR subtyping among 8 Salmonella serovars
根据图4聚类树,发现以相似度83%为基准时,8 种不同的沙门氏菌血清型分别聚在8 个不同的分支上,其中3 株肠炎沙门氏菌,2 株鸭沙门氏菌的ERIC-PCR图谱一致且其相似度均为100%。以相似度58%为基准,可以将菌株分为A、B、C三大簇,通过Hunter等
[25]提出的辛普森指数计算公式
![]() 式中:N为菌株总数;S为类型总数;n
j为第j类型菌株的数目)计算可得区分度指数D值为0.970 6,说明ERICPCR是不同沙门氏菌血清型的理想分型方法。
式中:N为菌株总数;S为类型总数;n
j为第j类型菌株的数目)计算可得区分度指数D值为0.970 6,说明ERICPCR是不同沙门氏菌血清型的理想分型方法。
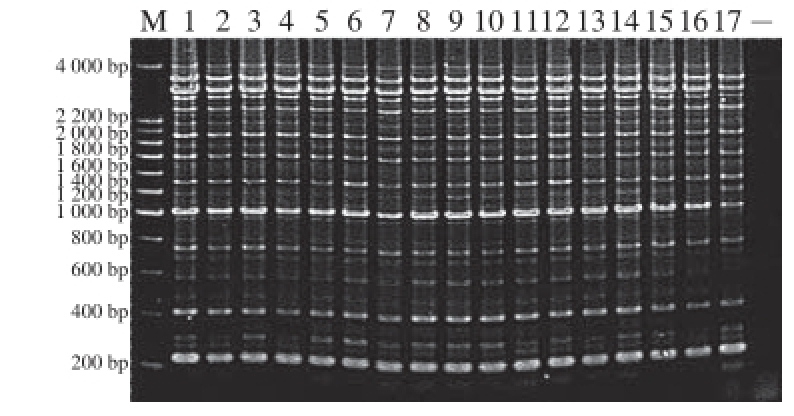
图5 部分肠炎沙门氏菌ERIC-PCR指纹图谱
Fig.5 ERIC-PCR profiles of part of Salmonella Enteritidis isolates
M. 200 bp DNA分子质量标准;1~16.不同来源肠炎沙门氏菌的ERIC-PCR图谱;17. S. Enteritidis ATCC13076;-.阴性对照。
随后,按照上述优化的循环程序,将90 株不同来源的上海肠炎沙门氏菌分离株进行ERIC-PCR扩增,部分结果如图5所示,每株肠炎沙门氏菌分离株ERIC-PCR扩增产物条带均清晰可辨,条带均匀分布,其中最大扩增片段接近4 000 bp,最小片段100 bp,所有条带大小在100~4 000 bp之间,每株菌的ERIC-PCR图谱都有22 条条带,其中大小为1 000 bp左右的条带最亮。本研究以标准菌株ATCC13076为阳性对照菌株,结果发现90 株肠炎沙门氏菌分离株不论是扩增条带数目还是条带大小均与标准菌株ATCC13076一致,ERIC-PCR指纹图谱并无差异,与Chmielewski等 [18]的结果存在较大差异,结果表明上海市医源性肠炎沙门氏菌分离株与食源性分离株之间,没有差异或者是差异微小以至于ERIC-PCR指纹图谱也难以辨别,一个可能的原因是在2008—2012年间上海地区肠炎沙门氏菌变异确实较小,以至于菌株同源性极高,另外一种可能是肠炎沙门氏菌分离株有基因组差异但是具有相同的ERIC分布及拷贝数,而ERIC-PCR不能甄别基因组其他位置上的基因差异。本研究也说明ERIC-PCR可能不适合作为高同源性菌株的理想分型方法,因此需要发掘新的分型方法,如单核苷酸多态性-PCR [26]和基于簇规律间隔短回文重复序列与毒力基因的多态性位点序列分型方法 [27]等,将成为高同源性肠炎沙门氏菌亚分型方法的未来发展方向。
肠炎沙门氏菌是重要的食源性致病菌,也是最主要的非伤寒沙门菌血清型之一,本研究对上海地区不同年份的肠炎沙门氏菌毒力基因携带情况进行了较为系统地探究,结果显示肠炎沙门氏菌毒力岛毒力基因invH、sopE、sugR、iacP、avrA、rhuM、prgK携带率达到98.0%~100.0%,毒力质粒毒力基因spvB、spvC携带率达到78.0%~86.0%,毒力基因高携带率对人群健康存在严重的威胁,须加强对食品卫生的监督与防控。此外,还进行了ERIC-PCR分子分型研究,阐述了ERIC-PCR对沙门氏菌血清型分型的适用性,证实ERIC-PCR对不同血清型具有快速、高效分型的优势,同时90 株不同来源肠炎沙门氏菌也阐释了ERIC-PCR对高同源性血清型菌株亚分型的局限性,需开发更适用于同一血清型或其他高同源性沙门氏菌菌株分型的分子分型方法,以适应流行病学研究和风险评估的需要。
参考文献:
[1] European Food Safety Authority & European Centre for Disease Prevention and Control. The European Union summary report on trends and sources of zoonoses, zoonotic agents and food-borne outbreaks in 2013[J]. EFSA Journal, 2015, 13(1): 3991-4152.
[2] 许学斌, 袁政安, 金汇明, 等. 上海市肠炎沙门菌流行特征和分子分型研究[J]. 上海预防医学杂志, 2009, 21(4): 149-152.
[3] 张艳红, 杜元钊, 吴延功, 等. 肠炎沙门氏菌快速检测方法的建立[J].中国动物检疫, 2002, 19(7): 25-28.
[4] FOLEY S L, LYNNE A M. Food animal-associated Salmonella challenges: pathogenicity and antimicrobial resistance[J]. Journal of Animal Science, 2008, 86(14): 173-187.
[5] FOLEY S L, JOHNSON T J, RICKE S C, et al. Salmonella pathogenicity and host adaptation in chicken-associated serovars[J]. Microbiology and Molecular Biology Reviews, 2013, 77(4): 582-607.
[6] 陈冬平, 罗薇. 沙门氏菌毒力相关因子研究进展[J]. 西南民族大学学报: 自然科学版, 2012, 38(5): 770-775.
[7] COLLAZO C M, GALAN J E. The invasion-associated type-Ⅲprotein secretion system in Salmonella: a review[J]. Gene, 1997,192(1): 51-59.
[8] BLANC-POTARD A B, SOLOMON F, KAYSER J, et al. The SPI-3 pathogenicity island of Salmonella enterica[J]. Journal of Bacteriology,1999, 181(4): 998-1004.
[9] 赵英伟, 孙萌. 肠炎沙门氏菌毒力质粒与食物中毒的关系研究[J].中国公共卫生学报, 1998, 17(3): 139-139.
[10] 方艳红, 孙裴, 魏建忠, 等. 沙门菌毒力基因研究进展[J]. 动物医学进展, 2010, 31(增刊1): 190-193.
[11] 程琼, 庞瑞亮, 王若晨, 等. 不同源沙门氏菌对小鼠致病力的比较与毒力基因检测[J]. 中国人兽共患病学报, 2013, 29(5): 460-465.
[12] 吴清平, 叶应旺, 张菊梅, 等. 结构功能及-技术的应用[J]. 中国卫生检验杂志, 2006, 16(4): 507-509.
[13] VERSALOVIC J, KOEUTH T, LUPSKI J R. Distribution of repetitive DNA sequences in eubacteria and application to fi ngerprinting of bacterial genomes[J]. Nucleic Acids Research, 1991, 19(24): 6823-6831.
[14] 晏群, 陈丽华, 刘文恩, 等. 分子生物学分型技术-的建立[J]. 实用医学杂志, 2003, 19(6): 688-689.
[15] 方伟, 杨杏芬, 柯昌文, 等. ERIC-PCR在肠道致病菌基因分型和流行病学中的应用[M]. 国外医学: 卫生学分册, 2009, 36(1): 46-50.
[16] LIM H, LEE K H, HONG C H, et al. Comparison of four molecular typing methods for the differentiation of Salmonella spp[J]. International Journal of Food Microbiology, 2005, 105(3): 411-418.
[17] 陈玲, 张菊梅, 杨小鹃, 等. 南方食品中沙门氏菌污染调查及分型[J].微生物学报, 2013, 53(12): 1326-1333.
[18] CHMIELEWSKI R, WIELICKO A, KUCZKOWSKI M, et al. Comparison of ITS profiling, REP- and ERIC-PCR of Salmonella Enteritidis isolates from Poland[J]. Journal of Veterinary Medicine Series B, 2002, 49(4): 163-168.
[19] LIU Bin, ZHOU Xiujuan, ZHANG Lida, et al. Development of a novel multiplex PCR assay for the identification of Salmonella enterica Typhimurium and Enteritidis[J]. Food Control, 2012, 27: 87-93.
[20] ZOU Wen, AL-KHALDI SF, BRANHAM WS, et al. Microarray analysis of virulence gene profi les in Salmonella serovars from food/ food animal environment[J]. Journal of Infection in Developing Countries, 2011, 5(2): 94-105.
[21] KARASOVA D, HAVLICKOVA H, SISAK F, et al. Deletion of sodCI and spvBC in Salmonella enterica serovar Enteritidis reduced its virulence to the natural virulence of serovars Agona, Hadar and Infantis for mice but not for chickens early after infection[J]. Veterinary Microbiology, 2009, 139 :304-309.
[22] HAN J, GOKULAN K, BARNETTE D, et al. Evaluation of virulence and antimicrobial resistance in Salmonella enterica serovar Enteritidis isolates from humans and chicken- and egg-associated sources[J]. Foodborne Pathogens and Disease, 2013, 10(12): 1008-1015.
[23] 杨劲松, 陈爱平, 陈建辉, 等. 年福建省分离自沙门菌病患者的肠炎沙门菌分子特征研究[J]. 预防医学论坛, 2013, 19(4): 241-242.
[24] 刘芳萍, 王德宁, 李昌文, 等. 鸡源沙门氏菌耐药性的分析及毒力基因的检测[J]. 中国兽医科学, 2013, 43(12): 1236-1239.
[25] HUNTER P R, GASTON M A. Numerical index of the discriminatory ability of typing systems: an application of Simpson's index of diversity[J]. Journal of Clinical Microbiology, 1988, 26(11): 2465-2466.
[26] OGUNGUNREMI D, KELLY H, DUPRAS A A, et al. Development of a new molecular subtyping tool for Salmonella enterica serovar Enteritidis based on single nucleotide polymorphism-polymerase chain reaction (SNP-PCR)[J]. Journal of Clinical Microbiology, 2014,52(12): 4275-4285.
[27] LIU Fenyun, KARIYAWASAM S, JAYARAO B M, et al. Subtyping Salmonella enterica serovar enteritidis isolates from different sources by using sequence typing based on virulence genes and clustered regularly interspaced short palindromic repeats (CRISPRs)[J]. Applied and Environmental Microbiology, 2011, 77(13): 4520-4526.
Screening of Virulence Genes and ERIC-PCR Sub-typing for Salmonella Enteritidis Isolates in Shanghai during 2008—2012
ZHUANG Xiaofei
1, ZHOU Xiujuan
1, XU Xuebin
2, SHI Xianming
1, SHI Chunlei
1,*
(1. MOST-USDA Joint Research Center for Food Safety, Bor Luh Food Safety Center, State Key Laboratory of Microbial Metabolism,School of Agriculture and Biology, Shanghai Jiao Tong University, Shanghai 200240, China;2. Center for Disease Control and Prevention of Shanghai, Shanghai 200336, China)
Abstract:In this study, a total of 90 isolates of Salmonella Enteritidis, including clinical and foodborne isolates, were collected from different areas of Shanghai during 2008 to 2012. Polymerase chain reaction (PCR) was used to investigate the carrying rates of 9 common virulence genes not only in SPIs (SPI-1 and SPI-3) but also in virulence plasmid. In addition, an optimized enterobacterial repetitive intergenic consensus (ERIC)-PCR was applied to subtype these isolates. The PCR results of 90 isolates showed that the carrying rate was 100.0% for virulence genes invH, sopE and sugR, and 98.9% for iacP, avrA,prgK and rhuM, while the carrying rate of spvB and spvC was 85.6% and 78.9% respectively. Seventeen strains belonging to eight different Salmonella serovars were clearly differentiated by ERIC-PCR, with the Simpson's Diversity Index up to 0.970 6. However, the ERIC-PCR profi les of 90 Salmonella Enteritidis isolates showed no difference. In conclusion, the carrying of these virulence genes, especially sugR, invH and sopE, is very common among these isolates, demonstrating that they pose a potential threat to human health and that ERIC-PCR can be applied to the molecular classifi cation of different Salmonella serovars, but not for Salmonella Enteritidis.
Key words:Salmonella Enteritidis; virulence genes; ERIC-PCR
中图分类号:TS201.6
文献标志码:A
文章编号:1002-6630(2015)14-0165-06
doi:10.7506/spkx1002-6630-201514032
收稿日期:2014-10-27
基金项目:国家高技术研究发展计划(863计划)项目(2012AA101601);“十二五”国家科技支撑计划项目(2012BAK17B10);上海市国际合作项目(14390711900)
作者简介:庄孝飞(1988—),男,硕士研究生,研究方向为食品安全与微生物。E-mail:zhuangxiaofeilinyi@163.com
*通信作者:施春雷(1977—),女,副教授,博士,研究方向为食品安全与微生物。E-mail:clshi@sjtu.edu.cn