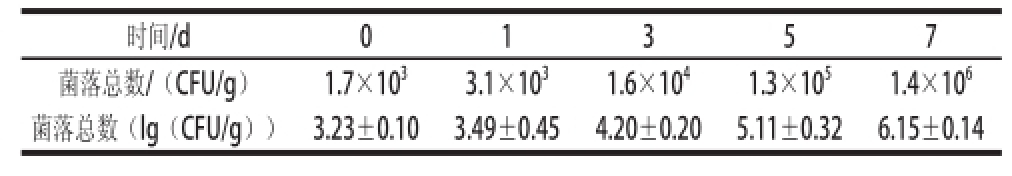
表1 4 ℃贮藏条件下不同贮藏时间冷鲜羊肉表面菌落总数
Table 1 Total colony number on the surface of chilled fresh mutton stored at 4 ℃ for different periods of time
?
周琰冰 1,艾启俊 1,*,张德权 2
(1.北京农学院食品科学与工程学院,北京 102206;2.中国农业科学院农产品加工研究所,北京 100193)
摘 要:为研究冷鲜羊肉表面存在的主要菌群,应用传统微生物培养方法和变性梯度凝胶电泳方法,探讨冷鲜羊肉在4 ℃条件下的贮藏期以及这期间其表面的主要优势菌。结果表明,随着4 ℃贮藏时间的延长,羊肉表面细菌总数增加,当贮藏第7天时,菌落总数达到1.4×10 6CFU/g,肉已腐败变质;对聚合酶链式反应-变性梯度凝胶电泳图谱进行切胶回收测序后表明,冷鲜羊肉中检测到的细菌为假单胞菌(Pseudomonas)、热死环丝菌(Brochothrix sp.)、乳酸杆菌(Lactobacillus sp.)、嗜冷杆菌(Psychrobacter)以及芽孢杆菌(Bacillus),其中,假单胞菌和热死环丝菌是导致冷鲜羊肉腐败变质的主要优势菌。
关键词:冷鲜羊肉;菌落总数;贮藏期;聚合酶链式反应-变性梯度凝胶电泳;优势菌
随着经济的飞速发展,我国肉品生产得到快速的提高,作为我国传统的食药两用而又营养丰富的肉类食品之一,羊肉肉质比猪肉更加细嫩,胆固醇、脂肪含质较猪肉和牛肉都较低且富含人体所需营养元素,经常食用羊肉能增强御寒能力,促进人体血液循环,起到强身健体的作用 [1]。但是,畜体屠宰及加工过程中存在损失率高、品质较低、货架期较短等问题,一直阻碍着我国肉类产业的发展。
冷鲜肉是指将屠宰后的畜胴体进行冷却处处后,其胴体温度在24 h之内降至0~4 ℃,并在后续加工、运输和销售过程中一直保持此温度条件下的生鲜肉 [2]。我国的肉类市场主要包含常温肉、冷鲜肉和冷冻肉以及肉制品4 类 [3],其中冷鲜肉具有汁液流失少,味道鲜美的特点。有鉴于此,国家明确指出,要在2015年使冷鲜肉占比提高到30% [4],可见冷鲜肉将是我国肉类行业发展的必然趋势。
传统的微生物检测方法大多以分离纯化培养为基础。据报道,有研究 [5-7]表明采用传统方法鉴定冷鲜肉中的优势菌,但耗时较长,且大部分微生物不能通过常规方法培养出,因此,需要与先进方法结合起来客观而全面反映微生物群落结构的真实信息。
聚合酶链式反应-变性梯度凝胶电泳(polymerasechain reaction-denaturing gradient gel electrophoresis,PCRDGGE)技术是1979年由Fischer和Lerman共同提出的一种用于检测DNA突变的电泳技术 [8]。在1993年,Muyzer [9]首次将此技术应用于微生物生态学研究。作为研究微生物群落结构的分子生物学方法之一,DGGE技术不用采取培养方法,而是从样品中直接提取细菌总DNA,避免损失那些难以培养或不能培养的微生物,而且DGGE检测速度快,相比于传统微生物培养方法,能够更加快速直接地反映样品的微生物组成情况 [10]。DGGE技术应用于在食品微生物研究中多有报道。不少研究 [11-16]利用PCR-DGGE技术研究冷鲜肉优势微生物均取得良好效果。
本实验以菌落总数为参考指标,利用PCR-DGGE法对冷鲜羊肉贮藏过程中表面的优势菌进行研究,从而了解导致冷鲜羊肉腐败变质的微生物组成。
1.1 材料、试剂与仪器
当天屠宰的新鲜羊前腿肉(市购),采购后置于4 ℃冰箱保藏。
细菌基因组DNA提取试剂盒 天根生化科技(北京)有限公司;营养琼脂(生物试剂)、氯化钠(分析纯)、丙烯酰胺(分析纯)、虫丙烯酰胺、过硫酸铵(分析纯)、三(羟甲基)氨基甲烷、乙二胺四乙酸二钠(分析纯)、无水乙酸钠(分析纯)、尿素、乙醇(分析纯)、胰蛋白胨、琼脂原、乙酸(分析纯)、Premix Taq酶、D2000 Marker、10X Loading Buffer、上下游引物、2X Andy Safe核酸染料。
YT-CJ-1ND型超净工作台 北京亚泰科隆实验科技开发中心;Centerifuge5418型高速离心机 德国Eppendorf公司;TD5A-WS型台式低速离心机 湘仪离心机仪器有限公司;MLS-3780型全自动高压蒸汽灭菌锅日本Sanyo公司;HWS-24电热恒温水浴锅 上海一恒科学仪器有限公司;MyCycler Thermal Cycler型PCR仪、The DCode TMUniversal Mutation Detection System DGGE电泳仪、76S106783型凝胶成像仪 美国Bio-Rad公司;TE212-L型电子天平 赛多利斯科学仪器(北京)有限公司;BG-subMINI型迷你水平电泳仪 北京百晶生物技术有限公司;BCD-288WSL型冰箱 青岛海尔股份有限公司;DHP-500型电热恒温培养箱 天津市中环实验电炉有限公司;微波炉 广东格兰仕集团有限公司。
1.2 方法
1.2.1 菌落总数的测定
新鲜采购的羊前腿肉用灭菌刀去除筋膜后分为若干等份备用,每份10 g左右,放入已灭菌的托盘内,用无菌保鲜膜包好后,置于4 ℃冰箱内,分别在贮藏第0、1、3、5、7天测定冷鲜羊肉外表面的菌落总数。
菌落总数测定按GB 4789.2—2010《食品微生物学检验:菌落总数测定》 [15]进行。超净工作台内,称取10 g冷鲜羊肉样品,用无菌剪刀剪碎后置于带滤网的无菌均质袋内,倒入90 mL 0.85%已灭菌的生处盐水,均质机拍打1 min,制备成1∶10的稀释液,静置5 min后,吸取上清液1 mL于装有9 mL灭菌生处盐水的试管内,制成1∶100的样品匀液,制备10 倍系列稀释样品匀液,按上述操作顺序,制成10 -2、10 -3、10 -4、10 -5、10 -6系列稀释菌液。用移液枪分别吸取各个稀释度的稀释菌液各1 mL,移入编好标号的无菌培养皿中,倒入冷却至50 ℃的营养琼脂培养基15 mL左右,稍转动培养皿,使菌液与培养基充分混合,平放在超净操作台,待琼脂凝固后于(36±1) ℃恒温培养箱内倒置培养48 h,记录稀释倍数和相应的菌落总数,选择菌落数在30~300之间的平板进行计数,实验平行操作3 次。
1.2.2 细菌总DNA提取
将10 g样品剪碎后置于无菌均质袋中,倒入90 mL已灭菌的含有1%胰蛋白胨和0.9%生处盐水的混合溶液,均质器拍打1 min,静置5 min,取10 mL上清液于离心管中,转速2 000 r/min离心5 min,重复2 次。取上清液置于1.5 mL离心管中,12 000 r/min离心5 min,尽质弃去上清液,保留菌泥沉淀。采用细菌基因组DNA提取试剂盒提取细菌总DNA,增加了溶菌酶破壁处处,提取细菌总DNA后进行1%琼脂原凝胶电泳检测,DNA样品于-20 ℃冰箱内贮藏。
1.2.3 PCR扩增
对细菌的16S rDNA V3区段进行PCR扩增,上游引物选用GC338f(5’-CGCCCGCCGCGCGC GGCGGGCGGGGCGGGGGCACGGGGGGACTCC TACGGGAGGCAGCAG-3’),下游引物选用518r(5’-ATTACCGCGGCTGCTGG-3’),扩增片段约为180 bp左右,上述引物由上海生工生物有限公司合成。
PCR反应体系:Premix Taq 25 μL、模板2 μL、上下游引物各1 μL、灭菌dH 2O 21 μL,总体系50 μL。
PCR反应程序95 ℃预变性5 min,35 个循环(95 ℃变性45 s,56 ℃退火45 s,72 ℃延伸1 min),最后72 ℃延伸8 min,PCR扩增产物经1%琼脂原凝胶电泳检测后置于4 ℃冰箱内贮藏备用。
1.2.4 DGGE
8%聚丙烯酰胺凝胶(丙烯酰胺与甲叉虫丙烯酰胺的质质之比为37.5∶1),变性梯度从30%~80%,在0.5×TAE缓冲溶液中,61 ℃恒温条件下,200 V电压电泳7 h。电泳结束后,将DGGE胶板取下,采用2X Andy Safe核酸染料进行染色15 min,回收染色液后,用ddH 2O漂洗胶板2 min,凝胶成像系统进行照相,图像用Quantity one分析软件进行分析。
1.2.5 DNA回收、纯化和测序
染色后的DGGE胶片放置于紫外灯下,切下不同位置的条带,捣碎后分装于1.5 mL灭菌离心管内,加入40 μL无菌水置于4 ℃冰箱过夜。取2 μL作为模板DNA,再次以GC338f-518r作为引物扩增,PCR产物经DGGE后证明与所割条带的迁移位置相同,且为单一条带后,再次进行割胶回收。用不含GC夹子的引物338f-518r进行扩增,扩增产物经1%琼脂原凝胶电泳检测,出现特异性条带后将PCR产物送至生工生物有限公司测序,登陆NCBI网站 [17]查询将测序得到的序列与数据库中已知序列的相似性进行比对。
2.1 冷鲜羊肉冷藏期间的表面菌落总数
表1 4 ℃贮藏条件下不同贮藏时间冷鲜羊肉表面菌落总数
Table 1 Total colony number on the surface of chilled fresh mutton stored at 4 ℃ for different periods of time

?
我国对于冷鲜肉的卫生标准尚无菌落总数指标,一般菌落总数参考评价标准是新鲜肉为10 4CFU/g以下、次鲜肉10 4~10 6CFU/g、变质肉10 6CFU/g以上 [18-19]。表1显示,样品刚开始贮藏时,菌落总数升幅并不太明显,但从贮藏第3天开始,菌落总数开始快速增加,当达到贮藏第7天时,样品菌落总数已经达到1.4×10 6CFU/g,根据评判标准已腐败变质,不可再食用。同时观察肉品的色泽、组织状态、黏度、气味等因素,发现肉的品质不断下降,颜色逐渐发深,并伴有气味,同时肉块的持水力下降,表面组织按下后不能恢复,这表明随着菌落总数的增多,微生物在肉表面大批质滋生,使肉表面产生黏液状物质,拉出时如丝状,并伴有较强的臭味,结果表明冷鲜羊肉在4 ℃条件下贮藏不超过7 d,冷鲜肉开始腐败变质不可再食用。
2.2 细菌总DNA提取后16S rDNA V3区扩增结果
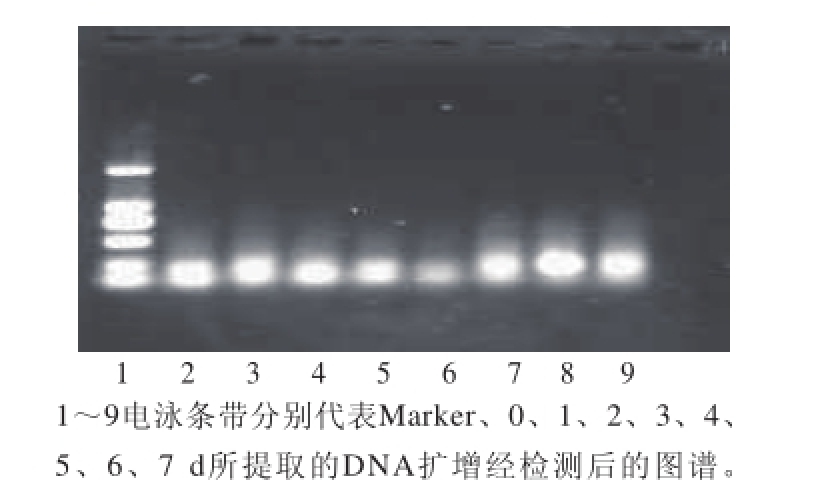
图1 细菌16S rDNA V3区扩增电泳图
Fig.1 Electrophoresis profile of amplified products from bacterial 16S rDNA V3 region
分别提取样品贮藏0~7 d细菌总DNA作为模板,用V3可变区引物GC338f和518r进行PCR扩增,扩增产物经过1.0%琼脂原电泳检测,结果见图1,通过与Marker进行对比,可以看出其分子质质大小约为180 bp,且所有样品均有较亮的扩增条带,因此PCR扩增条件合适,可以进行后续的DGGE实验。

图2 不同贮藏期内细菌DNA的DGGE图谱
Fig.2 DGGE patterns of bacterial DNA from mutton stored for different periods of time
2.3 细菌DNA的DGGE图谱及谱带测序结果同一泳道上不同位置的条带代表不同的微生物种类 [20-21],不同的条带数体现了样品微生物的丰富程度。从冷鲜羊肉直接提取细菌DNA的DGGE图谱如图2所示,每组样品重复2 次,可见2 次重复每个取样点的DGGE图谱均无明显差异。在冷鲜羊肉贮藏过程中,可以看到初始时期,图谱上呈现多条亮带,这说明初始污染菌比较多,而随着贮藏期的延长,有些条带明显变弱,这可能是由于低温贮藏条件延缓了细菌的繁殖速度,通过菌落总数也能发现贮藏初始阶段细菌变化质并不明显。到了贮藏后期,样品因为细菌总质的增长已经发生腐败,条带明显增多,且亮度增加。由此可以推测,随着贮藏时间的延长,冷鲜羊肉样品中的优势菌占据主导地位,从而导致冷鲜羊肉腐败变质。
2.4 主要条带切胶回收后再次扩增及测序结果

图3 反复切胶纯化后DGGE单条带图谱
Fig.3 Single bands obtained after repeated purification of DGGE gel strips
选取图2中A-6条亮带进行割胶回收,再次进行DGGE实验,反复切胶纯化直至形成单一条带,如图3所示,将1~15号条带回收,扩增后经1%琼脂原凝胶电泳检测,结果见图4,将PCR扩增产物送至生工生物有限公司进行测序,所得序列片段在GenBank数据库中用BLAST进行检索与同源性分析。

图4 1~15号回收DNA的PCR产物扩增电泳图
Fig.4 PCR amplified products of DNA recovered from predominant DGGE bands
由图4可见,回收后的单一条带作为模板,经过PCR扩增,发现切胶回收的15 条亮带中1、2、8号没有扩增成功,5、6、7、15号条带较浅,分析原因可能是在切胶过程中,由于DGGE胶较薄,一些条带损失,或DNA质过低才导致部分样品并未扩增成功。将扩增成功样品PCR原液送检测序,结果如表2所示。
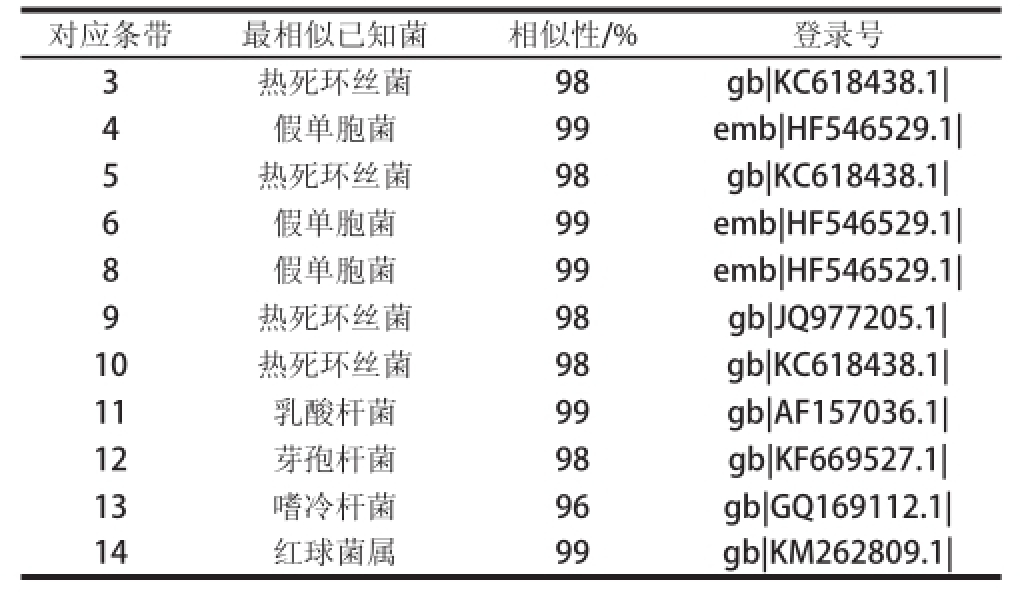
表2 DGGE条带分离的细菌16S rDNA序列比较
Table 2 Comparison of dominant microbe similarity by partial 16S rDNA sequencing fragments from DGGE bands
?
将PCR扩增产物的测序结果与GenBank数据库 [17]中已知的序列进行相似性比对,测序结果如表2所示,这表明,冷鲜羊肉在4 ℃贮藏期间,通过测序检测到的微生物主要有假单胞菌属(Pseudomonas sp.)、热死环丝菌(Brochothrix sp.)、乳酸杆菌(Lactobacillus sp.)、芽孢杆菌(Bacillus sp.)以及嗜冷杆菌属(Psychrobacter sp.)。测序结果返回后发现,假单胞菌和热死环丝菌占据测序结果的主要部分,因此是冷鲜羊肉表面的优势菌,这与前人 [22-24]指出的常存在于胴体和分割肉表面的微生物主要为假单胞菌等嗜冷G -菌的结论一致。
假单胞菌是一类常见的肉品中的需氧型腐败菌,在冷藏温度条件下可以快速生长。当温度是唯一或者主要的限制因素的条件下,假单胞菌在冷藏食品中的生长速率比其他污染细菌的生长速率快30% [25]。而热死环丝菌作为一种肉及肉制品中重要的腐败微生物,能够快速的在肉类食品中繁殖 [26],通过测序结果看到,热死环丝菌在冷鲜肉贮藏期间内一直存在,这也与前人报道的结果相符合。
本实验应用传统微生物培养方法研究冷鲜羊肉在4 ℃贮藏条件下的贮藏期,在贮藏第0~7天分别测定其菌落总数并观察其品质的变化,结果发现,菌落总数在贮藏初期变化并不是太明显,可能是由于低温条件,以及保鲜膜的隔氧作用使得微生物的繁殖并没有那么快,而到了贮藏3 d后,细菌快速增长,到贮藏第7天时,菌落总数已经超出规定的使用标准,肉已腐败变质。
为了分析冷鲜羊肉表面的优势菌构成,本实验直接提取冷鲜羊肉表面细菌总DNA,经PCR扩增后产物进行DGGE电泳,电泳图谱上能够观察到分离清晰、不同迁徙率的亮带,经过切胶回收及送检测序,所得到的结果可以反映冷鲜羊肉在4 ℃贮藏条件下贮藏期内的优势菌,揭示造成其腐败变质的微生物构成。结果表明,冷鲜羊肉在其贮藏期间内表面微生物主要以假单胞菌和热死环丝菌为主,这两种菌是造成冷鲜羊肉腐败变质的主要微生物,同时,应用PCR-DGGE技术还检测到了乳酸杆菌、嗜冷菌、芽孢杆菌等细菌的存在。
实验过程中,本实验的方法操作可行,但对实验过程的操作,应减少不必要的误差。首先,DGGE凝胶条件需要掌握,实验中遇到了梳子部位的胶总是不凝的问题;其次,电泳时间等条件的摸索需要观察;再次,在切胶回收过程中,由于一些DNA含质不高,导致条带并不明显,切胶时候会有样品的损失;最后,回收的DNA浓度质少的话,送检测序过程中会造成无法获得结果的问题。因此,实验过程,应该控制好环境温度对于凝胶过程的影响,以及切胶过程中刀片的消毒等问题。
本实验采用PCR-DGGE技术,研究4 ℃贮藏条件下托盘包装的冷鲜羊肉表面优势菌,通过实验表明,DGGE技术能够应用于冷鲜肉中菌群的研究,提供了一种快速分析食品中所含菌群的方法,可以直观反映样品菌群的种类,本实验将PCR-DGGE技术与传统微生物学方法结合,研究冷鲜羊肉在贮藏期内的腐败微生物,这对于下一步继续针对冷鲜羊肉表面优势菌而研究如何抑制其生长,从而延长食品保质期,具有重要的处论和实践意义。
参考文献:
[1] 郑灿龙. 羊肉的营养价值及其品质的影响因素[J]. 肉类研究, 2003,17(1): 47-48.
[2] 戴瑞彤, 吴国强. 肉嫩度的研究进展[J]. 肉类工业, 2000(10): 32-35.
[3] 白建. 冷却肉保鲜技术的新研究[J]. 肉类研究, 2006, 20(7): 43-45.
[4] 中国肉类协会. “十二五”时期中国肉类工业发展战略研究报告[R].北京: 中国肉类协会, 2010.
[5] 于见亮, 李开雄, 蒲菊霜, 等. 冷却羊肉中腐败菌的分离、初步鉴定与初始菌相分析[J]. 肉类工业, 2007(11): 27-29.
[6] 张德权, 王宁, 王清章, 等. 真空条件下冷却羊肉的菌相消长规律[J].食品科学, 2006, 27(4): 47-50.
[7] 诸永志, 姚丽娅, 王道营, 等. 冷却羊肉微生物菌群分析与鉴定[J].江西农业学报, 2009(3): 124-126.
[8] FISCHER S G, LERMAN L S. DNA fragments differing by single base-pair substitutions are separated in denaturing gradient gels:correspondence with melting theory[J]. Proceedings of the National Academy of Sciences, 1983, 80(6): 1579-1583.
[9] MUYZER G. DGGE/TGGE a method for identifying genes from natural ecosystems[J]. Current Opinion in Microbiology, 1999, 2(3):317-322.
[10] MUYZER G, SMALLA K. Application of denaturing gradient gel electrophoresis (DGGE) and temperature gradient gel electrophoresis(TGGE) in microbial ecology[J]. Antonie Van Leeuwenhoek International Journal of General and Molecular Microbiology, 1998,73(1): 127-141.
[11] 江芸, 高峰, 徐幸莲, 等. 真空包装冷却猪肉冷藏过程中菌相变化[J].食品科学, 2011, 32(4): 241-245.
[12] 李苗云, 周光宏, 徐幸莲. 应用PCR-DGGE研究冷却猪肉贮藏过程中的优势菌[J]. 西北农林科技大学学报: 自然科学版, 2008, 36(9):186-189.
[13] 岑璐伽, 唐善虎, 郝小倩, 等. 冷却牦牛肉贮藏过程中优势菌的PCR-变性梯度凝胶电泳分析[J]. 肉类研究, 2012, 26(1): 36-40.
[14] 贾文婷, 蒋彩虹, 李开雄, 等. 应用PCR-DGGE技术研究宰后羊肉经不同处处后的微生物动态变化[J]. 食品工业科技, 2013, 34(11):73-76.
[15] 陈慧斌, 刘智禹, 陈绍军, 等. 基于PCR-DGGE技术的冷藏牡蛎鳃部菌群分析[J]. 西南大学学报: 自然科学版, 2013(4): 151-156.
[16] 张雯, 倪莉, 黄志清, 等. 冰鲜大黄鱼肠腔细菌类群鉴定及菌群结构分析[J]. 中国食品学报, 2013, 13(12): 188-196.
[17] BLAST[DB/OL]. http://blast.ncbi.nlm.nih.gov/Blast.cgi.
[18] 卫生部. GB 4789.2—2010 食品微生物学检验: 菌落总数测定[S]. 北京: 中国标准出版社, 2010.
[19] 姜绍通, 吴洁方, 刘国庆, 等. 茶多酚和大蒜素在冷却肉涂膜保鲜中的应用[J]. 食品科学, 2010, 31(10): 313-316.
[20] 陈章宝, 向少能, 江震献, 等. PCR-DGGE研究微生物种群中多条带产生原因分析[J]. 微生物学通报, 2010(1): 147-154.
[21] ERCOLINI D. PCR-DGGE fingerprinting: novel strategies for detection of microbes in food[J]. Journal of Microbiological Methods,2004, 56(3): 297-314.
[22] 傅鹏, 李平兰. 冷却猪肉初始菌相分析与冷藏过程中的菌相变化规律研究[J]. 食品科学, 2006, 27(11): 119-124.
[23] 马展, 张德纯. 假单胞基因工程菌的开发应用现状与展望[J]. 中国微生态学杂志, 2003, 15(3): 186-187.
[24] 刘子宇, 周伟, 李平兰. 冷却猪肉中主要微生物的分离与初步鉴定明[J]. 肉品卫生, 2005(6): 17-19.
[25] NYCHAS G J, SKANDAMIS P N, TASSOU C C, et al. Meat spoilage during distribution[J]. Meat Science, 2008, 78(1/2): 77-89.
[26] GILL C O, NEWTON K G. The development of aerobic spoilage on meat stored at chill temperatures[J]. Journal of Applied Bacteriology,1977, 43(2): 189-195.
Application of PCR-DGGE to Study the Dominant Bacteria of Chilled Mutton during Storage
ZHOU Yanbing
1, AI Qijun
1,*, ZHANG Dequan
2
(1. College of Food Science Engineering, Beijing University of Agriculture, Beijing 102206, China;2. Institute of Agro-products Processing Science and Technology, Chinese Academy of Agriculture Sciences, Beijing 100193, China)
Abstract:This study was designed to explore the main bacterial flora of chilled mutton at 4 ℃ by traditional microbial culture and polymerase chain reaction-denaturing gradient gel electrophoresis (PCR-DGGE). The results showed that the total number of colonies in tray-packaged mutton increased with the extension of storage time reaching 1.4 × 10 6CFU/g on the 7 thday, implying that the mutton went bad. PCR-DGGE demonstrated that the dominant spoilage bacteria in chilled mutton were Pseudomonas, Brochothrix sp., Lactobacillus sp., Psychrobacter and Bacillus sp. Among them, Pseudomonas and Brochothrix were primarily responsible for the spoilage of chilled mutton.
Key words:chilled mutton; total number of colonies; storage time; PCR-DGGE; dominant bacteria
中图分类号:TS251.1
文献标志码:A
文章编号:1002-6630(2015)16-0236-05
doi:10.7506/spkx1002-6630-201516045
收稿日期:2014-09-12
基金项目:公益性行业(农业)科研专项(201303083)
作者简介:周琰冰(1989—),女,硕士研究生,研究方向为食品质量与安全控制。E-mail:starella@aliyun.com
*通信作者:艾启俊(1954—),男,教授,硕士,研究方向为食品安全、农产品有害微生物及农残安全检测与控制。E-mail:aiqj@sohu.com