基于内参的大肠埃希氏菌O157∶H7实时荧光定量PCR快速检测方法的建立
王建昌,王金凤,段永生,李 静,陈志敏,陈瑞春*
(河北出入境检验检疫局检验检疫技术中心,河北 石家庄 050051)
摘 要:针对大肠埃希氏菌O157∶H7 rfbE和Flic靶基因保守序列,设计特异性引物和探针,优化反应体系,并加入内参(internal amplification control,IAC),建立能够实时监控反应过程的荧光定量聚合酶链式反应(polymease chain reaction,PCR)检测方法。结果表明,该方法对E. coli O157∶H7基因组DNA的最低检测限为1 pg/μL;对含有靶基因质粒的最低检测限为10
3copies/μL;对细菌的最低检测限为5×10
3CFM/mL;Ct值与模板拷贝数均呈良好的线性关系(R
2=0.999)。人工污染实验结果表明,在初始菌量为7 CFM/25 g时,采用水洗加试剂盒法提取DNA,E. coli O157∶H7在增菌6 h后即可检出;而采用水煮法提取DNA,则在增菌10 h后方可检出。建立的E. coli O157∶H7实时荧光定量PCR方法,既能有效检测食品中O157∶H7,又能实时监测PCR反应过程,有效防止“假阴性”的发生。
关键词:大肠埃希氏菌O157∶H7;rfbE;Flic;实时荧光定量PCR;内参
大肠埃希氏菌O157∶H7(Escherichia coli O157∶H7,E. coli O157∶H7)是肠出血型大肠埃希氏菌的一种主要血清型,可引起人出血性结肠炎、溶血性尿毒综合征以及血栓形成性血小板减少性紫癜等
[1],是一种危害严重的食源性病原微生物。E. coli O157∶H7广泛分布于自然界,严重威胁公众健康
[2]。该菌在猪、牛粪便及猪肉、牛肉
[3]中分离率极高,其引起的疾病也与食用未烘烤的饼干面团
[4]、生菜
[5]等有关。E. coli O157∶H7多暴发于发达国家,在美国和加拿大监测的肠道致病菌中,该菌分别排在第2位和第3位。自1987年我国权太叔
[6]首次分离出该菌之后,相继有安徽、江苏、河南、湖北等十多个省份报道了该菌的感染情况
[7-8]。河北省疾病预防控制中心对随机抽取的530 份熟肉制品、生肉、蔬菜海产品的检测表明,E. coli O157∶H7均从生肉中检出,阳性检测率为0.9%
[9],说明食用生肉仍是其感染的主要途径。江苏省徐州市2006—2009年对E. coli O157∶H7的检出率为1.21%
[10]。E. coli O157∶H7时刻威胁着人们的健康,因而快速特异的检测方法有效控制该菌是十分必要的。
E. coli O157∶H7传统的分离鉴定法的操作步骤一般包括样品增菌、选择性增菌、生化特征鉴定与血清分型,操作复杂,一般需要5~6 d。胡慧
[11]、蒋彦君
[12]等分别建立了E. coli O157∶H7的荧光聚合酶链式反应(polymease chain reaction,PCR)和PCR检测方法,上述PCR方法多数对该菌纯培养物进行了灵敏性、检测限的分析,但未对增菌时间和增菌后样品核酸提取进行研究评估。而在PCR检测方法的实际应用中,大部分样品基质复杂,必定存在大量未知的复杂成分,可能会存在影响PCR扩增的物质,同时核酸提取过程中残留的物质也可能导致PCR扩增的抑制,从而出现“假阴性”结果或定量值较低。
本研究所建立的E. coli O157∶H7检测方法是一种基于内参的实时荧光定量PCR方法,在反应体系中添加IAC以对整个反应过程进行实时监测,从而有效地防止“假阴性”结果的产生,并通过水煮法和商业化试剂盒法对增菌后样品提取核酸进行靶基因检测,均取得了良好的检测结果。
1 材料与方法
1.1 实验菌株
本实验所用菌株见表1。
表1 本实验所用菌株
Table1 Strains used in this study
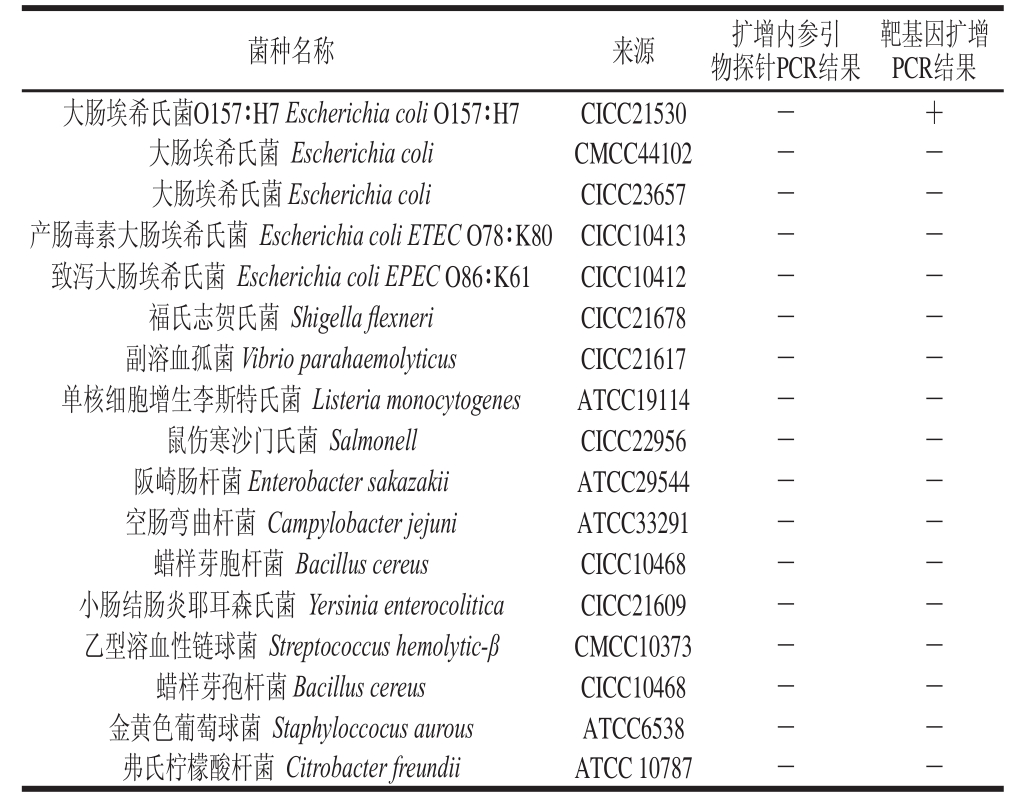
注:-.阴性;+.阳性。
靶基因扩增PCR结果大肠埃希氏菌O157∶H7 Escherichia coli O157∶H7CICC21530-+大肠埃希氏菌 Escherichia coliCMCC44102--大肠埃希氏菌 Escherichia coliCICC23657--产肠毒素大肠埃希氏菌 Escherichia coli ETEC O78∶K80CICC10413--致泻大肠埃希氏菌 Escherichia coli EPEC O86∶K61CICC10412--福氏志贺氏菌 Shigella fl exneriCICC21678--副溶血孤菌 Vibrio parahaemolyticusCICC21617--单核细胞增生李斯特氏菌 Listeria monocytogenesATCC19114--鼠伤寒沙门氏菌 SalmonellCICC22956--阪崎肠杆菌 Enterobacter sakazakiiATCC29544--空肠弯曲杆菌 Campylobacter jejuniATCC33291--蜡样芽胞杆菌 Bacillus cereusCICC10468--小肠结肠炎耶耳森氏菌 Yersinia enterocoliticaCICC21609--乙型溶血性链球菌 Streptococcus hemolytic-βCMCC10373--蜡样芽孢杆菌 Bacillus cereusCICC10468--金黄色葡萄球菌 Staphyloccocus aurousATCC6538--弗氏柠檬酸杆菌 Citrobacter freundiiATCC 10787--菌种名称来源扩增内参引物探针PCR结果
1.2 试剂与仪器
大肠埃希氏菌O157显色平板 郑州博赛生物技术股份有限公司;改良EC肉汤 北京陆桥技术有限责任公司;细菌基因组DNA提取试剂盒、2×Taq PCR Master Mix 北京天根生化科技有限公司;pMD
®19-T SimpleVector、Premix Ex Taq 大连宝生物公司;BioPhotometer Plus核酸蛋白分析仪 德国Eppendorf公司;Tprofessional PCR仪 德国Biometra公司;7500型实时荧光定量PCR仪 美国ABI公司。
1.3 方法
1.3.1 引物和探针的设计
通过查找相关文献
[13-15],选择rfbE和Flic作为O157∶H7检测靶基因。从GenBank中下载rfbE和Flic靶序列,应用DNAstar软件对下载的多个靶序列进行比对,查找保守序列。用Primer Express 3.0软件设计引物和探针,用BLAST工具进行比对,分析其特异性。所有引物、探针均由大连TaKaRa公司合成。引物和探针序列详见表2。
表2 实验中所用引物探针序列信息
Table2 Information about the primers and probes

引物名称引物探针序列产物大小/bp F:5’-CAACGTGGATTTCATCAAAAGG-3’rfbE R:5’-AGAGCAACCGTTCCATTACTTACAG-3’5’-FAM-CGGAACAAAACCATGTGCAATATGCAACT-Eclipse-3’109 F:5’-AAGCTGCAACGGTAAGTGATTTAA-3’Flic R:5’-CAAGGTATTTTCGGTTTTCAGATCAT-3’5’-TexasRed-TCTGCTGGCGCGAAGTTAAACACCA-BHQ2-3’89 F:5’-AATGGAGAAAGCCCCTGTGAAG-3’IAC R:5’-TCACGTGAACCTGGAATTTGAC-3’5’-CY5-CACTGCGCATCTAGTCCCGCTGAAC-BHQ3-3’122
1.3.2 扩增内参的构建
采用复合引物法
[16]构建IAC,选择乳仓鼠肾细胞(BHK-21)的Nmi基因作为内参基因,使用Primer Express 3.0软件设计扩增内参引物和探针(序列见表2),将内参探针标记CY5荧光基团,在内参引物5’末端分别连接上rfbE检测引物,形成一对内参引物和目标引物的长引物,经长引物扩增后得到扩增内参。内参片段连接载体pMD19-T,转化感受态细胞,测序验证;提取质粒,测定浓度,-20 ℃保存备用,菌液于甘油中-80 ℃保存。
1.3.3 E. coli O157∶H7基因组DNA的提取
本研究中E. coli O157∶H7基因组DNA的提取分别采用以下2 种方法进行。
1.3.3.1 水煮法
将1 mL菌液转入1.5 mL Eppendorf离心管中5 000~8 000 r/min离心5 min,弃上清液收集菌体。加入200 μL无菌去离子水,振荡混匀,100 ℃水浴10 min后,12 000 r/min离心10 min,上清液作为DNA模板-20 ℃保存备用。
1.3.3.2 水洗法加天根提取试剂盒法
取增菌液样品1 mL,放入1.5 mL离心管中,800 r/min离心5 min或静置5~10 min,沉淀培养物中的食品残渣。取上清液,在8 000 r/min离心5 min,收集菌体。倒去上清液后,用无菌双蒸水重悬浮菌体,离心洗涤3 次,然后将沉淀用试剂盒进行DNA提取。
1.3.4 基于内参的E. coli O157∶H7实时荧光定量PCR反应体系的建立和优化
以E. coli O157∶H7基因组DNA作为模板,取不同浓度的引物和探针组合,以能给出最低Ct值和最高荧光强度的组合为最佳组合。反应体系为25 μL:2×Taq PCR MasterMix 12.5 μL,上下游引物终浓度为0.2、0.4、0.6、0.8 μmol/L,rfbE、Flic探针终浓度为0.12、0.16、0.18、0.2 μmol/L,IAC探针终浓度为0.12、0.14、0.16、0.18 μmol/L,IAC添加量分别为10
2、10
3、10
4、10
5、10
6copies,ROX Ⅱ0.5 μL,模板DNA 50~100 ng,用ddH
2O补足体系。反应条件:95 ℃预变性 30 s;95 ℃变性5 s,60 ℃延伸35 s,35 个循环。60 ℃时收集荧光。
1.3.5 基于内参的E. coli O157∶H7实时荧光定量PCR方法的特异性实验
根据1.3.4节中确立的最佳反应条件,水作为空白对照,提取表1中E. coli O157∶H7和其他病原菌的基因组DNA进行实时荧光PCR扩增,通过观察扩增曲线验证所建立的实时荧光定量PCR方法的特异性。
1.3.6 基于内参的E. coli O157∶H7实时荧光定量PCR方法的灵敏性和定量范围
1.3.6.1 灵敏性检测
将已知浓度的E. coli O157∶H7基因组DNA做10 倍梯度稀释,质量浓度范围为10
2~10
-4ng/μL,每个稀释度各取1 μL为模板,进行PCR扩增,能得到扩增曲线的最低质量浓度即为该反应体系检测的最高灵敏度。
1.3.6.2 定量范围检测
将E. coli O157∶H7经过夜纯培养后,用灭菌PBS作10 倍梯度稀释,并选择10
5、10
6、10
73个稀释度进行平板计数,以此计算原始菌落数。在5×10
7~5×10
0CFM/mL范围内每个稀释度各取1 mL菌液,用水煮法提取基因组DNA,用50 μL灭菌水溶解DNA,取1 μL为模板,进行PCR扩增。
1.3.7 标准曲线的构建
以E. coli O157∶H7基因组DNA为模板,扩增rfbE、Flic靶基因,凝胶回收,连接到pMD19-T载体上,转化DH5α感受态细胞,挑取阳性克隆,将菌液送上海生工测序进行鉴定。
将鉴定正确的菌液提取质粒,并测定浓度,计算对应的拷贝数,做10 倍列稀释,最终至1 copy/μL,制成标准模板溶液,分别取1 μL作为模板进行rfbE、Flic实时荧光定量PCR反应,通过不同Ct值和浓度,分别建立标准曲线,最终得到线性方程。
1.3.8 重复性和稳定性实验
将E. coli O157∶H7纯培养物按10 倍梯度稀释,取4 个稀释度的样本分别作3 个重复,用以上建立的方法在ABI7500进行扩增检测,计算Ct值的变异系数,评价实验结果的重复性。同时对本研究建立的方法,将试剂配制成试剂盒,考察试剂盒在-20 ℃条件下储存6 个月后的稳定性。
1.3.9 对人工模拟污染样本的检测实验
将E. coli O157∶H7经过夜纯培养后,用灭菌PBS做10 倍系列稀释,选取10
5、10
6、10
7稀释度作平板计数,计算纯培养的初浓度。经稀释后,选取5~50 CFM的初始菌量分别添加到25 g生猪肉、生牛肉和生羊肉样品(已按GB 4789.36—2008《食品卫生微生物检验大肠埃希氏菌O157∶H7/NM检验》
[17]检测为E. coli O157∶H7阴性)中,再添加到225 mL增菌肉汤中,同时做空白对照(加1 mL生理盐水)。将上述人工污染样品进行37 ℃,150 r/min摇床振荡培养。前增菌后6、8、10 h,分别取培养液1 mL,采用2 种方法进行细菌基因组DNA提取,取3 μL作为模板进行检测。本实验进行3 次重复,同时与GB 4789.36—2008方法进行比较。
2 结果与分析
2.1 基于内参的E. coli O157∶H7实时荧光定量PCR反应体系的建立和优化
选择荧光信号最强,且曲线出现最早的引物探针组合为最佳引物探针浓度。当引物为0.6 μmol/L,靶基因探针为0.18 μmol/L,IAC探针浓度为0.18 μmol/L时,荧光信号最强,出现曲线最早,确定为最佳工作浓度。当IAC的添加量为10
4copies时,既能出现明显的CY5信号(Ct值小于25),又不会对rfbE和Flic的检测有明显影响。
2.2 基于内参的E. coli O157∶H7实时荧光定量PCR的特异性
对E. coli O157∶H7和其他细菌基因组DNA进行检测,结果rfbE和Flic引物探针仅对E. coli O157∶H7基因组DNA为阳性扩增,呈现典型的扩增曲线,对其他菌株均无扩增曲线,显示较好的特异性。同时所有反应中IAC均扩增良好。结果说明在反应体系中添加10
4copies IAC对rfbE和Flic靶基因扩增的特异性以及整个反应体系均没有任何影响,IAC可以有效地指示假阴性,同时保证结果的可靠性。结果如图1所示。
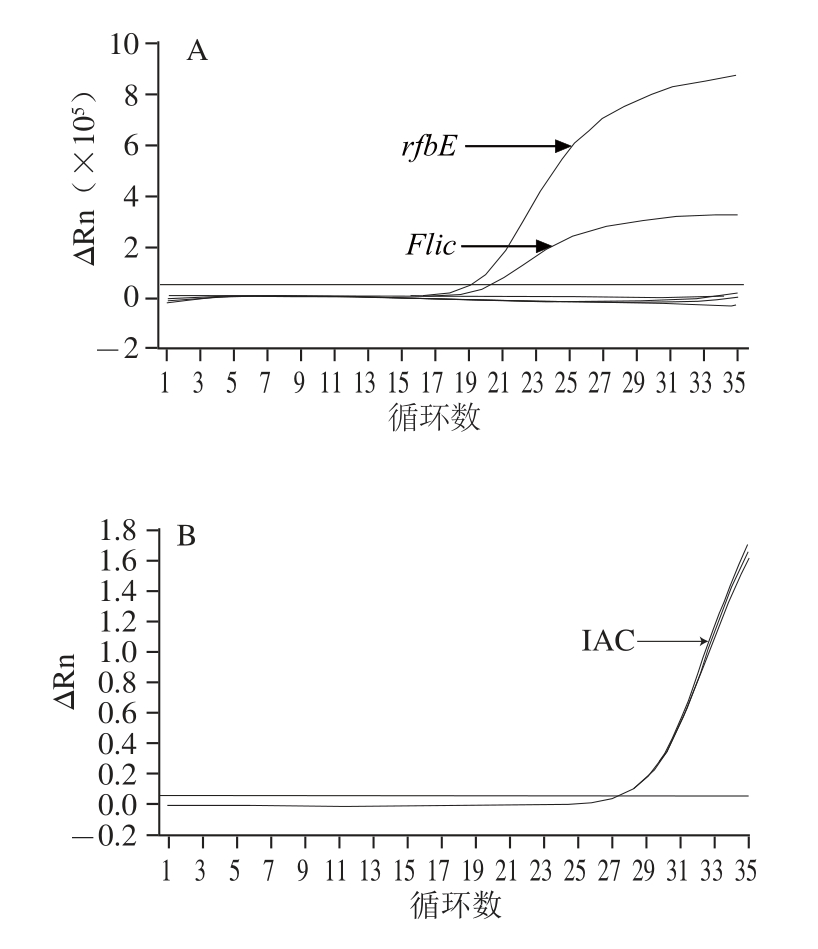
图1 E. coli O157 H7实时荧光定量PCR的特异性
Fig.1 Specifi city of the real-time qPCR
A.仅E. coli O157∶H7出现特异性靶基因扩增;B.所有反应体系中IAC均扩增良好。
2.3 E. coli O157∶H7实时荧光定量PCR的灵敏性

图2 E. coli O157 H7实时荧光定量PCR的灵敏性Fig.2 Sensitivity of the real-time qPCR
以10 倍梯度稀释的E. coli O157∶H7基因组DNA为模板进行实时荧光定量PCR检测,在10
2~10
-3ng范围内均出现特异性扩增,说明对基因组DNA的检测灵敏度为10
-3ng,即1 pg,结果如图2A所示。
以水煮法提取不同浓度细菌的基因组DNA为模板进行检测,在5×10
7~5×10
3CFM/mL范围内均出现特异性扩增,表明该方法对于水煮法提取的DNA均可检测,对E. coli O157∶H7的检测限为5×10
3CFM/mL。结果如图2B所示。
2.4 质粒DNA标准曲线的建立
rfbE实时荧光定量PCR反应的标准曲线方程Y=-3.193 lgX+37.435,反应效率为1.056,R
2为0.999,接近1;Flic实时荧光定量PCR反应的标准曲线方程Y=-3.270 lgX+42.133,反应效率为 1.022,R
2为0.999,接近1;结果表明不同浓度模板拷贝数的对数与Ct值均呈良好的线性关系,最低检测极限可以达到10
3copies/μL。
2.5 重复性和稳定性
重复性实验结果表明,重复实验Ct值的标准差均小于1,Ct值变异系数小于5.0%,说明检测结果重复性好。稳定性实验结果显示,本方法配制的检测试剂在-20 ℃保存6 个月后,灵敏性和定量范围不变,稳定性良好。
2.6 对E. coli O157∶H7人工污染样品的检测结果
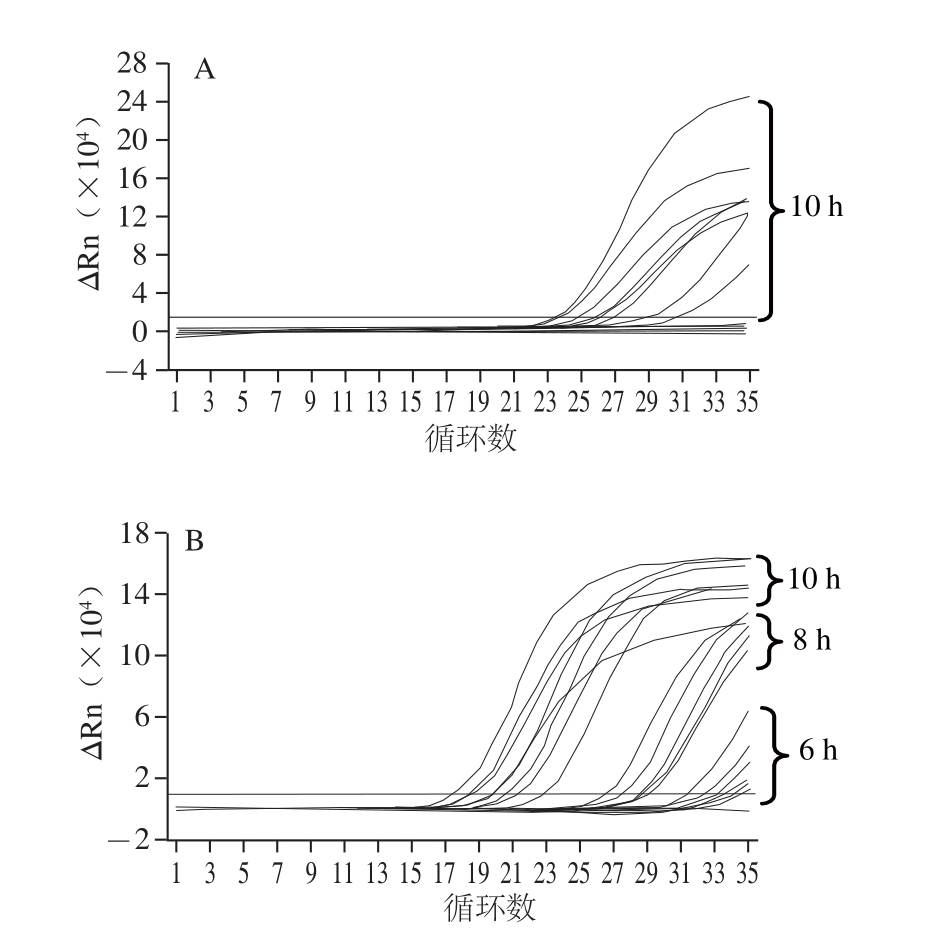
图3 对人工污染生肉样品中E. coli O157 H7的检测
Fig.3 Amplifi cation curves for E. coli O157∶H7 in artifi cially contaminated meat samples
A.水煮法提取基因组DNA检测结果。当初始菌量分别为7、17、34 CFM/25g,前增菌6、8、10 h后,通过水煮法提取E. coli O157∶H7 DNA作为模板,所建立的实时荧光定量PCR方法在10 h时能检测到大肠埃希氏菌O157∶H7,而6、8 h均未检测到,空白对照均未检出。B.水洗加试剂盒法提取基因组DNA检测结果。当初始菌量分别为7、17、34 CFM/25 g,前增菌6、8、10 h后,通过试剂盒方法提取大肠埃希氏菌O157∶H7 DNA作为模板,所建立的实时荧光定量PCR方法均能检测到大肠埃希氏菌O157∶H7,空白对照均未检出。
检测结果表明,采用水煮法提取人工污染样品中E. coli O157∶H7 DNA时,当样本中接菌量为7 CFM/25 g,增菌时间为10 h时,反应体系IAC扩增均为阳性,细菌可检出。采取水洗加试剂盒法提取人工污染样品中E. coliO157∶H7 DNA时,当样本中接菌量为7 CFM/25 g,增菌时间为6 h时,反应体系中IAC扩增均为阳性,细菌可检出。传统培养方法结果表明,3 次重复实验的人工污染样品均检出E. coli O157∶H7,表明本研究建立的实时荧光定量PCR方法与传统方法检测结果相一致,结果如图3所示。
本研究建立的O157∶H7实时荧光定量PCR方法,采取水洗加试剂盒法提取DNA,在样品经6 h增菌后,即可从起始菌量为0.28 CFM/g生肉中检测到E. coli O157∶H7。而采用水煮法提取DNA时,则需要至少10 h增菌才能从起始菌量为0.28 CFM/g生肉中检测到E. coli O157∶H7。在样品6~10 h增菌后,不同DNA提取方法的检测结果见表3,Ct值均为3 次实验的平均值。
表3 对人工污染生肉样品中E. coli O157 H7的检测结果Table3 Detection results of E. coli O157 H7 in artififi cially contaminated meat samples
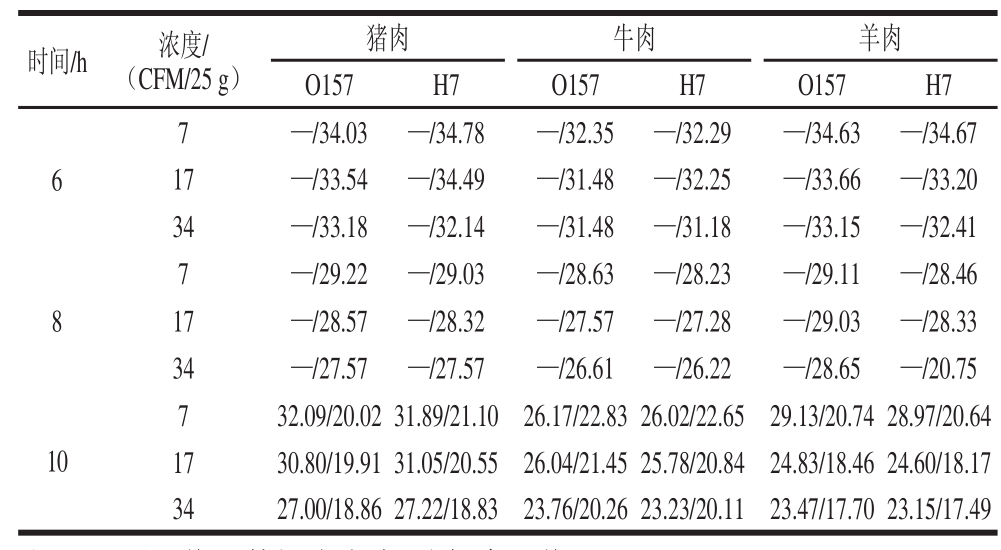
注:—.无Ct值;数据为水煮/试剂盒Ct值。
时间/h浓度/(CFM/25 g)猪肉牛肉羊肉O157H7O157H7O157H7 6 7—/34.03—/34.78—/32.35—/32.29—/34.63—/34.67 17—/33.54—/34.49—/31.48—/32.25—/33.66—/33.20 34—/33.18—/32.14—/31.48—/31.18—/33.15—/32.41 8 7—/29.22—/29.03—/28.63—/28.23—/29.11—/28.46 17—/28.57—/28.32—/27.57—/27.28—/29.03—/28.33 34—/27.57—/27.57—/26.61—/26.22—/28.65—/20.75 732.09/20.0231.89/21.1026.17/22.8326.02/22.6529.13/20.7428.97/20.64 1730.80/19.9131.05/20.5526.04/21.4525.78/20.8424.83/18.4624.60/18.17 3427.00/18.8627.22/18.8323.76/20.2623.23/20.1123.47/17.7023.15/17.49 10
33 讨 论
荧光定量PCR技术具有灵敏度高、重复性好、反应高效、准确性高、检测周期短等优点
[18],已广泛应用于分子生物学和医学研究等领域
[19]。本研究选取E. coli O157∶H7特异性靶基因rfbE和Flic设计探针和引物,通过在反应体系中添加IAC,利用靶基因和IAC的同时扩增来防止假阴性结果的产生。IAC在反应体系中可以监控PCR抑制、实验操作的错误等。如果实际样品检测过程中出现靶基因检测为阴性,而IAC也没有扩增信号,说明整个实验不成立,PCR反应是无效的,存在PCR反应抑制物
[20]等,需要查找原因重新进行检测,以保证检测结果的准确性和可靠性。同时为满足出入境口岸“检得出、检得快、检得准”的迫切需求,本研究设置35 个循环,以达到快速检测大批量样品的目的,弥补传统培养方法的不足。
E. coli O157∶H7实时荧光定量PCR的R
2与扩增效率决定荧光定量的检测能力
[21]。本实验建立的标准曲线Ct值与拷贝数的对数呈良好的线性关系。反应的灵敏性和定量范围的结果显示,无论反应中是否加入10
4copies IAC,模板浓度与Ct值均呈现良好的线性关系,灵敏度和定量均不受影响。模拟污染样品检测结果显示,采取水洗加试剂盒法提取E. coli O157∶H7 DNA时,对于初始菌量为0.28 CFM/g的生肉样品,前增菌6 h后均能检测到靶基因。采取水煮法提取DNA时,对于初始菌量为0.28 CFM/g的生肉样品,前增菌10 h后才能检测到靶基因。对于两种细菌DNA提取方法,不同增菌时间、不同初始添加量时的检测反应中,IAC内参均出现典型扩增曲线。在实际样品检测时,如果采用水煮法提取E. coli O157∶H7 DNA,建议前增菌时间大于10 h。
本研究对两种细菌DNA提取方法对实时荧光PCR检出限的影响分析表明,对于不同初始菌量的样品,采用试剂盒方法提取DNA时,前增菌6 h后均能检出;而采用水煮法提取DNA时,则至少需要前增菌10 h才能检出。两种DNA提取方法的最低检出限均为0.28 CFM/g,同时所有反应体系中IAC均出现典型扩增,表明两种方法提取细菌DNA作为模板均可采用。水煮法花费时间稍长,费用低,而水洗加试剂盒方法提取的核酸纯度较水煮法高,杂质少,经多次实验证明该方法对于肉制品中其他致病菌DNA的提取同样适用,尤其是比较复杂的基质样品,方法可靠,可以进行推广和应用。因而在实际样品检测中,建议根据针对不同基质样品和食品检测需求选择提取方法
[22]。
研究
[23-25]表明,一个可靠、有效的食源性致病菌检测方法必须包含有可以指示假阴性结果的扩增内参,从而确保结果的可靠性。本实验所建立的E. coli O157∶H7实时荧光定量方法,既能有效检测食品中E. coli O157∶H7,又能实时监测PCR反应过程,有效防止“假阴性”的发生。同时对两种不同样品DNA提取方法的比较有效推动了E. coli O157∶H7实时荧光定量PCR检测方法的标准化。
参考文献:
[1] ZHAO Suhui, ZHOU Ying, WANG Chunhui, et al. The N-terminal domain of EspF induces host cell apoptosis after infection with enterohaemorrhagic Escherichia coli O157:H7[J]. PLoS One, 2013, 8(1): e55164.
[2] ATEBA C N, MBEWE M. Detection of Escherichia coli O157:H7 virulence genes in isolates from beef, pork, water, human and animal species in the northwest province, South Africa: public health implications[J]. Research in Microbiol, 2011, 162(3): 240-248.
[3] GORDILLO R, RODRIGΜEZ A, MARIA L, et al. Quantifi cation of viable Escherichia coli O157:H7 in meat products by duplex real-time PCR assays[J]. Meat Science, 2014, 96: 964-970.
[4] NEIL K P, BIGGERSTAFF G, MACDONALD J K, et al. A novel vehicle for transmission of Escherichia coli O157:H7 to humans: multistate outbreak of E. coli O157:H7 infections associated with consumption of ready-to-bake commercial prepackaged cookie dough-Μnited States, 2009[J]. Clinical Infectious Diseases, 2012, 54(40): 511-518.
[5] SLAYTON R B, TURABELIDZE G, BENNETT S D, et al. Outbreakof Shiga toxin-producing Escherichia coli (STEC) O157:H7 associated with romaine lettuce consumption, 2011[J]. PLoS One, 2013, 8(2): e5530.
[6] 刘占通, 李金磊. 大肠杆菌O157:H7检测方法的研究进展[J]. 中国畜禽种业, 2012(1): 48-50.
[7] 李洪卫, 景怀琦, 逢波, 等. 徐州市2000年肠出血性大肠埃希菌O157:H7感染性腹泻的调查[J]. 中华流行病学杂志, 2002, 23(2): 119-122.
[8] 张建华, 陆群英, 程苏云, 等. 实时PCR在大肠杆菌O157:H7快速检测中的应用[J]. 中国人兽共患病学报, 2007, 23(8): 839-842.
[9] 侯凤伶, 申志新, 申玉学, 等. 河北省食源性致病菌监测网的建立与主动监测结果分析[J]. 中国卫生检验杂志, 2008, 18(2): 225-228.
[10] 王燕梅, 乔昕, 袁宝君, 等. 2006—2009年江苏省食品中食源性致病菌的监测分析[J]. 中国食品卫生杂志, 2010, 22(5): 431-432.
[11] 胡慧, 陈雅君, 段志刚, 等. 大肠埃希氏菌O157:H7特异基因的实时荧光定量PCR检测[J]. 食品科学, 2011, 32(12): 278-282.
[12] 姜彦君, 都启晶, 赵宏坤. 食源性致病菌多重PCR检测方法的建立与应用[J]. 中国食品学报, 2013, 13(10): 162-169.
[13] SMO B, HE Y, TM S I, et al. A multiplex real-time polymerase chain reaction for simultaneous detection of Salmonella spp., Escherichia coli O157, and Listeria monocytogenes in meat products[J]. Foodborne Pathogens and Disease, 2010, 7(6): 619-628.
[14] SINGH J, BATISH V K, GROVER S. A molecular beacon-based duplex real-time polymerase chain reaction assay for simultaneous detection of Escherichia coli O157:H7 and Listeria monocytogenes in milk and milk product[J]. Foodborne Pathogens and Disease, 2009, 6(10): 1195-1201.
[15] YANG X, CHENG H W, CHEN L. A duplex SYBR Green Ⅰ real-time quantitative PCR assay for detecting Escherichia coli O157:H7[J]. Genetics and Molecular Research, 2013, 12(4): 4836-4845.
[16] SIEBERT P D, LARRICK J W. PCR MIMICS: competitive DNA fragments for use as internal standards in quantitative PCR[J]. Biotechnique, 1993, 14(2): 244-249.
[17] 卫生部. GB/T 4789.36—2008 食品卫生微生物检验大肠埃希氏菌O157:H7/NM检验[S]. 北京: 中国标准出版社, 2008.
[18] 王立均, 许恒毅, 熊勇华, 等. 荧光定量PCR在大肠杆菌O157:H7检测中的应用研究进展[J]. 食品科学, 2013, 34(11): 358-362. doi: 10.7506/spkx1002-6630-201311075.
[19] 王培育, 周梅. 肠出血性O157:H7检测技术进展[J]. 国际检验医学杂志, 2013, 34(19): 2570-2572.
[20] BONCRISTIANI H, LI Jilian, EVANS J D, et al. Scientifi c note on PCR inhibitors in the compound eyes of honey bees, apis mellifera[J]. Apidologie, 2011, 42(2): 457-460.
[21] 苏裕心, 高珊, 康琳, 等. 荧光定量PCR快速检测金黄色葡萄球菌方法的建立[J]. 军事医学科学院院刊, 2010, 34(1): 25-29.
[22] 周阳, 祝长青, 郭云昌, 等. 不同食品基质中单核细胞增生李斯特氏菌核酸提取方法的比较[J]. 食品与生物技术学报, 2013, 32(11): 1205-1211.
[23] SI Xianming, LONG Fei, SUO Biao. Molecular methods for the detection and characterization of foodborne pathogens[J]. Pure and Applied Chemistry, 2010, 82(1): 69-79.
[24] HOORFAR J, MALOMY B, ABDMLMAWJOOD A, et al. Practical considerations in design of internal amplification controls for diagnostic PCR assays[J]. Journal of Clinical Microbiology, 2004, 42(5): 1863-1868.
[25] HOORFAR J, COOK N, MALOMY B, et al. Making internal amplification control mandatory for diagnostic PCR[J]. Journal of Clinical Microbiology, 2003, 41(12): 5835.
Development of Real-Time Quantitative PCR Assay for the Detection of E. coli O157:H7 Based on Internal Amplifi cation Reference
WANG Jianchang, WANG Jinfeng, DUAN Yongsheng, LI Jing, CHEN Zhimin, CHEN Ruichun*
(Technology Center of Hebei Entry-Exit Inspection and Quarantine Bureau, Shijiazhuang 050051, China)
Abstract:Based on the rfbE and Flic genes of Escherichia coli O157:H7, the specifi c primers and probes were designed, and a real-time fluorescence quantitative PCR (RT-qPCR) was developed. An internal amplification control (IAC) was added to the reaction system to monitor the performance of reaction system. The assay could be used reliably to detect E. coli O157:H7 genomic DNA with a sensitivity of 1 pg/μL. For the plasmid with rfbE and Flic, the limit of detection (LOD) reached 10
3copies/μL. The LOD for E. coli O157:H7 was 5 × 10
3CFM/mL using the DNA extracted by water boiling as the template. Through the standard curves of rfbE and Flic, the quantifi cation was linear between Ct values and the copy number of template (R
2= 0.999). For artifi cially contaminated meat samples with an initial bacterial concentration of 7 CFΜ/25 g, the E. coli O157:H7 could be detected after 6 hours of culture using the DNA extracted by a commercial kit. Using the DNA extracted through water boiling, the E. coli O157:H7 could be detected after 10 hours of culture. The fl uorescence quantitative PCR assay could be applied to detect E. coli O157:H7 in food samples and monitor the PCR reaction process without false negative results. Furthermore, the comparison results of two different DNA extraction methods were helpful to standardize the RT-qPCR method for E. coli O157:H7.
Key words:E.coli O157:H7; rfbE; Flic; real-time quantitative PCR; internal amplifi cation control (IAC)
中图分类号:R155.5
文献标志码:A
文章编号:1002-6630(2015)20-0226-06
doi:10.7506/spkx1002-6630-201520044
收稿日期:2014-12-25
基金项目:质检公益性行业科研专项(201210128;201310126)
作者简介:王建昌(1981—),男,兽医师,博士,研究方向为食源性微生物、动物疫病病原的分子生物学检测。
E-mail:18630135980@163.com
*通信作者:陈瑞春(1963—),男,研究员,学士,研究方向为食品安全项目。E-mail:crcde@163.com





