生鲜家畜肉类与水产鱼虾类物流过程中微生物菌相变化的研究进展
杨 超,车 有,宋存江*
(南开大学生命科学学院,分子微生物与技术教育部重点实验室,天津 30007 1)
摘 要:生鲜家畜肉类及水产鱼虾类物流过程中的腐败变质主要 由微生物引起。随着分子生物学技术的快速发展,分子生态技术已成为研究生鲜家畜肉类及水产鱼虾类物流过程中微生物菌相变化的主要手段。引起生鲜家畜肉类及水产鱼虾类在物流过程中腐败变质的微生 物也不断被发现。本文综述了生鲜家畜肉类及水产鱼虾类物流过程中常见微生物物种,包括肉食杆菌、乳酸菌、沙门氏菌属、假单胞菌属、明串珠菌属、热杀索丝菌、单增李斯特氏菌等细菌以及酵母菌、霉菌等真菌;介绍了2010年后多用于生鲜家畜肉类和水产鱼虾类菌相研究的分子生态技术,包括变性梯度凝胶电泳技术、末端限制性片段长度多态性技术、高通量测序技术和实时定量聚合酶链式反应技术,旨在为我国生鲜家畜肉类及水产鱼虾类的菌相研究 提供参考。
关键词:生鲜家畜肉类;水产鱼虾类;腐败变质;微生物菌相;分子生态技术
生鲜家畜肉类和水产鱼虾类作为生鲜肉类和水产品重要的组成部分,是我国居民日常消费的主要食品之一。我国是世界生鲜肉类和水产类第一生产大国
[1],但由于国内物流保鲜技术尚不完善,目前我国生鲜产品在物流过程中的腐败率超过35%,其中肉类和鱼类产品腐败率约占15%
[2],难以满足国民对食品质量的要求。为此,提高生鲜家畜肉类及水产鱼虾类在物流过程中的保鲜技术已迫在眉睫。在物流过程中,微生物的繁殖是引起生鲜家畜肉类及水产鱼虾类腐败变质的根源。因此,开展生鲜家畜肉类及水产鱼虾类中微生物的菌相研究对于生鲜家畜肉类及水产鱼虾类物流过程中保鲜技术的研究具有重要意义。
1983年Blickstad
[3]和Dainty
[4]等采用传统微生物培养的手段,对冷藏肉类中的微生物进行富集培养、筛选,在冷藏保鲜猪肉中筛得大量乳酸菌类。随后,Gill
[5]、Church
[6]和Borch
[7-8]等在冷藏保鲜的猪肉中分离得到了肉食杆菌(Carnobacterium)、乳杆菌(Lactobacillus)和热杀索丝菌(Brochothrix thermosphacta), 初步探究了生鲜家畜肉类及水产鱼虾类中微生物的物种。然而,采用传统微生物培养技术会受到培养条件的限制,并不能展示食物中真正的菌相分布。不同微生物对不同营养物质的偏好性不同、微生物间的竞争抑制、培养温度以及溶氧条件等诸多因素导致培养基中适宜生长的微生物数量增加,而不适宜培养的微生物最终减少直至消亡;同时,采用传统微生物培养策略,只能对食物中数量占优势的微生物进行鉴定,这些问题已成为菌相分析的技术难题。
近年,随着分子生态技术的发展,微生物的分析手段日益完善。特别是在食品微生物领域,以DNA指纹技术
[9]为基础所演生的分子生态技术被使用之后,生鲜家畜肉类和水产鱼虾类中微生物菌相分布研究取得了长足进步。Zhao Fan等
[10]利用高通量测序技术监测猪肉腐败过程中的菌相变化,发现了微球菌科(Micrococcaceae)、乳杆菌科(Lactobacillaceae)等微生物参与腐败过程,并在腐败末期检测到77%的微生物来自厚壁菌门(Firmicutes)。这表明利用灵敏度更高的分子生态技术不仅可以检测到传统培养法难以发现的微生物物种,同时亦可监测微生物的丰度水平,推动了该领域的研究发展。
为了促进分子生态技术在该领域的进一步发展,本文综述了近年利用分子生态技术在生鲜家畜肉类和水产鱼虾类物流过程中发现的常见微生物物种,并介绍了该领域常用的变性梯度凝胶电泳技术、末端限制性片段长度多态性分析技术和实时定量聚合酶链式反应(polymerase chain reaction,PCR)技术。此外,随着测序技术的快速发展,一种能够对微生物基因组测序并自动鉴定菌种的MicroSeq
[
11]微生物鉴定平台应运而生,使得食品微生物菌相分布的研究更加全面,自动化程度也大幅提高。
1 生鲜家畜肉类和水产鱼虾类物流过程中腐败微生物菌相分布的研究进展
1.1 生鲜家畜肉类物流过程中微生物的菌相分布
生鲜家畜肉类通常在0~4 ℃条件下运输或采用冰点以下的微冻技术长期冷藏
[12]。在该领域研究初期
[6-7,13],真空冷藏保鲜的肉类中以肉食杆菌、乳杆菌、明串珠菌(Leuconostoc spp.)、热杀索丝菌为肉类腐败的主要菌群。随着研究技术水平的提高,更多的微生物在生鲜家畜肉类中被发现,微球菌科、黄杆菌科(Flavobacteriaceae)、肠杆菌科(Enterobacteriaceae)、乳杆菌科和肉杆菌科(Carnobacteriaceae)等被鉴定为冷藏保鲜肉类中常见微生物
[14]。此外,由于屠宰加工环境及保藏条件的限制,一些常见微生物也经常在生鲜家畜肉类研究中被报道。如金黄色葡萄球菌(Staphylococcus aureus)
[15]、蜡状芽孢杆菌(Bacillus cereus)
[15]、沙门氏菌属(Salmonella spp.)
[15-16]、单增李斯特氏菌(Listeria monocytogenes)
[15-18]和假单胞菌属(Pseudomonas spp.)
[14,19]等均为生鲜家畜肉类中的常见微生物,是肉制品卫生安全的检测指标。除了细菌,多种真菌
[20-21]和酵母菌类
[21]微生物也在生鲜家畜肉类腐败过程中被检测到(表1)。
表1 生鲜家畜肉类及水产鱼虾类中常见致腐菌属及菌种
Table 1 Common genus and species of microorganism in fresh livestock meat and aquatic fish and shrimp during logistics
中文名 拉丁名 食物种类 保藏方式 参考文献肉食杆菌 Carnobacterium spp. 猪肉 托盘包装、真空包装 [7,10,13-14]热杀索丝菌 Brochothrix thermosphacta 猪肉、鱼类 托盘 包装、真空包装、气调包装 [7,13-14,22-24]金黄色葡萄球菌 Stap hylococcus aureus 猪肉、牛肉、虾类 托盘包装、真空包装 [15,18,25]蜡状芽孢杆菌 Bacillus cereus 猪肉 托盘包装、真空包装 [10,26]单增李斯特氏菌 Listeria monocytogenes 猪肉、鱼类、虾类 托盘包装、真空包装、气调包装 [15,17-18]大肠杆菌 Escherichia coli 猪肉、牛肉 托盘包装、真空包装、气调包装 [19,26-27]乳酸菌属 Lactobacillus spp. 猪肉、牛肉、虾类 托盘包装、真空包装、气调包装 [10,13,19,22,24-26]米酒乳酸杆菌 Lactobacillus sakei 猪肉、牛肉 真空包装 [7,13]弯曲乳酸杆菌 Lactobacillus curvatus 猪肉、牛肉 真空包装 [7,13]冷明串珠菌 Lactobacillus gelidum 猪肉、牛肉 真空包装 [7,13]嗜柠檬酸明串珠菌 Leuconostoc citreum 猪肉、牛肉 真空包装 [7,13]肠膜明串珠菌 Leuconostoc mesenteroides 猪肉、牛肉 真空包装 [7,13]乳酸明串珠菌 Leuconostoc lactis 猪肉、牛肉 真空包装 [5,11]库特氏菌属 Kurthia spp. 猪肉 真空包装 [10]不动杆菌属 Acinetobacter spp. 猪肉、鱼类 托盘包装 [28]棒状杆菌属 Coryneforms spp. 猪肉 真空包装 [10]希瓦氏菌属 Shewanella spp. 猪肉、鱼类 托盘包装、真空包装、气调包装 [23,28]木糖氧化无色杆菌 Achromobacter xylosoxidans 猪肉 真空包装 [10]沙门氏菌属 Salmonella spp. 猪肉 托盘包装、气调包装 [16,26]鼠伤寒沙门氏菌 Salmonella typhimurium 猪肉 托盘包装、气调包装 [29]肠炎沙门氏菌 Salmonella enteritidis 牛肉 托盘包装 [15]粘质沙雷氏菌 Serratia marcescens 猪肉 托盘包装 [14]假单胞菌属 Pseudomonas spp. 猪肉、牛肉、鱼类、虾类托盘包装、真空包装、气调 包 装 [7,10,14,19,22-23]荧光假单胞菌 Pseudomonas fuorescens 猪肉 托盘包装、气调包装 [14,19]梭杆菌属 Fusobacteria spp. 鱼类 托盘包装 [30]产气荚膜梭菌 Clostridium perfringens 猪肉 托盘包装 [26]曲霉属 Aspergillus spp. 猪肉、鱼类 托盘包装、真空包装、气调包装 [20-21,31]法夫红酵母 Xanthophyllom yces dendrorhous 猪肉、鱼类 托盘包装、真空包装、气调包装 [20-21,31]解脂耶氏酵母 Yarrowia lipolytica 猪肉、鱼类 托盘包装、真空包装、气调包装 [20-21,31]根霉属 Rhizopus spp. 猪肉、鱼类 托盘包装、真空包装、气调包装 [20-21,31]青霉属 Penicillium spp. 猪肉、鱼类 托盘包装、真空包装、气调包装 [20-21,31]
物流过程中肉类一旦发生腐败,肉类中的微生物组成也将发生动态变化。Fontana等
[13]发现在真空冷藏保鲜的牛肉中,保藏初期以米酒乳酸杆菌(Lactobacillus sakei)为主要菌群,随着保藏时间的推移,牛肉逐渐腐败变质,米酒乳酸杆菌逐渐消失,取而代之的是弯曲乳酸杆菌(Lactobacillus curvatus)、明串珠菌和热杀索丝菌。这表明肉类腐败的不同阶段,其微生物种类和丰度也在发生着动态变化。随着肉类腐败过程中营养物质、pH值等不断变化,保藏初期占优势的微生物其生长遭到抑制,而另一些微生物逐渐成为优势菌群,导致了生鲜家畜肉类中微生物的演替。因此,针对物流过程中肉类腐败的不同阶段采取不同的保鲜手段和使用防腐保鲜剂,将有效延长生鲜家畜肉类的货架期,为肉类保鲜技术和保鲜剂的开发提供参考。
此外,地域和保藏方式的差异也会影响肉类中的菌相分布(表1)。Mihaiu等
[16]在来自罗马尼亚的猪肉中发现沙门氏菌属为猪肉腐败末期菌相中主要致腐菌之一,这与前人报道的假单胞菌或乳酸菌类作为主要腐败菌略有不同。而Coton等
[23]发现在有氧条件下保藏的肉类其腐败末期以热杀索丝菌、假单胞菌属和希瓦氏菌(Shewanella spp.)为主,并发现在厌氧条件下假交替单胞菌(Pseudoalteromonas haloplanktis)也是参与肉类腐败变质的主要菌群之一。生鲜家畜肉类在物流过程中其微生物的菌相分布比较复杂,不同来源、不同保鲜方式的肉类,其菌相分布也各有差异。探究不同保鲜方式的微生物菌相分布,有利于保鲜技术的应用,从而延长生鲜家畜肉类的货架期。
1.2 水产鱼虾类物流过程中的微生物菌相分布
新鲜的水产鱼类、虾类等因捕捞后通常不会进行清洗,其表面、鳃内及消化系统内存在大量微生物,不同环境生长的鱼类其初始微生物菌相分布也大不相同。但经过低温保藏的水产鱼类腐败后,其微生物菌群主要以假单胞菌属、嗜冷菌属(Psychrobacter spp.)、沙雷氏菌属(Serratia spp.)、微杆菌属(Exiguobacterium spp.)、葡萄球菌属(Staphylococcus spp.)为主要菌群(表1)。此外,Noseda等
[24]在真空冷藏的鲶鱼中证实了乳酸菌类(lactic acid bacteria)和热杀索丝菌也是鱼肉腐败末期的主要菌群之一。而Bjornsdottir-Butler等
[32]从保藏的黄鳍金枪鱼、蓝鱼和假长鳍鱼中检测到了美人鱼发光杆菌(Photobacterium damselae)、志贺邻单胞菌(Plesiomonas shigelloides)、希瓦氏菌和摩氏摩根菌(Morganella morganii)等非常见微生物。
相比生鲜家畜肉类,水产鱼虾类物流过程中其微生物物种多样性更为复杂,对保鲜工艺要求更加严格,但其菌相变化规律却有相似之处。Boziaris等
[21]在研究水产变质过程中发现,随着食物pH值的降低,食物中革兰氏阴性菌逐渐减少,而乳酸菌类和酵母菌类微生物逐渐成为优势菌群。这与生鲜家畜肉类中的变化趋势基本一致。因此,针对水产鱼虾类中复杂菌相的潜在优势菌群进行防腐抑制,对延长水产鱼虾类的货架期有着重要指导意义。
2 生鲜家畜肉类和水产鱼类微生物引起腐败变质过程
2.1 生鲜家畜肉类微生物致腐过程
在肉类腐败初期,葡萄糖作为肉中微生物生长繁殖的主要碳源,随着肉中葡萄糖的消耗已不能满足微生物的生长繁殖,微生物开始利用肉中游离的氨基酸等小分子物质作为碳源,此过程会因降解氨基酸而产生酮酸和氨类,而因此产生恶臭味
[33]。肠杆菌属、假单胞菌属、沙门氏菌属和乳酸菌属等都是腐败过程中氨类物质的主要微生物源
[34]。
在肉类腐败中期和末期,此时微生物菌群数量达到一定规模,其分泌的蛋白酶足以降解肉中的蛋白质以进一步增殖,加速肉的腐败变质,肉本身也出现不同程度的腐烂、恶臭、颜色变暗或变绿,其表面也产生黏性物质,甚至滋生霉菌
[33,35]。
2.2 水产鱼虾类微生物致腐过程
水产鱼类腐烂前会因为机体死亡后停止呼吸,导致厌氧条件下糖酵解作用引发乳酸堆积,导致体内pH值下降,此时鱼体僵硬,但鲜度基本没有被破坏
[36]。随后鱼类开始进入腐败阶段,伴随微生物的生长繁殖,鱼体内蛋白质经微生物脱羧酶作用,催化氨基酸脱羧而产生生物胺(盐酸丁二胺、五甲烯二胺、组胺、酪胺等
[36])及小分子碳源开始积累。随着微生物的生长,鱼肉鲜度降低至最终完全腐败。
捕获后的新鲜水产鱼类一般不会进行细致清洗,其表面、鳃内和其他器官内存在着大量微生物,随着各种氨基酸及小分子碳源的增加,这些微生物开始快速繁殖,在降解氨基酸和其他小分子物质的同时产生一种名为“三甲胺”的物质,进而产生腥臭味
[36]。伴随微生物的大量繁殖,微生物侵入各个器官,引发鱼体内蛋白质和氨基酸的降解,从而最终造成水产鱼类的完全腐败,此时鱼已经失去食用营养价值。
3 生鲜家畜肉类及水产鱼虾类腐败研究中常用的微生物菌相分析方法
分子生物学技术通过对生命体进行分子水平的操作,以实现对生命体的改造
[37]。随着分子生物学技术的快速发展,微生物分子生态技术应运而生。微生物分子生态技术是利用分子生物学的技术方法对环境中的微生物进行分子水平的研究,从而了解环境中微生物的功能、多样性及群落结构
[37]。其中DNA指 纹识别技术作为分子生态技术中的基础研究方法,被广泛应用于食品微生物菌相的研究中。DNA指纹技术是一种利用不同个体间DNA片段差异性而对不同个体进行鉴定的分子生物学手段
[14]。细菌16S rDNA、真菌18S rDNA和真核ITS序列在不同微生物中具有特异性,常被用作微生物分类的依据,并可用于检测微生物的相对数量。为了对细菌16S rDNA、真菌18S rDNA和ITS序列进行鉴定,常常用到以下分子生态技术。
3.1 变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)技术
DGGE技术是1979年由Fischer等
[38]最先提出的鉴定DNA突变的一种电泳分离技术,而Muyze等
[39]首次将DGGE技术应用于微生物生态学研究,成为鉴定自然界微生物种群多样性常用的技术手段。
DGGE技术可以将长度相同而碱基不同的双链DNA片段(即突变序列)进行分离。利用双链DNA序列碱基排列方式影响解链温度的原理,将双链DNA片段在含梯度变性剂的聚丙烯酰胺凝胶中进行电泳,当双链DNA片段迁移到梯度凝胶一定距离且达到解链温度后,开始部分解链
[40]。DNA分子解链程度越小其迁移程度越大,从而使解链程度不同的双链DNA分子分离,获得纯合的DNA片段
[40]。为了在电泳过程中保证DNA双链不完全分离,DNA片段进行PCR扩增时,其5'端会被引入一段30~50 bp的富含G/C碱基的“GC夹板”(GC-clamp),以保证DNA片段在梯度凝胶电泳时能够充分分离
[40]。
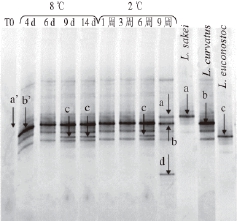
图1 利用DGGE技术鉴别真空冷藏牛肉中的菌相分布
[1133]
Fig.1 Bacterial community of vacuum-packaged beef evaluated by DGGE fingerprint
[13]
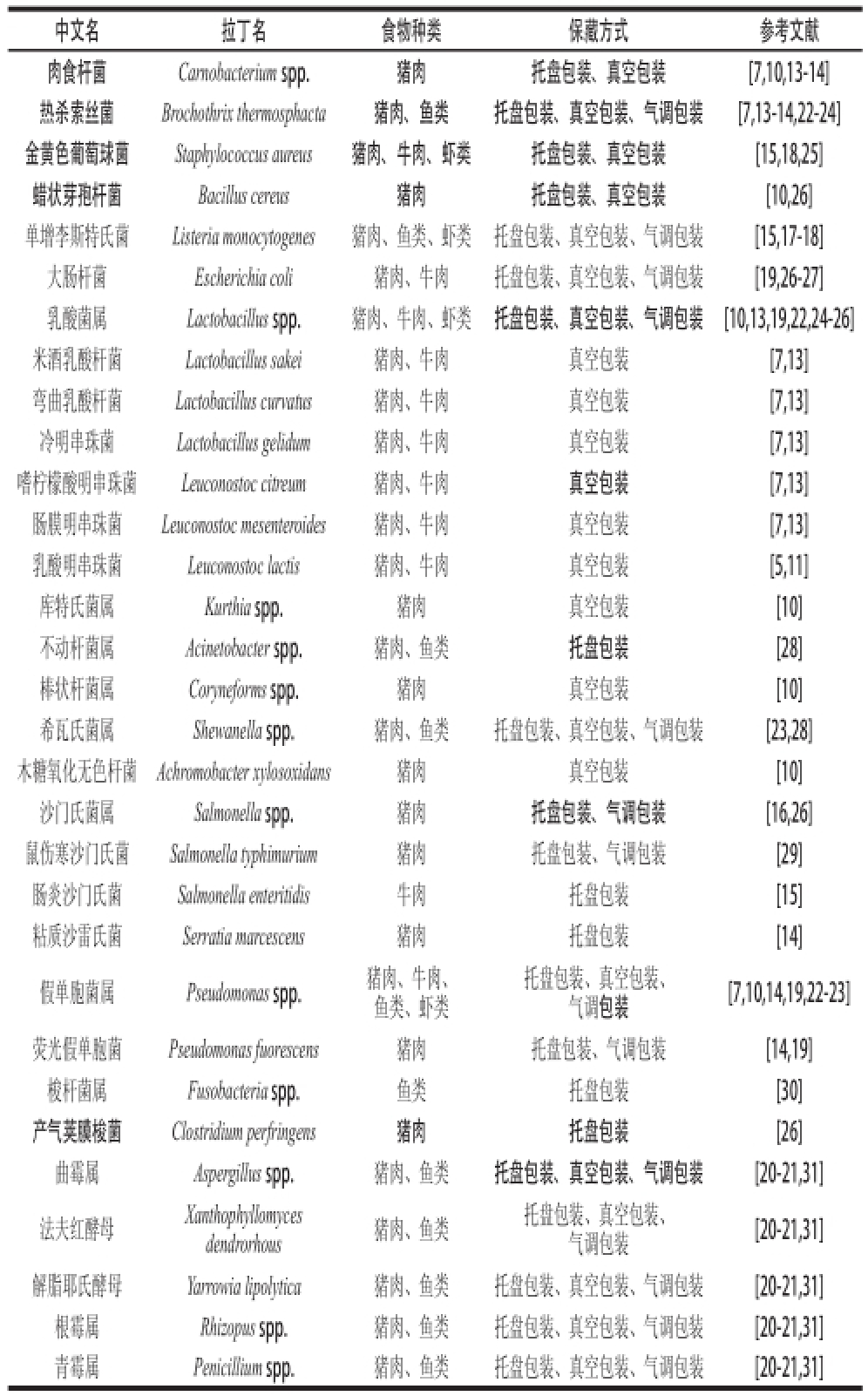
在生鲜家畜肉类和水产鱼虾类中,DGGE技术已被广泛应用于微生物菌相的多样性分析以及微生物菌相动态变化的监测。Fontana等
[13]首次采用DGGE技术,确定了保藏末期真空冷藏牛肉中主要微生物为乳酸菌类。Broekaert等
[28]则在冷冻保藏的水产鱼类中检测到培养基未能培养出的霉实假单胞菌(Pseudomonas fragi)和不动杆菌属(Acinetobac ter spp.)。DGGE技术作为分子生态学最基本的手段,已成为食品微生物菌相变化研究的常用手段。然而,尽管DGGE技术克服了传统培养基培养法不能反映食物中真实菌相分布的缺点,但该方法还存在一定局限性。一些稀有微生物的基因组含量较低,采用DGGE技术鉴定的结果(图1)只能反映优势微生物(清晰较深的条带)的分布状态,也不能将生鲜家畜肉类和水产鱼虾类中活菌和死菌的分布状态进行区分。因此,该方法只能用来定性分析生鲜家畜肉类和水产鱼虾类总体菌相变化,不能鉴定出全部微生物物种。
3.2 末端限制性片段长度多态性(terminal restriction fragment length polymorphism,TRFLP)技术
TRFLP技术结合荧光标记技术、DNA限制酶切技术和DNA自动测序仪技术,能够对特定核酸片段长度多态性进行测定,来鉴别环境中微生物的群落结构和功能
[41]。
TRFLP技术在目标基因保守区扩增时,在5'端引入荧光标记序列(HEX、TET、6-FAM等)
[42]。根据不同物种DNA保守区序列酶切位点不同的原理,对扩增后的保守区进行酶切,用荧光测序仪对带有荧光标记的不同长度的酶切产物进行检测
[43]。通过荧光测序仪所收集的荧光信息及生成的TRFLP图谱分析,便可分析得到微生物群落结构的物种种类、数量,确定微生物群落的结构变化
[37]。
TRFLP技术方便快捷,相比DGGE技术更加灵敏方便。Kiermeier等
[44]采用TRFLP和454测序技术,确定猪肉在真空和100% CO
2保藏条件下微生物菌相随保藏时间推移并不相同。Smith等
[45]则利用TRFLP在牙鳕表面检测到了发光细菌的存在。与DGGE技术相比,TRFLP技术可以定量检测微生物的相对含量(图2),可定量监测微生物的变化规律。

图2 利用TRFLP峰值数据对不同时期真空条件和110000% CCOO
2条件保藏的羊肉菌相进行典型相关分析
[4444]
Fig.2 Canonical analysis of principal coordinates of TRFLP peak data from lamb samples packaged in vacuum and 100% CO
2modified atmosphere
[44]
3.3 基于高通量测序技术的微生物鉴定系统(highthroughput sequencing)
随着高通量测序技术的快速发展,测序时间和成本大幅降低,为微生物种群物种鉴定奠定基础。以高通量测序技术为基础的主要测序平台包括Illumina的Hiseq和Miseq平台、Roche454平台以及Ion Torrent平台。Illumina高通量测序技术因其测序通量高、数据量大而被广泛使用,其中测序耗时更短的Miseq技术和测通量更高的Hiseq技术被广泛应用于微生物物种鉴定及丰度测定中。
该技术已广泛应用于污水处理等环境样本的菌相研究中
[46]。尽管还未见生鲜家畜肉类和水产鱼虾类中有过该技术报道,但在熟肉
[47]等微生物鉴定中已有报道。Połka等
[47]在最新的研究中利用高通量测序技术,在香肠发酵过程中成功鉴定到了多种葡萄球菌及乳酸菌类(图3)。通常采用高通量测序技术在生鲜家畜肉类和水产鱼虾类中可以鉴定到上百种微生物,这是传统平板培养法和DGGE技术所无法比拟的,此外,高通量测序技术可同时对多个样本进行平行测序,进而对环境样本物种分类、丰度、种群结构及群落变化等进行对比分析,从而系统地反映样品中微生物的菌相变化。值得一提的是,该系统需要与构建好的基因文库进行比对才能确定微生物物种。目前常用的基因文库有16S rDNA、18S rDNA以及某些抗性基因的基因文库,而其他具有特定功能的基因文库种类并不丰富,有待进一步完善。

图3 根据高通量测序结果对香肠中微生物进行聚类分析
[4477]
Fig.3 Hierarchical clustering of microbial classified sequences by highthroughput sequencing in Italian salami
[47]
3.4 实时定量荧光聚合酶链式反应技术(real-time polymerase chain reaction,real-time PCR)
实时定量荧光聚合酶链式反应(real-time PCR)技术是利用荧光信号作为探针以监测模板定量的PCR检测技术。通常采用SYBR Green作为荧光嵌合或TaqMan作为探针,在PCR扩增反应的每一个循环中,通过检测随机扩增产物同步积累的荧光信号,实现特异性扩增模板的进行定量分析
[48]。
由于Real-time PCR能够灵敏、快速地检测出模板量相对较少的DNA,被广泛应用于微生物的定量分析检测和功能基因的检查
[17]。Gattuso等
[17]采用Real-time PCR方法,在猪肉保藏期间,检测李斯特氏菌的增殖变化,确认李斯特氏菌是猪肉腐败末期的主要微生物之一。Kumar等
[49]则利用Real-time PCR技术,确定腐败末期的海鲜鱼类微生物以假单胞菌、嗜冷菌以及沙雷氏菌为主(图4)。Real-time PCR技术通过定量分析微生物物种DNA数量,更加直观地表现了不同微生物在微生物菌群中数量水平的变化,从而确定优势菌群。Real-time PCR技术不仅可以定量确定优势菌群,还可以检测稀有菌的存在。Bjornsdottir-Butler等
[32]利用Real-time PCR技术在保藏的金枪鱼中检测到少量存在的摩氏摩根菌,而使用DGGE技术未检测到该菌,证实了Real-time PCR的灵敏性。
Real-time PCR技术与其他技术不同,是对活菌中RNA进行分析的技术,是鉴定活菌菌相的有效技术手段。相信该技术今后在研究生鲜家畜肉类和水产 鱼虾类物流 过程中菌相变化的过程中发挥重要作用。
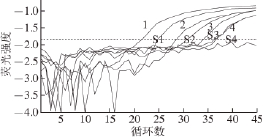
图4 利用Real-time PCR技术检测海鲜食品中沙门氏菌的数量变化
[4499]
Fig.4 Detection of Salmonella in seafood by real-time PCR assay
[49]
1~4、S1~S4为样品编号。
3.5 分子生态技术展望
以上4 种分子生态技术均为近年在生鲜家畜肉类和水产鱼虾类菌相研究中的常用手段。它们均能进行微生物的物种鉴定和丰度分析,反映生鲜家畜肉类和水产鱼虾类中细菌和真菌的分布状态。在生鲜家畜肉类和水产鱼虾类微生物总DNA的提取过程中,食物DNA混杂于其中,对微生物的丰度检测产生了干扰。为此,通过建立细菌16S rDNA文库和真菌18S rDNA及ITS文库,将样品总DNA与文库进行比对筛选,排除测序获得的肉类及水产鱼虾类DNA序列,提高微生物丰度分析的精度。
在食物腐败过程中,一些死亡微生物的DNA不能完全降解,采用基于微生物总DNA的研究方法不能区分食物中活菌与死菌。因此,采用基于DNA的分子生态技术并不能反映食物中活菌的分布状态。由于死亡微生物的RNA会快速降解,通过对微生物总RNA的分析可以更加可靠地反映出活菌菌相的分布状态,这对研究生鲜家畜肉类及水产鱼虾类微生物菌相变化有着重要意义。然而,目前该领域还只局限在对微生物总DNA水平的研究分析,而基于微生物总RNA的分析技术鲜见于生鲜家畜肉类及水产鱼虾类的菌相研究,这也限制了分子生态技术在生鲜家畜肉类和水产鱼虾类中微生物菌相研究的发展。
此外,DNA微阵列(DNA microarray)、自动核糖体间隔基因分析(automated ribosomal intergenic spacer analysis,ARISA)、单链构型多态性分析(single-strand conformation polymorphism,SSCP)、核糖体DNA扩增片段限制性分析(amplified ribosomal DNA restriction analysis,ARDRA)、荧光原位杂交(fluorescence in situ hybridization,FISH)等技术均为常用的分子生态技术。但根据近年报道
[17-49],这些技术尚未在生鲜家畜肉类及水产鱼虾类微生物菌相研究中被广泛应用。一些技术与本文介绍的4 种技术方法相似(如SSCP与DGGE技术,ARDRA与TRFLP技术等),同时某些技术随着分子生态技术的发展而被逐渐取代(如以DNA微阵列为基础发展而来的高通量测序技术因具有更高的测通量,将成为分子生态研究中的主流技术之一)。相信随着该领域研究的不断深入,更多的分子生态技术将被广泛应用。
4 结 语
本文综述了生鲜家畜肉类及水产鱼虾类物流过程中常见的微生物物种。其中乳酸菌类、肉食杆菌、沙门氏菌属、假单胞菌属、热杀索丝菌和单增李斯特氏菌等是腐败过程中常被报道的微生物,而一些发光细菌和酵母菌、霉菌等真菌也偶尔被发现。在腐败初期,乳酸菌类、肉食杆菌、假单胞菌属、热杀索丝菌等微生物被检测到的水平较低,而随着时间推移,在腐败末期这些微生物成为食物腐败的主要微生物,这表明随着生鲜家畜肉类和水产鱼虾类腐败的进行,微生物种类和丰度也在发生着动态变化。此外,微生物种类与物流过程中保藏方式密切相关。在真空保藏的条件下常检测到明串珠菌属、棒状杆菌属等微生物,而在托盘包装中大肠杆菌、不动杆菌及梭杆菌等微生物又常被发现于腐败的生鲜家畜肉类和水产鱼虾中。生鲜家畜肉类和水产鱼虾类在腐败过程中的菌相研究经历了由传统培养方法到分子生态学研究的重要转型时期。本文综述了近5 a常用于生鲜家畜肉类和水产鱼虾类相关分子生态技术,介绍了该领域作为基础研究手段而被广泛使用的DGGE技术、与荧光技术相结合的TRFLP技术、可以检测功能基因的Realtime PCR技术和新一代的高通量测序技术。
尽管人们对生鲜家畜肉类及水产鱼虾类中微生物的菌相变化进行了很多研究,但对食品变质过程中与腐败相关的功能基因及耐受基因的研究还有待深入。此外,采用基于微生物总DNA的研究策略并不能区分出活菌与死菌的分布水平。能够监测活菌的总RNA分析技术在生鲜家畜肉类及水产鱼虾类的菌相研究中有待推广使用。
生鲜家畜肉类和水产鱼虾类的菌相分析研究为预防食品变质、延长货架期等提供重要的理论依据。作为新一代的研究策略,高通量测序技术可以更加全面、准确地监测食品中微生物的菌相变化,特别是微生物高通量测序平台的建立,使得食品微生物菌种鉴定和菌相变化规律的研究更加自动化、精准化,将成为食品微生物菌相研究的重要研究手段。笔者正在对生鲜家畜肉类和水产鱼虾类中微生物总RNA的分析技术进行探索,结合高通量测序、Real-time PCR等分子生态技术对物流过程中微生物活菌的菌相变化进行研究。随着基于RNA分析技术的不断完善以及高通量测序技术在该领域的普及,相信生鲜家畜肉类和水产鱼虾类的菌相研究会更加深入,将为食品保鲜技术的发展提供理论上的保障。
参考文献:
[1] 钱乃余. 中国城镇居民食品消费与食品物流保障研究[D]. 成都: 西南财经大学, 2010.
[2] 王永锋. 生鲜产品在途配送质量控制及可追溯系统关键技术研究[D].重庆: 重庆大学, 2012.
[3] BLICKSTAD E, MOLIN G. Carbon dioxide as a controller of the spoilage flora of pork, with special reference to temperature and sodium chloride[J]. Journal of Food Protection, 1983, 46: 756-763.
[4] DAINTY R H, SHAW B G, ROBERTS T A. Microbial and chemical changes in chill-stored red meats[M]//ROBBERS T A, SKINNER F A. Food microbiology. London: Advances and Prospects Academic Press,1983: 151-178.
[5] GILL C D, MOLIN G. Modified atmospheres and vaccum packaging[M]//RUSELL N J, GOULD G W. Food preservatives. Glasgow: Blackie and Son Ltd., 1991: 172-199.
[6] CHURCH I, PARSONS A. Modified atmosphere packaging technology: a review[J]. Journal of the Science of Food and Agriculture, 1995, 67(2):143-152.
[7] BORCH E, KANT-MUERMANSM L, BLIXT Y. Bacterial spoilage of meat and cured meat product[J]. International Journal of Food Microbiology, 1996, 33(1): 103-120.
[8] BORCH E, NERBRINK E, SVENSSON P. Identification of major contamination sources during processing of emulsion sausage[J]. International Journal of Food Microbiology, 1988, 7(4): 317-330.
[9] BJ.RKROTH K J, KORKEALA H J. Use of rRNA gene restriction patterns to evaluate lactic acid bacterium contamination of vacuumpackaged sliced cooked whole-meat product in a meat processing plant[J]. Applied and Environmental Microbiology, 1997, 63(2): 448-453.
[10] ZHAO Fan, ZHOU Guanghong, YE Keping, et al. Microbial changes in vacuum-packed chilled pork during storage[J]. Meat Science, 2014,100: 145-149.
[11] 王博飞, 梁晓媛, 杨林森. 微生物测序分型系统: 基于美国应用生物系统公司基因分析仪的MicroSeq微生物鉴定系统[J]. 微生物与感染, 2008, 3(3): 191-192.
[12] 彭涛. 猪肉微冻保鲜技术研究[D]. 长沙: 湖南农业大学, 2010.
[13] FONTANA C, COCCONCELLI P S, VIGNOLO G. Direct molecular approach to monitoring bacterial colonization on accum-packaged beef[J]. Applied and Environmental Microbiology, 2006, 72(8):5618-5622.
[14] JIANG Yun, GAO Feng, XU Xinglian, et al. Changes in the composition of the bacterial flora on tray-packaged pork during chilled storage analyzed by PCR-DGGE and real-time PCR[J]. Journal of Food Science, 2011, 76(1): 27-33.
[15] CHANTARASATAPORN P, TEPKASIKUL P, KINGCHA Y, et al. Water-based oligochitosan and nanowhisker chitosan as potential food preservatives for shelf-life extension of minced pork[J]. Food Chemistry, 2014, 159: 463-470.
[16] MIHAIU L, LAPUSAN A, TANASUICA R, et al. First study of Salmonella in meat in Romania[J]. Journal of Infection in Developing Countries, 2014, 8(1): 50-58.
[17] GATTUSO A, GIANFRANCESCHI M V, SONNESSA M, et al. Optimization of a real time PCR based method for the detection of Listeria monocytogenes in pork meat[J]. International Journal of Food Microbiology, 2014, 184: 106-108.
[18] WEERAKKODY N S, CAFFIN N, DYKES G A, et al. Effect of antimicrobial spice and herb extract combinations on Listeria monocytogenes, Staphylococcus aureus, and spoilage microflora growth on cooked ready-to-eat vacuum-packaged shrimp[J]. Journal of Food Protection, 2011, 74(7): 1119-1125.
[19] LORENZO J M, SINEIRO J, AMADO I R, et al. Influence of natural extracts on the shelf life of modified atmosphere-packaged pork patties[J]. Meat Science, 2014, 96(1): 526-534.
[20] KAPETANAKOU A E, AGATHAGGELOU E I, SKANDAMIS P N. Storage of pork meat under modified atmospheres containing vapors from commercial alcoholic beverages[J]. International Journal of Food Microbiology, 2014, 178: 65-75.
[21] BOZIARIS I S, STAMATIOU A P, NYCHAS G J. Microbiological aspects and shelf life of processed seafood products[J]. Journal of the Science of Food and Agriculture, 2013, 93(5): 1184-1190.
[22] LI Miaoyun, TIAN Lu, ZHAO Gaiming, et al. Formation of biogenic amines and growth of spoilage-related microorganisms in pork stored under different packaging conditions applying PCA[J]. Meat Science,2014, 96(2): 843-848.
[23] COTON M, JOFFRAUD J J, MEKHTICHE L, et al. Biodiversity and dynamics of the bacterial community of packaged king scallop (Pecten maximus) meat during cold storage[J]. Food Microbiology, 2013,35(2): 99-107.
[24] NOSEDA B, ISLAM M T, ERIKSSON M, et al. Microbiological spoilage of vacuum and modified atmosphere packaged Vietnamese Pangasius hypophthalmus fillets[J]. Food Microbiology, 2012, 30(2): 408-419.
[25] SAMPAIO G S, PFLANZER-J.NIOR S B, RO.A R D, et al. Effects of polyethylene film wrap on cooler shrink and the microbial status of beef carcasses[J]. Meat Science, 2014, 100: 164-170.
[26] PEREIRA J A, DION.SIO L, PATARATA L, et al. Effect of packaging technology on microbiological and sensory quality of a cooked blood sausage, Morcela de Arroz, from Monchique region of Portugal[J]. Meat Science, 2014, 101: 33-41.
[27] CARAVEO O, ALARCON-ROJO A D, RENTERIA A, et al. Physicochemical and microbiological characteristics of beef treated with high-intensity ultrasound and stored at 4℃[J]. Journal of the Science of Food and Agriculture, 2014. doi: 10.1002/jsfa.6979.
[28] BROEKAERT K, HEYNDRICKX M, HERMAN L, et al. Seafood quality analysis: Molecular identification of dominant microbiota after ice storage on several general growth media[J]. Food Microbiology,2011, 28(6): 1162-1169.
[29] NISIOTOU A, CHORIANOPOULOS N G, GOUNADAKI A, et al. Effect of wine-based marinades on the behavior of Salmonella Typhimurium and background flora in beef fillets[J]. International Journal of Food Microbiology, 2013, 164(2/3): 119-127.
[30] KANDEL P P, PASTERNAK Z, van RIJN J, et al. Abundance, diversity and seasonal dynamics of predatory bacteria in aquaculture zero discharge systems[J]. FEMS Microbiology Ecology, 2014, 89(1): 149-161.
[31] ULBIN-FIGLEWICZ N, BRYCHCY E, JARMOLUK A. Effect of low-pressure cold plasma on surface microflora of meat and quality attribut es[J]. Journal of Food Science of Technology, 2015, 52(2):1228-1232.
[32] BJORNSDOTTIR-BUTLER K, JONES J L, BENNER R, et al. Development of a real-t ime PCR assay with an internal amplification control for detection of Gram-negative histamine-producing bacteria in fish[J]. Food Microbiology, 2011, 28(3): 356-363.
[33] GILL C O, GREER C G, DILTS B D. The aerobic growth of Aeromonas hydrophila and Listeria monocytogenes in broths and on pork[J]. International Journal of Food Microbiology, 1997, 35: 67-74.
[34] 李苗云. 冷却猪肉中微生物生态分析及货架期预测模型的研究[D].南京: 南京农业大学, 2010.
[35] ZWIETERING M H, JONGENBERGER, ROMOBOUTS F M, et al. Modeling of the bacterial growth curve[J]. Application Environment Microbiology, 1990, 56: 1875-1881.
[36] HU Yue, HUANG Zhiyong, LI Jian, et al. Concentrations of biogenic amines in fish, squid and octo pus and their changes during storage[J]. Food Chemistry, 2012, 135(4): 2604-2611.
[37] 江芸, 高峰, 徐幸莲, 等. PCR-TRFLP 研究托盘包装冷却猪肉冷藏过程中的菌相变化[J]. 浙江大学学报, 2011, 37(5): 565-572.
[38] FISHER S G, LERMAN L S. Length-independent separation of DNA restriction fragments in two dimensional gel electrophoresis[J]. Cell,1979, 16: 191-200.
[39] MUYZE S G, WAAL E C, UITTERLINDEN A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polyme r ase chain reaction-amplified genes coding for 16S rRNA[J]. Applied Environmental Microbiology, 1993,59: 695-700.
[40] MUYZER G. DGGE/TGGE a method for identifying genes from natural ecosystems[J]. Current Opinion in Microbiology, 1999, 2: 317-322.
[41] 江芸. 托盘和真空包装冷却猪肉冷藏过程中菌相变化规律研究[D].南京: 南京农业大学, 2010.
[42] MARSH T L, SAXMAN P, COLE J. Terminal restri ction fragment length polymorphism analysis program, a web-based research tool for microbial community analysis[J]. Applied and Environmental Microbiology, 2000, 66: 3616-3620.
[43] 任南琪, 赵阳国, 高崇洋. TRF LP在微生物群落结构与动态分析中的应用[J]. 哈尔滨工业大学学报, 2007, 39(4): 552-556.
[44] KIERMEIER A, TAMPLIN M, MAY D, et al. Microbial growth,communities and sensory characteristics of vacuum and modified atmosphere packaged lamb shoulders[J]. Food Microbiology, 2013,36(2): 305-315.
[45] SMITH C J, DANILOWICZ B S, MEIJER W G. Characterization of the bacterial community associated with the surface and mucus layer of whiting (Merlangius merlangus)[J]. FEMS Microbiology Ecology,2007, 62(1): 90-97.
[46] LI Bing, ZHANG Xuxiang, GUO Feng, et al. Characterization of tetracycline resistant bacterial community in saline activated sludge usin g batch stress incubation with high-throughput sequencing analysis[J]. Water Research, 2013, 47(13): 4207-4216.
[47] POŁKA J, REBECCHI A, PISACANE V, et al. Bacterial diversit y in typical Italian salami at different ripening stages as revealed by high-throughput sequencing of 16S rRNA amplicons[J]. Food Microbiology, 2015, 46: 342-356.
[48] WOO T H, PATEL B K, CINCO M, et al. Identification of Leptospira bifl exa by real-time homogeneous detection of rapid cycle PCR product[J] . Journal of Microbiological Methods, 1999, 35: 23-30.
[49] KUMAR R, LALITHA K V. Digoxigenin-labeled probe-based colony blotting assay for rapid quantification of Salmonella serovars in seafood and water[J]. Journal of AOAC International, 2012, 95(6):1652-1655.
Progress in Research on Microbial Flora in Fresh Livestock Meat and Aquatic Fish and Shrimp during Logistics
YANG Chao, CHE You, SONG Cunjiang*
(Key Laboratory of Molecular Microbiology and Technology, Ministry of Education, College of Life Sciences,Nankai University, Tianjin 300071, China)
Abstract:Spoilage of fresh livestock meat and aquatic fish and shrimp is mainly caused by microbial growth during logistics. In recen t years, with the rapid development of molecular ecological techniques, many kinds of microbes have been detected in fresh livestock meat and aquatic fish and shrimp, including Carnobacterium spp., Lactobacillus spp., Salmonella spp., Pseudomonas spp., Leuconostoc spp., Brochothrix thermosphacta, Listeria monocytogenes, yeasts, molds and so on. This review summarizes common molecular biology techniques that were applied during 2010 to 2015 to microbial flora in fresh livestock meat and aquatic fish and shrimp during log istics. Molecular ecological techniques including denaturing gradient gel electrophoresis, terminal restriction fragment length p olymorphism, high-throughput sequencing and realtime PCR are also described to provide a reference for studying microbial flora in fresh livestock meat and aquatic fish and shrimp during logistics.
Key words:fresh livestock meat; aquatic fish and shrimp; spoilage; microbial flora; molecular ecological techniques
中图分类号:TS201.3
文献标志码:A
文章编号:1002-6630(2015)23-0307-07
doi:10.7506/spkx1002-6630-201523056
收稿日期:2015-02-05
基金项目:“十二五”国家科技支撑计划项目(2015BAD16B04);国家自然科学基金面上项目(31470213;31170030);天津市重点支撑计划项目(13JCZDJC27800;13JCYBJC24900;13TXSYJC40100;14ZCZDSF00009)
作者简介:杨超(1991—),男,博士研究生,研究方向为食品微生物。E-mail:ychych3412@163.com
*通信作者:宋存江(1961—),男,教授,博士,研究方向为食品微生物。E-mail:songcj@nankai.edu.cn




