表1 牛肝菌样品信息
Table 1 Information aboutt Bolleettuuss sampplleess

?
杨天伟
1,2,张 霁
2,史云东
3,李 涛
3,王元忠
2,*,刘鸿高
1,*
(1.云南农业大学农学与生物技术学院,云南 昆明 650201;2.云南省农业科学院药用植物研究所,云南 昆明 650200;3.玉溪师范学院资源环境学院,云南 玉溪 653100)
摘 要:采用傅里叶变换红外光谱结合多元统计分析方法快速鉴别不同种类食用牛肝菌。采集10 个不同种类93 个牛 肝菌子实体的红外光谱,分析食用牛肝菌的红外光谱特征;用多元散射校正(multiplicative signal correction,MSC)、标准正态变量(standard normal variate,SNV)、二阶导数(second derivative,SD)、Norris平滑(ND)、正交信号校正(orthogonal signal correction,OSC)、小波压缩等方法对光谱进行优化处理;经优化处理的光谱数据分别建立马氏距离分类模型及偏最小二乘判别分析(partial least squares discriminant analysis,PLSDA)。结果显示,牛肝菌在3 325、2 934、2 927、1 637、1 547、1 402、1 375、1 259、1 453、1 081、1 029 cm -1等附近有多个吸收峰,主要归属为蛋白质、多糖、氨基酸等的特征吸收峰。MSC+SD+ND(15∶5)和SNV+SD+ ND(15∶5)两种预处理方式前10 个主成分累积贡献率分别为95.58%、95.54%,基于两种预处理方法建立马氏距离分类模型,验证集预测准确率分别为90%和95%。PLS-DA结果显示经MSC+SD+ND(15∶5)和SNV+SD+ND (15∶5)预处理不易于区分牛肝菌种类;原始光谱经正交信号校正及小波压缩(orthogonal signal correction wavelet compression,OSCW)、优化处理并进行PLS-DA分析,能够很好地区分不同种类牛肝菌。马氏距离分类模型不仅能反映样品的分类情况,同时计算出与测试样品相似度最大的物种,可为食用菌种类鉴别和未知物种鉴定提供可靠依据;OSCW预处理后进行 PLS-DA分析能有效鉴别不同种类牛肝菌,为野生食用菌的鉴别分类提供一种辅助方法。
关键词:红外光谱;牛肝菌;鉴别;马氏距离;偏最小二乘判别分析
食用菌营养丰富、食味独特,是集营养、保健、药用于一体的健康食品,大量研究表明食用菌富含蛋白质、膳食纤维、多糖、氨基酸、维生素、矿质元素等营养物质 [1-4],可作为人体8 种必需氨基酸和易缺乏微量元素Ca、Fe、Mg、Mn、Zn、Se等的重要来源 [3,5-6];多糖类物质具有抗氧化、防癌抗癌、防病治病、增强人体免疫力等功效 [7-9]。我国食用菌种类多、产量大,是世界上最大的食用菌生产国和出口国 [10];云南多样的立体气候和丰富的森林资源孕育了大量的野生食用菌资源,牛肝菌、松茸、块菌等珍稀食用菌深受消费者亲睐,已远销欧洲、日本等国家,其中牛肝菌成为菌类产品中出口量大、换汇率高的畅销产品。野生食用菌不与人争粮,不与农争时、争地的特征及人们日益增长的消费需求,市场价格不断增涨,推动了农民采集野生食用菌的积极性,成为增收的重要来源之一。然而食用菌种类繁多,种间形态相似性高,不易准确鉴别,因误食引起的中毒事件时有发生;市场上出现以假充真、以次充好的现象,甚至有不法商贩收购有毒牛肝菌,制成干片后混淆出售,严重威胁消费者健康、扰乱食用菌市场。
王向华 [11]从云南市场上收购了6 种当地称为“北风菌”的真菌,研究发现这6 种野生菌中有一类为皂味口蘑(Tricholoma saponaceum (Fr .) Kummer),该菌为有毒真菌不宜食用;食用菌市场上同名异物的“北风菌”仅为一例,其他形态相似极易混淆物种还大量存在。英国真菌学家Dentinger等 [12]报道用超市购买的中国云南出口美味牛肝菌进行DNA测序分析,发现同一包装袋内15 片牛肝菌中有3 种新牛肝菌物种,提出丰富多样的牛肝菌种类也许对消费者存在安全隐患。这些研究一方面表明真菌的物种多样性及中国野生食用菌的丰富多样性,另一方面提示人们准确鉴别不同种类野生食用菌的重要性。
野生食用牛肝菌的准确鉴别是保障消费者安全,深入开发、研究和市场质量控制的前提基础。传统野生食用菌鉴别主要凭借经验,根据牛肝菌生长特性、外观型貌、显微结构、菌肉菌管的颜色及变色反应等进行鉴别分类 [13],该方法受主观因素干扰较大。目前对食用菌鉴别分类研究主要有光谱法和分子生物学方法,本课题组依据牛肝菌紫外光谱特征结合化学计量学方法研究了牛肝菌不同产地、种类和部位的鉴别方法 [14-15],周在进 [13]、时有明 [16]等运用傅里叶红外光谱结合光谱检索、聚类分析法鉴别了部分食用菌。Feng Bang等 [17]通过分析牛肝菌3 个基因片段的DNA系列变异情况,揭示我国牛肝菌物种多样性;Mello等 [18]根据ITS片段设计引物,用分子生物学方法鉴别了铜色牛肝菌(Boletus aereus Bull.)和美味牛肝菌(Boletus edulis Bull.);李艳春等 [19]研究了市场上常见4 种牛肝菌的DNA条形码,结果表明4 种牛肝菌样品代表了12 个物种;Wu Gang等 [20]分析了牛肝菌属的DNA系列,并结合牛肝菌形态特征、显微结构等建立了牛肝菌科分子发育系统框架;分子生物学方法价格昂贵,操作复杂,不适宜推广应用。
傅里叶红外光谱具有操作简便、样品量少、分析快速、样品无损等优点 [21-22],已广泛应用于中药材产地、物种鉴别 [23-24],食品分析 [25-26],农业 [27]等领域。本研究通过采集10 种野生食用牛肝菌93 个子实体的红外光谱信息,对原始光谱进行平滑、求导、多元散射校正(multiplicative signal correction,MSC)、标准正态变量(standard normal variate,SNV)、正交信号校正(orthogonal signal correction,OSC)等优化处理;经优化处理的光谱数据分别建立马氏距离分类模型及进行偏最小二乘判别分析(partial least squares discriminant analysis,PLS-DA),为快速鉴别牛肝菌种类提供辅助方法。
1.1 实验材料
表1 牛肝菌样品信息
Table 1 Information aboutt Bolleettuuss sampplleess

?
10 种牛肝菌均于2012年采自云南省玉溪市易门县,由云南农业大学刘鸿高教授鉴定,保存于云南农业大学,详细信息见表1。
1.2 仪器与试剂
Frontier型傅里叶变换红外光谱仪(采用DTGS检测器,光谱分辨率为4 cm -1) 美国Perkin Elmer公司;YP-2型压片机 上海市山岳科学仪器有限公司;FW-100型高速粉碎机 天津市华鑫仪器厂;80 目标准筛盘浙江上虞市道墟五四仪器厂。
KBr(分析纯) 天津市风船化学试剂科技有限公司。
1.3 方法
1.3.1 采集光谱信息
牛肝菌样品采集后清洗干净,50 ℃烘干至质量恒定,粉碎,过80 目筛,备用。称取牛肝菌样品(1.5±0.2) mg、KBr(100
 2) mg,在红外线灯下充分混合研磨,混合均匀的粉末放入模具将其压成厚度均匀的透明薄片。设定光谱扫描范围4 000~400 cm
-1,累计扫描次数:16 次,每个样品测定3 次,取平均光谱。样品1
#重复称取7 份,分别压片测定,考察重复性,取同一片样品重复测定7 次,考察方法精密度,取同一片样品分别在0、10、20、30、40、50、60 min时测定(每次测完立即放到红外线灯下,以防吸水),考察稳定性。实验过程中时时扣除背景干扰。
2) mg,在红外线灯下充分混合研磨,混合均匀的粉末放入模具将其压成厚度均匀的透明薄片。设定光谱扫描范围4 000~400 cm
-1,累计扫描次数:16 次,每个样品测定3 次,取平均光谱。样品1
#重复称取7 份,分别压片测定,考察重复性,取同一片样品重复测定7 次,考察方法精密度,取同一片样品分别在0、10、20、30、40、50、60 min时测定(每次测完立即放到红外线灯下,以防吸水),考察稳定性。实验过程中时时扣除背景干扰。
1.3.2 光谱优化处理和数据分析
光谱数据采集时受仪器噪音、样品差异、环境等诸多因素影响;对原始光谱进行优化处理,可以降低或消除噪音、背景等的干扰,提高光谱分析的精度。常用的光谱预处理方法主要有平滑、求导、MSC、SNV、OSC等,这些方法各具优点,如光谱平滑能减弱噪音干扰,光谱数据进行求导可以消除基线漂移和噪音干扰,MSC可减小散射光带来的误差,OSC能有效去除与目标变量无关的干扰信息,提高光谱分析的准确度 [28-29]。优化处理后的红外光谱数据分别建立马氏距离判别分类模型和进行PLS-DA分析。
2.1 实验方法学考察
运用Omnic 8.2软件的光谱检索功能,建立重复性、精密度、稳定性的光谱数据库:A 1、A 2、A 3,分别计算用于方法学考察样品的匹配分值,匹配分值越大方法越可靠 [13]。结果显示重复性、精密度、稳定性的匹配值分别在99.42%~99.93%、99.90%~99.99%、99.21%~99.97%之间,表明方法稳定性好,精密度高。
2.2 不同种类牛肝菌红外光谱分析

图 1 不同种类牛肝菌平均红外光谱
Fig.1 Mean FTIR spectra of different species of boletus
由图1可看出,不同种类牛肝菌红外光谱的峰形、峰位基本一致,而峰高(吸光度)等具有差异,表明不同种类牛肝菌的化学组分基本相同,而对化学成分的积累量不同。牛肝菌在3 325 cm -1附近的强吸收峰归属为蛋白质、多糖、纤维素等的O—H伸缩振动和蛋白质中的N—H伸缩振动;2 934、2 927 cm -1附近明显的吸收峰主要为多糖、蛋白质等甲基的对称伸缩振动;1 637 cm -1附近吸收峰归属为蛋白质酰胺Ⅰ带和C=O伸缩振动;1 547 cm -1附近为蛋白质C=N、N—H的伸缩振动;1 453 cm -1附近归属为亚甲基的弯曲振动;1 402、1 375、1 259 cm -1等附近为多糖、蛋白质等的C—O—H弯曲振动和亚甲基的变形振动;1 081、1 029 cm -1附近分别为糖类的C—O和C—C伸缩振动;950~710 cm -1范围多个弱吸收峰,主要为糖类异构体的特征峰。
2.3 光谱预处理
运用TQ8.0软件对原始光谱进行平滑、求导、MSC、SNV等优化处理,运用SIMCA-P10.0软件对原始光谱进行SNV-小波压缩优化处理,以减弱噪音、散射光、基线漂移等的干扰,提高光谱分析的准确度,结果见图1和表2、3。
表2 不同预处理方法的主成分累积贡献率
Table 2 Principal component cumulative contribution rates of different pretreatment methods

注:MSC.多元散射校正;SNV.标准正态变量;FD.一阶导数;SD.二阶导数;ND. Norris平滑。
预处理方法 主成分数 累积贡献率/% NONE 10 92.87 MSC+FD+ND(15∶5) 10 94.28 MSC+SD+ND(15∶5) 10 95.58 SNV+FD+ND(15∶5) 10 94.15 SNV+SD+ND(15∶5) 10 9 5.54
由图1和表2可知,MSC+SD+ND(15∶5)和SNV+ SD+ND(15∶5)两种光谱优化方法,前10个主成分的累积贡献率均大于95%,效果较好。表3显示了正交信号校正及小波压缩(orthogonal signal correction wavelet compression,OSCW)预处理的参数,OSCW选取了特征值大于1的3 个主成分,选用多贝西小波函数和离散小波变换法进行压缩;结果显示,第3主成分的剩余平方和为26.07%,表明与牛肝菌分类变量无关的73.93%干扰信息已被消除。
表 3 OSCW预处理结果
Table 3 Results of OSCW pretreatment

主成分 正交角度/(°) 剩余平方和/% 特征值1 90.00 61.34 35.95 2 89.99 36.51 23.10 3 89.98 26.07 9.71
2.4 不同种类牛肝菌马氏距离分类模型
随机选取20 个样品为验证集,其余样品为训练集,将10 种不同牛肝菌种类定义为1~10类;用TQ8.0软件选择MSC+SD+ND(15∶5)和SNV+SD+ND(15∶5)两种光谱优化方法建立不同种类牛肝菌的马氏距离分类判别模型,结果见表4。由表4可知,基于MSC+SD+ND (15∶5)光谱优化处理方法建立的分类模型,20 个验证集中9 #和67 #样品分类错误,其余样品分类正确,样品分类预测正确率为90%;经SNV+SD+ND(15∶5)光谱 预处理建立 的模型,只有9 #样品分类错误,其余样品分类正确,预测正确率达95%。
表4不仅显示了20 个验证集样品的分类结果,验证集样品的计算类别与真实类别之间的马氏距离,还计算出验证集样品与下一个最接近类别的马氏距离,能反映两类样品的相似性。例如2 #样品真实类别为第1类(华丽牛肝菌),计算结果也为第1类,表明2 #样品分类正确,2 #样品到真实分类的马氏距离为0.811;除真实值外与2 #样品最近的类别为第4类,马氏距离为1.088,表明2 #(第1类)样品与第4类样品的相似度较大,可以推测两类牛肝菌样品化学组分及含量相似。MSC+SD+ND (15∶5)和SNV+SD+ND(15∶5)两种预处理方法建立的马氏距离分类模型结果基本一致,如23 #样品计算分类到真实分类的距离及与最接近类别的距离最大,53 #样品马氏距离最小。
表4 牛肝菌种类马氏距离分类模型预测结果
Table 4 Prediction results for boletus classififi cation by the Mahalanobisdistance classififi cation model

注:马氏距离 1:计算出的分类与实际类之间的马氏距离;最接近类别:与该样品最接近的下一个类别;马氏距离 2:样品到最接近下一个类别的马氏距离。*.分类错误。
编号MSC+SD+ND(15∶5) SNV+SD+ND(15∶5)真实类别计算类别马氏距离 1 最接近类别马氏距离 2 真实类别计算类别马氏距离 1 最接近类别马氏距离 22 # 1 1 0.811 4 1.088 1 1 0.802 4 1.115 9 # 1 6 * 0.890 4 1.226 1 6 * 0.913 4 1.202 13 # 2 2 0.588 4 1.575 2 2 0.570 5 1.551 15 # 2 2 0.957 5 1.937 2 2 0.962 5 1.842 20 # 2 2 1.026 5 1.477 2 2 1.016 5 1.472 23 # 3 3 1.440 1 2.014 3 3 1.446 1 2.025 29 # 3 3 0.880 1 1.552 3 3 0.889 1 1.530 34 # 4 4 1.211 1 1.595 4 4 1.192 1 1.556 36 # 4 4 1.112 1 1.515 4 4 1.105 1 1.593 42 # 5 5 0.642 9 1.588 5 5 0.650 9 1.525 47 # 6 6 0.842 7 1.012 6 6 0.851 7 1.011 53 # 6 6 0.297 7 0.950 6 6 0.313 7 0.979 59 # 7 7 0.869 6 1.377 7 7 0.895 6 1.425 6 4 # 7 7 1.015 9 1.427 7 7 1.011 9 1.348 67 # 8 5 * 1.016 8 1.346 8 8 1.111 6 1.130 71 # 8 8 0.774 6 1.218 8 8 0.766 6 1.219 77 # 9 9 0.925 8 1.661 9 9 0.925 8 1.605 82 # 9 9 0.915 8 1.585 9 9 0.878 5 1.566 88 # 10 10 0.675 9 1.748 10 10 0.589 9 1.740 92 # 10 10 0.655 7 1.329 10 10 0.673 7 1.354
2.5 PLS-DA
将MSC+SD+ND(15∶5)和SNV+SD+ND (15∶5)预处理后的光谱数据导入SIMCA-P软件进行PLS-DA,结果见图2。图2为前3 个主成分的三维得分图,由图2a可看出,不同种类牛肝菌样品只有部分样品可以很好的聚类,很多样品出现离散分布,不易区分不同种类牛肝菌样品,图2b反映出不同种类牛肝菌分类效果明显优于图2a的分类效果,但部分样品的聚类效果不明显如:砖红绒盖牛肝菌;不同种类牛肝菌样品互相堆叠,难以鉴别牛肝菌种类,表明SNV+SD+ND(15 ∶5)和MSC+SD+ND(15∶5)光谱预处理方法结合PLS-DA分析不适宜用于牛肝菌种类鉴别。

图 22 MMSSCC+SSDD+NNDD( 1155∶5)(a)和SSNNVV+SSDD+NNDD(1155∶55)(bb)预处理后PLS-DA分析的三维得分图
Fig.2 3D score plot of PLS-DA after MSC + SD + ND (15∶5) (a) and SNV + SD + ND (15∶5) (b) pretreatment
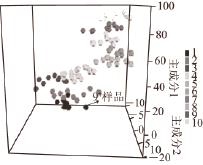
图3 OSCW预处理后PLS-DA分析的三维得分图
Fig.3 3D score plot of PLS-DA after OSCW pretreatment
OSCW预处理后的光谱数据进行PLS-DA分析,结果见图3,基于OSCW建立的PLS-DA分类模型能够很好地将不同种类牛肝菌样品区分开,可用于不同种类牛肝菌的鉴别分析。图中9 #样品与该类(华丽牛肝菌)其他样品的距离较远,结果与马氏距离分类判别模型中9 #样品分类错误的结果一致。与图2相比,OSCW红外光谱预处理方法能够更有效地去除与分类变量无关的信息,保留有用信息,为不同种类牛肝菌的鉴别分析提供更可靠的依据。
采用傅里叶变换红外光谱法测定了10 种牛肝菌的红外光谱,根据牛肝菌的红外吸收峰分析其成分特征。应用平滑、求导、MSC、SNV、OSC-小波压缩等方法对原始光谱进行优化处理。结果显示MSC+SD+ND (15∶5)和SNV+SD+ND(15∶5)两种预处理方法前10 个主成分累积贡献率均在95%以上,OSCW预处理方法能有效去除与牛肝菌种类无关的干扰信息提高光谱分析的准确度。
基于MSC+SD+ND(15∶5)和SNV+SD+ND (15∶5)预处理方法建立马氏距离分类判别模型,验证集样品的预测正确率分别为90%和95%,得到满意的结果。马氏距离分类模型不仅可以预测牛肝菌分类结果,而且可以看出与某一牛肝菌样品最相近的种类及到该种类的马氏距离,此方法可为牛肝菌等食药用真菌的物种鉴定、鉴别提供科学依据。经预处理后的光谱数据分别进行PLS-DA分析,结果显示,用MSC+SD+ND (15∶5)和SNV+SD+ND(15∶5)预处理结合PLS-DA分析,不能有效区分牛肝菌种类,不适宜牛肝菌种类鉴别分类;OSCW预处理后的光谱数据进行PLS-DA分析,能够明显区分不同种类牛肝菌样品,结果满意,可用于野生食用菌种类鉴别分析。
参考文献:
[1] KALAČ P. Chemical composition and nutritional value of European species of wild growing mushrooms: a review[J]. Food Chemistry,2009, 113(1): 9-16.
[2] 周玲仙, 殷建忠. 云南野生食用牛肝菌营养成分 分析及评价[J]. 食用菌, 2008(4): 61-62.
[3] LI Tao, WANG Yuanzhong, ZHANG Ji, et al. Trace element content of Boletus tomentipes mushroom collected from Yunnan, China[J]. Food Chemistry, 2011, 127(4): 1828-1830.
[4] ZHANG Yin, VENKITASAMY C, PAN Zhongli, et al. Recent developments on umami ingredients of edible mushrooms a review[J]. Trends in Food Science and Technology, 2013, 33(2): 78-92.
[5] WANG Xuemei, ZHANG Ji, WU Lihu, et al. A mini-review of chemical composition and nutritional value of edible wild-grown mushroom from China[J]. Food Chemistry, 2014, 151: 279-285.
[6] FALANDYSZ J. Selenium in edible mushrooms[J]. Journal of Environmental Science and Health Part C, 2008, 26(3): 256-299.
[7] CHANG S T, WASSER S P. The role of culinary-medicinal mushrooms on human welfare with a pyramid model for human health[J]. International Journal of Medicinal Mushrooms, 2012, 14(2):95-134.
[8] 倪宗耀. 食用菌的食疗作用与保健功效[J]. 食药用菌, 2013, 21(1):22-25.
[9] KAMIYAMA M, HORIUCHI M, UMANO K, et al. Antioxidant/antiinfl ammatory activities and chemical composition of extracts from the mushroom Trametes versicolor[J]. International Journal of Nutrition and Food Sciences, 2013, 2(2): 85-91.
[10] 张金霞. 食用菌产业发展需要科学研究的强力支撑[J]. 菌物学报,2014, 33(2): 175-182.
[11] 王向华. 滇中农贸市场上“北风菌”的鉴别[J]. 中国食用菌, 2000,19(4): 18-20.
[12] DENTINGER B T M, SUZ L M. What's for dinner? Undescribed species of porcini in a commercial packet[J]. Peer Journal, 2014, 2: e570.
[13] 周在进, 刘刚, 任先培. 中红外光谱和分层抽样法应用于野生菌的快速鉴别[J]. 激光与红外, 2010, 40(9): 970-975.
[14] 杨天伟, 崔宝凯, 张霁, 等. 食用牛肝菌不同部位紫外指纹图谱鉴别分析[J]. 菌物学报, 2014, 33(2): 262- 272.
[15] 杨天伟, 李涛, 张霁, 等. 紫外光谱结合欧氏距离和主成分分析法快速鉴别牛肝菌[J]. 食品科学, 2014, 35(16): 105-109. doi: 10.7506/ spkx1002-6630-201416020.
[16] 时有明, 刘刚, 孙艳琳, 等. FTIR光谱结合系统聚类分析鉴别松茸和姬松茸的研究[J]. 光散射学报, 2010, 22(2): 171-174.
[17] FENG Bang, XU Jianping, WU Gang, et al. DNA sequence analyses reveal abundant diversity, endemism and evidence for Asian origin of the porcini mushrooms[J]. PLoS One, 2012, 7(5): e37567.
[18] MELLO A, GHIGNONE S, VIZZINI A, et al. ITS primers for the identifi cation of marketable boletes[J]. Journal of Biotechnology, 2006,121(3): 318-329.
[19] 李艳春, 吴刚, 杨祝良. 我国云南食用牛肝菌的DNA条形码研究[J].植物分类与资源学报, 2013, 35(6): 725-732.
[20] WU Gang, FENG Bang, XU Jianping, et al. Molecular phylogenetic analyses redefi ne seven major clades and reveal 22 new generic clades in the fungal family Boletaceae[J]. Fungal Diversity, 2014, 69(1): 93-115.
[21] ALAMPRESE C, CASALE M, SINELLI N, et al. Detection of minced beef adulteration with turkey meat by UV-vis, NIR and MIR spectroscopy[J]. LWT-Food Science and Technology, 2013, 53(1):225-232.
[22] 赵艳丽, 张霁, 袁天军, 等. 近红外光谱快速鉴别不同产地药用植物重楼的方法研究[J]. 光谱学与光谱分析, 2014, 34(7): 1831-1835.
[23] JIANG Y, D AVID B, TU P, et al. Recent analytical approaches in quality control of traditional Chinese medicines: a review[J]. Analytica Chimica Acta, 2010, 657(1): 9-18.
[24] 孙鹏, 张小松, 范琦, 等. 不同产地青蒿的FTIR指纹图谱分析[J]. 中成药, 2008, 29(12): 1721-1724.
[25] CHE MAN Y B, MARINA A M, ROHMAN A, et al. A fourier transform infrared spectroscopy method for analysis of palm oil adulterated with lard in pre-fried french fries[J]. International Journal of Food Properties, 2014, 17(2): 354-362.
[26] JAWAID S, TALPUR F N, AFIRIDI I H, et al. Quick determination of melamine in infant powder and liquid milk by Fourier transform infrared spectroscopy[J]. Analytical Methods, 2014, 6(14): 5269-5273. [27] SMITH J P, SCHMIDTKE L M, MULLER M C, et al. Measurement of the concentration of nutrients in grapevine petioles by attenuated total reflectance Fourier transform infrared spectroscopy and chemometrics[J]. Australian Journal of Grape and Wine Research,2014, 20(2): 299-309.
[28] 蔡丽娟. 正交法在近红外光谱分析中的应用[D]. 哈尔滨: 哈尔滨理工大学, 2009.
[29] VISCARRA ROSSEL R A. ParLeS: software for chemometric analysis of spectroscopic data[J]. Chemometrics and Intelligent Laboratory Systems, 2008, 90(1): 72-83.
Infrared Spectroscopy Combined with Multivariate Statistical Analysis to Quickly Identify Different Species of Bolete Mushrooms
YANG Tianwei
1, 2, ZHANG Ji
2, SHI Yundong
3, LI Tao
3, WANG Yuanzhong
2,*, LIU Honggao
1,*
(1. College of Agronomy and Biotechnology, Yunnan Agricultural University, Kunming 650201, China;2. Institute of Medicinal Plants, Yunnan Academy of Agricultural Sciences, Kunming 650200, China;3. College of Resources and Environment, Yuxi Normal University, Yuxi 653100, China)
Abstract:Fourier transform infrared spectroscopy combined with multivariate statistical analysis was used to establish a rapid method for the identifi cation of different species of edible bolete mushrooms. The infrared spectral characteristics of 93 bolete samples of 10 different species were analyzed. The original infrared spectra were pretreated by multiplicative signal correction (MSC), standard normal variate (SNV), second derivative, Norris smooth, orthogonal signal correction (OSC) and wavelet compression. The optimized spectral data were used to establish a mahalanobis distance classifi cation model and a partial least squares discriminant analysis (PLS-DA) model. The results showed that the characteristic absorption peaks of protein, polysaccharide and amino acid appeared at wavenubmers around 3 325, 2 934, 2 927, 1 637, 1 547, 1 402, 1 375,1 259, 1 453, 1 081, and 1 029 cm -1. The cumulative contribution rates were 95.58% and 95.54% in t he PLS-DA model based on MSC + SD + ND (15:5) and SNV + SD + ND (15:5) pretreatment, respectively. The Mahalanobis distance classification model was established base on the two pretreatment m ethods and the prediction accuracies of validation
set were 90% and 95% respectively. Bolete species could not be well distinguished by the PLS-DA model, when the data were pretreated by the MSC + SD + ND (15:5) and SNV + SD + ND (15:5). PLS-DA analysis of the original spectra after optimization with orthogonal signal correction wavelet compression (OSCW) could distinguish different species of boletes. The Mahalanobis distance classification model could reflect the classification of the samples and compute the greatest similarity with the tested species, which can provide a reliable basis for the classifi cation of edible mushrooms and for the identifi cation of unknown species. OSCW pretreatment combined with PLS-DA analysis can effectively identify different species of boletes, providing an auxiliary method for the identifi cation of wild edible mushrooms.
Key words:infrared spectroscopy; boletes; discrimination; Mahalanobis distance; partial least squares discriminant analysis (PLS-DA)
收稿日期:2015-01-21
基金项目:国家自然科学基金地区科学基金项目(31260496;31160409;31460538);国务院农村综合改革专项(2014NG007-18);云南省教育厅科学研究基金项目(2013Z074)
作者简介:杨天伟(1989—),男,硕士研究生,主要从事野生食用菌资源研究。E-mail:yangtianweizj@126.com
*通信作者:王元忠(1981—),男,助理研究员,硕士,主要从事药用植物和真菌资源研究。E-mail:yzwang1981@126.com
刘鸿高(1974—),男,教授,博士研究生,主要从事野生食用菌资源研究。E-mail:honggaoliu@126.com
中图分类号:TS201.2
文献标志码:A
文章编号:1002-6630(2015)24-0116-06
doi:10.7506/spkx1002-6630-201524020