荧光假单胞菌、沙门氏菌和单增李斯特菌多重PCR检测方法的建立
胡冰雪
1,舒沿沿
1,潘道东
1,2,
*,曾小群
1,吴 振
1
(1.宁波大学 浙江省动物蛋白食品精深加工技术重点实验室,浙江 宁波 315211;2.南京师范大学 国家乳品加工技术研发分中心,江苏 南京 210097)
摘 要:针对肉制品中易污染的荧光假单胞菌、沙门氏菌和单增李斯特菌等有害微生物,通过3 种标准菌株及肉制品建立多重聚合酶链式反应方法,实现肉制品中这3 种菌的同时、快速检测。利用荧光假单胞菌的gyrB基因、沙门氏菌的invA基因和单增李斯特菌的hlyA基因设计3 对特异性引物,在确定引物特异性的基础上,对3 种标准菌株及在冷却肉上过夜富集后进行灵敏度检测。结果表明,该多重聚合酶链式反应方法对于同时检测这3 种有害微生物具有高度的特异性;同时检测这3 种菌时,纯菌DNA检测限可达1 pg/μL;将3 种菌一起接种到冷却肉中35 ℃过夜培养后,荧光假单胞菌、沙门氏菌和单增李斯特菌的检测限分别可达到9、5、70 CFU/mL。
关键词:多重聚合酶链式反应;检测方法;荧光假单胞菌;沙门氏菌;单增李斯特菌
肉制品因其需求量大、营养丰富及易于消化等特点而受到各个年龄阶层的欢迎。然而,由于其水分含量高、必需营养物质丰富,为各种腐败菌、致病菌的生长繁殖提供了基质,导致肉制品容易受到各种微生物污染
[1]。荧光假单胞菌(Pseudomonas fluorescens)是一种嗜冷微生物,易污染冷藏中的生肉、生牛乳等高脂肪、高蛋白的食品,在冷藏的生肉、碎肉中都是优势菌群。临床研究表明,P. fluorescens自溶后释放内毒素,内毒素中的磷脂成分污染了血制品,会导致病人输血后产生不可逆的休克、弥漫性血管内凝血
[2],是典型的条件致病菌。沙门氏菌(Salmonella)存在地域性广、发病率高和大范围食物污染等特点,在引起食物中毒的病原菌中一直占据首位
[3-4]。单核细胞增生李斯特氏菌(Listeria monocytogenes)在4 ℃仍可生长,主要通过肉乳及其制品引起人的感染发病,成为仅次于Salmonella感染引起的第二号致命的食源性感染疾病
[5]。因此,快速检测和鉴别冷却肉中有害微生物是保障肉品质量安全,防止食源性疾病爆发的有效手段。
对于食品微生物的检测方法主要有传统培养法、免疫学方法和分子生物学方法。传统培养法过程繁琐、耗时长、工作量大,一般需要4~7 d才能出检测结果。免疫学方法具有特异性好、敏感度高的优点,但由于微生物血清型复杂,检测中常出现假阳性结果,阳性结果需要其他实验进一步确认。目前,随着分子生物学技术的发展,聚合酶链式反应(polymerase chain reaction,PCR)成为肉制品中有害微生物检测的重要技术手段
[6]。在PCR基础上发展起来的多重-PCR(multiplex PCR,m-PCR)技术,是在普通PCR基础上加以改进,在同一PCR反应体系中加入多对特异性引物,同时扩增出多个目的核酸片段,快速检测或鉴定多种微生物的技术
[7],该技术不需要大量、昂贵的实验设备进行数据采集,同时对于肉制品中主要的有害微生物又能实现快速、经济、高特异性和高灵敏度的检测
[8]。然而,m-PCR方法的建立主要集中于Salmonella
[9]、L. monocytogenes
[10]等典型致病菌,对于极易污染肉乳制品的P. fl uorescens快速、有效的检测方法尚不成熟。虽然目前尚未出现由P. fl uorescens所引起的大规模食物中毒事件,但随着人们饮食习惯的改变、冰箱的普及和不合理使用,食品中该菌对健康的危害及引起食物中毒的可能性会越来越越大,因此建立该菌的快速检测方法迫在眉睫。但是假单胞菌属是一个很大的群体,Palleroni等
[11]研究表明其至少可以分为5 个生物变型,这就使得其分子生物学检测方法的建立存在一定难度,PCR方法是现阶段最快速最有效的检测P. fluorescens的技术,而其m-PCR检测方法的建立极不成熟。
鉴于此,本研究旨在建立一种应用于复杂食品基质如冷却肉中P. fluorescens、Salmonella、L. monocytogenes的同时、快速、高效的m-PCR检测方法,对于肉制品的常规监测及减少有害微生物的潜在危害具有重要意义。
1 材料与方法
1.1 材料、试剂与仪器
1.1.1 实验菌株
实验所用菌株名称和来源见表1,部分菌株从菌种库直接购买,另外一部分由浙江省疾病预防控制中心提供。实验菌株均用甘油管-40 ℃保藏。
表1 菌株及引物特异性验证
Table1 Specificity of the primers for different bacterial strains
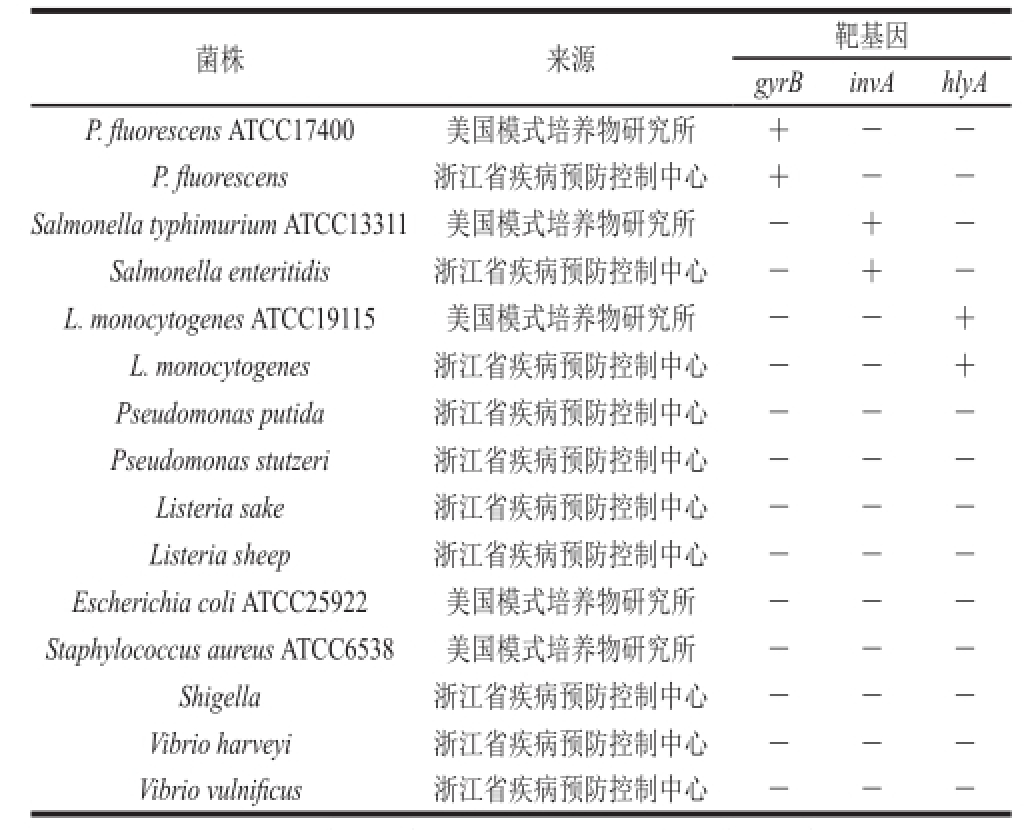
注:+. PCR扩增产物检测有条带;-. PCR扩增产物检测无条带。表3同。
菌株来源靶基因gyrBinvAhlyA P. fl uorescens ATCC17400美国模式培养物研究所+--P. fl uorescens浙江省疾病预防控制中心+--Salmonella typhimurium ATCC13311美国模式培养物研究所-+-Salmonella enteritidis浙江省疾病预防控制中心-+-L. monocytogenes ATCC19115美国模式培养物研究所--+ L. monocytogenes浙江省疾病预防控制中心--+ Pseudomonas putida浙江省疾病预防控制中心---Pseudomonas stutzeri浙江省疾病预防控制中心---Listeria sake浙江省疾病预防控制中心---Listeria sheep浙江省疾病预防控制中心---Escherichia coli ATCC25922美国模式培养物研究所---Staphylococcus aureus ATCC6538美国模式培养物研究所---Shigella浙江省疾病预防控制中心---Vibrio harveyi浙江省疾病预防控制中心---Vibrio vulnifi cus浙江省疾病预防控制中心---
1.1.2 培养基与试剂
营养肉汤培养基、脑心浸液培养基、营养琼脂培养基;实验所用引物序列由上海英潍捷基贸易有限公司合成,测序工作委托上海生工完成;DNA聚合酶等PCR反应试剂 北京全式金生物技术有限公司;细菌DNA抽提试剂盒(D3350) 大连宝生物工程有限公司。
1.1.3 仪器与设备
Multigene Thermal Cycler型PCR仪 美国Labnet公司;Universal HoodⅡ型凝胶成像分析系统 美国Bio-Rad公司;LDZX-50KBS型高压灭菌锅 上海申安医疗器械厂;TH-YJ-1450A/B型净化工作台 苏州华科净化设备有限公司;Centerifuge 5415R型台式高速冷冻离心机 德国Eppendorf公司;Mini Drop型超微量分光光度计 上海依科赛生物制品有限公司;H2500R-2型和H-2050R型高速冷冻离心机 长沙湘仪离心机仪器有限公司;DYCP-31DN型核酸电泳仪 北京六一生物科技有限公司;AL204型和PL4002型电子天平、FE20型实验室pH计 美国Mettler Toledo公司;GT16-3M型迷你离心机 杭州米欧仪器有限公司;HtPot40型恒温金属浴麦稷桥国际贸易有限公司;HJ-4型磁力搅拌器 常州申光仪器有限公司;QHZ-12A型恒温振荡培养箱 江苏盛蓝仪器制造有限公司。
1.2 方法
1.2.1 引物设计
根据文献[12-15]中提到的3 种菌的特异性靶基因,即P. fluorescens的gyrB基因、Salmonella的invA基因和L. monocytogenes的hlyA基因,应用Primer premier 5.0设计引物,确保引物之间无二聚体形成,并对各引物间的互补性、最佳退火温度进行了分析,获得了3 对特异性引物(表2)。3 对引物由上海英潍捷基贸易有限公司合成,扩增片段大小依次为271、482 bp和592 bp。
表2 3对引物序列及相关参数
Table2 Sequences and parameters of the primers
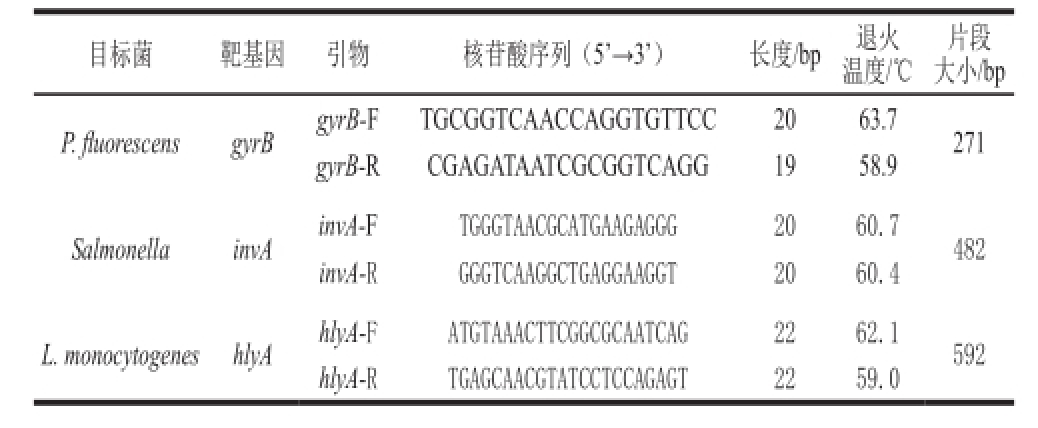
片段大小/bp P. fl uorescensgyrBgyrB-FTGCGGTCAACCAGGTGTTCC2063.7271 gyrB-RCGAGATAATCGCGGTCAGG1958.9 SalmonellainvAinvA-FTGGGTAACGCATGAAGAGGG2060.7482 invA-RGGGTCAAGGCTGAGGAAGGT2060.4 L. monocytogeneshlyAhlyA-FATGTAAACTTCGGCGCAATCAG2262.1592 hlyA-RTGAGCAACGTATCCTCCAGAGT2259.0目标菌靶基因引物核苷酸序列(5'→3')长度/bp退火温度/℃
1.2.2 细菌DNA提取
购买的标准菌株采用推荐培养基活化后涂平板,挑取单个菌落在35 ℃恒温振荡培养箱中过夜扩大培养,根据浑浊度判断细菌生长状况,待生长至稳定期后取3 mL菌液,按Omega biotek公司Bacteria DNA Kit试剂盒(D3350)的操作说明提取细菌基因组DNA,作为PCR反应的模板。
1.2.3 单重PCR反应体系的建立
单重PCR扩增采用50 μL反应体系:10×PCR缓冲液5 μL,各2.5 mmol/L的dNTPs 4 μL,2.5 U/μL的DNA聚合酶0.75 μL,上、下游引物各1 μL,模板1 μL,加ddH
2O补足至50 μL。反应条件如下:94 ℃预变性4 min,94 ℃变性30 s,62 ℃退火30 s,72 ℃延伸1 min,扩增30 个循环;72 ℃延伸10 min。
PCR扩增后的产物采用质量浓度为3 g/100 mL的琼脂糖、120 V电压进行凝胶电泳,割胶回收后的产物送上海生工测序鉴定。
1.2.4 m-PCR反应体系建立及优化
以P. fluorescens、Salmonella、L. monocytogenes提取的DNA为模板,与各种反应试剂混合在一起进行m-PCR反应条件和体系的优化,以建立最佳的m-PCR方法。优化的条件主要包括引物浓度配比、退火温度、退火时间、DNA聚合酶量等。
1.2.5 m-PCR特异性实验
m-PCR引物特异性实验分为两部分,首先是验证引物对目标菌DNA的特异性:分别向3 种目标菌的单一DNA、两两混合DNA、全部混合DNA中加入3 对混合引物,进行PCR扩增。其次是验证引物对非目标菌DNA没有非特异性扩增现象。
1.2.6 m-PCR灵敏度实验
首先,将3 种菌的DNA质量浓度调整为10
2ng/μL,再梯度稀释为10
1、10
0、10
-1、10
-2、10
-3、10
-4ng/μL,采用优化后的方法进行m-PCR扩增,测定能检测到PCR扩增条带的最低模板质量浓度。
1.2.7 人工模拟污染肉制品检测灵敏度
污染肉汤样品的方法参照Kumar等
[16]的方法稍作改进。取100 g冰鲜鸭肉加入100 mL营养肉汤培养液中,彻底漂洗后在无菌均质袋中均质1 min,分成10 等分,3 000 r/min离心5 min,悬浮液取出后用0.45 μm滤膜过滤,过滤好的肉汤冷藏于-20 ℃条件下待用。
将培养至稳定期的3 种菌梯度稀释,并对10
-6、10
-7、10
-8、10
-94个稀释度分别进行平板计数。同时接入3 种菌的梯度稀释液,使得每20 mL肉汤中3 种菌的最终浓度分别为10
0、10
1、10
2、10
3、10
4、10
5、10
6、10
7、10
8CFU/mL,对照组接入营养肉汤培养液。接种好菌液的肉汤在200 r/min条件下振荡10 min,使菌体充分悬浮,再往每份肉汤中加入80 mL营养肉汤培养液,使得样品体积∶总体积≈1∶10,将肉汤与培养基混合均匀后在35 ℃恒温振荡培养箱中过夜培养,各浓度梯度取1 mL菌液用Omega biotek公司bacteria DNA kit(D3350)提取DNA作为PCR模板,从而确定m-PCR方法检测3 种菌在肉制品中过夜富集后的灵敏度。
2 结果与分析
2.1 单重PCR特异性实验
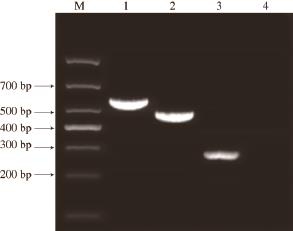
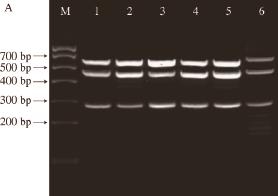
图1 单重PCR产物的凝胶电泳
Fig.1 Agarose gel electrophoresis of simplex PCR products
M. 1 000 bp DNA Marker;1. L. monocytogenes;2. Salmonella;3. P. fluorescens;4.阴性对照。
3 种目标菌进行单重PCR扩增,验证引物与模板之间的特异性,实验结果如图1所示。泳道1的扩增条带位于500~700 bp之间,与L. monocytogenes的PCR扩增产物片段长度的实验预期相符;泳道2的扩增条带位于400~500 bp之间,与Salmonella的PCR扩增产物片段长度的实验预期相符;泳道3的扩增条带位于200~300 bp之间,与P. fluorescens的PCR扩增产物片段长度的实验预期相符。将PCR扩增后的3 个目的片段测序结果在NCBI中进行BLAST比对分析,比对结果显示,测序结果与NCBI中L. monocytogenes相应基因序列的同源性为100%,与Salmonella相应基因序列的同源性为99%,与P. fl uorescens相应基因序列的同源性为99%。
2.2 m-PCR扩增及反应条件的优化
分别对3 种目标菌的3 对引物浓度配比、退火温度、退火时间、DNA聚合酶量进行优化,反应体系优化结果(图2)如下:10×PCR缓冲液5 μL,各2.5 mmol/L的dNTPs 4 μL,2.5 U/μL的DNA聚合酶1 μL,3 种菌的上、下游引物各1 μL,模板各1 μL,ddH
2O补足至50 μL。反应过程如下:94 ℃预变性4 min,94 ℃变性30 s,61.7 ℃退火20 s,72 ℃延伸1 min,扩增30 个循环,72 ℃延伸10 min。PCR产物用3%琼脂糖凝胶电泳鉴定,出现3 条清晰的条带,大小分别约为592、482、271 bp。
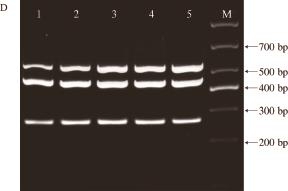
图2 m-PCR反应条件的优化
Fig.2 Optimization of multiplex PCR reaction conditions
A.引物浓度配比优化,Salmonella、L. monocytogenes、P. fluorescens引物浓度分别为1:0.1、0.1、0.1 μmol/L;2:0.2、0.2、0.1 μmol/L;3:0.1、0.2、0.2 μmol/L;4:0.2、0.1、0.2 μmol/L;5:0.2、0.2、0.2 μmol/L;6:0.2、0.2、0.3 μmol/L。B.退火温度优化,1~8分别为54.3、55.9、57.7、59.4、60.2、61.7、63.1、64.3 ℃。C.延伸时间优化,1~4分别为20、30、45、60 s。D. DNA聚合酶量优化,1~5分别为1.25、1.875、2.5、3.125、3.75 U。M. 1 000 bp DNA Marker。
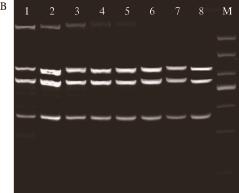
2.3 m-PCR反应特异性实验结果
为验证本实验所用引物的特异性,上述3 对引物先与P. fl uorescens、Salmonella、L. monocytogenes 3 种目标菌的DNA进行错配,按照上述优化后的m-PCR反应体系和条件进行扩增,结果如图3所示。其中,泳道1为三模板,2~4为双模板,5~7为单一模板,均加入3 对混合引物进行PCR扩增。结果表明每对引物只与对应模板结合,扩增出了相应大小的目的片段。
同时,验证3 对引物与其他非致病菌如浙江省疾病预防控制中心提供的Pseudomonas putida、Pseudomonas stutzeri、Listeria sake、Listeria sheep、Shigella、Vibrio harveyi、Vibrio vulnifi cus、Escherichia coli ATCC25922、Staphylococcus aureus ATCC6538进行PCR反应。以所有实验菌株提取的单一DNA为模板,同时加入3 对引物进行扩增,结果如图1所示。
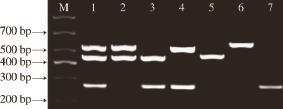
图3 m-PCR特异性实验
Fig.3 Specificity of the m-PCR assay
M. 1 000 bp DNA Marker。1. L. monocytogenes、Salmonella、P. fl uorescens;2. L. monocytogenes、Salmonella;3. Salmonella、P. fluorescens;4. L. monocytogenes、P. fluorescens;5. Salmonella;6. L. monocytogenes;7. P. fluorescens。
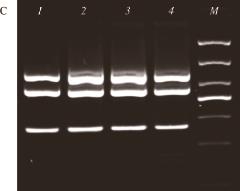
2.4 m-PCR灵敏度实验结果
首先将从3 种菌提取的DNA质量浓度调整为10
2ng/μL,再按照10
1、10
0、10
-1、10
-2、10
-3、10
-4ng/μL分别进行质量浓度梯度稀释,采用建立的m-PCR方法扩增后,结果显示模板质量浓度为10
-3ng/μL时能出现微弱的条带,当模板质量浓度低于10
-3ng/μL时条带消失,因此能同时检测到3 种菌的最低模板质量浓度为10
-3ng/μL(图4)。
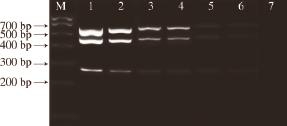
图4 m-PCR灵敏度实验
Fig.4 Sensitivity of the m-PCR assay
M. 1 000 bp DNA Marker。1~7质量浓度分别为10
2、10
1、10
0、10
-1、10
-2、10
-3、10
-4ng/μL。
2.5 人工模拟污染实际样品的灵敏度检测结果
考虑到冷却肉中原有腐败菌的数量和肉品基质对目标菌生长和检测的影响,本实验利用人工模拟实际样品污染,对m-PCR方法检测实际样品时的灵敏度进行了测定。通过富集培养之后,模拟肉汤中P. fluorescens、Salmonella、L. monocytogenes的检测限分别为9、5、70 CFU/mL(表3)。
表3 人工模拟肉制品污染的m-PCR灵敏度检测
Table3 Sensitivity of m-PCR for detection of artificially contaminated meat samples

稀释度P. fl uorescensSalmonellaL. monocytogenes菌落数/(CFU/mL) m-PCR菌落数/(CFU/mL)m-PCR菌落数/(CFU/mL)m-PCR 10
-1×10
8+×10
8+×10
8+ 10
-2×10
7+×10
7+×10
7+ 10
-3×10
6+×10
6+×10
6+ 10
-4×10
5+×10
5+×10
5+ 10
-5×10
4+×10
4+×10
4+ 10
-6(1.72±1.3)×10
3+(1.14±0.8)×10
3+(2.12±0.38)×10
3+ 10
-7(2.6±0.5)×10
2+(3.06±0.31)×10
2+(3.5±1.2)×10
2+ 10
-8(7±1.00)×10+(3.42±1.3)×10+(7±1.56)×10+ 10
-9(9±1.00)+(5.3±0.5)+(8.5±0.92)-
3 讨 论
传统的检测手段和PCR方法不利于带有多种微生物的复杂食品样品的检测,因此兼顾检测技术的自身优势和企业操作性的m-PCR已成为一种同时检测多种微生物的快速、可靠的手段,随着m-PCR技术的不断建立和发展,检测灵敏度将不断提高,在一定程度上,m-PCR可能发展成一种半定量甚至定量的检测方法
[17],在各大检测技术中心具有良好的应用前景。本研究利用该技术成功建立了同时检测P. fluorescens、Salmonella、 L. monocytogenes的方法,适用于肉乳制品中这3 种菌的同时快速检测。
m-PCR技术中,靶基因的选择直接决定着检测方法的可靠性和真实性。P. fluorescens的PCR检测方法还处于起步阶段,而其m-PCR检测方法的建立有待更多的研究。Scarpellini等
[18]针对16S rRNA基因设计了引物16SPSEFLuF/R,建立了区别P. fluorescens及第一组假单胞菌的方法;Martins等
[19]根据apr基因设计出一对PCR引物检测奶粉中的假单胞菌等嗜冷微生物;Irenehanning等
[20]利用gyrB基因分析导致冷冻鸡肉腐败的微生物,将P. fl uorescens和莓实假单胞菌这两种细菌和其他假单胞菌分开;杨一林
[12]通过生物信息学筛选共得到P. fl uorescens的4 个备选检测靶点gyrB、16S~23S rDNA基因区间序列、AraC、PFLU,设计引物并进行PCR验证,结果表明依据gyrB基因设计的引物扩增产物与预期片段一致,且无非特异性扩增。因此本实验选择P. fluorescens的gyrB基因作为检测的靶基因。目前检测Salmonella的靶基因有ssrA、ompC、invA、pefA、sefA、agfA等,研究表明特异性最强的有pefA、sefA和invA,其中报道的已有2 000多种Salmonella血清型的菌株带有侵袭蛋白基因invA,很多研究也表明该基因序列为属间特有
[13],因此本实验选择Salmonella的invA基因作为检测的靶基因。对于L. monocytogenes,很多研究针对不同的独立基因(iap、inlB、16S rRNA等)进行检测,应用最多的为编码溶血素的基因hlyA
[14],相关研究已证明其种属特异性
[15],因此本实验选择L. monocytogenes的hlyA基因作为检测的靶基因。根据选定的3 个靶基因设计3 对特异性引物,PCR扩增后的产物跑琼脂糖凝胶电泳的结果与预期目的片段长度相符,且上海生工测序后的序列在NCBI中进行BLAST比对分析,表明即为目标菌株。
对于一个有效的m-PCR方法,退火温度、时间、引物浓度配比、Mg
2+浓度与模板量的平衡等因素是非常重要的
[21]。该方法优化得到实验条件为引物浓度0.2 μmol/L、退火温度61.7 ℃、退火时间20 s、DNA聚合酶量2.5 U,这些条件的优化有利于m-PCR检测纯目标菌株及人工模拟肉制品同时污染3 种目标菌株时得到更明确、清晰的条带,从而得到更为准确的检测限。m-PCR方法具有灵敏度高的优点,刘贝贝
[22]根据无乳链球菌的sip基因、海豚链球菌的LctO基因以及P. fluorescens基因组的16SPSEFLuF/16SPSER基因序列设计引物,建立m-PCR方法,结果显示能同时检测到3 种菌的最低核酸浓度为6.557×10
-2ng/μL。Zheng Pingguan等
[23]用m-PCR特异性扩增L. monocytogenes的hlyA基因,Salmonella的invA基因,大肠杆菌O157:H7的rfbE基因,金黄色葡萄球菌的nuc基因以及小肠结肠炎耶尔森氏菌的ail基因,优化后的m-PCR方法在猪肉制品中的检测水平分别为9、142、670、51、33 CFU/mL。本研究建立的m-PCR方法同时检测3 种纯菌的DNA时,检测限可达1 pg/μL;人工模拟3 种菌同时污染肉制品,经过夜培养后,P. fluorescens、Salmonella、L. monocytogenes的检测限分别可达到9、5、70 CFU/mL。有报道表明大多数致病菌导致人体感染往往是其摄入量已超过10
3CFU/mL
[24],因此建立m-PCR方法的灵敏度对于威胁公共卫生安全的P. fluorescens、Salmonella和L. monocytogenes的检测是有效的。
4 结 论
本实验优化得到的m-PCR扩增条件为引物浓度0.2 μmol/L、退火温度61.7 ℃、退火时间20 s、DNA聚合酶量2.5 U,这些条件的优化提高了反应体系的特异性和灵敏度。建立的m-PCR方法同时检测到P. fluorescens、Salmonella、L. monocytogenes的DNA质量浓度为1 pg/μL;人工模拟3 种菌同时污染肉制品经过夜培养后,3 种菌的检测限分别可达到9、5、70 CFU/mL。
本研究建立的检测肉制品中常见的3 种有害微生物的m-PCR方法具有很高的特异性和灵敏度,该方法的建立相较于传统的培养检测方法可节约大量的劳力、试剂、时间等,对于某些企业或分析检验中心大批量样品的监测具有指导意义。
参考文献:
[1] LI M Y, ZHOU G H, XU X L, et al. Changes of bacterial diversity and main flora in chilled pork during storage using PCR-DGGE[J]. Food Microbiology, 2006, 23(7): 607-611. DOI:10.1016/j.fm.2006.01.004.
[2] PENA C, SUAREZ C, OCAMPO-SOSA A, et al. Effect of adequate single-drug vs combination antimicrobial therapy on mortality in Pseudomonas aeruginosa bloodstream infections: a post hoc analysis of a prospective cohort[J]. Clinical Infectious Diseases, 2013, 57(2):208-216. DOI:10.1093/cid/cit223.Epub2013Apr11.
[3] FREITAS C G, SANTANA A P, SILVA P H, et al. PCR multiplex for detection of Salmonella Enteritidis, Typhi and Typhimurium and occurrence in poultry meat[J]. International Journal of Food Microbiology, 2010, 139(1/2): 15-22. DOI:10.1016/ j.ijfoodmicro.2010.02.007.Epub2010 Feb13.
[4] OHL M E, MILLER S I. Salmonella: a model for bacterial pathogenesis[J]. Annual Review of Medicine, 2001, 52(1): 259-274. DOI:10.1146/annurev.med.52.1.259.
[5] GOULET V, HEDBERG C, MONNIER A, et al. Increasing incidence of listeriosis in France and other European countries[J]. Emerging Infectious Diseases, 2008, 14(5): 734-740. DOI:10.3201/ eid1405.071395.
[6] XU Y G, CUI L C, TIAN C Y, et al. A multiplex polymerase chain reaction coupled with high-performance liquid chromatography assay for simultaneous detection of six foodborne pathogens[J]. Food Control, 2012, 25(2): 778-783. DOI:10.1016/j.foodcont.2011.12.014.
[7] SEVERGNINI M, CREMONESII P, CONSOLANDI C, et al. Advances in DNA microarray technology for the detection of foodborne pathogens[J]. Food and Bioprocess Technology, 2011, 4(6):936-953. DOI:10.1007/s11947-010-0430-5.
[8] BAI S L, LI S C, YAO T, et al. Rapid detection of eight vegetable oils on optical thin-film biosensor chips[J]. Food Control, 2011, 22(10):1624-1628. DOI:10.1016/j.foodcont.2011.03.019.
[9] BABU L, REDDY P, MURALI H S, et al. Optimization and evaluation of a multiplex PCR for simultaneous detection of prominent foodborne pathogens of Enterobacteriaceae[J]. Annals of Microbiology, 2013, 63(4): 1591-1599. DOI:10.1007/s13213-013-0622-0.
[10] KUMAR A, GROVER S, BATISH V K. Exploring specific primers targeted against different genes for a multiplex PCR for detection of Listeria monocytogenes[J]. Biotech, 2015, 5(3): 261-269. DOI:10.1007/s13205-014-0225-x.
[11] PALLERONI N, KUNISAWA R, CONTOPOULOU R, et al. Nucleic acid homologies in the genus Pseudomonas[J]. International Journal of Systematic Bacteriology, 1973, 23(4): 333-339. DOI:10.1099/00207713-23-4-333.
[12] 杨一林. 荧光假单胞菌PCR检测靶点的筛选及PCR检测体系的建立[D].上海: 上海交通大学, 2011.
[13] D'SOUZA D H, CRITZER F J, GOLDEN D A. Real-time reversetranscriptase polymerase chain reaction for the rapid detection of Salmonella using invA primers[J]. Foodborne Pathogens and Disease, 2009, 6(9): 1097-1106. DOI:10.1089/fpd.2009.0322.
[14] CHURCHILL R L, LEE H, HALL J C. Detection of Listeria monocytogenes and the toxin listeriolysin O in food[J]. Journal of Microbiological Methods, 2006, 64(2): 141-170. DOI:10.1016/ j.mimet.2005.10.007.
[15] CHOI W S, HONG C H. Rapid enumeration of Listeria monocytogenes in milk using competitive PCR[J]. International Journal of Food Microbiology, 2003, 84(1): 79-85. DOI:10.1016/ S0168-1605(02)00401-4.
[16] KUMAR S, BALAKRISHNA K, BATRA H. Detection of Salmonella enterica serovar Typhi (S. Typhi) by selective amplification of invA, viaB, fl iC-d and prt genes by polymerase chain reaction in mutiplex format[J]. Letters in Applied Microbiology, 2006, 42(2): 149-154. DOI:10.1111/j.1472-765X.2005.01813.x.
[17] SETTANNI L, CORSETTI A. The use of multiplex PCR to detect and differentiate food and beverage-associated microorganisms: a review[J]. Journal of Microbiological Methods, 2007, 69(1): 1-22. DOI:10.1016/j.mimet.2006.12.008.
[18] SCARPELLINI M, FRANZETTI L, GALLI A. Development of PCR assay to identify Pseudomonas fluorescens and its biotype[J]. Fems Microbiology Letters, 2004, 236(2): 257-260. DOI:10.1111/j.1574-6968.2004.tb09655.x.
[19] MARTINS M L, DEARAUJO E F, MANTOVANI H C, et al. Detection of theaprgene in proteolytic psychrotrophic bacteria isolated from refrigerated raw milk[J]. International Journal of Food Microbiology, 2005, 102(2): 203-211. DOI:10.1016/j.ijfoodmicro.2004.12.016.
[20] HANNING I, JARQUIN R, OLEARY A, et al. Polymerase chain reaction based assays for the detection and differentiation of poultry significant Pseudomonas[J]. Journal of Rapid Methods and Automation in Microbiology, 2009, 17(4): 490-502. DOI:10.1111/ j.1745-4581.2009.00185.x.
[21] MARKOULATOS P, SIAFAKAS N, MONCANY M. Multiplex polymerase chain reaction: a practical approach[J]. Journal of Clinical Laboratory Analysis, 2002, 16(1): 47-51. DOI:10.1002/jcla.2058.
[22] 刘贝贝. 无乳链球菌、海豚链球菌及荧光假单胞菌多重PCR检测方法的建立和应用[D]. 成都: 四川农业大学, 2013.
[23] ZHENG P G, YUN J, FENG G, et al. Rapid and simultaneous analysis of five foodborne pathogenic bacteria using multiplex PCR[J]. European Food Research and Technology, 2013, 237(4): 627-637. DOI:10.1007/s00217-013-2039-1.
[24] CROOK J. EPA Guidelines for water reuse[C]//Washington: United States Environmental Protection Agency, 1991: 2907-2912.
A Multiplex PCR Method for Simultaneous Detection of Pseudomonas fluorescens, Salmonella and Listeria monocytogenes
HU Bingxue
1, SHU Yanyan
1, PAN Daodong
1,2,*, ZENG Xiaoqun
1, WU Zhen
1
(1. Key Laboratory of Animal Protein Food Deep Processing Techhology of Zhejiang Province, Ningbo University, Ningbo 315211, China;2. Baranch of National Dairy Processing Technology Developing Center, Nanjing Normal University, Nanjing 210097, China)
Abstract:Pseudomonas fluorescens (P. fluorescens), Salmonella and Listeria monocytogenes (L. monocytogenes) are 3 species of bacteria that contaminate meat products. The aim of this work was to establish a feasible multiplex polymerase chain reaction (m-PCR) protocol for simultaneous and rapid detection of these 3 species of bacteria. The specifi c primers for the gyrB gene of P. fl uorescens, the invA gene of Salmonella and the hlyA gene of L. monocytogenes were designed and their specifi cities were determined. In addition, the sensitivities for detecting 3 standard strains and these strains enriched on meat overnight were tested as well. The results indicated that the m-PCR method had high specifi city and sensitivity. For simultaneous detection of these target microorganisms, the limit of detection (LOD) for pure DNA was 1 pg/μL, and the LOD for P. fl uorescens, Salmonella and L. monocytogenes were 9, 5, and 70 CFU/mL, respectively, by inoculating these 3 strains onto duck meat and overnight culturing at 35 ℃.
Key words:multiplex PCR; detection method; Pseudomonas fl uorescens; Salmonella; Listeria monocytogenes
DOI:10.7506/spkx1002-6630-201620036
中图分类号:R155.31
文献标志码:A
文章编号:1002-6630(2016)20-0209-06
引文格式:
胡冰雪, 舒沿沿, 潘道东, 等. 荧光假单胞菌、沙门氏菌和单增李斯特菌多重PCR检测方法的建立[J]. 食品科学, 2016, 37(20): 209-214. DOI:10.7506/spkx1002-6630-201620036. http://www.spkx.net.cn
HU Bingxue, SHU Yanyan, PAN Daodong, et al. A multiplex PCR method for simultaneous detection of Pseudomonas fluorescens, Salmonella and Listeria monocytogenes[J]. Food Science, 2016, 37(20): 209-214. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201620036. http://www.spkx.net.cn
收稿日期:2016-03-09
基金项目:浙江省科技厅公益性项目(2014C32051);“十二五”国家科技支撑计划项目(2015BAD17B02-3)
作者简介:胡冰雪(1992—),女,硕士研究生,研究方向为畜产品质量安全。E-mail:1411085832@nbu.edu.cn
*通信作者:潘道东(1964—),男,教授,博士,研究方向为畜乳产品加工与安全。E-mail:daodongpan@163.com