基于PKS Ⅰ基因海芦笋内生真菌及其次级代谢产物筛选鉴定
张俊楠
1,彭 洁
1,刘天行
1,2,辛志宏
1,*
(1.南京农业大学食品科技学院,江苏 南京 210095;2.南京农业大学图书馆,江苏 南京 210095)
摘 要:一般研究内生真菌的方法都无法预测内生真菌次级代谢产物的类型,本实验以Ⅰ型聚酮合成酶(polyketide synthase,PKS)基因为探针,野生海芦笋(Salicornia bigelovii Torr.)为原料,筛选出菌株Salicorn 3,并对其发酵产物中的化合物进行分离鉴定。结果表明:经过ITS1-5.8S-ITS4 rDNA同源序列鉴定的菌株Salicorn 3为圆弧青霉(Penicillium cyclopium),通过系统发育树分析并结合文献数据比较,表明该菌株具有产生聚酮类化合物的潜力。通过常压硅胶柱层析和Sephadex LH-20柱层析从其发酵产物中分离得到两个单体化合物,采用核磁共振和电喷雾质谱鉴定其化学结构分别为二酮哌嗪生物碱和脑苷脂C。
关键词:海芦笋;内生真菌;Ⅰ型PKS基因;聚酮类化合物;核磁共振
植物内生真菌是指那些寄生在健康植物的根茎叶部位,在宿主的组织器官或细胞内完成其部分或全部生活周期并对寄主植物不产生明显病害症状的一类真菌
[1-2]。1993年,Strobel等
[3]从太平洋短叶紫杉中分离出了一株内生真菌,该菌能够产生独特的抗癌活性物质——紫杉醇,由此启发人们从植物内生真菌中探索寻找出更多的生物活性物质。此后,大量结构新颖、活性多样的内生真菌次级代谢产物被相继发现,有些化合物已经被广泛应用于医药、工业和农业。
一般研究内生真菌的方法包括化学筛选、活性筛选或者化学与活性结合筛选,但是这些筛选方法都无法准确预测内生真菌次级代谢产物的类型,难以实现以化学结构为导向的定向筛选。近些年来分子生物信息学在不断发展,随着越来越多的微生物次级代谢产物合成酶系及其相关基因的测序完成,许多生物合成机理越来越清晰,新型微生物筛选方法应运而生。基于聚酮合成酶(polyketide synthase,PKS)功能基因的定向筛选方法,目标直指聚酮化合物的合成基因,成为发现新型天然活性物质的重要策略之一。已有研究证实,许多新发现的抑菌、抗肿瘤和抗病毒化合物都是由PKS合成的
[4-6]。PKS是生物合成次级代谢产物过程中的关键酶,对其合成基因的深入研究不仅能够提高新化合物的发现概率,而且可以预测所产生化合物的结构类型
[7-8]。而目前,基于PKS基因筛选目标菌株,探索化学结构多型性的研究鲜有报道。
海芦笋(Salicornia bigelovii Torr.),藜科盐角草属,又名富贵菜、海鹿茸等
[9],其抗盐能力极强,嫩茎中含有高达37%的可溶性盐分(干质量)
[10-11],是一种有茎无叶的草本盐生植物。本实验以盐生海芦笋为实验材料,以Ⅰ型PKS功能基因为筛选探针,采用表面消毒、平板分离纯化技术,获得4 株含有Ⅰ型PKS基因的海芦笋内生真菌Salicorn 1~4,进一步筛选出菌株Salicorn 2和Salicorn 3,利用分子生物学方法确定其属种地位;后对一株编号为Salicorn 3的真菌进行发酵,发酵产物经溶剂提取、柱层析分离纯化得到两个单体化合物,利用现代波谱学方法鉴定了这两个化合物的结构。
1 材料与方法
1.1 材料、培养基与试剂
野生盐生海芦笋采自新疆盐湖。
马铃薯葡萄糖琼脂培养基(potato dextrose agar,PDA)固体培养基(g/L):土豆200、葡萄糖20、琼脂20、NaCl 30、海水素10、海芦笋提取液250;真菌发酵液体培养基(g/L):土豆200、麦芽糖20、甘露醇20、葡萄糖10、味精5、蛋白胨5、酵母膏3,pH 6.0;细菌基础培养基(Luria-Bertani,LB)液体培养基(g/L):胰蛋白胨10、酵母粉5、NaCl 10,pH 7.2。
D3390-01 E.Z.N.A真菌DNA微量提取试剂盒 美国Omega公司;DR001AM PCR试剂、Ver.3.0 D823A琼脂糖凝胶、DNA提取试剂盒、D102A pMD19-T载体 日本TaKaRa公司;无水乙醇、NaClO 南京寿德实验器材有限公司;其他试剂为国产分析纯。
1.2 仪器与设备
生化恒温培养箱 上海新苗医疗器械制造有限公司;EPED-S3-D 型超纯水器 南京易普易达科技发展有限公司;Microfuge 22R台式微量冷冻离心机 美国Beckman Coulter公司;TP600梯度PCR仪 日本TaKaRa公司;JS-380C全自动数码凝胶成像分析仪 上海培清科技有限公司;JNM-ECP600型核磁共振仪 日本JEOL公司;Mariner API-TOF型质谱仪 美国应用生物系统公司;EYELAN-N型立式旋转蒸发仪 日本Tokyo Rikakikai有限公司;ULVAC DTC-22B型隔膜真空泵上海万库真空设备有限公司。
1.3 方法
1.3.1 海芦笋内生真菌的分离纯化及保藏
一般采取表面消毒法分离植物内生真菌
[12]。取一根新鲜干净无污染的野生海芦笋,将根茎部位剪成每段5~8 cm长,在超净工作台中无菌操作以下步骤:处理好的根茎置于为70%(体积分数,下同)酒精溶液中1 min,1% NaClO溶液中30 s,70%酒精1 min,无菌水1 min漂洗浸泡,反复3 次,无菌滤纸吸干表面水分,再接种于PDA固体培养基中,每板放3~5 段,于28 ℃恒温培养箱中培养3~8 d,待观察到切口处的培养基上长有菌丝后,挑取菌丝转移到新的PDA固体培养基上划线分离得到单菌落,挑取单菌落得到纯培养物,然后转移纯培养物到PDA斜面试管中,标记并于4 ℃冰箱保存备用。
1.3.2 Ⅰ型PKS基因的聚合酶链式反应(polymerase chain reaction,PCR)扩增
从保藏菌种的斜面培养基中挑取菌丝转移到新鲜PDA固体培养基上,于28 ℃培养箱培育3~5 d,待菌体几乎全部长满平板后备用。利用真菌DNA试剂盒提取PKS菌株基因组DNA,具体步骤详见操作说明书。将收集到的DNA样品进行1%琼脂糖凝胶电泳检测,4 ℃条件下保存备用。
参考文献:[9,13]选择3 对简并引物进行Ⅰ型PKS基因的筛选。3 对引物分别为:KAF
1/KAR
1、KAF
1/KAR
2、LC
1/LC
2C。KAF
1(5’-GAR KSI CAY GGI ACI GGI AC-3’);KAR
1(5’-CCA YTG IGC ICC RTG ICC IGA RAA-3’);KAR
2(5’-CCA YTG IGC ICC YTG ICC IGT RAA-3’);LC
1(5’-GAY CCI MGI TTY TTY AAY ATG-3’);LC
2C(5’-GTI CCI GTI CCR TGC ATY TC-3’)。PCR反应条件为:预变性95 ℃ 4 min;95 ℃变性30 s,50 ℃退火1 min, 72 ℃延伸2 min,共35 个循环;最后72 ℃延伸10 min
[9,14]。
从盐生海芦笋分离出4 株含Ⅰ型PKS基因的内生真菌,分别命名为Salicorn 1、Salicorn 2、Salicorn 3和Salicorn 4。
1.3.3 ITS rDNA基因片段的扩增
利用总DNA为模板,对真核生物ITS区域进行扩增,选用ITS通用扩增引物:IT S 1(5’-T C C G TAG GTGAACCTGCGG-3’)、ITS4(5’-TCCTCCGCTTATTGATATGC-3’)。PCR反应条件为:预变性94 ℃ 2 min; 94 ℃变性30 s, 59 ℃退火30 s,72 ℃延伸90 s,共35 个循环;最后72 ℃延伸7 min
[15]。
1.3.4 目的基因片段的测序及系统发育学分析
将PCR产物交由上海美吉生物医药科技有限公司进行测序。测序所得核酸序列在国家国立生物技术信息中心(National Center for Biotechnology Information,NCBI)数据库上进行BLAST比对,并下载同源性高的序列,利用Mega 5.0软件,以邻接法(Neighbor-Joining)构建系统进化树
[16],并通过Bootstrap进行置信度检测,自展数为1 000。
1.3.5 菌株Salicorn 3的发酵
将活化好的Salicorn 3菌株分别接种至5 瓶含400 mL液体发酵培养基的1 000 mL三角瓶中,静置培养3 个月,共发酵2 L。
1.3.6 化合物的分离与纯化
2 L发酵液用纱布过滤后得到发酵液和菌丝体,发酵液减压浓缩至0.5 L,加入等体积的乙酸乙酯,萃取3 次,合并乙酸乙酯提取液、减压浓缩至0.2 L得到发酵液浓缩液,菌丝体用等量的80%丙酮提取两次,减压浓缩无水后用等量的乙酸乙酯提取3 次,合并提取液并减压浓缩至0.3 L得到菌丝体浓缩液,合并发酵液和菌丝体浓缩液减压蒸干得到乙酸乙酯提取总浸膏(20 g),总浸膏经正相减压硅胶色谱柱,用石油醚-丙酮-甲醇梯度洗脱分离,洗脱的组分减压浓缩后经薄层层析(thin layer chromatography,TLC)检测后合并得到2 个组分(fraction,Fr):Fr.A~Fr.B。
Fr.A(2.1 g)经正相硅胶色谱柱,用环己烷-乙酸乙酯(100∶1,V/V)洗脱,得到Fr.A
1与Fr.A
2两个亚组分;Fr.A
2(187 mg)继续经正相硅胶色谱柱纯化,用纯氯仿洗脱,得到化合物1(15 mg)。
Fr.B(1.8 g)经正相硅胶色谱柱,用石油醚-丙酮(15∶1,V/V)进行洗脱,得到Fr.B
1组分;Fr.B
1(125 mg)经Sephadex LH-20纯化,用氯仿-甲醇(1∶1,V/V)洗脱,得到化合物2(20 mg)。
2 结果与分析
2.1 菌株Salicorn 1~4 Ⅰ型PKS基因扩增结果分析
以分离纯化得到的4 株内生真菌的DNA为模板,利用Ⅰ型PKS片段扩增引物进行PCR扩增,4 株菌株都扩增得到与预期值600~800 bp相近的Ⅰ型PKS基因片段,具体结果见表1,其中有两对引物从Salicorn 2和Salicorn 3中扩增到PKS基因,而从其余2 株菌中仅有一对引物扩增到PKS基因,提示产生更多种类的聚酮化合物,因此利用分子生物学方法对这两株菌进行鉴定。
表1 PKS基因的PCR扩增结果
Table 1 PCR amplification results of PKS gene
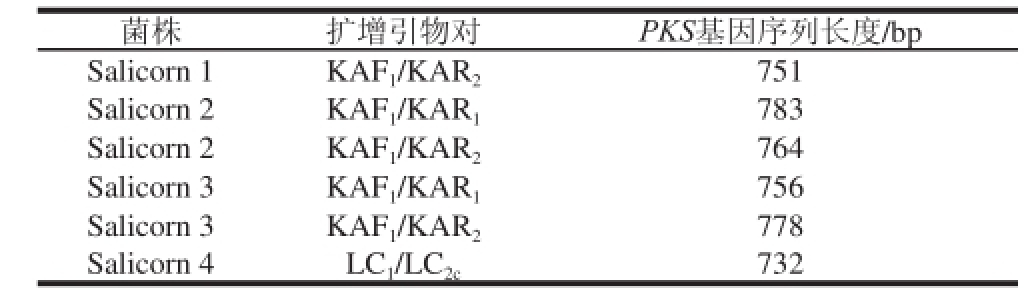
菌株扩增引物对PKS基因序列长度/bp Salicorn 1KAF
1/KAR
2751 Salicorn 2KAF
1/KAR
1783 Salicorn 2KAF
1/KAR
2764 Salicorn 3KAF
1/KAR
1756 Salicorn 3KAF
1/KAR
2778 Salicorn 4LC
1/LC
2c732
2.2 ITS进化树的构建与分析
菌株Salicorn 2经PCR扩增测序后获得的ITS基因序列长度为523 bp,该基因序列在GenBank上注册后获得登录号KJ810665。测序所得ITS基因序列在NCBI上进行BLAST比对,并利用Clastal X与Mega 5.0软件构建ITS系统发育树。如图1所示,从该进化树中可看出,同属不同种的菌株单独聚为一枝,菌株Salicorn 2与Aspergillus fl avus AF548060、Aspergillus fl avus D63696亲缘关系最接近,自展值为99。由此将菌株Salicorn 2鉴定为散囊菌纲、散囊菌目、发菌科、曲霉属、黄曲霉(Aspergillus fl avus)。
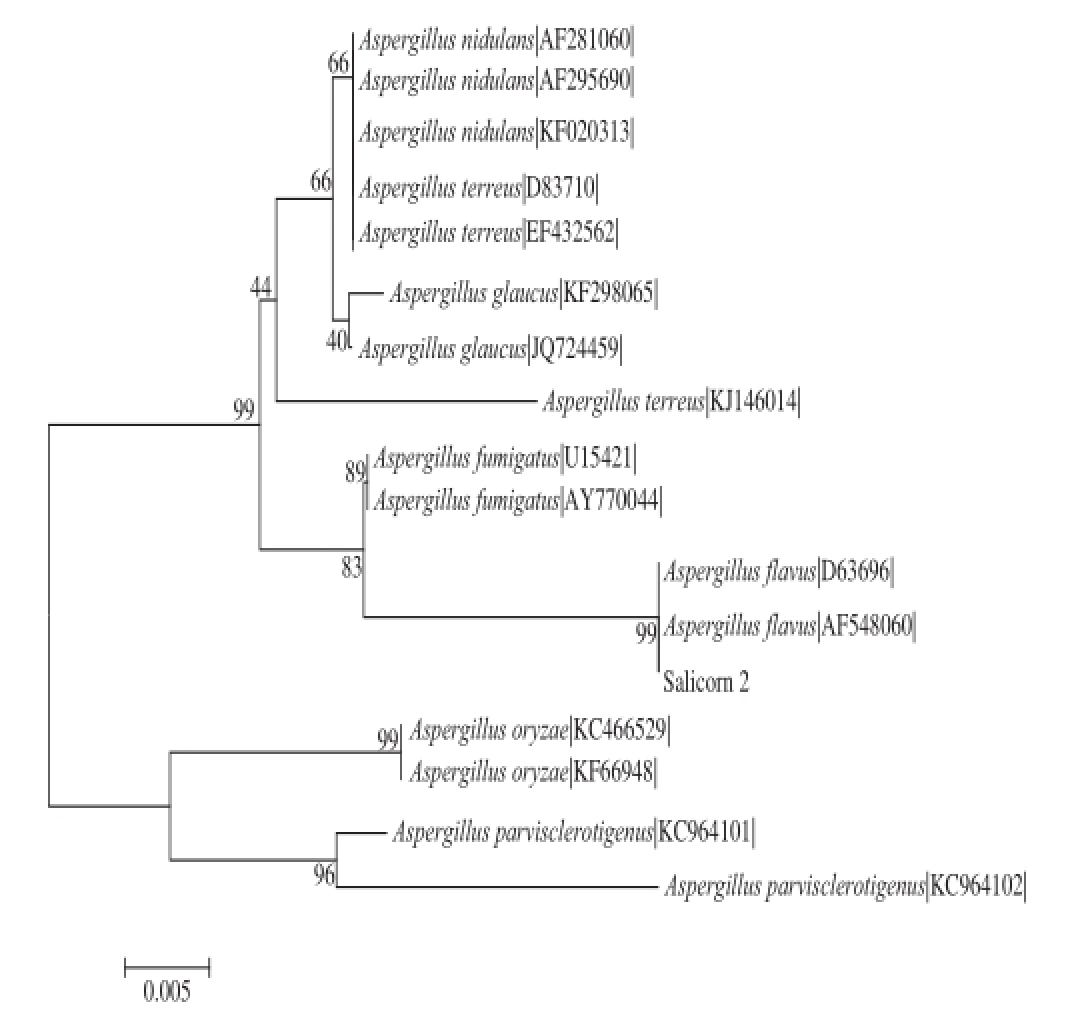
图1 Salicorn 2基于ITS基因序列构建的邻接树
Fig.1 Neighbor-Joining tree based on ITS gene sequences of strain Salicorn 2
菌株Salicorn 3的ITS基因序列经PCR扩增测序后获得基因序列长度为585 bp。将测序所得序列在NCBI上进行BLAST比对,发现菌株Salicorn 3的ITS基因序列与青霉属Penicillium sp.同源关系最近;利用Clastal X与Mega 5.0软件构建系统发育树,如图2所示,菌株Salicorn 3与圆弧青霉(Penicillium cyclopium JN942746、Penicillium cyclopium JN942747)聚为一枝,自展值为98,表现了相当近的亲缘关系,因此鉴定菌株Salicorn 3为半知菌纲、壳霉目、杯霉科、圆弧青霉属、圆弧青霉(Penicillium cyclopium)。
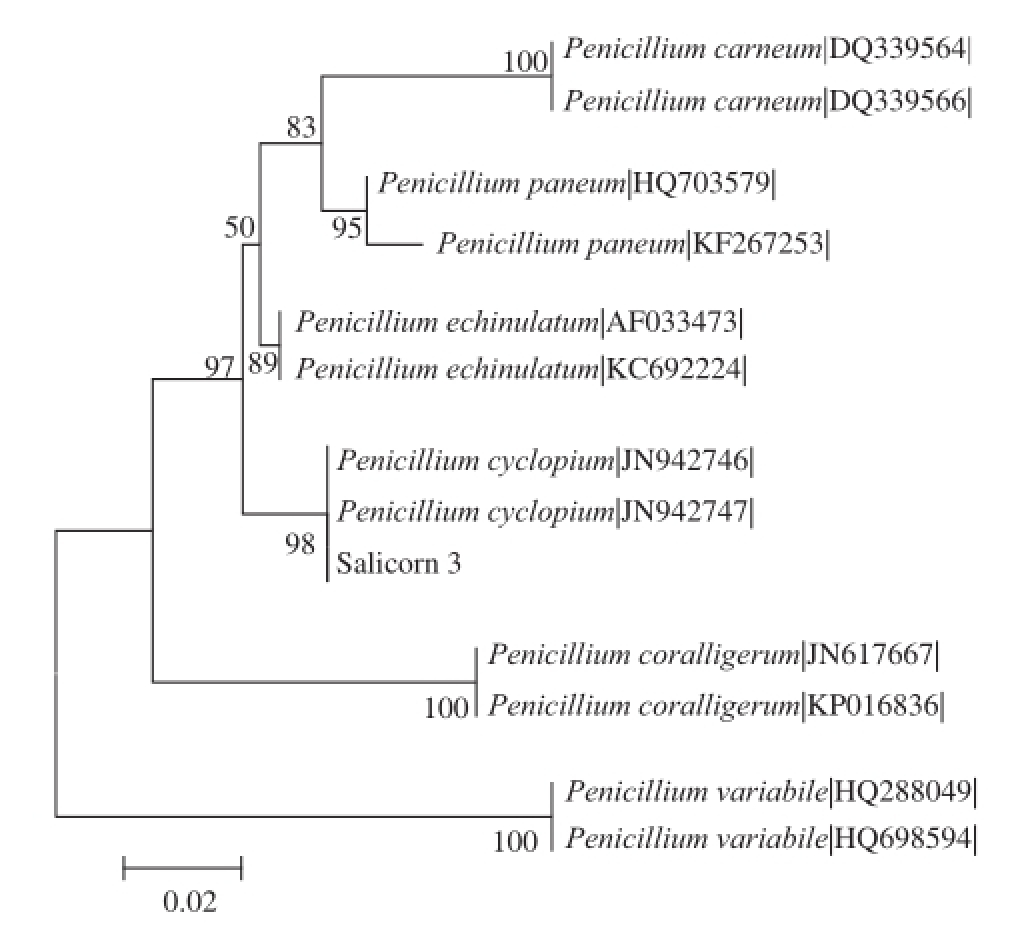
图2 Salicorn 3基于ITS基因序列构建的邻接树
Fig.2 Neighbor-Joining tree based on ITS gene sequences of strain Salicorn 3
由此,本实验确定了菌株Salicorn 2与Salicorn 3的种属分类地位分别为黄曲霉(Aspergillus flavus)以及圆弧青霉(Penicillium cyclopium)。鉴于黄曲霉能产生强致癌物——黄曲霉毒素,本实验选择对圆弧青霉进行发酵,从其发酵产物分离鉴定纯化聚酮化合物。
2.3 化合物结构解析
2.3.1 化合物1
白色固体,阴离子低分辨ESI-MS在m/z 691.4 [M-H]
-处给出准离子峰,提示该化合物的相对分子质量为692.4,结合
1H NMR和
13C NMR推测该化合物分子式为C
42H
40N
6O
4。
1H NMR(400 MHz,CDCl
3,TMS)谱中:δ 6.97(d,J=7.55 Hz,1H,H-5)、6.69(t,J = 7.45 Hz,3H,H-6)、7.06(t,J=7.35 Hz,1H,H-7)、6.55(d,J=7.90 Hz,3H,H-8)和7.13(d,J=7.15 Hz,1H,H-19)、7.54(t,J=6.85 Hz,1H,H-20)、7.06(t,J=7.35 Hz,1H,H-21)、7.48(t,J=7.25 Hz,1H,H-22)显示为两个苯环上质子信号,δ 5.85(s,1H,H-1)为酰胺键上的氢质子,δ 4.82(s,1H,H-2)、4.25(s,1H,H-15)、3.66(dd,J =3.75、11.55 Hz,1H,H-11)、3.02(s,3H,H-24)分别为3 个连氮次甲基和1 个连氮甲基上的质子信号。
13C NMR谱中:δ 150.1(C-9)为苯环上与氮相连的碳信号,δ 165.4(C-13)、163.9(C-16)为酰胺羰基碳信号。分析氢核化学位移相关谱(
1H-
1H correlation spectroscopy,
1H-
1H COSY)、异核多级量子相关谱(heteronuclear multiple quantum coherence,HMQC)及多量子跃迁谱(heteronuclear multiple bond correlation,HMBC)的耦合关系,使所有的碳氢耦合得到全面归属,
1H-
1H COSY使片段C5~C8和C19~C23顺次连接起来,HMBC谱显示一组关键的远程耦合关系,包括:H-2到C-3、C-4、C-9;H-12到C-2、C-3、C-4、C-11、C-13;H-17到C-15、C-16、C-18、C-19、C-23;H-24到C-13和C-15。C-3为一季碳,若要与该化合物的分子质量匹配,则该化合物须为C3—C3’相连接的对称结构,以上数据与文献[17]数据一致(表2),确定化合物1是二酮哌嗪生物碱,结构如图3所示。
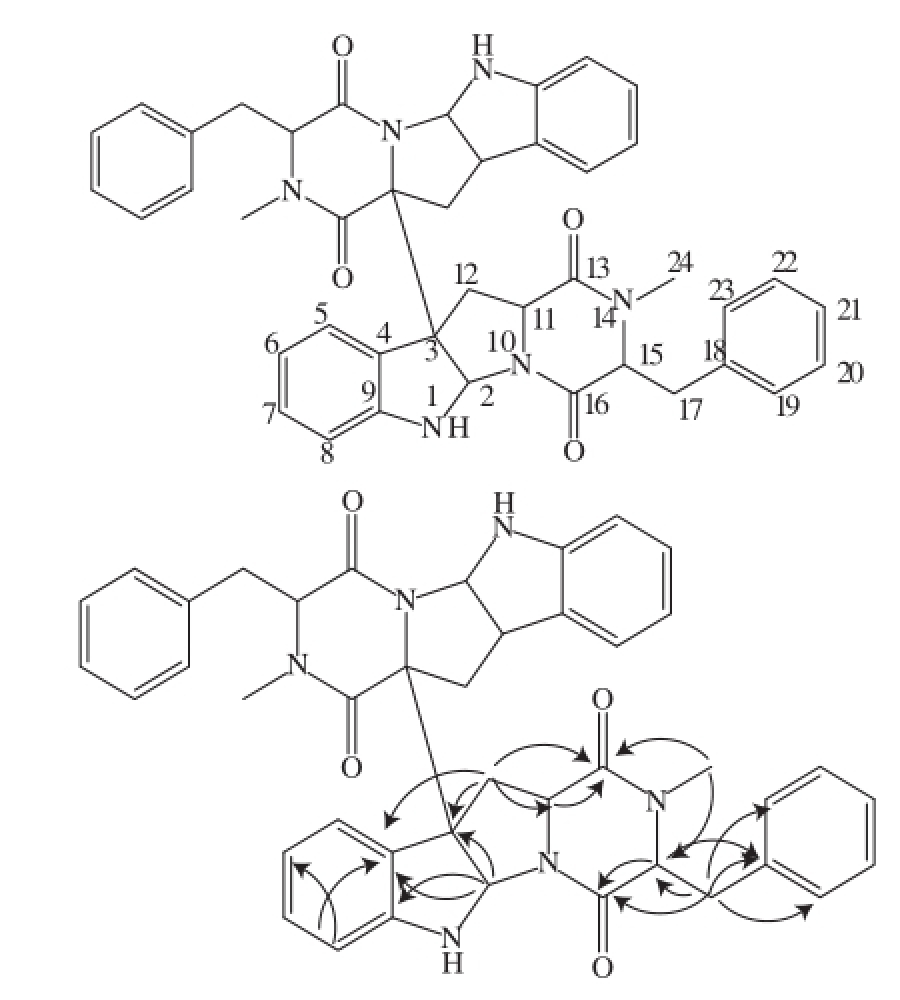
图3 化合物1的结构与关键的HMBC耦合关系
Fig.3 Structure of compound 1 and the key coupling relationship with HMBC
表2 化合物1的核磁数据
Table 2 NMR Spectral data of compound 1

位置H谱C谱HMBC谱(H→C)
1H-
1H COSY谱1-NH5.85(s,1H)2 4.82(s,1H)78.6C-3、C-4、C-9 3 58.9 4 126.4 56.97(d,J=7.55 Hz,1H)125.7C-3、C-7、C-9H-6 66.69(t,J=7.45 Hz,3H)118.9C-8、C-4 77.06(t,J=7.35 Hz,1H)129.6C-5、C-9H-8、H-6 86.55(d,J=7.90 Hz,3H)109.6C-6、C-4 9 150.1 10 113.66(dd,J=3.75、11.55 Hz,1H)58.5C-13H-12 12a2.02(dd,J=4.80、12.50 Hz,1H)C-11、C-3、C-2、C-4H-12b 12b1.57(t,J=12.05 Hz,1H)35.9C-11、C-3、C-2、C-4、C-13 13165.4 14 154.25(s,1H)63.1C-18、C-16 16163.9 17a3.25(dd,J=4.40、14.35 Hz,3H)36.2C-15、C-19、C-18、C-16、C-23 17b3.53(dd,J=2.30、14.30 Hz,3H)H-17a 18134.4 197.13(d,J=7.15 Hz,1H)129.3C-17、C-20、C-21 207.54(t,J=6.85 Hz,1H)125.4C-21、C-18H-19 217.06(t,J=7.35 Hz,1H)129.6C-20、C-19 227.48(t,J=7.25 Hz,1H)127.9C-19、C-21 237.13(d,J=7.15 Hz,1H)129.3 243.02(s,3H)32.6C-13、C-15
2.3.2 化合物2
白色无定形粉末,阴离子低分辨ESI-MS在m/z 752.6 [M-H]
-处给出准离子峰,表示该化合物相对分子质量为753,结合核磁谱图分析其分子式为C
43H
79NO
9。分析
1H-NMR和
13C-NMR谱提示该化合物为典型的脑苷脂类化合物。
1H-NMR谱中δ 7.36(d,J=9.25 Hz,1H)处的质子信号结合
13C-NMR谱中δ 171.9(C-1’)的羰基碳信号,提示分子中含有一个酰胺键,
1H-NMR中,δ 5.68(m,1H,H-4’)、5.57(m,1H,H-5)、5.42(m,1H,H-3’)、5.38(m,1H,H-4)、5.09(t,J=5.70 Hz,1H,H-8)显示为烯烃质子信号,δ 0.85(t,J= 6.65 Hz,3H,H-18,H-18’)、1.54(s,3H,H-19)提示为3 个甲基信号;
13C-NMR中,δ 134.8(C-9)、130.9(C-4’)、130.8(C-4)、128.9(C-3’)、123.4(C-8),123.4(C-9)为3 个双键碳信号,
1H-NMR谱和
13C-NMR谱显示出一组吡喃糖信号,包括:δ 4.12(H-1”)、2.96(H-2”)、3.13(H-3”)、3.05(H-4”)、3.08(H-5”)、3.67(H-6a”)、3.45(H-6b”);δ 103.4(C-1”)、73.3(C-2”)、76.5(C-3”)、70.0(C-4”)、76.8(C-5”)、61.0(C-6”)。根据以上波谱数据,并查阅文献[18],可确定该化合物2为脑苷脂C,结构如图4所示。

图4 化合物2的结构
Fig.4 Structure of compound 2
3 讨 论
微生物能否产生结构新颖、活性特别的次级代谢产物取决于其基因组中是否含有相关的合成基因。分子生物学的飞速发展逐渐阐释了相关酶系合成微生物次级代谢产物的生物合成途径
[19],奠定了从基因到预测化合物结构类型的理论基础
[20],成为了研究和开发现代天然产物的新型战略。基于Ⅰ型PKS功能基因分析方法可以筛选出可能产生聚酮化合物的菌株,大大有助于发现微生物中聚酮类化合物的概率。
本研究以Ⅰ型PKS功能基因为导向,从野生海芦笋中分离得到4 株含有Ⅰ型PKS基因的内生真菌,编号分别为Salicorn 1、Salicorn 2、Salicorn 3、Salicorn 4,进一步筛选出菌株Salicorn 2和Salicorn 3进行ITS1-5.8S-ITS4 rDNA同源序列鉴定,确定其种属地位分别为:Salicorn 2为黄曲霉、Salicorn 3为圆弧青霉。有研究显示,这些含有PKS功能基因的菌株均能产生结构新颖的活性次级代谢产物:Lin等
[21]从海绵体黄曲霉C-F-3菌株的发酵产物中分离得到1 个新天然产物异-α-环匹阿尼酸、2 个二酮哌嗪生物碱和3 个真菌毒素,其中新化合物对细胞HL-60、MOLT-4以及BEL-7402具有细胞毒活性;Holzapfel
[22]从圆弧青霉中首次分离出具有生物活性的霉菌毒素——α-环匹阿尼酸(α-cyclopiazonic acid,α-ACP),自此,人们也从其他青霉与曲霉属菌株中发现了α-ACP的衍生物。这些化合物均是含有相似化学骨架的聚酮类化合物,提示微生物产生的次级代谢产物大都是结构相似的系列衍生物,极有可能是同一菌株产生次级代谢产物的生物合成途径基本相同。由此,基于Ⅰ型PKS基因的定向筛选方法十分有望成为寻找大量产生天然活性产物——聚酮类化合物菌株的有效途径。
本研究初步从盐生海芦笋来源真菌Salicorn 3发酵提取物中分离得到2 个单体化合物,分别鉴定为二酮哌嗪生物碱和脑苷脂C。据报道
[17-18],这2 个化合物都有较强的生物活性和药理功效。化合物1是一种二酮哌嗪生物碱,属环肽类化合物,Wang等
[23]从分离自海绵体黄曲霉的发酵产物中提取到一个新的真菌毒素(黄曲霉毒素B
2b)和二酮哌嗪生物碱,其中二酮哌嗪生物碱对癌细胞有抑制作用。化合物2是脑苷脂类化合物,属聚酮类化合物,是一类普遍存在于动植物、菌类、海洋生物组织细胞膜中的内源性生物活性物质
[24]。Valeria等
[25]发现,脑苷脂类产品用于神经恢复的治疗,国外已应用于开发改善老年性脑功能衰退的药物。本研究中化合物脑苷脂C是从含有Ⅰ型PKS功能基因的菌株中分离得到的,这一结果为定向筛选盛产具有生物活性的聚酮类化合物菌株提供了一个新的思路。
参考文献:
[1] SAIKONEN K, WÄLL P R, HELANDER M, et al. Evolution of endophyte plant symbioses[J]. Trends in Plant Science, 2004, 9(6): 275-280. DOI:10.1016/j.tplants.2004.04.005.
[2] JACOB M, BHAT D J. Two new endophytic conidial fungi from India[J]. Ctyptogamie Mycologie, 2000, 21(2): 81-88. DOI:10.1016/ S0181-1584(00)00116-0.
[3] STROBEL G, STIERLE A, STIERLE D, et al. Taxomyces andreanae a propposed new Taxon for a Bulbilliferous hyphomycete associated with Pacific yew(Taxus brevifolia)[J]. Mycotaxon, 1993, 47: 71-80.
[4] SUN W, PENG C S, ZHAO Y Y, et al. Functional gene guided discovery of type II polyketides from culturable actinomycetes associated with soft coral Scleronephthya sp.[J]. PLoS ONE, 2012, 7(8): e42847. DOI:10.1371/journal.pone.0042847.
[5] ZHOU K, ZHANG X, ZHANG F L, et al. Phylogenetically diverse cultivable fungal community and polyketide synthase (PKS), nonribosomal peptide synthase (NRPS) genes associated with the South China Sea sponges[J]. Microbial Ecology, 2011, 62(3): 644-654. DOI:10. 1007/s00248-011-9859-y.
[6] NIKOLOULI K, MOSSIALOS D. Bioactive compounds synthesized by non-ribosomal peptide synthetases and type-I polyketide synthases discovered through genome-mining and metagenomics[J]. Biotechnology Letters, 2012, 34(8): 1393-1403. DOI:10.1007/s10529-012-0919-2.
[7] SCHNEEMANN I, NAGEL K, KAJAHN I, et al. Comprehensive investigation of marine Actinobacteria associated with the sponge Halichondria panicea[J]. Applied and Environmental Microbiology, 2010, 76(11): 3702-3714. DOI:10.1128/AEM.00780-10.
[8] WATANABE A, FUJII I, SANKAWA U, et al. Re-identification of Aspergillus nidulans wA gene to code for a polyketide synthase of naphthopyrone[J]. Tetrahedron Letters, 1999, 40(1): 91-94. DOI:10.1016/S0040-4039(98)80027-0.
[9] BINGLE L E H, SIMPSON T J, LAZARUS C M. Ketosynthase domain probes identify two subclasses of fungal polyketide synthase genes[J]. Fungal Genetics and Biology, 1999, 26(3): 209-223. DOI:10.1006/fgbi.1999.1115.
[10] 洪立洲, 丁海荣, 杨智青, 等. 盐生植物海蓬子的研究进展及前景展望[J]. 江西农业学报, 2008, 20(7): 46-48. DOI:10.1111/j.1440-1797.2009.01182.x.
[11] ATTIA F M, ALSOBAYEL A A, KRIADEES M S, et al. Nutrient composition and feeding value of Salicornia bigelovii torr meal in broiler diets[J]. Animal Feed Science and Technology, 1997, 65(1/4): 257-263. DOI:10.1016/S0377-8401(96)01074-7.
[12] GUO L D, HUANG G R, WANG Y, et al. Molecular identification of white morphotype strains of endophytic fungi from Pinus tabulaeformis[J]. Mycological Research, 2003. 107(6): 680-688. DOI:10.1017/S0953756203007834.
[13] AMNUAYKANJANASIN A, PUNYA J, PAUNGMOUNG P, et al. Diversity of type I polyketide synthase genes in the wood-decay fungus Xylaria sp. BCC 1067[J]. FEMS Microbiology Letters, 2005, 251(1): 125-136. DOI:10.1016/j.femsle.2005.07.038.
[14] NICHOLSON T P, RUDD B A, DAWSON M, et al. Design and utility of oligonucleotide ene probes for fungal polyketide synthases[J]. Chemistry & Biology, 2001, 8(2): 157-178. DOI:10.1016/S1074-5521(00)90064-4.
[15] 王惠, 湛东锐, 李连强, 等. 18S rDNA和ITS相结合鉴定一株盐生海芦笋内生真菌的研究[J]. 食品科学, 2012, 33(21): 173-176.
[16] TAMURA K, PETERSON D, PETERSON N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods[J]. Molecular Biology and Evolution, 2011. 28(10): 2731-2739. DOI:10.1093/ molbev/msr121.
[17] COLIN J. 1-(2-phenyl-ethylene)-Ditryptophenaline, a new dimeric diketopiperazine from Aspergillus flavus[J]. Journal of Natutal Products, 1994, 57(9): 1239-1244. DOI:10.1021/np50111a008.
[18] SHU R G, WANG F W, YANG Y M, et al. Antibacterial and xanthine oxidase inhibitory cerebrosides from Fusarium sp. IFB-121, an endophytic fungus in Quercus variabilis[J]. Lipids, 2004, 39(7): 667-673. DOI:10.1007/s11745-004-1280-9.
[19] 白林泉, 邓子新. 微生物次级代谢产物生物合成基因簇与药物创新[J]. 中国抗生素杂志, 2006, 31(2): 80-99. DOI:10.3969/ j.issn.1001-8689.2006.02.004.
[20] 朱孟沼, 崔晓龙, 李铭刚, 等. 发现微生物药物的新途径: 从基因到新化合物[J]. 天然产物研究与开发, 2006, 18(5): 854-857. DOI:10.3969/j.issn.1001-6880.2006.05.037.
[21] PEDRAS M S C, SMITH K C. Sinalexin, a phytoalexin from white mustard elicited by destruin B and Alternaria brassicae[J]. Phytochemistry, 1997, 46(5): 833-837. DOI:10.1016/S0031-9422(97)00362-2.
[22] HOLZAPFEL C W. The isolation and structure of cyclopiazonic acid, a toxic metabolite of Penicillium cyclopium Westling[J]. Tetrahedron, 1968, 24(5): 2101-2119. DOI:10.1016/0040-4020(68)88113-X.
[23] WANG H, LU Z Y, QU H J, et al. Antimicrobial aflatoxins from the marine-derived fungus Aspergillus flavus 0092008[J]. Archives of Pharmacal Research, 2012, 35(8): 1387-1392. DOI:10.1007/s12272-012-0808-1.
[24] 陈颖, 吕洁丽, 段金厫, 等. 从生物进化看脑苷脂类化合物的分布及其生物活性研究进展[J]. 国际药学研究杂志, 2009, 36(2): 121-126. DOI:10.3969/j.issn.1674-0440.2009.02.009.
[25] CAREAGA V P, MAIER M S. Chapter 3-cerebrosides from marine organisms[J]. Studies in Natural Products Chemistry, 2014, 42(3): 59-61.
Isolation and Identification of Endophyte and Its Secondary Metabolites from Salicornia bigelovii Torr. Based on Type I Polyketide Synthase (PKS I) Gene
ZHANG Junnan
1, PENG Jie
1, LIU Tianxing
1,2, XIN Zhihong
1,*
(1. College of Food Science and Technology, Nanjing Agricultural University, Nanjing 210095, China; 2. Library of Nanjing Agricultural University, Nanjing 210095, China)
Abstract:In this study, we aimed to predict the type of secondary metabolites of the endophyte Salicorn 3 isolated from wild Salicornia bigelovii Torr. by using the type I polyketide synthase (PKS I) gene as a probe. As a result, two polyketide compounds were isolated from the fermentation product of Salicorn 3. Based on the ITS1-5.8S-ITS4 rDNA sequence analysis, the strain Salicorn 3 was identified as Penicillium cyclopium. The isolate was found to have the potential to produce polyketide compounds by phylogenetic analysis and comparing the data with the reference. Two pure compounds were obtained from the fermentation product of Salicorn 3 using open silica gel column chromatography and Sephadex LH-20, and their chemical structures were elucidated as (1) ditryptophenaline and (2) cerebrosid e C by nuclear magnetic resonance (NMR) and electrospray ionization mass spectrometry (ESI-MS).
Key words:Salicornia bigelovii Torr.; endophytic fungi; type I PKS functional gene; polyketide compounds; nuclear magnetic resonance
DOI:10.7506/spkx1002-6630-201601021
中图分类号:TS201.2
文献标志码:A
文章编号:1002-6630(2016)01-0114-06
引文格式:
张俊楠, 彭洁, 刘天行, 等. 基于PKS Ⅰ基因海芦笋内生真菌及其次级代谢产物筛选鉴定[J]. 食品科学, 2016, 37(1): 114-119.
DOI:10.7506/spkx1002-6630-201601021. http://www.spkx.net.cn
ZHANG Junnan, PENG Jie, LIU Tianxing, et al. Isolation and identification of endophyte and its secondary metabolites from Salicornia bigelovii Torr. based on type Ⅰ polyketide synthase (PKS Ⅰ) gene[J]. Food Science, 2016, 37(1): 114-119. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201601021. http://www.spkx.net.cn
收稿日期:2015-03-11
基金项目:国家自然科学基金面上项目(31071586)
作者简介:张俊楠(1989—),女,硕士,研究方向为食品营养与化学。E-mail:2012108057@njau.edu.cn
*通信作者:辛志宏(1974—),男,教授,博士,研究方向为食品营养与化学。E-mail:xzhfood@njau.edu.cn





