肉制品真伪鉴别技术研究进展
任君安
1,2,黄文胜
1,葛毅强
2,3,陈 颖
1,*
(1.中国检验检疫科学研究院,北京 100123;2.中国农业大学食品科学与营养工程学院,北京 100083;3.中国农村技 术开发中心,北京 100045)
摘 要:肉类制假、掺假问题严重侵害了消费者权益、身体健康和进出口贸易。目前,肉类掺假检验技术已经逐渐成为研究热点,主要的研究方法有形态学、基于代谢物的检测和基于蛋白水平及核酸水平的检测等几大 类,主要包括酶联免疫吸附实验、色谱、质谱和聚合酶链式反应等。本文综述了食品中肉类成分鉴别技术的研究进展,并对其发展趋势进行讨论。
关键词:肉制品;真伪;鉴别;检测技术
肉及其制品营养丰富,蛋白质含量约为10%~20%,畜肉中含丰富的B族维生素,禽肉中不饱和脂肪酸含量较高,如人体必需的亚油酸,约占总脂肪含量的20%
[1]。全球范围内肉制品的产量和消费量持续升高,2013年我国成为全球肉类生产第一大国,年产量达8 536 万t,伴随而来是近年来肉制品的掺假掺杂事件频繁发生。2013年,我国部分地区开展了肉 及其制品的掺假情况调查
[
2],结果显示有25.6%的样品与标识不符,肉制品掺假情况较明显,熟制牛肉和食用羊肉卷是肉类掺假高危品。目前,肉制品的掺假主要表现在:1)原料肉的掺假;2)组织的替换,如用其 他动物组织(脂肪等)替换肌肉组织成分;3)非肉成分的添加,如注水以及掺入植物蛋白等
[3]。这些问题的出现严重干扰了全球范围内的肉业生产,不仅成为制约肉品质量提升的主要因素,而且严重损害了消费者的经济利益和知情权,甚至涉及宗教信仰等问题(例如在清真食品中掺杂进猪肉或其他血浆成分等非清真食品)。
为保护消费者权益,全球不同国家和地区对肉制品的标识要求都做了不同程度的规定。欧盟定量成分声明(quantitative ingredient declaration,QUID)强制要求肉制品需要标识出所有成分及其净含量,详细规定了净含量的计算方法,并规定肉的主要成分为骨骼肌蛋白、结缔组织蛋白和脂肪等成分需单独列出且规定了最高含量,其中猪肉的脂肪最高限量为30%,结缔组织蛋白限量为25%,鸟类和兔肉的脂肪最高限量为15%,结缔组织蛋白限量为10%,其他哺乳动物纯肉及混 合肉制品脂肪含量和结缔组织蛋白限量为25%,这是最严格的肉类成分标识制度
[4]。美国食品药品监督管理局(Food and Drug Administration,FDA)食品标签指南规定肉制品中需要标识出所有成分,并在食品外包装的主展示面上根据质量按照降序顺序罗列所有成分。我国于2011年发布预包装食品标签通则,也规定各种配料应按制造或加工食品时加入量的递减顺序一一排列。
综上所述,各国已有肉制品标识的法律法规来约束生产者和经营者的行为,但要保障相关法规的有效执行,还需 要借助科学的检测方法
[5]。各国学者近年来基于各类技术平台研究肉制品中动物种类的定性定量检测方法,大致可分为形态学方法、基于代谢物的检测方法以及基于蛋白、核酸的检测方法。本文就各种肉类掺假鉴别方法的研究进展进行分析和总结,并对该领域的未来发展方向进行探讨。
1 形态学检测方法
形态学方法是以组织学特性为基础,通过立式显微镜和复合光学显微镜观察样品粗糙颗粒和细小颗粒形态学构造来区分不同动物组织的一种检测方法,此种方法仍然是目前欧盟官方唯一认定的可用于肉骨粉仲裁检测的标准方法
[6],可以在几乎不存在假阴性的情况下检测出低于0.1%的肉骨粉含量
[7],检测假阳性率低,检出限低,分析热稳定性好。但是显微镜分析结果有一定的主观性,与检测人员的经验有较大关系,例如在有细 胞存在的 肉骨粉样品中尽管能够检出微量的动物源性成分,但无法区分动物种类,只可以区分哺乳动物、禽类和鱼类
[8],因此迫切需要寻找分辨率和灵敏度更高的鉴定方法。近几年,基于电镜与数字图像分析相结合的组织学技术已成功地应用在肉类加工产品中肌肉组织含量的评价和检测,前提是肉在微切片后的形态学改变并不明显
[9],该技术成功用于分析美国热狗中的骨骼肌含量及结缔组织和 骨组织等其他成分,结果表明大部分品牌的热狗产品中肉(骨骼肌)含量均低于10%,水分占50%以上,所有品牌的热狗中均含有其他组织成分。基于光镜的图像分析技术也被用来检 测4 个商业品牌的意大利饺子馅中存在的各种动物组织及其骨骼 肌含量
[10],并检测了不同品牌和批次样品的骨骼肌密度、骨骼肌蛋白大小和不同批次的均一性,结果表明该方法的检测结果可靠,可以对加工肉制品中的微量动物组织成分和含量进行鉴定。
2 基于代谢物的检测方法
广义上,代谢物包括了生物体内除核酸、蛋白之外的所有化合物,如糖类大分子、脂类大分子及各种小分子代谢物,但目前代谢物检测只涉及分子质量小于2 000 D的小分子代谢物。一些研究者报道了根据肉类中的氨基酸、糖和酚类物质等生物代谢物组成进行动物种属鉴别的方法
[11-13]。代谢物检测的常用技术有光谱、色 谱-质谱及各类传感器(如电子鼻和电子舌)等。光谱检测常用于研究物质的分子结构和组成,由于拉曼散射光谱和红外吸收光谱可以有效表征小分子代谢物中的C、N、O、H之间的化学键及相应官能团,因此自20世纪70年代起被广泛用于有机物和农产品品质成分的定性定量检测。电子鼻和电子舌是基于电化学传感器和模式识别的气体、液体分子分析仪器,能同时与样品中的各类分子相互作用,获取样品的整体信息,近年也已用于食品农产品的品质 检测和种类鉴别。
由于各种代谢物往往含有多种官能团且多有相似分子结构,因此各物质光谱、电子鼻舌等的测量信号交叉重叠,无法直接测得食品农产品中各物质的含量,因而这种定量也被称为“黑匣子” 定量。需要运用化学计量学将测量信号与典型样品的已知质量参数进行关联,通过大量典型样品的测量数据建立定量分析模型,才能用于样品定量检测,目前常用的建模方法有主成分分析、判别分析、偏最小二乘回归分析和人工神经网络等。
2.1 光谱学技术
光谱学方法是一种快速、无损的食品饲料动物成分检测鉴别方法,其突出特点是分析速率快,样品制备简单,可同时测定多种成分,简单无污染,只需对比已知谱图,即可进行快速、简单、可重复、无损伤的定性定量分析。拉曼光谱和近红外光谱检测的都是物质分子的各种振动、转动频率和能级的信息,反映了样品分子中有机物结构和官 能团类型,且拉曼光谱与红外光谱在应用对象上各具特色。近几年,国内外很多实验室开展了拉曼光谱法和近红外光谱法鉴别不同动物源性成分的方法研究。
拉曼光谱是由物质分子对光源的散射产生的一种非破坏性的指纹成像技术。Ellis等
[11]首次运用傅里叶变换拉曼光谱技术,鉴别了鸡肉和火鸡鲜肉,同时对不同部位的鸡肉,如鸡胸肉和鸡腿肉进行了有效地区分。Sowoidnich等
[12]应用转移激发拉曼光谱(shifted-excitationraman difference spectroscopy,SERDS)技术原位鉴定牛肉、猪肉、鸡肉和火鸡的鲜肉切片,对两个激发光波长的光谱数据进行主成分分析,结果表明4 种肉类可被明确区分。但比之于红外光谱,拉曼光谱的灵敏度较低,待测物需要较高的含量才可检测。
常用波长在780~2 526 nm范围的近红外光谱法。Ding等
[14]用近红外技术研究牛肉汉堡包掺假问题,采用400~2 500 nm波长范围的红外光对生、熟及碎肉进行分析,并通过建立偏最小二乘回归(partial least-squares regression,PLS)模型判断掺假程度,发现掺假量与准确度成正比例关系,其中,掺入量为5%~25%的牛肉样品,判断准确率在92.7%以上。Restaino等
[15]将近红外光谱及化学计量学相结合,应用线性判别分析(spatially smooth linear discriminant analysis,SLDA)鉴别意大利肉块的动物种类,对牛肉、猪肉肉块的鉴别准确率为100%。Morsy等
[16]利用近红外光谱对新鲜的解冻的牛肉尝试进行了定量检测。最近Kamruzzaman等
[17]应用近红外高频谱图像分析法检测碎羊肉中掺杂的猪肉含量,该技术的最大特点为光谱技术与成像技术有机结合,通过光谱鉴定分析物的动物种类,通过图像分析计算样品切片中各种肉类的空间分布。笔者选择4 个波长进行分析并建立了多重线性回归(multi-linear regression,MLR)模型,在4%~40%的范围内可检测掺杂肉的含量。孙淑敏等
[18]用近红外光谱快速鉴别羊肉产地来源。对来自国内5 个产区共99 份羊肉样品的光谱数据进行分析,建立了羊肉产地来源的定性判别模型。结果表明,不同地区羊肉的近红外光谱有显著差异,整体正确判别率达到100%。证明近红外光谱结合化学计量学方法可以作为羊肉产地溯源的一种有效技术手段。近红外 光谱法的缺点是:1)需要大量有代表性且测量值已知的样品建立模型。2)模型不通用,每台仪器的模型都不相同,限制它的使用范围。
2.2 电子鼻和电子舌技术
电子鼻采用氧化物半导体和固体电解质等气敏传感器对气味进行捕捉和检测,模拟人鼻的感觉功能,属于气味指纹检测方法。电子舌以电化学传感器模拟人的味蕾功能,用于检测液体中 风味相关物质,属于嗅觉指纹检测方法。电子鼻和电子舌已在食品品质控制、新鲜度评价及原料种类鉴别等方面得到了广泛应用
[19],在检测肉品时具有样品预处理简单、操作方便、成本低、重现性好及可同时检测多种成分等优点。
贾洪锋等
[20]尝试用电子鼻进行肉类掺假识别,对牦牛肉、牛肉和猪肉样品进行分析。结果表明,电子鼻能够有效识别样品中的猪、牛肉,并能对牦牛肉和普通牛肉的不同部位进行有效区分,但不能区分猪肉的不同部位。在牛肉馅中掺入不同比例的猪肉馅时,电子鼻响应信号和猪肉馅掺入比例之间有很好的相关性,PLS模型预测误差在1.27%~7.00%之间。García等
[21]采用多传感器阵列电子鼻系统,在传感器阵列中溅射入SnO
2薄膜,结合主成分分析(principal component analysis,PCA)和概率神经网络(probabilistic neural network,PNN)数据分析技术可以鉴别4 种品牌的火腿肉,准确率为100%。Tian等
[22]利用电子鼻鉴别羊肉中掺杂的猪肉成分,结果表明,采用线性判别分析可以区分掺入不同比例猪肉的羊肉样品,并采用PLS、MLR和反向传播神经网络(back propagation neural network,BPNN)分析建立预测模型,能有效区分掺入羊肉中的猪肉比例。
田晓静等
[23]报道了利用电子舌方法对羊肉中混入不同比例鸡肉的样品进行定性和定量检测。通过主成分分析和典型判别分析,电子舌能区分混入不同比例鸡肉的羊肉糜样品;采用多元线性回归分析和偏最小二乘回归分析建立的定量预测模型能有效预测混入的鸡肉比例。韩剑众等
[24]将自行开发的多频脉冲电子舌应用于鲈鱼、鳙鱼、鲫鱼等3 种淡水鱼和马鲇鱼、小黄鱼、鲳鱼等3 种海水鱼的鉴别实验中,表明电子舌不仅可以有效区分淡水鱼和海水鱼,而且还可以辨识不同品种淡水鱼或海水鱼之间的差异。
3 基于蛋白质的检测方法
蛋白质是生命的物质基础,氨基酸序列和三维结构通常具有一定种内保守性和种间特异性,是适用于物种鉴别技术的理想分析对象。Wilkins等
[25]首次提出了蛋白质组的概念,随后,研究人员逐渐开始采用电泳方法、基于抗原抗体特异性反应的免疫学方法或质谱分析等方法等对肉及其制品中 的总蛋白或蛋白标志物进行分析检测
[26]。每种方法都有其优缺点,需要对不同样品找到最适合的检测方法。
3.1 蛋白质电泳技术
电泳方法是一种传统的动物源性成分检测方法,至今仍得到广泛的应用。动物的品种、组织结构、保存时间和加工技术都会对蛋白质产生影响。利用蛋白质组学对肉类进行鉴别的电泳技术主要包括聚丙烯酰胺凝胶电泳(polyacrylamide gel electrophoresis,PAGE)、十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-PAGE,SDS-PAGE)、双向凝胶电泳(two-dimensional gel electrophoresis technology,2-DE)等。
2-DE根据蛋白质的等电点(pI)和分子质量(M
w)对蛋白质进行分离纯化,能在一张凝胶上同时鉴定10 000 个蛋白点
[27],并且可以实现与质谱技术的结合,对不同品种的肉制品做定性或定量分析,成为蛋白质组学研究的主要支撑技术之一。19世纪80年代双向凝胶电泳技术第一次应用到了动物源性食品鉴别领域,初步鉴定了屠宰后的生肉样品。随后,2-DE技术在过去20 a中得到了飞速的发展,部分物种的肌 肉2-DE图谱已经公布,包括猪
[28-29]、牛
[30]和鸡
[31]的骨骼肌蛋白图谱,一些图谱已被用于获得与肉类种属性状相关的分子标记。研究人员分析了猪的不同品种
[32-33]、不同性别
[34]和不同肌肉部位
[35]的蛋白表达量差异,但是表达量的差异并不能作为不同动物源性食品鉴别的依据
[36]。随后,Montowska等
[37]鉴定出牛、猪和鸡等生鲜及加工样品可以差异表达的蛋白质,可用作动物源性成分鉴别的蛋白靶标,包括调节蛋白、代谢酶和肌纤维血浆蛋白。双向凝胶电泳技术本身存在局限性,操作自动化程度低,在分离过程中分子质量高于2 000 kD和低于1 000 kD的蛋白质易丢失、对于极酸、极碱、难溶、低丰度的蛋白质分离效果差等
[38],因此也对检测结果的准确性产生一定的影响。
3.2 免疫学技术
免疫学技术是通过抗原和抗体的特异性结合反应和信号放大技术达到鉴别动 物源性成分的方法。自从Warneck和Saffle用肌肉水浸液做兔体免疫抗原,解决了血清学实验中最重要的抗体制备问题之后,肉类食品品种掺假的免疫学检测方法不断涌现,例如试管沉淀法、琼脂扩散法、对流免疫电泳法、放射免疫法以及酶联免疫吸附实验(enzyme-linked immunosorbent assay,ELISA)等,其中以ELISA方法的应用最为广泛
[39]。
酶联免疫是将抗原抗体反应的高度特异性和酶的高效催化作用相结合发展建立的一种免疫分析方法
[40]。利用抗原-抗体反应的显著特异性,进行成分的鉴别和血清学分型。目前,用于肉类等食品鉴别的ELISA方法主要包括间接酶联免疫和夹心酶联免疫分析,一些公司(Neogen、ELISA Technologies、SDI和Stamford)已经开发出一系列肉类检测的ELISA试剂盒,可以选择性的进行生熟肉样品的检测。20世纪90年代,Martin等
[41]就采用ELISA方法对牛肉中掺杂0%到80%的猪肉成分绘制标准曲线,定量检测猪肉成分含量,最低检测限为1%,变异系数18%,检测的牛肉样品中,最高含有的猪肉成分为48%。之后Renčová等
[42]通过将兔肌肉蛋白100 ℃或120 ℃加热30 min后制备热稳定性多克隆抗体,成功检测了62 种市售热加工产品中的家禽肉、马肉、袋鼠肉和鼠肉, 检测灵敏度为1%~5%。随后,Liu等
[43]利用抗体夹心法,以猪tnI 蛋白为抗原制备了两种单克隆抗体(MABs 8F10和5H9),最低能够检测鸡肉和牛肉中0.05%和0.1%的猪肉成分,并可以检出经过高温处理后大豆蛋白基料 饲料中和肉骨粉中的肉成分。Giovannacci等
[44]采用ELISA试剂盒成功鉴别了牛肉、猪肉和羊肉,但是无法区分禽肉样品的鸡肉和火鸡肉。
ELISA法的优点是操作简单方便,非专业人士也可以熟练掌握,使用范围广泛;成本低,灵敏度和通量较高,可以同时检测大量样品。难点是必须找到一个热稳定的抗原或者使特异性蛋白复性,研制出针对此抗原的特异性单克隆抗体。骨骼肌中的肌钙蛋白Ⅰ具有高特异性和热稳定性,是理想的抗原之一。如果抗体特异性低,则在鉴别同一物种的不同品种动物源性食品时,容易出现交叉反应,产生假阳性结果。另外,该方法对于加工后样品成分的鉴定的准确度会降低,这是由于环境因素的改变都会对蛋白抗原决定簇的立体结构产生影响,因此常常需要其他方法辅助检测。
3.3 色谱和质谱技术
液相色谱(气相色谱)可根据有机物的分子质量、疏水性和电负性,将其在色谱柱中分开,而后串联的质谱仪可根据荷质比确定测待分子的种类,因而液相色谱-质谱联用和气相色谱-质谱联用也广泛用于生物有机物和多肽的定性定量检测。
Chou等
[45]利用高效液相色谱法鉴别牛肉、猪肉等15 种生熟肉品,变异系数小于6%。Aristoy等
[13]以高效液相色谱法对肉制品来源的反刍动物饲料进行特异性检测,通过确定肌肽和鹅肌肽的含量鉴定加入饲料中的哺乳动物源性成分,当肌肽和鹅肌肽含量之比大于0.3时,认为饲料中含有哺乳动物成分,最低检测限为0.5%,证明高效液相色谱(high performance liquid chromatography,HPLC)可作为哺乳动物源性成分检测的筛选手段,但还需用其他方法进一步确定哺乳动物种类。Giaretta等
[46]利用超高效液相色谱技术(ultra performance liquid chromatogra phy,UPLC),以肌红蛋白作为靶蛋白检测生牛肉汉堡中的猪源性成分含量。生肉样品需要通过亚硝酸钠预处理,将氧合肌红蛋白和脱氧肌红蛋白转化为较稳定的高铁肌红蛋白,然后再上柱检测,最低检测限可达到5%(25 mg/500 mg)。然而,由于肉制品中蛋白质含量变化范围广、干扰因素多,导致此类技术灵敏度较低、易产生假阳性结果;对仪器、试剂和样品处理均有较高要求,在分析混合成分时难度较大,因此在实际应用中较少,标准化与推广应用的前景均受到很大局限
[47],因此,一般不作为标准检测方法或仲裁鉴定方法,而作为快速筛查方法使用。
目前,将鸟枪法和质谱法相结合对肉制品中不同动物源性成分进行鉴别成为了新的发展方向。这种方法操作简单,不需要在质谱检测前通过双向凝胶电泳法分离动物源性蛋白,只需将总蛋白酶解成肽段,通过质谱分析鉴别特征肽,并以质谱多反应监测方法检测特征肽含量,可实现不 同动物源性成分的定量检测。Christoph等
[48]首次采用此方法,鉴别了牛肉中的马肉和猪肉成分。他们以鸡肉、羊肉和牛肉为阴性对照,筛选出马和猪的特征肽,并确定牛肉中马肉和猪肉的最低定量检测限为0.55%。此方法利用三级质谱(multiple reaction monitoring,MRM3)降低基质效应,提高信噪比,有效提高定量灵敏度,可以定量检测牛肉中掺有0.13%的猪肉成分。随后,Montowska等
[49]采用电喷雾解吸电离质谱技术(desorption electrospray ionization,DESI-MS)与液滴萃取表面分析质谱(liquid extraction surface analysis-mass spectrometry,LESA-MS)技术首次检测牛、猪、马、鸡和火鸡肉等5 种肉类的骨骼肌蛋白,根据信噪比和多元分析法(multivariate analysis,MVA)成功检测鉴别5 种肉类成分的特征肽,并发现LESA-M S方法的稳定性和灵敏度优于DESI-MS方法。
4 基于核酸的检测方法
近几十年来,以核酸为标志物的动物种类鉴别检测方法飞速进步,并获得了广泛应用。常用技术有核酸杂交、聚合酶链式反应(polymerase chain reaction,PCR)及其衍生方法、DNA条码及新兴的数字PCR技术等。与蛋白质相比,DNA不易受到食品加工方式的影响,稳定性更高;存在于大部分的组织细胞中,即使动物组织部位不同,也可得到可靠的鉴定信息。同时,这些分析方法与红外光谱法
[50]、ELISA
[51]、毛细管电泳等方法相比,更加灵敏,且可以检测经过热处理的肉制品
[52],因此成为动物源性成分鉴定的主流技术。
4.1 核酸杂交技术
早期研究中多用DNA杂交完成动物源性成分鉴定,该方法将用放射性同位素标记的动物全基因组DNA物种特异性探针与固定到尼龙膜上的样品DNA进行杂交,根据产生的放射信号鉴定样品的物种组成
[53]。利用这种方法对加工过的鸡肉、猪肉样品
[54]以及牛肉和猪肉的混合制品
[55]进行同源性鉴定,得到了良好的效果。Kirsten等
[56]利用此方法鉴定了牛肉中掺杂的猪肉,将提取出的猪DNA固定在尼龙膜上,使其与
32P标记的猪DNA探针杂交,建立信号强度与DNA数量的函数关系,可在牛肉样品中检测出0.1%含量的猪肉成分,在加工后的肉制品中最低可检出0.5%的猪肉成分。这种方法耗时长、有放射性、对DNA的纯度要求高,探针信号的强度常受到样品组织来源或其加工方式等因素的影响,且灵敏度不高,定量困难,因此在实际应用中受到了限制。
4.2 常规PCR技术
PCR的突出特点是其具有极高的灵敏度,这在检测食品中的猪成分和饲料中的牛羊源性成分时极为重要,因食品中只要污染了微量的猪成分就不能作为清真食品,目前其他方法不具备如此高的灵敏度。PCR方法的另一特点是它的极高的分辨率,由于采用种属特异性的DNA序列作为靶标,PCR方法不仅可分辨动物的种类甚至可用于动物的品种鉴别和个体身份识别,这在肉类的溯源体系中已获得广泛应用。PCR检测方法的适用范围广,因为基因组DNA序列决定了动物的物种、品种和个体差异,且每个动物个体的DNA序列都具有唯一性,且终生不变,因此动物不同组织或器官的DNA 序列完全相同,无论肉块、骨头、皮、角等,都能从中提取DNA进行检测。由于DNA不具有挥发性且比大多数蛋白的热稳定性高,因此,PCR方法能用于各种加工肉品和产品的检测。肉种类鉴定中常选用线粒体基因作为PCR检测靶标,包括线粒体细胞色素b基因、D-Loop区、12S rRNA、16S rRNA等。这是由于线粒体DNA进化速度快,具有广泛的种内、种间多态性和高度的物种特异性,且拷贝数多,加热时不容易被彻底破坏,存活机率大,更容易被扩增,不会发生基因重组
[47,57]。陈颖等
[58-59]根据牛、羊和马属动物(马和驴)mtDNA序列,设计了牛、羊和马属动物源性成分的特异性扩增引物,建立了牛、羊源性成分和马属动物(马和驴)成分的快速检测方法,并首次通过选用不同的内切酶区分食品和饲料中的马和驴成分,在食品真伪鉴别中具有很好的实用价值和现实意义。Martin等
[60]利用线粒体12S rRNA序列设计特异性引物对鸭肉混合物进行检测,扩增的靶基因片段长度小于100 bp,检测限为0.1%~1.0%,因此适用于经高压或热加工后DNA出现高度降解的肉制品定性分析。Soares等
[61]选取线粒体cyt b和12S rRNA作为目的基因,对猪肉和家禽肉进行双重PCR鉴定,灵敏度为0.1%,具有良好的重复性。Safdar
[62]和Amaral
[63]等分别运用PCR方法对马肉、大豆蛋白、家禽、香肠同时鉴定和香肠中野兔、家兔、马鹿、猪和母牛成分鉴别进行了研究。近几年的文献报道中多选用线粒体基因组编码序列作为肉类定性鉴别的目的基因,对于定量检测来说,线粒体基因组在不同组织中数量变异较大,在动物组织种类未知的情况下以之作为定量检测的目标基因,可能导致定量结果的偏差。核基因DNA在各个组织中拷贝数恒定,在定量方面更加准确,但由于拷贝数低,导致灵敏度降低,因此对实验设计要求较高
[57]。Aslan等
[64]对75~100 ℃加热条件下牛肉中的GAPDH、CAST和CTSB基因降解程度做了初步研究,发现在100 ℃的高温加热后仍然可以检测到大于150 bp的片段,为以核基因为靶基因的定性定量检测提供依据。
PCR方法也用于动物的品种鉴别和个体身份识别,欧盟的管理法规强制要求自2005年1月1日起销售的肉品都必须具备可追溯性。加拿大、澳大利亚等国家也都相继建立肉类溯源管理系统。当肉制品发生污染时,对其DNA 标记进行检测可以快速地追溯到它的产地和亲本来源,完成肉品的从生产到餐桌的全程溯源
[65]。Vázquez等
[66]建立3 个微卫星多态性标记,用DNA指纹图谱技术对鲜牛肉片进行溯源。Heaton等
[67]用SNP标记对牛肉进行溯源,错误识别率仅为百万分之一。
4.3 PCR-限制性片段长度多态性(restriction fragment length polymorphism,RFLP)和PCR-单链构象多态性(single strand conformation polymorphism,SSCP)技术
限制性片段长度多态性分析是指利用限制性内切酶识别双链DNA分子的特定序列并在此将DNA切开,形成长度不等的限制片段作为指纹图的分析技术。PCR-RFLP是结合了RFLP与PCR技术各自的特点,通过第一步对基因组DNA进行PCR特定引物扩增,然后对扩增产物进行限制性内切酶酶切,用凝胶电泳进行多态性分析
[68]。Chikuni等
[69]根据微卫星DNA序列设计绵羊和山羊的特异性引物,并对PCR产物进行测序,结果表现出92%的同源性,然后通过4 个限制性酶切位点区分绵羊和山羊。Sharma等
[70]运用RFLP技术鉴别了生猪肉、高温蒸煮处理后和高温高压处理后的猪肉样品,检测结果均与生肉样品一致,并没有受到加热处理的影响。Doosti等
[71]利用RFLP技术研究建立牛肉、羊肉、猪肉、鸡肉、驴肉和马肉的鉴别方法并进行了实际样品检测验证。与核酸序列分析相比,这项技术的优点在于省去了直接测序的环节,价格低廉。缺点是操作中DNA用量大,纯度要求高,DNA制品中的污染(如蛋白质、酚、氯仿、乙醇、乙二胺四乙酸(ethylene diamine tetraacetic acid,EDTA)、SDS、高浓度盐)均能抑制酶切活性,但可以通过增加酶浓度、增大反应体积稀释可能的抑制剂和延长反应时间加以克服;此外,由于操作环节多、周期长、容易受到目标基因序列中酶切位点随机突变的影响,易产生检测结果的不确定性,如有些酶由于受旁侧核苷酸甲基化的影响,导致在特异位点的切割效率降低,这些都限制了RFLP技术大范围地应用。
常规PCR方法通常要求具有特异性的引物,单链构象多态性分析则可使用通用引物。多数方法选择线粒体细胞色素b基因(cyt b)进行扩增,将扩增后的目的片段经高温处理后解链并自我复性,在合适的条件下进行聚丙烯酰胺凝胶电泳,由于复性后单链DNA长度、空间构象差异,最后形成不同的谱图,通过此方法,可以区分序列中小至单个碱基的差异。陈颖团队的吴亚君等
[72]采用PCR-CE-SSCP技术建立了梅花鹿产品的快速筛查方法,在cyt b基因上设计出可以扩增出梅花鹿、白唇鹿等7 个鹿种样品的鹿科通用引物,实现了大批量梅花鹿保健品中掺假鹿种成分的快速检测。但影响SSCP稳定性的因素较多,实验过程中须严格控制实验条件,鉴定时要求同时使用标准品以保证实验的准确性,实际应用较少
[73]。
4.4 实时荧光定量PCR技术(real-time PCR)
传统PCR技术对扩增产物进行终点检测,而实时荧光PCR是通过荧光化合物在整个反应过程中检测和收集扩增信号。由于指数增长期是分析数据的最佳时期,样品中DNA含量与PCR产物在任意的循环数内存在定量关系,因此real-time PCR可通过标准曲线对样品模板浓度进行定量分析
[74-75]。
实时荧光定量PCR主要包括两大类:利用双荧光标记探针的荧光基团解离产生荧光强度变化的TaqMan realtime PCR和利用染料分子插入双链DNA产生荧光变化的SYBR Green real-time PCR。SYBR Green法的优势在于DNA扩增后通过溶解曲线检测出单碱基突变
[76]。López-Andreo等
[77]用之鉴别牛肉、猪肉、马肉和袋鼠,最低检测限达到了0.04 pg DNA,对实验室人工掺杂的肉制品均有良好的检测效果,随后进行的二重PCR的检测灵敏度略有降低。郭云霞等
[78]利用SYBR Green实时荧光PCR方法建立了食品中的鲨鱼源性成分鉴定方法,对9 种鲨鱼样品和48 种非鲨鱼类动植物样品进行检测,检测限为0.000 1 ng/μL DNA,并用此方法对20 种常见鲨鱼产品进行了检测,检测特异性强,灵敏度高。Soares等
[79]利用SYBR Green实时荧光PCR法检测出了禽肉制品 中掺入的猪肉成分。杨丽霞等
[80]利用SYBR Green 实时荧光PCR法检测出了肉类产品中的鸭肉成分。
TaqMan real-time PCR技术通过TaqMan探针与模板的结合与水解极大地增强了反应特异性,且实时检测荧光信号,省去了电泳的环节,使得同一反应中多种肉类同时鉴别的可靠性大大提升。这种方法在新鲜样品和加工品如灭菌罐头中均可以获得较好的效果,但一些深加工肉制品如明胶、肉汤类产品由于DNA被高度破坏造成检测难度较大。现阶段,国内外运用实时荧光PCR技术鉴别肉类的常见应用如表1所示。陈颖团队的Wang等
[81]根据牦牛的细胞色素b基因设计特异性引物探针,对8 个不同地方品种的牦牛和9 种其他动物DNA扩增,只有牦牛可以产生特异性的扩增曲线且与其他动物DNA无交叉反应,在混合肉品中检测灵敏度达到了0.001%,并在对市售牦牛制品检测时,发现部分商品以黄牛肉冒充牦牛肉。Ulca等
[82]针对土耳其的牛肉、鸡肉和火鸡肉等42 种肉制品中掺假情况进行了检测,其中40 种产品没有掺假情况,2 种产品不同程度的掺杂其他肉制品,检测限低于0.1%。张弛等
[83]针对市售羊肉中常常掺杂鸭肉现象,根据鸭cyt b基因中的保守序列,建立了肉制品中鸭源性成分定性和定量检测技术,检测灵敏度达到1.78 pg DNA。López-Andreo等
[84]采用MGB探针对65~126 ℃条件下加热10~30 min后的猪肉和牛肉混合样品中的猪肉成分进行定量检测,结果显示,处理后与处理前的猪肉样品Ct值相差2,处理后的DNA降解至大约100 bp左右,证明加热对检测结果产生了一定程度的影响,所建立的方法可以最低定量检测到5%的加热处理后的猪肉成分。Soares等
[85]利用TaqMan实时荧光PCR法检测出了肉制品中掺入的大豆成分。Kesmen等
[86]通过基于TaqMan探针的双重实时荧光PCR法定量检测了生的和热加工过的驴肉和猪肉。陈颖团队的吴亚君等
[87]利用实时荧光PCR方法建立了加工工艺复杂的阿胶产品中的马驴成分检测方法,为市售商品的监管及原料品质控制提供了快速有效的技术手段。
我国也于近几年研究建立了利用PCR方法和实时荧光PCR法鉴别畜、禽、鱼类的一系列国家和行业标准,包括进出口食品和饲料中常见禽类
[88](火鸡、鸡、鹅、鹌鹑、鸭、鸽子、鹧鸪)的品种鉴定标准,马、驴、鹿、骆驼
[89]和牛
[90]等畜类产品的鉴定标准和三文鱼、鳕、河豚、黄鱼、金枪鱼等
[91]鱼类产品鉴定标准等,为鉴别和检测饲料中的不同动物源性成分提供一种实用、有效的分子生物学方法。
尽管近年来实时荧光PCR技术给生熟肉制品及肉类加工品掺假的定性定量检测带来了长足的飞跃,但是相关方法的完善和推广仍然面临着诸多挑战。由于不同动物样品性别、种别、年龄、组织器官和肌肉类型的DNA总量,特别是线粒体DNA总量存在很大差异,使得较难确定标准样品,并且扩增的靶序列不同,PCR反应效率之间的差异也会对检测结果产生影响,阻碍方法的标准化。
表1 以实时荧光PCR技术为基础的常见肉类鉴别方法国内外研究现状
Table 1 An overview of species identification of common meat by realtime PCR
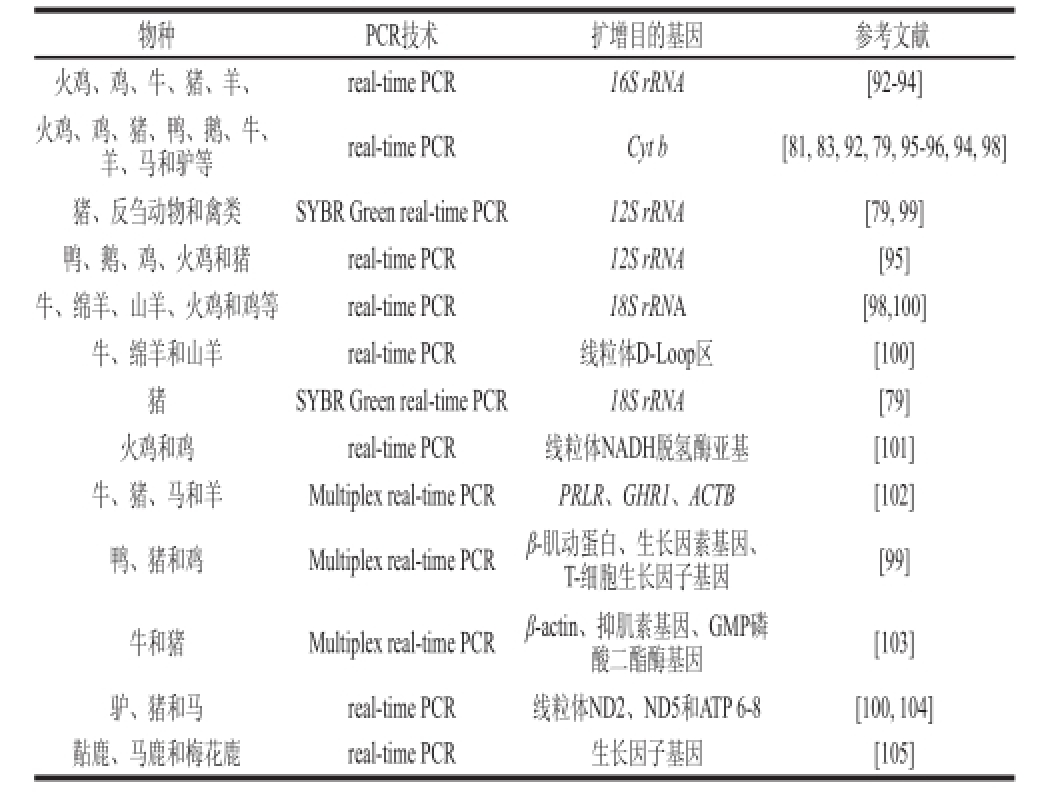
物种PCR技术扩增目的基因参考 文献火鸡、鸡、牛、猪、羊、real-time PCR16S rRNA [92-94]火鸡、鸡、猪、鸭、鹅、牛、羊、马和驴等real-time PCRCyt b [81, 83, 92, 79, 95-96, 94, 98]猪、反刍动物和禽类SYBR Green real-time PCR12S rRNA [79, 99]鸭、鹅、鸡、火鸡和猪real-time PCR12S rRNA [95]牛、绵羊、山羊、火鸡和鸡等real-time PCR18S rRNA [98,100]牛、绵羊和山羊real-time PCR线粒体D-Loop区 [100]猪SYBR Green real-time PCR18S rRNA [79]火鸡和鸡real-time PCR线粒体NADH脱氢酶亚基 [101]牛、猪、马和羊Multiplex real-time PCRPRLR、GHR1、ACTB [102]鸭、猪和鸡Multiplex real-time PCRβ-肌动蛋白、生长因素基因、T-细胞生长因子基因 [99]牛和猪Multiplex real-time PCRβ-actin、抑肌素基因、GMP磷酸二酯酶基因 [103]驴、猪和马real-time PCR线粒体ND2、ND5和ATP 6-8 [100, 104]黇鹿、 马鹿和梅花鹿real-time PCR生长因子基因 [105]
4.5 DNA条码技术
在2003年发展起来的DNA条码技术是一种快速、可靠、低成本的鉴定技术
[106]。该方法利用生物体内一段几百个碱基的DNA序列作为条形码进行物种鉴定。目前,常用线粒体细胞色素c氧化酶亚基Ⅰ(COX I)基因的部分序列作为动物的条形码。Eaton等
[107]利用基因条码技术对来自非洲中部及南美的哺乳动物和爬行动物进行分子鉴定,对23 个野生动物品种设计了一段扩增长度为645 bp的通用引物,204 份样品中有179 份(87.7%)获得了高质量的条码序列,COX I基因片段的种内多态性普遍较低,平均值为0.24%,而在同属物种之间的差异达到了9.77%,证明基因条码技术是一种可以用来监管濒危物种的偷猎和非法交易的有效手段,尤其是针对不易用形态学区分的半加工产品和野生动物产品。
4.6 数字PCR技术
PCR方法具有准确、灵敏、快速、简便的优点,实时荧光PCR更将原有的常规PCR技术发展成为一种高特异性、高灵敏的半定量基因分析技术。尽管实时荧光PCR方法在肉及其制品的定性和半定量检测中发挥了巨大作用,但是在PCR过程中影响扩增效率的因素很多,反应过程中扩增效率的差异,实际样品和标准样品以及不同样品之间的扩增效率的变化,均会导致循环阈值发生改变,进而使定量检测结果不准确,因此实时荧光PCR的定量只是半定量,准确度和重现性依然不能满足行政执法和贸易商品检测所用方法的要求。
数字PCR是一项可以精确定量DNA拷贝数的核酸检测新技术。它相较实时荧光PCR来说,不需要使用标准物质生成标准曲线,可以实现DNA拷贝数的绝对定量。数字PCR实验过程一般包括两步,即PCR扩增和荧光信号分析。在PCR扩增阶段,要将样品DNA平均分配到几千至几万个单元中,稀释到单分子水平进行反应,并在扩增期间或扩增结束后采集每个反应单元的荧光信号,最后通过直接技术或泊松分布计算样品DNA的原始浓度和含量
[108]。这种方法不同于real-time PCR对每个循环的实时荧光进行测定的方法,可消除real-time PCR中本底信号的影响,提高低丰度靶标的扩增灵敏度,简单计算出DNA模板拷贝数,而不需要采用标准物质作为外标。数字PCR在临床诊断医学、单细胞表达、转基因定量、环境微生物检测和下一代测序等方面发挥了重要作用
[109],但尚未见应用数字PCR进行肉类定量检测的研究报道,但是由于数字PCR技术具有拷贝绝对定量的优点,相信在肉制品的掺假检测中有良好的应用前景。
5 结 语
动物源性食品的掺假、掺杂是食品产业中的重要问题。以上方法,是近几年应用于肉制品种属鉴别的主要方法。形态学方法发展较早,已经从最初的更多依赖实验操作人员的光学显微镜技术发展为将电镜和数据图像分析相结合的新型鉴别方法;光谱学技术样品制备简单,分析速度快,但常需要大量已知样品建立模型,且模型通用性不强;基于蛋白水平的免疫技术操作简单,通量高,成本低,但是准确度不高,常用作快速筛查手段;核酸检测方法特异性、灵敏度高,结果较为准确,成为了国际上肉类掺假检验的主流方法,实时荧光PCR技术和数字PCR技术为肉类成分检测开辟了新的途径。目前,肉类产品的定性检测方法研究较为充分,但是如何判定掺假成分是添加还是污染所致,如何确定掺假严重程度都需要可靠的定量技术作为依托,因此研发精准的定量检测技术将成为检测方法的发展方向。
参考文献:
[1] 杨志敏. 应用近红外光谱技术快速检测原料肉新鲜度及掺假的研究[D]. 杨凌: 西北农林科技大学, 2011.
[2] 金萍, 丁洪流, 李培, 等. 2013年苏州地区肉及其制品掺假情况调查[J]. 中国食品卫生杂志, 2014, 26(2): 168-172. DOI:10.13590/ j.cjfh.2014.02.016.
[3] NAKYINSIGE K, MAN Y B C, SAZILI A Q. Halal authen ticity issues in meat and meat products[J]. Meat Science, 2012, 91(3): 207-214. DOI:10.1016/j.meatsci.2012.02.015.
[4] ZUKÁL E, KÖRMENDY L. On calculation of ‘meat content’according to the quantitative ingredient decl arations (QUID)[J]. Journal of Food Engineering, 2007, 78(2): 614-621. DOI:10.1016/ j.jfoodeng.2005.10.033.
[5] 陈颖, 吴亚君. 基因检测技术在食品物种鉴定中的应用[J]. 色谱, 2011, 29(7): 594-600. DOI:10.3724/SP.J.1123.2011.00594.
[6] European Commission (EC). EC-152-2009. Method of analysis for the determination of constituents of animal origin for the official control of feed[S]. Europe: In official journal of the European communities, 2009.
[7] EENGLING F P, JORGENSON J S, PARADIES, et al. Evidence of animal meal in feeds[J]. Kraftfutter, 2000, 1: 14-17.
[8] 吕二盼. 动物源性食品中各种动物源性肉及肉制品鉴别检验的研究[D]. 保定: 河北农业大学, 2012.
[9] PRAYSON B E, MCMAHON J T, PRAYSON R A. Applying morphologic techniques to evaluate hotdogs: what is in the hotdogs we eat?[J]. Annals of Diagnostic Pathology, 2008, 12(2): 98-102. DOI:10.1016/j.anndiagpath.2007.04.012.
[10] GHISLENI G, STELLA S, RADAELLI E, et al. Qualitative evaluation of tortellini meat fi lling by histology and image analysis[J]. International Journal of Food Science & Technology, 2010, 45(2): 265-270. DOI:10.1111/j.1365-2621.2009.02130.x.
[11] ELLIS D I, BROADHUR ST D, CLARKE S J, et al. Rapid identifi cation of closely related muscle foods by vibrational spectroscopy and machine learning[J]. Analys t, 2005, 130(12): 1648-1654. DOI: 10.1039/B511484E.
[12] SOWOIDNICH K, KRONFELDT H D. Shifted excitation Raman difference spectroscopy at multiple wavelengths for in-situ meat species differentiation[J]. Applied Physics B, 2012, 108(4): 975-982. DOI:10.1007/s00340-012-5160-0.
[13] ARISTOY M C, TOLDRÁ F. His tidine dipeptides HPLC-based test for the detection of mammalian orig in proteins in feeds for ruminants[J]. Meat Science, 2004, 67(2): 211-217. DOI:10.1016/ j.meatsci.2003.10.008.
[14] DING H B, XU R J. Near-infrared spectroscopic technique for detection of beef hamburger adulteration[J]. Journal of Agricultural and Food Chemi stry, 2000, 48(6): 2193-2198. DOI:10.1021/jf9907182.
[15] RESTAINO E, FASSIO A, COZZOLINO D. Discrimination of meat patés according to the animal species by mea ns of near infrared spectroscopy and chemometrics[J]. CyTA: Journal of Food, 2011, 9(3): 210-213. DOI:10.1080/19476337.2010.512396.
[16] MORSY N, SUN D W. Robust linear and non-linear models of NIR spectroscopy for detection and quantification of adulterants in fresh and frozen beef[J]. Meat Science, 2013, 93(2): 292-302. DOI:10.1016/ j.meatsci.2012.09.005.
[17] KAMRUZZAMAN M, SUN D W, ELMASRY G, et al. Fast detection and visualization of minced lamb meat adulteration using NIR hyperspectral imaging and multivariate image analysis[J]. Talanta, 2013, 103(1): 130-136. DOI:10.1016/j.talanta.2012.10.020.
[18] 孙淑敏, 郭波莉, 魏益民, 等. 近红外光谱指纹分析在羊肉产地溯源中的应用[J]. 光谱学与光谱分析, 2011, 31(4): 937-941. DOI:1013964/j1issn11000-0593 (2011) 04-0937-05.
[19] Ś LIWINSKA M, WISNIEWSKA P, DYMERSKI T, et al. Food analysis using artificial senses[J]. Journal of Agricultural and Food Chemistry, 2014, 62(7): 1423-1448. DOI:10.1021/jf403215y.
[20] 贾洪锋, 卢一, 何江红, 等. 电子鼻在牦牛肉和牛肉猪肉识别中的应用[J]. 农业工程学报, 2011, 27(5): 358-363. DOI:10.3969/ j.issn.1002-6819.2011.05.062.
[21] GARCIA M, ALEIXANDRE M, GUTIERREZ J, et al. Electronic n ose for ham discrimination[J]. Sensors and Actuators B: Chemical, 2006, 114(1): 418-422.DOI:10.1016/j.snb.2005.04.045.
[22] TIAN X J, WANG J, CUI S Q. Analysis of pork adulteration in minced mutton using electronic nose of metal oxide sensors[J]. Journal of Food Engineering, 2013, 119(4): 744-749. DOI:10.1016/ j.jfoodeng.2013.07.004.
[23] 田晓静, 王俊, 崔绍庆. 羊肉纯度电子舌快速检测方法[J]. 农业工程学报, 2013, 29(20): 255-262. DOI:10.3969/j.issn.1002-6819.2013.20.033.
[24] 韩剑众, 黄丽娟, 顾振宇, 等. 基于电子舌的鱼肉品质及新鲜度评价[J]. 农业工程学报, 2008, 24(12): 141-144. DOI:10.3321/ j.issn:1002-6819.2008.12.030.
[25] WILKINS M R, SANCHEZ J C, GOOLEY A A, et al. Progress with proteome projects: why all proteins expressed by a genome should be identi fied and how to do it[J]. Biotechnology and Genetic Engineering Reviews, 1996, 13(1): 19-50. DOI:10.1080/02648725. 1996.10647923.
[26] BENDIXEN E. The use of proteomics in meat science[J]. Meat Science, 2005, 71(1): 138-149. DOI:10.1016/j.meatsci.2005.03.013.
[27] 赵方圆, 吴亚君, 韩建勋, 等. 蛋白质组学技术在食品品质检测及鉴伪中的应用[J]. 中国食品学报, 2012, 12(11): 128-135. DOI:1009-7848(2012)11-0128-08.
[28] LAMETSCH R, ROEPSTORFF P, BENDIXEN E. Identification of protein degradation during post-mortem storage of pig meat[J]. Journal of Agricultural and Food Chemistry, 2002, 50(20): 5508-5512. DOI:10.1021/jf025555n.
[29] MORZEL M, CHAMBON C, HAMELIN M, et al. Proteome changes during pork meat ageing following use of two different presl aughter handling procedures[J]. Meat Science, 2004, 67(4): 689-696. DOI:10.1016/j.meatsci.2004.01.008.
[30] BOULEY J, CHAMBON C, PICARD B. Mapping of bovine skeletal muscle proteins using two-dimensional gel electrophoresis and mass spectrometry[J]. Proteomics, 2004, 4(6): 1811-1824. DOI:10.1002/ pmic.200300688.
[31] DOHERTY M K, MCLEAN L, HAYTER J R, et al. The proteome of chicken skeletal muscle: changes in soluble protein expression during growth in a layer strain[J]. Proteomics, 2004, 4(7): 2082-2093. DOI:10.1002/pmic.200300716.
[32] HOLLUNG K, GROVE H, FAERGESTAD E M, et al. Comparison of muscle proteome profi les in pure breeds of Norwegian Landrace and Duroc at t hree different ages[J]. Meat Science, 2009, 81(3): 487-492. DO I:10.1016/j.meatsci.2008.10.003.
[33] X U Y J, JIN M L, WANG L J, et al. Differential proteome analysis of porcine skeletal muscles between Meishan and Large White[J]. Journal of Animal Science, 2009, 87(8): 2519-2527. DOI:10.2527/jas.2008-1708.
[34] KWASIBORSKI A, SAYD T, CHAMBON C, et al. Pig longissimus lumborum proteome: Part I. Effects of genetic background, rearing environment and gender[J]. Meat Science, 2008, 80(4): 968-981. DOI:10.1016/j.meatsci.2008.04.028.
[35] KIM N-K, JOH J H, PARK H R, et al. Differential expression profi ling of the proteomes and their mRNAs in porcine white and red skeletal muscles[J]. Proteomics, 2004, 4(11): 3422-3428. DOI:10.1002/ pmic.200400976.
[36] SENTANDREU M Á, SENTANDREU E. Authenticity of meat products: tools against fraud[J]. Food Research International, 2014, 60(6): 19-29. DOI:10.1016/j.foodres.2014.03.030.
[37] MONTOWSKA M, POSPIECH E. Species-specific expression of various pr oteins in meat tissue: proteomic analysis of raw and cooked meat and meat products made from beef, pork and selected poult ry species[J]. Food Chemistry, 2013, 136(3): 1461-1469. DOI:10.1016/ j.foodchem.2012.09.072.
[38] RIGHETTI P G, BOSSI A. Isoelectric focusing in immobilized pH gradients: an update[J]. Journal of Chromatography B: Biomedical Sciences and Applications. 1997, 699(1): 77-89. DOI:10.1016/S0378-4347(96)00151- X.
[39] 马永征 , 马冬, 白娣斯, 等. 免疫学检测肉类制品掺假研究进展[J].肉类研究, 2012, 26(9): 26-29. DOI:1001-8123(2012)09-0026-04.
[40] 张占军, 王富花. 酶联免疫吸附技术及其在食品安全检测中的应用[J]. 食品研究与开发, 2011, 32(1): 157-160. DOI:10. 3969/j. issn.1005-6521. 2011. 01. 045.
[41] MARTIN D R, CHAN J, C HIU J Y. Quantitative evaluation o f pork adulteration in raw ground beef by radial immunodiffusion and enzyme-linked immunosorbent assay[J]. Journal of Food Protection, 1998, 61(12): 1686-1690.
[42] RENČOVÁ E, SVOB ODA I, NECIDOVA L. Identifi cation by ELISA of poultry, horse, kangaroo, and rat muscle specifi c proteins i n heatprocessed products[J]. Veterinarni Medicina, 2000, 45(12): 353-356.
[43] LIU L H, CHEN F C, DORSEY J L, et al. Sen sitive monoclonal antibody-based sandwich elisa for the detection of porcine skeletal muscle in meat and feed products[J]. Journal of Food Science, 2006, 71(1): M1-M6. DOI:10.1111/j.1365-2621.2006.tb12393.x.
[44] GIOV ANNACCI I, GUIZARD C, CARLIER M, et al. Species identification of meat products by ELISA[J]. International Journal of Food Science & Technology, 2004, 39(8): 863-867. DOI:10.1111/ j.1365-2621.2004.00859.x.
[45] CHOU C C, LIN S P, LEE K M, et al. Fast differentiation of meats from fifteen animal species by liquid chromatography with electrochemical detection using copper nanoparticle plated electrodes[J]. Journal of Chromatography B, 2007, 846(1): 230-239. DOI:10.1016/j.jchromb.2006.09.006.
[46] GIARETTA N, di GIUSEPPE A, LIPPERT M, et al. Myoglobin as marker in meat adulteration: a UPLC method for determining the presence of pork meat in raw beef burger[J]. Food Chemistry, 2013, 141(3): 1814-1820. DOI:10.1016/j.foodchem.2013.04.124.
[47] 何玮玲, 黄明, 张驰. 食品中肉类成分种属鉴别技术研究进展[J]. 食品科学, 2012, 33(3): 304-307.
[48] von BARGEN C, DOJAHN J R, WAIDELICH D, et al. New sensitive high-performance liquid chromatography-tandem mass spectrometry method for the detection of horse and pork in halal beef[J]. Journal of Agricultural and Food Chemistry, 2013, 61(49): 11986-11994. DOI:10.1021/jf404121b.
[49] MONTOWSKA M, RAO W, ALEXANDER M R, et al. Tryptic digestion coupled with a mbient desorption electrospray ioniza tion and liquid extraction surface analysis mass spectrometry enabling identifi cation of skeletal muscle proteins in mixtures and distinguishing between beef, por k, horse, chicken, and turkey meat[J]. Analytical Chemistry, 2014, 86(9): 4479-4487. DOI:10.1021/ac5003432.
[50] MCELHINNEY J, DOWNEY G, FEARN T. Chemometric processing of visible and near infrared refl ectance spectra for species identifi cation in select ed raw homogenised meats[J]. Journal of Near Infrared Spectroscopy, 1999, 7(3): 145-154.
[51] ASENSIO L, GONZÁLEZ I, GARCÍA T, et al. Determination of food authenticity by enzyme-linked immunosorbent assay (ELISA)[J]. Food Control, 2008, 19(1): 1-8. DOI:10.1016/j.foodcont.2007.02.010.
[52] WALKER J A, HUGHES D A, ANDERS B A, et al. Quantitative intra-short interspersed element PCR for species-specific DNA identification[J]. Analytical Biochemistry, 2003, 316(2): 259-269. DOI:10.1016/S0003-2697(03)00095-2.
[53] EBBEHØJ K F, THOMSEN P D. Differentiation of closely related species by DNA hybridization[J]. Meat Science, 1991, 30(4): 359-366. DOI:10.1016/0309-1740(91)90044-Q.
[54] CHIKUNI K, OZUTSUMI K, KOISHIKAWA T, et al. Species identifi cation of cooked meats by DNA hybridization assay[J]. Meat Science, 1990, 27(2): 119-128. DOI:10.1016/0309-1740(90)90060-J.
[55] WINTERØ A, THOMSEN P, DAVIES W. A comparison of DNA-hybridization, immunodiffusion, countercurrent immunoelectrophoresis and isoelectric focusing for detecting the admixture of pork to beef[J]. Meat Science, 1990, 27(1): 75-85. DOI:10.1016/0309-1740(90)90030-A.
[56] EBBEHØJ K F, THOMSEN P D. Species differentiation of heated meat products by DNA hybridization[J]. Meat Science, 1991, 30(3): 221-234. DOI:10.1016/0309-1740(91)90068-2.
[57] BALLIN N Z, VOGENSEN F K, KARLSSON A H. Species determination: can we detect and quantify meat adulteration?[J]. Meat Science, 2009, 83(2): 165-174. DOI:10.1016/j.meatsci.2009. 06.003.
[58] 陈颖, 钱增敏, 徐宝梁, 等. 保健品中牛羊源性成分的PCR检测[J].食品科学, 2004, 25(10): 215-218.
[59] 陈颖, 吴亚君, 徐宝梁, 等. 食品及饲料中马属动物源性成分的PCR检测研究[J]. 中国生物工程杂志, 2004, 24(5): 78-83. DOI:10. 3969/ j.issn.1671-8135.2004.05.018.
[60] MARTÍN I, GARCÍA T, FAJARDO V, et al. Mitochondrial markers for the detection of four duck species and the specific identification of Muscovy duck in meat mixtures using the polymerase chain reaction[J]. Meat Science, 2007, 76(4): 721-729. DOI:10.1016/ j.meatsci.2007.02.013.
[61] SOARES S, AMARAL J S, MAFRA I, et al. Quantitative detection of poultry meat adulteration with pork by a duplex PCR assay[J]. Meat Science, 2010, 85(3): 531-536. DOI:10.1016/j.meatsci.2010.03.001.
[62] SAFAR M, JUNEJO Y, ARMAN K, et al. Ahighly sensitive and specific tetraplex PCR assay for soybean, poultry, house and pork species identifi cation in sausages: development and validation[J]. Meat Science, 2014, 98(2): 296-300. DOI:10.1016/j.meatsci.2014.06.006.
[63] ARMARAL J S, SANTOS C G, MELO V S, et al. Authentication of a traditional meat sausage (Alheira) by species- specifi c PCR assay to detect hare, rabbit, red deer, pork and cow meats[J]. Food Research International, 2014, 60: 140-145. DOI:10.1016/j.foodres.2013.11.003.
[64] ASLAN Ö, HAMILL R M, SWEENEY T, et al. Integrity of nuclear genomic deoxyribonucleic acid in cooked meat: implications for food tra ceability[J]. Journal of Animal Science, 2009, 87(1): 57-61. DOI:10.2527/jas.2008-0995.
[65] WILSON D W, BEERS P T. Global trade requirements and co mpliance with World Trade Organization agreements: the role of tracing animals and animal products[J]. Revue Scientifi que et Technique, 2001, 20(2): 379-382.
[66] VAZQUEZ J F, PEREZ T, URENA F, et al. Practical application of DNA fi ngerprinting to trace beef[J]. Journal of Food Protection, 2004, 67(5): 972-979.
[67] HEATON M P, KEEN J E, CLAWSON M L, et al. Use of bovine single nucleotide polymorphism markers to verify sample tracking in beef processing[J]. Journal of the American Veterinary Medical Association, 2005, 226(8): 1311-1314. DOI:10.2460/ javma.2005.226.1311
[68] 陈颖, 葛毅强. 现代食品分子检测鉴别技术[M]. 北京: 中国轻工业出版社, 2008: 196-208.
[69] CHIKUNI K, TABATA T, KOSUGIYAMA M, et al. Polymerase chain reaction assay for detection of sheep and goat meats[J]. Meat Science, 1994, 37(3): 337-345. DOI:10.1016/0309-1740(94)90051-5.
[70] SHARMA A, KUMAR S, SHARMA N, et al. Prospects in bioscience: addressing the issues[M]. India: Springer, 2013: 49-54.
[71] DOOSTI A, DEHKORDI P G, RAHIMI E. Molecular assay to fraud identification of meat products[J]. Journal of Food Science and Technology, 2014, 51(1): 148-152. DOI:10.1007/s13197-011-0456-3.
[72] 吴亚君, 韩建勋, 王斌, 等. 采用PCR-CE-SSCP技术快速筛查梅花鹿产品的鹿种真伪[J]. 食品科技, 2011, 36(11): 279-283. DOI:1005-9989(2 011)11-0279-04.
[73] 李文静, 李燕俊. 分子学方法鉴定肉制品种属来源的研究进展[J]. 国外医学(卫生学分册), 2009: 36(3): 151-158. DOI:1001-1226(2009)03-0151-08.
[74] SINGH V, NEELAM S. Meat species specifications to ensure the quality of meat: a review[J]. International Jou rnal of Meat Science, 2011, 1(1): 15-26. DOI:10.3923/ijmeat.2011.15.26.
[75] PRIMROSE S, WOOLFE M, ROLLINSON S. Food forensics: methods for determining the authenticity of foodstuffs[J]. Trends in Food Science & Technology, 2 010, 21(12): 582-590. DOI:10.1016/ j.tifs.2010.09.006.
[76] BALLIN N Z, MADSEN K G. Sex determination in beef by melting curve analysis of PCR amplicons from the amelogenin locus[J]. Meat Science, 2007, 77(3): 384-348. DOI:10.1016/j.meatsci.2007.04.029.
[77] LÓPEZ-ANDREO M, GARRIDO-PERTIERRA A, PUYET A. Evaluation of post-polymerase chain reaction melting temperature anal ysis for meat species identification in mixed dna samples[J]. Journal of Agricultural and Food Chemistry, 2006, 54(21): 7973-7938. DOI:10.1021/jf0615045.
[78] 郭云霞, 张舒亚, 谌鸿超, 等. SYBR Green实时荧光PCR检测食品中鲨鱼源性成分真实性方法的建立[J]. 食品与生物技术学报, 2012: 31(12): 1300-1306. DOI:10.3969/j.issn.1673-1689.2012.12.011.
[79] SOARES S, AMARAL J S, OLIVERIA M B P P, et al. A SYBR Green real-time PCR assay to detect and quantify pork meat in processed poultry meat products[J]. Meat Science, 2013, 94(1): 115-120. DOI:10.1016/j.meatsci.2012.12.012.
[80] 杨丽霞, 宋涛平, 谢晓红, 等. SYBR Green I 实时PCR技术鉴定鸭源性成分的研究[J]. 中国农学通报, 2013, 29(11): 16-19.
[81] WANG Ping, HU Yue, YANG Hairong, et al. DNA-based authentication method for d etection of yak (bos grunniens) in meat products[J]. Journal of AOAC International, 2013, 96(1): 142-146. DOI:http://dx.DOI.org/10.5740/jaoacint.11-398.
[82] ULCA P, BALTA H, ÇAĞIN İ, et al. Meat species identifi cation and Halal authentication using PCR analysis of raw and cooked traditional Turkish foods[J]. Meat Science, 2013, 94(3): 280-284. DOI:10.1016/ j.meatsci.2013.03.008.
[83] 张驰, 邱皓璞, 张筠. 荧光定量PCR检测肉制品中鸭源性成分[J]. 食品科学, 2013, 34(18): 154-157.
[84] LÓPEZ-ANDREO M, ALDEGUER M, GUILLÉN I, et al. Detection and quantification of meat species by qPCR in heat-processed food containing highly fragmented DNA[J]. Food Chemistr y, 2012, 134(1): 518-523. DOI:10.10 16/j.foodchem.2012.02.111.
[85] SOARES S, AMARAL J S, OLIVEIRA M B P P, et al. Quantitive detection of soybean in meat products by TaqMan real-time PCR assay[J]. Meat Scien ce, 2014, 98(1): 41-46. DOI:10.1016/ j.meatsci.2014.04.002.
[86] KESMEN Z, AYTEN G, MUSTAFA T Y, et al. TaqMan-based duplex real-time polymerase chain reaction approach for the detection and quantific ation of donkey and pork adulterations in raw and heatprocessed meats[J]. International Journal of Food Properties, 2014, 17(3): 629-638. DOI:10.1080/10942912.2012.654569.
[87] 吴亚君, 王斌, 刘鸣畅, 等. 阿胶中马驴成分的实时荧光PCR检测方法[J]. 食品科学, 2014, 35(8): 85-88. DOI:10.7506/spkx1002-6630-201408016.
[88] 中国检验检疫科学研究院. SN/T 373 1—2013 食品及饲料中常见禽类品种的鉴定方法[S]. 北京: 中国标准出版社, 2013.
[89] 中国检验检 疫科学研究院. SN/T 3730—2013 食品及饲料中常见畜类品种的鉴定方法[S]. 北京: 中国标准出 版社, 2013.
[90] 中国检验检疫科学研究院. SN/T 2557—2010 畜肉食品中牛成分定性检测方 法[S]. 北京: 中国标准出版社, 2010.
[91] 中国检验检疫科学研究院. SN/T 3589—2013 出口食品中常见鱼类及其制品的鉴伪方法[S]. 北京: 中国标准出版社, 2013.
[92] CAMMÀ C, di DOMENICO M, MONACO F. Development and validation of fast real-time pcr assays for species identifi cation in raw and cooked meat mixtures[J]. Food Control, 2012, 23(2): 400-404. DOI:10.1016/j.foodcont.2011.08.007.
[93] MIGUEL A, GARCÍA T, GONZÁLEZ I, et al. TaqMan realtime PCR for the detection and quantitation of pork in meat mixtures[J]. Meat Science, 2005, 70(1): 113-120. DOI:10.1016/ j.meatsci.2004.12.005.
[94] CHISHOLM J, CONYERS C, BOOTH C, et al. The detection of horse and donkey using real-time PCR[J]. Meat Science, 2005, 70(4): 727-732. DOI:10.1016/j.meatsci.2005.03.009.
[95] KÖPPEL R, DANIELS M, FELDERER N, et al. Multiplex real-time PCR for the detection and quantifi cation of DNA from duck, goose, chicken, turkey and pork[J]. European Food Research and Technology, 2013, 236(6): 1093-1098. DOI:10.1007/s00217-013-1973-2.
[96] DOOLEY J J, PAINE K E, GARRETT S D, et al. Detection of meat species using TaqMan real-time PCR assays[J]. Meat Science, 2004, 68(3): 431-438. DOI:10.1016/j.meatsci.2004.04.010.
[97] CHISHOLM J, CONYERS C, BOOTH C, et al. The detection of horse and donkey using real-time PCR[J]. Meat Science, 2005, 70(4): 727-732. DOI:10.1016/j.meatsci.2005.03.009.
[98] LÓPEZ-ANDREO M, LUGO L, GARRIDO-PERTIERRA A, et al. Identifi cation and quantitation of species in complex DNA mixtures by real-time polymerase chain reaction[J]. Analytical Biochemistry, 2005, 339(1): 73-82. DOI:10.1016/j.ab.2004.11.045.
[99] CHENG X, HE W L, HUANG F, et al. Multiplex real-time PCR for the identification and quantification of DNA from duck, pig and chicken in Chinese blood curds[J]. Food Research International, 2014, 60: 30-37. DOI:10.1016/j.foodres.2014.01.047.
[100] PEGELS N, GONZÁLEZ I, MARTÍN I, et al. Applicability assessment o f a real-time PCR assay for the specifi c detection of bovine, ovine and caprine material in feedstuffs[J]. Food Control, 2011, 22(8): 1189-1196. DOI:10.1016/j.foodcont.2011.01.015.
[101] KESMEN Z, YETIMAN A E, ŞAHIN F, et al. Detection of chicken and turkey meat in meat mixtures by using real-time PCR Assays[J]. Journal of Food Science, 2012, 77(2): C167-C173. DOI:10.1111/ j.1750-3841.2011.02536.x.
[102] KÖPPEL R, RUF J, RENTSCH J. Multiplex real-time PCR for the detection and quantification of DNA from beef, pork, horse and sheep[J]. European Food Research and Technology. 2011, 232(1): 151-155. DOI:10.1007/s00217-010-1371-y.
[103] IWOBI A, SEBAH D, KRAEMER I, et al. A multiplex real-time PCR method for the quantification of beef and pork fractions in minced meat[J]. Food Chemistry, 2015, 169: 305-313. DOI:10.1016/ j.foodchem.2014.07.139.
[104] KESMEN Z, GULLUCE A, SAHIN F, et al. Identification of meat species by TaqMan-based real-time PCR assay[J]. Meat Science, 2009, 82(4): 444-449. DOI:10.1016/j.meatsci.2009.02.019.
[105] DRUML B, GRANDITS S, MAYER W, et al. Authenticity control of game meat products: a single method to detect and quantify adulteration of fallow deer (Dama dama), red deer (Cervus elaphus) and sika deer (Cervus nippon) by real-time PCR[J]. Food Chemistry, 2015, 170: 508-517. DOI:10.1016/j.foodchem.2014.08.048.
[106] LUO A R, ZHANG A B, HO S Y, et al. Potential efficacy of mitochondrial genes for animal DNA barcoding: a case study using eutherian mammals[J]. BMC Genomics, 2011, 12(1): 84. DOI:10.1186/1471-2164-12-84.
[107] EATON M J, MEYERS G L, KOLOKOTRONIS S O, et al. Barcoding bushmeat: molecular identification of Central African and South American harvested vertebrates[J]. Conservation Genetics, 2010, 11(4): 1389-1404. DOI:10.1007/s10592-009-9967-0.
[108] 林彩琴, 姚波. 数字PCR技术进展[J]. 化学进展, 2012, 24(12): 2415-2423. [109] 李亮, 隋志伟, 王晶, 等. 基于数字PCR的单分子DNA定量技术研究进展[J]. 生物化学与生物物理进展, 2012, 39(10): 1017-1023. DOI:10. 3724/SP. J. 1206. 2012. 00003.
Progress in Meat Adulteration Detection Techniques
REN Junan
1,2, HUANG Wensheng
1, GE Yiqiang
2,3, CHEN Ying
1,*
(1. Chinese Academy of Inspection and Quarantine, Beijing 100123, China; 2. College of Food Science and Nutritional Engineering, China Agricultural University, Beijing 100083, China; 3. China Rural Technology Development Center, Beijing 100045, China)
Abstract:Currently, meat adulteration issues occur frequently, and severely impact import and export trade, consumers’rights and people’s health. The development of reliable and accurate testing methods has become a hot research topic in many countries. The most reported meat authenticity detection techniques for meat products include microscopic examination, enzyme-linked immunosorbent assay (ELISA), liquid chromatography, tandem mass spectrometry and polymerase chain reaction, based on the characteristics of morphology, metabolites, proteins and nucleic acids. This paper reviews the progress in identification techniques for meat ingredients and their future trends are discussed.
Key words:meat products; authenticity; identification; detection technology
DOI:10.7506/spkx1002-6630-201601043
中图分类号:TS207.3
文献标志码:A
文章编号:1002-6630(2016)01-0247-11
引文格式:
任君安, 黄文胜, 葛毅强, 等. 肉制品真伪鉴别技术研究进展[J]. 食品科学, 201 6, 37(1): 247-257. DOI:10.7506/spkx1002-6630-201601043. http://www.spkx.net.cn
REN Junan, HUANG Wensheng, GE Yiqiang, et al. Progress in meat adulteration detection techniques[J]. Food Science, 2016, 37(1): 247-257. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201601043. http://www.spkx.net.cn
收稿日期:2015-01-27
基金项目:国家高技术研究发展计划(863计划)项目(2011AA100807);中国检验检疫科学研究院基本科研业务费专项(2014JK027)
作者简介:任君安(1988—),女,博士研究生,研究方向为食品质量安全检测与控制。E-mail:renja126@126.com
*通信作者:陈颖(1972—),女,研究员,博士,研究方向为食品质量安全与控制。E-mail:chenyingcaiq@163.com
