
图1 苯甲酸类化合物1~13的化学结构
Fig.1 Chemical structures of benzoic acid and its analogues
李慕紫 1,王庆华 2,*,王晓艺 1,何 云 1
(1.广东药学院中药学院,广东 广州 510006;2.广东药学院基础学院,广东 广州 510006)
摘 要:以AutoDock 4.2和iGEMDOCK 2.1分子对接软件对13 种苯甲酸类似物与酪氨酸酶进行模拟对接研究,探讨苯甲酸类似物对接结合自由能与实验测得的酶抑制活性的关系,并对分子对接结果进行分析。结果表明:AutoDock 4.2程序中Cu电荷数的设置对结合自由能有显著影响,铜电荷数为2.0时,苯甲酸类似物结合自由能和pIC 50(-lgIC 50)线性相关系数可达0.803 6。iGEMDOCK 2.1预测苯甲酸类似物的结果线性较差,即AutoDock 4.2较iGEMDOCK 2.1预测酪氨酸酶抑制剂的可靠性更高。
关键词:酪氨酸酶;苯甲酸;抑制剂;分子对接
酪氨酸酶(EC 1.14.18.1)是结构复杂的多亚基含铜氧化还原酶,是人体黑色素合成的限速酶,通过抑制酪氨酸酶活性从而抑制黑色素生成 [1],可预防和治疗色素沉着、黑色素瘤等疾病,在化妆品领域也有广泛应用,使得越来越多的酪氨酸酶抑制剂得到研究开发 [2-3]。
随着计算机辅助药物设计的深入,分子对接的应用越来越广,现已有多个分子对接软件应用于药物筛选。利用分子对接软件对这些抑制剂与酪氨酸酶的相互作用进行预测,可以为了解这些化合物的酶活性抑制机理提供参考 [4]。AutoDock是美国Scripps研究所的Olson实验室开发与维护的一款分子对接模拟软件,主要应用于配体-蛋白质分子对接 [5]。iGEMDOCK软件是台湾国立交通大学药物设计和系统生物学实验室开发的分子对接软件,是一种集分子对接、筛选、对接或筛选后分析及图形可视化于一体的药物辅助设计工具 [6],其对接过程简单,选择受体分子、导入配体后选择合适的对接模式即可进行对接,且能同时对接多个配体,常用于快速对接和活性筛选。在具体研究中,不同的对接软件得到的对接结果不尽相同,且与对接过程中各参数的选取密切相关。本研究利用AutoDock和iGEMDOCK软件,模拟酪氨酸酶与13 种具有酪氨酸酶抑制活性的苯甲酸类似物的对接,分析小分子对酪氨酸酶的作用模式和机理,探讨对接结合自由能与抑制活性的相关性,以及两种对接软件用于预测酪氨酸酶抑制剂强度的可靠性。
1.1 AutoDock 4.2软件分子对接步骤及对接参数的设置
AutoDock Tools 1.5.4软件从官网(http://autodock. scripps.edu/)下载,编号2y9x的酪氨酸酶晶体结构文件来自Protein Data Bank,2y9x为双孢菇酪氨酸酶的X衍射三维晶体结构 [7],有8 个亚基A~H,对接位点选在A亚基活性中心小分子抑制剂环庚三烯酚酮(tropolone)所在区域。为充分暴露其活性中心,删去其余亚基及配体tropolone,得到用于对接的受体三维结构,保存为pdb格式 [8-9]。配体抑制剂结构在ZINC数据库下载,其三维结构坐标文件由GlycoBioChem PRODRG2 Server(http://davapc1.bioch. dundee.ac.uk/prodrg)转化。13 种苯甲酸类似物的结构式如图1所示 [10]。用AutoDock Tools 1.5.4软件处理受体分子:去水加氢,计算Gasteiger电荷,合并非极性氢原子,保存为pdbqt格式。同样,AutoDock Tools可对配体进行处理,其非极性氢原子被合并,Gasteiger电荷被加上,使能量最小化 [11]。AutoDock Tools 1.5.4软件中的Ligand子程序包可自动检测可旋转键的个数,在对接过程中,这些键可以灵活旋转与受体分子对接。AutoDock 4.2可以进行柔性对接,但选取柔性基团的不确定因素较多,因此本实验仍采用刚性对接形式。

图1 苯甲酸类化合物1~13的化学结构
Fig.1 Chemical structures of benzoic acid and its analogues
在格点盒子(gridbox)中,格点间距为0.375 Å,大小为60 Å×60 Å×60 Å,格子的中心为默认受体活性中心,坐标值为(-7.397, -23.551, -32.508)。配体构象搜索过程使用半经验打分函数与拉马克遗传算法 [12](Lamarckian genetic algorithm,LGA),算法对接的轮数(number of GA runs)设为100,对接次数越多,所耗时间会越长,但对接结果更可靠。在分子对接运行过程中,能量评估的最大数目(max number of evals)设为250万,平移步骤大小(rate of crossover)为0.8,其他参数取默认值 [13]。
酪氨酸酶为含Cu氧化还原酶,Cu电荷数的大小对其活性对接有影响,但Cu不是AutoDock 4.2的默认原子类型,需要在对接前自行添加Cu和设置电荷数,因此,Cu电荷数是需确定的重要参数之一 [14]。实验中分别设置受体坐标文件Cu电荷数为0、0.5、1.0、1.5、2.0、2.5、3.0,以苯甲酸为例,运行得到对接结合自由能,探讨Cu电荷最佳参数。
1.2 iGEMDOCK 2.1软件分子对接步骤及对接参数的设置
iGEMDOCK 2.1软件在导入酪氨酸酶受体分子时,不必删去多余的亚基,可自行将配体小分子tropolone所在区域作为活性位点 [15]。直接导入配体分子进行对接。对接参数设置:通用进化方法中群体大小(population size)设为300,子代数目(generations)设为80,解的个数(number of solutions)设为10,运行docking。
2.1 对接准确性验证
为验证AutoDock 4.2参数设置与对接结果的准确性,将酪氨酸酶晶体活性中心中的小分子tropolone作为配体,与删去配体的酪氨酸酶进行对接 [16]。对接的最优构象与在Protein Data Bank下载带有tropolone结构的酪氨酸酶三维X衍射图进行对比,发现模拟对接的小分子tropolone与三维X衍射图中的小分子结合在酪氨酸酶活性中心的相同部位,其构象大部分重合(图2),可见本实验中设置的对接参数合理,能较好地建立抑制剂与酶在活性中心的结合模型。
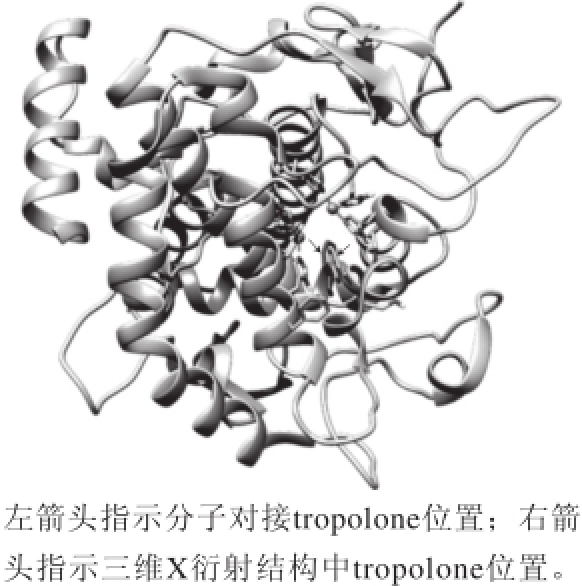
图2 分子对接与三维X衍射结构对比
Fig.2 Comparison between docking and three-dimensional X-ray structure
2.2 Cu电荷数改变对AutoDock对接结果的影响
图3显示了苯甲酸与酪氨酸酶对接结合自由能与所设置Cu电荷数大小的关系。Cu电荷数为0时,结果偏差大,Cu电荷数越大,对接结合自由能与Cu电荷数的线性关系越好。综合考虑Cu的价态,本实验中酪氨酸酶的Cu电荷数设为2.0。

图3 对接结合自由能与Cu电荷数关系
Fig.3 Relationship between binding free energy and Cu charge
2.3 AutoDock 4.2和iGEMDOCK 2.1软件对接结果
本研究筛选了13 种苯甲酸类似物的酪氨酸酶抑制剂,由文献[9,17-21]获得其抑制常数和半抑制浓度(IC 50),抑制剂的活性数据及两种软件预测的最低对接结合自由能见表1。
表1 苯甲酸类似物对酪氨酸酶的抑制作用及对接结合自由能
Table1 Inhibitory effect and binding free energy of benzoic acid and its analogues against tyrosinase
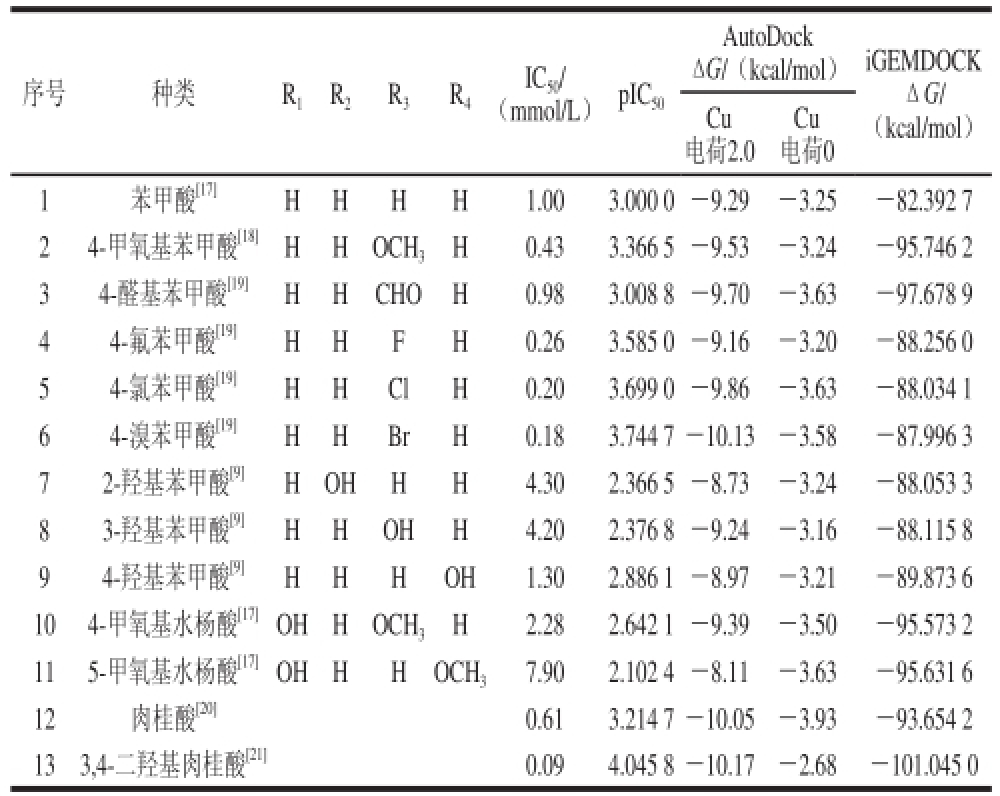
注:Δ G.对接结合自由能;肉桂酸与3,4-二羟基肉桂酸的结构见图1。
序号种类R 1R 2R 3R 4Cu电荷0 1苯甲酸 [17]HHHH1.003.000 0-9.29-3.25-82.392 7 24-甲氧基苯甲酸 [18]HHOCH 3H0.433.366 5-9.53-3.24-95.746 2 34-醛基苯甲酸 [19]HHCHOH0.983.008 8-9.70-3.63-97.678 9 44-氟苯甲酸 [19]HHFH0.263.585 0-9.16-3.20-88.256 0 54-氯苯甲酸 [19]HHClH0.203.699 0-9.86-3.63-88.034 1 64-溴苯甲酸 [19]HHBrH0.183.744 7-10.13-3.58-87.996 3 72-羟基苯甲酸 [9]HOHHH4.302.366 5-8.73-3.24-88.053 3 83-羟基苯甲酸 [9]HHOHH4.202.376 8-9.24-3.16-88.115 8 94-羟基苯甲酸 [9]HHHOH1.302.886 1-8.97-3.21-89.873 6 104-甲氧基水杨酸 [17]OHHOCH 3H2.282.642 1-9.39-3.50-95.573 2 115-甲氧基水杨酸 [17]OHHHOCH 37.902.102 4-8.11-3.63-95.631 6 12肉桂酸 [20]0.613.214 7-10.05-3.93-93.654 2 133,4-二羟基肉桂酸 [21]0.094.045 8-10.17-2.68-101.045 0 IC 50/(mmol/L)pIC 50Cu电荷2.0 AutoDock ΔG/(kcal/mol)iGEMDOCK ΔG/(kcal/mol)
以抑制剂pIC 50(-lgIC 50)为横坐标,对接结合自由能(取各对接构象中的最低对接结合自由能)为纵坐标,得到AutoDock 4.2软件对接的苯甲酸类似物与对接结合自由能相关性图。Cu电荷数设置为2.0时,其回归方程为y=-0.798 5x-6.950 7(r=0.803 6);Cu电荷数设置为0时,其回归方程为y=0.107 1x-3.705 4(r=0.203 2)。
苯甲酸及其类似物与酪氨酸酶iGEMDOCK 2.1的对接结合自由能见表1,其pIC 50与对接结合自由能的回归方程为y=-0.981 3x-88.674(r=0.112 2)。
2.4 酪氨酸酶对接的成键作用与活性中心分析
通过13 种抑制剂与酪氨酸酶的相互作用分析,发现与抑制剂作用的氨基酸残基主要有:His85、His263、His259、His296、His61、Phe292、Val283、Ala286、Gly281、Ser283等。选取苯甲酸预测所得对接结合自由能最低的构象作为最佳构象进行分析。苯甲酸与酪氨酸酶关键残基的作用机理如图4所示。由苯甲酸与受体的结合模型可推测其他抑制剂作为底物占据酶活性中心的作用机理。

图4 苯甲酸与酪氨酸酶活性中心结合构象
Fig.4 Conformation of benzoic acid combined with active sites of tyrosinase
在酪氨酸酶对接研究中,Cu电荷数的设置对苯甲酸类抑制剂自由能的影响很大。Cu是稳定酶活性中心和进行催化反应的重要辅助因子,设置较大的Cu电荷数,能够提高抑制剂与受体的亲和力,使对接结合自由能更小,根据Cu离子的氧化价态为+2,将对接的Cu电荷数设为2.0,并获得了较好的对接结果。13 种苯甲酸类似物对接结合自由能与抑制剂实验测定的抑制活性具有较好的线性相关性,基本能够反映抑制剂与酶结合力越大,对酶活性抑制作用越强。对接研究中曾以多种酪氨酸酶抑制剂与2y9x结构对接,发现若不按抑制剂的结构类似性分类研究,对接结合自由能与pIC 50无统计学上的相关性。苯甲酸类化合物的羧基具有络合作用,可能与酪氨酸酶的铜离子形成络合物,抑制酶的活性 [22]。抑制剂与酶对接结合自由能越小,结合能力越强,抑制效果更明显,则实验测得的IC 50越大。
AutoDock 4.2软件在进行构象搜索时使用的是半经验打分函数,对接结合自由能考虑因素有范德华力、氢键、静电作用力、去溶剂化和扭转作用能。肉桂酸是苯丙烯酸类化合物,其结构与苯甲酸相似,因苯环上的碳链延长,可旋转的键增多,与酶的亲和力更强,对接结合自由能较苯甲酸要小,相应IC 50也较小。而3,4-二羟基肉桂酸与酪氨酸酶的天然底物L-多巴(3,4-二羟基苯丙氨酸)结构非常类似,其IC 50在13 种苯甲酸类似物中最小,表现出非常强的酶活性抑制作用,而通过AutoDock 4.2对接得到的结合自由能亦是最小。酪氨酸酶非竞争性抑制剂4-氟苯甲酸、4-氯苯甲酸、4-溴苯甲酸的结合自由能与pIC 50相关性很好,卤素原子的电负性越小,IC 50越低,结合自由能值越小,其机理可能是酶催化时在底物与氧之间形成氢氧桥,卤素原子能影响氢氧桥的形成 [23],且卤素原子的电负性越小,卤代烃的活性越强,抑制效果越好,与AutoDock 4.2的对接结果一致。
iGEMDOCK 2.1软件打分时考虑范德华力、氢键和静电作用力,苯甲酸及其类似物与酪氨酸酶的对接结果显示范德华力约占对接总能量的60%。但iGEMDOCK 2.1对接结合自由能与pIC 50的线性相关性不高。
本研究采用AutoDock 4.2软件,通过添加Cu并调整其电荷数,成功将苯甲酸及其类似物等13 种抑制剂与酪氨酸酶进行分子对接,发现对接结合自由能与抑制剂实验测定的抑制活性具有较好的线性相关性,可用于结构相似的同类化合物中活性预测和相互作用分析。相比而言,iGEMDOCK 2.1软件在本研究所采用的13 种抑制剂中的对接结果与实验测定活性的相关性不如AutoDock 4.2。因此,在虚拟分子对接中应根据具体的体系和需要,选择适宜的对接软件。
参考文献:
[1] 李娜, 鲁晓翔. 酪氨酸酶抑制剂的研究进展[J]. 食品工业科技, 2010, 31(7): 406-409.
[2] 郑宗平, 兰山, 秦川, 等. 天然酪氨酸酶抑制剂研究进展: 种类及其构效关系[J]. 食品工业科技, 2014, 35(8): 374-378.
[3] 毕云枫, 宋凤瑞, 刘志强. 天然酪氨酸酶抑制剂的种类及其对酪氨酸酶抑制作用的研究进展[J]. 吉林大学学报(医学版), 2014, 40(2): 454-459. DOI:10.13481/j.1671-587x.20140247.
[4] KHAN M T H. Molecular design of tyrosinase inhibitors: a critical review of promising novel inhibitors from synthetic origins[J]. Pure and Applied Chemistry, 2007, 79(12): 2277-2295. DOI:10.1351/ pac200779122277.
[5] KANADE S R, SUHAS V L, CHANDRA N, et al. Functional interaction of diphenols with polyphenol oxidase[J]. FEBS Journal, 2007, 274(16): 4177-4187. DOI:10.1111/j.1742-4658.2007.05944.x.
[6] YANG J M, CHEN C C. GEMDOCK: a generic evolutionary method for molecular docking[J]. Proteins: Structure, Function, and Bioinformatics, 2004, 55(2): 288-304. DOI:10.1002/prot.20035.
[7] ISMAYA W T, ROZEBOOM H J, WEIJN A, et al. Crystal structure of Agaricus bisporus mushroom tyrosinase: identity of the tetramer subunits and interaction with tropolone[J]. Biochemistry, 2011, 50(24): 5477-5486. DOI:10.1021/bi200395t.
[8] 朱杰华, 宋胜玉, 谷万港. 基于结构的HIV-1整合酶抑制剂的虚拟筛选[J]. 中国生物制品学杂志, 2014, 27(1): 78-81.
[9] 张海玲, 光翠娥, 江波, 等. 皂苷类似物与肾素的分子对接和结合能分析[J]. 食品与生物技术学报, 2014, 33(10): 1056-1062. DOI:10.3969/j.issn.1673-1689.2014.10.008.
[10] 陈清西, 林建峰, 宋康康. 酪氨酸酶抑制剂的研究进展[J]. 厦门大学学报(自然科学版), 2007, 46(2): 274-282. DOI:10.3321/ j.issn:0438-0479.
[11] YIN S J, SI Y X, CHEN Y F, et al. Mixed-type inhibition of tyrosinase from agaricus bisporus by terephthalic acid: computational simulations and kinetics[J]. Protein Journal, 2011, 30(4): 273-280. DOI:10.1007/ s10930-011-9329-x.
[12] KHATIB S, NERYA O, MUSA R, et al. Enhanced substituted resorcinol hydrophobicity augments tyrosinase inhibition potency[J]. Journal of Medicinal Chemistry, 2007, 50(11): 2676-2681. DOI:10.1021/jm061361d.
[13] 李伟. 蛋白质配体结合位点柔性的系统分析及分子柔性对接方法的发展和应用[D]. 北京: 北京协和医学院, 2012: 78-107.
[14] SENDOVSKI M, KANTEEV M, BEN-YOSEF V S, et al. First structures of an active bacterial tyrosinase reveal copper plasticity[J]. Journal of Molecular Biology, 2011, 405(1): 227-237. DOI:10.1016/ j.jmb.2010.10.048.
[15] 刘文, 蒋世云, 李强, 等. 酚类抑制剂抗5-脂氧合酶的分子对接研究[J]. 生物技术通讯, 2012, 23(4): 527-531. DOI:10.3969/ j.issn.1009-0002.
[16] 伟尧, 朱琛, 张卓, 等. 硝基咪唑类似物作为尿素酶抑制剂的分子对接研究[J]. 扬州大学学报(农业与生命科学版), 2010(2): 6-9; 95.
[17] 张丽娟. 苯甲酸类似物对酪氨酸酶的效应及其抑菌作用[D]. 厦门:厦门大学, 2006: 52-76.
[18] 林建成, 邱凌, 张丽娟, 等. 茴香醛与茴香酸对蘑菇酪氨酸酶单酚酶的抑制作用[J]. 厦门大学学报(自然科学版), 2007(2): 254-257. DOI:10.3321/j.issn:0438-0479.
[19] 张春乐. 抑制剂对蘑菇氨酸的抑制效应及抗菌活性[D]. 厦门: 厦门大学, 2007: 30-48.
[20] 龚盛昭, 杨卓如, 程江. 肉桂酸抑制酪氨酸酶催化反应的动力学研究[J]. 高校化学工程学报, 2007, 21(2): 345-349. DOI:10.3321/ j.issn:1003-9015.
[21] 闫军, 李昌生, 陈声利, 等. 咖啡酸, 阿魏酸和香草酸对酪氨酸酶活性的影响[J]. 中国临床药理学与治疗学, 2004, 9(3): 337-339. DOI:10.3969/j.issn.1009-2501.
[22] 邹先伟, 蒋志胜. 植物源酪氨酸酶抑制剂研究进展[J]. 中草药, 2004, 35(6): 702-705. DOI:10.3321/j.issn:0253-2670.
[23] WANG Q, SHI Y, SONG K K, et al. Inhibitory effects of 4-halobenzoic acids on the diphenolase and monophenolase activity of mushroom tyrosinase[J]. Protein Journal, 2004, 23(5): 303-308. DOI:10.1023/ B:JOPC.0000032649.60933.58.
Molecular Docking of Benzoic Acid and Its Analogues with Tyrosinase
LI Muzi
1, WANG Qinghua
2,*, WANG Xiaoyi
1, HE Yun
1
(1. School of Traditional Chinese Medicine, Guangdong Pharmaceutical University, Guangzhou 510006, China; 2. School of Basic Courses, Guangdong Pharmaceutical University, Guangzhou 510006, China)
Abstract:Benzoic acid and its 12 analogues were docked with tyrosinase by using AutoDock 4.2 and iGEMDOCK 2.1 software, respectively. The linear correlation between docking binding free energy and experimental inhibitory activity of benzoic acid analogues was studied. The results from AutoDock 4.2 showed that the change in Cu charge had a significant effect on binding free energy, and the linear correlation coefficient between binding free energy and pIC 50was up to 0.803 6 when Cu charge was 2.0, but the linear correlation coefficient from iGEMDOCK 2.1 was poor. The prediction capacity of AutoDock 4.2 was more reliable than that of iGEMDOCK 2.1.
Key words:tyrosinase; benzoic acid; inhibitors; molecular docking
DOI:10.7506/spkx1002-6630-201603017
中图分类号:Q74
文献标志码:A
文章编号:1002-6630(2016)03-0087-04
引文格式:
李慕紫, 王庆华, 王晓艺, 等. 苯甲酸及其类似物与酪氨酸酶分子对接研究[J]. 食品科学, 2016, 37(3): 87-90. DOI:10.7506/spkx1002-6630-201603017. http://www.spkx.net.cn
LI Muzi, WANG Qinghua, WANG Xiaoyi, et al. Molecular docking of benzoic acid and its analogues with tyrosinase[J]. Food Science, 2016, 37(3): 87-90. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201603017. http://www.spkx.net.cn
收稿日期:2015-04-20
作者简介:李慕紫(1992—),女,硕士研究生,主要从事中药活性成分研究与新药开发。E-mail:muzilee_12@163.com
*通信作者:王庆华(1969—),男,教授,硕士,主要从事生物化学与分子生物学研究。E-mail:wqhwj@163.com