红球菌及其生物降解作用研究进展
邱孜博
1,汪 荣
1,张 杨
1,吴 茜
1,谢笔钧
1,杨季芳
2,陈吉刚
2,孙智达
1,
*
(1.华中农业大学食品科学技术学院,湖北 武汉 430070;2.浙江万里学院生物与环境学院,浙江 宁波 315100)
摘 要:红球菌是一类可以从土壤、深海等各种环境中分离得到的革兰氏阳性菌,属于放线菌门、放线菌纲、放线菌亚纲、放线菌目、诺卡氏菌科;含有独特的细胞壁结构、大型基因组与环状质粒,可分泌大量的活性酶,能够充分利用有机化合物作为能源和碳源。本文首先讨论了红球菌的分类,然后主要就红球菌对石油烃、有机腈和霉菌毒素的降解作用进行了阐述,现有研究结果表明红球菌有着很好的生物修复作用,可广泛应用于环境污染和工农业生产中。
关键词:红球菌;分类;生物降解
红球菌(Rhodococcus)广泛存在于土壤、海洋和极地环境,被认为是增强环境污染地区生物修复的理想之选。2008年日本学者Takaichi等
[1]从日本岛屿海洋表面分离出一株棒状杆菌Gordoniɑ terrɑe AIST-1,根据16S rRNA基因序列分析鉴定,该菌株被归到红球菌属。2014年,Liu Hui等
[2]从中国黑龙江省五常市凤凰山一种千足虫中分离出一株细菌,经多相分类研究发现其含有红球菌基因特征,后经16S rRNA基因序列相似性分析证明该菌株属于红球菌,并将其作为红球菌的新物种,命名为R. kronopolitis。
红球菌能够降解石油烷烃、芳香烃、多环芳烃、有机农药残留等环境污染物,还可以分解脂肪酶、腈水合酶和胆固醇氧化酶等,其中腈水合酶和脂肪酶可以运用到环境的生物修复中。红球菌中通常包含大型线性质粒和环状质粒,它们可以容纳大量的分解代谢基因,这些基因通过高频率的重组有助于其基因组的灵活性和获得新的基因,从而产生新的酶活性
[3]。越来越多的研究数据表明,红球菌中多种途径代谢和基因同源物的存在都可以进一步提高分解代谢的多功能性和效率。
本文对以往关于红球菌的研究进行了汇总,并主要就红球菌的分类及其生理作用进行阐述,如红球菌对石油烃、有机腈和霉菌毒素的降解作用等。
1 红球菌的分类及分布特征
红球菌细胞形态复杂,属于革兰氏阳性菌,一般是化能营养型,在37 ℃培养基上生长良好;菌落可以是粗糙的、光滑的或黏质的,色泽呈乳酪色、淡黄色、橙色、黄色或红色,也可以产生无色的变异菌
[4]。红球菌属在1977年作为R. rhodochrous新物种重新定义
[5]。近年来,红球菌属的数量大幅度增加,随着多相分类法的进一步发展和基于16S rRNA基因序列的分析,红球菌属的分类越来越清晰,红球菌属可以分为4~6 个亚目
[6-7]。通过对红球菌的分类进行汇总,发现有36 个种取得了分类学的合法地位,分别是:R. aetherivorans、R. aerolatus、R. baikonurensis、R. biphenylivorans、R. cercidiphylli、R. coprophilus、R. corynebacteroides、R. equi、R. erythropolis、R. fascians、R. globerulus、R. gordoniae、R. imtechensis、R. jostii、R. koreensis、R. kronopolitis、R. kroppenstedtii、R. kunmingensis、R. kyotonensis、R. maanshanensis、R. marinonenascens、R. opacus、R. percolatus、R. phenolicus、R. pyridinivorans、R. qingshengii、R. rhodnii、R. rhodochrous、R. ruber、R. triatomae、R. tukisamuensis、R. wratislaviensis、R. yunnanensis、R. zopfii、R. kronopolitis、R. soli。尽管红球菌的研究逐渐完善和清晰,其名称的合法性还是再次遭到了质疑
[8],红球菌不同物种的分类也遭到了质疑,如关于R. jialingiae
[9]和R. equi
[10]的重新分类。原来属于红球菌属的R. aechiensis、R. sputi、R. terrae等14 个种被分到其他的属,同时也有少量种分到红球菌属
[11]。
红球菌分布广泛,功能多样,常见的有R. erythropolis、R. equi、R. rhodochrous和红色红球菌R. ruber等。Pirog等
[12]研究了R. erythropolis EK-1在亲水性(乙醇和葡萄糖)和疏水性(液体石蜡和十六烷)底物上生长时产生表面活性剂的能力,发现该菌产生的表面活性剂具有乳化和表面活性的特性。红平红球菌还被发现是很好的脱硫细菌,并应用到实际生产运用中。研究表明,R. erythropolis IGTS8具有特异性脱硫作用
[12],DNA序列分析显示,脱硫相关基因包含dszA、dszB和dszC 3 个基因。2005年熊小超等
[13]从土壤中分离得到一株R. erythropolis LSSE8-1,经研究发现其脱硫基因与R. erythropolis IGTS8基因是高度同源的脱硫基因,测序结果已提交到GenBank数据库。马红球菌于1923年发现并被命名为马棒状杆菌,后经细胞壁结构分析发现该菌与棒杆菌属有较大差异,因此将其归属为红球菌属,命名为R. equi
[14]。它是一种机会致病菌,极易导致人类感染,以呼吸道感染最常见,最普通的病变是慢性化脓性支气管肺炎和广泛性肺部脓肿。临床上常见发热、咳嗽、呼吸困难及胸痛等
[15]。
2 红球菌的生物降解作用
2.1 红球菌降解石油烷烃的作用
红球菌能够适应各种各样的底物环境,具有极强的有机溶剂耐受性和很宽的降解谱。石油烃主要是由烷烃、环烷烃以及芳香烃三类烃组成,红球菌能够通过产生表面活性剂和改变细胞表面组成结构来提升自身对疏水环境的适应能力,从而有效地降解这3 种烃。据现有报道,红球菌属被视为主要的生物降解微生物,它们可以转化多种外源性化合物,包括多氯联苯、硝基芳香化合物,故而被认为是降解多环芳烃污染物的主要微生物
[16]。
红球菌的细胞膜上含有霉菌酸,霉菌酸的长脂肪链能够摄取疏水性物质进入细胞,且可产生表面活性剂,从而进一步提高疏水性物质的电位,降低细胞表面张力,使疏水性物质在两相系统中吸附于疏水相,更易进入菌体细胞
[10]。同时表面活性物质可以驱散亲水性化合物,使红球菌更易进入石油有机相内部。李丹等
[17]报道了R. erythropol能产生表面活性剂,并对该表面活性物质的理化性质进行了研究。R. rhodochrous CF222菌株产生的表面活性物质包括酸性多糖和酯,从而可增强其对十六烷的耐受性
[18]。王建宁等
[8]对北极表层海水进行低温(4 ℃)和常温(25 ℃)富集,结果发现红球菌属为优势菌属,并表明红球菌具有烷基脱卤酶基因,是极地环境中常见的石油烃降解菌。
然而,在红球菌降解石油烷烃的过程中,只有合适的浓度才有利于有机物的降解。当石油烷烃浓度过高时则会形成胶束,导致微生物无法直接与石油烷烃作用,从而降低了红球菌的降解过程
[19]。此外,研究还发现红球菌的表面特性对其降解石油烃的作用也有很大影响,具体的机制尚待进一步研究
[20]。
2.2 红球菌对有机腈的降解
2.2.1 腈的生物降解
腈是具有通式R-CN的化合物,有机腈是主要的化学化石燃料处理的中间产物、副产物或废弃物。腈的水解有2 种不同的路线,可以被腈水合酶或酰胺酶催化。丙烯腈和其他脂族腈的毒性被认为是由于部分氰化物释放在体内的原因,但由微粒体酶生物转化的不饱和腈也有可能产生毒性代谢物而不是氰化物。例如,丙烯腈的致癌作用可能归因于它的生物转化产物2-氰环氧乙烷
[21]。随着腈水解酶和腈水合酶的发现,微生物和植物降解腈的潜力也开始被发现应用,且许多微生物和酶对腈的转化表现出较高的活性和广泛的底物特异性,如红球菌腈水合酶表现出对丙烯腈的高活性,并且此活性与酶的类型无关(表1)。腈水合酶和腈水解酶都可以在红球菌属或某单一菌株中找到,它们的催化性能有很大的不同,腈水解酶具有相对不稳定的性质,腈水合酶可能妨碍生物催化和生物修复应用。普通酶的最适温度一般适中(37 ℃左右),与相同属的腈水解酶(<50 ℃)相比,红球菌腈水合酶需要较低的温度(<40 ℃)(表1)。因此,在实际生产运用中,应注意控制酶的温度。Rhodococcus sp. AJ270菌株被广泛地用作有机化学合成中的生物催化剂,也适用于原位丙烯腈的特异性生物修复
[15]。
表1 红球菌中腈水解酶的催化性
[22]
Table 1 Catalytic properties of purified nitrilases from the genus Rhodococcus
[22]

注:na.无法分析;上标a.重新分类(原诺卡氏菌);上标b.从10%饱和硫酸铵存在下的聚合亚单位中发现丙烯腈的活性;上标c.仅通过完整细胞测。
酶型 菌株 特殊活力/(U/mg pro)(底物)底物特异性/%最佳 稳定性pH T/℃ pH T/℃R. rhodochrous J1 14.6(苯甲腈) 苯甲腈:100丙烯腈
b:58.8 7.5 45 na <50 R. rhodochrous PA-34 24.5(苯甲腈)7.5 35 6.0~7.0 30~50芳香族苯甲腈:100丙腈:5丁腈:5丙烯腈:22 Rhodococcus sp. ATCC 39484 84.0(苯甲腈) 苯甲腈:100丙烯腈
b:1 7.5 30 5.5~9.5 25~40 Rhodococcus sp. NCIMB11216
abenzonitrile-induced 2.0(苯甲腈) 苯甲腈:100丙腈
c:5 8.0 30 na na Rhodococcus sp. NCIMB11216
apropionitrile-induced 10.6(苯甲腈) 苯甲腈:100丙腈
c:5 8.0 30 na na脂肪族 R. rhodochrous K22 2.6(丙烯腈)丙烯腈:100乙腈:8丙腈:4丁腈:5苯甲腈:8 5.5 50 6.0~8.0 <50
红球菌属细菌细胞壁对腈有一定的渗透性,它可以通过将腈转化为毒性较低的腈或通过腈水合酶或水解酶降解腈
[23]。研究发现,目前只有假单胞菌及其相关菌被报道与红球菌的生物降解作用有可比性,且红球菌的生理生化特性和遗传特性对其分解代谢起关键作用
[22]。
2.2.2 红球菌中的腈转化酶降解机制
根据底物特异性,腈水解酶分为三种:芳香族、脂肪族和芳基。研究发现,R. rhodochrous J1中位于腈水解酶基因(NITA)下游的调节基因(NITR)能够编码蛋白质,并对NITA基因转录有积极的调节作用
[24]。
腈水合酶一般分为Fe
3+型腈水合酶和Co
3+腈水合酶。通过对红球菌属不同菌株中Fe
3+型腈水合酶进行测序发现,不同的酶显示出高水平的相似性(>90%),当底物不同时它们的差异性也较大
[22]。携带Co
3+辅因子的腈水合酶包括两个亚型:低分子质量和高分子质量的腈水合酶
[25],它们同样也表现出不同底物的特异性。用不同的腈水合酶对不同活性的苄腈进行测定发现酶活性结果不同,其差异的程度较大。Brandao等
[26]认为这是由于不同的活性位点的氨基酸残基会影响酶的活性和底物特异性,如Rhodococcus sp. AJ 270和R. erythropolis A4中一些酶对芳香族、(芳基)脂肪族、脂环族和支链腈表现出广泛的特异性。此外,Fe
3+型腈水合酶中α和β亚基高度保守的编码基因,能够形成醛肟脱水酶和酰胺酶的基因簇。该基因簇的NHR
3基因可作为腈水合酶的活化剂,对功能性Fe
3+型腈水合酶的生物合成起着关键作用
[27]。NHR
1同系物的编码基因也可以与编码Co
3+型腈水合酶α和β亚基的基因
[28]和位于R. jostii RHA1中pRHL2大质粒上编码乙腈水合酶α和β亚基的基因形成基因簇
[20],编码酰胺酶的基因,尤其是脂肪酰胺(乙酰胺、丙酰胺、丁酰胺和苯乙酰胺)的基因,一般位于质粒pRHL2的腈水合酶亚基基因上游。根据图1中的红球菌中丁腈的代谢机制,发现经一系列酶的催化作用(醛肟脱水酶、α亚单位腈水合酶等),丁腈最终被代谢转化为丁酸,也因此会减少工业生产中对环境的污染,有利于现代工业化生产发展。
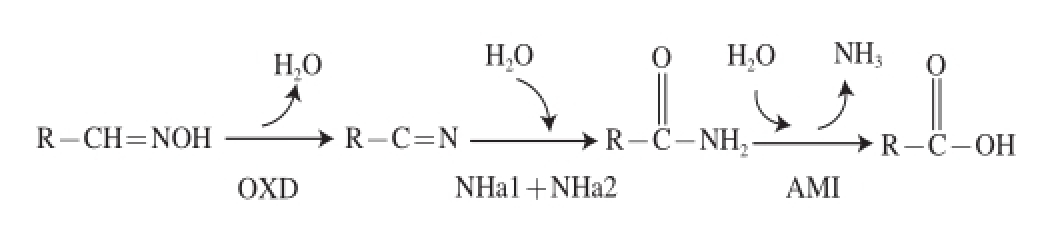
图1 红球菌中丁腈的代谢机制
[29-30]
Fig.1 Nitrile metabolism in Rhodococci
[29-30]
OXD. 醛肟脱水酶(aldoxime dehydratase);NHa1. α亚单位腈水合酶(nitrile hydratase-α-subunit);NHa2. β亚单位腈水合酶(nitrile hydratase-β-subunit)AMI.酰胺酶(amidase)。下同。
2.2.3 红球菌降解有机腈的应用
红球菌中腈水解酶能够将丙烯腈水解为毒性较小的丙烯酸胺,将丁腈降解为丁酸(图1),对聚合物的生产很有价值
[10]。红球菌Rhodococcus UKMP-5M被证明对含氰化物的废水降解效果显著,并且废水中金属的存在对红球菌降解氰化物不产生影响,这对废水氰化物的生物处理提供了参考
[31]。
有学者从R. jostii RHA1中分离纯化出一种可作用于脂肪腈的腈水合酶(乙腈水合酶),并得到了该酶的基因序列
[32]。该腈水合酶中(32%)的α和β亚单位之间存在着不同的同源性,且亚基的大小(63~56 kD)比其他腈水合酶(26~35 kD)的高,对乙腈有明显的特异性。Kato等
[30]对R. globerulus A-4、R. rhodochrous sp. N-771、R. erythropolis A4红球菌进行分析,发现其对腈的生物合成和生物代谢的基因合成通路如下(图2)。其中,NHR2为基因表达产物。

图2 红球菌中参与腈生物合成和生物降解的基因分布和通路
[29-30]
Fig.2 Organization of the genes involved in nitrile biosynthesis and biodegradation in Rhodococci and corresponding pathway
[29-30]
NHR1.腈水合酶(nitrile hydratase regulator 1);NHR2.腈水合酶调节因子2(nitrile hydratase regulator 2);NHR4.腈水合酶调节因子4(nitrile hydratase regulator 4);NHR3.腈水合酶催化剂(nitrile hydratase regulator 3)。
2.3 红球菌对霉菌毒素的降解
霉菌毒素是一些常见谷类植物中丝状真菌产生的高度有毒的次生代谢产物,常见的霉菌毒素有黄曲霉毒素B
1(Aspergillus flavus B
1,AFB
1)、赭曲霉毒素A(ochratoxins A,OTA)、玉米赤霉烯酮(zearalenone,ZON)和T2毒素。黄曲霉毒素能够干扰蛋白质合成转录抑制,赭曲霉毒素能够抑制代谢过程,玉米赤霉烯酮具有产雌激素和致畸作用
[33]。近年来,抗霉菌毒素的方法主要有化学、物理和生物3 种方法(表2)。
表2 抗霉菌毒素的方法
Table 2 Methods for the removal of mycotoxins

方法类型 原理 缺点化学法 加入氢氧化钙、臭氧、氨等选择性吸附,只对个别霉菌毒素有效;且过量化学物质的残留不利于动物的生长物理法将不同的吸附剂(黏土、硅铝酸盐类、木炭、酵母细胞壁等)加入饲料中选择性吸附,吸附力小、添加量大生物法利用微生物对真菌毒素的生物转化和生物降解可能产生有害的中间体和副产物,如玉米赤霉烯酮的生物转化中产生雌激素,AFB1环氧化物代谢中产生细胞毒性物(8,9-二氢-8,9-二羟基-黄曲霉毒素AFB
1)
红球菌能够与黄曲霉毒素的基础结构,即香豆素发生作用,从而降解黄曲霉毒素。通过对R. ruber、R. globerulus、R. rhodochrous、R. coprophilus、R. gordoniae、R. pyridinivorans和R. erythropolis等红球菌属菌株对霉菌毒素的降解进行研究,结果发现红球菌菌株降解AFB
1的能力由低到高依次为:R. ruber<R. globerulus<R. coprophilus<R. gordoniae<R. pyridinivorans<R. erythropolis。但种内的降解能力差异很大,例如在R. globerulus AK36菌株能降解黄曲霉毒素的效率为100%的情况下,R. globerulusN58的降解效率只有20%
[34]。
3 结 语
红球菌能够降解石油烃、有机腈和霉菌毒素的特性与其细胞壁、细胞膜及基因构成有着密切的关系,如红球菌菌体中大型基因组和线性质粒,由于它们可容纳大量的氧化酶和其他酶,使得红球菌能够充分利用来自有机化合物的能源和碳源。此外,红球菌还被发现能够产特殊的类胡萝卜素
[1,35],且通过拉曼显微光谱方法研究发现类胡萝卜素的积累发生在红球菌生物膜生长的整个阶段,通过控制红球菌生物膜的生长可以控制类胡萝卜素的积累
[34]。
本文对红球菌降解石油烃、有机腈和霉菌毒素的研究进行了简单的阐述,但关于它们的降解机制及红球菌特殊的基因组和环形质粒涉及较少。红球菌的分类学至今未完善和统一,红球菌产类胡萝卜素的基因位点等研究也有待进一步深入和完善。相信随着科学技术的进步和人类对红球菌研究兴趣的增加,以上各个问题可以得到解决,红球菌本身及其应用的研究也将更加全面,同时也将发现更多的红球菌生理功能,其特殊的生理生化结构及生理功能必将给人类和社会发展发挥推动作用。
参考文献:
[1] TAKAICHI S, MAOKA T, AKIMOTO N, et al. Carotenoids in a Corynebacterineae, Gordoniɑ terrɑe AIST-1: carotenoid glucosyl mycoloyl esters[J]. Bioscience, Biotechnology, and Biochemistry, 2008, 72(10): 2615-2622. DOI:10.1271/bbb.80299.
[2] LIU H, ZHANG Y, LIU C, et al. Rhodococcus kronopolitis sp. nov., a novel actinobacterium isolated from a millipede (Kronopolites svenhedind Verhoeff)[J]. Antonie van Leeuwenhoek, 2014, 106(6): 1207-1214. DOI:10.1007/s10482-014-0290-5.
[3] FINNERTY W R. The biology and genetics of the genus Rhodococcus[J]. Annual Reviews in Microbiology, 1992, 46(1): 193-218. DOI:10.1146/annurev.micro.46.1.193.
[4] 华苟根, 郭坚华. 红球菌属的分类及应用研究进展[J]. 微生物学通报, 2003, 30(4): 107-11. DOI:10.13344/j.microbiol.china.2003.04.027.
[5] GOODFELLOW M, ALDERSON G. The actinomycete-genus Rhodococcus: a home for the ‘rhodochrous’ complex[J]. Journal of General Microbiology, 1977, 100(1): 99-122. DOI:10.1099/00221287-100-1-99.
[6] ZHAO G Z, LI J, ZHU W Y, et al. Rhodococcus artemisiae sp. nov., an endophytic actinobacterium isolated from the pharmaceutical plant Artemisia annua L.[J]. International Journal of Systematic and Evolutionary Microbiology, 2012, 62(4): 900-905. DOI:10.1099/ ijs.0.031930-0.
[7] JONES A L, SUTCLIFFE I C, GOODFELLOW M. Prescottiɑ equi gen. nov., comb. nov.: a new home for an old pathogen[J]. Antonie van Leeuwenhoek, 2013, 103(3): 655-671. DOI:10.1007/s10482-012-9850-8.
[8] 王建宁, 董纯明, 赖其良, 等. 北极表层海水中氯代十六烷降解菌的多样性[J]. 微生物学报, 2012, 52(8): 1011-1020. DOI:10.13343/ j.cnki.wsxb.2012.08.011.
[9] TÁNCSICS A, BENEDEK T, FARKAS M, et al. Sequence analysis of 16S rRNA, gyrB and catA genes and DNA-DNA hybridization reveal that Rhodococcus jialingiae is a later synonym of Rhodococcus qingshengii[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(1): 298-301. DOI:10.1099/ijs.0.059097-0.
[10] KAMPFER P, DOTT W, MARTIN K, et al. Rhodococcus defluvii sp. nov., isolated from wastewater of a bioreactor and formal proposal to reclassify [Corynebacterium hoagii] and Rhodococcus equi as Rhodococcus hoagii comb. nov.[J]. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(3): 755-761. DOI:10.1099/ ijs.0.053322-0.
[11] GŰRTLER V, SEVIOUR R J. Systematics of members of the genus Rhodococcus(Zopf 1891) emend goodfellow et al. 1998[M]// ALVAREZ H M. Biology of rhodococcus. Berlin: Springer berlin heidelberg, 2010: 1-28. DOI:10.1007/978-3-642-12937-7_1.
[12] PIROG T, SHEVCHUK T, VOLOSHINA I, et al. Use of clayditeimmobilized oil-oxidizing microbial cells for purification of water from oil[J]. Applied Biochemistry and Microbiology, 2005, 41(1): 51-55. DOI:10.1007/s10438-005-0010-z.
[13] 熊小超, 李望良, 李信, 等. 专一性脱硫菌脱硫活性比较与基因保守性研究[J]. 微生物学报, 2005, 45(5): 733-737. DOI:10.13343/j.cnki. wsxb.2005.05.016.
[14] 李艳, 彭少华. 23株马红球菌的生物学性状及药敏分析[J].中华医院感染学杂志, 2000, 10(3): 232-233. DOI:10.3321/ j.issn:1005-4529.2000.03.037.
[15] BAXTER J, GARTON N, CUMMINGS S. The impact of acrylonitrile and bioaugmentation on the biodegradation activity and bacterial community structure of a topsoil[J]. Folia Microbiologica, 2006, 51(6): 591-597. DOI:10.1007/BF02931624.
[16] DUA M, SINGH A, SETHUNATHAN N, et al. Biotechnology and bioremediation: successes and limitations[J]. Applied Microbiology and Biotechnology, 2002, 59(2/3): 143-152. DOI:10.1007/s00253-002-1024-6.
[17] 李丹, 黄磊, 李国强, 等. 烃降解菌株 T7-2 产生的生物乳化剂及其理化性质研究[J]. 微生物学通报, 2008, 35(5): 653-660. DOI:10.13344/j.microbiol.china.2008.05.022.
[18] IWABUCHI N, SUNAIRI M, ANZAI H, et al. Relationships between colony morphotypes and oil tolerance in Rhodococcus rhodochrous[J]. Applied and Environmental Microbiology, 2000, 66(11): 5073-5077. DOI:10.1128/AEM.66.11.5073-5077.2000.
[19] 张光军, 方萍. 红球菌在石油烃类物质降解中的作用[J].环境科学与技术, 2013, 36(2): 93-99. DOI:10.3969/ j.issn.1003-6504.2013.02.019.
[20] OBUEKWE C O, AL-JADI Z K, AL-SALEH E S. Hydrocarbon degradation in relation to cell-surface hydrophobicity among bacterial hydrocarbon degraders from petroleum-contaminated Kuwait desert environment[J]. International Biodeterioration & Biodegradation, 2009, 63(3): 273-279. DOI:10.1016/j.ibiod.2008.10.004.
[21] SAILLENFAIT A, SABATE J. Comparative developmental toxicities of aliphatic nitriles: in vivo and in vitro observations[J]. Toxicology and Applied Pharmacology, 2000, 163(2): 149-163. DOI:10.1006/ taap.1999.8839.
[22] MARTÍNKOVÁ L, UHNÁKOVÁ B, PÁTEK M, et al. Biodegradation potential of the genus Rhodococcus[J]. Environment International, 2009, 35(1): 162-177. DOI:10.1016/j.envint.2008.07.018.
[23] LARKIN M J, KULAKOV L A, ALLEN C C. Biodegradation and Rhodococcus: masters of catabolic versatility[J]. Current Opinion in Biotechnology, 2005, 16(3): 282-290. DOI:10.1016/ j.copbio.2005.04.007.
[24] KOMEDA H, KOBAYASHI M, SHIMIZU S. Characterization of the gene cluster of high-molecular-mass nitrile hydratase (H-NHase) induced by its reaction product in Rhodococcus rhodochrous J1[J]. Proceedings of the National Academy of Sciences, 1996, 93(9): 4267-4272. DOI:10.1073/pnas.93.9.4267.
[25] HUGHES J, ARMITAGE Y C, SYMES K C. Application of whole cell rhodococcal biocatalysts in acrylic polymer manufacture[J]. Antonie van Leeuwenhoek, 1998, 74(1/3): 107-118. DOI:10.1023/ A:1001716332272.
[26] BRANDAO P F, CLAPP J P, BULL A T. Diversity of nitrile hydratase and amidase enzyme genes in Rhodococcus erythropolis recovered from geographically distinct habitats[J]. Applied and Environmental Microbiology, 2003, 69(10): 5754-5766. DOI:10.1128/ AEM.69.10.5754-5766.2003.
[27] LU J, ZHENG Y, YAMAGISHI H, et al. Motif CXCC in nitrile hydratase activator is critical for NHase biogenesis in vivo[J]. FEBS Letters, 2003, 553(3): 391-396. DOI:10.1016/S0014-5793(03)01070-6.
[28] KOMEDA H, HORI Y, KOBAYASHI M, et al. Transcriptional regulation of the Rhodococcus rhodochrous J1 nitA gene encoding a nitrilase[J]. Proceedings of the National Academy of Sciences, 1996, 93(20): 10572-10577. DOI:10.1073/pnas.93.20.10572.
[29] XIE S X, KATO Y, KOMEDA H, et al. A gene cluster responsible for alkylaldoxime metabolism coexisting with Nitrile hydratase and amidase in Rhodococcus globerulus A-4[J]. Biochemistry, 2003, 42(41): 12056-12066. DOI:10.1021/bi035092u.
[30] KATO Y, YOSHIDA S, XIE S X, et al. Aldoxime dehydratase coexisting with nitrile hydratase and amidase in the iron-type nitrile hydratase-producer Rhodococcus sp. N-771[J]. Journal of Bioscience and Bioengineering, 2004, 97(4): 250-259. DOI:10.1016/S1389-1723(04)70200-5.
[31] MANIYAM M N, SJAHRIR F, IBRAHIM A L, et al. Biodetoxification of cyanide-containing industrial wastewaters by Rhodococcus UKMP-5M[J]. Biologia, 2014, 69(12): 1635-1643. DOI:10.2478/s11756-014-0487-0.
[32] OKAMOTO S, ELTIS L D. Purification and characterization of a novel nitrile hydratase from Rhodococcus sp. RHA1[J]. Molecular Microbiology, 2007, 65(3): 828-838. DOI:10.1111/j.1365-2958.2007.05834.x.
[33] RICHARD J L. Some major mycotoxins and their mycotoxicoses: an overview[J]. International Journal of Food Microbiology, 2007, 119(1): 3-10. DOI:10.1016/j.ijfoodmicro.2007.07.019.
[34] CSERHÁTI M, KRISZT B, KRIFATON C, et al. Mycotoxindegradation profile of Rhodococcus strains[J]. International Journal of Food Microbiology, 2013, 166(1): 176-185. DOI:10.1016/ j.ijfoodmicro.2013.06.002.
[35] 郑晓冬, 王友永. 用红球菌生产类胡萝卜素的研究[J]. 浙江大学学报(农业与生命科学版), 2000, 26(5): 516-520. DOI:10.3321/ j.issn:1008-9209.2000.05.013.
Recent Progress in Studies of Rhodococcus and Its Application of in Biodegradation
QIU Zibo
1, WANG Rong
1, ZHANG Yang
1, WU Qian
1, XIE Bijun
1, YANG Jifang
2, CHEN Jigang
2, SUN Zhida
1,
*
(1. College of Food Science and Technology, Huazhong Agricultural University, Wuhan 430070, China; 2. College of Biological and Environmental Sciences, Zhejiang Wanli University, Ningbo 315100, China)
Abstract:Rhodococcus are gram-positive bacteria isolated from a variety of environments, such as soil and deep sea. They belong to the family Nocardiaceae of the order Actinomycetales of the subclass Actinobacteridae of the class Actinobacteria of the phylum Actinobacteria. The metabolic diversity of Rhodococci is associated with unique cell wall structure, large genome size and the presence of circular plasmids which accommodate large sets of oxidases along with other enzymes, which make these microbes highly competitive in the race to utilize energy and carbon sources derived from organic compounds. This paper discusses the classification of Rhodococcus and explains the role of Rhodococcus in the degradation of petroleum hydrocarbons, organic nitrile and mycotoxins. The existing studies suggest that Rhodococcus have good bioremediation ability and can be widely used in environmental pollution and industrial and agricultural production.
Key words:Rhodococcus; classification; biodegradation
DOI:10.7506/spkx1002-6630-201607045
中图分类号:Q819
文献标志码:A
文章编号:1002-6630(2016)07-0254-05
引文格式:
邱孜博, 汪荣, 张杨, 等. 红球菌及其生物降解作用研究进展[J]. 食品科学, 2016, 37(7): 254-258. DOI:10.7506/spkx1002-6630-201607045. http://www.spkx.net.cn
QIU Zibo, WANG Rong, ZHANG Yang, et al. Recent progress in studies of Rhodococcus and its application of in biodegradation[J]. Food Science, 2016, 37(7): 254-258. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201607045. http://www.spkx.net.cn
收稿日期:2015-05-01
基金项目:公益性行业(海洋)科研专项(201405015-5)
作者简介:邱孜博(1991—),女,硕士研究生,研究方向为天然产物化学。E-mail:ziboqiu@163.com
*通信作者:孙智达(1963—),男,教授,博士,研究方向为天然产物化学。E-mail:sunzhida@mail.hzau.edu.cn


