微滴数字PCR法对肉制品中牛源和猪源成分的定量分析
苗 丽
1,张秀平
2,陈 静
2,李 轲
2,王永杰
2,白 杰
2
(1.河南出入境检验检疫局,河南 郑州 450003;2.河南检验检疫鉴定咨询中心,河南 郑州 450003)
摘 要:为准确检测肉及肉制品中肉源成分的含量,实验基于微滴数字聚合酶链式反应(droplet digital polymerase chain reaction,ddPCR)技术,建立了定量检测肉及肉制品中牛肉和猪肉含量的方法。由ddPCR结果可知,在一定范围内生鲜肉质量与DNA含量、DNA含量与DNA拷贝数之间均呈现明显的线性关系,并以DNA含量为中间值计算出DNA拷贝数(C)与生鲜肉质量之间的换算公式M
牛=0.062C-0.943,M
猪=0.045C-1.72。应用建立的数字ddPCR方法对已知目标肉种含量的混合肉量进行检测,结果表明测量值和真实值基本一致,且不受外源物种的干扰。通过对市售量品的检测,能够准确检测出不同量品中牛肉和猪肉的含量,并发现存在掺假现象,说明该检测方法具有良好的市场应用前景。本实验建立的ddPCR方法在肉及肉制品中牛肉和猪肉含量的定量检测方面具有较大的应用潜力,可为肉制品真伪鉴别日常检测提供有力的科学依据。
关键词:微滴数字PCR;肉制品;DNA拷贝数;掺杂使假
苗丽, 张秀平, 陈静, 等. 微滴数字PCR法对肉制品中牛源和猪源成分的定量分析[J]. 食品科学, 2016, 37(8): 187-191.
MIAO Li, ZHANG Xiuping, CHEN Jing, et al. Quantitative analysis of bovine and porcine ingredients in meat products by droplet digital PCR[J]. Food Science, 2016, 37(8): 187-191. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201608033. http://www.spkx.net.cn
肉及肉制品的“掺杂使假”是食品加工、流通和餐饮中存在的重要问题,由于价格差异带来的利益诱惑,驱使一些不法商贩和企业在牛、羊等高价肉制品中掺杂一些低廉肉种,用低价肉经过香精处理后冒充高价肉,严重侵害消费者权益
[1-2]。三聚氰胺、瘦肉精、假牛肉事件以及2013年的马肉风波
[3]等食品问题层出不穷,政府及检验检疫部门应加强对肉及肉制品“掺杂使假”行为的监督,而可靠的定性和定量检测方法是检验的关键。
在所有动物源性成分的检验技术中,聚合酶链式反应(polymerase chain reaction,PCR)技术广泛应用于肉及肉制品的定性检测
[4-6],特别是荧光定量PCR,不但能用于定性检测
[7],还可以根据反应中的循环数Ct值与初始DNA模板浓度进行相对定量
[8-9]。但是由于普通PCR的扩增效率低、定量PCR需建立标准曲线及容易出现假阳性等因素,使得其定量体系需进一步完善
[10-11]。
微滴数字PCR(droplet digital PCR,ddPCR)是准确定量核酸的一门新的核酸扩增技术
[12-13]。将含有DNA或RNA的PCR反应体系分成约20 000 个微滴,经PCR扩增后,逐个对每个微滴反应进行检测,有荧光信号的微滴判读为1,没有荧光信号的微滴判读为0,根据泊松分布原理及阳性微滴的个数与比例即可得出靶分子的起始拷贝数
[14]。由于其不需要建立标准曲线,在低浓度的核酸含量下,也可高度灵敏和准确的对核酸的拷贝数进行绝对定量,使得ddPCR技术广泛用于基因表达分析
[15]、拷贝数变异分析
[16-17]、致病菌检测
[18]、转基因食品检测
[19]等,具有较好的应用前景。
本研究将具有绝对定量特性的ddPCR应用于肉及肉制品中牛肉和猪肉的定量检测,旨在探索肉的质量与拷贝数之间的关系,通过检测量品中不同肉种的质量来达到甄别恶意掺假与轻微沾染的目的,为质检部门打击不法商贩,维护消费者权益,提供有力科学依据。
1 材料与方法
1.1 材料
生鲜牛肉、猪肉、鸭肉、鸡肉、羊肉、鹅肉、鹌鹑肉、貂肉、狐狸肉等肉量;猪肉松、牛肉松、火腿肠、速冻撒尿牛肉丸等牛肉和猪肉制品、小麦粉、大豆粉、玉米粉等购自郑州某农贸市场。
1.2 试剂
Tris-饱和酚 索莱宝生物科技有限公司;蛋白酶K(20mg/mL) 日本Takara公司;ddPCR Master Mix 美国Bio-Rad公司;组织裂解液(1 mol/L Tris-HCl、0.5 mol/L乙二胺四乙酸、10%十二烷基硫酸钠)、氯仿-异戊醇(24∶1,V/V)、3 mol/L醋酸钠溶液均为自配试剂。
1.3 仪器与设备
DT500电子天平 江苏常熟长青仪器厂;Z36HK高速冷冻离心机 德国Hermle公司;NTS-4000AM恒温振荡水槽 日本Tokyo Tikakikai公司;BioSpec-nano微量核酸蛋白仪 岛津企业管理(中国)有限公司;Quest LabCycler梯度PCR仪 德国Senso公司;QX200 ddPCR微滴生成仪 美国Bio-Rad公司。
1.4 方法
1.4.1 引物与探针的设计
根据GenBank中公布的牛肉单拷贝β-actin基因序列,通过软件Meglign对其特异区域进行多序列的比对,针对其保守区域设计牛肉的特异性引物和探针,用FAM荧光染料基团标记牛探针的5’端,非荧光淬灭基团BHQ标记牛探针的3’端,并满足数字PCR的要求。猪源引物和探针引自文献[20],引物和探针均由上海生物工程技术有限公司合成。详细序列见表1。
表1 ddPCR的引物和探针
Table 1 Primer and probe sequences for quantitative ddPCR assays
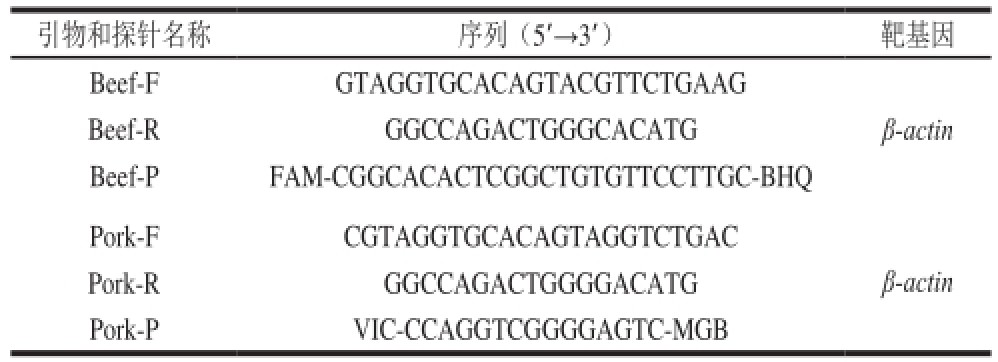
引物和探针名称序列(5’→3’)靶基因Beef-FGTAGGTGCACAGTACGTTCTGAAG β-actin Beef-RGGCCAGACTGGGCACATG Beef-PFAM-CGGCACACTCGGCTGTGTTCCTTGC-BHQ Pork-FCGTAGGTGCACAGTAGGTCTGAC Pork-RGGCCAGACTGGGGACATG Pork-PVIC-CCAGGTCGGGGAGTC-MGB β-actin
1.4.2 肉量制备
取新鲜肉量的肌肉组织,绞碎后于烤箱中80 ℃烘干72 h,于组织匀浆机中进行匀质处理,作为实验的标准品,并用于制作模量混合量品,匀质过程中不同肉类分开处理,防止不同动物组织粉末交叉污染。
1.4.3 DNA提取
采用苯酚-氯仿法提取基因组DNA
[20-21]。向50 mg肉粉里加入700 μL组织裂解液和20 μL蛋白酶K,涡旋振荡混匀,56 ℃水浴2 h,加入等量饱和酚,上下颠倒混匀,10 000 r/min离心10 min;取上清液,加入1/2体积的饱和酚和1/2体积的氯仿-异戊醇(24∶1,V/V),混匀,10 000 r/min离心10 min;取上清转移至一干净的EP管中,加入等量氯仿-异戊醇(24∶1,V/V),10 000 r/min离心10 min;取上清液,加入等体积的氯仿,10 000 r/min离心10 min;取上清液约400 μL,加入2.5 倍体积的无水乙醇及1/10体积的3 mol/L的醋酸钠溶液,混匀,-20 ℃沉淀2 h后4 ℃ 12 000 r/min离心30 min;弃上清液,加入1mL 75%乙醇溶液,4 ℃、12 000 r/min离心10 min;弃上清液,自然干燥后加入ddH
2O 100 μL溶解DNA,采用核酸定量测定仪测定提取肉量的DNA含量,确保提取DNA的OD
260
nm/OD
280
nm比值均在1.8~2.0之间,于-20 ℃保存备用。
1.4.4 ddPCR反应程序
20 μL反应体系:上下游引物各1.8 μL(900 nmol/L),探针0.5 μL(250 nmol/L),ddPCR Master Mix 10 μL,DNA模板4 μL,其余用无菌水补齐。通过微滴生成仪约生成20 000 个微滴。PCR反应条件:95 ℃ 10 min;94 ℃ 30 s,60 ℃ 1 min,98 ℃ 10 min,40 个循环。最后用微滴分析仪对扩增产物进行分析。
1.4.5 引物与探针的特异性
为验证所建ddPCR方法的特异性,将通过试剂盒测定的牛肉和猪肉阳性量品作为阳性对照,以羊、鸡、鸭、鹅、鹌鹑、貂、狐狸肉等常见肉及小麦粉、大豆粉、玉米粉等植物物种等提取的DNA作为模板进行检测。
1.4.6 肉量质量与拷贝数换算公式的确定
1.4.6.1 肉量质量与DNA含量的关系
分别称量10 个质量梯度的牛肉和猪肉(含量依次从5~50 mg)量品,提取基因组DNA,运用核酸定量测定仪测定DNA含量。每个梯度重复3 次。相关系数R
2的计算用Excel自动生成。
1.4.6.2 DNA含量与拷贝数的关系
将提取的牛肉和猪肉的DNA进行系列稀释后,分别以10 个DNA质量浓度梯度(25~250ng/μL)进行ddPCR的检测,分析DNA含量与拷贝数之间的关系。每个梯度重复3 次。相关系数R
2的计算用Excel自动生成。
1.4.7 ddPCR的抗干扰实验
为验证所建ddPCR方法的抗干扰性,以总质量为50 mg,分别称量10 个质量分数(10%~100%)的牛肉粉与猪肉粉制成两两混合量品;同时分别在牛肉粉与猪肉粉最低含量(10%)时添加不同质量分数的其他肉种,制成多肉量混合量品。按1.4.3节的方法提取混合肉量DNA,进行10 倍稀释后,取4 μL进行ddPCR检测,进行抗干扰实验。
1.4.8 市售量品的检测
分别从某市场中购买牛肉和猪肉制品(猪肉松、速冻肉包、火腿肠、速冻撒尿牛肉丸等),用建立的ddPCR方法对肉制品中牛源和猪源成分的质量进行检测,进一步验证所建方法的准确性和实际应用能力。
1.5 数据分析
所有结果采用SPSS 17.0进行统计学分析,利用Excel进行图表编辑。
2 结果与分析
2.1 引物和探针特异性
为验证本实验选用的牛肉和猪肉引物和探针的特异性,选择常见肉量及部分植物物种作为DNA模板进行ddPCR检测,结果显示选取的引物和探针对其他肉量及植物物种均无交叉反应,表明引物和探针的特异性良好,能够用于牛肉和猪肉含量的检测。
2.2 DNA提取及含量的测定
为摸索不同肉量质量与DNA含量的关系,提取牛肉和猪肉各10 个质量梯度(质量依次从5~50 mg)量品的DNA,运用核酸定量测定仪检测提取肉量的DNA含量。通过3 次批间重复,结果显示肉的质量与其所测定的DNA含量之间有明显的线性关系。牛肉和猪肉的相关系数R
2分别是0.995 9(图1)和0.997 7(图2),表明肉的质量在5~50 mg范围内与DNA含量有明显的线性关系,即y
牛=11.406x+13.602和y
猪=11.957x-3.160 7,其中x代表肉的质量/mg;y代表所测得的DNA含量/(ng/μL)。

图1 牛肉的质量与DNA含量的关系
Fig.1 Linear relationship between meat quantity and nucleic acid content of beef
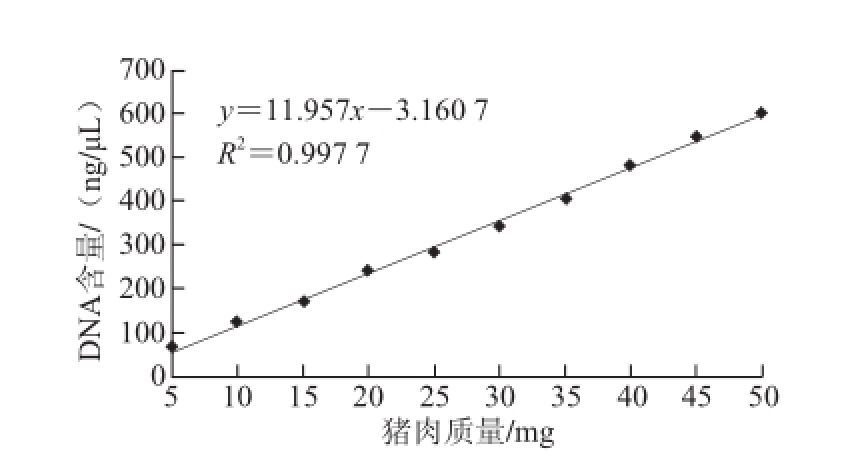
图2 猪肉的质量与DNA含量的关系
Fig.2 Linear relationship between meat quantity and nucleic acid content of pork
2.3 DNA含量与DNA拷贝数的关系
为摸索DNA含量与DNA拷贝数之间的关系,在ddPCR检测最高核酸质量浓度范围内,将提取的牛肉和猪肉DNA进行10 个梯度的系列稀释(25~250 ng/μL),取4 μL模板进行ddPCR的检测。结果显示在ddPCR检测过程中,每个量品的微滴生成均在15 000以上,满足了绝对定量的要求,且DNA含量与其所生成的DNA拷贝数之间有明显的线性关系。牛肉和猪肉的相关系数R
2分别是0.999 0(图3)和0.999 2(图4),表明核酸的含量在100~1 000 ng范围内与拷贝数呈明显的线性关性,即y
牛=3.526 2x-4.008 2和y
猪=4.626 4x+43.857,其中x代表所加模板DNA总量/ng;y代表所测得的DNA拷贝数/(copies/μL)。

图3 牛肉DNA含量与DNA拷贝数的关系
Fig.3 Linear relationship between nucleic acid content and DNA copy number of beef

图4 猪肉DNA含量与DNA拷贝数的关系
Fig.4 Linear relationship between nucleic acid content and DNA copy number of pork
2.4 肉量质量与拷贝数换算公式的确定结果
根据肉量的质量与所提取DNA含量之间的线性关系,及DNA含量与所测定DNA拷贝数之间的线性关系,选择DNA含量作为中间值,计算肉量的质量与拷贝数之间的关系,从而达到通过测定DNA拷贝数来计算原始肉量质量的目的。牛肉质量与DNA拷贝数之间的关系式:M
牛=0.062C-0.943;猪肉质量与DNA拷贝数之间的关系式:M
猪=0.045C-1.72,其中C代表每微升的DNA拷贝数/(copies/μL);M代表肉量的质量/mg。
2.5 已知质量混合肉量的数字PCR抗干扰实验结果
表2 已知质量的多种混合肉样的ddPCR定量分析
Table 2 Results of quantification of samples with known concentration

量品编号相对标准偏差/% 14544.24-1.6855.285.600 24040.892.221011.8618.600 33534.77-0.661515.956.3300 43029.58-1.42021.8900 52524.57-1.722527.3911.600 62019.13-4.353030.351.1700 71514.16-5.63536.975.6300 8109.4-64040.330.8300 954.63-7.44545.330.7300 1054.27-5.4000450 1155.020.4000045 1255.285.61515.835.531515 1300055.357450 1400055.6613.2045 151514.334.4755.361515实际质量/mg测量值/mg牛肉猪肉鸡肉实际质量/mg相对标准偏差/%实际质量/mg测量值/mg鸭肉实际质量/mg
为验证实验方法的准确性,分别对已知目标肉量质量分数的混合量品提取DNA,进行10 倍稀释后,取4 μL进行ddPCR检测,将DAN拷贝数值代入肉量质量与拷贝数关系式中计算肉量质量。结果显示测得的数值与真实肉量基本一致,并且不受外源肉量的干扰(表2),表明所建立的ddPCR方法可以应用于不同肉制品中牛源和猪源成分的定量检测。
2.6 市售量品的检测结果
表3 市售样品的ddPCR定量分析
Table 3 Results of quantification of samples from local supermarket %

量品测得牛肉含量测得猪肉含量儿童牛肉松27.80速冻撒尿牛丸2.4110.64火腿肠08.7儿童猪肉松026.6卤牛肉93.70牛肉卷2.536腊肠088.6猪肉卷091.2猪肉脯072.8牛肉干630
使用本研究建立的ddPCR方法,对市售肉制品中的牛源和猪源成分进行检测,结果如表3所示,其中儿童牛肉松和儿童猪肉松中牛肉和猪肉含量约为30%,经ddPCR测定后牛肉和猪肉的含量分别为27.8%和26.6%;速冻撒尿牛肉丸成分包含猪肉、鸡肉和牛肉,经检测牛肉含量仅为2.41%,相对含量较少。对牛肉卷的检测发现,牛肉含量占2.5%,而猪肉含量占36%,这表明市售肉制品存在掺假现象。通过一系列的市售食品的定量检测,验证了建立的牛肉和猪肉定量ddPCR检测体系的实际应用能力,可以用来甄别无意沾染与故意掺假以及衡量肉制品的掺假程度。
3 讨 论
食品安全、质量和原料组成成分等问题已经越来越多的受到公众的关注
[22-23],而传统的依靠感官与经验鉴别的手段已远不能满足对市售肉制品掺假现象进行控制和监管的需要,建立有效地测定肉源成分的检测方法是诸多研究人员研究的热点。
定量PCR是依靠标准曲线或参照基因来测定核酸含量
[9,24-25],而ddPCR能够直接数出DNA分子的个数,是对起始量品的绝对定量
[12,14]。因此特别适用于检测依靠Ct值不能很好分辨、以及目的核酸含量较低的量品。
本研究通过ddPCR分析了牛肉和猪肉量品的质量与拷贝数的关系,发现肉质量与DNA含量、DNA含量与DNA拷贝数之间均具有明显的线性关系,建立了肉质量与DNA拷贝数之间的换算公式,证明了ddPCR技术可以应用于肉及肉制品中种源成分的定量分析。这对于市售量品中某种肉源成分的无意沾染或有意添加可以进行准确的判断,由于检测过程不需要构建标准曲线,直接增加了检测效率,使得肉制品中掺假行为能够快速准确的检出。
本研究建立的检测体系主要着眼于市售肉及肉制品,鉴于其成分复杂,肉种多量,本研究制备了已知目标肉种质量分数的混合肉量来检验所建体系的抗干扰能力,结果显示本研究建立的牛源和猪源的检测体系无论在两两混合还是多肉量混合的情况下,抗干扰能力都很强,与实际质量相差不大。
为进一步验证所建方法的实际应用能力,选取含有目标肉种的市售加工食品进行检测。牛肉和猪肉ddPCR检测体系都能够特异性地检出各种肉制品中的目标肉种的含量。通过两种检测体系的联用,还可以进一步发现是否掺杂标签未注明肉类,如本研究发现某品牌牛肉卷中牛肉含量占2.5%,而猪肉含量占36%,说明牛肉卷中掺杂有大量的猪肉成分,证明市场上确实存在用低价肉冒充高价肉的掺假现象。
综上所述,本研究所建立的ddPCR检测体系弥补了传统检测方法的缺陷,更加快速、准确、灵敏,且可以进行绝对定量,可满足当前食品中种源性成分掺假的定量检测,尤其是应用于清真食品检测,以及保护对某种肉(牛、猪)成分过敏者,为质检部门打击不法商贩的食品掺假、造假行为,维护消费者合法利益提供有力的科学依据。
参考文献:
[1] 李巧玲, 刘景艳. 市场鲜猪肉掺假状况的调查监测[J]. 食品科学,2004, 25(10): 273-276. DOI:10.3321/j.issn:1002-6630.2004.10.068.
[2] LÓPEZ-CALLEJA I, GONZÁLEZ I, FAJARDO V. Quantitative detection of goats' milk in sheep's milk by real-time PCR[J]. Food Control, 2007, 8(11): 1466-1473. DOI:10.1016/j.foodcont.2006.11.006.
[3] OMAHONY P J. Finding horse meat in beef products-a global problem[J]. QJM-International Journal of Medicine, 2013, 106(6): 595-597. DOI:10.1093/qjmed/hct087.
[4] 高琳, 徐幸莲, 周光宏. PCR技术用于食品中原料肉物种鉴别的研究进展[J]. 肉类研究, 2006, 20(10): 19-21. DOI:10.3969/ j.issn.1001-8123.2006.10.008.
[5] STROMMENGER B, KETTLITZ C, WERNER G, et al. Multiplex PCR assay for simultaneous detection of nine clinically relevant antibiotic resistance genes in Staphylococcus aureus[J]. Journal Clinical Microbiology, 2003, 41: 4089-4094. DOI:10.1128/JCM.41.9.4089-4094.2003.
[6] KOPPEL R, JURG R, JURG R. Multiplex real-time PCR for the detection and quantification of DNA from beef, pork, horse and sheep[J]. European Food Research and Technology, 2011, 232(6): 151-155. DOI:10.1007/s00217-010-1371-y.
[7] 李林, 陈继明, 王志亮. Real-time PCR和常规PCR方法快速鉴定肉骨粉中的牛源性成分[J]. 中国预防兽医学报, 2005, 27(1): 50-54. DOI:10.3969/j.issn.1008-0589.2005.01.013.
[8] CALVO J H, OSTA R, ZARAGOZA P. Quantitative PCR detection of pork in raw and heated ground beef and pate[J]. Journal of Agricultural and Food Chemistry, 2002, 50(19): 5265-5267. DOI:10.1021/ jf0201576.
[9] ALI M E, HASHIM U, MUSTAFA S, et al. Analysis of pork adulteration in commercial meatballs targeting porcine-specific mitochondrial cytochrome b gene by TaqMan probe real-time polymerase chain reaction[J]. Meat Science, 2012, 91(4): 454-459. DOI:10.1016/j.meatsci.2012.02.031.
[10] PEKIN D, SKHIRI Y, BARET J C, et al. Quantitative and sensitive detection of rare mutations using droplet-based microfluidics[J]. Lab on a Chip, 2011, 11: 2156-2166. DOI:10.1039/C1LC20128J.
[11] JOTHIKUMAR P, HILL V, NARAYANAN J. Design of FRETTaqMan probes for multiplex real-time PCR using an internal positive control[J]. Biotechniques, 2009, 46(7): 519-524. DOI:10.2144/000113127.
[12] HINDSON B J, NESS K D, MASQUELIER D A, et al. Highthroughput droplet digital PCR system for absolute quantitation of DNA copy number[J]. Analytical Chemistry, 2011, 83: 8604-8610. DOI:10.1021/ac202028g.
[13] SANDERS R, HUGGETT J F, BUSHELL C A, et al. Evaluation of digital PCR for absolute DNA quantification[J]. Analytical Chemistry,2011, 83: 6474-6484. DOI:10.1371/journal.pone.0075296.
[14] MASSANELLA M, SINGHANIA A, BELIAKOVA B N, et al. Differential gene expression in HIV-infected individuals following ART[J]. Antiviral Research, 2013, 100(2): 420-428. DOI:10.1016/ j.antiviral.2013.07.017.
[15] BHAT S, POLANOWSKI A M, DOUBLE M C, et al. The effect of input DNA copy number on genotype call and characterising SNP markers in the humpback whale genome using a nanofluidic array[J]. PLoS ONE, 2012, 7(6): 1-6. DOI:10.1371/journal.pone.0039181.
[16] 姜羽, 胡佳莹, 杨立桃. 利用微滴数字PCR分析转基因生物外源基因拷贝数[J]. 农业生物技术学报, 2014, 22(10): 1298-1305. DOI:10.3969/j.issn.1674-7968.2014.10.013.
[17] BRUNETTO G S, MASSOUD R, LEIBOVITCH E C, et al. Digital droplet PCR (ddPCR) for the precise quantification of human T-lymphotropic virus 1 proviral loads in peripheral blood and cerebrospinal fluid of HAM/TSP patients and identification of viralmutations[J]. Journal of Neurovirology, 2014, 20(4): 341-351. DOI:10.1007/s13365-014-0249-3.
[18] 董莲华, 张玲, 姜君, 等. 大肠杆菌O157:H7微滴数字PCR定量方法的建立[J]. 分析化学, 2015, 43(3): 319-324. DOI:10.11895/ j.issn.0253-3820.140569.
[19] MORISSET D, STEBIH D, MILAVEC M, et al. Quantitative analysis of food and feed samples with droplet digital PCR[J]. PLoS ONE,2013, 8(5): 1-9. DOI:10.1371/Journal.pone.0062583.
[20] CAI Yicun, LI Xiang, LÜ Rong, et al. Quantitative analysis of pork and chicken products by droplet digital PCR[J]. BioMed Research International, 2014, 2014: 810209. DOI:10.1155/2014/810209.
[21] SANTELLA R M. Approaches to DNA/RNA extraction and whole genome amplification[J]. Cancer Epidemiology Biomarkers and Prevention, 2006,15(9): 1585-1587. DOI:10.1158/1055-9965.EPI-06-0631.
[22] 李家鹏, 乔晓玲, 田寒友, 等. 食品和饲料中动物源性成分检测技术研究进展[J]. 食品科学, 2011, 32(9): 340-347.
[23] 何伟玲, 黄明, 张弛. 食品中肉类成分种属鉴别技术研究进展[J]. 食品科学, 2012, 33(3): 304-307.
[24] 张弛, 邱皓璞, 张筠. 实时荧光定量PCR检测肉制品中鸭源性成分[J].食品科学, 2013, 34(18): 154-157. DOI:10.7506/spkx1002-6630-201318031.
[25] 赵新, 王勇, 兰青阔, 等. 荧光定量PCR方法鉴别肉制品中羊源性成分[J]. 食品工业科技, 2015, 36(1): 299-302. DOI:10.13386/ j.issn1002-0306.2015.01.054.
Quantitative Analysis of Bovine and Porcine Ingredients in Meat Products by Droplet Digital PCR
MIAO Li
1, ZHANG Xiuping
2, CHEN Jing
2, LI Ke
2, WANG Yongjie
2, BAI Jie
2
(1. Henan Entry-Exit Inspection and Quarantine Bureau, Zhengzhou 450003, China;2. Identification Consulting Center of Henan Inspection and Quarantine, Zhengzhou 450003, China)
Abstract:A highly precise quantitative method based on the droplet digital polymerase chain reaction (ddPCR) technique was developed to detect bovine and porcine ingredients in meat products. By using the ddPCR method, we found good linear relationships between the raw meat weight and DNA content and between the DNA content and DNA copy number. Using the DNA content as an intermediate value, we established the following formulae for calculating the weight of the original raw meat from the specific DNA copy number: M
beef= 0.062C - 0.943,M
pork= 0.045C - 1.72. By examining the mixed meat samples of known compositions, we found that the final quantitative results for the mixed samples were similar to the true raw meat weights, free from interference from exogenous species. Analysis of commercial samples showed that the ddPCR quantification system enables determination of the proportions of bovine and porcine ingredients in meat products with good practicability. Quantitative analysis indicates that ddPCR is highly precise in quantifying beef and pork in meat products and therefore has the potential to be used in routine analysis and meat adulteration of various species.
Key words:droplet digital polymerase chain reaction; meat products; DNA copy number; meat adulteration
DOI:10.7506/spkx1002-6630-201608033
中图分类号:TS207.3
文献标志码:A
文章编号:1002-6630(2016)08-0187-05
DOI:10.7506/spkx1002-6630-201608033. http://www.spkx.net.cn
收稿日期:2015-07-10
基金项目:国家质检总局科技计划项目(2014IK089)
作者简介:苗丽(1971—),女,高级兽医师,硕士,研究方向为分子生物学及食品微生物学。E-mail:ml5628@163.com
引文格式:






