市售酸奶中乳酸菌耐药性及耐药基因的检测
于 涛
1,姜晓冰
2,*,李 磊
1,王 辉
1,逯胜哲
1,张萌萌
1,祈真平
1,于明月
1
(1.新乡学院生命科学技术学院,河南 新乡 453000;2.河南师范大学生命科学学院,河南 新乡 453007)
摘 要:为探讨酸奶中乳酸菌所携带耐药基因对人类健康的潜在影响,对市售酸奶中的乳酸菌进行分离和鉴定,并通过药物敏感性实验确定菌株的耐药谱,同时利用聚合酶链式反应(polymerase chain reaction,PCR)扩增技术调查链霉素、庆大霉素、磺胺类和四环素等耐药基因的分布情况。结果表明:25 份市售酸奶样品中共分离得到56 株乳酸菌,包括26 株德氏乳杆菌保加利亚亚种、3 株植物乳杆菌、2 株嗜酸乳杆菌以及25 株嗜热链球菌。药敏结果显示,31 株乳杆菌对链霉素(87.1%)、庆大霉素(80.6%)、环丙沙星(74.2%)和四环素(61.3%)的耐药率较高,对头孢菌素类则较为敏感;而25 株嗜热链球菌同样对链霉素的耐药率最高,达76.0%;其次分别为万古霉素(32.0%)、环丙沙星(32.0%)和四环素(20.0%)。56 株乳酸菌中共检出5 种不同的耐药基因,分别为链霉素耐药基因ant(6)(检出率1.8%)、庆大霉素耐药基因aac(6')-aph(2'')(检出率7.1%)、四环素耐药基因tetM(检出率5.4%)以及磺胺类耐药基因sulⅠ(检出率14.3%)和sulⅡ(检出率1.8%)。受试的乳酸菌中共有13 株检出耐药基因,其中有4 株携带两种不同的耐药基因。长期以来被认为安全并广泛应用于发酵食品领域的乳酸菌可能成为潜在的耐药基因贮存库。
关键词:酸奶;乳酸菌;耐药;耐药基因
乳酸菌是指能够发酵糖类,主要代谢产物为乳酸的一类无芽孢、革兰氏阳性细菌的总称
[1]。除极少数外,绝大多数乳酸菌都是人体内必不可少且具有重要生理功能的菌群,广泛存在于人体肠道中。乳酸菌作为发酵剂或益生菌菌种被广泛应用于食品、农业、制药等行业。长期以来,发酵产品中所使用的乳酸菌普遍被人们认为是安全菌株
[2],其中德氏乳杆菌保加利亚亚种和嗜热链球菌是目前酸奶生产工艺中的主要发酵剂。
随着抗生素在临床、畜牧业、养殖业等领域的广泛应用甚至是滥用,细菌耐药性问题愈发严重。早期有关细菌耐药的研究主要集中于病原微生物
[3-4],但近年来对环境中共生细菌及乳酸菌的耐药性研究越来越受到学者的关注
[5-9]。研究表明,乳酸菌对临床使用的多种抗菌药物表现出不同程度的耐药性,多数乳酸菌对氨基糖苷类、糖肽类、氟喹诺酮类以及磺胺类等抗生素具有天然耐受能力
[10]。并且,已有学者在乳酸菌耐药菌株中检测到相应的耐药基因
[8,11-12]。Thumu等
[13]在分离自发酵食品的乳酸菌耐药株中检测到红霉素和四环素耐药基因。另外,研究者在生产酸奶用的乳杆菌、双歧杆菌和嗜热链球菌耐药菌株中也检测到相关的耐药基因
[8,14]。已有研究证实,在体外培养条件下耐药基因能够在不同种属的乳酸菌菌株中发生转移
[15]。这就意味着,当乳酸菌耐药菌株经食物摄入进入人体后,可能会通过基因水平转移等途径将耐药基因传递给肠道内其他共生菌甚至是致病菌,从而对人类健康构成潜在威胁。因此,是否携带耐药基因可做为评价乳酸菌安全性的指标之一。
鉴于此,本研究对市售酸奶中的乳酸菌进行分离和鉴定,通过药物敏感性实验确定菌株的耐药谱,同时调查链霉素、环丙沙星、庆大霉素、四环素、万古霉素和磺胺等耐药基因在这些菌株中的分布情况,为乳酸菌的安全评价提供参考依据。
1 材料与方法
1.1 材料、菌株与试剂
实验所用样品采购于河南省新乡市区4 个大型超市,共选购8 个厂家生产的保质期内酸奶25 份。样品在2 h内运送至实验室进行后续操作。
药敏实验用质控菌株Escherichia coli ATCC 25922、Staphylococcus aureus ATCC 25923和Pseudomonas aeruginosa ATCC 27853由中国人民解放军海军总医院检验科惠赠。
MRS培养基、MC琼脂培养基、革兰氏染色试剂盒、乳酸菌生化鉴定试剂盒 北京陆桥技术有限责任公司;11 种药敏纸片:庆大霉素(gentamicin,GEN,10 μg)、链霉素(streptomycin,STR,10 μg)、万古霉素(vancomycin,VAN,30 μg)、红霉素(erythromycin,ERY,15 μg)、四环素(tetracycline,TET,30 μg)、复方新诺明(trimethoprim/ sulfamethoxazole,SXT,1.25 μg+23.75 μg)、氨苄西林(ampicillin,AMP,10 μg)、环丙沙星(ciprofloxacin,CIP,5 μg)、头孢曲松(ceftriaxone,CRO,30 μg)、头孢他啶(ceftazidime,CAZ,30 μg)、头孢噻肟(cefotaxime,CTX,30 μg) 杭州天和微生物有限公司;细菌基因组DNA提取试剂盒 北京天根生化科技有限公司;Taq DNA聚合酶、dNTP、10×聚合酶链式反应(polymerase chain reaction,PCR)反应缓冲液、10×Loading Buffer 大连宝生物工程有限公司。
1.2 仪器与设备
Bactron I厌氧培养箱 美国Shellab公司;XSP-4C光学显微镜 上海宇隆仪器有限公司;ETC-811基因扩增仪 苏州东胜兴业科学仪器有限公司;DYY-8C电泳仪 北京六一生物科技有限公司;Universal Hood II凝胶成像分析系统 美国Bio-Rad公司。
1.3 方法
1.3.1 乳酸菌的分离与鉴定
参照GB 4789.35—2010《食品安全国家标准 食品微生物学检验 乳酸菌检验》中的检验方法:取酸奶样品25 mL(g)置于装有225 mL生理盐水的无菌锥形瓶中,充分混匀,制成1∶10的样品匀液。吸取样品匀液1 mL,用生理盐水按10 倍递增进行梯度稀释,选取3 个适宜的稀释度分别涂布于MRS和MC固体培养基上,36 ℃厌氧培养24~48 h。挑取典型单菌落划线于MRS或MC平板上进行纯化,纯化后的菌株进行革兰氏染色并镜检。利用乳酸菌生化鉴定试剂盒对所分离菌株进行生化鉴定。
1.3.2 DNA模板的制备
乳酸菌DNA的提取按照细菌基因组DNA提取试剂盒的产品说明书方法进行。
1.3.3 16S rDNA的PCR扩增
以提取的基因组DNA为模板,选用细菌通用引物进行扩增。正向引物27F:5’-AGAGTTTGAT CCTGGCTCAG-3’,反向引物1492R:5’-GGTTACCTT GTTACGACTT-3’
[16]。PCR反应程序为:95 ℃预变性3min;95 ℃变性1 min,55 ℃退火1 min,72 ℃延伸1 min,30 个循环;72 ℃延伸10 min。PCR产物送至上海英骏生物技术有限公司进行DNA序列双向测序,将测序结果在Ribosomal Database Project(RDP)数据库(http:// rdp.cme.msu.edu)进行比对确定菌株的种属。
1.3.4 药物敏感性实验
按照世界卫生组织(World Health Organization,WHO)推荐的K-B纸片扩散法检测菌株对抗生素的敏感性,结果判定依照美国临床实验室标准化委员会(Clinical and Laboratory Standards Institute,CLSI)2015版方法手册
[17]进行。目前关于乳酸菌对抗生素的耐药折点浓度尚缺乏统一的标准,在本研究中乳酸菌对氨苄西林、万古霉素、红霉素、四环素和环丙沙星的抑菌圈直径解释参照肠球菌属的标准;对头孢类、庆大霉素和复方新诺明的抑菌圈直径解释参考葡萄球菌属的标准;对链霉素的抑菌圈直径解释参考肠杆菌科的标准(CLSI中没有肠球菌属和葡萄球菌属对链霉素的抑菌圈直径解释标准)。药敏结果分为敏感(S)、中介(I)和耐药(R)3 种。每批实验均用Escherichia coli ATCC 25922、Staphylococcus aureus ATCC 25923和Pseudomonas aeruginosa ATCC 27853进行质量控制。
1.3.5 PCR扩增耐药基因
所有菌株分别经PCR扩增检测链霉素耐药基因strA、strB、ant(6),喹诺酮类(环丙沙星)质粒介导耐药基因qnrA、qnrB、qnrS,庆大霉素耐药基因aac(6')-aph(2''),四环素耐药基因tetM、tetS,万古霉素耐药基因vanA、vanB和磺胺甲基异噁唑耐药基因sulⅠ、sulⅡ,引物序列及扩增条件见表1。选择部分PCR产物送至上海英骏生物技术有限公司进行基因测序,结果在GenBank基因库中比对分析。
表1 PCR扩增耐药基因所用引物及扩增条件
Table 1 Primers for screening antimicrobial resistance genes and PCR conditions used in this study

参考文献strAstrA-FCTTGGTGATAACGGCAATTC54855[8] strA-RCCAATCGCAGATAGAAGGC strBstrB-FATCGTCAAGGGATTGAAACC50956[8] strB-RGGATCGTAGAACATATTGGC ant(6)ant6-FACTGGCTTAATCAATTTGGG59753[8] ant6-RGCCTTTCCGCCACCTCACCG qnrAqnrA-FTCAGCAAGAGGATTTCTCA62748[18] qnrA-RGGCAGCACTATTACTCCCA qnrBqnrB-FATGACGCCATTACTGTATAA56253[18] qnrB-RGATCGCAATGTGTGAAGTTT qnrSqnrS-FACCTTCACCGCTTGCACATT57157[18] qnrS-RCCAGTGCTTCGAGAATCAGT aac(6')-aph(2'')基因引物序列(5’→3’)扩增产物长度/bp退火温度/℃aac-FCCAAGAGCAATAAGGGCATACC67558[8] aac-RACCCTCAAAAACTGTTGTTGC tetMtetM-FGTGGACAAAGGTACAACGAG40655[19] tetM-RCGGTAAAGTTCGTCACACAC tetStetS-FCATAGACAAGCCGTTGACC66755[19] tetS-RATGTTTTTGGAACGCCAGAG vanAvanA-FGCAAGTCAGGTGAAGATGG1 02962[15] vanA-RACCTCGCCAACAACTAACGC vanBvanB-FACCCTGTCTTTGTGAAGCCGGCAC45759[15] vanB-RCAAAAAAAGATCAACACGAGCAAGCCC sulⅠsulⅠ-FTCACCGAGGACTCCTTCTTC33154[20] sulⅠ-RCAGTCCGCCTCAGCAATATC sulⅡsulⅡ-FCCTGTTTCGTCCGACACAGA43554[20] sulⅡ-RGAAGCGCAGCCGCAATTCAT
2 结果与分析
2.1 酸奶样品乳酸菌的分离与鉴定
本研究共采集市售酸奶样品25 份,每份样品对应的平板上均有细菌生长。从MRS和MC平板上挑取单菌落进行革兰氏染色,结果显示菌体呈紫色,可判定为革兰氏阳性。在显微镜下可观察到乳杆菌属的菌体形态多样,呈长杆状、弯曲杆状和短杆状;嗜热链球菌的菌体呈球形。通过生化鉴定和16S rDNA序列比对可知,分离得到的56 株乳酸菌中26 株为德氏乳杆菌保加利亚亚种(Lactobacillus delbrueckii subsp. bulgaricus)、3 株为植物乳杆菌(Lactobacillus plantarum)、2 株为嗜酸乳杆菌(Lactobacillus acidophilus)、25 株为嗜热链球菌(Streptococcus thermophilus)。
2.2 药敏实验结果
3 株质控菌株对11 种抗生素的抑菌圈直径均在质控范围内,表明药敏实验结果可靠。56 株乳酸菌对11 种抗生素的药物敏感性结果见表2(本研究中耐药率为耐药和中介耐药的菌株所占的比例),31 株乳杆菌对链霉素的耐药率最高,达87.1%;其次分别为庆大霉素(80.6%)、环丙沙星(74.2%)、四环素(61.3%)和万古霉素(48.4%);对头孢噻肟最敏感,无耐药菌株出现。25 株嗜热链球菌对链霉素的耐药率最高,达76.0%;其次分别为万古霉素(32.0%)、环丙沙星(32.0%)和四环素(20.0%)。
表2 乳酸菌药物敏感性结果
Table 2 Antimicrobial resistance phenotypes of LAB strains
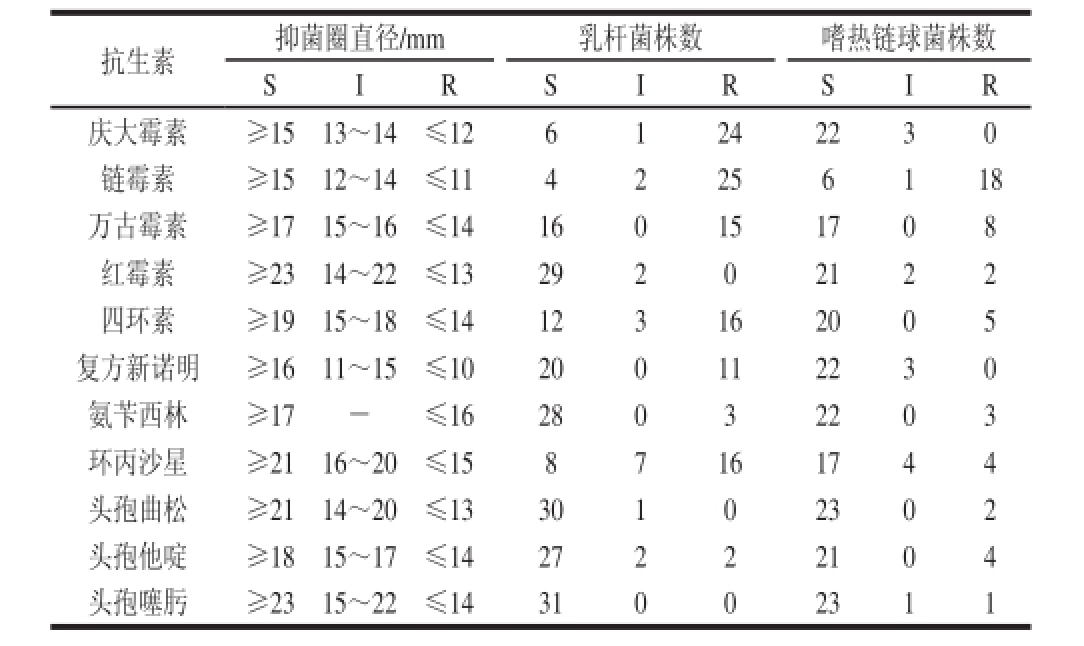
注:—. 无中介耐药状态。
抗生素抑菌圈直径/mm乳杆菌株数嗜热链球菌株数SIRSIRSIR庆大霉素≥1513~14≤1261242230链霉素≥1512~14≤1142256118万古霉素≥1715~16≤14160151708红霉素≥2314~22≤1329202122四环素≥1915~18≤14123162005复方新诺明≥1611~15≤10200112230氨苄西林≥17-≤1628032203环丙沙星≥2116~20≤1587161744头孢曲松≥2114~20≤1330102302头孢他啶≥1815~17≤1427222104头孢噻肟≥2315~22≤1431002311
总体而言,本研究分离得到的56 株乳酸菌对供试的抗生素均表现出不同程度的耐药性,其中对链霉素(82.1%)、环丙沙星(55.4%)、庆大霉素(50.0%)、四环素(42.9%)、万古霉素(41.1%)和复方新诺明(25.0%)的耐药情况较为严重;头孢噻肟和头孢曲松的对受试菌株表现出较好的抑菌效果(图1)。Zhou Ning等
[8]从我国多个地区采集的酸奶样品中分离得到43 株乳酸菌,药敏结果显示这些菌株对链霉素、庆大霉素、卡那霉素、四环素和氯霉素的耐药率较高。石磊等
[21]对广州市售酸奶中乳酸菌的耐药性进行调查,发现48 株乳酸菌对卡那霉素、链霉素、庆大霉素、万古霉素和环丙沙星的耐药水平较高。本研究中乳酸菌对链霉素、庆大霉素、万古霉素和环丙沙星的高耐药率与先前报道的结果相似。
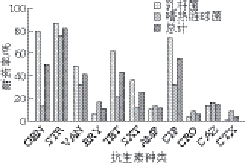
图1 分离菌株对11 种抗生素的耐药率
Fig. 1 Frequency of drug resistance in all isolates
2.3 耐药基因检测结果

图2 乳酸菌中耐药基因PCR扩增结果
Fig. 2 Amplifi cation of PCR products of antimicrobial resistance genes from LAB strains
如图2和表3所示,56 株乳酸菌中有13 株携带耐药基因,占受试菌株的23.2%。其中,1 株德氏乳杆菌保加利亚亚种携带链霉素耐药基因ant(6);2 株植物乳杆菌、1 株德氏乳杆菌保加利亚亚种和1 株嗜热链球菌携带庆大霉素耐药基因aac(6')-aph(2'');磺胺甲基异噁唑耐药基因sulⅠ在受试菌株中的检出率最高(8/56,14.3%),而sulⅡ基因仅在1 株嗜热链球菌中检出;3 株德氏乳杆菌保加利亚亚种携带四环素耐药基因tetM,而tetS基因在受试菌株中没有检出。所有菌株中均未检出环丙沙星和万古霉素相关耐药基因。
链霉素是一种氨基糖苷类抗生素,能够与细菌核糖体结合从而抑制蛋白质合成,最终导致细菌死亡。细菌对链霉素产生耐药的主要机制是通过产生多种作用于特定氨基或羟基的钝化酶,抑制药物与细菌核糖体结合,使药物无法干扰菌体蛋白质的合成过程,从而导致耐药。编码修饰链霉素钝化酶的耐药基因主要包括strA、strB、ant(6)和aadA,其中aadA主要分布于革兰氏阴性菌中。本研究中所有菌株均未检测到strA和strB,仅在1 株保加利亚乳杆菌中检出ant(6),这与先前的报道一致
[8]。
aac(6')-aph(2'')编码双功能酶AAC(6')-APH(2''),该酶具有乙酰转移酶和磷酸转移酶两种活性,可介导除链霉素外的大多数氨基糖苷类抗生素的高水平耐药。许多文献表明,aac(6')-aph(2'')基因与肠球菌对庆大霉素耐药具有高度相关性,并且该基因常位于耐药质粒上
[22]。
表3 乳酸菌中耐药基因的检出情况
Table 3 Determination of antimicrobial resistance genes from LAB strains

编号菌株名称耐药表型耐药基因1R-1Lactobacillus bulgaricusCIP-STR-SXT-TETsulⅠ、tetM 3R-3Lactobacillus plantarumAMP-CAZ-GEN-SXT-VANaac(6')-aph(2'')、sulⅠ7R-3Lactobacillus acidophilusCIP-GEN-SXTsulⅠ8R-3Lactobacillus bulgaricusCIP-GEN-STR-TETaac(6')-aph(2'') 9R-2Lactobacillus bulgaricusAMP-CIP-GEN-STR-SXT-VANsulⅠ10R-2Lactobacillus bulgaricusCIP-SXT-TETsulⅠ、tetM 16RLactobacillus bulgaricusCIP-GEN-STRant(6) 21R-2Lactobacillus plantarumCIP-GEN-STR-VANaac(6')-aph(2'') 25RLactobacillus bulgaricusCIP-TET-VANtetM 4S-5Streptococcus thermophilusSTR-SXTsulⅠ6S1Streptococcus thermophilusCIP-SXTsulⅠ7S1Streptococcus thermophilusCIP-GEN-STRaac(6')-aph(2'') 24SStreptococcus thermophilusSTR-SXT-TETsulⅠ、sulⅡ
磺胺类耐药基因编码二氢叶酸合成酶,介导细菌对此类药物的耐药。本研究中sulⅠ的阳性率最高,并且有一株嗜热链球菌同时携带sulⅠ和sulⅡ。研究表明,磺胺耐药基因多位于可移动遗传元件上,如整合子、质粒等,能够通过水平转移在细菌种内甚至种间传播,从而加速耐药以及多重耐药菌株的出现
[23]。
目前已发现的四环素耐药基因达40多种,其中tetM和tetS在革兰氏阳性菌中分布较为广泛,而在本研究中未检出tetS基因。Nawaz等
[24]在22 株乳酸菌中检出tetM阳性菌株8 株,tetS阳性菌株3 株;Zhou Ning等
[8]对23 株乳酸菌四环素耐药菌株进行研究,发现有3 株携带tetM,未检出其他种类的四环素耐药基因。以上研究表明,在乳酸菌四环素耐药菌株中tetM基因的分布可能更为普遍。报道显示tet基因常与质粒、转座子、接合型转座子等可移动成分相连,通过质粒和转座子在菌群之间进行转移
[25]。
本研究中,菌株携带的耐药基因与耐药表型之间呈现对应关系。例如,保加利亚乳杆菌1R-1和10R-2均携带sulⅠ和tetM基因,相应的这两株菌均对复方新诺明和四环素表现耐药;植物乳杆菌3R-3携带aac(6')-aph(2'')和sulⅠ基因,该菌株对庆大霉素和复方新诺明耐药。值得注意的是,在部分耐药菌株中并没有检出相应的耐药基因,表明在这些耐药菌株中可能还存在其他的耐药机制。本研究中,菌株对环丙沙星和万古霉素耐药率较高,但是扩增结果显示在环丙沙星和万古霉素耐药菌株中均未检出相关耐药基因。环丙沙星为第3代喹诺酮类抗菌药物,目前普遍认为细菌对此类药物的耐药机制主要有3 种,即药物靶蛋白突变、主动外排系统表达增强及质粒介导喹诺酮耐药
[18]。乳酸菌环丙沙星耐药菌株中未检出质粒介导的喹诺酮耐药基因,表明这些菌株的耐药性可能由其他机制介导。万古霉素为糖肽类抗生素,主要通过抑制细胞壁的合成实现杀菌作用。细菌对万古霉素的耐药机制包括基因突变导致细胞壁变厚或成分改变和耐药基因的获得。研究显示,万古霉素耐药基因vanA和vanB能够在肠球菌和葡萄球菌之间发生转移,从而介导细菌对万古霉素的高水平耐药
[26]。而在本研究中,所有万古霉素耐药菌株均未携带vanA和vanB,说明这些菌株的耐药不是由这两个基因所介导的。
56 株乳酸菌中共检出5 种不同的耐药基因,其中有4 株菌株携带两种不同的耐药基因。已有体内实验证明,某些耐药基因能够在肠道正常菌之间以及肠道正常菌与致病菌之间发生转移
[15],而食品链就是耐药基因在肠道内传播的主要途径。虽然大部分与食品有关的乳酸菌都已经获得相对安全认证,但是它们仍然存在潜在的安全隐患。Nawaz等
[24]在分离自发酵食品的乳酸菌中检测到多种耐药基因的存在,并通过接合转移实验证明红霉素耐药基因emrB和四环素耐药基因tetM能够从乳杆菌转移至受体菌Enterococcus faecalis 181。我国学者也对杭州地区发酵乳制品中乳酸菌发生耐药传播的安全性进行了评估,结果显示18.8%的乳酸菌能够通过质粒接合将耐药基因转移至受体菌Escherichia coli J53
[27]。这些研究表明分离自发酵食品的乳酸菌很有可能会扮演耐药基因贮存宿主的角色,并通过食品链将耐药性转移到肠道其他正常菌群或者致病菌中,造成耐药性的传播
[28]。本研究中乳酸菌携带的耐药基因能否在不同种属的细菌间发生转移则需要进一步的深入研究。
3 结 论
本研究从25 份酸奶样品中分离得到56 株乳酸菌,包括31 株乳杆菌和25 株嗜热链球菌。药敏结果显示,这些菌株对链霉素、庆大霉素、环丙沙星和四环素耐药率较高。56 株乳酸菌中共检出5 种不同的耐药基因,即链霉素耐药基因ant(6)、庆大霉素耐药基因aac(6')-aph(2'')、四环素耐药基因tetM以及磺胺类耐药基因sulⅠ和sulⅡ。本研究中多种耐药基因的存在表明用于发酵食品的乳酸菌可能成为潜在的耐药基因贮存库。由于这些耐药基因多位于可移动遗传元件上,这就意味着它们可能会转移到肠道中的其他正常菌群或者致病菌中,进而造成耐药性的传播,最终威胁人类健康。因此,应当加强对乳酸菌等食品工业用菌的耐药监测,合理评估其安全性,保证食品质量与安全。
参考文献:
[1] BOURDICHON F, CASAREGOLA S, FORROKH C, et al. Food fermentations: microorganisms with technological beneficial use[J]. International Journal of Food Microbiology, 2012, 154: 87-97. DOI:10.1016/j.ijfoodmicro.2011.12.030.
[2] MATHUR S, SINGH R. Antibiotic resistance in food lactic acid bacteria: a review[J]. International Journal of Food Microbiology, 2005, 105: 281-295. DOI:10.1016/j.ijfoodmicro.2005.03.008.
[3] DALLAL M M S, DOYLE M P, REZADEHBASHI M, et al. Prevalence and antimicrobial resistance profiles of Salmonella serotypes, Campylobacter and Yersinia spp. isolated from retail chicken and beef, Tehran, Iran[J]. Food Control, 2010, 21: 388-392. DOI:10.1016/j.foodcont.2009.06.001.
[4] GOUSIA P, ECONOMOU V, SAKKAS H, et al. Antimicrobial resistance of major fooborne pathogens from major meat products[J]. Foodborne Pathogens and Disease, 2011, 8: 27-38. DOI:10.1089/ fpd.2010.0577.
[5] BOK E, MAZUREK J, STOSIK M, et al. Prevalence of virulence determinants and antimicrobial resistance among commensal Escherichia coli derived from dairy and beef cattle[J]. International Journal of Environmental Research and Public Health, 2015, 12: 970-985. DOI:10.3390/ijerph120100970.
[6] D’AIMMO M R, MODESTO M, BIAVATI B. Antibiotic resistance of lactic acid bacteria and Bifidobacterium spp. isolated from dairy and pharmaceutical products[J]. International Journal of Food Microbiology, 2007, 115: 35-42. DOI:10.1016/ j.ijfoodmicro.2006.10.003.
[7] FRAQUEZA M J. Antibiotic resistance of lactic acid bacteria isolated from dry-fermented sausages[J]. International Journal of Food Microbiology, 2015, 212: 76-88. DOI:10.1016/ j.ijfoodmicro.2015.04.035.
[8] ZHOU Ning, ZHANG Jianxin, FAN Mingtao, et al. Antibiotic resistance of lactic acid bacteria isolated from Chinese yogurts[J]. Journal of Dairy Science, 2012, 95: 4775-4783. DOI:10.3168/jds.2011-5271.
[9] ZHANG Hongmei, XIE Lisi, ZHANG Wenyan, et al. The association of biofi lm formation with antibiotic resistance in lactic acid bacteria from fermented foods[J]. Journal of Food Safety, 2013, 33: 114-120. DOI:10.1111/jfs.12030.
[10] 张灼阳, 刘畅, 郭晓奎. 乳酸菌耐药性的研究进展[J]. 中国微生态学杂志, 2007, 19(5): 478-480. DOI:10.13381/j.cnki.cjm.2007.05.015.
[11] HUMMEL A S, HERTEL C, HOLZAPTEL W H, et al. Antibiotic resistances of starter and probiotic strains of lactic acid bacteria[J]. Applied and Environmental Microbiology, 2007, 73: 730-739. DOI:10.1128/AEM.02105-06.
[12] TOOMEY N, BOLTON D, FANNING S. Characterisation and transferability of antibiotic resistance genes from lactic acid bacteria isolated from Irish pork and beef abattoirs[J]. Research in Microbiology, 2010, 161: 127-135. DOI:10.1016/ j.resmic.2009.12.010.
[13] THUMU S C R, HALAMI P M. Presence of erythromycin and tetracycline resistance genes in lactic acid bacteria from fermented foods of Indian origin[J]. Antonie van Leeuwenhoek, 2012, 102: 541-551. DOI:10.1007/s10482-012-9749-4.
[14] 秦宇轩, 李晶, 王秋涯, 等. 市售酸奶中乳酸菌的鉴定与耐药性[J].微生物学学报, 2013, 53(8): 889-897. DOI:10.13343/j.cnki. wsxb.2013.08.010.
[15] MIELE A, BANDERA M, GOLDSTEIN B P. Use of primers selective for vancomycin resistance genes to determine van genotype in enterococci and to study gene organization in VanA isolates[J]. Antimicrobial Agents and Chemotherapy, 1995, 39(8): 1772-1778. DOI:10.1128/aac.39.8.1772.
[16] MORENO C, ROMERO J, ESPEJO R T. Polymorphism in repeated 16S rRNA genes is a common property of type strains and environmental isolates of the genus Vibrio[J]. Microbiology, 2002, 148: 1233-1239. DOI:10.1099/00221287-148-4-1233.
[17] Clinical and Laboratory Standards Institute. Performance Standards for Antimicrobial Susceptibility Testing; 25th Informational Supplement, M100-S25[S]. Wayne, PA: Clinical and Laboratory Standards Institute, 2015.
[18] JIANG X, YU T, WU N, et al. Detection of qnr, aac(6')-Ib-cr and qepA genes in Escherichia coli isolated from cooked meat products in Henan, China[J]. International Journal of Food Microbiology, 2014, 187: 22-25. DOI:10.1016/j.ijfoodmicro.2014.06.026.
[19] NG L K, MARTIN I, ALFO M, et al. Multiplex PCR for the detection of tetracycline resistance genes[J]. Molecular and Cellular Probes, 2001, 15: 209-215. DOI:10.1006/mcpr.2001.0363.
[20] CHEN S, ZHAO S, WHITE D G, et al. Characterization of multipleantimicrobial-resistant Salmonella serovars isolated from retail meats[J]. Applied and Environmental Microbiology, 2004, 70: 1-7. DOI:10.1128/aem.70.1.1-7.2004.
[21] 石磊, 李艳莉, 梁暖意, 等. 广州市售酸奶中乳酸菌的耐药性评估[J]. 现代食品科技, 2014, 30(10): 250. DOI:10.13982/ j.mfst.1673-9078.2014.10.041.
[22] 冯霞, 王蓉, 单斌, 等. 高耐庆大霉素肠球菌多重耐药性与aac(6')-aph(2'')基因的相关性研究[J]. 实用检验医师杂志, 2011, 3(4): 210-214. DOI:10.3969/j.issn.1674-7151.2011.04.005.
[23] ENNE V I, LIVERMORE D M, STEPHENS P, et al. Persistence of sulphonamide resistance in Escherichia coli in the UK despite national prescribing restriction[J]. Lancet, 2001, 357: 1325-1328. DOI:10.1016/ S0140-6736(00)04519-0.
[24] NAWAZ M, WANG J, ZHOU A, et al. Characterization and transfer of antibiotic resistance in lactic acid bacteria from fermented food products[J]. Current Microbiology, 2011, 62: 1081-1089. DOI:10.1007/s00284-010-9856-2.
[25] KOO H, WOO G. Distribution and transferability of tetracycline resistance determinants in Escherichia coli isolated from meat and meat products[J]. International Journal of Food Microbiology, 2011, 145: 407-413. DOI:10.1016/j.ijfoodmicro.2011.01.003.
[26] 王敬华. 携带vanA基因万古霉素耐药金黄色葡萄球菌研究进展[J]. 国际检验医学杂志, 2011, 32(6): 658-660. DOI:10.3969/ j.issn.1673-4130.2011.06.020.
[27] 刘传杰, 谈千千, 朱凯妮, 等. 杭州地区发酵乳制品中乳酸菌发生耐药传播的安全性研究[J]. 中国微生态学杂志, 2014, 26(5): 526-533. DOI:10.13381/j.cnki.cjm.201405008.
[28] van REENEN C A, DICKS L M T. Horizontal gene transfer amongst probiotic lactic acid bacteria and other intestinal microbiota: what are the possibilities? A review[J]. Archives of Microbiology, 2011, 193: 157-168. DOI:10.1007/s00203-010-0668-3.
Antimicrobial Resistance and Resistance Genes in Lactic Acid Bacteria Isolated from Commercial Yogurt
YU Tao
1, JIANG Xiaobing
2,*, LI Lei
1, WANG Hui
1, LU Shengzhe
1, ZHANG Mengmeng
1, QI Zhenping
1, YU Mingyue
1
(1. School of Life Science and Technology, Xinxiang University, Xinxiang 453000, China; 2. College of Life Sciences, Henan Normal University, Xinxiang 453007, China)
Abstract:In order to investigate the potential public health risk of resistance genes in lactic acid bacteria (LAB), LAB strains isolated from commercial yogurts were identified and investigated for antibiotic resistance profiles and the prevalence of streptomycin, gentamycin, sulfamethoxazole, and tetracycline resistance genes by PCR amplifi cation. A total of 56 LAB strains were isolated from 25 yogurt samples, including Lactobacillus delbrueckii subsp. bulgaricus (n = 26), Lactobacillus plantarum (n = 3), Lactobacillus acidophilus (n = 2), and Streptococcus thermophilus (n = 25). Antimicrobial susceptibility tests showed that 31 Lactobacillus strains were frequently resistant to streptomycin (87.1%), gentamycin (80.6%), ciprofl oxacin (74.2%), and tetracycline (61.3%), whereas cephalosporin exhibited good activity against the strains. 25 Streptococcus thermophilus also showed the highest resistance rate to streptomycin (76.0%), followed by vancomycin (32.0%), ciprofl oxacin (32.0%) and tetracycline (20.0%). Five different resistance genes were detected among the tested strains, including ant(6) (accounting for 1.8% of the strains), aac(6')-aph(2'') (7.1%), tetM (5.4%), sul I (14.3%), and sul II (1.8%). Of the 56 LAB strains, thirteen were positive for resistance genes, among which four strains harbored two different resistance genes, indicating LAB strains which have been recognized as safe and widely used in fermented foods for a long time may act as a reservoir of resistance genes.
Key words:yogurt; lactic acid bacteria; antimicrobial resistance; resistance gene
DOI:10.7506/spkx1002-6630-201611023
中图分类号:TS252.7
文献标志码:A
文章编号:1002-6630(2016)11-0131-06
引文格式:
于涛, 姜晓冰, 李磊, 等. 市售酸奶中乳酸菌耐药性及耐药基因的检测[J]. 食品科学, 2016, 37(11): 131-136. DOI:10.7506/spkx1002-6630-201611023. http://www.spkx.net.cn
YU Tao, JIANG Xiaobing, LI Lei, et al. Antimicrobial resistance and resistance genes in lactic acid bacteria isolated from commercial yogurt[J]. Food Science, 2016, 37(11): 131-136. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201611023. http://www.spkx.net.cn
收稿日期:2015-07-24
基金项目:河南省高等学校重点科研项目(15A180006);新乡市重点科技攻关计划项目(ZG15007);河南师范大学博士科研启动费支持课题;河南师范大学青年科学基金项目
作者简介:于涛(1977—),男,讲师,博士,研究方向为食品微生物污染与控制。E-mail:yutao@xxu.edu.cn
*通信作者:姜晓冰(1984—),女,讲师,博士,研究方向为食品微生物污染与控制。E-mail:jxb841001@163.com




