出入境动物源性食品中单增李斯特菌的多位点序列分型分析
刘二龙1,袁慕云2,邓建英3,幸 芳3,吕英姿1,蒋 原4,薛 峰4,邵景东4,许龙岩2,*
(1.黄埔出入境检验检疫局,广东 广州 510730;2.广东出入境检验检疫局,广东 广州 510623;3.广州市番禺质量技术监督检测所,广东 番禺 511400;4.江苏出入境检验检疫局,江苏 南京 210001)
摘 要:对出入境动物源性食品中分离的单增李斯特菌进行多位点序列分型(mutilocus sequence typing,MLST)分析,了解其序列型分布特点及不同菌株之间的亲缘关系。提取单增李斯特菌基因组DNA,选择其7 个管家基因进行聚合酶链式反应扩增并测序。将测序结果截成标准序列的长度后上传到MLST数据库进行比对分析,获得7 个管家基因的等位基因谱和序列分型编码,并将结果采用不加权算术平均组对(unweighted pair group method using arithmetic averages,UPGMA)法进行聚类分析。89 株单增李斯特菌共获得51 个STs,其中26 个为新获得的STs (STnew1~STnew26);数量最多的5 个STs为ST8(9.0%),ST121(9.0%)、ST7(5.6%)、ST87(5.6%)及新发现的STnew3(7.8%);其中ST456、ST34、ST343、ST19、ST517、ST201、ST98、ST330和ST73为在国内首次获得。采用UPGMA算法得到的进化树可将89 株菌株分为3 大类群,分类的结果与单增李斯特菌血清学家系分类结果一致。MLST结果对了解出入境动物源性食品中分离的单增李斯特菌的亲缘关系及流行病学溯源有重要意义。
关键词:单增李斯特菌;多位点序列分型;亲缘关系
引文格式:
刘二龙, 袁慕云, 邓建英, 等. 出入境动物源性食品中单增李斯特菌的多位点序列分型分析[J]. 食品科学, 2016, 37(12): 212-216. DOI:10.7506/spkx1002-6630-201612038. http://www.spkx.net.cn
LIU Erlong, YUAN Muyun, DENG Jianying, et al. Multilocus sequence typing of Listeria monocytogenes isolated from imported and exported animal-derived foods[J]. Food Science, 2016, 37(12): 212-216. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201612038. http://www.spkx.net.cn
单增李斯特菌(Listeria monocytogenes)是一种人畜共患病的病原菌,感染后主要表现为败血症、脑膜炎和单核细胞增多,可以通过污染奶及奶制品、蔬菜、水产品、肉制品等食物导致人群感染[1-2]。该菌在4 ℃的环境中仍可生长繁殖,是冷藏食品中威胁人类健康的主要病原菌之一[3-5]。单增李斯特菌有13 个不同的血清型,依据其可将单增李斯特菌分成Lineage Ⅰ、Lineage Ⅱ和Lineage Ⅲ 三大家系[6-7]。但单增李斯特氏菌的分子亚型有100多个,近年国外应用随机扩增多态性DNA、脉冲场凝胶电泳等方法对单增李斯特菌进行分型研究[8-11]。
随着测序费用的大幅降低及测序速度的加快,多位点序列分型(multilocus sequence typing,MLST)逐渐被应用于病菌原微生物流行病学研究[12]。MLST通过聚合酶链式反应(polymerase chain reaction,PCR)扩增7 个管家基因内部片段并测定其序列,经MLST数据库分析后获得管家基因的等位基因图谱及STs编码,可以获得菌株间精确的进化关系数据[13-14]。它由Maiden等[15]于1998年首次运用于脑膜炎奈瑟菌的分型,后来由于其结果精确并且数据易于实验室间传递和比较而广泛应用于其他致病菌、真菌以及一些非致病菌分子分型[16]。
本研究对进出境动物源性食品中分离的88 株单增李斯特氏菌及标准参考菌株ATCC19115,采用法国巴斯德研究所的单增李斯特菌MLST方案[17]进行MLST分型分析,初步探讨我国进出境动物源性食品中单增李斯特菌STs分布特点及菌株间亲缘关系。
1 材料与方法
1.1 材料
1.1.1 菌株来源
本研究共选用单增李斯特氏菌菌株89 株,其中88 株菌株为广东出入境检验检疫局技术中心和中国检验检疫科学研究院分离保存,另1 株为单增李斯特菌标准菌株ATCC19115。
1.1.2 试剂
dNTPs、高保真PrimerSTAR HS DNA酶、dNTPmix、5×PrimeSTAR缓冲液、DL2000 DNA Marker、琼脂糖 宝生物工程(大连)有限公司;Goldview、DNA提取试剂盒 北京天根生化科技有限公司;脑心浸液肉汤(brain heart infusion,BHI)培养基北京陆桥技术股份有限公司;Tris、乙二胺四乙酸 美国Sigma公司。
1.1.3 仪器与设备
CT15RE高速冷冻离心机 日本Hitachi公司;ND2000C微量分光光度计 美国Thermo Scientific公司;S1000 PCR仪、164-5050电泳仪 美国Bio-Rad公司;Tanon 4200凝胶成像系统 上海天能公司。
1.2 方法
1.2.1 菌株培养及基因组DNA提取
将保存于-80 ℃单增李斯特菌菌株接种于BHI肉汤中,36 ℃过夜培养。取培养后的单增李斯特菌菌液1 mL离心去培养基后,用DNA提取试剂盒提取基因组DNA作为PCR扩增反应的模板,-20 ℃保存备用。
1.2.2 MLST管家基因选择及PCR扩增
参考法国巴斯德研究所的单增李斯特菌多位点序列分型数据库操作方案[17],确定用于分型的7 个管家基因abcZ、bglA、cat、dapE、dat、ldh和lhkA进行PCR扩增[18],扩增的引物信息见表1,测序上游引物为GTTTTCCCAGTCACGACGTTGTA,下游引物为TTGTGAGCGGATAACAATTT。引物委托宝生物合成。
表1 用于MLST分析的单增李斯特菌7 个管家基因的引物信息
Table 1 Primers sequences in the MLST of Listeria monocytogenes

引物名称 引物序列(5’→3’) 截成标准序列的长度/bp abcZ-F GTTTTCCCAGTCACGACGTTGTATCGCTGCTGCCACTTTTATCCA 537 abcZ-R TTGTGAGCGGATAACAATTTCTCAAGGTCGCCGTTTAGAG bglA-F GTTTTCCCAGTCACGACGTTGTAGCCGACTTTTTATGGGGTGGAG 399 bglA-R TTGTGAGCGGATAACAATTTCCGATTAAATACGGTGCGGACATA catF GTTTTCCCAGTCACGACGTTGTAATTGGCGCATTTTGATAGAGA 486 catR TTGTGAGCGGATAACAATTTCAGATTGACGATTCCTGCTTTTG dapE-F GTTTTCCCAGTCACGACGTTGTACGACTAATGGGCATGAAGAACAAG 462 dapE-R TTGTGAGCGGATAACAATTTCATCGAACTATGGGCATTTTTACC datF GTTTTCCCAGTCACGACGTTGTAGAAAGAGAAGATGCCACAGTTGA 471 datR TTGTGAGCGGATAACAATTTCTGCGTCCATAATACACCATCTTT ldhF GTTTTCCCAGTCACGACGTTGTAGTATGATTGACATAGATAAAGA 453 ldhR TTGTGAGCGGATAACAATTTCTATAAATGTCGTTCATACCAT lhkA-F GTTTTCCCAGTCACGACGTTGTAAGAATGCCAACGACGAAACC 480 lhkA-R TTGTGAGCGGATAACAATTTCTGGGAAACATCAGCAATAAAC
PCR扩增反应体系为50 μL:5×PrimeSTAR PCR缓冲液10 μL,dNTPmix(2.5 mmol/L)4 μL,上、下游引物(10 μmol/L)各1 μL,DNA聚合酶(5 U/μL)0.5 μL,DNA模版1.5 μL,超纯水32 μL。abcZ、cat、dapE、dat和ldh基因的反应条件为:98 ℃变性10 s,52 ℃退火12 s,72 ℃延伸1 min,35 个循环。lhkA基因的反应条件为:98 ℃变性10 s,52 ℃退火18 s,72 ℃延伸1 min,35 个循环。bglA基因的反应条件为:98 ℃变性10 s,45 ℃退火15 s,72 ℃延伸1 min,35 个循环。
1.2.3 PCR产物的检测、测序、序列拼接
扩增产物阳性者,委托Invitrogen纯化并进行双向测序。利用DNAStar中的Sequence模块进行双向测序序列的拼接,并截齐成标准长度(abcZ,537 bp;bglA,399 bp;cat,486 bp;dapE,462 bp;dat,471 bp;ldh,453 bp;lhkA,480 bp)。
1.2.4 数据分析
依次将测序得到的7 个管家基因提交MLST网站[17],可以得到所有菌株的7 个管家基因的等位基因编号。将菌株所得的等位基因数据按abcZ、bglA、cat、dapE、dat、ldh和lhkA顺序提交,获得每株菌的等位基因图谱确定序列的STs,按顺序(abcZ-bglA-cat-dapE-dat-ldh-lhkA)将管家基因序列进行连接,利用MEGA4软件,按Construct Phylogeny的UPGMA算法,构建系统进化树,得到菌株间的亲缘关系。
2 结果与分析
2.1 7 个管家基因的PCR扩增结果
以单增李斯特菌标准菌株ATCC19115基因组DNA为模板,PCR扩增7 个管家基因。扩增片断大小与预期相符(图1)。
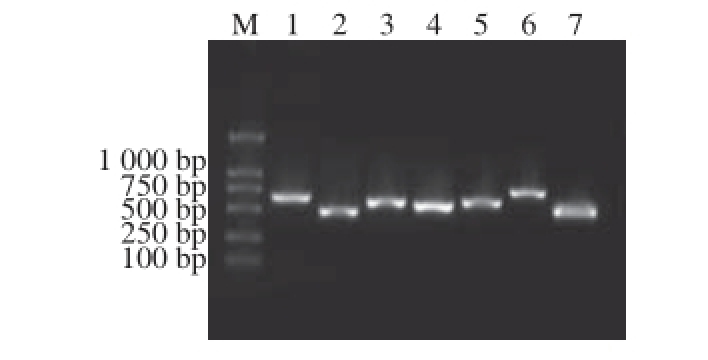
图1 管家基因电泳图
Fig. 1 Electrophoretogram of the PCR products of seven housekeep genes
M. Marker;1~7分别为ATCC19115菌株的abcZ、bglA、cat、dapE、dat、ldh和lhkA基因PCR扩增产物。
2.2 MLST中7 个管家基因的等位基因情况

图2 89 株单增李斯特菌中管家基因等位基因的数量
Fig. 2 Number of alleles in seven loci from all 89 strains of Listeria monocytogenes
管家基因等位基因值的数量反映该基因的变异程度,由图2可知,在89 株分离株中,等位基因数量最多的是ldh,为22 个,其次是dapE 16 个、bglA 15 个、cat 15 个、lhkA基因最稳定,等位基因为10 个,变化较小。
2.3 STs的分布情况
MLST基因分型分析发现,89 株单增李斯特菌共呈现51 个ST型别,其26 个为新发现的STs(暂命名为STnew1~STnew26)。将经MLST分型的单增李斯特菌分离株各STs的分布情况如图3所示:ST8、ST121、ST7、ST87及新发现的STnew3(暂命名)为优势STs,所占比例分别为:9.1%、9.1%、5.7%、5.7%和8%。本实验发现的新STs将提交MLST数据库管理员进行确认。
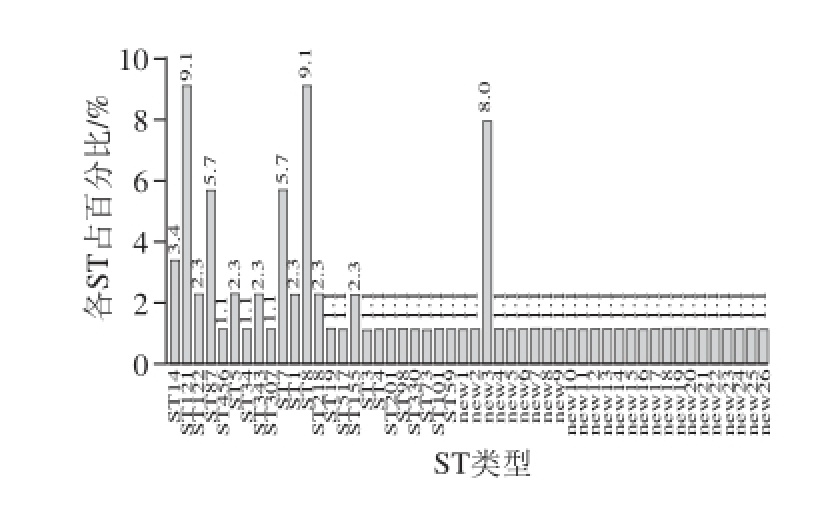
图3 89 株菌株各STs占比例
Fig. 3 Proportion of STs obtain from 89 strains of Listeria monocytogenes
2.4 UPGMA分析
将各菌株的等位基因序列按顺序(abcZ-bglA-catdapE-dat-ldh-lhkA)进行连接,利用MEGA4软件,按Construct Phylogeny的UPGMA算法进行聚类,得到菌株间的进化关系如图4所示。89 株实验菌株经UPGMA分析得到聚类可分为3 大类群Group Ⅰ、Group Ⅱ和Group Ⅲ。其中Group Ⅰ和Group Ⅱ中STs的分布分别与血清学分类的Lineage Ⅰ、Lineage Ⅱ中STs分布一致[18]。

图4 89 株单增李斯特菌UPGMA聚类分析
Fig. 4 UPGMA dendrogram for 89 strains of Listeria monocytogenes
3 讨 论
研究显示,食入被单增李斯特菌污染的食品是目前引起人类单增李斯特菌病暴发和散发的重要原因[19-20]。自1981年加拿大沿海省份的李斯特菌病的暴发证实单增李斯特菌可能通过食物在人群中传播以来[21],各国相继报道了多起李斯特菌病的暴发事件(其中美国1983—2011暴发6 起,加拿大2008年暴发1 起,欧洲1983—1992年暴发了5 起)[18],单李增斯特菌被世界卫生组织列为重点监测的食源性病原菌之一[22]。中国目前虽然未曾爆发人感染单增李斯特菌病,但多位研究者从各地多种食品中均分离出单增李斯特菌[23-27],其风险性不容忽视。
本实验采用Pasteur网站的MLST方案,对单增李斯特菌的7 个管家基因采用高保真的Taq酶进行PCR扩增,降低碱基的错配几率。此外对扩增体系退火延伸时间进行了优化(abcZ、cat、dapE、dat和ldh基因为52 ℃退火12 s,lhkA基因为52 ℃退火18 s,bglA基因为45℃退火15 s),达到了良好的扩增效果,无非特异性扩增条带且条带清晰(特别是对于lhkA基因),与之前报道的PCR扩增退火条件略有不同[28-29]。此外,本研究在合成引物时,在上游、下游引物的5’端分别加上相同的测序引物,使后序测序时引物准备的工作量减少且不易出错,也便于测序仪对于退火温度较低bglA基因顺利进行测序反应。
据文献[30-31]显示,全球已获得的单增李斯特菌STs主要为ST9、ST1、ST2、ST3、ST5、ST87、ST121、ST8、ST7、ST59、ST4、ST6、ST122、ST101、ST155、ST101、ST199。法国和瑞典在1995年曾暴发分子亚型为ST1的单增李斯特菌感染事件,1997年在意大利暴发的单增李斯特菌型别为ST2[19]。Wang Yan 等[18]对中国国内单增李斯特菌进行了MLST分型,共获得36 个STs,发现国内主要STs为ST9(29.1%)、ST8 (11.7%)、ST87(10%),并认为是国内潜在可能传播和暴发的STs型别。马爱静等[28]调查了北京市一些地区生肉标本中单增李斯特菌的分子流行病学特征,将分离到的21 株单增李斯特菌经MLST得到7 个STs,其中ST9数量最多(10 株)。刘萍萍等[29]将由动物源性食品中分离到的33 株单增李斯特菌株通过MLST得到8 个STs,其中属于ST121型别菌株最多,所占比例为45.5%。
本研究从我国出入境口岸动物源性食品中分离单增李斯特菌共获得51个STs,其中26个为新STs(暂命名为STnew1-STnew26,尚需递交MLST网站确认),ST8、ST121、ST7、ST87及新发现的STnew3为优势STs,所占比例分别为:9.0%、9.0%、5.6%、5.6%和7.8%。此外,本实验获得了国内尚无报道的9种STs:ST456、ST34、ST343、ST19、ST517、ST201、ST98、ST330、ST73。
按传统的血清学分类,单增李斯特菌分为3 大家系:LineageⅠ、LineageⅡ和Lineage Ⅲ(最近有文献报道发现Lineage Ⅳ)[32]。本实验将89 株单增李斯特菌7 个管家基因序列连接后根据UPGMA算法进行聚类分析,可分成3 大类:GroupⅠ、GroupⅡ、Group Ⅲ。其中GroupⅠ内分布的STs均属LineageⅠ、GroupⅡ内分布的STs均属LineageⅡ,分类的结果与血清学分类一致。
随着国际贸易的日益频繁,单增李斯特菌可能随着进出境动物源性食品贸易而进行传播。本实验通过MLST分析获得的优势STs如ST8、ST121、ST7和STnew3可能成为潜在传播和暴发单增李斯特菌病的分子亚型。本实验通过对出入境口岸动物源性食品中分离的单增李斯特氏菌进行的MLST分析,了解其STs分布情况及菌株间亲缘关系,这对于单增李斯特菌病的临床诊断,流行病学溯源、疾病的防控有着重要意义。
参考文献:
[1] Centers for Disease C(2008) Prevention. Outbreak of Listeria monocytogenes infections associated with pasteurized milk from a local dairy-Massaschusetts[J]. Morbidity and Mortality Weekly Report, 2008, 57(40): 1097-1100.
[2] 李爱华, 叶长芸. 单核细胞增生性李斯特菌致病相关机制的研究进展[J]. 疾病监测, 2011, 26(11): 914-919.
[3] SCHLECH W F. Foodborne listeriosis[J]. Clinical Infectious Diseases, 2000, 31(3): 770-775. DOI:10.1086/314008.
[4] VAZQUEZ-BOLAND J A, KUHN M, BERCHE P, et al. Listerian pathogenesis and molecular virulence determinants[J]. Clinic Microbiology Reviews, 2001, 14(3): 583-640. DOI:10.1128/ CMR.14.3.584-640.2001.
[5] BODRO M, PATERSO D L. Listeriosis in patients receiving biologic therapies[J]. European Journal of Clinical Microbiology and Infectious Diseases, 2013, 32(9): 1225-1230. DOI:10.1007/s10096-013-1873-1.
[6] WIEDMANN M, BRUCE J L, KEATING C, et al. Ribotypes and virulence gene polymorphisms suggest three distinct Listeria monocytogenes lineages with differences in pathogenic potential[J]. Infection and Immunity, 1997, 65: 2707-2716.
[7] WARD T J, GORSKI L, BORUCKI M K, et al. Intraspecific phylogeny and lineage group identification based on the prfA virulence gene cluster of Listeria monocytogenes[J]. Journal of Bacteriology, 2004, 186: 4994-5002. DOI:10.1128/JB.186.15.4994-5002.2004.
[8] 曾海燕. 基于ActA基因的单核细胞增多性李斯特菌的序列分型研究[J]. 青海大学学报(自然科学版), 2006, 24(3): 29-33. DOI:10.3969/ j.issn.1006-8996.2006.03.009.
[9] 郭明星, 陈晨, 赵晖, 等. 湖北省水产品中单增李斯特菌基因分型研究[J]. 中国人兽共患病学报, 2008, 24(9): 855-858. DOI:10.3969/ j.issn.1002-2694.2008.09.014.
[10] GRAVES L M, SWANINATHAN B. PulseNet standardized protocol for subtyping Listeria monocytogenes by macrorestriction and pulsedfield gel electrophoresis[J]. International Journal of Food Microbiology, 2001, 65: 55-62. DOI:10.1016/S0168-1605(00)00501-8.
[11] 贺春月, 王艳, 王天姝, 等. 中国部分食品来源单增李斯特菌中致病相关基因的分布研究[J]. 疾病监测, 2013, 28(5): 384-388. DOI:10.3784/j.issn.1003-9961.2013.5.014.
[12] 王中强, 邱少富, 王勇, 等. 多位点序列分型技术及其研究进展[J]. 军事医学科学院院刊, 2010(1): 76-79. DOI:10.3969/ j.issn.1674-9960.2010.01.021.
[13] CHEN Ti. DNA sequence-based subtyping of epidemic clones and outbreak clones of Listeria monocytogems[D]. USA, Pennsylvania State: The Pennsylvania State University, 2007.
[14] SALCEDO C, ARREAZA L, ALCALA B, et al. Development of a multilocus sequence typing method for analysis of Listeria monocytogenes clones[J]. Journal of Clinical Microbiology, 2003, 41(2): 757-762. DOI:10.1128/JCM.41.2.757-762.2003.
[15] MAIDEN M C J, BYGRAYES J A, FEIL E, et al. Multilocus sequence typing: a portable approach to the identification of clones within populations of pathogenic microorganisms[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(6): 3140-3145. DOI:10.1073/pnas.95.6.3140.
[16] ENRIGHT M C, SPRATT B G. A multilocus sequence typing scheme for Streptococcus pneumoniae: identification of clones associated withserious invasive disease[J]. Microbial, 1998, 144(11): 3049-3060. DOI:10.1099/00221287-144-11-3049.
[17] Primers used for MLST of Listeria monocytogenes[DB/OL]. http:// bigsdb.web.pasteur.fr/listeria/primers_used.html.
[18] WANG Yan, ZHAO Ailan, ZHU Renfa, et al. Genetic diversity and molecular typing of Listeria monocytogenes in China[J]. British Medical Council Microbiology, 2012, 12(1): 119. DOI:10.1186/1471-2180-12-119.
[19] HOELZER K, POUILLOT R, DENNIS S. Listeria monocytogenes growth dynamics on produce: a review of the available data for predictive modeling[J]. Foodborne Pathogens and Disease, 2012, 9: 661-673. DOI:10.1089/fpd.2011.1087.
[20] VISWANATH P, MURUGESANL, KNABEL S, et al. Incidence of Listeria monocytogenes and Listeria spp. in a small-scale mushroom production facility[J]. Foodborne Pathogens and Disease, 2013, 9(7): 661-673. DOI:10.4315/0362-028X.JFP-12-292.
[21] FLEMING D W, COCHI S L, MACDONALD K L, et al. Pasteurized milk as a vehicle of infection in an outbreak of listeriosis[J]. The New England Journal of Medicine, 1985, 312: 404-407.
[22] 炊慧霞, 李文杰, 张秀丽, 等. 2012年河南省食源性单核细胞增生李斯特菌血清分型及耐药性分析[J]. 中国卫生检验杂志, 2014(12): 1800-1803.
[23] 田静. 熟肉制品中单增李斯特菌的风险评估及风险管理措施的研究[D]. 北京: 中国疾病预防控制中心, 2010: 21; 24.
[24] 张丽萍, 高涛, 张克检, 等. 宝鸡市单核细胞增生性李斯特菌污染状况监测结果分析[J]. 实用预防医学, 2012, 19(5): 710-711. DOI:10.3969/j.issn.1006-3110.2012.05.025.
[25] 靳晓燕, 韩军, 于宏伟, 等. 食品中单核增生性李斯特菌(Listeria monocytogenes)污染状况研究[J]. 中国食品学报, 2009, 9(1): 226-231. DOI:10.3969/j.issn.1009-7848.2009.01.038.
[26] 方叶珍, 包芳珍, 徐丹戈. 江干区部分食品中李斯特菌污染状况分析[J]. 中国食品卫生杂志, 2009, 21(2): 142-143.
[27] 蒋兴祥, 赵霞贊, 何停停. 生鸡肉中单核细胞增生李斯特菌的污染监测[J]. 中国卫生检验杂志, 2007, 17(1): 119; 147. DOI:10.3969/ j.issn.1004-8685.2007.01.052.
[28] 马爱静, 王艳, 王毅, 等. 北京市一些地区生肉标本中单增李斯特菌的分离及其分子流行病学特征分析[J]. 中国人兽共患病学报, 2015, 31(5): 403-407. DOI:10.3969/cjz.j.issn.1002-2694.2015.05.003.
[29] 刘萍萍, 王少辉, 赵秋华, 等. 上海市动物源性食品中单增李斯特菌的MLST分析[J]. 中国动物传染病学报, 2013, 21(4): 18-22. DOI:10.3969/j.issn.1674-6422.2013.04.004.
[30] RAGON M, WIRTH T, HOLLANDT F, et al. A new perspective on Listeria monocytogenes evolution[J]. PLoS Pathog, 2008, 4: e1000146. DOI:10.1371/journal.ppat.1000146.
[31] VIVIANE C F, JODIE L, THOMAS C, et al. Worldwide distribution of major clones of Listeria monocytogenes[J]. Emerging Infectious Diseases, 2011, 17: 1110-1112. DOI:10.3201/eid/1706.101778.
[32] ORSI R H, BAKKER H C, WIEDMANN M. Listeria monocytogenes lineages: genomics, evolution, ecology, and phenotypic characteristics[J]. International Journal of Medical Microbiology, 2010, 301: 79-96. DOI:10.1016/j.ijmm.2010.05.002.
Multilocus Sequence Typing of Listeria monocytogenes Isolated from Imported and Exported Animal-Derived Foods
LIU Erlong1, YUAN Muyun2, DENG Jianying3, XING Fang3, LÜ Yingzi1, JIANG Yuan4, XUE Feng4, SHAO Jingdong4, XU Longyan2,* (1. Huangpu Entry-Exit Inspection and Quarantine Bureau, Guangzhou 510730, China; 2. Guangdong Entry-Exit Inspection and Quarantine Bureau, Guangzhou 510623, China;
3. Guangzhou Panyu Quality and Technology Supervision and Testing Institute, Guangzhou 511400, China;
4. Jiangsu Entry-Exit Inspection and Quarantine Bureau, Nanjing 210001, China)
Abstract: This study was devised to understand the sequence types (STs) and the genetic and evolutionary relationship of different Listeria monocytogenes isolated from imported and exported animal-derived food samples. Seven house-keeping genes were selected as target genes for PCR amplification. Sequencing, analysis and comparison of the 7 housekeeping genes were conducted. Their alleles and STs codes were obtained, and their evolutionary relationships were analyzed by unweighted pair group method using arithmetic average (UPGMA). A total of 51 STs were obtained from 89 strains by multilocus sequence typing (MLST), among which 26 were novel. The 5 most common STs were ST8 (9.0%), ST121 (9.0%), ST7 (5.6%) and ST87 (5.6%) as well as the novel STnew 3 (7.8%). ST456, ST34, ST343, ST19, ST517, ST201, ST98, ST330 and ST73 were found for the first time in China. The evolutionary relationship analyzed by UPGMA method showed that the isolates were grouped into 3 clusters, which were consistent with three lineages classified by serotype of L. monocytogenes. MLST is a useful method to evaluate genetic evolution. This method is applicable to study the population structure and epidemiological traceability of L. monocytogene isolates from imported and exported animal-derived food samples.
Key words: Listeria monocytogenes; mutilocus sequence typing (MLST); evolutionary relationship
收稿日期:2015-08-29
基金项目:广东省科技计划项目(2013B040402004)
作者简介:刘二龙(1978—),男,工程师,硕士,研究方向为微生物与分子生物学。E-mail:erlongliu@126.com
*通信作者:许龙岩(1970—),男,研究员,硕士,研究方向为微生物与分子生物学。E-mail:xlyciq@126.com
DOI:10.7506/spkx1002-6630-201612038
中图分类号:R377
文献标志码:A
文章编号:1002-6630(2016)12-0212-05




