荧光PCR和数字PCR法检测转基因DAS-44406-6品系大豆
于晓帆
1,高宏伟
2,*,孙 敏
2,肖西志
2,李荣贵
1,*
(1.青岛大学生命科学学院,山东 青岛 266071;2.山东出入境检验检疫局检验检疫技术中心,山东 青岛 266002)
摘 要:目的:建立实时荧光聚合酶链式反应(polymerase chain reaction,PCR)检测转基因DAS-44406-6品系大豆的定性检测方法和使用数字PCR检测转基因DAS-44406-6品系大豆的定量检测方法。方法:针对转基因DAS-44406-6大豆品系,进行5’-RACE,测定该品系转基因大豆外源片段与大豆染色体重组的边界序列,并根据该边界序列设计引物和探针。使用23 种非DAS-44406-6品系转基因植物作为阴性对照测试实时荧光PCR引物和探针的特异性,以DAS-44406-6品系样品制备6 个含量梯度的样品进行检测低限实验。使用数字PCR技术进行定量检测,并确定定量检测的低限。结果:建立的转基因DAS-44406-6大豆品系的实时荧光PCR特异性检测方法品系鉴定特异性较强,实时荧光PCR检测方法的检测低限在模板DNA浓度为100 ng/反应时,为0.01%的转基因大豆含量,约为16.6 个拷贝的DAS-44406-6基因组DNA;数字PCR检测方法的检测低限在模板DNA浓度为0.5 ng/反应、转基因大豆含量为1%时,相对标准偏差为0.7%。因此,建立的转基因DAS-44406-6大豆品系实时荧光PCR和数字PCR特异性检测方法符合转基因检测的要求。
关键词:转基因大豆;DAS-44406-6品系;品系鉴定;实时荧光PCR;数字PCR
转基因作物,是通过基因工程的方法在原有作物基因组中添加其他生物的基因,敲除对作物生长不利的基因,从而培育出性状更加优良的作物。根据国际农业生物技术应用服务组织报告,到2014年,全球转基因作物的种植面积为1.815亿hm
2。与1996年转基因作物的种植面积1 700万hm
2相比增幅约100 倍。2014年中国转基因作物种植面积达到390万hm
2[1]。
正是由于现在转基因食品大量出现于市场,因此越来越多的消费者开始注意到转基因食品的安全性问题
[2]。为保障公众的合法知情权,自2001年开始,世界各国都相继出台了关于标识转基因产品的标准法规,还设定了阀值
[3]。我国也相应地出台一系列法规条例,2001年6月6日,国务院颁布了《农业转基因生物安全管理条例》。2002年1月5日,农业部颁布了《农业转基因生物标识管理办法》等3 个相应法律法规条例,严格转基因标识准入制度从而把控进入中国市场的相关产品
[4]。只有对相应的转基因产品进行定性或定量的检测才能真正地贯彻标识制度,所以市场准入的关键就在于转基因检测
[5]。
转基因检测技术包括蛋白检测技术、核酸检测技术、酶活性检测方法、实时荧光聚合酶链式反应(polymerase chain reaction,PCR)技术、数字PCR技术等。转基因品系蛋白质检测技术包括试纸法、酶联免疫吸附测定法、蛋白质印迹法、蛋白质单向电泳、蛋白质双向电泳、Western杂交分析等。转基因品系核酸检测技术包括PCR(多重PCR、定性PCR、定量PCR、电化学发光PCR)、连接酶链式反应、指纹图谱法(限制性片段长度多态性、扩增片段长度多态性及随机扩增多态性DNA)等技术
[6]。成熟的实时荧光定量PCR技术通过对PCR扩增反应中每一个循环产物荧光信号的实时检测,可以定量及定性分析起始模板。添加一种荧光化学物质在本技术中,这样在反应中PCR产物不断增加,与此相对应的荧光信号的量也不断被检测到增长。仪器可以检测到每过一个循环荧光信号的变化量,最后得到一条荧光扩增曲线图。实时荧光定量PCR技术有着特异性强、可重复性好、定量分析较准确、PCR污染少、自动化程度高等传统PCR技术不具有的特点
[7]。转基因检测技术可以分为转基因筛选检测和转基因品系检测两个层次。转基因筛选检测是在一批样品中筛选区分出转基因品系和非转基因品系,转基因品系检测是根据相关检测标准确定出具体的转基因品系。
数字PCR(digital PCR,dPCR)是近些年来兴起的一项针对单分子目标DNA的定量分析技术。dPCR要求稀释核酸模板,然后分配到大量独立的反应室中使之具有单个模板分子并且发生扩增反应,扩增结束采用泊松概率分布公式等统计学公式分析单个反应室的阳性信号,来定量DNA拷贝数。由于dPCR是通过先扩增后定量的方法,因此可以不依赖扩增曲线的循环阈值(Ct值),也不需要采用内参基因和标准曲线,准确度高、重现性好,可以实现绝对定量分析。dPCR由于有相对于传统技术的明显优点,在医学诊断、转基因定量分析、单细胞基因表达分析、环境微生物检测和下一代基因测序等研究领域未来有较好的应用空间
[8-10]。
大豆是人类重要的油料作物,富含多种营养物质,可以作为粮食饲料以及营养品的来源,大豆油是中国需求最多的食用油,大豆加工产物豆粕是可以作为饲料添加物,因此大豆在我国粮食与饲料市场占有不可替代的重要地位。从1988年第一株转基因大豆问世至今,现在国际市场上转基因大豆种类可达30余种,囊括抗转基因除草剂、转基因高油酸、低亚麻酸、低棕榈酸、高硬脂酸、高棕榈酸、表达抗癌蛋白及富含脱敏因子等多种功能的转基因大豆品种。其中,孟山都公司研发的抗除草剂转基因大豆GTS40-3-2品系在全球近半数国家获得批准进行商业化种植,类似的还有拜尔公司的A2704-12抗除草剂品系等
[1]。
转基因大豆品种DAS-44406-6是美国陶氏益农公司研发的能耐受草铵膦、草甘膦还有2,4-二氯苯氧乙酸(2,4-滴)除草剂3 种成分的转基因大豆品系。其中含有的主要外来基因为来源于食酸戴尔福特菌的aad-12基因、来源于玉米的2mepsps基因和来源于绿色产色链霉菌的pat基因
[11]。自从2013年加拿大批准转基因种植以来,截止到2015年10月30日,已经先后有美国、日本、阿根廷等国家商业化种植,加拿大、南非、新西兰、澳大利亚、美国、墨西哥、日本、韩国、中国台湾等国家和地区批准用于食品和饲料的食用和加工。本研究通过建立DAS-44406-6特异性检测方法,从而对该品系进行有效地检测和监管,便于转基因标识制度的实施
[11]。
1 材料与方法
1.1 材料与试剂
本实验样品包括转基因DAS-44406-6大豆和23 个阴性对照样品,样品来源见表1。
GenoDNA Plant Mini Kit-Genomic DNA Isolation kit试剂盒 长沙Ambiogen公司;RealMaster Mix(Probe)试剂盒 北京Tiangen公司;特异性引物、探针由上海生物工程有限公司合成;LA Taq(DRR02AG)、2×GC Buffer(DRR02AG) 大连宝生物工程有限公司;Marker(B600032) 加拿大BioBasic公司;6×DNA Loading Dye、柱式DNA胶回收试剂盒(B518131)上海生物工程有限公司。
表1 实验用转基因阳性和阴性样品来源
Table 1 Sources of GM positive and negative samples

注:*.欧洲标准物质中心;#.美国油脂化学家学会。
样品名称 来源 货号转基因DAS-44406-6大豆粉 ERM* ERM-BF436b转基因DAS-68416-4大豆粉 ERM ERM-BF432d转基因305423大豆粉 ERM ERM-BF426d转基因MON87701大豆粉 AOCS
# AOCS 0809-B转基因MON87705大豆粉 AOCS AOCS 0210-A转基因MON87708大豆粉 AOCS AOCS 0311-A转基因BPS-CV127-9大豆粉 AOCS AOCS 0911-C转基因GTS40-3-2大豆粉 山东出入境检验检疫局技术中心 验出样品转基因T304-40棉花籽 ERM ERM-BF429c转基因GHB119棉花籽 ERM ERM-BF428c转基因DAS-40278-9玉米粉 ERM ERM-BF433d转基因98140玉米粉 ERM ERM-BF427d转基因MIR162玉米粉 AOCS AOCS1208-A转基因Bt11玉米粉 山东出入境检验检疫局技术中心 验出样品转基因H7-1甜菜 ERM ERM-BF419b转基因73496油菜籽 ERM ERM-BF434b转基因281-24-236×3006-210-23棉花籽 ERM ERM-BF422d转基因MON 15985-7棉花籽 AOCS AOCS 0804-D转基因EH92-527-1马铃薯粉 AOCS AOCS 0806-C转基因AV43-6-G7马铃薯粉 ERM ERM-BF431b转基因AM04-1020马铃薯粉 ERM ERM-BF430b转基因PH05-026-0048马铃薯粉 ERM ERM-BF435b油菜籽 山东出入境检验检疫局技术中心 验出样品酒糟蛋白饲料DDGS 山东出入境检验检疫局技术中心 验出样品
1.2 仪器与设备
5810R台式离心机、Master Cycle Realplex 4实时PCR仪、微量移液器 德国Eppendorf公司;TissueLyser II研磨机 德国Qiagen公司;PCR反应扩增仪 加拿大BBI公司;SW-CJ-1D洁净工作台 江苏苏洁净化设备厂;DK-8D型电热恒温水槽 上海森信实验仪器有限公司;DYY-8型稳压稳流电泳仪 上海琪特分析仪器有限公司;YXJ-2离心机 湘仪离心机仪器有限公司;H6-1微型电泳槽 上海精益有机玻璃制品仪器厂;凝胶成像系统 美国Gene Genius公司;U-3010紫外-可见分光光度计 日本Hitachi公司;QX 100微滴式dPCR系统美国Bio-Rad公司。
1.3 方法
1.3.1 试剂盒法提取DNA
将非转基因大豆样品冷冻干燥,用研磨机在样品里面放钢珠研磨5~10 min研磨成60 目粉末。分别取50 mg转基因大豆DAS-44406-6样品粉末与非转基因大豆粉末于离心管中,采用安比奥植物核酸提取试剂盒,按照操作说明提取基因组DNA。
将转基因大豆DAS-44406-6基因组提取液与非转基因大豆基因组提取液按照一定的体积分数进行混合,分别配制成转基因大豆含量(体积分数)为100%、10%、1%、0.1%、0.01%和0.001%的预混液。梯度配制方法为:100%的转基因大豆预混液是用1 000 μL的转基因大豆基因组提取液的上清液和0 μL的非转基因大豆提取液的上清液进行混合获得。10%的转基因大豆预混液是用100 μL的转基因大豆基因组提取液的上清液和900 μL的非转基因大豆提取液的上清液进行混合获得。以此类推,按转基因大豆基因组提取液的上清液占总预混液体积的100%、10%、1%、0.1%、0.01%和0.001%进行添加。将DNA预混液分别取少部分用U-3010紫外-可见分光光度计测量质量浓度及纯度,在波长260 nm处测量预混液DNA浓度,并用OD
260 nm/OD
280 nm的比值作为衡量DNA纯度的标志,当OD
260 nm/OD
280 nm比值在1.7~1.9之间时可以用于本实验,配制成质量浓度为40 ng/μL的工作液,为荧光PCR做准备。
1.3.2 外源基因5’-RACE测定边界序列
根据DAS-44406-6品系大豆的外源插入基因RB7 MAR基因序列信息,用5’-RACE方法测定转基因大豆DAS-44406-6的5’端侧翼序列。使用基因特异性引物和适配性引物,通过cDNA末端快速扩增技术(rapidamplification of cDNA ends,RACE)得到5’末端基因序列。对5’-RACE来说,用基因特异性外引物D44406-R1和引物AP1进行PCR。DNA样品扩增在0.2 mL离心管中进行,最终反应体系是25 µL,分别包含引物D44406-R1和引物AP1 200 nmol/L;2×GC Buffer,2 mmol/L氯化镁、dATP、dCTP、dGTP、和dTTP各200 nmol/L,一个单位的LA Taq酶和100 ng的提取的DNA模板。PCR程序为预变性95 ℃,5 min;94 ℃,30 s;68 ℃,30 s;72 ℃,150 s;10 个循环(每个循环降0.8 ℃);94 ℃,30 s;60 ℃,30 s;72 ℃,150 s;30 个循环;终延伸72 ℃,10 min。
用基因特异性外引物D44406-R2和引物AP2进行巢式PCR。DNA样品扩增在0.2 mL离心管中进行,最终反应体系是25 µL,分别包含引物D44406-R2和引物AP2 200 nmol/L;2×GC Buffer,2 mmol/L氯化镁,dATP、dCTP、dGTP和dTTP各200 nmol/L,1 U的LA Taq酶和100 ng的提取的DNA模板。PCR程序为预变性95 ℃,5 min;94 ℃,30 s;58 ℃,30 s;72 ℃,150 s;28 个循环(每个循环降0.8 ℃);94 ℃,30 s;60 ℃,30 s;72 ℃,150 s;30 个循环;终延伸72 ℃,10 min。
1.3.3 实时荧光PCR
1.3.3.1 实时荧光PCR特异性引物探针设计
根据转基因DAS-44406-6大豆品系基因序列,使用Primer Express Version 3.0软件,设计出转基因DAS-44406-6大豆品系5’端边界序列特异性寡核苷酸引物和探针,并在表2中列出,由生工生物工程(上海)股份有限公司合成。对大豆而言lectin基因被选为内源参考基因。以特异性DNA序列为靶序列设计出来的相关引物对和探针如表2所示。
表2 转基因DAS-44406-6大豆品系实时荧光PCR特异性引物探针
Table 2 Primers and probes of real-time PCR for transgenic soybean DAS-44406-6

引物及探针名称 序列和荧光标记(5’-3’) 来源大豆内源基因Lectin-F CCAGCTTCGCCGCTTCCTTC [12]大豆内源基因Lectin-R GAAGGCAAGCCCATCTGCAAGCC [12]大豆内源基因Lectin-P FAM-CTTCACCTTCTATGCCCCTGACAC-TAMRA [12]大豆DAS-44406-6F CGTACAATATTACTCACCGGATCCT 本研究大豆DAS-44406-6R TGGTTTGGTTCGAATTTGTTTTAC 本研究大豆DAS-44406-6P FAM-TCATGGGCCGCGATTAAAAATCTCAATTA-TAMRA 本研究
1.3.3.2 实时荧光PCR特异性实验
大豆内源基因Lectin和转基因DAS-44406-6大豆品系特异性序列分别在大豆DAS-44406-6和表1中23 个样品提取的DNA。每个样品做两个重复。
使用实时PCR仪进行实时PCR。每个DNA样品在0.2 mL PCR反应管中进行扩增,最终反应体系为25 μL,包括:正向引物(10 μmol/L)0.3 μL、反向引物(10 μmol/L)0.9 μL、探针(10 μmol/L)0.9 μL、模板DNA 100 ng/反应,2.5×RealMaster Mix(with ROX)10 μL,20×Probe Enhancer Solution 1.25 μL,超纯水补足至25 μL。实时PCR反应程序为:预变性95 ℃,10 min;95 ℃,10 s;60 ℃,45 s;45 个循环。
1.3.3.3 实时荧光PCR检测低限测定
转基因大豆含量为100%、10%、1%、0.1%、0.01% 和0.001%的预混液每个梯度设置4 个重复,进行转基因大豆DAS-44406-6荧光PCR检测,同时设置空白对照。
使用实时PCR仪进行实时PCR。每个DNA样品在0.2 mL PCR反应管中进行扩增,最终反应体系为25 μL,包括:正向引物(10 μmol/L)0.3 μL、反向引物(10 μmol/L)0.9 μL、探针(10 μmol/L)0.9 μL、模板DNA(达到100 ng)5 μL,2.5×RealMaster Mix (with ROX)10 μL,20×Probe Enhancer Solution 1.25 μL,超纯水补足至25 μL。实时PCR反应程序为:预变性95 ℃,2 min;95 ℃,15 s;58 ℃,40 s;45 个循环。
1.3.4 dPCR
转基因大豆含量为100%、50%、10%、1%及0.1%的样品,进行相对灵敏度实验。绝对灵敏度样品的制备:将转基因含量为100%的大豆粉末提取基因组DNA,配制成10 ng/μL的DNA溶液,进行梯度稀释。每个反应加5 μL模板DNA。
微滴式dPCR(QX 100微滴式dPCR系统)反应体系为:dd PCR
TMSupermix for Probes 10 μL,上、下游引物各1 μL(10 μmol/L),探针1 μL(10 μmol/L),DNA模板5 μL,加双蒸水补足总体积至20 μL。反应体系充分混匀后加入Bio-Rad公司的微滴生成卡中,油包水微滴生成按照厂家的操作说明进行。将生成的微滴全部转入96 孔荧光PCR反应板中,进行PCR扩增。荧光PCR包括3 个步骤:95 ℃预变性10 min;94 ℃变性30 s,60 ℃退火1 min,45 个循环;98 ℃固化微滴10 min。最后用微滴分析仪对扩增产物进行计数分析。
2 结果与分析
2.1 边界序列测定结果
转基因大豆DAS-44406-6的外源基因5’边界序列进行5’-RACE得到确定的DNA片段。转基因大豆DAS-44406-6边界序列外源基因5’-RACE第2次PCR结果如图1所示。PCR引物为基因特异性外引物D44406-R2和引物AP2。

图1 转基因大豆DAS-44406-6外源基因边界序列5’-RACE第2次PCR结果电泳图
Fig. 1 Electrophoresis of the second PCR products of exogenous gene 5’-RACE from flanking region of transgenic soybean DAS-44406-6
2.2 实时荧光PCR
2.2.1 特异性引物探针设计
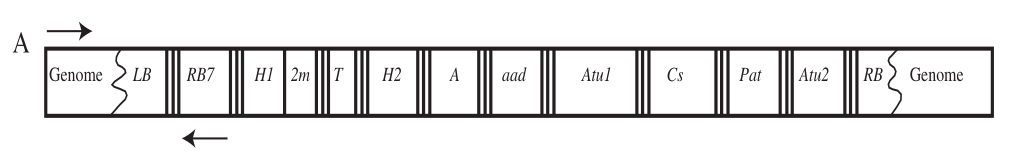
根据遗传物质修饰委员会(Commissie Genetische Modificatie,CoGEM)网站(http://www.cogem.org/ index.cfm/en)查询到的“Import of genetically modified soybean DAS-44406-6 with three herbicide tolerance traits”(COGEM advisory report CGM/130627-01)得到转基因大豆DAS-44406-6的外源插入基因的信息,进行特异性引物探针设计。结果如图2所示。

图2 DAS-44406-6品系大豆外源基因框架结构图
Fig. 2 Organization of transgene elements in soybean DAS-44406-6 genome
2.2.2 特异性实验结果
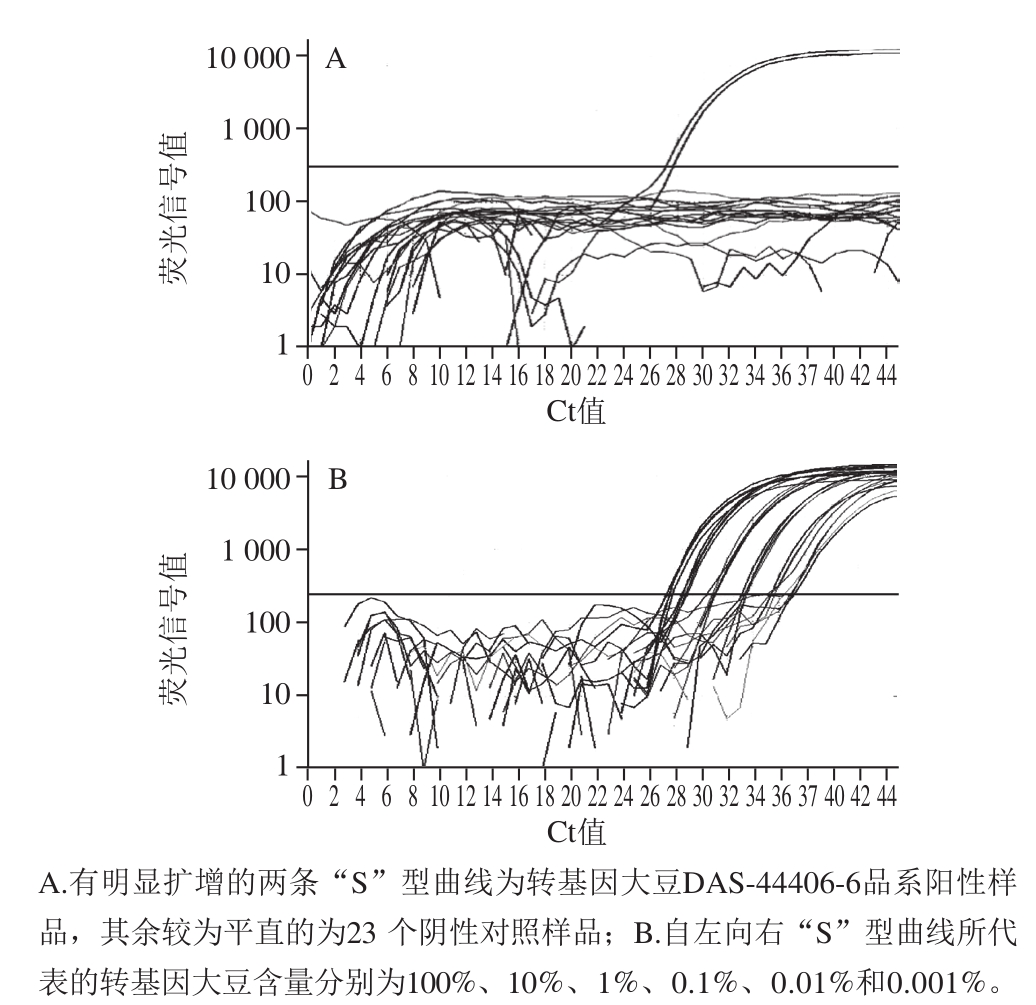
图3 实时荧光PCR方法检测转基因大豆DAS-44406-6的特异性与浓度梯度实验扩增曲线
Fig. 3 Specificity tests and standard curves of real time PCR method to detect transgenic soybean DAS-44406-6 and amplification curves from concentration gradient experiment
根据5端边界基因序列设计出的特异性引物探针建立荧光实时PCR反应,Lectin基因作大豆内源基因。如图3A所示,只有转基因大豆DAS-44406-6基因组DNA成功扩增出转基因DAS-44406-6大豆品系特异性序列,其他23 种DNA样本阴性对照在45 个循环内没有明显的扩增曲线,没有荧光增幅现象。这个结果表明针对转基因大豆DAS-44406-6特异性序列设计的引物探针特异性较好。
2.2.3 检测低限测定结果
转基因大豆DAS-44406-6基因组提取液与非转基因大豆基因组提取液混合配制成转基因大豆含量分别为100%、10%、1%、0.1%、0.01%和0.001%的预混液。对不同转基因含量样品DNA,进行实时荧光PCR扩增。结果如图3B所示,100 ng DNA模板中转基因大豆含量分别为100%、10%、1%、0.1%和0.01%的样品存在明显的扩增曲线,Ct值分别为27.65±0.15、28.80±0.03、30.86±0.11、33.36±0.09、35.60±0.20,Ct值均低于36
[3]。结果表明建立的转基因大豆DAS-44406-6特异性品系实时荧光PCR检测方法在检测模板DNA浓度为100 ng/反应时检测底限可达0.01%,根据大豆基因组DNA大小
[13](1 115 Mbp)计算,建立的转基因大豆DAS-44406-6特异性品系检测方法,在检测模板DNA浓度为100 ng/反应时,检测低限为0.01%的转基因大豆含量,相当于16.6 个拷贝数,符合转基因品系鉴定标识制度的要求
[14]。
2.3 dPCR定量检测转基因DAS-44406-6大豆品系
表3 使用dPCR检测转基因DAS-44406-6大豆的相对灵敏度实测数据
Table 3 Relative sensitivity of event-specific digital PCR for transgenic soybean DAS-44406-6

含量/% DAS-44406-6转基因大豆DNA浓度/(ng/反应)dPCR反应总DNA模板浓度/(ng/反应)基因组拷贝数实测含量/%相对偏标准差/% 100 50 50 83 000 93.19 1.1 50 25 50 41 500 57.58 1.8 10 5 50 8 300 9.84 2.5 1 0.5 50 830 1.17 0.7 0.1 0.05 50 83 0.23 16.0
在相对灵敏度实测实验中,总DNA模板浓度为50 ng/反应时,DAS-44406-6转基因大豆含量为100%、50%、10%、1%及0.1%的的样品每个稀释拷贝数为83 000、41 500、8 300、830、83拷贝数(表3)。转基因DAS-44406-6大豆品系dPCR检测方法对重复4 次的1%含量的转基因样品定量结果为1.17%。即当其转基因含量大于等于1%时,定量结果准确且精密度良好
[15]。转基因含量小于1%时,可以检出转基因靶标,但是不能够实现准确定量。
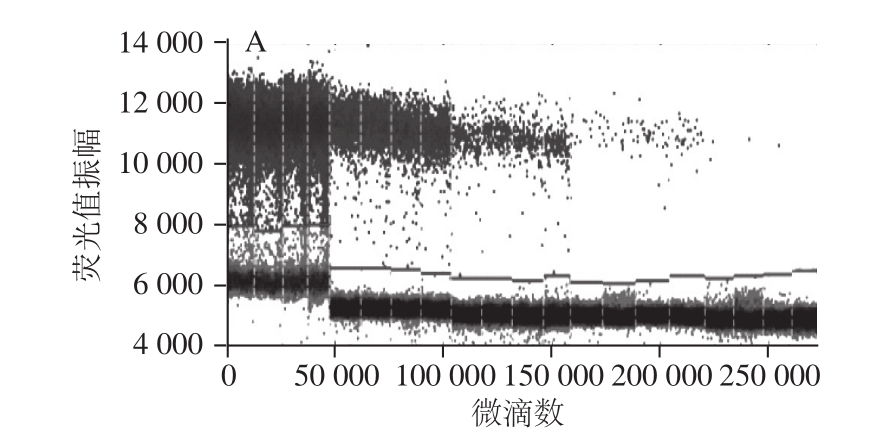
由图4、表4可知,在绝对灵敏度实测实验中,转基因DAS-44406-6大豆品系dPCR检测方法对重复4 次的模板DNA质量浓度为0.1 ng/μL的转基因样品定量结果为98.06%,相对标准差为3.9%;模板DNA质量浓度为0.01 ng/μL的转基因样品定量结果为96.34%,相对标准差为9.8%。即当模板DNA质量浓度大于等于0.01 ng/μL时,定量结果准确且精密度良好
[16-17]。
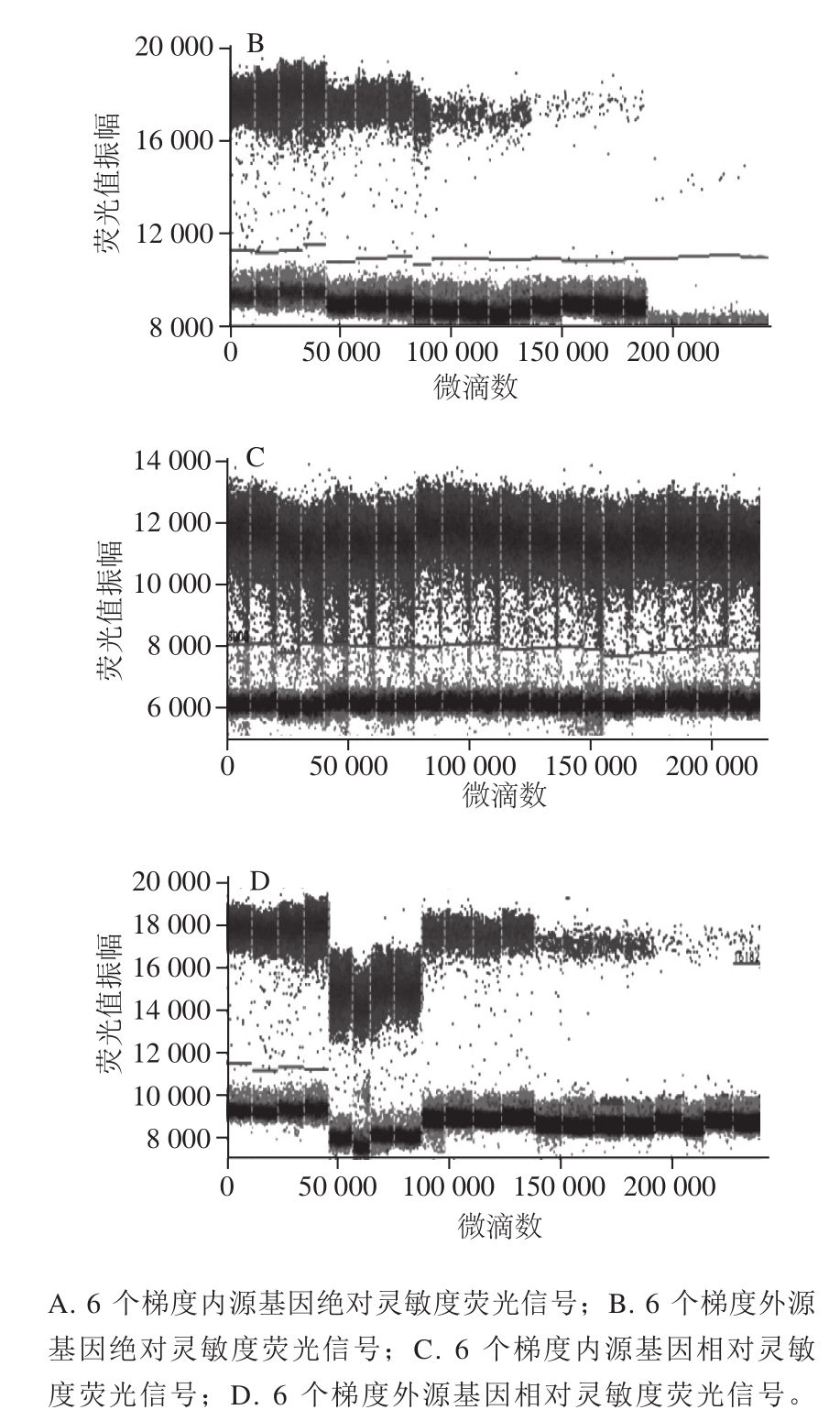
图4 dPCR检测转基因DAS-44406-6大豆绝对灵敏度与相对灵敏度荧光信号密度
Fig. 4 Absolute sensitivity and relative sensitivity fluorescence signal density diagram of digital PCR method to detect transgenic soybean DAS-44406-6
表4 使用dPCR检测转基因DAS-44406-6大豆的绝对灵敏度实测数据
Table 4 Absolute sensitivity of event-specific digital PCR for transgenic soybean DAS-44406-6
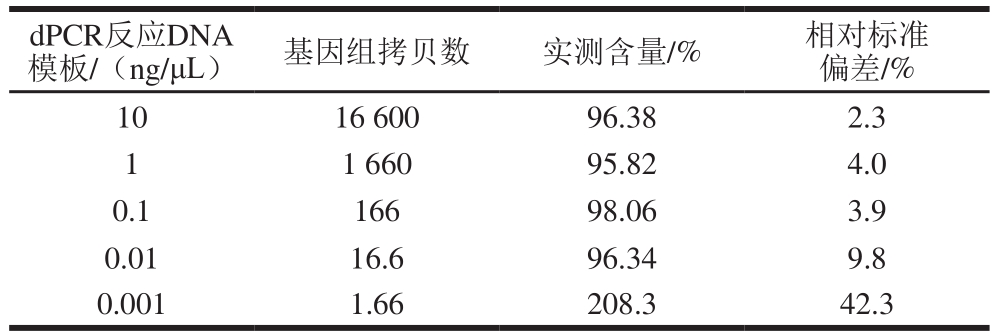
模板/(ng/μL) 基因组拷贝数 实测含量/% 相对标准偏差/% 10 16 600 96.38 2.3 1 1 660 95.82 4.0 0.1 166 98.06 3.9 0.01 16.6 96.34 9.8 0.001 1.66 208.3 42.3 dPCR反应DNA
3 讨 论
现在世界上种植面积最大的4 种转基因农作物包括大豆、玉米、棉花和油菜,而中国种植的主要转基因作物主要有棉花、木瓜、白杨、番茄和甜椒,2014年我国进口大豆超7 100万 t,其中大部分为转基因品系
[18]。现在中国政府已经许可10 种转基因大豆品系(A2704-12、A5547-127、CV127、DP305423、DP305423×GTS40-3-2、DP356043、GTS40-3-2、MON87701、MON87701×MON89788、MON89788)为加工原料进入国内市场,随着转基因作物进入人们的视野和餐桌,为了保障公众对于转基因食品的知情权,国家相关监管部门有必要建立和完善对进入国内市场的包括转基因大豆在内的转基因食品相关标识制度,而这其中的关键就是精确、有效、快速的转基因检测技术。
实时荧光定量PCR通过在PCR过程中引入一种荧光化学物质,在每个循环结束后,实时监测荧光信号累积的变化,从而定量定性推断出模板的初始量,绘制出荧光扩增的标准曲线。相对于普通PCR技术,无需将产物进行电泳,也避免了不必要的污染,准确直观地反映出产物的变化情况
[19-21]。非特异性SYBR Green Ⅰ染料法是在PCR反应体系中,加入过量荧光染料,掺入DNA双链后,发射出荧光信号,TaqMan探针技术相对于非特异性SYBR Green Ⅰ染料法探针识别具有更强的特异性以及灵敏度,适用性也很好,因此更加广泛地应用在各类转基因品系检测领域
[16-17]。与本研究类似,转基因MON89788大豆品系的实时荧光PCR检测方法已经有文献报道
[4]。
dPCR技术采用微滴化的方法,将含有DNA模板的微量样品进行稀释以及分液,分布到大量的独立反应室中,使每个样品中所含分子数不超过一个,然后各自在相同条件下进行PCR扩增,最后逐个分析发生扩增反应的分子个数
[22-24]。dPCR不使用扩增曲线的Ct值,也与内参基因和标准曲线无关,可以对样品做绝对定量分析,比定性PCR仅仅依靠标准曲线来确定核酸量更加准确
[8-10]。
在QX 100微滴式dPCR系统的反应条件较为宽松,dPCR与实时荧光定量PCR使用的引物和探针相同,也可以获得准确结果。建立的转基因大豆DAS-44406-6实时荧光定量PCR特异性品系检测方法检测低限在模板DNA浓度为100 ng/反应时,为0.01%的转基因大豆含量,约为16.6 个拷贝的DAS-44406-6基因组DNA。转基因DAS-44406-6大豆品系dPCR相对灵敏度检测方法在总DNA模板浓度为50 ng/反应时,能准确定量到转基因大豆含量为1%,模板DNA浓度为0.5 ng/反应的转基因样品
[25-28]。当转基因大豆含量为0.1%时,能检测出转基因样品,已超过准确定量范围。绝对灵敏度检测方法能准确定量到模板DNA质量浓度为0.01 ng/μL的转基因样品,为16.6 个拷贝。但是由于我国转基因品系检测标准为定性筛选检测,没有定量标准。因此dPCR方法相比于荧光定量PCR检测方法同样具有简便、高效等特点,对于转基因DAS-44406-6大豆品系的实际检测具有很大的应用价值。
参考文献:
[1] Clive James. 2014年全球生物技术/转基因作物商业化发展态势[J].中国生物工程杂志, 2015, 34(1): 1-14.
[2] 曹际娟, 徐君怡, 曹冬梅, 等. 实时荧光聚合酶链式反应检测转基因小麦B73-6-1、B72-8-11b和B102-1-2品系[J]. 食品科学, 2014,35(8): 156-159. DOI:10.7506/spkx1002-6630-201408031.
[3] 曹际娟, 朱水芳, 曹远银. GA21转基因玉米实时荧光PCR检测方法的建立[J]. 中国生物工程杂志, 2003, 23(8): 87-91; 95. DOI:10.3969/ j.issn.1671-8135.2003.08.019.
[4] 刘欣, 张国丛, 周兴虎, 等. 转基因大豆MON89788实时荧光PCR检测方法的建立[J]. 食品科学, 2015, 36(4): 193-197. DOI:10.7506/ spkx1002-6630-201504038.
[5] 李鹏, 潘爱虎, 贾军伟, 等. 转基因香石竹Moonlite品系特异性定性PCR检测方法的建立[J]. 中国农业科技导报, 2010, 12(6): 109-113.
[6] 邓平建, 赵锦, 刘建军, 等. 转基因食品安全性检验的核酸检测技术研究[J]. 卫生研究, 2002, 31(1): 37-40. DOI:10.3969/ j.issn.1000-8020.2002.01.020.
[7] 周晓丽, 朱国坡, 李雪华, 等. 实时荧光定量PCR技术原理与应用[J].中国畜牧兽医, 2010, 37(2): 87-89.
[8] 李亮, 隋志伟, 王晶, 等. 基于数字PCR的单分子DNA定量技术研究进展[J]. 生物化学与生物物理进展, 2012, 39(10): 1017-1023.
[9] 李春勇. 数字PCR技术原理及应用[J]. 生物技术世界, 2014(11): 10-13.
[10] 林彩琴, 姚波. 数字PCR技术进展[J]. 化学进展, 2012, 24(12): 2415-2423.
[11] 国际农业生物技术应用服务组织. GM approval database[EB/OL]. (2015-01-01) [2015-12-28]. http://www.isaaa.org/ gmapprovaldatabase/event.
[12] TAKABATAKE R, ONISHI M, KOIWA T, et al. Development and evaluation of event-specific quantitative PCR method for genetically modified soybean MON89788[J]. Journal of Biotechnology, 2010,150(6): 482-483. DOI:10.1016/j.jbiotec.2010.09.734.
[13] ARUMUGANATHAN K, EARLE E D. Nuclear DNA content of some important plant species[J]. Plant Molecular Biology Reporter,1991, 9(3): 208-218. DOI:10.1007/BF02672016.
[14] NELSON M, ELENA M, MARIOLINA G, et al. Methods for detection of GMOs in food and feed[J]. Analytical and Bioanalytical Chemistry, 2008, 392(3): 369-384. DOI:10.1007/s00216-008-2303-6.
[15] Codex Alimentarius Commission. CAC/GL 74-2010 Guidelines on performance criteria and validation of methods for detection,identification and quantification of specific DNA sequences and specific proteins in foods[S]. Rome: Codex Alimentarius Liment, 2010.
[16] DOBNIK D, SPILSBERG B, BOGOŽALEC KOŠIR A, et al. Multiplex quantification of 12 European Union authorized genetically modified maize lines with droplet digital polymerase chain reaction[J]. Analytical Chemistry, 2015, 87(16): 8218-8226. DOI:10.1021/acs. analchem.5b01208.
[17] LI P, JIA J W, JIANG L X, et al. Event-specific qualitative and quantitative PCR detection of the GMO carnation (Dianthus caryophyllus) variety Moonlite based upon the 5’-transgene integration sequence[J]. Genetics and Molecular Research Gmr, 2012, 11(2): 1117-1129. DOI:10.4238/2012.April.27.11.
[18] 韩俊. 2014年我国进口大豆超7 100万 吨大部分为转基因[EB/ OL]. (2015-02-03) [2015-12-28]. http://www.gov.cn/2015-02/03/ content_2813986.htm.
[19] 刘小荣, 张笠, 王勇平. 实时荧光定量PCR技术的理论研究及其医学应用[J]. 中国组织工程研究与临床康复, 2010, 14(2): 329-332.
[20] 陈旭, 齐凤坤, 康立功, 等. 实时荧光定量PCR技术研究进展及其应用[J]. 东北农业大学学报, 2010, 41(8): 148-155. DOI:10.3969/ j.issn.1005-9369.2010.08.029.
[21] 王小红. 荧光定量PCR技术研究进展[J]. 医学分子生物学杂志,2001, 23(1): 42-45. DOI:10.3870/j.issn.1672-8009.2001.01.015.
[22] OGUCHI T, ONISHI M, MANO J, et al. Development of multiplex PCR method for simultaneous detection of four events of genetically modified maize: DAS-59122-7, MIR604, MON863 and MON88017[J]. Shokuhinseigaku Zasshi Journal of the Food Hygienic Society of Japan, 2010, 51(3): 92-100. DOI:10.3358/shokueishi.51.92. [23] REONA T, MARI O, TOMOHIRO K, et al. Development and interlaboratory validation of quantitative polymerase chain reaction method for screening analysis of genetically modified soybeans[J]. Biological and Pharmaceutical Bulletin, 2013, 36(1): 131-134.
DOI:10.1248/bpb.b12-00766.
[24] LEE D, MURA M L, ALLNUTT T R, et al. Detection of genetically modified organisms (GMOs) using isothermal amplification of target DNA sequences[J]. Bmc Biotechnology, 2009, 9(3): 7.
DOI:10.1186/1472-6750-9-7.
[25] LI Xiang, WANG Xiuxiu, YANG Jielin, et al. A novel quadruplex real-time PCR method for simultaneous detection of Cry2Ae and two genetically modified cotton events (GHB119 and T304-40)[J]. BMC Biotechnology, 2014, 14(4): 1-10. DOI:10.1186/1472-6750-14-43.
[26] SARA J, ELENA N, CHRISTIAN S, et al. Development, optimization,and single laboratory validation of an event-specific real-time PCR method for the detection and quantification of Golden Rice 2 using a novel taxon-specific assay[J]. Journal of Agricultural and Food Chemistry, 2015, 63(6): 1711-1721. DOI:10.1021/jf505516y.
[27] RABIEI M, MEHDIZADEH M, RASTEGAR H, et al. Detection of genetically modified maize in processed foods sold commercially in iran by qualitative PCR[J]. Iranian Journal of Pharmaceutical Research, 2013, 12(1): 25-30.
[28] LIU M, LUO Y, TAO R, et al. Sensitive and rapid detection of genetic modified soybean (Roundup Ready) by loop-mediated isothermal amplification[J]. Agricultural and Biological Chemistry, 2009, 73(11): 2365-2369. DOI:10.1271/bbb.80723.
Detection of Genetically Modified Soybean Event DAS-44406-6 by Real-Time PCR Method and Digital PCR Method
YU Xiaofan
1, GAO Hongwei
2,*, SUN Min
2, XIAO Xizhi
2, LI Ronggui
1,*
(1. College of Life Sciences, Qingdao University, Qingdao 266071, China; 2. Center of Inspection and Quarantine Technology,Shandong Entry-Exit Inspection and Quarantine Bureau, Qingdao 266002, China)
Abstract:Purpose: To determine the flanking sequence between exogenous and endogenous fragments using rapid amplification of cDNA ends (5’-RACE) and consequently to examine genetically modified soybean (GMS) DAS-44406-6. Methods: Specific primers and probes were designed based on the flanking sequences of exogenous fragments of DAS-44406-6. The specificity of the developed method was validated by using it to detect a variety of other GM samples and non-GM samples. DAS-44406-6 was used to prepare 6 content gradients to test the sensitivity of the method. At last, digital PCR was applied to measure nucleic acid molecules for absolute quantification. Conclusions: A real-time PCR method has been established for the identification of GMS soybean DAS-44406-6 with high specificity. The limit of detection (LODs) of the real-time PCR method at template DNA concentration of 100 ng/reaction and 0.01% GM soybean content was 16.6 copies of genomic DNA from DAS-44406-6. As for the LOD of the digital PCR method, the relative standard deviation (RSD) was 0.7% at template DNA concentration of 0.5 ng/reaction. The highly specific method combining real-time PCR and digital PCR could meet the requirements for the detection of GMS DAS-44406-6.
Key words:genetically modified soybean; DAS-44406-6 event; event-specific detection; real-time PCR; digital PCR
DOI:10.7506/spkx1002-6630-201616038
中图分类号:S565.1
文献标志码:A
文章编号:1002-6630(2016)16-0235-07
引文格式:
于晓帆, 高宏伟, 孙敏, 等. 荧光PCR和数字PCR法检测转基因DAS-44406-6品系大豆[J]. 食品科学, 2016, 37(16): 235-241. DOI:10.7506/spkx1002-6630-201616038. http://www.spkx.net.cn
YU Xiaofan, GAO Hongwei, SUN Min, et al. Detection of genetically modified soybean event DAS-44406-6 by realtime PCR method and digital PCR method[J]. Food Science, 2016, 37(16): 235-241. (in Chinese with English abstract)
DOI:10.7506/spkx1002-6630-201616038. http://www.spkx.net.cn
收稿日期:2016-01-07
基金项目:公益性行业(质检)科研专项(201410014)
作者简介:于晓帆(1992—),男,硕士研究生,研究方向为微生物及食品基因检测。E-mail:wallace38@126.com
*通信作者:高宏伟(1975—),女,研究员,博士,研究方向为农业转基因检测及动植物基因检测。E-mail:ghw75@126.com
李荣贵(1969—),男,教授,博士,研究方向为生物活性分子的结构与功能。E-mail:lrg@qdu.edu.cn