Salmonella hadar对喹诺酮类药物耐药性及其耐药基因分析
吴浩天,武 运
*,尹明远,古丽娜孜,王 威,张亚南,田 歌,马文瑞
(新疆农业大学食品科学与药学学院,新疆 乌鲁木齐 830052)
摘 要:目的:研究新疆乌鲁木齐市部分农贸市场内检出的21 株Salmonella hadar对2 种喹诺酮类抗生素的药敏性及相关耐药基因,更好地了解耐药性的产生和传播途径,确保食品安全。方法:用琼脂稀释法测定Salmonella hadar的药敏性,用聚合酶链式反应和测定基因序列的方法确定耐药沙门氏菌中与喹诺酮类抗生素耐药性相关的喹诺酮类抗性决定区突变基因以及质粒携带的耐药基因。结果:21 株Salmonella hadar对萘啶酮酸的耐药率达100%,对环丙沙星表现为敏感;qnrB、qnrA、qnrS基因的检出率分别为52.30%、4.76%、4.76%;21 株Salmonella hadar均为gyrA和parC基因同时突变,gyrA基因的突变类型是Ser83Phe,parC基因的突变类型是Thr57Ser。结论:新疆乌鲁木齐Salmonella hadar对喹诺酮类药物的耐药状况应当予以关注,其耐药决定区突变基因及质粒携带的耐药基因在一定程度上会影响Salmonella hadar的耐药机制。
关键词:沙门氏菌;喹诺酮类药物;耐药性;聚合酶链式反应;耐药基因
吴浩天, 武运, 尹明远, 等. Salmonella hadar对喹诺酮类药物耐药性及其耐药基因分析[J]. 食品科学, 2016, 37(17): 150-155. DOI:10.7506/spkx1002-6630-201617025. http://www.spkx.net.cn
WU Haotian, WU Yun, YIN Mi ngyuan, et al. Quinolone resistance and related genes in Salmonella hadar[J]. Food Science, 2016, 37(17): 150-155. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201617025. http://www.spkx.net.cn
沙门氏菌是目前最常见的食源性致病菌,也是引起我国食源性疾病的主要病原体之一。我国每年大概3亿 人因沙门氏菌感染而患病,占病原菌型食源性疾病总数的70%~80%
[1]。目前,在人类临床治疗、畜牧业及农业生产中,抗生素依然是抵抗各种病原菌的首选药物
[2]。然而,病原菌耐药性的耐药谱型增宽,对食品用动物生产和公共卫生安全产生了极大危害,同时对人类健康和全球食品安全也造成巨大威胁
[3-6]。肉制品是人们摄取动物蛋白的主要来源,并在人们的日常饮食生活中占据重要地位。在喂养过程中大量使用抗生素是商业化生产中普遍存在的问题,由此必然会引发Salmonella hadar的耐药谱型增宽。了解Salmonella hadar的耐药性及其耐药机制可以有效预防与控制该病原菌,具有重要意义。
喹诺酮类药物耐药机制主要是由染色体介导的靶位点拓扑异构酶的改变和细胞内药物蓄积的减少引起的,前者通过降低靶位与喹诺酮类药物的亲合力引起耐药,后者通过孔通道蛋白缺失、主动外排引起系统亢进导致细胞内有效药物浓度减少而耐药
[7]。沙门氏菌对喹诺酮类抗生素的耐药表型与突变点检出率之间存在着一定关系。喹诺酮类抗生素被广泛用于治疗呼吸道感染、尿路感染、腹腔感染等疾病,但随着临床应用的增多,沙门氏菌对喹诺酮类药物的耐药性上升迅速。研究表明沙门氏菌对喹诺酮类的耐药机制主要为质粒介导、靶位改变及主动外排,后两者均为染色体介导。抗生素作用靶位编码基因突变、质粒介导和外排泵是引起沙门氏菌对喹诺酮类药物产生耐药性的主要因素
[8-12]。
目前食源性沙门氏菌的耐药性及其相关耐药性基因研究已经在国外取得了高度关注,其耐药分子的作用机理及耐药基因方面的研究也相对较多,但是在国内相关研究较少
[13-14]。有关乌鲁木齐市Salmonella hadar的喹诺酮类抗生素耐药性、相关突变基因及耐药质粒携带基因的研究也鲜见报道。本实验主要对2013—2014年采集分离于新疆乌鲁木齐市的21 株Salmonella hadar对2 种喹诺酮类药物的耐药状况及相关耐药基因进行分析,以期为保障食品安全提供部分参考依据。
1 材料与方法
1.1 材料
1.1.1 菌株来源
2013—2014年采集自乌鲁木齐市部分农贸市场内的鸡肉、羊肉和猪肉的1 414 份样品中分离的96 株沙门氏菌,其中21 株为Salmonella hadar;沙门氏菌标准菌株Salmonella Typhimurium LT2,药敏实验质控菌株大肠埃希菌ATCC 25922和粪肠球菌ATCC 29212均由西北农林科技大学食品科学与工程微生物实验室保存。
1.1.2 培养基与试剂
蛋白胨缓冲液、四硫磺酸盐煌绿增菌液、XLT4培养基、SS琼脂培养基、亚硫酸铋琼脂(BS)、氯化镁孔雀绿肉汤增菌液、木糖赖氨酸脱氧胆盐琼脂培养基、Luria-Bertani琼脂 青岛日水生物技术有限公司;尿素琼脂、Luria-Bertani(LB)肉汤、麦康凯琼脂 北京陆桥技术有限责任公司;MH琼脂(MHA)培养基、沙门氏菌显色培养基 青岛海博生物技术有限责任公司;沙门氏菌属O多价抗血清A-F 宁波天润生物药业有限公司;聚合酶链式反应(polymerase chain reaction,PCR)用引物(表1)均使用Primer Premier 5.0软件设计,由上海捷锐生物工程有限公司合成。
表1 PCR扩增用引物
Table 1 The primers for PCR amplification

基因引物序列(5’→3’)退火温度/℃产物长度/bp qnrA正向:AGAGGATTTCTCACGCCAGG60580反向:TGCCAGGCACAGATCTTGAC qnrB正向:GATCGTGAAAGCCAGAAAGG56469反向:ATGAGCAACGATGCCTGGTA qnrS正向:GCAAGTTCATTGAACAGGGT57428反向:TCTAAACCGTCGAGTTCGGCG gyrA正向:CGTTGGTGACGTAATCGGTA56190反向:CCGTACCGTCATAGTTATCC parC正向:CTATGCGATGTCAGAGCTGG54270反向:TAACAGCAGCTCGGCGTATT
1.1.3 仪器与设备
超净工作台 青岛海尔特种电器有限公司;隔水式培养箱 江苏东鹏仪器制造有限公司;鼓风干燥箱宝康电器设备有限公司;-30 ℃低温冰箱、-80 ℃超低温冰箱 日本三洋株式会社;三用恒温水箱 常州中捷实验仪器制造有限公司;高压灭菌锅 上海申安医疗器械厂;漩涡振荡器 海门市其林贝尔仪器制造有限公司;分析天平 上海民桥精密科学仪器有限公司。
1.2 方法
1.2.1 2 种喹诺酮类药物药敏实验
1.2.1.1 喹诺酮类药物贮存液的制备
配制抗生素母液及相应质量浓度的稀释液时,按照不同种类的抗生素所需溶剂不同的原理,将各种抗生素配制并稀释到实验所需质量浓度(母液均5 120 μg/mL),如使用过程中因质量浓度过高而导致添加体积过小不能实现或取样误差太大时,可用无菌超纯水或相应的缓冲液将母液贮存液稀释成低质量浓度抗生素溶液,如:2 560、1 280、640、320、160、80 µg/mL,然后使用孔径为0.2 μm的无菌滤器对抗生素溶液进行过滤,除菌后母液和各稀释度的抗生素溶液均贮存在-30 ℃冰箱,备用。
1.2.1.2 抗生素平板制备
称取一定质量的MH琼脂培养基后,分别加入已经准备好的13 个三角瓶中,加入适量的蒸馏水,放置于磁力加热搅拌器上煮沸1 min左右,使培养基颗粒充分溶解,然后121 ℃高压灭菌15 min。灭菌完毕后,将培养基置于50 ℃恒温水浴锅中,使其降温并保持在50 ℃左右,再将适量抗生素溶液分别加入相应的培养基中。其中,第13瓶为对照,第1~12瓶抗生素终质量浓度分别为0.25、0.5、1、2、4、8、16、32、64、128、256、512 μg/mL。同一抗生素质量浓度重复倒3 个琼脂平板。
1.2.2 PCR检测相关耐药基因
1.2.2.1 DNA模板制备
用无菌棉签擦拭适量的新鲜沙门氏菌液在LB平板上培养24 h,将其均匀洗剂于无菌生理盐水中制成麦氏标准为0.5的菌悬液,取菌悬液800 μL于1.5 mL 的无菌离心管中,100 ℃左右加热煮沸10 min,13 200 r/min离心5~8 min后,小心吸取上清液,留作备用。
1.2.2.2 PCR反应
将25 μL PCR混合反应体系所需的13.25 μL水,2.5 μL 10×PCR Buffer,2.0 μL dDTP,1.5 μL MgCl
2,0.25 μL的Taq酶、上游引物、下游引物和5 μL DNA模板依次加入置于冰浴上的无菌PCR 管中,离心混匀后,设置PCR扩增反应条件为94 ℃预变性10 min,1 个循环;94 ℃变性1 min,60 ℃退火1min,72 ℃延伸1 min,35 个循环;最后72 ℃延伸10 min。退火温度随着扩增用引物DNA熔解温度的不同而改变,不同基因扩增过程的退火温度应由相应的引物序列来确定(表1)。PCR产物检测:PCR扩增之后取出8 μL产物,加入2 μL上样缓冲液混匀,点样于1%的琼脂糖凝胶,100 V电泳30 min,电泳后用凝胶成像系统照相并观察结果
[15]。
1.2.2.3 DNA序列确定
将扩增得到的gyrA和parC PCR粗产物在低温条件下送至上海桑尼生物技术有限责任公司纯化后测序,测定得到的gyrA和parC基因DNA序列输入基因库,采用基因库在线比对软件BLAST程序进行比对,确定标准菌株Salmonella Typhimurium LT2的上述DNA序列与基因库序列完全吻合后,分析比对供试菌株相应的DNA序列,确定突变点和相应的氨基酸突变种类。质粒耐药基因检测:由于抗性细菌因其质粒上的抗性基因导致对喹诺酮类抗生素产生抗性,根据其对喹诺酮类药物产生抗性的特点,通过PCR检测喹诺酮类药物的耐药基因qnr
[16]。
2 结果与分析
2.1 Salmonella hadar对2 种喹诺酮类药物的耐药性
表2 2 Salmonella hadar adar耐药检测结果
Table 2 Detection results of drug resistance in Salmonella haaddaarr

(中介耐药率/%) 敏感数(敏感率/%)萘啶酮酸≥3219(100)00环丙沙星≥402(9.5)19(90.5)抗生素耐药折点耐药数(耐药率/%)中介耐药数
由表2可知,21 株Salmonella hadar对萘啶酮酸的耐药率达100%,9.5%的Salmonella hadar对环丙沙星的耐药率为中度敏感、90.5%的Salmonella hadar对环丙沙星的耐药率敏感,说明Salmonella hadar对第一代喹诺酮类药物表现很强的耐药性,对第三代喹诺酮类药物表现为敏感。
2.2 Salmonella hadar中与喹诺酮类药物耐药决定区突变基因检测
表3 突变基因检测结果
Table 3 Results of mutant gene detection
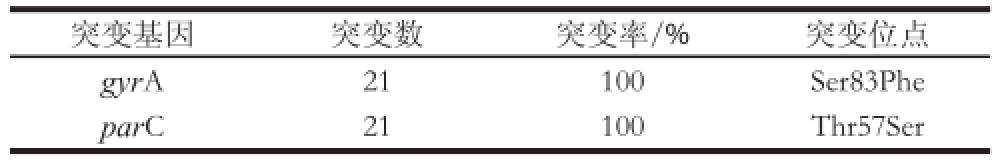
突变基因突变数突变率/%突变位点gyrA21100Ser83Phe parC21100Thr57Ser

图1 1 gyrgyrA基因的PCR扩增产物电泳图谱
Fig. 1 Electrophoresis map of gyrA gene
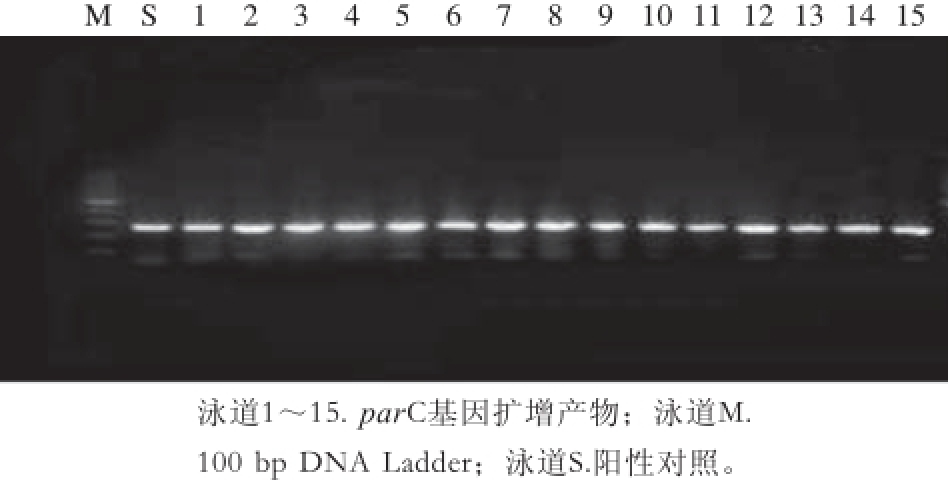
图2 2 parparC基因的PCR扩增产物电泳图谱
Fig. 2 Electrophoresis map of parC gene
本实验主要检测的是21 株Salmonella hadar的gyrA和parC基因突变。图1、2结果显示gyrA、parC基因的DNA大小分别为190 bp和270 bp。由表3可知,gyrA基因的突变位点是Ser83Phe,检出率为100%;parC基因的突变位点是Thr57Ser,检出率为100%,说明21 株Salmonella hadar均为Ser83Phe(gyrA)-Thr57Ser(parC)同时突变菌株。
2.3 Salmonella hadar中与喹诺酮类药物耐药质粒携带基因检测
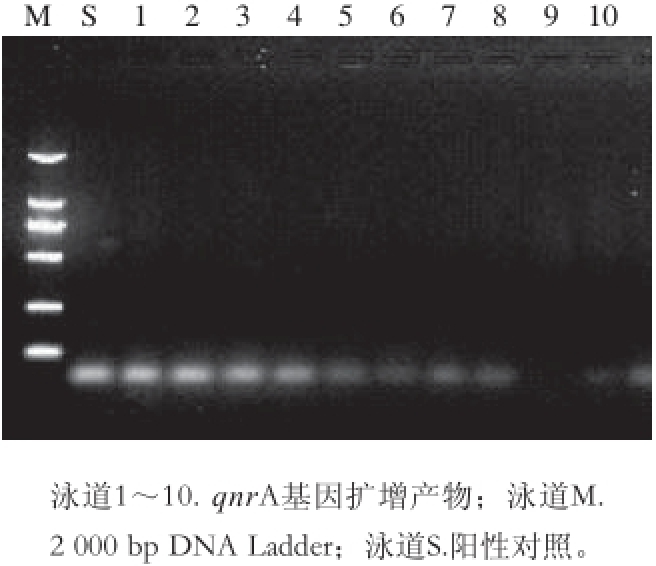
图3 3 qnrqnrA基因的PCR扩增产物电泳图谱
Fig. 3 Electrophoresis map of qnrA gene

图4 4 qnrqnrB基因的PCR扩增产物电泳图谱
Fig. 4 Electrophoresis map of qnrB gene

图5 5 qnrqnrS基因的PCR扩增产物电泳图谱
Fig. 5 Electrophoresis map of qnrS gene
表4 耐药相关质粒基因检出结果
Table 4 Detection results of drug resistance related plasmids
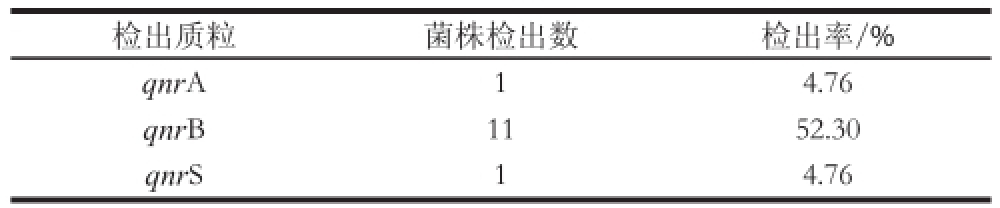
检出质粒菌株检出数检出率/% qnrA14.76 qnrB1152.30 qnrS14.76
qnr是一种质粒介导水平传播的喹诺酮类耐药基因
[17]。由图3~5可知,通过PCR扩增基因产物进行电泳后得qnrA、qnrB、qnrS基因的DNA大小分别为580、469、428 bp。由表4可知,分离鉴定的21 株 Salmonella hadar qnr基因阳性菌株为12 株,qnr基因检出率为57.14%。其中qnrA和qnrS基因阳性菌株均为1 株,qnrB阳性菌株为11 株,qnr阳性菌株中编号为YR5的菌株同时检测到qnrA和qnrB基因,用喹诺酮类耐药基qnr特异性引物qnrA、qnrB、qnrS分别检测其可移动遗传元件上携带耐药基因的情况,21 株Salmonella hadar中qnrA、qnrB、qnrS基因的检出率分别为4.76%、52.3%、4.76%,可知qnrB基因的检出率明显高于qnrA、qnrS基因的检出率。
3 讨 论
21 株Salmonella hadar对2 种喹诺酮类药物的耐药情况,说明21 株Salmonella hadar对喹诺酮类药物表现出了一定的耐药性,对萘啶酮酸的耐药率达100%,9.5%的Salmonella hadar对环丙沙星的耐药率为中度敏感、90.5%的Salmonella hadar对环丙沙星的耐药率为敏感。林居纯等
[18]研究结果显示食源沙门氏菌对萘啶酮酸的耐药率达85.45%、环丙沙星的耐药率达12.35%。本实验结果表明Salmonella hadar对第一代喹诺酮类药物表现很强的耐药性,对第三代喹诺酮类药物未表现出耐药,与林居纯等
[18]的研究结果相符合,这一结果为乌鲁木齐市研究Salmonella hadar对喹诺酮类药物的耐药性的研究提供了重要依据。
喹诺酮类药物的作用机制是通过干扰沙门氏菌DNA复制从而具有杀菌作用,其作用的靶位点是拓扑异构酶(DNA解旋酶)和DNA促旋酶,二者均属于沙门氏菌正常生长所必需的H型拓扑异构酶,两者任意一种受到抑制,都会影响沙门氏菌的生长,最终引起死亡。在DNA的复制与转录过程中,拓扑异构酶Ⅳ的作用是解开DNA分子天然存在的负超螺旋状态,使得复制叉能够顺利地向前移动,完成DNA分子的负超螺旋,从而使得复制或转录后的DNA分子恢复天然的负超螺旋状态。喹诺酮类药物通过细菌膜通透屏障后可与DNA复制与转录过程中形成的DNA-拓扑异构酶Ⅳ(parC)或DNA-促旋酶(gyrA)复合物相结合,引起酶的构象发生改变,而形成副喹诺酮-DNA-拓扑异构酶Ⅳ与DNA分子的分离,还能够有效阻止DNA复制交叉的前进从而达到DNA的复制和细菌的生长,最终导致细菌的死亡
[19-21]。目前已报道
[22]与沙门氏菌的喹诺酮类药物耐药耐药性有关的靶基因突变位点有:gyrA的突变包括:67位Ala突变为Pro、72位Asp突变Gly、73位Val突变为Ile、82位Asp突变为Asn、83位Ser突变为Ala/Tyr/Phe、98位Leu突变为Val、114位Met突变为Leu、119位Ala突变为Ser/Val/Glu、121位Arg突变为Cys、131位Ala突变为Gly、139位Ala突变为Ser;parC的突变包括:57位Thr突变为Ser、66位Thr突变为Ile、80位Ser突变为Arg
[23]。本实验中gyrA基因的突变位点是Ser83Phe;parC基因的突变位点是Thr57Ser,符合已有的相关报道。抗生素靶位基因突变是导致细菌产生耐药的重要原因之一。沙门氏菌通过改变抗生素作用靶位使抗菌药物不能识别,从而产生耐药性。gyrA基因是氟喹诺酮类药物的最初靶位,是沙门氏菌DNA复制与转录中起重要作用的DNA旋转酶亚基。位于gyrA蛋白第67(Ala)和106(Gln)位氨基酸残基之间喹诺酮耐药决定区中第87位的Asp经常突变为Gly、Asn或Tyr,第83位的Ser经常突变为Phe、Tyr或Ala而引起对氟喹诺酮类药物的抗性
[24]。据报道
[25],沙门氏菌中parC基因是拓扑异构酶Ⅳ编码基因,也可能是喹诺酮类药物的作用靶位,如parC中第57位Thr突变为Ser、第80位Ser突变为Arg或Ile均可引起抗性产生
[26]。本实验中21 株Salmonella hadar均为Ser83Phe(gyrA)-Thr57Ser(parC)同时突变菌株,符合已有的相关报道。说明Salmonella hadar喹诺酮类药物耐药区gyrA、parC基因的突变在一定程度上会影响Salmonella hadar的喹诺酮类药物的耐药机制,需要进一步研究。
表4是对喹诺酮类耐药基因qnr类基因qnrA、qnrB、qnrS进行检测,结果表明,21株Salmonella hadar中qnrA和qnrS基因检出率较低,均仅检出1 株,而qnrB阳性菌株为11 株,整体来说,qnr基因的扩散并不算广泛,可知并非所有对喹诺酮类药物具有抗性的Salmonella hadar菌株质粒都携带有qnr基因,有可能是这些耐药菌株基因组中喹诺酮耐药区基因产生突变
[27]引起的,还需要进一步深化研究。
4 结 论
本实验对2013—2014年间采集的样本中分离鉴定出21 株阳性Salmonella hadar,对2 种喹诺酮类药物耐药情况及相关耐药突变基因和相关耐药质粒基因进行检测研究。结果显示21 株Salmonella hadar对萘啶酮酸的耐药率达100%,环丙沙星表现为敏感,其中9.5%为中度敏感、90.5%为敏感;qnrB、qnrA、qnrS基因的检出率分别为52.30%、4.76%、4.76%;21 株Salmonella hadar均为gyrA和parC基因同时突变,gyrA基因的突变类型是Ser83Phe,parC基因的突变类型是Thr57Ser。结果表明21株Salmonella hadar对第一代喹诺酮类药物具有很高的耐药性,对第三代喹诺酮类药物未产生耐药,但具有较高的敏感性,Salmonella hadar耐药决定区突变基因可能是影响Salmonella hadar耐药的重要因素,Salmonella hadar耐药决定区突变基因及耐药质粒基因与Salmonella hadar的耐药性的相关关系需要进一步深化研究。
研究新疆乌鲁木齐市Salmonella hadar对2 种喹诺酮类药物的耐药性及耐药突变基因与相关耐药质粒基因,有助于从食物链的源头和食品性动物生产中采取合理的干预措施,坚持合理用药,减少和防止沙门氏菌耐药性的产生,保障食品安全。同时,也为更好地了解沙门氏菌的耐药机理和有效控制日趋严重的沙门氏菌耐药性问题提供依据。
参考文献:
[1] SCHUCHAT A, TAPPERO J, BLANDFORD J. Global health and the US Centers for Disease Control and Prevention[J]. The Lancet, 2014, 384: 98-101. DOI:10.1016/s0140-6736(14)60570-5.
[2] SU L H, CHIU C H, CHU C, et al. Antimicrobial resistance in nontyphoid Salmonella serotypes: a global challenge[J]. Clinical Infectious Disease, 2004, 39(4): 546-551. DOI:10.1086/422726.
[3] 郭云昌, 刘秀梅. 市售鸡肉中沙门氏菌分离株多重耐药谱测定[J]. 中国食品卫生杂志, 2005, 17(2): 100-103.
[4] 张增峰, 孟晓风, 杨保伟, 等. 鸡肉源沙门氏菌对(氟)喹诺酮类抗生素的耐药性及相关基因[J]. 中国食品学报, 2015, 15(3): 158-165. DOI:10.16429/j.1009-7848.2015.03.021.
[5] BYWATER R, DELUYKER H, DERCOOVER E, et al. A European survey of antimicrobial susceptibility among zoonotic and commensal bacteria isolated from food-producing animals[J]. Journal of Antimicrobial Chemotherapy, 2004, 54(4): 744-754. DOI:10.1093/jac/ dkh422.
[6] CRUMP J A, MEDALLA F M, JOYCE K W, et al. Antimocrobial resistance among invasive nontyphoidal Salmonella enterica isolates in the United States: national antimocrobial resistance monitoring system, 1996 to 2007[J]. Antimocrobial Agents and Chemotherapy, 2011, 55(3): 1148-1154. DOI:10.1128/aac.01333-10.
[7] 陈冬平, 罗薇. 沙门氏菌毒力相关因子研究进展[J]. 西南民族大学学报(自然科学版), 2012(5): 770-775. DOI:10.3969/ j.issn.1003-4271.2012.05.17.
[8] 郝宏珊, 杨保伟, 师俊玲, 等. 鸡肉源沙门氏菌对喹诺酮和氟喹诺酮类抗生素耐药状况及相关基因[J]. 微生物学报, 2011, 51(10): 1413-1420.
[9] 刘芳萍, 王德宁, 李昌文, 等. 鸡源沙门氏菌耐药性的分析及毒力基因的检测[J]. 中国兽医科学, 2013(12): 1236-1239.
[10] 侯雪娇, 莫国东, 焦鹏涛, 等. 市售鲜畜肉中沙门菌的血清型及耐药性研究[J]. 黑龙江畜牧兽医, 2015(15): 281-284.
[11] 周佳, 刘书亮, 侯小刚, 等. 四川省动物性食品源沙门氏菌的耐药性监测与分析[J]. 中国畜牧兽医, 2011(3): 188-191.
[12] 唐攀, 崔恩慧, 刘万华, 等. 鸡源沙门菌PFGE分型及耐药性研究[J].动物医学进展, 2013(11): 1-5.
[13] FONSECA E L, MYKYTCZUK O L, ASENSI M D, et al. Clonality and antimicrobial resistance gene profi les of multidrug resistant Salmonella enterica serovar infantis isolates from four public hospitals in Rio de Janeiro, Brazil[J]. Journal of Clini cal Microbiology, 2006, 44(8): 2767-2772. DOI:10.1128/jcm.01916-05.
[14] LYNNE A M, RHODES-CLARK B S, BLIVEN K, et al. Antimicrobial resistance genes associated with Salmonella enterica serovar Newport isolates from food animals[J]. Antimicrobial Agents and Chemotherapy, 2008, 52(1): 353-356. DOI:10.1128/aac.00842-07.
[15] 杨保伟, 曲东, 申进玲, 等. 陕西食源性沙门氏菌耐药及相关基因[J].微生物学报, 2010, 50(6): 788-796.
[16] VELDMAN K, CAVACO L M, MEVIUS D, et al. International collaborative study on the occurrence of plasmid-mediated quinoloneresistance in Salmonella enterica and Escherichia coli isolated from animals, humans, food and the environment in 13 European countries[J]. Journal of Antimicrobial Chemotherapy, 2011, 66(6): 1278-1286. DOI:10.1093/jac/dkr084.
[17] 王明华. qnr介导细菌对喹诺酮类耐药机制的研究[D]. 上海: 复旦大学, 2010: 51-59.
[18] 林居纯, 覃春红, 赖婧, 等. 食品动物源沙门氏菌质粒介导喹诺酮类耐药基因的检测与分析[J]. 畜牧兽医学报, 2012, 43(5): 803-809.
[19] 陈文标, 佘菲菲. 细菌对喹诺酮类药物耐药机制的研究进展[J]. 检验医学与临床, 2007, 4(3): 200-201. DOI:10.3969/ j.issn.1672-9455.2007.03.025.
[20] JIN H, JI H K, JONG H P, et al. Molecular and virulence characteristics of multi-drug resistant Salmonella Enteritidis strains isolated from poultry[J]. The Veterinary Journal, 2011, 189(3): 306-311. DOI:10.1016/j.tvjl.2010.07.017.
[21] DALLAL M M S, DOYLE M P, REZADEHBASHI M, et al. Prevalence and antimicrobial resistance profiles of Salmonella serotypes, Campylobacter and Yersinia spp. isolated from retail chicken and beef, Tehran, Iran[J]. Food Control, 2010, 21(4): 388-392. DOI:10.1016/j.foodcont.2009.06.001.
[22] YANG B, ZHENG J, BROWN E W, et al. Characterisation of antimicrobial resistance associated integrons and mismatch repair gene mutations in Salmonella serotypes[J]. International Journal of Antimicrobial Agents, 2009, 33: 120-124. DOI:10.1016/ j.ijantimicag.2008.08.016.
[23] 曾振灵, 林居纯. 氟喹诺酮类药物耐药性的研究进展[J]. 广东畜牧兽医科技, 2005, 30(1): 11-15. DOI:10.3969/j.issn.1005-8567.2005.01.003.
[24] EAVES D J, RANDALL L, GRAY D T, et al. Prevalence of mutation within the quinolone resistance-determining region of gyrA, gyrB, parC and parE and association with antibioticresistance in quinolone-resistant Salmonella enterica[J]. Antimicrob Agents Chemother, 2004, 48(10): 4012-4015. DOI:10.1128/aac.48.10.4012-4015.2004.
[25] CASIN I, BREUIL J, DARCHIS J P, et al. Fluoroquinolone resistancelinked to GyrA, GyrB, and ParC mutations in Salmonella enterica typhimurium isolates in humans[J]. Emerging Infectious Diseases, 2003, 9(11): 1455-1457. DOI:10.3201/eid0911.030317.
[26] CHEN S, CUI S, MCDERMOTT P F, et al. Contribution of target gene mutation and efflux to decrease susceptibility of Salmonella enterica serovar typhimurium to fluoroquinolones and other antimicrobial[J]. Antimicrobial Agents & Chemotherapy, 2007, 51(1): 535-542. DOI:10.1128/aac.00600-06.
[27] 苑丽. 鸡源分离菌超广谱β-内酰胺酶基因分型和整合子分子特征[D].郑州: 河南农业大学, 2010: 80-88. DOI:10.7666/d.y1729058.
Quinolone Resistance and Related Genes in Salmonella hadar
WU Haotian, WU Yun*, YIN Mingyuan, GULINAZI, WANG Wei, ZHANG Yanan, TIAN Ge, MA Wenrui (College of Food Science and Pharmaceutical Science, Xinjiang Agricultural University, Ürümqi 830052, China)
Abstract:Objective: To characterize the antimicrobial resistance to 2 kinds of quinolones and the related genes in 21 Salmonella hadar isolates obtained from poultry and meat samples from some farmer’s markets in Ürümqi for the purpose of getting a better understanding of the pathways of generation and transmission of antimicrobial resistance and consequently ensuring food safety. Methods: The drug sensitivity of Salmonella hadar was evaluated by agar dilution method. In addition, PCR and gene sequencing were used to detect the presence of mutations in the quinolone resistance-determining region (QRDR) and plasmid-mediated quinolone resistance genes. Results: The resistance rates of these 21 Salmonella hadar isolates were 100% to nalidixic acid and they were sensitive to ciprofl oxacin. The plasmid-mediated quinolone resistance genes including qnrB (52.3%), qnrA (4.76%), and qnrS (4.76%) were identifi ed. The 21 Salmonella hadar isolates had mutations in the gyrA and parC genes at the same time. Among 21 Salmonella hadar isolates, the common mutations appeared at Ser83Phe and Thr57Ser in the gyrA and parC genes, respectively. Conclusion: There should be concerns about the serious situation of antimicrobial resistance in Salmonella hadar isolates from poultry and meat in Ürümqi. In addition, the quinolone resistance-determining region (QRDR) mutations and the plasmid-mediated quinolone resistance genes may affect the antimicrobial resistance of Salmonella hadar.
Key words:Salmonella; quinolone drugs; drug resistance; polymerase chain reaction (PCR); drug resistance gene
DOI:10.7506/spkx1002-6630-201617025
中图分类号:TS201.3
文献标志码:A
文章编号:1002-6630(2016)17-0150-06
收稿日期:2015-12-27
基金项目:新疆维吾尔自治区高技术研究发展计划项目(201317106)
作者简介:吴浩天(1994—),男,硕士研究生,研究方向为食品生物技术。E-mail:1076650961@qq.com
*通信作者:武运(1965—),女,教授,硕士,研究方向为食品生物技术。E-mail:wuyunster@sina.com
引文格式:








