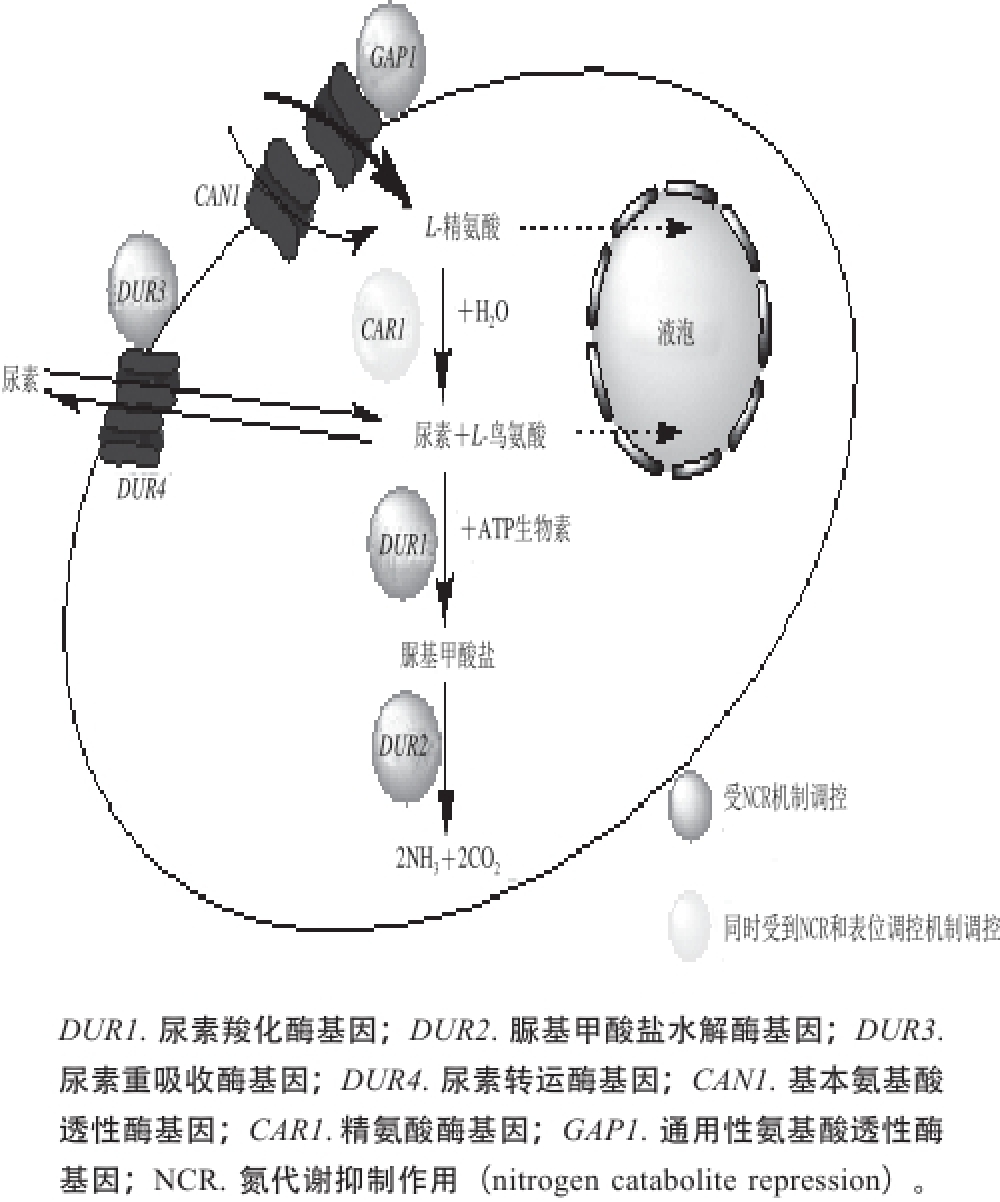
图1 精氨酸、尿素在酿酒酵母细胞内的代谢调控机制
Fig. 1 Proposed metabolism of urea and arginine by S. cerevisiae
焦志华,陈启和 *
(浙江大学生物系统工程与食品科学学院,浙江 杭州 310058)
摘 要:氨基甲酸乙酯(ethyl carbamate,EC)又称脲烷,是一种广泛存在于发酵酒精饮品中的氨基甲酰类物质。由于EC目前被证实能对实验动物造成多位点致癌,并且对人类具有潜在致癌性,因此其对发酵食品安全的影响是目前发酵工业中面临的重要问题。EC主要由氨基甲酰类物质和乙醇自发作用形成,其中重要的前体物包括尿素和瓜氨酸,并且二者均来源于精氨酸的分解代谢,因此对这三者代谢调控机制的研究能够为EC抑制途径的探索提供新的思路,本文主要就尿素、瓜氨酸及精氨酸分别在酿酒酵母及乳酸菌中的代谢调控机制进行系统的概述,并对未来EC的相关研究进行展望。
关键词:氨基甲酸乙酯;尿素;瓜氨酸;精氨酸;代谢调控机制
发酵酒精饮品不仅风味独特,还含有许多对人体健康起调控作用的营养因子,目前被世界各地的消费者大量饮用。事实上,发酵酒精饮品作为传统发酵的一个重要分支,主要是由一些发酵微生物所分泌的降解酶将发酵底物分解成为小分子的营养物质所形成 [1]。尽管不同发酵酒精饮品中所包含的营养物质不同,但发酵过程大致相似,都包含一个复杂的微生物体系。复杂的发酵环境使得发酵酒精饮品为消费者提供大量有益营养物质的同时,难免因副反应的存在而产生对人体有害的物质,目前被大量报道的有生物胺、亚硝胺及氨基甲酸乙酯(ethyl carbamate,EC)等,有趣的是这些物质都是由发酵过程中含氮物质的不完全代谢产生 [2]。
据流行病与统计学调查发现,发酵酒精饮品中的EC能够对实验动物造成多位点致癌,并且对人体具有潜在致癌性。2007年EC被国际癌症研究机构提升为2A级别 [3]。目前针对EC的致癌机理已有相当系统且深入的研究,主要归纳为3 种机制:1)EC的代谢物可以与核酸形成化合物 [4];2)EC的代谢物能够使DNA脱嘌呤 [5];3)EC本身可以诱导姐妹染色体的互换。随着对EC致癌性的认识,加拿大首先对进口发酵酒精饮品中EC的最高浓度作出限定,之后西欧及美洲国家相继制定了各种发酵酒精饮品中EC的限定浓度 [6]。随着各国对发酵酒精饮品中EC的重视,相应的分析检测方法也在不断改进,固相微萃取结合气相色谱-质谱联用技术已经能满足较低浓度的检测要求 [7-8],高效液相色谱及傅里叶红外光谱的应用大大提高了EC的检测效率 [9-10]。通过对各种发酵酒精饮品中EC浓度的检测以及比较,研究结果发现水果白兰地及中国的传统黄酒中EC的浓度较高 [11-12]。
发酵酒精饮品中EC主要是由乙醇和氨甲酰类化合物自发作用形成,前体物主要有尿素和瓜氨酸。目前对EC的抑制途径主要集中在发酵原料及发酵过程的优化选择、外源添加脲酶、基因工程手段修饰发酵菌株以及外源添加EC降解酶等手段 [13-16]。发酵原料及发酵过程的优化对EC浓度的降低贡献较小,不适用于规模较小的发酵厂;外源添加酸性脲酶容易受到发酵液中多种物质的干扰,成本较高 [17]。采用基因工程手段能够为EC的调控提供有效且安全的途径,其主要机理在于抑制EC前体物的形成,因此对于EC前体物的代谢调控机制的研究能够为EC抑制方法提供新的思路。目前对于EC致癌机理、检测方法、形成途径、抑制策略已有大量的综述,而对于发酵酒精饮品中EC前体物的代谢调控机制的概述却很少,本文将着重概括EC前体物(尿素、瓜氨酸、精氨酸)的代谢调控机制,并为基因工程手段改造发酵菌株提供新的靶点及方向。
尿素被认为是各种发酵饮品中最主要的EC前体物 [16]。科研工作者们目前在葡萄酒、中国黄酒和日本的米酒方面,对尿素在酿酒酵母中的代谢进行了大量研究。发酵饮品中尿素一方面来自于发酵原料,另一方面来源于精氨酸酶催化作用下的精氨酸降解 [18]。实际发酵环境下生成的尿素会在细胞内积累,当浓度较高的时候,通过易化扩散的方式排出到细胞外 [19]。

图1 精氨酸、尿素在酿酒酵母细胞内的代谢调控机制
Fig. 1 Proposed metabolism of urea and arginine by S. cerevisiae
尿素在细胞内的代谢主要是通过尿素羧化酶和脲基甲酸盐水解酶来完成(图1),尿素首先在尿素羧化酶的作用下降解为脲基甲酸盐,进而在脲基甲酸盐水解酶的作用下,降解为铵盐和二氧化碳 [20]。其中尿素羧化酶和脲基甲酸盐水解酶是由多功能基因DUR1/2编码,且DUR1/2基因的转录受NCR的调控,即较好氮源存在时,DUR1/2基因的转录被抑制 [21]。在实际的发酵条件下尿素的降解也是受到氮代谢抑制的,因为发酵原料中,大量存在的精氨酸相对于尿素来讲是一种更好的氮源。
尽管精氨酸降解产生的尿素不能被酿酒酵母立即分解代谢,酿酒酵母具有将分泌到胞外的尿素重吸收的功能。酿酒酵母对尿素的重吸收功能对于降低发酵饮品中EC浓度具有重要意义 [15]。目前认为,尿素从胞外转运到胞内主要通过两种方式 [22]:当胞外尿素浓度高于0.5 mmol/L时,尿素进入酿酒酵母细胞内通过易化扩散的方式,由DUR4基因编码的蛋白参与该过程,且不需要能量的存在;当胞外尿素的浓度较低时(K m=14 μmol/L),尿素通过主动运输的方式转运进入细胞内,这个过程需要钠-尿素协同载体,需要能量的参与,载体由受氮代谢抑制调控的DUR3编码 [23]。另外,已有研究报道实际发酵条件下,发酵液中尿素浓度低于异化扩散K m值,因此实际发酵条件下,尿素的重吸收主要是通过主动运输完成 [24]。
影响尿素形成、释放及重新利用的因素主要包括发酵原料的成分、发酵菌株和酿造条件等 [25]。由于尿素主要来源于精氨酸的降解,因此精氨酸吸收降解速率越高的菌株,其释放到胞外的尿素就越多;酒精体积分数高会抑制酿酒酵母细胞的生长,进而影响精氨酸的吸收利用,这使得释放到胞外的尿素就会相对较少;另外研究报道,当发酵培养基中添加的精氨酸浓度越高时,释放到胞外的尿素就越多,但是酵母细胞重新利用的尿素却不多。这主要是由于精氨酸相对于尿素而言更容易被酵母细胞吸收利用,因此在发酵培养基中添加高浓度精氨酸会影响尿素的重新利用 [19]。
尽管在发酵饮品中酿酒酵母产生的尿素是EC形成的最主要前体物,瓜氨酸对EC形成也起到一定的作用。研究表明在葡萄酒的酿造过程中,虽然大部分精氨酸是由酿酒酵母分解代谢,但在酒精发酵后期,发酵液中仍会含有0.1~2.3 g/L的精氨酸 [26],这部分精氨酸可以通过乳酸发酵途径来降解。发酵液中的瓜氨酸主要来自于乳酸菌对精氨酸的降解作用 [27],具体代谢途径如图2所示。

图2 瓜氨酸在乳酸菌细胞中的代谢途径
Fig. 2 Proposed metabolism of urea and citrulline by lactic acid bacteria
首先,精氨酸在精氨酸脱亚胺酶的作用下降解为瓜氨酸和氨,瓜氨酸可以和磷酸基在鸟氨酸氨甲酰基转移酶的作用下生成鸟氨酸和氨甲酰基磷酸(ADI途径),与精氨酸降解第一步不同的是该过程是一个可逆反应。生成的氨甲酰基磷酸和ADP在氨基甲酸激酶的作用下进一步生成ATP、NH
3和
 。从发酵食品的安全方面考虑,该过程产生的瓜氨酸会与乙醇作用生成EC,对人体的健康不利。但是从发酵食品的风味及对发酵菌株本身生长状况而言,该过程产生的ATP和NH
3可以为乳酸菌自身的生长提供能量和调节pH值,并且可以改善发酵酒精饮品的风味
[28]。
。从发酵食品的安全方面考虑,该过程产生的瓜氨酸会与乙醇作用生成EC,对人体的健康不利。但是从发酵食品的风味及对发酵菌株本身生长状况而言,该过程产生的ATP和NH
3可以为乳酸菌自身的生长提供能量和调节pH值,并且可以改善发酵酒精饮品的风味
[28]。
目前已有多种细菌被发现通过ADI途径降解精氨酸,如酒明串珠菌、希氏乳杆菌 [26]和布氏乳杆菌 [29]等。通常乳酸菌和酒球菌被认为是参与乳酸发酵的最主要菌株 [30]。随着发酵过程中产生的NH 3不断增加,发酵环境中的pH值不断增加,当pH>3.5时,就会有更多种类的乳酸菌参与乳酸发酵过程,其中希氏乳杆菌容易造成酒体的腐败 [31],因此在研究过程中需要对其特别注意。
与尿素分泌到细胞外不同的是,瓜氨酸的分泌作用主要是由于瓜氨酸的生成和消耗作用不平衡,即精氨酸在ADI途径第一步中生成瓜氨酸的速率比第二步中瓜氨酸与磷酸基生成鸟氨酸和氨甲酰基磷酸的速率快,因此瓜氨酸与磷酸基的反应被认为是不符合热力学反应规则 [26]。也有研究报道瓜氨酸向胞外分泌是由于能量的解偶联或者能量过溢。尤其是在乳酸菌的对数生长后期和稳定期,精氨酸水解过程生成的ATP远远超过细胞生长需要的能量。另外,研究报道由ATP介导的鸟氨酸氨甲酰基转移酶活性抑制也可能最终引起瓜氨酸的积累 [32]。对于瓜氨酸的重吸收作用,通过对分泌瓜氨酸的乳酸菌自身及其他腐败菌的研究结果表明,许多菌株都具有重吸收瓜氨酸的作用,这对于降低发酵过程中产生的EC具有重要作用,但是目前对这方面的研究还相对较少 [33]。
另外与尿素转运作用不同的是,瓜氨酸的释放需要一个更为复杂的体系。目前研究中报道了几种不同的交换模型,如酒球菌中,瓜氨酸的释放是通过精氨酸/瓜氨酸交换作用完成 [34]。布氏乳杆菌中,瓜氨酸的分泌是通过瓜氨酸/鸟氨酸交换作用完成,即在转运之后,瓜氨酸转变为鸟氨酸 [34]。Morita等 [35]的研究报道,瓜氨酸在布氏乳杆菌CUC-3中的转运是通过瓜氨酸/鸟氨酸反向转运蛋白完成的。另外,Vrancken等 [36]的研究发现发酵乳杆菌IMDO 130101中含有另外一个精氨酸/鸟氨酸(A/O)逆向转运载体,并且其编码基因位于ADI启动子的下游。A/O逆向转运载体在其他的乳酸菌中也有报道 [37]。尽管目前对于瓜氨酸的转运系统已有一定的研究,但是每个转运载体所特有的功能仍然较含糊,需要进一步的研究。该领域的研究可以为瓜氨酸重吸收作用提供新的思路,并且对降低EC作用有重要意义。此外,在精氨酸丰富的发酵培养基中,细胞的溶菌作用也会使得胞内大量的瓜氨酸释放到发酵液中 [33]。
在过去的几十年里,针对EC前体物的研究已有很大进展。值得注意的问题是,EC最主要的前体物——尿素和瓜氨酸,都来自于精氨酸的降解作用。因此阐明精氨酸的代谢调控机制对于EC抑制途径具有重要意义。
3.1 精氨酸在酿酒酵母中的分布及转运
对于精氨酸在细胞内的分布和代谢,已有的研究结果均表明,精氨酸及其他的一些氨基酸会有部分贮存在液泡内,这部分的氨基酸不同于细胞质中的氨基酸,不会被用来合成蛋白质 [38]。另外精氨酸在细胞内的分布主要是通过液泡贮存、蛋白质合成、细胞质内少量贮存来维持平衡。研究报道细胞内精氨酸的来源主要有2 种途径:1)液泡中含有大量的蛋白酶,在细胞特定阶段蛋白酶降解大量的蛋白质,生成的氨基酸是液泡内精氨酸的一个重要来源 [39];2)酿酒酵母细胞从培养基中吸收的精氨酸是细胞内精氨酸的最主要来源。与贮存在细胞质中的氨基酸不同的是,液泡中氨基酸的转运过程需要2 步过程:1)精氨酸从培养基中转运到细胞质中;2)精氨酸从细胞质中转运到液泡,这个过程需要一个更为复杂的体系 [40]。
目前对于酿酒酵母细胞中精氨酸转运酶的认识主要是基于Grenson等 [41-42]的一系列相关研究。通常包括基本氨基酸透性酶和通用性氨基酸透性酶。基本氨基酸透性酶是由CANI基因编码的,它可以识别所有的基本氨基酸,其中精氨酸的K m值为10 -5mol/L。通用性氨基酸透性酶由GAPI基因编码,受氮代谢抑制作用的调控,与基本氨基酸透性酶相比较,通用性氨基酸透性酶具有更少的特异性和更高的转运效率,其中精氨酸的K m值为7.6×10 -6mol/L。NCR对通用性氨基酸透性酶的调控机制表明,在氮源丰富的条件下,精氨酸的转运通过效率高的通用性氨基酸透性酶进行转运。而在氮源不足的条件下,精氨酸的转运依赖于基本性氨基酸透性酶。
研究报道,酿酒酵母细胞中精氨酸大量贮存在液泡中这一现象主要与多聚磷酸盐有关,可能的机制为多聚磷酸盐与液泡膜上选择性透性酶结合,最终导致精氨酸被限制在液泡内 [43]。对于精氨酸在液泡内的转运,Boller等 [40]的最初研究结果表明,内源性精氨酸的外流和精氨酸的吸收是同时存在的,但是当外部的精氨酸被消耗完时,贮存在液泡中的精氨酸作为储备氮源不会转运到液泡外。目前研究报道有多种转运模型参与了精氨酸的交换。首先是Ohsumi等 [44]提出主动运输参与精氨酸交换,该过程依赖能量的存在,并且具有较高的特异性,精氨酸的K m值为0.6 mmol/L [40]。另外位于酿酒酵母液泡膜的囊泡中的精氨酸/组氨酸交换转运体系也参与了精氨酸在液泡内的转运 [45]。
3.2 精氨酸在酿酒酵母细胞中的代谢及调控机制
精氨酸在酿酒酵母细胞内的代谢途径主要包括精氨酸水解成为鸟氨酸和尿素、鸟氨酸转化为谷氨酸盐以及尿素降解为谷氨酸盐和二氧化碳(图1) [46]。其中前面两步分别是在精氨酸酶和鸟氨酸转氨酶的催化作用下完成。值得注意的是精氨酸的水解作用并非伴随发酵过程的始终。事实上,精氨酸的代谢受到多种调控机制的影响。目前大量的研究结果均表明NCR是调控精氨酸代谢的最主要因素,并且对EC的形成具有间接影响。作为一个转录调控体系,NCR将氮源分为不同的等级。即酿酒酵母选择性利用较好氮源如谷氨酸盐、天冬酰胺和铵,同时抑制不太好氮源的利用,如精氨酸、瓜氨酸及尿素等 [47]。在实际的发酵环境中,因为较好氮源比较缺乏,而精氨酸存在较多,因此在发酵后期,精氨酸作为主要氮源被大量降解 [48-49]。

图3 Gln3p在NCR中的具体调控作用
Fig. 3 Regulation mechanism of Gln3p in NCR pathway
目前酿酒酵母中NCR调控氮源的代谢主要是通过调控因子及调控蛋白如Gln3p、Gzf3p、Gat1p、Dal80p、Nil1p、Nil2p和Ure2p的全局调控作用而实现,其中Gln3p、Gat1p和Nil1p为阳性转录因子,Nil2p、Gzf3p和Dal80p是阴性转录因子,即在Gln3p、Nil1P和Gat1p蛋白存在的条件下,尿素、精氨酸等氮源的转录被激活,而在Dal80p、Nil2P和Gzf3p蛋白存在的条件下,较差氮源的转录被抑制 [50]。另外Gzf3p和Gat1p蛋白主要在NCR状态下发挥作用,而Gln3p和Dal80p蛋白主要在去阻遏的情况下发挥作用 [50]。不同调控因子分别对特定的氮源代谢酶及转运酶起调控作用,同时各调控因子之间也起到相互调控作用 [51]。目前许多调控因子的具体调控机制还在不断的探索中,其中调控因子Gln3p在NCR及去阻遏条件下的信号转导途径已被阐明(图3),在NCR的条件下,上游调控因子Tor1p可以与Gln3p蛋白结合,使得Gln3p中的一些丝氨酸磷酸化,磷酸化的Gln3p能够与Ure2p结合形成复合物并且被限制在细胞质中,不能进入细胞核内激活与精氨酸、尿素等降解有关的酶。在去阻遏的条件下,磷酸化的Gln3p蛋白容易在磷酸酶Sit4p及Cip的作用下去磷酸化,并且与调控因子Srp1p结合进而转运进入细胞核内 [16],Gln3p蛋白在细胞核内能够激活与氮代谢抑制有关的启动子,进而激活相关氮源代谢酶及转运酶基因的转录及表达,目前已被报道的转运酶基因有DAL5、DAL7、PUT1和GLN1 [52]。
另外,有研究报道,添加雷帕霉素靶蛋白(target of rapamycin,TOR)途径抑制剂也会使得酿酒酵母细胞处于去阻遏状态,并且该作用是由于与TOR相关的Sit4磷酸酶对Gln3p的去磷酸化起重要作用 [53]。但是目前越来越多的研究表明,TOR信号途径与氮代谢抑制之间不存在信号转导作用,首先,亮氨酸饥饿所引起的TorC1酶活力下降并没有引起Gln3p从细胞核中转移到核外 [54]。其次,当通过改变某个氨基酸对Ure2p和Gln3p进行突变时,Gln3p对不同氮源的作出的反应没有影响,然而突变后添加雷帕霉素后却不能再使Gln3p的位置发生变化 [55-57]。最后,TorC1的激活依赖Gtr-Ego,而Gln3p在较好氮源存在条件下转移到细胞核外却不依赖Gtr-Ego [58]。这说明TOR信号途径和氮代谢抑制途径可能是2 条平行的代谢途径。因此未来的研究中需要对氮代谢抑制途径中的信号转导做进一步的研究,这将为EC的抑制提供更多的借鉴。
另一个相关的精氨酸代谢调控机制为“epiarginasic regulation” [59],值得注意的是该调控途径仅在精氨酸和鸟氨酸同时存在的前提下才起作用,该调控机制的作用机制主要是精氨酸酶和鸟氨酸氨甲酰基转移酶可逆性结合,精氨酸酶保持其原有活性不变,而鸟氨酸氨甲酰基转移酶完全丧失其活性作用。因此在此调控机制作用下,当精氨酸进行降解时,其合成作用受到抑制。这也与精氨酸的合成作用与水解作用不同时进行相一致,因为该循环对酿酒酵母细胞生长代谢没有有益作用,并且消耗能量。另外研究报道,该调控机制的存在与酵母细胞内能量状态有关 [60]。精氨酸合成途径中的一些中间产物如瓜氨酸和氨基甲酰磷酸也会与乙醇作用形成EC,但是在实际的发酵环境中,精氨酸的合成途径通常被其分解途径所抑制,因此不需要对其进行深入研究。
通过对精氨酸分布、转运和代谢调控的整体描述,我们会发现一个问题,即大量的精氨酸贮存在液泡中,而精氨酸的降解作用发生在细胞质中,这两者之间看似相互矛盾。一个可能的机制为精氨酸从胞外吸收进入细胞质中的速率高于精氨酸从细胞质中转运到液泡的速率,这使得精氨酸在细胞质中短暂积累,从而使得精氨酸降解作用在细胞质中发生 [61]。
基因工程手段改造发酵菌株能够为发酵酒精饮品中EC的调控提供安全有效且经济的方案,该途径很大程度上依赖于对EC前体物代谢机制的认识。目前对于中国传统发酵黄酒,尿素被公认为最直接的前体物,而精氨酸作为间接前体物,这两者在酿酒酵母及乳酸菌中代谢调控机制的研究将为基因工程手段改良发酵菌株提供新的靶点。
针对酿酒酵母细胞中精氨酸及尿素的代谢调控机制,前期研究主要集中在尿素及精氨酸的胞内分布、代谢途径、分解酶以及氮代谢抑制等方面,而目前研究主要着重于探索氮代谢抑制途径中具体调控因子分别对精氨酸及尿素分解的作用途径。另外由于发酵培养基中添加雷帕霉素能够使酿酒酵母细胞处于氮饥饿状态,因此酿酒酵母中TOR途径及氮源代谢之间关系也是目前研究的热点。尽管当前对于酿酒酵母中氮代谢已有较为系统的研究,但是对于具体的调控机制及信号转导途径仍需要细致地探索。
与酿酒酵母中EC前体物的代谢研究相比较,目前有关这些前体物在乳酸菌中的研究还较少,主要集中在对精氨酸代谢途径的认识上,另外也有研究者对瓜氨酸在乳酸菌胞内积累的机制进行了探索,但就这些氮源在乳酸菌细胞内的代谢调控机制的研究目前还较少见,这需要在未来的研究中不断探索。
参考文献:
[1] STEINKRAUS K H, Fermentations in world food processing[J]. Comprehensive Reviews in Food Science and Food Safety, 2002, 1(1): 23-32. DOI:10.1111/j.1541-4337.2002.tb00004.x.
[2] THIBON C, MARULLO P, CLAISSE O, et al. Nitrogen catabolic repression controls the release of volatile thiols by Saccharomyces cerevisiae during wine fermentation[J]. Federation of European Microbiological Societies Yeast Research, 2008, 8(7): 1076-1086. DOI:10.1111/j.1567-1364.2008.00381.x.
[3] BAAN R, STRAIF K, GROSSE Y, et al. Carcinogenicity of alcoholic beverages[J]. The Lancet Oncology, 2007, 8(4): 292-293.
[4] GUENGERICH F P, KIM D H. Enzymic oxidation of ethyl carbamate to vinyl carbamate and its role as an intermediate in the formation of 1, N6-ethenoadenosine[J]. Chemical Research in Toxicology, 1991, 4(4): 413-421. DOI:10.1021/tx00022a003.
[5] SAKANO K, OIKAWA S, HIRAKU Y, et al. Metabolism of carcinogenic urethane to nitric oxide is involved in oxidative DNA damage[J]. Free Radical Biology and Medicine, 2002, 33(5): 703-714. DOI:10.1016/S0891-5849(02)00969-3.
[6] ALEXANDER J, AUOUNSSON G A, BENFORD D, et al. Ethyl carbamate and hydrocyanic acid in food and beverages-scientific opinion of the panel on contaminants[J]. EFSA Journal, 2007, 5(10): 1-44.
[7] HORII S, GOTO K. Determination of ethyl carbamate in Sake using headspace solid phase microextraction[J]. Journal of the Institute of Brewing, 2010, 116(2): 177-181. DOI:10.1002/j.2050-0416.2010. tb00415.x.
[8] ZHANG Y, ZHANG J. Optimization of headspace solid-phase microextraction for analysis of ethyl carbamate in alcoholic beverages using a face-centered cube central composite design[J]. Analytica Chimica Acta, 2008, 627(2): 212-218. DOI:10.1016/j.aca.2008.08.014.
[9] FU M L, LIU J, CHEN Q H, et al. Determination of ethyl carbamate in Chinese yellow rice wine using high-performance liquid chromatography with fluorescence detection[J]. International Journal of Food Science & Technology, 2010, 45(6): 1297-1302. DOI:10.1111/j.1365-2621.2010.02279.x.
[10] JARYJ E, LORENZ K, SPANGENBERG B. A simple method for the quantification of urethane in spirits[J]. Journal of Liquid Chromatography & Related Technologies, 2008, 31(13): 1969-1976. DOI:10.1080/10826070802197370.
[11] WU P, PAN X, WANG L, et al. A survey of ethyl carbamate in fermented foods and beverages from Zhejiang, China[J]. Food Control, 2012, 23(1): 286-288. DOI:10.1016/j.foodcont.2011.07.014.
[12] HA M S, KWON K S, KIM M, et al. Exposure assessment of ethyl carbamate in alcoholic beverages[J]. Journal of Microbiology and Biotechnology, 2006, 16(3): 480-483.
[13] WU D, LI X, LU J, et al. Constitutive expression of the DUR1, 2 gene in an industrial yeast strain to minimize ethyl carbamate production during Chinese rice wine fermentation[J]. Federation of European Microbiological Societies Microbiology Letters, 2016, 363(1): 174-183. DOI:10.1093/femsle/fnv214.
[14] YANG Y, KANG Z, ZHOU J, et al. High-level expression and characterization of recombinant acid urease for enzymatic degradation of urea in rice wine[J]. Applied Microbiology and Biotechnology, 2015, 99(1): 301-308. DOI:10.1007/s00253-014-5916-z.
[15] LI X, SHEN C, WU D, et al. Enhancement of urea uptake in Chinese rice wine yeast strain N85 by the constitutive expression of DUR3 for ethyl carbamate elimination[J]. Journal of the Institute of Brewing, 2015, 121(2): 257-261. DOI:10.1002/jib.208.
[16] ZHAO X, ZOU H, FU J, et al. Metabolic engineering of the regulators in nitrogen catabolite repression to reduce the production of ethyl carbamate in a model rice wine system[J]. Applied and Environmental Microbiology, 2014, 80(1): 392-398. DOI:10.1128/AEM.03055-13.
[17] 焦志华, 陈启和. 发酵饮品中氨基甲酸乙酯的研究进展[J]. 食品安全质量检测学报, 2013, 4(5): 1299-1307.
[18] WU D, LI X, SHEN C, et al. Decreased ethyl carbamate generation during Chinese rice wine fermentation by disruption of CAR1 in an industrial yeast strain[J]. International Journal of Food Microbiology, 2014, 180: 19-23. DOI:10.1016/j.ijfoodmicro.2014.04.007.
[19] AN D, OUGH C S. Urea excretion and uptake by wine yeasts as affected by various factors[J]. American Journal of Enology and Viticulture, 1993, 44(1): 35-40.
[20] WHITNEY P A, COOPER T G. Urea carboxylase and allophanate hydrolase two components of adenosine triphosphate: urea amidolyase in Saccharomyces cerevisiae[J]. Journal of Biological Chemistry, 1972, 247(5): 1349-1353.
[21] ZHAO X, ZOU H, FU J, et al. Nitrogen regulation involved in the accumulation of urea in Saccharomyces cerevisiae[J]. Yeast, 2013, 30(11): 437-447. DOI:10.1002/yea.2980.
[22] SUMRADA R, GORSKI M, COOPER T. Urea transport-defective strains of Saccharomyces cerevisiae[J]. Journal of Bacteriology, 1976, 125(3): 1048-1056.
[23] COOPER T G, SUMRADA R. Urea transport in Saccharomyces cerevisiae[J]. Journal of Bacteriology, 1975, 121(2): 571-576
[24] MONTEIRO F F, BISSON L F. Amino acid utilization and urea formation during vinification fermentations[J]. American Journal of Enology and Viticulture, 1991, 42(3): 199-208.
[25] OUGH C S, STEVENS D, SENDOVSKI T, et al. Factors contributing to urea formation in commercially fermented wines[J]. American Journal of Enology and Viticulture, 1990, 41(1): 68-73.
[26] ARENA M E, SAGUIR F M, de NADRA M C M. Arginine, citrulline and ornithine metabolism by lactic acid bacteria from wine[J]. International Journal of Food Microbiology, 1999, 52(3): 155-161. DOI:10.1016/S0168-1605(99)00133-6.
[27] SPONHOLZ W R. The breakdown of arginine by lactic acid bacteria and its relation to ethyl carbamate production[J]. BiolOggi, 1992, 6: 15-24.
[28] WIBOWO D, ESCHENBRUCH R, DAVIS C R, et al. Occurrence and growth of Lactic acid bacteria in wine: a review[J]. American Journal of Enology and Viticulture, 1985, 36(4): 302-313.
[29] LIU S Q, PRITCHARD G G, HARDMAN M J, et al. Citrulline production and ethyl carbamate (urethane) precursor formation from arginine degradation by wine lactic acid bacteria Leuconostocoenos and Lactobacillus buchneri[J]. American Journal of Enology and Viticulture, 1994, 45(2): 235-242.
[30] GRANCHI L, PAPERI R, ROSELLINI D, et al. Strain variation of arginine catabolism among malolactic Oenococcus oeni strains of wine origin[J]. Italian Journal of Food Science, 1998, 10(4): 351-357.
[31] CLAISSE O, LONVAUD-FUNEL A. Primers and a specific DNA probe for detecting lactic acid bacteria producing 3-hydroxypropionaldehyde from glycerol in spoiled ciders[J]. Journal of Food Protection, 2001, 64(6): 833-837.
[32] RUSSELL J B, COOK G M. Energetics of bacterial growth: balance of anabolic and catabolic reactions[J]. Microbiological Reviews, 1995, 59(1): 48-62.
[33] de ORDUÑA R M, LIU S Q, PATCHETT M L, et al. Ethyl carbamate precursor citrulline formation from arginine degradation by malolactic wine Lactic acid bacteria[J]. Federation of European Microbiological Societies Microbiology Letters, 2000, 183(1): 31-35. DOI:10.1111/ j.1574-6968.2000.tb08929.x.
[34] de ORDUÑA R M, PATCHETT M L, LIU S Q, et al. Growth and arginine metabolism of the wine lactic acid bacteria Lactobacillus buchneriand Oenococcusoeni at different pH values and arginine concentrations[J]. Applied and Environmental Microbiology, 2001, 67(4): 1657-1662. DOI:10.1128/AEM.67.4.1657-1662.2001.
[35] MORITA H, TOH H, FUKUDA S, et al. Comparative genome analysis of Lactobacillus reuteri and Lactobacillus fermentum reveal a genomic island for reuterin and cobalamin production[J]. DNA Research, 2008, 15(3): 151-161. DOI:10.1093/dnares/dsn009.
[36] VRANCKEN G, RIMAUX T, WECKX S, et al. Environmental pH determines citrulline and ornithine release through the arginine deiminase pathway in Lactobacillus fermentum IMDO 130101[J]. International Journal of Food Microbiology, 2009, 135(3): 216-222. DOI:10.1016/j.ijfoodmicro.2009.07.035.
[37] MILLS D A, RAWSTHORNE H, PARKER C, et al. Genomic analysis of Oenococcusoeni PSU-1 and its relevance to winemaking[J]. Federation of European Microbiological Societies Microbiology Reviews, 2005, 29(3): 465-475. DOI:10.1016/j.fmrre.2005.04.011.
[38] WIEMKEN A, NURSE P. Isolation and characterization of the aminoacid pools located within the cytoplasm and vacuoles of Candida utilis[J]. Planta, 1973, 109(4): 293-306. DOI:10.1007/BF00387098.
[39] WIEMKEN A, SCHELLENBERG M, URECH K. Vacuoles: the sole compartments of digestive enzymes in yeast (Saccharomyces cerevisiae)?[J]. Archives of Microbiology, 1979, 123(1): 23-35. DOI:10.1007/BF00403499.
[40] BOLLER T, DÜRR M, WIEMKEN A. Characterization of a specif c transport system for arginine in isolated yeast vacuoles[J]. European Journal of Biochemistry, 1975, 54(1): 81-91. DOI:10.1111/j.1432-1033.1975.tb04116.x.
[41] GRENSON M. Study of the positive control of the general amino-acid permease and other ammonia-sensitive uptake systems by the product of the NPR1 gene in the yeast Saccharomyces cerevisiae[J]. European Journal of Biochemistry, 1983, 133(1): 141-144. DOI:10.1111/j.1432-1033.1983.tb07439.x.
[42] GRENSON M, HOU C, CRABEEL M. Multiplicity of the amino acid permeases in Saccharomyces cerevisiae IV, Evidence for a general amino acid permease[J]. Journal of Bacteriology, 1970, 103(3): 770-777.
[43] DÜRR M, URECH K, BOLLER T, et al. Sequestration of arginine by polyphosphate in vacuoles of yeast (Saccharomyces cerevisiae)[J]. Archives of Microbiology, 1979, 121(2): 169-175. DOI:10.1007/ BF00689982.
[44] OHSUMI Y, ANRAKU Y. Active transport of basic amino acids driven by a proton motive force in vacuolar membrane vesicles of Saccharomyces cerevisiae[J]. Journal of Biological Chemistry, 1981, 256(5): 2079-2082.
[45] SATO T, OHSUMI Y, ANRAKU Y. An arginine/histidine exchange transport system in vacuolar-membrane vesicles of Saccharomyces cerevisiae[J]. Journal of Biological Chemistry, 1984, 259(18): 11509-11511.
[46] BRANDRISS M C, MAGASANIK B. Proline: an essential intermediate in arginine degradation in Saccharomyces cerevisiae[J]. Journal of Bacteriology, 1980, 143(3): 1403-1410.
[47] 张伟平, 赵鑫锐, 堵国成, 等. 酿酒酵母氮代谢物阻遏效应及其对发酵食品安全的影响[J]. 应用与环境生物学报, 2012, 18(5): 862-872. DOI:10.3724/SP.J.1145.2012.00862.
[48] CHIVA R, BAIGES I, MAS A, et al. The role of GAP1 gene in the nitrogen metabolism of Saccharomyces cerevisiae during wine fermentation[J]. Journal of Applied Microbiology, 2009, 107(1): 235-244. DOI:10.1111/j.1365-2672.2009.04201.x.
[49] BELTRAN G, NOVO M, ROZÈS N, et al. Nitrogen catabolite repression in Saccharomyces cerevisiae during wine fermentations[J]. Federation of European Microbiological Societies yeast research, 2004, 4(6): 625-632. DOI:10.1016/j.femsyr.2003.12.004.
[50] ZHAO X, ZOU H, CHEN J, et al. The modification of Gat1p in nitrogen catabolite repression to enhance non-preferred nitrogen utilization in Saccharomyces cerevisiae[J]. Scientif c Reports, 2016, 6: 21603. DOI:10.1038/srep21603.
[51] DEED N K, van VUUREN H J J, GARDNER R C. Effects of nitrogen catabolite repression and di-ammonium phosphate addition during wine fermentation by a commercial strain of S. cerevisiae[J]. Applied Microbiology and Biotechnology, 2011, 89(5): 1537-1549. DOI:10.1007/s00253-011-3084-y.
[52] GEORIS I, TATE J J, COOPER T G, et al. Tor pathway control of the nitrogen-responsive DAL5 gene bifurcates at the level of Gln3 and Gat1 regulation in Saccharomyces cerevisiae[J]. Journal of Biological Chemistry, 2008, 283(14): 8919-8929. DOI:10.1074/ jbcM708811200.
[53] GEORIS I, FELLER A, TATE J J, et al. Nitrogen catabolite repressionsensitive transcription as a readout of Tor pathway regulation: the genetic background, reporter gene and GATA factor assayed determine the outcomes[J]. Genetics, 2009, 181(3): 861-874. DOI:10.1534/ genetics.108.099051.
[54] TATE J J, COOPER T G. Five conditions commonly used to downregulate tor complex 1 generate different physiological situations exhibiting distinct requirements and outcomes[J]. Journal of Biological Chemistry, 2013, 288(38): 27243-27262. DOI:10.1074/jbc. M113.484386.
[55] FELLER A, GEORIS I, TATE J J, et al. Alterations in the Ure2 αCap domain elicit different GATA factor responses to rapamycin treatment and nitrogen limitation[J]. Journal of Biological Chemistry, 2013, 288(3): 1841-1855. DOI:10.1074/jbc.M112.385054.
[56] RAI R, TATE J J, NELSON D R, et al. GLN3 mutations dissociate responses to nitrogen limitation (nitrogen catabolite repression) and rapamycin inhibition of TorC1[J]. Journal of Biological Chemistry, 2013, 288(4): 2789-2804. DOI:10.1074/jbc.M112.421826.
[57] RAI R, TATE J J, SHANMUGANATHAM K, et al. A domain in the transcription activator Gln3 specifically required for rapamycin responsiveness[J]. Journal of Biological Chemistry, 2014, 289(27): 18999-19018. DOI:10.1074/jbc.M114.563668.
[58] TATE J J, GEORIS I, RAI R, et al. GATA factor regulation in excess nitrogen occurs independently of Gtr-Ego complex-dependent TorC1 activation[J]. G3(Bethesda), 2015, 5(8): 1625-1638. DOI:10.1534/ g3.115.019307.
[59] VISSERS S, URRESTARAZU L A, JAUNIAUX J C, et al. Inhibition of ornithine carbamoyltransferase by arginase among yeasts: correlation with energy production, subcellular localization and enzyme synthesis[J]. Microbiology, 1982, 128(6): 1235-1247. DOI:10.1099/00221287-128-6-1235.
[60] WIAME J M. Evolving of arginase-ornithine carbamoyltransferase interaction[C]//Proceedings of the FEBS Meeting. Oxford: Pergamon Press, 1974: 161-177.
[61] JAUNIAUX J C, URRESTARAZU L A, WIAME J M. Arginine metabolism in Saccharomyces cerevisiae: subcellular localization of the enzymes[J]. Journal of Bacteriology, 1978, 133(3): 1096-1107.
Progress in Understanding Metabolic Mechanisms of Ethyl Carbamate Precursors in Fermented Alcoholic Beverages
JIAO Zhihua, CHEN Qihe
*
(College of Biosystems Engineering and Food Science, Zhejiang University, Hangzhou 310058, China)
Abstract:Ethyl carbamate (EC), commonly found in fermented alcoholic beverages, has been verified to be a multisite carcinogen in experimental animals and a potential carcinogen to humans. Because of its threat to human safety, the presence of EC may be a big challenge for the alcoholic beverage industry. EC primarily results from the reaction of ethanol and compounds containing carbamyl groups. The main EC precursors are commonly generated from arginine metabolism by Saccharomyces cerevisiae or Lactobacillus accompanied by the fermentation process. This review comprehensively summarizes the metabolic mechanisms of urea, citrulline, and arginine, which may shed light on new strategies for reducing EC. Meanwhile, this paper presents future prospects of research in this f eld.
Key words:ethyl carbamate; urea; citrulline; arginine; metabolic mechanism
DOI:10.7506/spkx1002-6630-201623043
中图分类号:TS201.6
文献标志码:A
文章编号:1002-6630(2016)23-0260-07
引文格式:
焦志华, 陈启和. 发酵酒精饮品中氨基甲酸乙酯前体物的代谢调控机制研究进展[J]. 食品科学, 2016, 37(23): 260-266.
DOI:10.7506/spkx1002-6630-201623043. http://www.spkx.net.cn
JIAO Zhihua, CHEN Qihe. Progress in understanding metabolic mechanisms of ethyl carbamate precursors in fermented alcoholic beverages[J]. Food Science, 2016, 37(23): 260-266. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201623043. http://www.spkx.net.cn
收稿日期:2016-03-05
基金项目:国家自然科学基金面上项目(31171734;31371825)
作者简介:焦志华(1988—),女,博士研究生,研究方向为发酵食品安全。E-mail:jzhihua@zju.edu.cn
*通信作者:陈启和(1975—),男,教授,博士,研究方向为食品生物安全与生物技术。E-mail:chenqh@zju.edu.cn