钱 辉,陆兆新,张 充,别小妹,赵海珍,吕凤霞*
(南京农业大学食品科技学院,江苏 南京 210095)
摘 要:为提高鱼腥藻脂肪氧合酶(Anabaena sp. PCC 7160 lipoxygenase,Ana-LOX)的热稳定性,探索其热稳定性机制。通过同源建模预测了Ana-LOX的三维结构,并通过分子动力学模拟技术手段分别模拟了Ana-LOX于298 K和320 K条件下在水溶液中的运动轨迹,计算均方根偏移与均方根涨落后,以均方根涨落为热稳定性评价指标,结合作用力分析,预测不稳定区域。结果表明:Ana-LOX的T94、I96、A325~Q327区域柔性较大,是导致Ana-LOX热不稳定的关键区域。通过I96V定点突变后,半衰期提高到野生型的2.5 倍,验证了原预测结果的有效性,从而为脂肪氧合酶分子的热稳定性改造提供了理论依据。
关键词:脂肪氧合酶;同源建模;分子动力学模拟;热稳定性
脂肪氧合酶(Lipoxygenase,LOX,EC1.13.1.13)属于氧化还原酶,广泛存在于动物、植物、藻类、真菌以及少数细菌中 [1]。LOX能够专一催化氧化含有顺,顺-1,4-戊二烯结构的多元不饱和脂肪酸,形成具有共轭双键的脂肪酸氢过氧化物 [2],这些氢过氧化物性质活泼,在食品加工中具有重要的应用价值 [3]。此外,许多不饱和脂肪酸的氢过氧化物是食品风味物质的前体,能够通过酶解进一步转化为酮、醛和醇类,较化学合成香料具有更高的商业价值 [4]。
目前市场上商品级LOX产品大多数以富含LOX的大豆粉作为酶源。大豆中存在多种LOX同工酶,导致LOX的产品质量不稳定 [5]。因此,构建高产重组菌成为发酵法生产LOX的关键。本实验室前期已克隆了鱼腥藻(Anabaena sp. PCC 7120)LOX基因,并在Escherichia coli和Bacillus subtilis中获得了表达 [6-7];后续研究发现其在面粉加工中有着重要的应用价值 [8],并进一步阐释了其强筋漂白机理 [9-10]。但鱼腥藻脂肪氧合酶(Anabaena sp. PCC 7120 lipoxygenase,Ana-LOX)热稳定性较差,限制了其在食品工业中的应用。因此,需要对其热稳定性机制进行解析,从而制定出相应的策略来提高其热稳定性,如loop环的改造 [11]、定点突变 [12]、融合自组装双亲短肽 [13]等。
近年来,大量蛋白质晶体结构被解析及同源建模算法的不断完善,使得利用计算机模拟来完成蛋白质结构与功能的分析工作、更高效地研究热稳定性机制并对其进行分子改造成为可能。Ana-LOX的晶体结构尚无报道,不宜采用基于晶体结构解析来分析其热稳定性。因此,应用计算机辅助技术,采取同源建模、分子动力学(molecular dynamics,MD)模拟等方法,以均方根偏移(root mean square deviation,RMSD)与均方根涨落(root mean square fluctuation,RMSF)值来评估酶分子局部空间结构的改变,探索其热不稳定区域 [14]及酶热稳定性机制。本实验将MD用于LOX的研究,锁定Ana-LOX的热不稳定区域,探索酶热稳定性机制,为LOX的热稳定性改造提供理论依据。
1.1 材料与试剂
pET32a-Ana-LOX重组质粒由南京农业大学酶工程实验室构建。
LB培养基:胰蛋白胨10 g/L、酵母提取物5 g/L、NaCl 10 g/L、pH 7.0,121 ℃条件下灭菌20 min。
限制性内切酶Dnp Ⅰ、primeSTAR聚合酶 宝生物工程(大连)有限公司;氨苄青霉素、异丙基-β-D-硫代半乳糖苷(isopropyl β-D-thiogalactoside,IPTG) 生工生物工程(上海)股份有限公司;亚油酸 美国Sigma公司;其他试剂均为国产分析纯。
1.2 仪器与设备
JY91ⅡDN超声波破碎仪 宁波新芝生物科技股份有限公司;UV-2600紫外-可见分光光度计 日本岛津公司。
1.3 方法
1.3.1 Ana-LOX蛋白三维结构构建
将Ana-LOX基因序列提交phyre2 [15]数据库(http:// www.sbg.bio.ic.ac.uk/phyre2),与晶体结构获得解析的同源蛋白质进行比对,将一致性最高的序列作为同源建模的模板,构建目的蛋白质的三维结构。将构建好的模型提交PSVS服务器(http://psvs-1_5-dev.nesg.org/),用Procheck [16]程序进行模型评估,借助Pymol软件进行结构分析 [17]。
1.3.2 Amber 12软件分子动力学模拟步骤及参数设置
1.3.2.1 模拟体系构建
利用Amber [18]程序包和ff99SB [19]力场,加载小分子gaff力场与金属离子参数,使用Leap模块添加氢原子,选用TIP3P八面体水分子盒模型,蛋白质分子置于10 Å的水溶剂箱内,加入Na +平衡电荷。
1.3.2.2 能量最小化
用sander模块进行3 000步能量最小化,执行最陡下降法500 步后转为共轭梯度法,以消除不合理的能量势垒。
1.3.2.3 加热
对系统加热使其从0 K升温至298 K或320 K,并在保持体积不变的状态下运行50 ps带有位置限制的动力学,将非键相互作用阈值设为8 Å,使用弱耦合的算法来控制温度。
1.3.2.4 平衡
在常压条件下系统分别处在298 K和320 K条件下,进行500 ps的分子动力学模拟,以平衡系统。
1.3.2.5 动力学模拟
恒温恒压系统下进行MD模拟,所用步长为2 fs,以每1 000 步采集一次能量与坐标信息。分析动力学模拟过程中RMSD值变化,再选择RMSD值平衡阶段的数据,计算RMSF值,形成轨迹文件,最后通过VMD软件分析动力学模拟轨迹。
1.3.3 定点突变
为了检验MD预测的准确性,针对模拟预测位点,采取质粒聚合酶链式反应(polymerase chain reaction,PCR)突变法进行定点突变。引物为I96V-f(5’-ACCGAT GTTCCTGACAGCTACTTTTTAG-3’)和I96V-r(5’-TGT CAGGAACATCGGTGGGATCAGTA-3’)。PCR程序为:96 ℃预变性5 min;98 ℃变性10 s,55 ℃退火5 s,72 ℃延伸90 s,循环25 次;72 ℃延伸5 min。全质粒PCR产物经限制性内切酶DpnⅠ消化后转化E. coli BL21(DE3)。
1.3.4 LOX活力测定
采用紫外-分光光度法测酶活力,反应体系3 mL:取pH 9.0的Tris-HCl缓冲液2.79 mL、酶液10 μL、1.733 mmol/L亚油酸钠底物200 μL混合均匀后放入30℃水浴中并开始计时,以1 min内3 mL反应体系在234 nm波长处的吸光度增加0.001为一个酶活力单位(U)。
1.3.5 酶学性质表征
1.3.5.1 半衰期测定
IPTG诱导表达目的蛋白,经镍柱亲和层析纯化后透析去除咪唑。将突变酶与野生型酶液调整至相同质量浓度,在50 ℃条件下保温,每隔5 min取样,然后置于冰上冷却10 min,按照1.3.4节测定LOX酶的残余活力。再以ln(残余活力)对时间作图,并进行线性拟合,所得斜率即为酶在该温度条件下的一级失活常数(k d)。酶在一定温度条件下的半衰期(t 1/2)按以下公式计算。
1.3.5.2 酶相对活力对温度的变化曲线
将缓冲液加热至15、20、30、35、40、45、50、55、60 ℃,然后在各温度条件下加入10 μL待测酶液,测定酶活力。以最适反应温度条件下的酶活力为100%,以不同温度条件下酶的相对活力作图,从而获得该酶相对活力对温度的变化曲线。
1.3.5.3 酶动力学参数的测定
在3 mL反应体系中以亚油酸为底物,以浓度梯度0.047、0.023、0.019、0.014、0.009 mmol/L添加底物,进行酶活力测定。以摩尔消光系数ε 234 nm为2.5×10 4(mol/L·cm) -1,计算产物生成量。采用Lineweaver-Burk双倒数法作图,计算动力学参数 [20]。
2.1 蛋白模型建立与评估
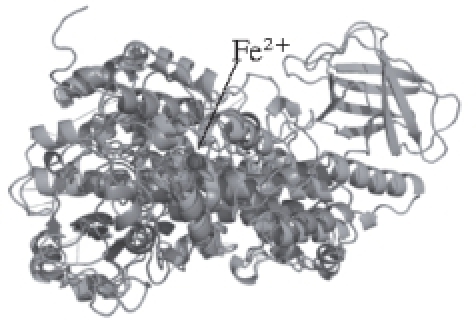
图1 Ana-LOX结构与模板结构比对
Fig. 1 Comparison between Ana-LOX and template structure
采用同源建模技术预测Ana-LOX三维结构。Ana-LOX序列提交phyre2,选择蓝杆藻(Cyanothece sp.)PCC 8801脂肪氧合酶晶体结构(PDB ID:5EK8)为模板 [21],构建成如图1所示的三维空间主体图。将模板与Ana-LOX在pymol中比对,定位出铁离子活性中心。在建模过程中,Ana-LOX与模板序列一致性为20%,低于30%,因此选择phyre2数据库建模,phyre2数据库采取隐马可夫模型比对,并结合了从头折叠模拟,使得即使在序列一致性较低(15%~25%)的情况下,依然可以提供精准的结构信息 [15]。

图2 Ana-LOX拉氏构象图
Fig. 2 Ramachandran plot of Ana-LOX
表1 Procheck的拉氏构象图概要
Table1 Ramachandran plot summary from Procheck

结构质量得分/%最佳合理区74.0额外合理区22.4一般合理区1.7不合理区1.9
采用Procheck对建好的模型结构进行评估,检查最终模型的二面角构象和立体化学参数,获得其拉氏构象图(图2)。结果表明,仅有1.9%的氨基酸位于不合理区域(表1),由此可以判断模型质量较高,该模型可以用于下一步的分子动力学模拟实验。
2.2 RMSD与RMSF值计算
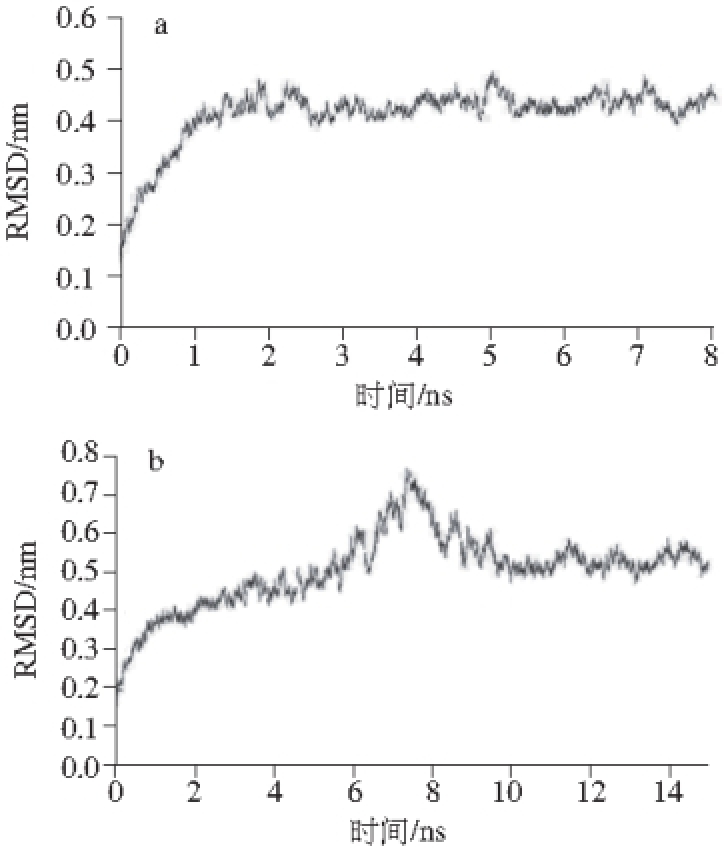
图3 Ana-LOX在298 K(a)与320 K(b)条件下RMSSDD值
Fig. 3 RMSD values for Ana-LOX at 298 (a) and 320 K (b)
RMSD值是蛋白质每个原子运动偏移的均值,反映蛋白质整体的运动状况。RMSD值平衡,反映蛋白质此时运动处于平衡状态,此时即可抽取这一时间段来计算RMSF值。298 K条件下MD 1 ns后Ana-LOX就迅速进入了平衡状态(图3a),取3~8 ns计算RMSF值。320 K条件下15 ns的MD过程中发现6~9 ns时RMSD值发生了跃迁,跃迁后又下降到达平衡状态(图3b)。RMSD值的跃迁说明在6~9 ns时,蛋白质的构象发生了较大的改变,然后又恢复平衡状态,在这一过程中发生构象较大变化的位置可能是潜在的不稳定的临界位点,分别取6~9 ns和10~15 ns计算RMSF值。
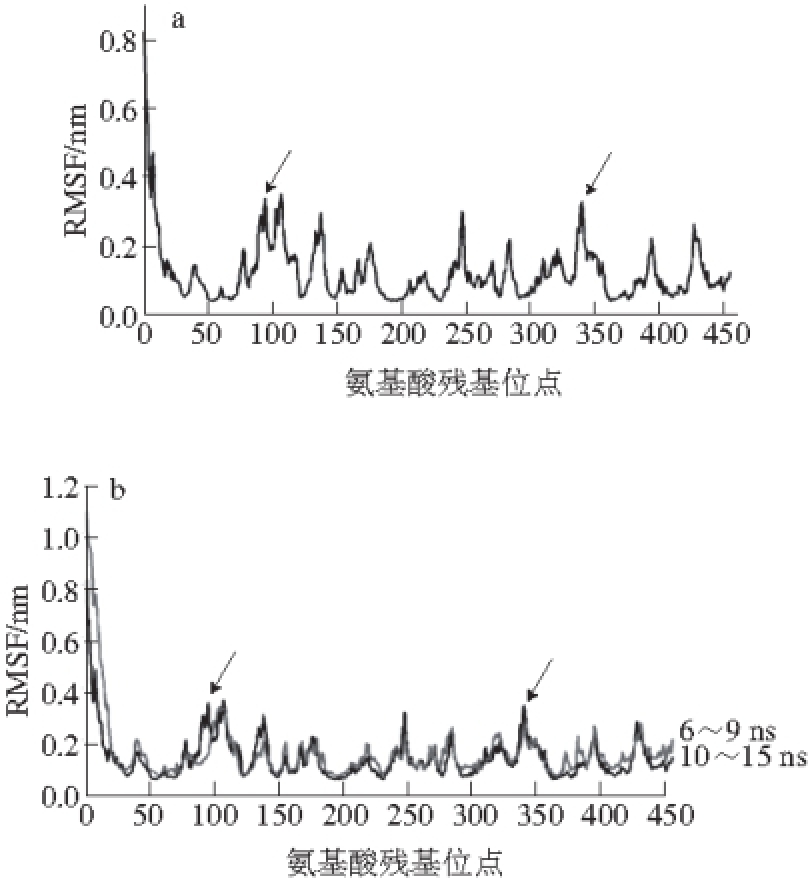
图4 Ana-LOX在298 K(3~8 ns )(a)与320 K(b)条件下RMSF值
Fig. 4 RMSF values for Ana-LOX at 298 (a) and 320 K (b)
如图4所示,RMSF值高的区域振幅较大,可能是潜在柔性较大的不稳定区域。N端的氨基酸RMSF值最高,但这是因N端位于蛋白质末端的一侧处于游离状态,导致天然振幅较大,不将其视为不稳定区域。320 K条件下6~9 ns RMSD发生了跃迁,表明在MD过程中,存在部分位置氨基酸发生较大的位置偏移,使得RMSD发生跃迁。比较320 K条件下6~9 ns与平衡后的RMSF,选择发生较大偏移的位点。选择图4a中出现RMSF峰值的氨基酸残基位点与图4b中6~9 ns条件下出现RMSF值跃迁的残基位点作为候选,逐一分析各氨基酸与周围氨基酸氢键、离子键、π键堆积等相互作用,确定T94、I96、A325~Q327区域(箭头所示)为Ana-LOX不稳定区域(图5)。
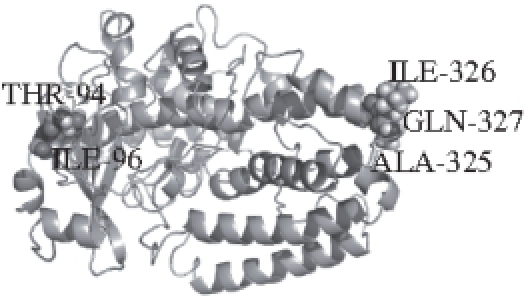
图5 Ana-LOX不稳定区域的位置
Fig. 5 Location of the unstable regions of Ana-LOX
2.3 突变体性质鉴定
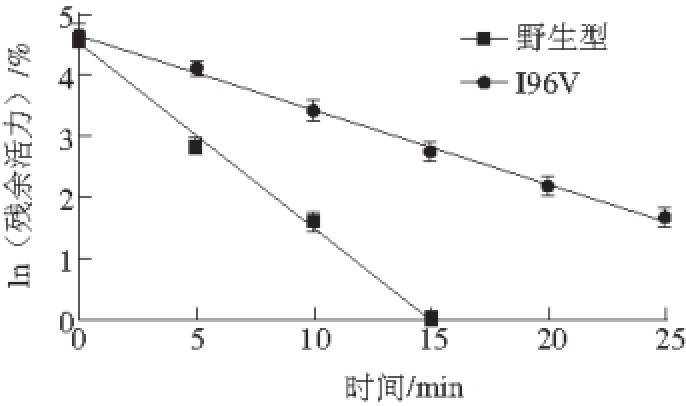
图6 Ana-LOX与I96V突变热失活动力学
Fig. 6 Thermal inactivation kinetics of Ana-LOX and I96V mutant
为了验证预测的准确性,选择MD预测的I96号位点突变成缬氨酸。突变体I96V经纯化,酶热处理每隔5 min取样测酶活力,以ln(残余活力)(y)对时间(x)作图,并进行线性拟合(图6)。野生型为y=-0.298 7x+4.515,R²=0.995 6;突变体I96V为y=-0.121 7x+4.686 6,R²=0.993。
经计算,野生型t 1/2为2.3 min,突变体t 1/2为5.7 min,是野生型的2.5 倍。由此判断MD模拟对酶热敏感区域的预测有效,可进一步对预测出的T94、I96、A325~Q327区域进行局部的刚性增强,如定点突变引入更多的作用力或删除柔性部分,以提高Ana-LOX的热稳定性。
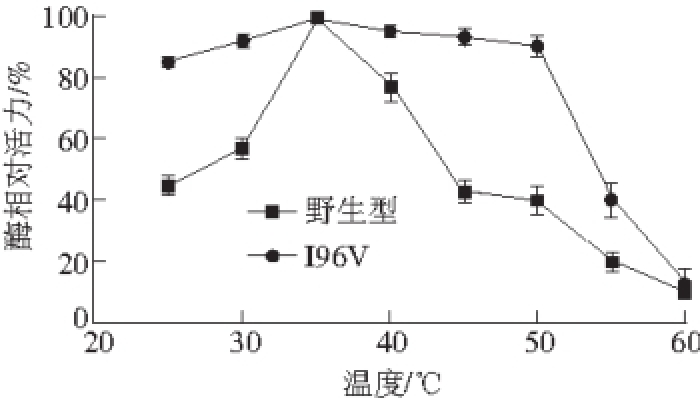
图7 温度对AAnnaa-LOX与I96V酶相对活力的影响
Fig. 7 Effect of temperature on the activity of Ana-LOX and I96V mutant
由图7可知,I96位发生突变后,温度对酶相对活力的影响发生了较大改变,在25~55 ℃范围内突变体可以保持80%以上的活力,而野生型对温度的变化较为敏感,仅在33~37 ℃范围内酶相对活力可以维持80%以上。说明定点突变拓宽了突变体I96V对温度的适应范围。
表2 动力学参数测定
Table2 Kinetic parameters of AAnnaa-LOX and I96V mutant

比活力/(IU/mg)K m/(mmol/L)k cat/(1/s)k cat/K m/(L/(mmol·s))野生型10.4±0.20.056 0±0.003 030.3±1.3 541.1突变体I96V25.8±0.80.011 9±0.000 675.8±1.86 369.2
与野生型相比,突变体I96V在热稳定性提高的同时,酶的比活力也得到了提高,发生了活力与稳定性的共进化。如表2所示,突变体I96V的米氏常数(K m)相对野生型显著降低,因此突变体I96V与底物更具有亲和力,同时催化速率(k cat)均与k cat/K m高于野生型,比活力达到了野生型的2.48 倍。
影响蛋白质热稳定性的因素有疏水相互作用、氢键、离子键、芳香环的相互作用、二硫键、金属离子、氨基酸组成和外界因素等 [22]。但酶的热稳定性往往受到诸多因素的协同影响,因此对于酶热稳定性的改造和机制研究一直是生物学和化学领域的难点。MD提供了在原子水平上探索蛋白质动力学特性的有力方法,作为一种计算机辅助手段,已被用于鉴定蛋白质中可以作为增强稳定性靶点的柔性区域。现研究已表明,将分子动力学模拟应用到蛋白质热稳定性的研究中,可以明显提高蛋白质的热稳定性 [23]。Pikkemaat等 [24]通过MD鉴定出脱卤素酶的一段helix-loop-helix的高柔性区域,引入二硫键后使热稳定性及尿素变性都产生了巨大改变。高新星等 [25]鉴定出枯草芽孢杆菌氨肽酶存在非催化的loop区域,并对其删除,使半失活温度(T 50)从71 ℃提高到77 ℃。而有关鱼腥藻脂肪氧合酶热稳定性提高的分子动力学模拟研究国内外尚未见报道。
本研究为了比较298 K和370 K运动状态下的构象差异,分别在298 K和370 K条件下进行了分子动力学模拟;在MD过程中,当活性中心缺少铁离子时,RMSD无法平衡,蛋白结构发生崩塌(数据未给出),表明LOX活性中心的铁离子对维持结构稳定、发挥催化活性不可缺少。而同源建模构建的模型并没有给出铁离子信息,因此需要将模板与Ana-LOX模型比对,定位出铁离子位置。同时,在MD过程中需要添加小分子力场与金属离子参数。结果显示,在298 K条件下1 ns后RMSD值就达到平衡,而320 K条件下6~9 ns之间RMSD值发生了跃迁后又恢复平衡(图3)。整体上320 K条件下的RMSD高于298 K条件下的RMSD,RMSD变化较大证明在320 K 较高温度下,主链Cα变化较为剧烈,分子柔性变大。比较两个温度下的RMSF,以及320K条件下6~9 ns和平衡时的RMSF,选取RMSF较大的氨基酸,并逐一结合蛋白质结构与作用力分析,预测出T94、I96、A3~Q327区域为Ana-LOX不稳定区域(图5),均位于Loop环,且位于蛋白质表面。研究已表明,影响蛋白质热稳定性的氨基酸也主要位于蛋白质表面 [26],Loop环在蛋白质中未形成有序二级结构的部分,是柔性较大的区域,不利于蛋白质的稳定 [27]。另外,从氨基酸组成角度分析,5 个氨基酸(T94、I96、A3、I326、Q327)中有4 个属于疏水氨基酸,而疏水氨基酸一般分布于蛋白质内部,蛋白表面的疏水氨基酸不利于其稳定性 [28]。其中苏氨酸(threonine,Thr)与谷氨酰胺(glutamine,Gln)也属于热不稳定氨基酸,Thr可以和蛋白质周围的水分子发生相互作用,但在高温条件下结合的水分子会释放出来,从而破坏了蛋白质和水分子结合部位的局部结构,导致整个蛋白质的失稳 [29]。而暴露状态的Gln,在高温条件下容易产生脱酰氨基反应,也不利于蛋白质空间构象保持稳定 [30]。
综上所述,本研究运用Amber 12软件,对Ana-LOX进行了MD模拟,计算了蛋白质分子在水溶液中的运动轨迹,发现Ana-LOX的T94、I96、A3~Q327区域柔性较大,为Ana-LOX不稳定区域,并对I96号位进行了突变,验证了预测的有效性,探索了增强酶热稳定性的机制,结果表明通过蛋白质工程分子改造手段,可以增强上述区域的刚性,提高Ana-LOX热稳定性。
参考文献:
[1] HANSEN J, GARRETA A, BENINCASA M, et al. Bacterial lipoxygenases, a new subfamily of enzymes? a phylogenetic approach[J]. Applied Microbiology and Biotechnology, 2013, 97(11): 4737-4747. DOI:10.1007/s003-013-4887-9.
[2] ALBERTI J C, MARIANI M, de CARAFFA V B B, et al. A functional role identif ed for conserved charged residues at the active site entrance of lipoxygenase with double specificity[J]. Journal of Molecular Catalysis B: Enzymatic, 2016, 123: 167-173. DOI:10.1016/ j.molcatb.2015.10.011.
[3] ZHANG Chong, ZHANG Shuang, LU Zhaoxin, et al. Effects of recombinant lipoxygenase on wheat flour, dough and bread properties[J]. Food Research International, 2013, 54(1): 26-32. DOI:10.1016/j.foodres.2013.05.0.
[4] GOUNARIS Y. Biotechnology for the production of essential oils, flavours and volatile isolates. a review[J]. Flavour and Fragrance Journal, 2010, 25(5): 367-386. DOI:10.1002/ffj.1996.
[5] HUGHES R K, WU Z C, ROBINSON D S, et al. Characterization of authentic recombinant pea-seed lipoxygenases with distinct properties and reaction mechanisms[J]. Biochemical Journal, 1998, 333: 33-43.
[6] 张充, 周孝伟, 吕凤霞, 等. 重组鱼腥藻脂肪氧合酶基因的克隆表达、分离纯化及活性分析[J]. 生物工程学报, 2012, 28(4): 440-456. DOI:10.13345/j.cj b.2012.04.006.
[7] ZHANG C, TAO T, YING Q, et al. Extracellular production of lipoxygenase from Anabaena sp. PCC 7120 in Bacillus subtilis and its effect on wheat protein[J]. Applied Microbiology and Biotechnology, 201 2, 94(4): 949-958. DOI:10.1007/s003-012-3895-5.
[8] 任娣, 谢亚娟, 陆兆新, 等. 重组脂肪氧合酶对面团流变性质及面包品质的影响[J]. 食品科学, 2015, 36(13): 1-6. DOI:10.7506/spkx1002-6630-201513001.
[9] WANG X M, LU F X, ZHANG C, et al. Peroxidation radical formation and regiospecif city of recombinated Anabaena sp. lipoxygenas e and its effect on modifying wheat proteins[J]. Journal of Agricultural and Food Chemistry, 2014, 62(7): 1713-1719. DOI:10.1021/jf503238h.
[10] WANG X M, LU F X, ZHANG C, et al. Effects of recombinated Anabaena sp. lipoxygenase on the protein component and dough property of wheat f our[J]. Journal of Agricultural and Food Chemistry, 2014, 62(40): 9885-9892. DOI:10.1021/jf503238h.
[11] LU X, LIU S, FENG Y, et al. Enhanced thermal stability of Pseudomonas aeruginosa lipoxygenase through modif cation of two highly f exible regions[J]. Applied Microbiology and Biotechnology, 2014, 98(4): 1663-1669. DOI:10.1007/s00253-013-5039-y.
[12] GUO F F, ZHANG C F, BIE X M, et al. Improving the thermostability and activity of lipoxygenase from Anabaena sp. PCC 7120 by d irected evolution and site-directed mutagenesis[J]. Journal of Molecular Catalysis B: Enzymatic, 2014, 107: 23-30. DOI:10.1016/ j.molcatb.2014.05.016.
[13] LU X, LIU S, ZHANG D, et al. Enhanced thermal stability and specif c activity of Pseudomonas aer uginosa lipoxygenase by fusing with self-assembling amphipathic peptides[J]. Applied Microbiology and Biotechnology, 2013, 97(21): 9419-9427. DOI:10.1007/s00253-013-4751-y.
[14] CHRISTENSEN N J, KEPP K P. Stability mechanisms of a thermophilic laccase probed by molecular dynamics[J]. PLoS ONE, 2013, 8(4): e61985. DOI:10.1371/journal.pone.0061985.
[15] KELLEY L A, MEZULIS S, YATES C M, et al. The Phyre2 web porta l for protein modeling, prediction and analysis[J]. Nature Protocols, 2015, 10(6): 845-858. DOI:10.1038/nprot.2015.053.
[16] LASKOWSKI R A, MACARTHUR M W, MOSS D S, et al. Procheck-a program to check the stereochemical quality of protein struct ures[J]. Journal of Applied Crystallography, 1993, 26(2): 26283-26291. DOI:10.1107/S0021889892009944.
[17] SEELIGER D, de GROOT B L. Ligand docking and binding site analysis with PyMOL and Autodock/Vina[J]. Journal of Computer-Aided Molecular Design, 2010, 24(5): 417-422. DOI:10.1007/s10822-010-9352-6.
[18] SALOMON-FERRER R, CASE D A, WALKER R C. An overview of the Amber biomolecular simulation package[J]. Wiley Interdisciplinary Reviews-Computational Molecular Science, 2013, 3(2): 198-210. DOI:10.1002/wcms.1121.
[19] L INDORFF-LARSEN K, PIANA S, PALMO K, et al. Improved sidechain torsion potentials for the Amber ff99SB protein force field[J]. Proteins-Structure Function and Bioinformatics, 2010, 78(8): 1950-1958. DOI:10.1002/prot.22711.
[20] BRODHUN F, CRISTOBAL-SARRAMIAN A, ZABEL S, et al. An iron 13S-lipoxygenase with an α-linolenic acid specific hydroperoxidase activity from Fusarium oxysporum[J]. PLoS ONE, 2013, 8(5): e64919-e64919. DOI:10.1371/journal.pon e.0064919.
[21] NEWIE J, ANDREOU A, NEUMANN P, et al. Crystal structure of a lipoxygenase from Cyanothece sp. may reveal novel features for substrate acquisition[J]. Journal of Lipid Res earch, 2016, 57(2): 276-287. DOI:10.1194/jlr.M064980.
[22] 田健, 王平, 伍宁丰, 等. 理性设计提高蛋白质热稳定性的研究进展[J]. 生物技术进展, 2012, 2(4): 233-239. DOI:10.3969/ j.issn.2095-2341.2012.04.01.
[23] ZHANG C, ZHANG L, ZHANG Y, et al. Study on the stabil ity and enzymatic property improvement of porcine pancreas lipase modif ed by ionic liquids using molecular simulation[J]. Acta Chimica Sinica, 2016, 74(1): 74-80. DOI:10.6023/a15070496.
[24] PIKKEMAAT M G, LINSSEN A B, BERENDSEN H J, et al. Molecular dynamics simulations as a tool for improving protein stability[J]. Protein Engineering, 2002, 15(3): 185-192. DOI:10.1093/ protein/15.3.185.
[25] GAO X X, LIU Z M, CU I W J, et al. Enhanced thermal stability and hydrolytic ability of Bacillus subtilis aminopeptidase by removing the thermal sensitive domain in the non-catalytic region[J]. PLoS ONE, 2014, 9(3): e92357. DOI:10.1371/journal.pone.0092357.
[26] MORLEY K L, KAZLAUSKAS R J. Improving enzyme properties: when are closer mutations better?[J]. Trends in Biotechnology, 2005, 23(5): 231-237. DOI:10.1016/j.tibtech.2005.03.005.
[27] LI B C, YANG G Y, WU L, et al. Role of the N C-Loop in catalytic activity and stability in lipase from Fervidobacterium changbaicum[J]. PLoS ONE, 2012, 7(10): e46881. DOI:10.1371/journal.pone.0046881.
[28] SCHWEHM J M, KRISTYANNE E S, BIGGERS C C, et al. Stability effects of increasing the hydrophobicity of solvent-exposed side chains in staphylococcal nuclease[J]. Biochemistry, 1998, 37(19): 6939-6948. DOI:10.1021/b i9725069.
[29] NAGENDRA H G, SUKUMAR N, VIJAYAN M. Role of water in plasticity, stability, and action of proteins: the crystal structures of lysozyme at very low levels of hydration[J]. Proteins-Structure Function and Bioinformatics, 1998, 32(2): 229-240. DOI:10.1002/ (SICI)1097-0134(19980801)32:2<229::AID-PROT9>3.0.CO;2-F.
[30] KANEKO H, MINAGAWA H, SHIMADA J. Rational design of thermostable lactate oxidase by analyzing quaternary structure and p revention of deamidation[J]. Biotechnology Letters, 2005, 27(22): 1777-1784. DOI:10.1007/s10529-005-3555-2.
Molecular Dynamics Simulation of Thermal Stability Improvement of Lipoxygenase from Anabaena sp. PCC 7160
QIAN Hui, LU Zhaoxin, ZHANG Chong, BIE Xiaomei, ZHAO Haizhen, L☒ Fengxia*
(College of Food Science and Technology, Nanjing Agricultural University, Nanjing 210095, China)
Abstract:Homology modeling was used to predict the 3D structure of lipoxygenase from Anabaena sp. PCC 7160 (Ana-LOX) and to simulate its motion trajectory in aqueous solution by molecular dynamics simulation at 298 and 320 K, respectively. After root mean square deviation (RMSD) and root mean square f uctuation (RMSF) values were calculated, thermal stability was evaluated based on the RMSF value. Force analysis was also performed to predict the unstable region. Results indicated that the T94, I96, and A325 to Q327 areas of Ana-LOX were f exible and they played a crucial role in the thermal instability of Ana-LOX. The half-life of I96V mutant constructed by site-directed mutagenesis increased 2.5 folds compared with that of the wild-type enzyme, conf rming the validity of the prediction. Our research can provide a theoretical basis for the improvement of the thermal stability of lipoxygenase.
Key words:lipoxygenase; homology modeling; molecular dynamics simulation; thermal stability
DOI:10.7506/spkx1002-6630-201702001
中图分类号:TS202.3
文献标志码:A
文章编号:1002-6630(2017)02-0001-06
引文格式:
钱辉, 陆兆新, 张充, 等. 鱼腥藻脂肪氧合酶热稳定性提高的分子动力学模拟[J]. 食品科学, 2017, 38(2): 1-6. DOI:10.7506/spkx1002-6630-201702001. http://www.spkx.net.cn
QIAN Hui, LU Zhaoxin, ZHANG Chong, et al. Molecular dynamics simulation of thermal stability improvement of lipoxygenase from Anabaena sp. PCC 7160[J]. Food Science, 2017, 38(2): 1-6. (in Chinese with English abstract)
DOI:10.7506/spkx1002-6630-201702001. http://www.spkx.net.cn
收稿日期:2016-03-30
基金项目:国家自然科学基金面上项目(31470095)
作者简介:钱辉(1990—),男,硕士研究生,主要从事食品生物技术研究。E-mail:2013108028@njau.edu.cn
*通信作者:吕凤霞(1963—),女,教授,博士,主要从事酶工程研究。E-mail:lufengxia@njau.edu.cn