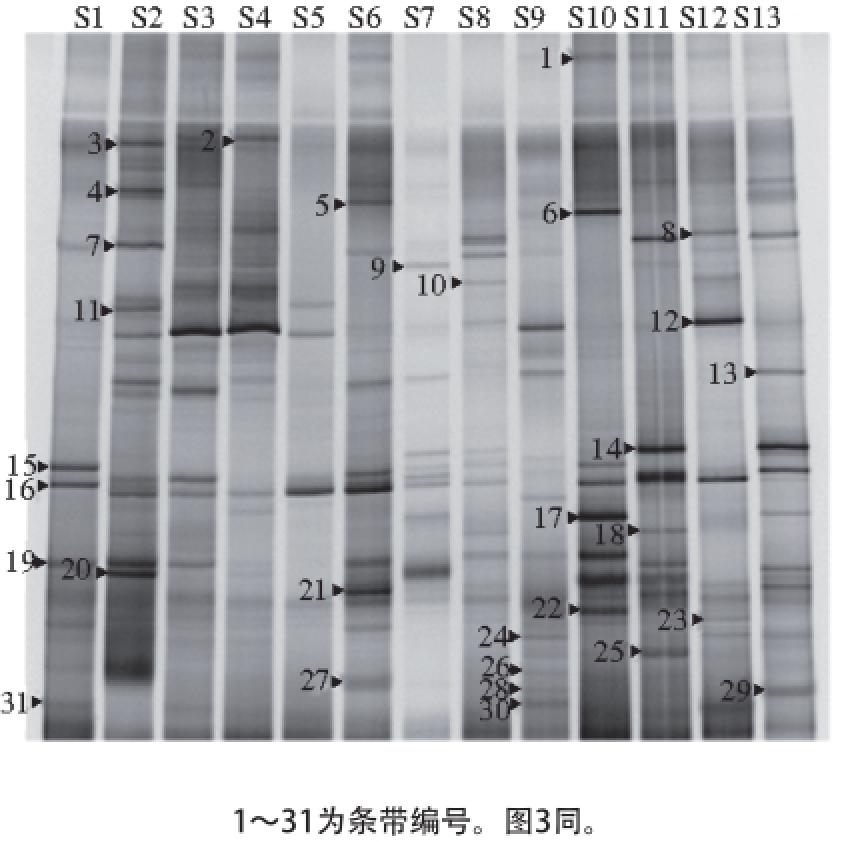
图1 郫县豆瓣酱真菌群落DGGE图
Fig. 1 DGGE fingerprints of fungal communities in broad bean paste
朱永清,李治华,李华佳,王雪涛,董 玲,黄巧莲
(四川省农业科学院农产品加工研究所,四川 成都 610066)
摘 要:以不同品牌的郫县豆瓣酱为研究对象,通过聚合酶链式反应-变性梯度凝胶电泳免培养技术分析样品中微生物真菌菌群结构,并对典型条带进行测序,构建系统发育进化树。结果表明:郫县豆瓣酱真菌菌群种类较丰富,不同品牌的郫县豆瓣酱真菌菌群既包含共有菌群又存在一定差异。假丝酵母(Candida sp.)、曲霉(Aspergillus)、鲁氏酵母(Zygosaccharomyces rouxii)、奥默柯达酵母菌(Kodamaea ohmeri)是优势种群,尤其是黄曲霉(Aspergillus f avus)共有10 个条带,占整个测序条带32.25%,表明郫县豆瓣酱的生产应特别注意防控黄曲霉。
关键词:郫县豆瓣;真菌;聚合酶链式反应-变性梯度凝胶电泳;多样性
郫县豆瓣酱因产于四川省成都市的郫县而得名,已有近300年历史,因其独特的风味闻名于世 [1]。2008年,“郫县豆瓣传统制作技艺”被列为国家非物质文化遗产名录,是到目前为止涉及川菜的唯一的国家级非物质文化遗产。郫县豆瓣制作工艺较为复杂,主要包括制椒、制瓣、后熟等过程,制椒即红辣椒经盐渍制成辣椒胚;制瓣即蚕豆制曲后制成甜豆瓣;后熟即辣椒胚和甜豆瓣按一定比例混合入缸经较长时间(6 个月以上)的翻、晒、露后熟发酵,然后制成成品。
郫县豆瓣酱风味物质的形成十分复杂,其来源主要是原料中的蛋白质、淀粉、脂类等大分子物质经微生物酶水解后产生的各种次级产物和小分子最终产物 [2]。微生物对郫县豆瓣风味品质的形成起着重要作用,一方面,不同种类的微生物所具有的酶系不同,代谢产物就有所差异,这就导致产品的风味有所不同 [3-4];另一方面,微生物菌群新陈代谢所产生的挥发性成分是产品中香味物质的主要来源之一 [4-7]。
传统培养手段主要基于培养基的选择,具有很大的局限性,而据估计大部分微生物是不可培养的。聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reactiondenatured gradient gel electrophoresis,PCR-DGGE)作为一种有效的免培养方法,基于DNA分析,可以有效避免上述不足 [8]。前人已将该方法应用到泡菜 [9]、豆酱 [10]、酱油 [11]、镇江香醋 [12]等传统发酵食品微生物菌群的分析上。
本研究采用PCR-DGGE研究不同品牌的成品郫县豆瓣真菌多样性,加深对郫县豆瓣微生物菌群的认识,为郫县豆瓣酱安全性及改进生产工艺提供依据。
1.1 材料与试剂
13 个郫县豆瓣酱样品分别购自四川省郫县不同厂家,均为传统工艺生产,分别编号为S1~S13。
Gel Extraction Kit试剂盒 美国Omega Bio-Tek公司;溶菌酶 美国Amresco公司;蛋白酶K 德国Merck公司;PCR mixture 生工生物工程(上海)股份有限公司。
1.2 仪器与设备
5804R冷冻高速离心机 德国Eppendorf公司;PCR仪、凝胶成像仪、电泳仪 美国Bio-Rad公司。1.3 方法
1.3.1 郫县豆瓣酱中微生物总DNA的提取
参考李治华等 [13]的方法提取郫县豆瓣酱中微生物的总DNA。
1.3.2 PCR扩增
第1轮PCR以基因组DNA为模板扩增18S rRNA全序列,琼脂糖凝胶电泳胶回收之后以18S rRNA基因为模板扩增SSU rRNA。第1轮PCR引物序列为 [10]:FR1(5’-AICCATTCAATCGGTAIT-3’)、NS1(5’-GTAGTCATATGCTTGTCTC-3’);体系(50 μL)为:模板D N A 4 μ L、上下游引物各2 μ L、2×Taq PCR MasterMix(包含500 μmol/L dNTP、0.1 U/μL Taq聚合酶、20 mmol/L Tris-HCl、100 mmol/L KCl 和 3 mmol/L MgCl 2)25 μL、ddH 2O 17 μL;扩增条件:95 ℃预变性8 min,95 ℃变性30 s,47 ℃退火45 s,72 ℃延伸3 min,35 个循环;最后72 ℃延伸10 min。阴性对照用ddH 2O作为模板。扩增结束后,用1.5%琼脂糖凝胶电泳检测,以凝胶成像系统采集图片并进行结果分析,然后切胶回收,回收后的DNA作为第2轮PCR的模板。
第2轮PCR引物序列为 [10]:FR1-GC(5’-CC CCCGCCGCGCGCGGCGGGCGGGGCGGGGG CACGGGCCGAICCATTCAATCGGTAIT-3’),FF390(5’-CGATAACGAACGAGACCT-3’);体系(50 μL)为:模板DNA 4 μL、上下游引物各2 μL、2×Taq PCR MasterMix(包含500 μmol/L dNTP、0.1 U/μL Taq聚合酶、20 mmol/L Tris-HCl、100 mmol/L KCl和 3 mmol/L MgCl 2)25 μL,ddH 2O 17 μL;扩增条件:95 ℃预变性8 min;95 ℃变性30 s,50 ℃退火45 s,72 ℃延伸2 min,30 个循环;最后72 ℃延伸10 min。阴性对照用ddH 2O作为模板。扩增结束后,用1.5%琼脂糖凝胶电泳检测,以凝胶成像系统采集图片并进行结果分析。
1.3.3 DGGE及数据分析
制备8%聚丙烯酰胺凝胶,变性梯度为35%~65%,呈线性增加,梯度方向与电泳方向垂直,加约40 μL含有适量上样缓冲液的PCR扩增产物。电泳时,温度为60 ℃,电压为120 V,运行6 h。电泳后用SYBR Green Ⅰ进行染色,染料用1×TAE以1∶10 000(V/V)稀释,每次染液用量15 mL,染色15 min,共染3 次。染色完后用凝胶成像系统采集图片并用Quantity One 4.6.9软件分析结果。
测得的序列在NCBI数据库进行相似性比较。将测序结果和代表性菌株的rRNA部分序列用程序ClustalX 2.0.11 [14]进行序列比对排序,采用UPGMA算法在软件Mega 6.06构建进化树 [15],分析亲缘关系及相似性。
2.1 真菌ITS区域PCR扩增产物DGGE图谱分析
13 个不同品牌样品SSU rRNA产物的DGGE结果如图1所示。郫县豆瓣酱样品中真菌种群多样性较高,不同品牌的豆瓣酱样品真菌种群结构有所不同。整体上,13 个样品经DGGE后都分离出清晰的电泳条带。相对而言,样品S5、S7、S8、S9的条带丰度较低,且条带亮度较弱,说明这4 个品牌豆瓣酱中的真菌多样性有可能较低。图1中各条带的相对强度和迁移率不完全相同,表明这些样品中既存在一些共有的真菌种群,又存在一些特有真菌,不同样品间在真菌种群丰度上存在一定的差别。另外,强度高的条带的迁移率也不完全相同,表明每个品牌真菌的优势种群也不完全一样。如条带3、4、7、11在样品S2中是优势菌群,而在S10样品中很弱或没有检测到;条带14在S11和S13中是优势真菌,但在S3、4、5却很弱或没有检测到。

图1 郫县豆瓣酱真菌群落DGGE图
Fig. 1 DGGE fingerprints of fungal communities in broad bean paste
表1 郫县豆瓣酱真菌SSU亚基DGGE条带测序结果
Table1 Sequencing results for DGGE bands of fungal SSU in broad bean paassttee

注:a.编号对应图1的编号。
测序条带编号 a与NCBI中同源性比较(序列登录号)相似度/% 2 Candida sp.(FJ153078.1)99 3 Candida sp.(FJ153078.1)99 4 C. sophiae-reginae(HQ651949.1)98 6Uncultured basidiomycete(AY934694.1)99 7 Zygosaccharomyces(AB087385.1)96 8 Z. rouxii(KT199372.1)99 9 C. catenulate(KT336721.1)99 10Malassezia obtuse(EU192365.1)100 12Kodamaea ohmeri(KM006493.1)99 13Aspergillus f avus(KU060796.1)98 14Uncultured Sebacinales(FN663658.2)99 15Capnobotryella sp. MA 4565(AJ972852.1)99 16Fungal sp. EL002650(KU354735.1)99 17A. f avus(KU060796.1)99 18A. f avus(KU060796.1)100 20A. f avus(KU060796.1)99 23A. f avus(KU060796.1)99 26A. f avus(KU060796.1)99 27A. f avus(KU060796.1)99 29A. f avus(KU060796.1)99 30A. f avus(KU060796.1)100 31A. f avus(KU060796.1)99
由表1可知,总共对31 条序进行了测序,其中有9 条序列与NCBI中序列的最大相似度低于95%(未列出)。在余下的22 条序列中,除条带7,其余23 条序列最大相似度都在98%~100%。其中有两个及以上条带检测到分别为假丝酵母(Candida)、鲁氏酵母(Zygosaccharomyces rouxii)、黄曲霉(Aspergillus f avus)。奥默柯达酵母菌(Kodamaea ohmeri)已在豆类发酵产品 [16]、啤酒 [17]、腌制蔬菜中分离得到 [18]。由图1和表1可知,A. f avus为郫县豆瓣酱的优势菌种,存在于所有品牌的样品中,尽管目前生产的多数郫县豆瓣黄曲霉毒素符合国家标准,但仍然有个别黄曲霉毒素B 1超标的报道 [19]。考虑到郫县豆瓣传统工艺主要在开放环境下进行,结合本研究的结果,表明郫县豆瓣的生产应该把防控黄曲霉放在非常重要的位置,避免成品中黄曲霉毒素超标。
2.2 聚类分析
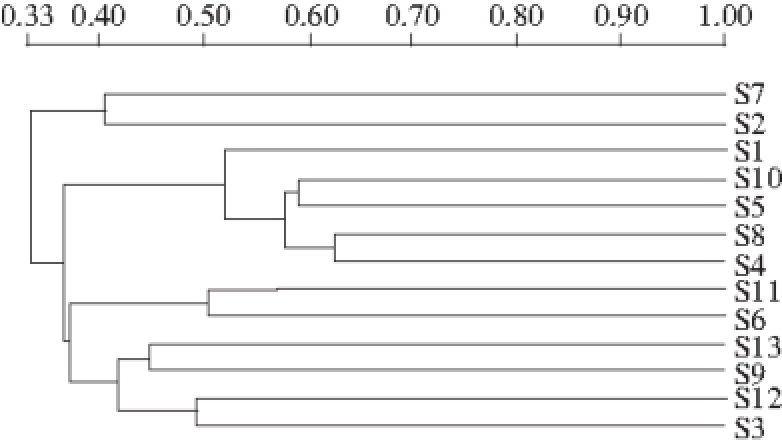
图2 郫县豆瓣酱中真菌的DGGE指纹图谱聚类分析图
Fig. 2 Cluster analysis of DGGE profiles for fungal communities of broad bean paste
从图2可知,初步可以将13 个郫县豆瓣样品分为4 类:第1类(S7、S2)、第2类(S1、S4、S5、S8、S10)、第3类(S6和S11)、第4类(S3、S9、S12、S13),这与DGGE电泳图(图1)的直观反映一致,即这些郫县豆瓣样品中既存在一些共有的真菌种群,又存在一些特有真菌,或者不同样品间在真菌种群丰度上存在一定的差别。
2.3 系统发育分析

图3 郫县豆瓣酱中真菌的系统发育分析
Fig. 3 Phylogenetic analysis of fungi in broad bean paste
从图3可知,测序序列可以分为4 个类别,类别Ⅰ将大多数的序列聚在了一起,主要为Candida、Zygosaccharomyces属和K. ohmeri,包含条带2、4、7、8、9、12;类别Ⅱ为Malassezia obtuse,包含条带10;类别Ⅲ为Capnobotryella sp.,包含条带15;类别Ⅵ为A. flavus,包含13、17、18、22、23、26、27、29、30、31。表明从系统发育的角度,郫县豆瓣真菌微生物具有一定多样性。
PCR-DGGE技术能有效的分析微生物菌群变化 [8,16]。前人研究表明,郫县豆瓣自然发酵过程中的优势真菌有米曲霉(A. oryzae)、淀粉丝菌(Amylomyces rouxii)、米根霉(Rhizopus oryzae)、异常毕赤酵母(Pichia anomala)、汉逊德巴利酵母(Debaryomyces hansenii)的近缘种 [20];董丹等 [21]采用纯培养对两个不同发酵时期豆瓣中微生物多样性进行了分析,发现Pichia、Candida、Wickerhamomyces属是真菌的优势菌群。本研究发现Candida sp.、Aspergillus、Z. rouxii、K. ohmeri是郫县豆瓣酱的优势种群,与上述结论存在一些异同,这可能与取样时间不同有关,如汪先丁等 [20]在制曲阶段(1、4、7 d)和发酵初期(14 d和30 d)取样,而本研究的样品都在后熟发酵6个月以上,后熟时间不同导致真菌菌群不一样;也有可能是生产的原料有差异,导致原料自身所带微生物菌群不同,在发酵过程中也有可能影响优势真菌菌群结构。
前人研究发现,Candida sp.和Z. rouxii在日本鱼酱和酱油中作为优势菌种在发酵过程中出现 [11,22-24],具有较强的耐盐能力,能在发酵过程中促进风味的形成 [25],表明上述两种酵母是传统发酵食品中常见的优势功能微生物菌群。张先琴等 [18]在分析四川泡菜微生物多样性时发现,K. ohmeri是四川泡菜的优势真菌,但是其功能和作用还有待进一步研究。
聚类分析可以将13 个郫县豆瓣样品分为4 类,而这13 个企业尽管都坐落于郫县镜内,但是分布于郫县的不同位置,不同位置的企业成品豆瓣酱在真菌多样性上也可聚为一类,表明外界环境对郫县豆瓣特有真菌群落结构的形成作用不明显。系统发育分析也可以将有效测序序列分为4 个类别,其中,Candida、Zygosaccharomyces属和K. ohmeri聚为Ⅰ类,这些真菌已在其他传统发酵食品广泛报道 [11,16-17],而A. f avus单独归为Ⅳ类。
特别需要注意的是,代表A. f avus的条带有10 条,占整个测序条带的32.25%,表明郫县豆瓣酱样品易受到黄曲霉菌的污染。尽管目前生产的多数郫县豆瓣黄曲霉毒素符合国家标准,但由于郫县豆瓣酱的生产工艺较复杂,包含制瓣、制椒、后熟等环节,其中多个环节都在开放的发酵环境下进行,而且相关检测机构抽检发现存在少数厂家样品黄曲霉毒素B 1超标的现象,因此,进一步研究郫县豆瓣生产中易受黄曲霉菌污染的关键环节,并建立相关黄曲霉污染控制模型和相关防控措施十分必要。
参考文献:
[1] 李幼筠. “郫县豆瓣”剖析[J]. 中国酿造, 2008, 14(11): 83-86. DOI:10.3969/j.issn.0254-5071.2008.06.027.
[2] 王静, 孙宝国. 中国主要传统食品和菜肴的工业化生产及其关键科学问题[J]. 中国食品学报, 2011, 11(9): 1-7. DOI:10.16429/j.1009-7848.2011.09.004.
[3] LEE S J, AHN B. Comparison of volatile components in fermented soybean pastes using simultaneous distillation and extraction (SDE) with sensory characterisation[J]. Food Chemistry, 2009, 114: 600-609. DOI:10.1016/j.foodchem.2008.09.091.
[4] ZHAO J X, DAI X J, LIU X M, et al. Comparison of aroma compounds in naturally fermented and inoculated Chinese soybean pastes by GC-MS and GC-olfactometry analysis[J]. Food Control, 2011, 22: 1008-1013. DOI:10.1016/j.foodcont.2010.11.023.
[5] 黄梅丽, 王俊卿. 食品色香味化学[M]. 2版. 北京: 中国轻工业出版社, 2008: 305-318.
[6] 张玉玉, 孙宝国, 冯军, 等. 不同发酵时间的郫县豆瓣酱挥发性成分分析[J]. 食品科学, 2010, 31(4): 166-170. DOI:10.7506/spkx1002-6300-201004037.
[7] 李治华, 黄驰, 王自鹏, 等. 不同后熟发酵时间郫县豆瓣酱挥发性成分分析[J]. 食品科学, 2014, 35(16): 180-184. DOI:10.7506/spkx1002-6630-201211033.
[8] 高永超, 王加宁, 迟建国, 等. 污染土壤微生物群落结构分子鉴定技术研究进展[J]. 生物技术, 2012, 22(2): 91-96. DOI:10.3969/ j.issn.1004-311X.2012.02.052.
[9] CHANG H W, KIM K H, NAM Y D, et al. Analysis of yeast and archaeal population dynamics in kimchi using denaturing gradient gel electrophoresis[J]. International Journal of Food Micro biology, 2008, 126: 159-166. DOI:10.1016/j.ijfoodmicro.2008.05.013.
[10] KIM T W, LEE J H, KIM S E, et al. Analysis of microbial communities in doenjang, a Korean fermented soybean paste, using nested PCR-denaturing gradient gel electrophoresis[J]. International Journal of Food Microbiology, 2009, 131: 265-271. DOI:10.1016/ j.ijfoodmicro.2009.03.001.
[11] TANAKA Y, WATANABE J, MOGI Y. Monitoring of the microbial communities involved in the soy sauce manufacturing process by PCR-denaturing gradient gel electrophoresis[J]. Food Microbiology, 2012, 31: 100-106. DOI:10.1016/j.fm.2012.02.005.
[12] XU W, HUANG Z Y, ZHANG X J, et al. Monitoring the microbial community during solid-state acetic acid fermentation of Zhenjiang aromatic vinegar[J]. Food Microbiology, 2011, 28: 1175-1181. DOI:10.1016/j.fm.2011.03.011.
[13] 李治华, 黄驰, 王自鹏, 等. 发酵豆瓣酱微生物宏基因组DNA提取研究[J]. 西南农业学报, 2013, 26(6): 2663-2665. DOI:10.16213/j.cnki. scjas.2013.06.005.
[14] LARKIN M A, BLACKSHIELDS G, BROWN N P, et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics, 2007, 23: 2947-2948. DOI:10.1093/bioinformatics/btm404.
[15] TAMURA K, STECHER G, PETERSON D, et al. MEGA6: molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology and Evolution, 2013, 30: 2725-2729. DOI:10.1093/molbev/mst197.
[16] EZEOKOLI O T, GUPTA A K, MIENIE C, et al. PCR-denaturing gradient gel electrophoresis analysis of microbial community in soydaddawa, a Nigerian fermented soybean (Glycine max (L.) Merr.) condiment[J]. International Journal of Food Microbiology, 2016, 220: 58-62. DOI:10.1016/j.ijfoodmicro.2016.01.003.
[17] N’GUESSAN K F, BROU K, JACQUES N, et al. Identification of yeasts during alcoholic fermentation of tchapalo, a traditional sorghum beer from Côte d’Ivoire[J]. Antonie van Leeuwenhoek, 2011, 99: 855-864. DOI:10.1007/s10482-011-9560-7.
[18] 张先琴, 张小平, 敖晓琳, 等. PCR-DGGE分析四川地区家庭制作泡菜中微生物多样性[J]. 食品科学, 2013, 34(12): 129-134. DOI:10.7506/spkx1002-6630-201312027.
[19] 中国质量新闻网. 成都食药监局: 10 批次豆瓣酱被检出黄曲霉毒素B 1超标[EB/OL]. (2014-12-18)[2016-08-15]. http://www.cqn.com.cn/ news/xfpd/ccgg/dfcc/2014/984632.html.
[20] 汪先丁, 刘敏, 高鹏, 等. 郫县豆瓣自然发酵过程中真菌群落的演替及黄曲霉毒素B 1的消长[J]. 食品科学, 2012, 33(11): 142-146.
[21] 董丹, 关统伟, 赵辉平, 等. 两个不同发酵时期豆瓣中微生物多样性的差异对比[J]. 中国酿造, 2014, 33(11): 55-58. DOI:10.11882/ j.issn.0254-5071.2014.11.012.
[22] YOSHIKAWA S, YASOKAWA D, NAGASHIMA K, et al. Microbiota during fermentation of chum salmon (Oncorhynchus keta) sauce mash inoculated with halotolerant microbial starters: analyses using the plate count method and PCR-denaturing gradient gel electrophoresis (DGGE)[J]. Food Microbiology, 2010, 27: 509-514. DOI:10.1016/j.fm.2009.12.008.
[23] QI W, HOU L H, GUO H L, et al. Effect of salt-tolerant yeast of Candida versatilis and Zygosaccharomyces rouxii on the production of biogenic amines during soy sauce fermentation[J]. Journal of the Science of Food and Agriculture, 2014, 94: 1537-1542. DOI:10.1002/ jsfa.6454.
[24] SUN S Y, JIANG W G, ZHAO Y P. Profile of volatile compounds in 12 Chinese soy sauces produced by a high-salt-diluted state fermentation[J]. Journal of the Institute of Brewing, 2010, 116: 316-328. DOI:10.1002/j.2050-0416.2010.tb00437.x.
[25] WAH T T, WALAISRI S, ASSAVANING A, et al. Co-culturing of Pichia guilliermondii enhanced volatile flavor compound formation by Zygosaccharomyces rouxii in the model system of Thai soy sauce fermentation[J]. International Journal of Food Microbiology, 2013, 160: 282-289. DOI:10.1016/j.ijfoodmicro.2012.10.022.
Analysis of Fungal Diversity of Different Commercial Brands of Pixian Broad Bean Paste Using PCR-DGGE Method
ZHU Yongqing, LI Zhihua, LI Huajia, WANG Xuetao, DONG Ling, HUANG Qiaolian
(Institute of Agro-Products Processing Science and Technology, Sichuan Academy of Agricultural Sciences, Chengdu 610066, China)
Abstract:In this study, the fungal diversity and community structures of 13 brands of Pixian broad bean paste were analyzed using polymerase chain reaction-denatured gradient gel electrophoresis (PCR-DGGE). The typical DGGE bands were sequenced, and a phylogenetic tree was constructed. The results showed that fungal diversity was abundant in the samples and both unique and common bands were identified. Candida sp., Aspergillus, Zygosaccharomyces rouxii and Kodamaea ohmeri were the predominant fungi. Especially, a total of 10 bands, accounting for 32.25% of the entire sequenced bands, were assigned to Aspergillus flavus. Thus, special attention should be paid to the prevention and control of A. flavus in the broad-bean paste industry.
Key words:broad bean paste; fungi; polymerase chain reaction-denatured gradient gel electrophoresis (PCR-DGGE); diversity
DOI:10.7506/spkx1002-6630-201702017
中图分类号:TS201.3
文献标志码:A
文章编号:1002-6630(2017)02-0104-05
引文格式:
朱永清, 李治华, 李华佳, 等. 基于PCR-DGGE分析不同品牌郫县豆瓣酱真菌多样性[J]. 食品科学, 2017, 38(2): 104-108. DOI:10.7506/spkx1002-6630-201702017. http://www.spkx.net.cn
ZHU Yongqing, LI Zhihua, LI Huajia, et al. Analysis of fungal diversity of different commercial brands of Pixian broad bean paste using PCR-DGGE method[J]. Food Science, 2017, 38(2): 104-108. (in Chinese with English abstract)
DOI:10.7506/ spkx1002-6630-201702017. http://www.spkx.net.cn
收稿日期:2016-09-06
基金项目:四川省财政创新能力提升专项(2016GXTZ-008);四川省农业科学院青年基金项目(2016QNJJ-020);国家自然科学基金四川省农业科学院补助项目(2015KXJJ-012)
作者简介:朱永清(1968—),男,副研究员,博士,研究方向为果蔬加工与食品发酵。E-mail:zhuyongqing68@sina.com