传统发酵锦州小菜中主要微生物多样性分析
孙慧君
1,2,张 颖
1,王洪玉
3,杨 梅
1,岳喜庆
1,乌日娜
1,4,*
(1.沈阳农业大学食品学院,辽宁 沈阳 110866;2.辽宁省农业经济学校现代农业技术系,辽宁 锦州 121001;3.沈阳农业大学园艺学院,辽宁 沈阳 110866;4.江南大学 食品科学与技术国家重点实验室,食品学院,江苏 无锡 214122)
摘 要:通过探究锦州小菜的微生物群落组成,以期为改善其品质提供依据。利用聚合酶链式反应-变性梯度凝胶电泳对采集自10 份锦州地区传统发酵小菜中的微生物多样性进行研究,结果表明:共鉴定出5 种细菌,分别为植物乳杆菌(Lactobacillus plantarum)、食窦魏斯氏菌(Weissella cibaria)、Lactobacillus fabifermentans、希腊魏斯菌单菌(Weissella hellenica)、假单细胞菌(Psychromonas arctica);真菌菌属有3 种,分别为热带假丝酵母(Candiada tropicalis)、弯曲黏膜菌(Campylobacter mucosalis)、白色念珠菌(Candida albicans)。其中,主要细菌为植物乳杆菌、食窦魏斯氏菌,主要真菌为热带假丝酵母。
关键词:锦州小菜;聚合酶链式反应-变性梯度凝胶电泳;多样性
我国传统发酵食品有着悠久的历史,且有各自独特的生产工艺,发酵过程涉及微生物种类较多,赋予食品特有的风味
[1]。锦州小菜是辽宁锦州市特色的传统发酵食品,创始于清朝初年,距今已有三百多年的历史
[2-3],为人们熟知的腌菜如虾油类的虾油黄瓜、什锦菜、虾酱菜,盐渍类的盐渍豇豆等等。传统的锦州小菜采用黄瓜、圆青椒、小茄子、豇豆等新鲜蔬菜经食盐、酱油、食醋、糖等调料腌制而成,因当地的独特气候及水质使酱菜具有特殊风味。制作出来的小菜质地脆嫩,味道可口,受到许多人们的喜爱。传统发酵食品由于微生物的发酵作用经过分解、消除和过滤过程后更具有安全性
[4]。食用这些发酵蔬菜除可提高营养利用外
[5],同时还可以摄入乳酸菌及其代谢产生的有机酸等,可以促进宿主肠道内菌群生态平衡、防止细胞老化、降低胆固醇,对人体代谢产生有益的调节作用
[6-7]。
20世纪初,人们对发酵蔬菜中微生物进行研究发现,乳酸菌及酵母菌等益生菌对发酵蔬菜中的风味及质地起到至关重要的作用
[8]。在国内利用益生菌发酵的泡菜产品酸鲜可口,营养丰富,天然风味浓郁,具有良好的保健功能,富含益生菌和天然植物化学物
[9]。发酵蔬菜中的主要微生物有:植物乳杆菌、肠膜明串珠菌、乳酸乳球菌、魏斯氏菌、酵母菌(主要为酿酒酵母、汉逊得巴利酵母、热带假丝酵母)等。而国外的发酵食品多为发酵奶类及肉制品,其中的主要微生物为粪肠球菌、嗜酸乳杆菌、副干酪乳杆菌、鼠李糖乳杆菌、干酪乳酸杆菌、长双岐杆菌和分叉双歧杆菌等
[10]。
变性梯度凝胶电泳(denatured gradient gel electrophoresis,DGGE)最初是Lerman等
[11]于20世纪80年代初期发明的。Muyzer等在1993年首次将其应用于微生物群落结构研究,并证实了这种技术在揭示自然界微生物领域的遗传多样性和菌群差异方面具有独特的优越性,目前已经发展成为研究微生物群落结构的主要分子生物学方法之一
[12-13]。与传统分离培养的方法相比,DGGE技术可以反映微生物群体结构的原始状态,可以比较不同的发酵样品,进而探究发酵食品中微生物的变化规律
[14]。
锦州小菜风味独特,其中赋予小菜风味的菌种有待于进一步去探究开发,而目前相关研究甚少。本实验采用聚合酶链式反应(polymerase chain reaction,PCR)结合DGGE方法对10 种传统发酵锦州小菜中微生物多样性进行初步探究,为改善其品质提供依据。
1 材料与方法
1.1 样品采集
选取常见的锦州传统发酵制作的特色小菜作为研究材料,样品自同一日上午采集于辽宁锦州市内3 区的5 个地点。采集地点及样品编号为:古塔区石桥子早市咸芥菜丝(ZS1)、红芥菜丝(ZS2)、早市虾油黄瓜(ZS3)、腌蒜(ZS4);古塔区大润发超市虾油黄瓜(DRF1)、辣萝卜(DRF2)、凌河区金城农贸市场辣白菜(JL1)、辣萝卜(JL2)、凌河区城市生活广场辣白菜(CS1)、太和区华联超市白醋蒜(HL1)。将其发酵液存入灭菌的离心管中封口,平行采集3 份。样品采集的同时记录采集温度、发酵时间并做好标识。样品用液氮处理后保存于-80 ℃冷库。
1.2 试剂
聚丙烯酰胺、三羟甲基氨基甲烷、过硫酸铵、四甲基乙二胺 北京艾徳莱生物有限公司;磷酸二氢钾、十二水合磷酸氢二钾、氯化钠、10×TE缓冲液、1×TAE电泳缓冲液、十二烷基硫酸钠、十六烷基三甲基溴化铵(cetyltrimethyl ammonium bromide,CTAB)、乙酸钠、异丙醇、冰醋酸、乙醇 国药集团化学试剂有限公司;琼脂糖 北京沃比森科技有限公司;液氮 沈阳嘉和气体有限公司;蛋白酶K、脱氧核糖核苷三磷酸(deoxyribonucleotide triphosphate,dNTP)、10×PCR buffer、r-Taq酶、Loading buffer、Marker DM2000、Gel-red核酸染料、引物 北京艾比根生物有限公司。
1.3 仪器与设备
PL303/01电子天平 上海卓精电子科技有限公司;GMSX-280手提式压力蒸汽灭菌锅 北京永光明医疗仪器有限公司;SH2100 pH计 奥豪斯仪器(常州)有限公司;ZHJH-C1112C无菌操作台 北京瑞尔欣德科技有限公司;DHG-9070S电热鼓风干燥箱、电热恒温水浴锅上海精宏实验设备有限公司;VS-1涡旋仪 北京中时维兴仪器设备公司;CR-21G离心机 日本日立公司;TDL-SA离心机 上海菲恰尔分析仪器有限公司;超低温冰箱、TGL-168高速台式离心机、5810高速冷冻离心机、微量紫外-分光光度计 德国Eppendorf公司;DYY-6C电泳仪 北京六一仪器厂;PCR扩增仪、凝胶成像系统、DGGE仪 美国伯乐公司。
1.4 方法
1.4.1 发酵液的处理
取20 mL发酵液,12 000 r/min离心15 min,弃去上清液,加入10 mL灭菌水清洗沉淀,重复多次,直至上清液清澈。在沉淀中加入700 μL TE缓冲液回溶,用于总DNA提取。
1.4.2 发酵小菜的处理
取20 g样品小菜捣碎,用30 mL PBS缓冲液4 ℃浸提过夜
[15]。之后操作同1.4.1节。
1.4.3 样品的总DNA提取
采用CTAB法对总DNA的进行提取
[16],每份样品提取3 次。用微量紫外-分光光度计测定DNA质量浓度和A
260
nm/A
280
nm(表示所提取DNA的纯度),将A
260
nm/A
280
nm在1.8~2.2范围内的DNA样品进行下一步PCR扩增。
1.4.4 PCR扩增
1.4.4.1 细菌DNA的PCR扩增
以提取的总DNA为模板,采用16S rDNA基因V3区具有特异性的引物(V3F+GC:5’-CGCCC GCCGCGCGCGGCGGGCGGGGCGGGGGCA CGGGGGGACTCCTACGGGAGGCAGCAG-3’和V3R:5’-ATTACCGCGGCTGCTGG-3’)进行16S rDNA的PCR扩增,扩增产物片段长约250 bp。扩增反应体系(50 μL)包括:1.5 μL DNA模板、5.0 μL 10×PCR buffer、4.0 μL dNTP、上游引物1.5 μL、下游引物1.5 μL、DNA Taq酶0.5 μL、36 μL超纯水。反应参数:95 ℃预变性4 min;95 ℃变性1 min,55 ℃退火45 s,72 ℃延伸1 min,循环30 次;72 ℃延伸7 min。PCR产物用1%琼脂糖凝胶电泳检测。
1.4.4.2 真菌DNA的PCR扩增
以提取的总DNA为模板,采用26SrDNA D1/D2区具有特异性的引物(正向引物:5’-CTTGGTCATTTAGAGGAAGTAA-3’,反向引物:5’-TCCTCCGCTTATTGATATGC-3’)进行26S rDNA的PCR扩增
[17],扩增产物片段长约600 bp
[18]。扩增反应体系(50 μL)包括:5.0 μL DNA模板、5.0 μL buffer、4.0 μL dNTP、上游引物1.0 μL、下游引物1.0 μL,剩余体系用超纯水混匀。反应参数:95 ℃预变性5 min; 94 ℃变性1 min,54 ℃退火1 min,72 ℃延伸90 s,循环36 次;72 ℃延伸10 min,最后4 ℃条件下保存。
1.4.5 DGGE
细菌的变性剂浓度范围为35%~50%
[19],在150 V电压、60 ℃条件下电泳7 h,真菌的变性剂浓度范围为35%~50%,在120 V电压下,60 ℃电泳5.25 h。电泳完毕后,利用Gel-red染色法对凝胶进行染色。将染好的凝胶用扫描仪成像,得到PCR-DGGE图谱,对扫描后的图片进行标注分析,并标注特异性条带,回收条带。
1.4.6 DGGE胶条回收测序
用刀片在酒精灯外延加热灭菌后切割胶条,将胶条放入1.5 mL离心管中,并做好标记。向其中加入100 μL去离子水冲洗胶条(3~4 次),弃去洗水后,再用100 μL超纯水溶解胶条中的DNA,4 ℃条件下静置约12 h。将充分溶解的DNA样品进行PCR检验。其中,细菌回收DNA扩增反应体系(50 μL)包括:5.0 μL回收DNA模板、5.0 μL 10×PCR buffer、4.0 μL dNTP、上游引物(F338)1.5 μL、下游引物1.5 μL、DNA Taq酶0.5 μL、超纯水32.5 μL,反应参数同1.4.4.1节。真菌回收DNA扩增反应体系,反应参数同1.4.4.2节。测序由上海派森诺生物公司完成。
2 结果与分析
2.1 DNA提取结果
由表1可知,10 种样品的DNA质量浓度在均100 ng/μL以上,同时这些样品DNA的A
260
nm/A
280
nm值基本保持在1.8~2.2的范围内,可进行PCR扩增。
表1 锦州小菜提取总DNA结果
Table1 Extraction of total DNA from Jinzhou pickles
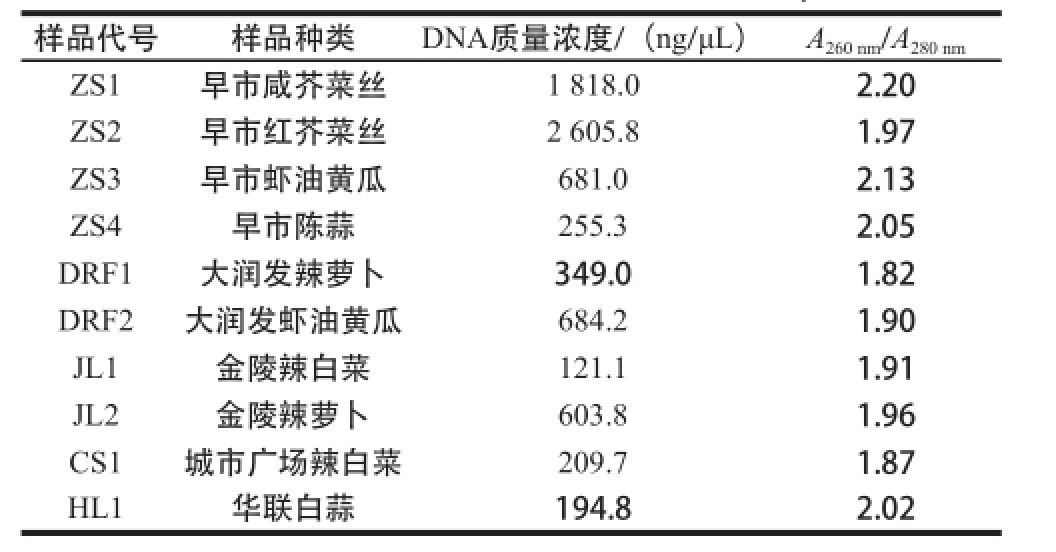
样品代号样品种类DNA质量浓度/(ng/μL)A
260
nm/A
280
nmZS1早市咸芥菜丝1 818.02.20 ZS2早市红芥菜丝2 605.81.97 ZS3早市虾油黄瓜681.02.13 ZS4早市陈蒜255.32.05 DRF1大润发辣萝卜349.01.82 DRF2大润发虾油黄瓜684.21.90 JL1金陵辣白菜121.11.91 JL2金陵辣萝卜603.81.96 CS1城市广场辣白菜209.71.87 HL1华联白蒜194.82.02
2.2 PCR扩增结果
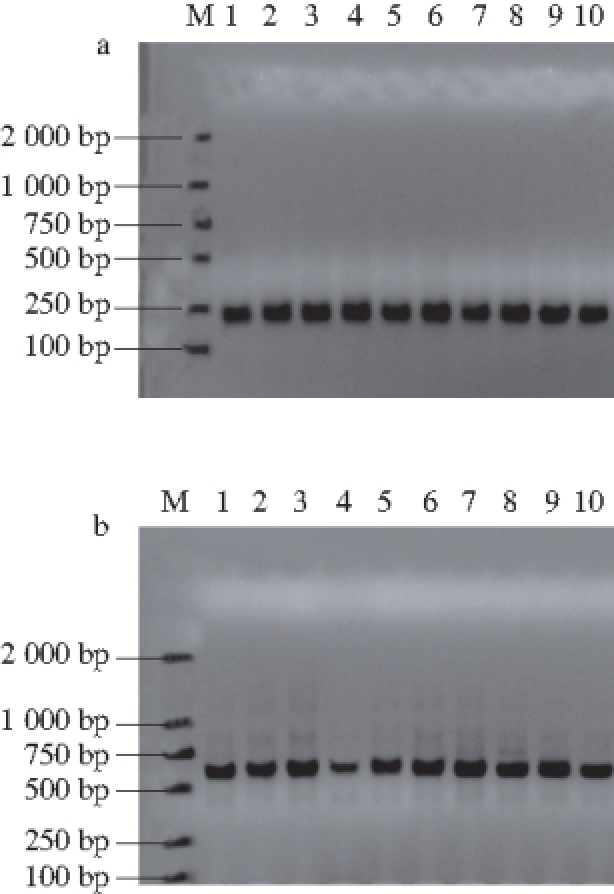
图1 细菌(a)及部分真菌(b)PCR扩增结果
Fig. 1 PCR amplification results of total DNA extracted from the bacterial (a) and fungal (b) strains
分别以提取的细菌和真菌总DNA做模板,进行PCR扩增,然后采用1%琼脂糖凝胶电泳对其产物进行检测。所扩增的产物分别在250 bp(图1a)和600 bp(图1b)左右处有明显条带,说明PCR扩增成功,可以进行DGGE。
2.3 DGGE检测结果
2.3.1 细菌多样性检测结果
表2 锦州小菜样品细菌16S rDNA V3区PCR-DGGE特异性条带比对结果
Table2 Sequence alignment of PCR-DGGE specific bands of bacterial 16S rDNA V3 regions from samples

条带编号鉴定结果同源性/%序列号A植物乳杆菌(Lactobacillus plantarum) 97NC 004567.2 B假单细胞菌(Psychromonas arctica) 99NE KI 519478.1 C食窦魏斯氏菌(Weissella cibaria)99NZ AEKT01000037 D Lactobacillus fabifermentans100NZ KK036522.1 E希腊魏斯氏菌(Weissella hellenica) 100NZ BBIK01000014.1

图2 锦州小菜样品细菌的PCR-DGGE图谱
Fig. 2 PCR-DGGE profiles of bacterial populations from each pickle sample
采用CTAB法提取传统发酵小菜样品的总DNA,通过特异性扩增细菌16SrDNA V3区,其PCR-DGGE图谱如图2所示。锦州小菜细菌DNA的PCR-DGGE图谱中共找到5 条特异性条带,对其编号切割、回收测序,并登录NCBI数据库进行BLAST同源性对比分析(http://www. ncbi.nlm.nih.gov/BLAST/)。
由图2、表2可知,条带A鉴定结果为植物乳杆菌(Lactobacillus plantarum),共有7 份样品(ZS3、HL1、ZS2、ZS4、ZS1、JL2、DRF2)检测到该条带;条带B为一种假单细胞菌(Psychromonas arctica),共有1 份样品(JL1)检测到该条带;条带C为食窦魏斯氏菌(Weissella cibaria),有6 份样品(ZS3、ZS2、DRF1、ZS1、JL1、CS1)检测到该条带;条带D为Lactobacillus fabifermentans,共有2 份样品(ZS3、ZS2)检测到该条带;条带E为希腊魏斯氏菌(Weissella hellenica),共有3 份样品(DRF1、JL1、CS1)检测到该条带。推测细菌植物乳杆菌、食窦魏斯氏菌可能为传统发酵锦州小菜中的优势菌群。
魏斯氏菌主要存在于泡菜、发酵食品等,并在发酵初期成为主要优势菌群,而在后期发酵中被乳酸杆菌等代替
[20]。Lee等
[21]探究了人粪便中分离的8 种魏斯氏菌的益生属性,初步验证了食窦魏斯氏菌等对低pH值、6.5%低盐浓度和0.3%胆盐有一定的耐受性,对卡那霉素、链霉素有一定的耐药性;相比于黏附性较好的鼠里糖乳杆菌(Lactobacillus rhamnosus),食窦魏斯氏菌等对Caco-2细胞模型具有更好的吸附能力等益生属性。Sang等
[22]发现魏斯氏菌在泡菜的发酵过程中其菌落数量会因添加胡椒粉而增加,而明串珠菌和乳杆菌则会有所降低。2009年,Bruyne等
[23]在发酵的加纳可可豆堆中分离到2 种新菌株Lactobacillus fabifermentans sp.和Lactobacillus cacaonum sp.,并通过16S rDNA基因测序证明Lactobacillus fabifermentans是植物乳杆菌的一种;Treu等
[24]在自然发酵的葡萄渣中分离到了Lactobacillus fabifermentans T30PCM01,表明这类乳杆菌可能具有潜在的碳水化合物利用及转录调节功能。
此外,在东北特色发酵食品酸菜中,也分离得到大量菌株。乌日娜等
[25]在东北自然发酵酸菜中通过PCRDGGE分离到了明串珠菌属(Leuconostoc)、芽孢杆菌属(Bacillus)、假单细胞菌属(Pseudomonas)等乳酸菌菌属;丛敏等
[26]从酸菜的前期阶段分离到乳酸乳球菌(Lactococcus lactis)、戊糖乳杆菌(Lactobacillus pentosus);杨洪岩等
[27]对酸菜发酵中细菌的动态变化分析发现,弯曲乳杆菌(Lactobacillus curvatus)为发酵中的优势菌群;曹碧璇等
[28]利用16S rRNA基因克隆文库分析自然发酵酸菜中片球菌属(Pediococcuss)和枸橼酸杆菌属(Citrobacter)等亦是特色发酵食品中的优势菌属,但在本实验并未分离到。本实验分离到的2 种魏斯氏菌和Lactobacillus fabifermentans可以在以后的实验中继续探究。
2.3.2 真菌多样性检测结果

图3 锦州小菜样品真菌的PCR-DGGE图谱
Fig. 3 PCR-DGGE profiles of fungal populations from each pickle sample
表3 锦州小菜样品真菌26SrDNAD1/D2区PCR--DDGGGGEE特异性条带比对结果
Table3 Sequence alignment of specific bands obtained by PCR-DGGE of fungal 26S rDNA D1/D2 regions from samples

条带编号鉴定结果同源性/%序列号A 热带假丝酵母菌(Candiada tropicalis)86EU543673.1 B弯曲黏膜菌(Campylobacter mucosalis strain)96NZ JHQQ01000018.1 I白色念珠菌(Candida albicans)99KC405619.1
采用CTAB法提取小菜样品的总DNA,通过特异性扩增真菌26S rDNA D1/D2区,其PCR-DGGE图谱如图3所示。传统发酵锦州小菜真菌DNA的PCR-DGGE图谱中共找到9 条特异性条带,对其编号切割、回收测序,并登录NCBI数据库进行BLAST同源性对比分析(http://www. ncbi.nlm.nih.gov/BLAST/)。
由图3、表3可知,条带A鉴定结果为热带假丝酵母(Candiada tropicalis),共有5 份样品(JL1、CS1、ZS3、ZS1、ZS2)检测到该条带;条带B为弯曲黏膜菌(Campylobacter mucosalis strain),有4 份样品(JL1、CS1、DRF2、HL1)检测到该条带;条带I为白色念珠菌(Candida albicans),共有1 份样品(ZS2)检测到该条带。条带C、D、E、F、G、H未鉴定出结果,10号样品没有检测到特异性条带。推测热带假丝酵母可能为传统发酵锦州小菜中的优势菌群。
东北传统发酵酸菜中的主要真菌菌群有汉逊得巴利酵母(Debaryomyces hansenii)、热带假丝酵母(Candida tropicalis)、扩展青霉菌(Penicillium expansum)等
[16];除此之外,武俊瑞
[18]通过PCRDGGE从传统发酵豆酱中还分离到了二孢接合酵母(Zygosaccharomyces bisporus)、近平滑假丝酵母(Candida parapsilosis)、解脂耶氏酵母(Yarrowia lipolytica)、黏质红酵母(Rhodotorula mucilaginosa)、涎沫假丝酵母(Candida zelanoides)等真菌;田甜
[29]从东北传统发酵豆酱中分离到了赤霉菌属(Gibberella)、拟青霉菌属(Paecilomyces)、青霉菌属(Penicillium)等真菌。
3 结 论
采用CTAB法提取总DNA,通过PCR-DGGE方法对10 份传统发酵锦州小菜中微生物的多样性进行分析,对其中可能富含的优势菌群组成进行初步研究。结果发现,传统发酵锦州小菜中的微生物丰富多样:传统发酵锦州小菜中可能存在的优势菌有植物乳杆菌、魏斯氏菌、热带假丝酵母。其中,7 份样品检测到植物乳杆菌;6 份样品检测到食窦魏斯氏菌;共有3 份检测到希腊魏斯氏菌;2 份样品检测到Lactobacillus fabifermentans;此外,1 份样品检测到假单细胞菌;5 份样品检测到热带假丝酵母;4 份样品检测到弯曲黏膜菌;1 份样品检测到白色念珠菌。通过对传统发酵锦州小菜中微生物多样性的研究,以期为锦州小菜的品质提供依据。
参考文献:
[1] 聂志强, 王敏, 郑宇. 3 种分子生物学技术在传统发酵食品微生物多样性研究中的应用[J]. 食品科学, 2012, 33(23): 346-350.
[2] 荀凤英. 锦州小菜史话[J]. 兰台世界, 2002(11): 40-41.
[3] 曲玲童, 牛文静, 孙征, 等. 锦州腌渍小菜中耐盐乳酸菌的筛选与产酸性能研究[J]. 中国酿造, 2012, 31(6): 83-87. DOI:10.3969/ j.issn.0254-5071.2012.06.022.
[4] 白凤翎. 微生物的发酵作用对传统酿造食品安全性的影响[J]. 中国酿造, 2009, 28(2): 5-8. DOI:10.3969/j.issn.0254-5071.2009.02.002.
[5] CAGNO R D, CODA R. Fermented vegetable products[M]// Encyclopedia of Food Microbiology, 2014: 875-883. DOI:10.1016/ B978-0-12-384730-0.00115-4.
[6] 刘志文, 袁伟静, 张三燕, 等. 三江镇腌菜中降解亚硝酸盐乳酸菌的筛选和初步鉴定[J]. 食品科学, 2012, 33(1): 166-199.
[7] 徐鑫, 王茜茜, 王晓蕊, 等. 传统农家大酱中耐盐性乳酸菌的分离与鉴定[J]. 食品与发酵工业, 2014, 40(11): 33-40.
[8] 乌日娜, 孟令帅, 王茜茜, 等. 延边泡菜品质评价以及耐盐酵母菌的筛选[J]. 食品科学, 2015, 36(5): 83-88. DOI:10.7506/spkx1002-6630-201505016.
[9] 谢明勇, 熊涛, 关倩倩. 益生菌发酵果蔬关键技术研究进展[J].中国食品学报, 2014, 14(10): 1-9. DOI:10.16429/j.1009-7848.2014.10.010.
[10] 胡晓清, 潘露, 王汝毅. 发酵蔬菜中乳酸菌的功能性与安全性研究进展[J]. 现代食品科技, 2012, 28(11): 1606-1611. DOI:10.13982/ j.mfst.1673-9078.2012.11.045.
[11] 朱扬玲. 采用PCR-DGGE方法研究浙江玫瑰醋酿造过程中的微生物多样性[D]. 杭州: 浙江工商大学, 2009: 3-14.
[12] 张伟. 温度梯度凝胶电泳(TGGE)分析消毒剂对微宇宙细菌群落结构的影响[D]. 武汉: 华中科技大学, 2006: 7-8.
[13] DUNYACH-REMY C, CADIÈRE A, RICHARD J L, et al. Polymerase chain reaction-denaturing gradient gel electrophoresis (PCR-DGGE): a promising tool to diagnose bacterial infections in diabetic foot ulcers[J]. Diabetes & Metabolism, 2014, 40(6): 476-480. DOI:10.1016/j.diabet.2014.03.002.
[14] 梁新乐, 朱扬玲, 蒋予箭, 等. PCR-DGGE法研究泡菜中微生物群落结构的多样性[J]. 中国食品学报, 2008, 8(3): 133-137. DOI:10.3969/ j.issn.1009-7848.2008.03.024
[15] 孔令琼, 管政兵, 陆健, 等. 绍兴黄酒成品麦曲中微生物胞外酶的双向电泳技术的建立[J]. 食品与生物技术学报, 2011, 5(3): 453-457.
[16] 乌日娜, 于美玲, 孟令帅, 等. PCR-DGGE分析东北自然发酵酸菜中的微生物多样性[J]. 现代食品科技, 2014, 30(10): 8-13.
[17] 武俊瑞, 王晓蕊, 唐筱杨, 等. 辽宁传统发酵豆酱中乳酸菌及酵母菌分离鉴定[J]. 食品科学, 2015, 36(9): 78-84. DOI:10.7506/spkx1002-6630-201509015.
[18] 武俊瑞. 东北传统发酵特色食品中主要微生物多样性研究[D].沈阳: 沈阳农业大学, 2013: 41-42.
[19] 于美玲. DGGE分析自然发酵酸菜中微生物的多样性[D]. 沈阳:沈阳农业大学, 2015: 20-21.
[20] JEONG S H, JUNG J Y, LEE S H, et al. Microbial succession and metabolite changes during long-term storage of kimchi[J]. Journal of Food Science, 2013, 78(5): 763-769. DOI:10.1111/1750-3841.12095.
[21] LEE K W, PARK J Y, JEONG H R, et al. Probiotic properties of Weissella strains isolated from human faeces[J]. Anaerobe, 2011, 18(1): 96-102. DOI:10.1016/j.anaerobe.2011.12.015.
[22] SANG H J, LEE H J, JI Y J, et al. Effects of red pepper powder on microbial communities and metabolites during kimchi fermentation[J]. International Journal of Food Microbiology, 2013, 160(3): 252-259. DOI:10.1016/j.ijfoodmicro.2012.10.015.
[23] BRUYNE K D, CAMU N, VUYST L D, et al. Lactobacillus fabifermentans sp. nov. and Lactobacillus cacaonum sp. nov. isolated from Ghanaian cocoa fermentations[J]. International Journal of Systematic & Evolutionary Microbiology, 2009, 59(1): 7-12. DOI:10.1099/ijs.0.001172-0.
[24] TREU L, VENDRAMIN V, BOVO B, et al. Genome sequence of Lactobacillus fabifermentans strain T30PCM01, isolated from fermenting grape marc[J]. Genome Announcements, 2014, 2(1): 1-2. DOI:10.1128/genomeA.00060-14.
[25] WU R N, YU M L, LIU X M, et al. Changes in flavour and microbial diversity during natural fermentation of suan-cai, a traditional food made in Northeast China[J]. International Journal of Food Microbiology, 2015, 211: 23-31. DOI:10.1016/ j.ijfoodmicro.2015.06.028.
[26] 丛敏, 李欣蔚, 武俊瑞, 等. PCR-DGGE分析东北传统发酵酸菜中乳酸菌多样性[J]. 食品科学, 2016, 37(7): 78-82. DOI:10.7506/ spkx1002-6630-201607015.
[27] 杨洪岩, 李超, 刘通, 等. 传统东北酸菜发酵过程中的细菌动态及多样性[J]. 食品工业科技, 2015, 36(1): 154-159. DOI:10.13386/ j.issn1002-0306.2015.01.024.
[28] 曹碧璇, 胡滨, 刘爱平. 利用16S rRNA基因克隆文库分析东北自然发酵酸菜中细菌多样性[J]. 食品与发酵工业, 2015, 41(11): 76-80. DOI:10.13995/j.cnki.11-1802/ts.201511014.
[29] 田甜. 东北豆酱自然发酵过程中风味品质与微生物变化规律研究[D].沈阳: 沈阳农业大学, 2015: 51-52.
Microbial Diversity of Traditional Fermented Jinzhou Pickles
SUN Huijun
1,2, ZHANG Ying
1, WANG Hongyu
3, YANG Mei
1, YUE Xiqing
1, WU Rina
1,4,*
(1. College of Food Science, Shenyang Agricultural University, Shenyang 110866, China; 2. College of Morden Agricultural Technology, Liaoning Agricultural Economic School, Jinzhou 121001, China; 3. College of Horticulture, Shenyang Agricultural University, Shenyang 110866, China; 4. State Key Laboratory of Food Science and Technology, College of Food Science, Jiangnan University, Wuxi 214122, China)
Abstract:In order to improving the quality of Jinzhou pickles, this experiment studied the microbial community composition of Jinzhou pickles. Polymerase chain reaction-denaturing gradient gel electrophoresis (PCR-DGGE) was adopted to analyze the microbial diversity of ten traditional pickle samples in Jinzhou area, Liaoning province. The results showed that f ve bacterial strains were identif ed namely Lactobacillus plantarum, Weissella cibaria, Lactobacillus fabifermentans, Weissella hellenic and Psychromonas arctica as well as three fungal species i.e., Candiada tropicalis, Campylobacter mucosalis and Candida albicans. The predominant bacteria were Lactobacillus plantarum, Weissella cibaria and the predominant fungus was Candiada tropicalis.
Key words:Jinzhou pickles; polymerase chain reaction-denaturing gradient gel electrophoresis (PCR-DGGE); diversity
DOI:10.7506/spkx1002-6630-201702003
中图分类号:Q93
文献标志码:A
文章编号:1002-6630(2017)02-0015-05
引文格式:
孙慧君, 张颖, 王洪玉, 等. 传统发酵锦州小菜中主要微生物多样性分析[J]. 食品科学, 2017, 38(2): 15-19. DOI:10.7506/ spkx1002-6630-201702003. http://www.spkx.net.cn
SUN Huijun, ZHANG Ying, WANG Hongyu, et al. Microbial diversity of traditional fermented Jinzhou pickles[J]. Food Science, 2017, 38(2): 15-19. (in Chinese with English abstract)
DOI:10.7506/spkx1002-6630-201702003. http://www.spkx.net.cn
收稿日期:2016-06-27
基金项目:国家自然科学基金面上项目(31471713);中国博士后科学基金项目(2014M560395);辽宁省农业领域青年科技创新人才培养计划项目(2014048);辽宁省高等学校优秀人才支持计划项目(LR2015059);江苏省博士后科研资助计划项目(1402071C)
作者简介:孙慧君(1990—),女,硕士研究生,研究方向为食品生物技术。E-mail:jun_sh666@163.com
*通信作者:乌日娜(1979—),女,副教授,博士,研究方向为食品生物技术。E-mail:wrn6956@163.com





