表 1 EGDe-ΔprfA构建及鉴定所需引物
Table 1 Primer sequences used in the construction and identification of EGDe-ΔprfA
注:下划线表示酶切位点。
郭 亮,陈国薇,谢曼曼,刘武康,丁承超,王淑娟,罗 勤,刘 箐*
(上海理工大学医疗器械与食品学院,上海 200093)
摘 要:单核细胞增生性李斯特菌(Listeria monocytogenes,Lm)是一种风险较高的食源性致病菌,其绝大多数毒力基因的表达均受到由prfA基因编码的PrfA(positive regulatory factor A)蛋白全部或部分调控。使用同源重组的方法敲除Lm野生菌株EGDe的prfA基因,并通过测定生长曲线、毒力基因表达和侵袭Caco-2细胞能力等探讨单缺失菌株EGDe-ΔprfA生 物学特性。通过测定生长曲线显示prfA敲除株和野生型EGDe二者生长状态无差异;运用实时定量荧光聚合酶链式反应检测EGDe-ΔprfA的主要毒力基因表达,结果显示plcA、plcB毒力基因的表达量分别上升至原来的2.5 倍和2 倍,inlA、inlB、inlC、actA、vip等均呈下降趋势,prfA基因表达量趋向于零;侵袭Caco-2细胞结果显示EGDe-ΔprfA侵袭数为野生株的1/5。该基因缺失菌株的构建及生物学特性研究对食源性致病菌EGDe致病机理研究具有重要意义并且提供了重要材料。
关键词:单核细胞增生性李斯特菌;prfA;基因敲除;生物学特性
单核细胞增生性李斯特菌(Listeria monocytogenes,Lm)是普遍存在的一种微生物,广泛分布在各种环境中,人类通过摄入感染Lm的乳制品、肉类、鱼类、蔬菜等发生李氏杆菌病,李氏杆菌病主要影响免疫力低下的老年人、孕妇和新生儿,具有较高的致死率(20%~30%)[1-3]。单核细胞增生李斯特氏菌可以在较宽的温度范围内(1~45 ℃)生长,最佳温度为30~37 ℃;此外,它可以在恶劣环境下生存,例如低pH值和高质量分数盐(10% NaCl)[4]。不管是人类还是动物,Lm均可以感染多种组织,包括脾、肝、脑,一般认为Lm穿过肠道屏障进入淋巴和血液,在肝细胞或脾细胞中复制然后通过血液可以到达大脑和胎盘[5-7]。
Lm致病过程由多种不同的毒力基因编码的毒力因子调控,前期研究表明,Lm通过触发两种膜表面蛋白内化素A(internalin A,InlA)和内化素B(internalin B,InlB),和宿主细胞的特异性受体结合,引导Lm被吞噬细胞吞噬后[8-9],细胞溶解素酶O(listeriolysin O,LLO,由hly编码)在plcA和plcB编码的磷脂酰肌醇特异性磷脂酶(phosphatidylinositol-specific phospholipase C,PIPLC)和卵磷脂酶(phosphatidylcholine-PLC,PC-PLC)协助下裂解吞噬胞膜,帮助Lm逃离吞噬小体[10-11];Lm利用磷酸己糖(hexose phosphate,HP)在细胞浆中繁殖,HP的摄取是由hpt编码的己糖磷酸盐转运蛋白 (hexose phosphate transporter,Hpt)完成,Hpt酶的缺失会导致Lm毒力受损和侵入小鼠能力下降,Hpt是第一个被确定参与细胞内病原菌增值的毒力因子[12];与此同时,actA基因编码的肌动蛋白A(actin assembly-inducing protein,ActA)诱导宿主细胞肌动蛋白纤丝聚合在细菌末端,推动细菌移动,进入新的细胞裂解繁殖[13]。上述所有毒力基因的表达均受到PrfA蛋白全部或部分调控[14-16]。hly、plcA、plcB、mpl、actA和prfA基因组成的基因簇是Lm入侵宿主的关键因素,它的转录表达依赖PrfA蛋白[17]。Fang Chun等[18]将菌株EGDe和M7的prfA基因进行相互替换得到EGDe-prfAM7和M7-prfAEGDe,发现较低致病性的M7携带的prfA基因替换到EGDe后,菌株的InlA、InlB、LLO和ActA蛋白表达量上调,同时细胞的侵袭和黏附能力上升。除此之外,prfA基因的缺失也会导致细菌菌膜形成能力下降[19]。
为了解毒力基因prfA对EGDe菌株起到的作用,本研究使用温敏型穿梭质粒pKSV7,运用同源重组技术构建单缺失菌株EGDe-ΔprfA[20],该基因缺失菌株的构建及生物学特性研究为食源性致病菌Lm致病机理研究提供重要依据。
1.1 材料与试剂
1.1.1 菌株、质粒和细胞
野生株EGDe、结肠癌Caco-2细胞株为上海理工大学系统生物医学研究中心保存;pKSV7(温度敏感性穿梭载体,含有氯霉素(chloramphenicol,Cam)标记)由华中师范大学副教授罗勤馈赠;大肠杆菌感受态细胞DH5α和pMD19-T Vector购自大连宝生物公司。
1.1.2 试剂
限制性内切酶(SmaⅠ、XbaⅠ和SalⅠ)、Taq DNA聚合酶、dNTPs、DNA Ladder Marker、T4 DNA连接酶大连宝生物公司;脑心浸液(brain heart infusion,BHI)培养基、LB培养基 北京陆桥技术股份有限公司;细胞培养基、胎牛血清 上海赛齐生物工程有限公司;氨苄青霉素(ampicillin,Amp)、Cam 上海朝瑞生物科技有限公司;细菌基因组抽提试剂盒、核酸凝胶回收试剂盒、质粒抽提试剂盒 天根生化科技(北京)有限公司。
1.2 仪器与设备
9500型普通聚合酶链式反应(polymerase chain reaction,PCR)仪、7500型实时荧光定量PCR仪 美国ABI公司;SpectraMax M2型多功能全波长酶标仪 美国Molecular Devices公司;核酸电泳设备、电转化仪、凝胶成像仪及相关软件 美国Bio-Rad公司;Nano Drop微量生化测定仪 美国Thermo Fisher Scienti c公司;台式离心机 德国Eppendorf公司。
1.3 方法
1.3.1 引物序列
引物设计是根据NCBI相关报道使用DNAman软件设计,由生工生物工程(上海)股份有限公司合成,序列见表1。
表 1 EGDe-ΔprfA构建及鉴定所需引物
Table 1 Primer sequences used in the construction and identification of EGDe-ΔprfA

注:下划线表示酶切位点。
1.3.2 EGDe基因组DNA和总RNA的提取
EGDe基因组DNA和总RNA提取分别用冻融法和苯酚法。
1.3.3 prfA基因上下游同源臂的扩增及连接
分别用引物A1/A2和A3/A4扩增prfA基因的上下游片段,产物进行1.5%琼脂糖凝胶电泳,割胶回收并测定浓度,以OD260nm/OD280nm判断纯度,割胶回收具体步骤参考核酸凝胶回收试剂盒说明书。
割胶回收产物使用限制性内切酶XbaⅠ单酶切处理,37 ℃水浴5~6 h,电泳后割胶回收;酶切产物使用T4DNA连接酶16 ℃金属浴14~16 h进行上下游同源臂连接,电泳并割胶回收。
1.3.4 同源臂连接T载体并导入DH5α感受态细胞
DH5α感受态细胞制备:取过夜培养的DH5α E. coli
转接到100 mL LB培养基中,培养至OD600nm0.4~0.6;用0.1 mol/L CaCl210 mL洗涤2 次,离心;使用10 mL含10%~20%甘油的0.1 mol/L CaCl2重悬;1.5 mL离心管每管200 μL分装,冰浴5 h后-80 ℃保存。
连接pMD19-T vector:pMD19-T vector、上下游连接产物、solubionⅠ置于16 ℃金属浴30 min,转入DH5α感受态细胞中,冰浴30 min;42 ℃ 90 s热激,再冰浴1 min;加入890 μL LB培养基37 ℃摇床培养60 min;离心弃上清液,剩余100 μL重悬,涂布含100 μg/mL Amp的LB平板上,37 ℃培养。
1.3.5 阳性克隆鉴定
将抗性平板上的单菌落挑下,于含100 μg/mL Amp的LB培养基中摇床培养;取菌液使用引物A1/A4进行PCR鉴定上下游连接片段,电泳后将阳性克隆对应的菌液扩大培养抽提质粒。
1.3.6 同源臂与pKSV7连接
以pKSV7和连有同源臂的T载体为样本,使用两种内切酶,37 ℃水浴5~6 h,电泳后分离产物并割胶回收;割胶回收后的产物使用T4 DNA连接酶16 ℃金属浴15~16 h,后导入DH5α感受态细胞中,涂布含100 μg/mL Amp的LB平板并鉴定,方法同T载体;脱盐的质粒样本约20 μL送测序并比对。
1.3.7 重组质粒电转化EGDe感受态细胞
EGDe感受态细胞制备:用终质量浓度为5 μg/mL的青霉素G和3.5×SMHEM溶液(952 mmol/L蔗糖、3.5 mmol/L MgCl2和7 mmol/L HEPES溶于50 mL双蒸水,使用0.22 μm滤膜过滤除菌)处理EGDe菌体,将感受态细胞浓度控制在1.1×1011个/mL 左右,并以200 μL每管分装至小离心管中,-80 ℃保存。
电转参数:电击杯加入8 μL质粒和200 μL Lm感受态细胞,电转场强2.5 kV,时间4 ms;电转后菌体移入3~5 mL BHI中,37 ℃培养1~2 h,离心弃上清液重悬涂布于含10 μg/mL Cam BHI平板,30 ℃摇床培养48~72 h。1.3.8 prfA基因缺失突变株的筛选与鉴定
挑取平板中单菌落于含10 μg/mL Cam BHI培养基中培养41 ℃过夜,按1∶100(V/V)转接8~10 次,一天两次仍在41 ℃条件下培养,末代细菌划线接种于含10 μg/mL Cam BHI平板,41 ℃过夜培养;挑取上述平板中的单菌落于BHI液体中培养,30 ℃过夜后按1∶100(V/V)转接6~8 次,一天两次在相同条件下培养;末代培养物划线BHI平板30 ℃培养,挑单菌落使用引物A1/A4和A5/A6 PCR鉴定上下游连接片段和prfA基因片段,并将疑似株分别划线抗性和无抗性平板培养,抗性平板不生长、无抗性平板生长的为可疑突变株,扩增相应基因片段进行测序。
1.3.9 突变株生长曲线
将EGDe、EGDe-ΔprfA接种于BHI液体培养基中过夜培养,次日取1 mL饱和菌液转接到100 mL新鲜BHI液体培养基中摇床培养,每隔1 h取200 μL菌液加入96 孔板中测定OD600nm,共测定12 h,设置3 个平行组,重复3 次实验,用数据处理软件绘制生长曲线。
1.3.10 RT-PCR
将提取的EGDe、EGDe-ΔprfA总RNA反转录获得cDNA(反转录试剂盒中含有去除基因组DNA的相关试剂)、以cDNA为模板进行SYBR Green荧光染料法实时定量荧光PCR(real time-polymerase chain reaction,RTPCR),以EGDe的16S rRNA为内参基因,测定EGDe的9个主要毒力基因表达变化,以2-ΔΔCt表示所有基因的表达量,即EGDe所有基因的表达量均为1,EGDe-ΔprfA的各基因表达量大于1的为表达上调,小于1的为表达下调。
1.3.11 EGDe、EGDe-ΔprfA侵袭Caco-2细胞
将Caco-2细胞转移到12 孔板中培养,显微镜观察生长至铺满孔板;挑取EGDe、突变株单菌落过夜培养,调菌至OD600nm为0.3,菌数约108CFU/mL;孔板废液取出加入2 mL非完全培养基,加100 μL菌液,37 ℃培养箱4 h;吸掉废液用生理盐水洗涤,加庆大霉素1 mL/孔,放37 ℃培养箱30 min;1% Triton室温处理30 min,裂解后溶液取出放入1.5 mL离心管振匀,稀释10 倍涂BHI平板37 ℃培养并计数。
1.4 数据处理
本实验数据经Microsoft Excel与GraphPad Prism 5处理。
2.1 prfA基因上下游同源臂的扩增

图 1 上下游同源臂扩增
Fig. 1 Electrophoretogram of amplification products of the upstream and downstream flanking sequences
以EGDe基因组为模板扩增prfA基因的上下游同源臂,进行电泳后结果见图1。上游同源臂626 bp,下游同源臂636 bp,扩增的片段与预期的片段大小相符且特异性良好无杂带。
2.2 prfA基因上下游同源臂的连接

图 2 上下游同源臂连接
Fig. 2 Electrophoretogram showing successful ligation of the upstream and downstream flanking sequences
上下游同源臂连接结果见图2。上下游连接片段大小为1 262 bp,由图2可知,prfA基因上下游同源臂均连接成功。
2.3 导入DH5α感受态细胞
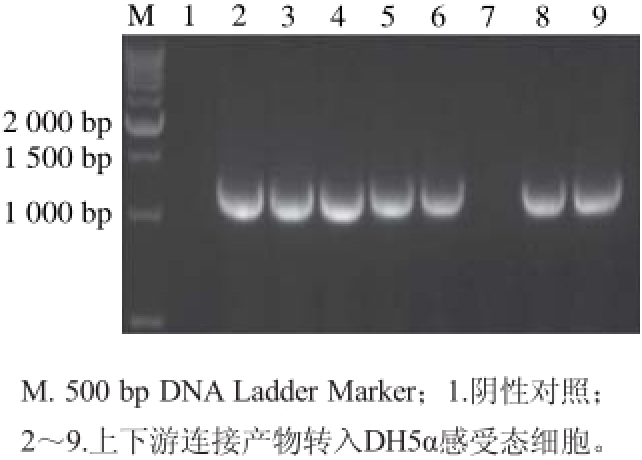
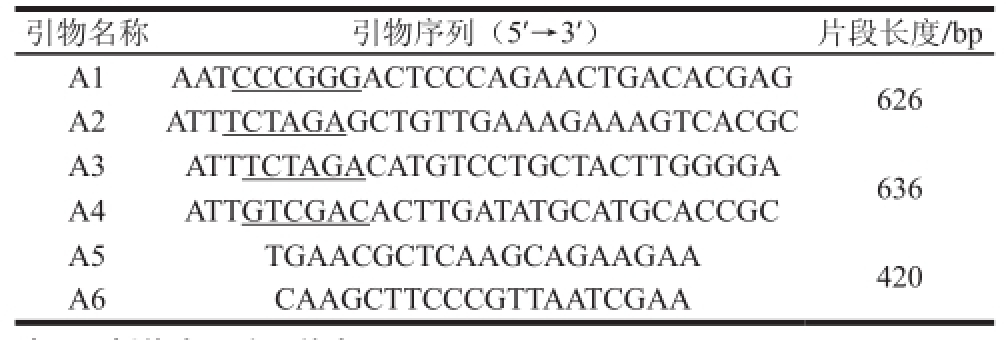
图 3 导入DH5α感受态细胞鉴定
Fig. 3 Identi fication of DH5α competent cells
上下游连接产物转入DH5α感受态细胞中鉴定结果见图3。图3为菌落PCR鉴定,其中阳性片段大小1 200 bp左右,为筛选菌株,进行扩大培养并-80℃冻存。
2.4 pKSV7重组质粒的构建
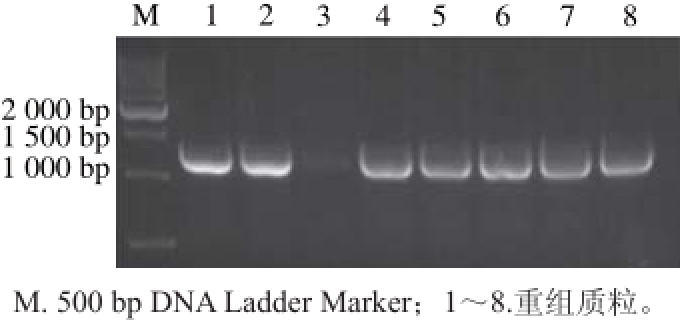
图 4 pKSV7重组质粒鉴定
Fig. 4 Identification of recombinant plasmid PKSV7
pKSV7重组质粒的鉴定结果见图4。泳道阳性片 段在1 200 bp附近,表明prfA上下游连接片段成功插入质粒pKSV7中。提取质粒送测序,比对结果显示序列相似度为100%,可以进行后续实验。
2.5 prfA基因缺失突变株的筛选与鉴定
电转后的菌体在一定条件下进行培养筛选,预期得到筛去质粒的目的菌株,挑取单菌落使用同源臂引物和目的基因引物进行PCR鉴定,鉴定结果见图5。同源臂引物A1/A4结合位点位于目的基因的外侧,即目的基因缺失后扩增产物应明显小于EGDe野生株为模板的阳性对照扩增产物;A5/A6位于目的基因内侧,目的基因缺失后无法扩增出相应片段,疑似菌株的电泳结果应为空白。
图 5 prfA基因缺失菌株的筛选
Fig. 5 Identification of the deletion mutant ΔprfA
图5 中,2号泳道为以EGDe基因组DNA作为模板的阳性对照,19号同源臂引物扩增产物比阳性对照片段小约1 000 bp左右,而用A5、A6引物则未扩增出任何产物,说明19号泳道菌株可能为突变菌株。将疑似突变菌株扩大培养涂布抗性平板和无抗性平板,结果抗性平板无菌落生长,将产物进行测序结果表明prfA基因已经缺失,prfA基因缺失菌株构建完成,并命名为EGDe-ΔprfA,菌液冻存-80 ℃用于后续实验。
2.6 生长曲线

图 6 EGDe、EGDe-ΔprfA生长曲线
Fig. 6 Growth curves of EGDe and EGDe-ΔprfA
取EGDe、EGDe-ΔprfA 1 mL饱和菌液转接到100 mL新鲜BHI液体培养基中摇床培养测定OD600nm比较2 种菌株的活力是否不同,结果见图6。根据图6显示,prfA基因缺失后的菌株生长趋势与野生株并无明显差异。
2.7 RT-PCR结果
EGDe-ΔprfA的9 个主要毒力基因RT-PCR实验结果见图7。以野生EGDe毒力基因的表达水平1为基准,EGDe-ΔprfA中的prfA基因几乎没有表达,说明基因缺失菌株构建成功;inlA、inlB、inlC、actA、vip表达量均有明显下调(P<0.05);sigB基因的表达量无明显变化;plcA、plcB表达量分别上升至原来的2.5 倍和2 倍。由于基因调控的复杂性和本实验中RT-PCR针对的毒力基因有一定的局限性,EGDe-ΔprfA毒力的具体变化情况,还需要进行后续的细胞侵袭实验进行验证。
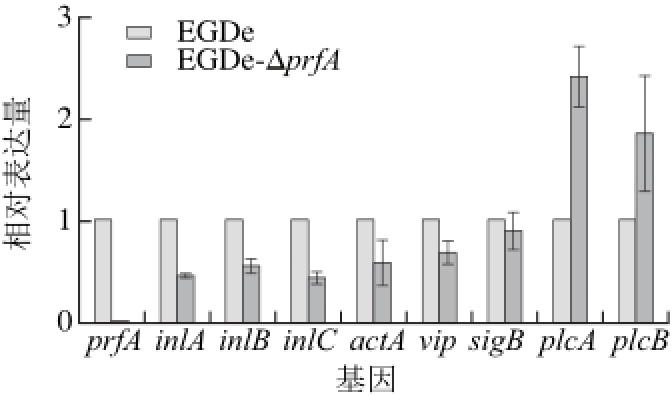
图 7 EGDe-ΔprfA毒力基因RT-PCR
Fig. 7 RT-PCR amplification of virulence genes of EGDe-ΔprfA
2.8 EGD e、EGDe-ΔprfA侵袭Caco-2细胞
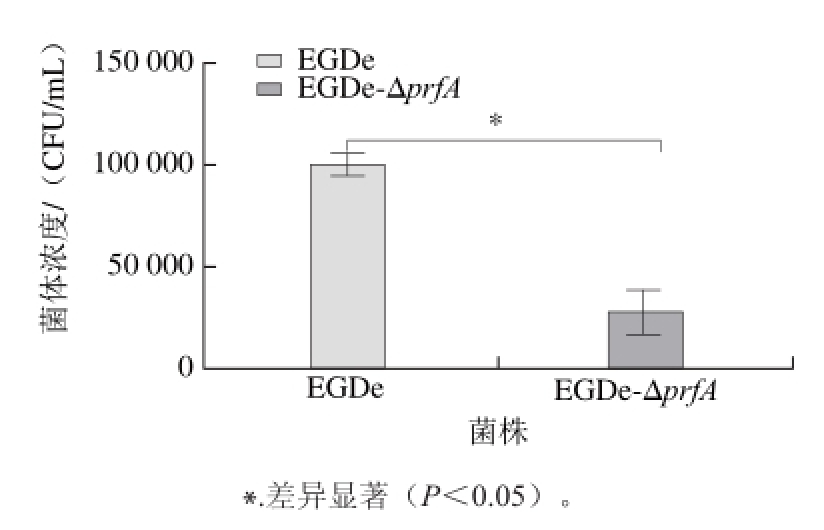
图 8 各菌株菌落总数
Fig. 8 Numbers of bacterial colonies of EGDe and EGDe-ΔprfA after invasion into Caco-2 cells
分别对EGDe、EGDe-ΔprfA侵袭Caco-2细胞后细胞裂解液稀释涂平板,对单菌落计数结果见图8。细胞侵袭实验结果表明,与EGDe侵袭能力相比,EGDe-ΔprfA基因缺失株的侵袭能力明显下降,EGDe-ΔprfA细菌侵袭数量仅为野生株1/5,说明prfA基因敲除会降低EGDe的毒力。
InlA 、InlB、LLO、PlcA、PlcB、Mpl、Hpt、ActA、InlC 9 个主要的毒力因子严格受PrfA调控,除此之外多达145 个其他的单核细胞增生李斯特菌基因在转录组学和蛋白组学分析中受到PrfA影响,这些间接调控的基因包括转运、代谢、调节、应激反应调控和其他未知功能蛋白[21-24]。目前对PrfA的功能有了一定的了解,但是很多机理尚不清楚,有待进一步的研究。
PrfA对毒力基因的调控是一个复杂的机制,用EGDe-ΔprfA、EGDe-ΔinlA、EGDe-ΔinlB和EGDe-Δhly(hly,编码LLO蛋白)几种毒力基因缺失株感染鸡胚
胎,发现prfA基因缺失和hly基因缺失降低了EGDe的毒力,然而EGDe-ΔinlA和EGDe-ΔinlB两种基因缺失株对鸡胚胎的致死率与野生株相比并未下降,这证明了PrfA对
EGDe毒力起到调控作用[25]。本研究通过同源重组技术成功对prfA基因进行了敲除,同时研究了单缺失菌株EGDe-ΔprfA部分生物学特性。生长曲线结果表明prfA基因缺失后的菌株生长趋势与野生株并无太大差别,生长能力没有影响;但prfA基因缺失造成多个毒力基因表达下降,细胞侵袭实验表明prfA基因的缺失使EGDe的毒力有明显的下降,证明了prfA基因的调控作用,为深入研究prfA基因介导EGDe入侵宿主细胞过程中的作用机制的研究提供基础参考数据。
参考文献:
[1] MAGALH☒ES R, MENA C, FERREIRA V, et al. Bacteria: Listeria monocytogenes[J]. Encyclopedia of Food Safety, 2014, 1: 450-461. DOI:10.1016/B978-0-12-378612-8.00101-3.
[2] GRAY M J, FREITAG N E, BOOR K J. How the bacterial pathogen Listeria monocytogenes mediates the switch from environmental Dr. Jekyll to pathogenic Mr. Hyde[J]. Infection and Immunity, 2006, 74(5): 2505-2512. DOI:10.1128/IAI.74.5.2505-2512.2006.
[3] 丁建英, 韩剑众. 食品中单增李斯特菌的存在现状及检测方法研究进展[J]. 食品研究与开发, 2008, 29(12): 171-174. DOI:10.3969/ j.issn.1005-6521.2008.12.050.
[4] CHATURONGAKUL S, RAENGPRADUB S, WIEDMANN M, et al. Modulation of stress and virulence in Listeria monocytogenes[J]. Trends in Microbiology, 2008, 16(8): 388-396. DOI:10.1016/j.tim.2008.05.006.
[5] COSSART P, TOLEDO-ARANA A. Listeria monocytogenes, a unique model in infection biology: an overview[J]. Microbes and Infection, 2008, 10(9):1041-1050. DOI:10.1016/j.micinf.2008.07.043.
[6] 李文艳, 董明倩, 王亚光, 等. 单增李斯特菌感染过程中炎性体激活的研究进展[J]. 微生物学通报, 2016, 43(3): 634-639. DOI:10.13344/ j.microbiol.china.150487.
[7] 任防振, 徐国勋. 兼氧/好氧膜生物反应器处理食品废水研究[J].上海理工大学学报, 2007, 29(3): 285-288. DOI:10.3969/ j.issn.1007-6735.2007.03.018.
[8] COSSART P, PIZARRO-CERDA J, LECUIT M. Invasion of mammalian cells by Listeria monocytogenes: functional mimicry to subvert cellular functions[J]. Trends in Cell Biology, 2003, 13(1): 23-31. DOI:10.1016/S0962-8924(02)00006-5.
[9] 殷月兰, 董慧, 焦新安, 等. 产单核细胞李斯特菌actA基因在大肠杆菌中的表达及其单克隆抗体的研制[J]. 微生物学报, 2006, 46(6): 999-1002. DOI:10.3321/j.issn:0001-6209.2006.06.027.
[10] VAZQUEZ-BOLAND J A, KUHN M, BERCHE P, et al. Listeria pathogenesis and molecular virulence determinant[J]. Clinical Microbiology Reviews, 2001, 14(3): 584-640. DOI:10.1128/ CMR.14.3.584-640.2001.
[11] BAVDEK A, KOSTANJSEK R, ANTONINI V, et al. pH dependence of listeriolysin O aggregation and pore-forming ability[J]. FEBS Journal, 2012, 279(1): 126-141. DOI:10.1111/j.1742-4658.2011.08405.x.
[12] CHICO-CALERO I, SUAREZ M, GONZALEZ-ZORN B, et al. Hpt, a bacterial homolog of the microsomal glucose-6-phosphate translocase, mediates rapid intracellular proliferation in Listeria[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(1): 431-436. DOI:10.1073/pnas.012363899.
[13] KNOWLES H, HEIZER J W, LI Y, et al. Transient receptor potential melastatin 2 (TRPM2) ion channel is required for innate immunity against Listeria monocytogenes[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(28): 11578-11583. DOI:10.1073/pnas.1010678108.
[14] SCORTTI M, MONZO H J, LACHARME-LORA L, et al. The PrfA virulence regulon[J]. Microbes and Infection, 2007, 9(10): 1196-1207. DOI:10.1016/j.micinf.2007.05.007.
[15] HERAS A D L, CAIN R J, BIELECKA M K, et al. Regulation of Listeria virulence: PrfA master and commander[J]. Current Opinion in Microbiology, 2011, 14(2): 118-127. DOI:10.1016/j.mib.2011.01.005. [16] SWAMINATHAN B, GERNER-SM IDT P. The epidemiology of human listerio sis[J]. Microbes and Infection, 2007, 9(10): 1236-1243. DOI:10.1016/j.micinf.2007.05.011.
[17] KREFT J, VAZQUEZ-BOLAND J A. Regulation of virulence genes in Listeria[J]. International Journal of Medical Microbiology, 2001, 291(2): 145-157. DOI:10.1078/1438-4221-00111.
[18] FANG C, CAO T, CHENG C Y, et al. Activation of PrfA results in overexpression of virulence factors but does not rescue the pathogenicity of Listeria monocytogenes M7[J]. Journal of Medical Microbiology, 2015, 64(8): 818-827. DOI:10.1099/jmm.0.000101.
[19] LUO Q, SHANG J L, FENG X Q, et al. PrfA led to reduce d bio lm formation and contributed to altered gene expression patterns in biofilm-forming Listeria monocytogenes[J]. Current Microbiology, 2013, 67: 372-378. DOI:10.100 7/s00284-013-0377-7.
[20] HUANG Y Y, SUO Y J, SHI C L, et al. Mutations in gltB and gltC reduce oxativeid stress tolerance and biofilm formation in Listeria mono cytogenes 4b G[J]. International Journal of Food Microbiology, 2013, 163(2/3): 223-230. DOI:10.1016/j.ijfoodmicro.2013.02.023.
[21] XAYARATH B, SMART J I, M UELLER K J, et al. A novel C-terminal mutation resulting in constitutive activation of the central virulence regulatory factor PrfA[J]. Microbiology, 2011, 157(Pt 11): 3138-3149. DOI:10.1099/mic.0.049957-0.
[22] 马婷, 李芳, 单大亚. 基于物联网技术的食品冷链物流跟踪及追溯问题研究[J]. 上海理工大学学报, 2013, 35(6): 557-562. DOI:10.3969/j.issn.1007-6735.2013.06.009.
[23] BÉCAVIN C, BOUCHIER C, LECHAT P, et al. Comparison of widely used Listeria monocytogenes strains EGD, 10403S, and EGD-e highlights genomic variations underlying differences in pathogenicity[J]. mBio, 2014, 5(2): e00969-14. DOI:10.1128/ mBio.00969-14.
[24] CHE N J, CHEN Q, JIANG L, et al. Internalin pro ling and multilocus sequence typing suggest four Listeria innocua subgroups with different evolutionary distances from Listeria monocytogenes[J]. BMC Microbiology, 2010, 10(1): 97. DOI:10.1186/1471-2180-10-97.
[25] ANDERSSON C, GRIPENLAND J, JOHANSSON J. Using the chicken embryo to assess virulence of Listeria monocytogenes and to model other microbial infections[J]. Nature Protocols, 2015, 10(8): 1155-1164. DOI:10.1038/nprot.2015.073.
Construction and Biological Activity of a Strain of Listeria monocytogenes Deleted for Positive Regulatory Factor A Gene (prfA)
GUO Liang, CHEN Guowei, XIE Manman, LIU Wukang, DING Chengchao, WANG Shujuan, LUO Qin, LIU Qing*
(School of Medical Instrument and Food Engineering, University of Shanghai for Science and Technology, Shanghai 200093, China)
Abstract:Among the known Listeria strains, only Listeria monocytogenes (Lm) can cause human disease. The expression of most virulence genes is regulated fully or partially by positive regulatory factor A (PrfA) protein encoded by prfA gene. In this study, we adopted homologous recombination method to knock out the prfA gene of the wild strain EGDe, and investigated the biological characteristics of EGDe-ΔprfA by testing growth state, virulence gene expression and Caco-2 cell invasion. There was no difference in the growth status of EGDe-ΔprfA and EGDe as demonstrated by the growth curves. Real time quantitative PCR results showed that the expression levels of plcA and plcB genes increased by 2.5 times and twice, respectively, while the expression levels of inlA, inlB, inlC, actA and vip declined. The expression level of prfA gene was nearly zero. The invasion of Caco-2 cells by EGDe-ΔprfA was onefth as high as that by the wild strain. The gene-deleted strain can provide an important tool for studying the pathogenic mechanism of the foodborne pathogen Listeria monocytogenes.
Key words:Listeria monocytogenes; prf A; gene knock-out; biological characteristics
DOI:10.7506/spkx1002-6630-201710003
中图分类号:TS201.3
文献标志码:A
文章编号:1002-6630(2017)10-0012-06
引文格式:
郭亮, 陈国薇, 谢曼曼, 等. 单增李斯特菌prfA基因缺失菌株的构建及其生物学特性鉴定[J]. 食品科学, 2017, 38(10): 12-17. DOI:10.7506/spkx1002-6630-201710003. http://www.spkx.net.cn
GUO Liang, CHEN Guowei, XIE Manman, et al. Construction and biological activity of a strain of Listeria monocytogenes deleted for positive regulatory factor A gene (prfA)[J]. Food Science, 2017, 38(10): 12-17. (in Chinese with English abstract)
DOI:10.7506/spkx1002-6630-201710003. http://www.spkx.net.cn
收稿日期:2016-07-22
基金项目:国家自然科学基金面上项目(31371776);上海市科委“科技创新行动计划”长三角科技联合攻关领域项目(15395810900);上海理工大学研究生创新基金资助项目;上海理工大学微创励志创新基金项目
作者简介:郭亮(1992—),男,硕士研究生,研究方向为食源性致病菌致病机理。E-mail:guoliangyxh@163.com
*通信作者:刘箐(1970—),男,教授,博士,研究方向为食源性致病菌致病机理及快速检测技术。E-mail:liuq@usst.edu.cn