表 1 正常反应中的引物序列
Table 1 Pr imers used for normal RT-qPCR

注:下划线标识碱基发生错配的位置,下同。
李金春,李家鹏*,周 彤,乔晓玲*,许随根,戚 彪,米瑞芳,曲 超,许 典
(中国肉类食品综合研究中心,肉类加工技术北京市重点实验室,北京 100068)
摘 要:以错配反应与正常反应之间的ΔCt值为指示,研究不同碱基错配类型、位置与个数条件下引物3´端与模板之间非特异性扩增的发生规律,并构建ΔCt值的二次多项式回归模型。结果表明,碱基错配种类、位置、个数对实时荧光定量聚合酶链式反应(quantitative real-time polymerase chain reaction,RT-qPCR)的非特异性扩增都有显著影响,并呈现出较强的规律性。碱基类型上,研究的各个位置上异型错配较易发生非特异性扩增,而同型错配则较难发生,并且两种错配类型之间有显著性差异(P<0.05);错配位置上,相同条件下引物3’端第1位碱基错配对ΔCt值影响最大,错配位置远离3’端时对ΔCt值的影响逐渐减小,非特异性扩增也更容易发生;错配个数上,随着错配碱基个数的增加对ΔCt值得影响程度不断增大,非特异性扩增较难发生。构建ΔCt值预测模型R2达到0.837 1,根据此模型可以预测出不同位置、不同错配类型条件下ΔCt值,从而可以判断RT-qPCR非特异性扩增的发生的难易程度。
关键词:肉类掺假;循环阈值;碱基错配;非特异性扩增;引物特异性
实时荧光定量聚合酶链式反应(quantitative realtime polymerase chain reaction,RT-qPCR)技术具有准确度高、稳定性好、高效便捷等优点,已广泛应用于食品真伪与掺假鉴别[1-4]、食品转基因成分检测[5-6]、食品致病菌检测[7-9]等多个检测项目中,已成为肉类品种鉴别的主流方法[10-11],在食品安全监管中发挥着重要作用。引物特异性是RT-qPCR方法体系中关键性能指标之一,特别是对用于食品真伪检测的RT-qPCR方法来说更是核心指标[12-13]。设计和选取的引物体系对扩增目标DNA序列需要具有唯一性[14],即在具有多种相近序列的复杂DNA模板中,引物体系只扩增出目标模板,而不扩增其他的相似序列DNA模板。但当存在相似DNA序列的复杂样本时,引物和模板之间容易发生非特异性扩增,导致出现假阳性结果,对检测结果判定带来干扰[15]。
国内外对引物与模板之间非特异性扩增的研究相对较少。Wu等[16]将引物中每个碱基分别替换成其余3 种碱基后得到一系列新引物,新引物再与模板进行非特异性扩增,发现碱基错配位置对PCR反应扩增效率有较大影响,并提出一种评价引物扩增能力的方法;Brett等[17]研究了引物中间位置和靠近引物5’端的单个或多个碱基与模板发生非特异性扩增对RT-qPCR扩增准确性的影响,发现中间位置和引物5’端的碱基错配也能较大程度的降低RT-qPCR定量准确性。Süß等[18]发现在RT-qPCR引物和探针中的单碱基错配都能够引起较大的定量误差,误差甚至高到63.12%。Klungthong等[10]发现引物3’端单一碱基错配便可较大程度的降低方法灵敏度。目前,尚缺少系统考察3个因素:碱基错配类型、位置、个数对RT-qPCR非特异性扩增发生规律影响的报道,而现有报道也多集中于对其中某一因素的研究,以及碱基错配对扩增效率、定量准确性和方法灵敏度等方面的研究,因此研究影响非特异性扩增反应发生的关键因素及其作用规律就显得尤为迫切。
为探究引物和模板之间非特异性扩增的发生规律,筛选本实验室属性相近、序列各异的引物作为研究基础。由于引物3’端核苷酸序列对RT-qPCR特异性扩增起到至关重要的作用,因此选取引物3’端核苷酸序列作为研究对象,在正常引物的基础上人为设计一系列错配引物,研究引物中错配碱基种类、个数、位置对RT-qPCR非特异性扩增发生规律的影响,并通过错配反应与正常反应之间的Ct值之差(ΔCt值)的预测模型来判断非特异性扩增的发生的难易程度。研究错配引物与目标物种模板之间RT-qPCR非特异性扩增的发生规律,对于建立引物评价体系和设计技术都具有重要意义。
1.1 材料与试剂
猪四号肉 北京资源公司;牛肉 北京御香苑畜牧有限公司;Qiagen69504血液、组织DNA提取试剂盒德国Qiagen公司;琼脂糖 西班牙Biowest公司;D2000 DNA Marker 天根生化科技有限公司;AxyPrep DNA凝胶回收试剂盒 美国Axygen公司;TaKaRa 2×酶体系预混液 大连宝生物工程(大连)有限公司;SYBR GreenⅠMaster 德国Roche公司;RT-qPCR 96 孔板罗氏诊断产品(上海)有限公司。实验中涉及引物均由实验室自行设计,并由英潍捷 基(上海)有限公司合成。
1.2 仪器与设备
Prep多样品匀浆仪 美国Omni公司;BT224S分析天平 赛多利斯科学仪器(北京)有限公司;LightCycler 480荧光PCR仪 罗氏诊断产品(上海)有限公司;PCR仪、Biorad GelDoc XR凝胶成像仪 美国Bio-Rad公司;S-100涡旋振荡器 大洋科学工业株式会社;Eppendorf 5417型离心机 艾本德中国有限公司;Synergy H4全功能酶标仪 美国BioTek公司;DYCP-31F琼脂糖水平电泳仪 北京六一仪器厂;GI54DW全自动高压灭菌器 美国致微(厦门)仪器有限公司;Cascada BIO超纯水系统 美国Pall公司。
1.3 方法
1.3.1 DNA提取方法
将肉和水按照1∶4(m/m)的比例进行准确称量并进行匀浆,13 500 r/min匀浆10 min。吸取100 μL匀浆液于1.5 mL离心管中,按照试剂盒说明书操作提取基因组DNA,总DNA溶解于100 μL TE缓冲液中,通过酶标仪测定280 nm和260 nm波长处的吸光度,计算DNA纯度和质量浓度。然后用TE缓冲液稀释至5 ng/μL,作为普通PCR反应模板。
1.3.2 RT-qPCR模板制备方法
模板通过普通PCR反应进行扩增,PCR产物用1%的琼脂糖凝胶进行电泳分离,然后用凝胶试剂盒回收得到。普通PCR反应为25 μL体系,其中TaKaRa 2×酶体系预混液12.5 μL;浓度为5 μmol/L的上、下游引物各1.5 μL,引物序列如表1所示;质量浓度为5 ng/μL全基因组模板溶液2.0 μL;灭菌双蒸水补足体积至25 μL。普通PCR循环条件为95 ℃预变性5 min;95 ℃变性30 s,60 ℃退火30 s和72 ℃延伸30 s,共计35 个循环;最后72 ℃延伸5 min。
表 1 正常反应中的引物序列
Table 1 Pr imers used for normal RT-qPCR

注:下划线标识碱基发生错配的位置,下同。
扩增结束后按照凝胶回收试剂盒操作流程对PCR产物进行回收,然后通过酶标仪测定回收得到的目的片段质量浓度,用TE缓冲液稀释至2×10-3ng/μL,作为RT-qPCR的模板。
1.3.3 错配反应引物设计与合成
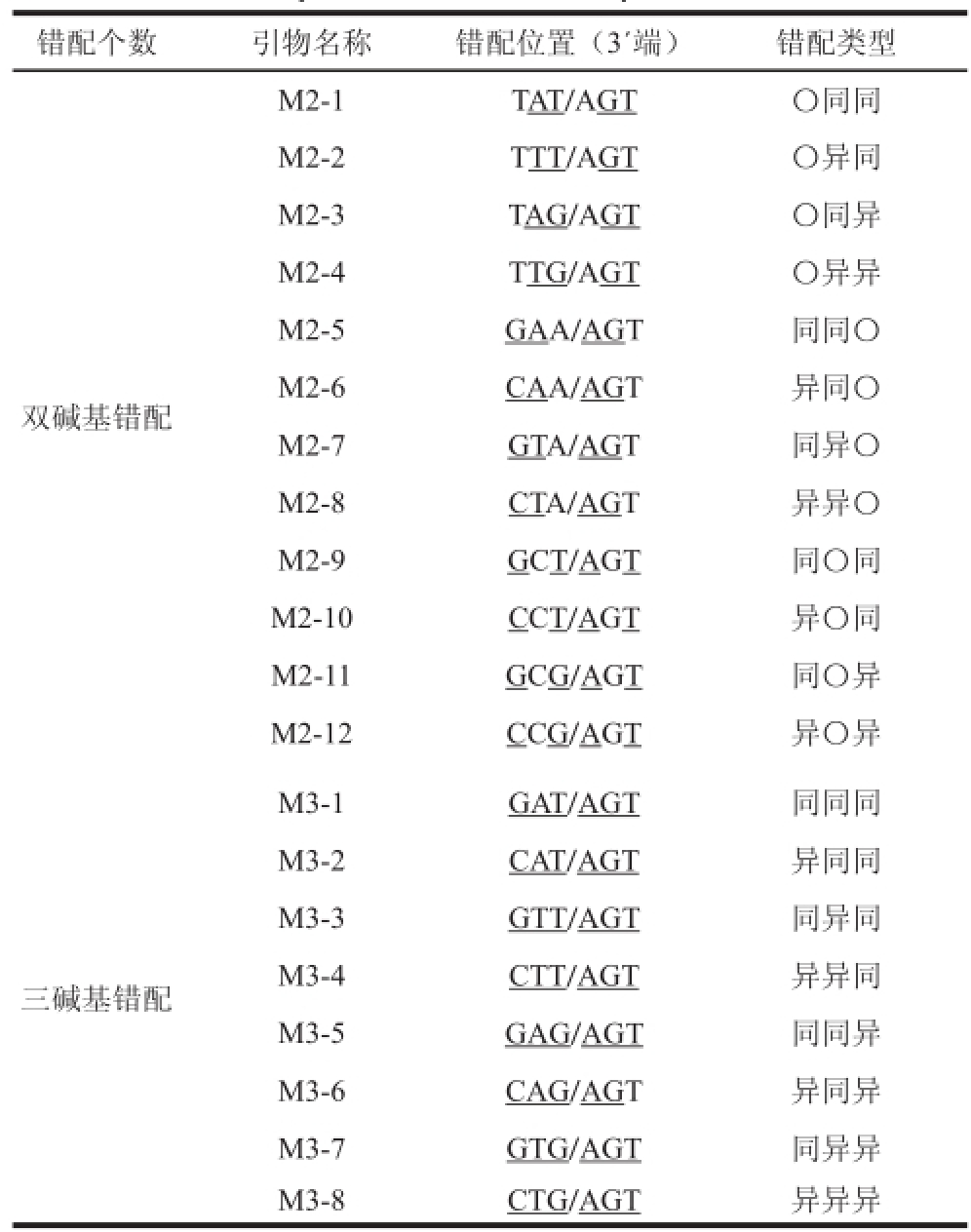
表 2 单碱基错配实验方案
Table 2 Experimental program of single base-pair mismatches in RT-qPCR

注:a.同型指配对碱基同为嘌呤或者同为嘧啶,异型则指配对碱基一个为嘌呤另一个为嘧啶;b.“TGA/ACA”指引物上碱基A与模板上碱基A发生错配反应,“/”前的碱基为引物上碱基,“/”后的碱基为模板上碱基,下同。
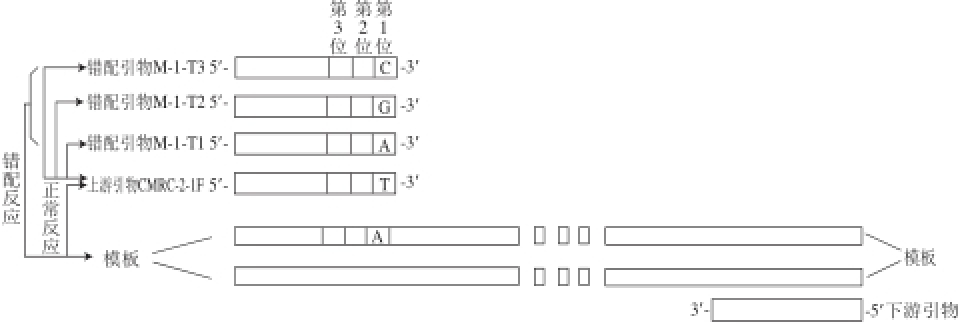
图 1 RT-qPCR反应过程中的非特异性扩增
Fig. 1 Schematic diagram of base pair mismatch in RT-qPCR
以引物CMRC-2-1F为例描述PCR反应非特异性扩增过程,具体如图1所示。筛选本实验室属性相近、序列各异的引物作为基础(表1),按照表2和表3方案合成系列错配引物。其中表2是单碱基错配方案,涵盖了3 个位置不同碱基配对的所有36 种情况。表3列出了2 个和3 个碱基典型错配方案,包含典型错配类型。
表 3 双碱基、三碱基错配实验方案
Table 3 Experimental program of double base-pair and triple basepair mismatches in RT-qPCR
注:“〇”指该位置碱基未发生错配,“同”指该位置引物碱基与模板发生同型配对,“异”指该位置碱基发生异型配对。下同。
1.3.4 RT-qPCR反应体系
用1.3.3节中合成的引物与其对照引物,及对应模板组成RT-qPCR反应扩增体系,分别记录错配引物及其正常引物的Ct值,并计算其Ct值之差,作为衡量非特异性扩增反应发生难易程度的衡量标准。ΔCt值越大代表非特异性扩增越难发生,反之则容易发生。
RT-qPCR扩增体系为10 μL体系,其中Roche 2×酶体系预混液5 μL;浓度为5 μmol/L的上、下游引物各0.6 μL;质量浓度为2×10-3ng/μL模板溶液2.0 μL;灭菌双蒸水补足体积至10 μL。RT-qPCR循环条件为:95 ℃预变性5 min;95 ℃变性10 s,退火和延伸合并为一步,温度为60 ℃,时间为45 s,共计40 个循环。
1.3.5 ΔCt值预测模型构建
由单碱基错配结果可将碱基错配对ΔCt值得影响程度由小到大分为5 个等级,然后给所有单碱基、双碱基和三碱基错配情况(共计56 种情况)的不同位置按照错配等级赋予分值,而不同的错配情况又对应不同的ΔCt值,则可以得到引物3’端3 个位置与ΔCt值之间的数量对应关系。然后通过二次多项式逐步回归的方法便可得到ΔCt值与引物3’端3 个位置上不同错配类型之间的数学预测模型,模型公式见式(1)[20]:
式中:x、y、z分别表示引物3’端第3位、第2位、第1位;pi(i=1,2,310)为多项式回归系数。
1.4 数据处理与统计分析
用DPS 7.05软件(中国,唐启义)对ΔCt预测模型进行数据处理和模型的构建;用PASWStatistics 18.0软件(美国SPSS公司)进行位置以及个数对ΔCt影响的显著性分析;t检验和R2以及数据处理通过Microsoft Excel 2007软件(美国微软公司)进行数据处理分析。
2.1 碱基错配类型对非特异性扩增的影响
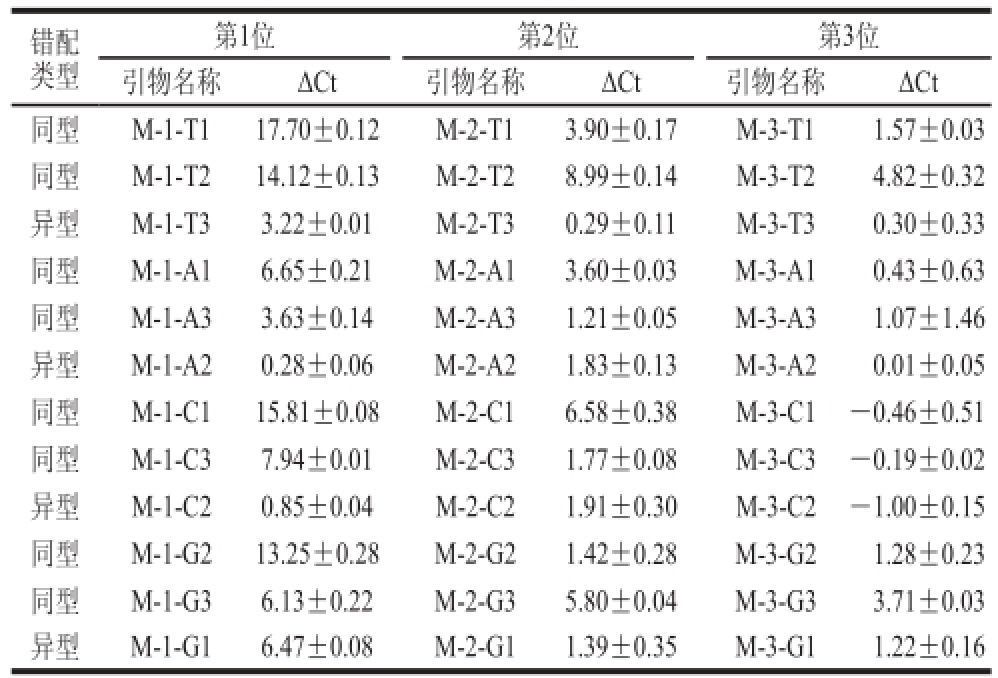
结果显示,碱基错配种类对非特异性扩增出现的难易程度有显著影响。基本上,引物与模板对应位置上出现同类型碱基错配(嘌呤/嘌呤,或嘧啶/嘧啶)会对非特异性扩增产生明显的抑制作用,而异型碱基错配(嘌呤/嘧啶,或嘧啶/嘌呤)对抑制非特性扩增的作用不大。由表4可知,当将引物M-1-T1 3’端第1位T被替换成A(A/A)和G时(G/A),ΔCt分别达到了17.70和14.12,而替换成C时,ΔCt仅为3.63,前者发生非特异性扩增的概率不足后者的千分之一。t检验结果显示,3’端第1位同型错配和异型错配之间对ΔCt值影响有极显著差异(P<0.01),无论是第2位还是第3位两种错配类型之间对ΔCt值的影响有显著差异(P<0.05),且该影响程度差距逐步缩小。这可能与4种核苷酸的分子结构有关,同类核苷酸之间的错配DNA合成酶的容错率更低一些。实验结果证实了异型错配发生的难易程度明显低于同型错配,而异型错配之所以容易发生,可能是由于核苷酸的5-甲基胞嘧啶脱氨基反应相对比较容易发生导致的[21-22],这与生物遗传中突变类型多为转换结果一致[23]。另外由表4可知,错配反应中有碱基A参与的,一般ΔCt值都会偏大,说明碱基种类A对非特异性扩增产生明显的抑制作用。
表 4 单碱基错配RT-qPCR反应中ΔCt值
Table 4 Δ Ct values of single base-pair mismatches in RT-qPCR
2.2 碱基错配位置对非特异性扩增的影响
表 5 双碱基、三碱基错配RT-qPCR中ΔCt值
Table 5 ΔCt values of double and triple base-pair mismatches in RT-qPCR
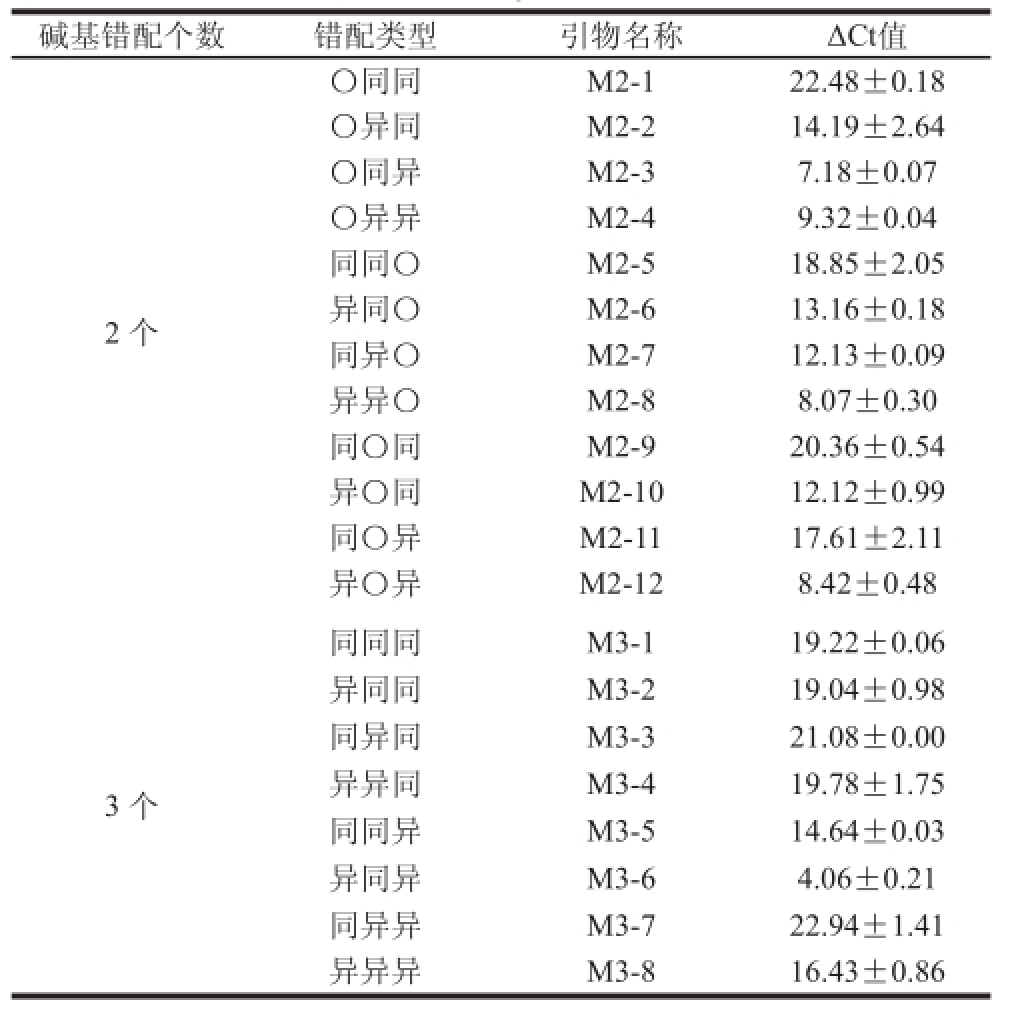
碱基错配位置对非特异性扩增发生的难易程度有显著影响,且越是远离3’端错配位置对ΔCt值的影响呈现逐渐减小的趋势。由表4可知,单碱基错配反应自3’端起3 个位置ΔCt的平均值依次为8.04、3.22、1.06,最大值依次为17.70、8.99、4.82,均呈现逐渐减小的趋势。这说明越是远离3´端错配位置对ΔCt值的影响呈现逐渐减小的趋势,此时RT-qPCR非特异性扩增越是容易发生,这与Ayyadevara等[24]研究的结果相一致。而由t检验可知,错配类型为异型错配时,3’端第1位和第2位之间对ΔCt值影响不显著(P=0.11),但两者与第3位相比均具有显著性差异(P<0.05);当为同型错配时,3’端3 个位置间对ΔCt值影响具有极显著的差异(P<0.01),说明碱基错配位置对非特异性扩增发生的难易程度有显著影响。而由表5可知,双碱基错配中第1、2位碱基错配最大ΔCt值为22.48,第1、3位的为20.36,而第2、3位的为18.85,同样说明碱基错配位置越靠近3’端对ΔCt值的影响越大,非特异性扩增越难以发生,这与单碱基错配结果相一致。而由t检验结果可知,三者之间并无显著差异,说明当碱基错配个数增加后错配位置对Δ Ct值影响减弱。
2.3 碱基错配个数的影响
由表4、5可知,碱基错配个数对ΔCt值得影响同样明显。引物3’端3 个碱基发生的错配反应中,单碱基、双碱基以及三碱基错配ΔCt值的平均值依次为4.10、13.66、17.15,ΔCt值的最大值依次为17.70、22.48、22.94,两者均呈现逐渐增大的趋势,说明随着错配碱基个数的不断增加,对ΔCt值的影响程度不断增大,对非特异性扩增的抑制也不断增强。而由t检验结果显示,错配类型为异型错配时,单个、双个以及3 个碱基错配时对ΔCt值的影响存在极显著差异(P<0.01);而当错配类型为同型错配时,单碱基错配分别与双碱基错配和三碱基错配之间存在显著性差异(P<0.05),但双碱基错配与三碱基错配之间对ΔCt值的影响并无显著差异(P=0.16),说明错配碱基个数增加到一定程度后对ΔCt值的影响程度便会减弱。
2.4 因素交互作用研究与Δ Ct值预测模型构建
为研究因素之间的交互作用和数量相关关系,用三元二次多项式对上述结果进行回归分析。由单碱基错配结果可知,同型错配的ΔCt值明显高于异型错配的,并且相同错配类型条件下含有碱基A的ΔCt值也相对偏大,据此将碱基错配对ΔCt值得影响程度由小到大分为5 个等级,分别为“未发生错配”、“异型错配”、“异型错配(含碱基A)”、“同型错配”以及“同型错配(含碱基A)”。并且给每个等级依次赋值“0、1、2、3、4”。将所有单碱基、双碱基和三碱基错配情况(共计56种情况)的不同位置按照错配等级进行赋分,而不同的错配情况又对应不同的ΔCt值,则可以得到引物3’端3 个位置与Δ Ct值之间的数量对应关系,如表6所示。
通过二次多项式逐步回归的方法对表6中数据进行拟合,得到3 个位置上不同错配类型与ΔCt的预测模型,模型公式见式(2):
ΔCt=-8.49+5.41x+8.32y+8.62z-0.52x2-1.26y2-0.76z2-0.21xy-0.40xz-0.62yz (2)
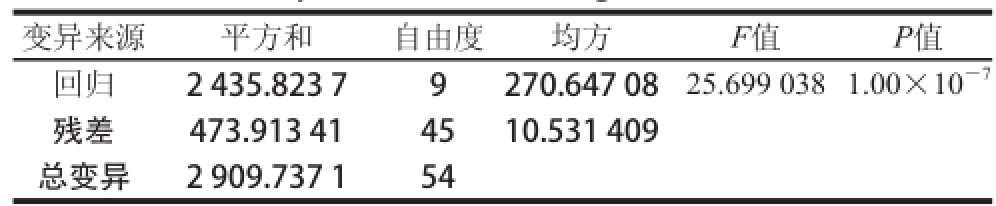
通过公式(2)可知,引物3’端3 个位置上不同错配类型对ΔCt值都有较大的影响,而从公式中二次项xy、yz以及xz可知,3 个位置中的任意两个之间都存在明显的交互作用。对表6的实验结果进行统计分析,得到的方差结果如表7所示,模型F值为25.70,P<0.000 1,模型是极显著的,模型 拟合成功。回归模型R2达到0.837 1,根据此模型在一定程度上可以预测出不同位置、不同错配类型条件下ΔCt值。
表 6 ΔCt值与3’端3 个位置上不同错配类型之间的数量关系
Table 6 Relationship between ΔCt value and mismatch types

注:a.引物M3-6的Δ Ct值为异常值,拟合模型时弃之不用。
表 7 Δ Ct值回归模型方差分析结果
Table 7 Analysis of variance for regression model for ΔCt
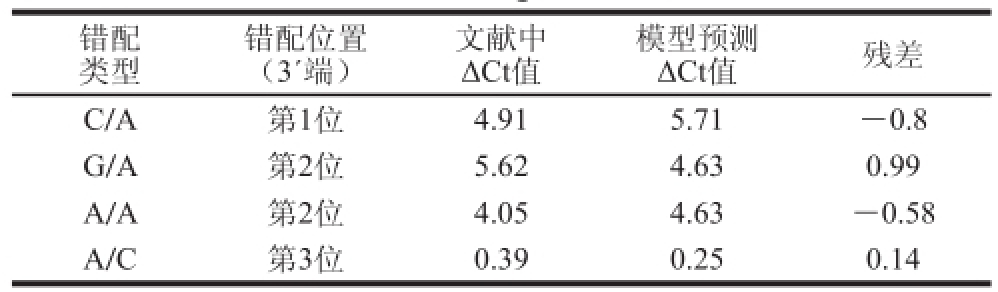
将文献[25]报道的单碱基错配ΔCt值与通过模型得到的预测值进行比较验证,结果如表8所示,可以看出模型残差值的绝对值均小于1,上下浮动于零左右,说明该模型预测的ΔCt值是可信的。因此,应用二次多项式回归模型能够预测出引物3’端不同位置、不同错配类型条件下Δ Ct值,从而可以判断RT-qPCR非特异性扩增的发生规律。
表 8 ΔCt值回归模型的残差值
Table 8 Residuals of regression model for ΔCt
肉类掺假售假事件近年来频频发生,2013年更是爆发了欧洲“马肉风波”事件[26-27],引起了全球关注,基于RT-qPCR的动物源性成分检测技术也随之成为研究热点[28-30]。由于物种之间DNA序列相近,极易产生非特异性扩增,导致假阳性结果出现,因此该检测技术对引物特异性要求较高,为引物设计和筛选带来了较大难度,并且引物设计完成后通常需要做特异性评价实验,该评价不但费时费力,而且需要收集尽可能多的物种肌肉或血液等生物样本,极大增加了方法开发成本。
本实验通过引物3’端一个或者多个碱基与模板发生错配反应引起Ct值变化来评价RT-qPCR中非特异性扩增的发生规律,发现引物上某些碱基发生错配对ΔCt值影响较小,甚至毫无影响,说明引物碱基发生置换后仍能与模板发生非特异性扩增,间接说明用于设计引物的该段模板特异性较差。引物3’端不同种类、位置、个数碱基错配对RT-qPCR非特异性扩增发生规律的影响截然不同,本实验构建了3’端3 个位置上不同碱基错配类型与ΔCt之间预测模型,根据此模型在一定程度上可以预测出不同位置、不同错配类型条件下ΔCt值,提出了一种评价RT-qPCR非特异性扩增发生难以程度的方法,从而可以描述非特异性扩增的发生规律。设计引物时,掌握引物3’端碱基错配对RT-qPCR非特异性扩增的发生规律,避免选择DNA序列中易于发生非特异性扩增的碱基位置、碱基种类,确保用于设计引物的特异序列具有真实的特异性,对于提高特异性引物设计效率,降低基于RT-qPCR的动物源性成分检测方法的开发成本具有重要的实用性价值。
除引物3’端核苷酸序列外,DNA聚合酶保真性、探针序列、试剂体系、扩增条件等因素都会对非特异性扩增的产生有影响,因此将会进行持续系统的研究,建立完整的评价体系,为该领域的引物设计提供参考。根据碱基错配反应ΔCt值评价RT-qPCR非特异性扩增的发生规律,可用于指导特异性引物的设计,为肉制品真伪鉴别方法的建立提供技术支持,有利于维护肉制品安全,保障消费者、肉制品加工企业的合法权益。
参考文献:
[1] UMMI K H, MOHD N M, AMIN I, et al. A higher sensitivity and ef ciency of common primer multiplex PCR assay in identi cation of meat origin using NADH dehydrogenase subunit 4 gene[J]. Journal of Food Science and Technology, 2015, 2(7): 4166-4175. DOI:10.1007/ s13197.014.1459.7.
[2] MD E A, MD A R, SHARIFAH B A H. Multiplex PCR in species authentication: probability and prospects: a review[J]. Food Analytical Method, 2014, 7: 1933-1949. DOI:10.1007/s12161.014.9844.4.
[3] SÓNIA S, JOANA S A, ISABEL M, et al. Quantitative detection of poultry meat adulteration with pork by a duplex PCR assay[J]. Meat Science, 2010, 85: 531-536. DOI:10.1016/j.meatsci.2010.03.001.
[4] 周彤, 李家鹏, 田寒友, 等. 一种基于实时荧光聚合酶链式反应的肉及肉制品中猪源性成分含量测定[J]. 肉类研究, 2013, 27(12): 11-15. [5] 魏霜, 陈贞, 芦春斌, 等. 多重PCR检测转基因水稻的转基因成分[J].食品科学, 2012, 33(12): 159-162.
[6] 王小花, 李建祥, 王国卿, 等. SYBR Green实时荧光定量PCR检测大豆转基因成分[J]. 食品科学, 2009, 30(8): 171-175.
[7] RODRÍGUEZ A, GORDILLO R, ANDRADE M J, et al. Development of an ef cient real-time PCR assay to quantify enterotoxin-producing staphylococci in meat products[J]. Food Control, 2016, 60: 302-308. DOI:10.1016/j.foodcont.2015.07.040.
[8] FERNÁNDEZ I C, GUARDDON M, BÖHME K, et al. Detection and quantification of spoilage and pathogenic Bacillus cereus, Bacillus subtilis and Bacillus licheniformis by real-time PCR[J]. Food Microbiology, 2011, 28: 605-610. DOI:10.1016/j.fm.2010.10.014.
[9] ALMEIDA C, CERQUEIRA C, AZEVEDO N F, et al. Detection of Salmonella enterica serovar Enteritidis using real time PCR, immunocapture assay, PNA FISH and standard culture methods in different types of food samples[J]. International Journal of Food Microbiology, 2013, 161: 16-22. DOI:10.1016/ j.ijfoodmicro.2012.11.014.
[10] 吴亚君, 王斌, 刘鸣畅, 等. 阿胶中马和驴成分的实时荧光PCR检测[J]. 食品科学, 2014, 35(8): 85-88. DOI:10.7506/spkx1002-6630-201408016.
[11] 陈颖, 吴亚君. 基因检测技术在食品物种鉴定中的应用[J]. 色谱, 2011, 29(7): 594-600. DOI:10.3724/SP.J.1123.2011.00594.
[12] MARIA T B, ALESSANDRA D.Animal species identification in food products: evolution of biomolecular methods[J]. The Veterinary Journal, 2011, 190: 34-38. DOI:10.1016/j.tvjl.2010.09.024.
[13] 李家鹏, 乔晓玲, 田寒友, 等. 食品和饲料中动物源性成分检测技术研究进展[J]. 食品科学, 2011, 32(9): 340-347.
[14] MUHAMMAD S, YASMEEN J. A multiplex-conventional PCR assay for bovine, ovine, caprine andsh species identi cation in feedstuffs: highly sensitive and specific[J]. Food Control, 2015, 50: 190-194. DOI:10.1016/j.foodcont.2014.08.048.
[15] 申志勇. PCR引物特异性评估体系及多重PCR引物设计系统的构建与应用[D]. 北京: 中国人民解放军军事医学科学院, 2009: 4-6.
[16] WU J H, HONG P Y, LIU W T. Quantitative effects of position and type of single mismatch on single base primer extension[J]. Journal of Microbiological Methods, 2009, 77: 267-275.
[17] BRETT M L, SUSAN K D L. The effect of multiple primer-template mismatches on quantitative PCR accuracy and development of a multiprimer set assay for accurate quantification of pcrA gene sequence variants[J]. Journal of Microbiological Methods, 2013, 94: 224-231.
[18] SÜß B, FLEKNA G, WAGNER M, et al. Studying the effect of single mismatches in primer and probe binding regions on ampli cation curves and quanti cation in real-time PCR[J]. Journal of Microbiological Methods, 2009, 76: 316-319. DOI:10.1016/ j.mimet.2008.12.003.
[19] KLUNGTHONG C, CHINNAWIROTPISAN P, HUSSEM K, et al. The impact of primer and probe-template mismatches on the sensitivity of pandemic in uenza A/H1N1/2009 virus detection by real-time RTPCR[J]. Journal of Clinical Virology, 2010, 48: 91-95. DOI:10.1016/ j.jcv.2010.03.012.
[20] SUTHERLAND J P, BAYLISS A J, BRAXTON D S, et al. Predictive modelling of Escherichia coli O157:H7: inclusion of carbon dioxide as a fourth factor in a pre-existing model[J]. International Journal of Food Microbiology, 1997, 37: 113-120.
[21] CHRISTY M W, ROBERT E, BENJAMIN D C. Kinetic characterization of primer mismatches in allele-specific PCR: a quantitative assessment[J]. Biochemical and Biophysical Research Communications, 2002, 299: 715-722.
[22] TORAHIKO T, NOBUYUKI K, MAKOTO H,et al. Base transitions and base transversions seen in mutations among various types of the hepatitis C viral genome[J]. Federation of European Biochemical Societies, 1993, 315(2): 201-203.
[23] 赵辉, 李启寨, 李俊, 等. 相邻碱基组分与产生SNP的转换或颠换在植物基因组中的研究[J]. 中国科学: C辑, 2006, 36(1): 1-8.
[24] AYYADEVARA S, THADEN J J, REIS R J. Discrimination of primer 39-nucleotide mismatch by Taq DNA polymerase during polymerase chain reaction[J]. Analytical Biochemistry, 2000, 284: 11-18. DOI:10.1006/abio.2000.4635.
[25] STADHOUDERS R, PAS S D, ANBER J, et al. The effect of primertemplate mismatches on the detection and quantification of nucleic acids using the 5’nuclease assay[J]. Journal of Molecular Diagnostics, 2010, 12(1): 109-117. DOI:10.2353/jmoldx.2010.090035.
[26] IWOBI A, SEBAH D, KRAEMER I, et al. A multiplex real-time PCR method for the quantification of beef and pork fractions in minced meat[J]. Food Chemistry, 2015, 169: 305-313. DOI:10.1016/ j.foodchem.2014.07.139.
[27] SENTANDREU M A, SENTANDREU E. Authenticity of meat products: tools against fraud[J]. Food Research International, 2014, 60: 19-29. DOI:10.1016/j.foodres.2014.03.030.
[28] SOARES S, AMARAL J S, OLIVEIRA M B P P, et al. A SYBR green real-time PCR assay to detect and quantify pork meat in processed poultry meat products[J]. Meat Science, 2013, 94: 115-120. DOI:10.1016/j.meatsci.2012.12.012.
[29] DRUMMOND M G, BRASIL B S A F, DALSECCOA L S, et al. A versatile real-time PCR method to quantify bovine contamination in buffalo products[J]. Food Control, 2013, 29: 131-137. DOI:10.1016/ j.foodcont.2012.05.051.
[30] BALLIN N Z. Authentication of meat and meat products[J]. Meat Science, 2010, 86: 577-587. DOI:10.1016/j.meatsci.2010.06.001.
Pattern of Occurrence of Nonspecific Amplification in Real-Time Fluorescent PCR with Different Base Pair Mismatches at the Primer 3’ End
LI Jinchun, LI Jiapeng*, ZHOU Tong, QIAO Xiaoling*, XU Suigen, QI Biao, MI Ruifang, QU Chao, XU Dian
(Beijing Key Laboratory of Meat Processing Technology, China Meat Research Center, Beijing 100068, China)
Abstract:Primer specificity is one of the key figures of merit for the identification of meat adulteration by polymerase chain reaction (PCR). In this study, the pattern of occurrence of non-specific amplification in real-time quantitative polymerase chain reaction (RT-qPCR) was evaluated in terms of the Δ Ct value between base pair matches and mismatches. Three independent variables including the type, position and number of base pair mismatches were investigated for building a quadratic polynomia l to predict the Δ Ct value. Results showed that the type, location, and number of base pair mismatches had a significant impact on non-specific RT-qPCR amplification in a very regular pattern. As far as different types of base pair mismatches were concerned, nonspecific amplification between purine and pyrimidine, but not between different purines or between different pyrimidines, occurred easily, with a significant difference being observed between two types of base pair mismatches. For base position, the first base of the 3’ end of the primer had the greatest impact on the Δ Ct value; the mismatch position gradually far away from the 3’ end had a decreased effect on the Ct value, making nonspecific amplification occur more easily. The greater the number of base pair mismatches was, the more significant the influence on the Δ Ct value was, leading to reduced occurrence of nonspecific amplification. The prediction model for Δ Ct was fitted using a stepwise method with correlation coefficient of more than 0.80, which could provide a useful and accurate method to predict the ΔCt value within certain limits.
Key words:meat adulteration; cycle threshold value; base pair mismatch; nonspecific amplification; primer specificity
DOI:10.7506/spkx1002-6630-201710045
中图分类号:TS207.3
文献标志码:A
文章编号:1002-6630(2017)10-0277-07
收稿日期:2016-11-01
基金项目:国家自然科学基金面上项目(31271877);“十三五”国家重点研发计划重点专项(2016YFD0401203)
作者简介:李金春(1987—),男,工程师,硕士,研究方向为食品生物技术。E-mail:lijinchun1987@163.com
*通信作者:李家鹏(1979—),男,高级工程师,硕士,研究方向为食品生物技术。E-mail:ljp7915@126.com
乔晓玲(1964—),女,教授级高级工程师,本科,研究方向为肉品科学和加工技术。E-mail:cmrcsen@126.com
引文格式:
李金春, 李家鹏, 周彤, 等. 引物3’端不同碱基错配情况下实时荧光定量PCR非特异性扩增的发生规律[J]. 食品科学, 2017, 38(10): 277-283. DOI:10.7506/spkx1002-6630-201710045. http://www.spkx.net.cn
LI Jinchun, LI Jiapeng, ZHOU Tong, et al. Pattern of occurrence of nonspecific amplification in real-time fluorescent PCR with different base pair mismatches at the primer 3’ end[J]. Food Science, 2017, 38(10): 277-283. (in Chinese with English abstract)
DOI:10.7506/spkx1002-6630-201710045. http://www.spkx.net.c n