表1 基于ALA序列合成的多肽
Table 1 A series of peptides synthesized based on the sequence ofα-lactalbumin

丛艳君1,陈 澍1,李 晔1,于晓凤1,李林峰2
(1.北京工商大学食品学院,食品添加剂与配料北京高校工程研究中心,北京 100048;2.北京友谊医院皮肤科,北京 100050)
摘要:α-乳白蛋白是引起牛乳过敏的主要过敏原之一。识别α-乳白蛋白作用表位及影响致敏性的关键氨基酸,对于揭示α-乳白蛋白致敏机理及低致敏乳制品的开发具有重要的意义。本研究采用固相合成技术合成α-乳白蛋白系列多肽,以牛乳过敏患者血清为探针,通过酶联免疫吸附分析法识别α-乳白蛋白的作用表位和关键氨基酸。结果表明:免疫球蛋白(immunoglobulins,Ig)E作用表位的氨基酸序列定位为aa1-15、aa6-20、aa46-60、aa71-85和aa101-115。α-乳白蛋白IgE作用表位关键氨基酸为第8位的缬氨酸、第9位的苯丙氨酸、第10位的精氨酸、第103位的酪氨酸、第105位的亮氨酸和第107位组氨酸。本研究可以为过敏原cDNA克隆以激活T细胞、降低IgE结合能力提供重要思路。
关键词:牛乳过敏;α-乳白蛋白;作用表位;关键氨基酸
牛乳过敏(cow milk allergy,CMA)是一种常见的疾病,它通常是暂时性的,2 岁以下儿童的发生率为2.5%[1]。大多数婴幼儿在3~4 岁时开始对牛乳产生耐受,不再受过敏症困扰,但是,仍然有15%的由免疫球蛋白(immunoglobulins,Ig)E介导的患有CMA症的婴幼儿持续这种疾患[2]。
牛乳中主要含有酪蛋白和乳清蛋白,分别占总蛋白含量的80%和20%。其中,最主要的过敏原为αs1-、αs2-、β-、κ-酪蛋白、α-乳白蛋白(α-lactalbumin,ALA)和β-乳球蛋白(β-lactoglobulin,BLG)。ALA是一种球蛋白,由123 个氨基酸残基构成,相对分子质量约为14 200,有4 个二硫键[3]。同源性研究表明牛乳与人乳中的ALA同源性为74%,另有6%的氨基酸残基化学性质相似。ALA与钙有较高的亲和力,使之二级结构相当稳定。根据ALA亲水区域推测其主要的抗原结合位点位于肽链第5~18位的氨基酸片段,与BLG分子中氨基酸序列124~134具有同源序列[4]。
表位是抗原中能够被B细胞和T细胞识别的氨基酸序列,分为IgE作用表位和IgG作用表位。作用表位的识别对于开发特异性更强的CMA诊断方法具有重要的理论意义。本研究中,将能被65%以上过敏患者血清识别的多肽定义为作用表位[5-6]。
对其他食物过敏原(比如花生过敏原Ara h1、Ara h2和Ara h3)的基因突变分析报告表明,用丙氨酸依次替换每个IgE结合表位的单个的氨基酸可以显著降低花生过敏患者血清IgE抗体与花生过敏原结合的能力[7-9]。本课题组之前的研究找到了影响BLG和αs1-酪蛋白致敏性的IgE和IgG作用表位的关键氨基酸残基[10-11]。
本研究以婴幼儿CMA患者血清为探针识别ALA的IgE作用表位,在此基础上,通过丙氨酸扫描方法即用丙氨酸依次取代作用表位的氨基酸合成新的多肽,然后用酶联免疫吸附分析(enzyme linked immunosorbent assay,ELISA)法识别降低ALA致敏性的关键氨基酸。
1.1 材料与试剂
链霉亲和素 北京博奥森生物技术有限公司;
ALA、辣根过氧化物酶(horseradish peroxidase,HRP)标记的鼠抗人IgE(ε链特异性)、4-氯-1-萘酚、三氟乙酸美国Sigma公司;96 孔聚苯乙烯酶标板 美国Costar公司;ALA多肽由北京食品添加剂与配料重点实验室合成。
1.2 仪器与设备
550型酶标仪 美国Bio-Rad公司;PSH500A生化培养箱 中国重庆银河实验仪器有限公司;Th-80D-2B型冻干机 北京天地精仪科技有限公司;高效液相C18柱美国Vydac公司;PSI200多通道多肽合成仪 美国多肽科技有限公司 。
1.3 方法
1.3.1 CMA患者血清的收集
ALA CMA患者血清及阴性血清由北京友谊医院提供。8 例CMA患者(6 个月~3 岁,平均年龄为1 岁)血清用来鉴别IgE表位和这些表位上的关键氨基酸。通过ALA试剂盒、免疫印迹实验测定8 例血清ALA特异性IgE抗体含量为25~100 kU/L。8 例CMA患者的临床症状表现为特异性皮炎。5 位非CMA症幼儿(1~3 岁)的血清用作阴性对照。
1.3.2 ALA多肽的合成、纯化及鉴定
以ALA氨基酸序列为模板,用9-芴甲氧羰基(fiuorenylmethoxy carbony,Fmoc)固相合成法错位合成系列ALA多肽(长度为15 个氨基酸)[12]。
合成的多肽通过C18反向高效液相色谱纯化,以0.1%三氟乙酸-水和0.1%乙腈-水作为流动相进行梯度洗脱,流速为12.0 mL/min,上样量为50 mg/mL的合成肽。通过质谱测定多肽分子质量鉴定合成多肽的准确性,质谱条件为:ESI离子源;喷雾压力:15 psi;干燥气温度:350 ℃;流速:5 L/min;扫描质量范围:m/z 500~2 200。
1.3.3 IgE作用表位和关键氨基酸的识别
参考Cocco等[13]的研究方法,通过直接ELISA识别IgE作用表位和关键氨基酸。首先将连接链霉亲和素的ALA多肽加入到96 孔酶标板中4 ℃包被12 h。洗涤后,用含1%牛血清白蛋白的Tris缓冲溶液封闭,之后加入个体血清用以识别IgE作用表位或者加入混合血清用于识别关键氨基酸,个体血清、混合血清均用含1%牛血清白蛋白的10 mmol/L Tris-HCl(pH 7.5)缓冲溶液以1∶10稀释。以HRP标记的单克隆鼠抗人IgE为二抗,用含1%牛血清白蛋白的10 mmol/L Tris-HCl(pH 7.5)缓冲溶液以1∶2 000稀释。4-氯-1-萘酚为显色液,450 nm波长处检测其OD值。以非CMA患者血清作为对照。
将被65%以上CMA患者个体血清IgE识别的多肽定义为作用表位。将被丙氨酸取代而导致失去致敏性的氨基酸称为关键氨基酸。
1.4 数据分析
统计分析采用微软Excel 2010软件进行。所有实验均做3 次平行。数据间的差异水平使用ANOVA进行分析,以Kruskal-Wallis检验置信水平,95%作为显著水平。ALA的三维结构图用Pymol软件制作。
2.1 多肽合成
表1 基于ALA序列合成的多肽
Table 1 A series of peptides synthesized based on the sequence ofα-lactalbumin

由表1可知,合成了23 条多肽,属于ALA大部分氨基酸序列区域,这些多肽的纯度均在85%以上,用于识别ALA IgE作用表位。
2.2 IgE作用表位的识别
表2 ALA IgE作用表位
Table 2 IgE-binding epitopes ofα-lactalbumin

由表2可知,用8 位CMA患者血清识别多肽,其中编号为P1的多肽(aa1-15)和编号为P21的多肽(aa101-115)的识别率为100%(8/8,即与8 份血清均发生特异性反应)。P2(aa6-20)和P10(aa46-60)的识别率为87.5%(7/8)。P15(aa71-85)的识别率为75%(6/8)。P11、P14、P20和P22的识别率为37.5%(3/8)。多肽P3、P4、P6、P7、P8、P9、P12、P13、P16、P17、P18、P19和P23与血清也发生了免疫反应,多肽P5未与血清发生免疫反应。识别率在65%以上的多肽为作用表位。表2中以灰度深浅表示免疫反应程度强弱。因此IgE的作用表位在ALA氨基酸序列中的定位为aa1-15、aa6-20、aa46-60、aa71-85和aa101-115,在ALA三维结构中的位置见图1。非CMA患者血清识别多肽表现为弱反应(本实验未附阴性对照的具体数据)。
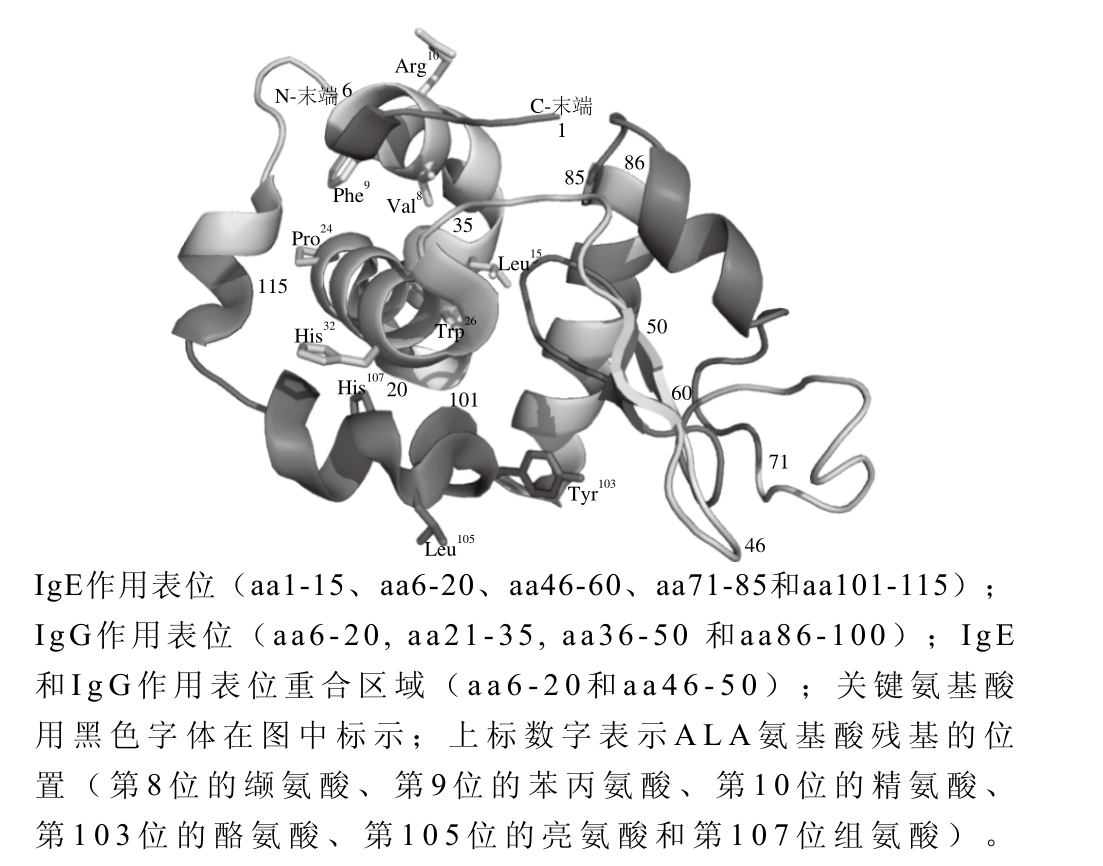
图1 ALA的三维结构图
Fig. 1 3D structure of α-lactalbumin
2.3 ALA IgE作用表位(aa1-15和aa101-115)关键氨基酸的识别
用丙氨酸依次取代aa1-15肽段上的氨基酸合成多肽,以8 名CMA患者的混合血清为抗体通过ELISA识别新合成的多肽,以aa1-15为对照,450 nm波长处测定OD值(图2),第8位的缬氨酸,第9位的苯丙氨酸,第10位的精氨酸分别被丙氨酸取代后合成的多肽(V8A、F9A、R10A)OD值为0,多肽致敏性消失。其他绝大多数氨基酸被丙氨酸取代后的多肽OD值与对照(aa1-15)差异不显著。
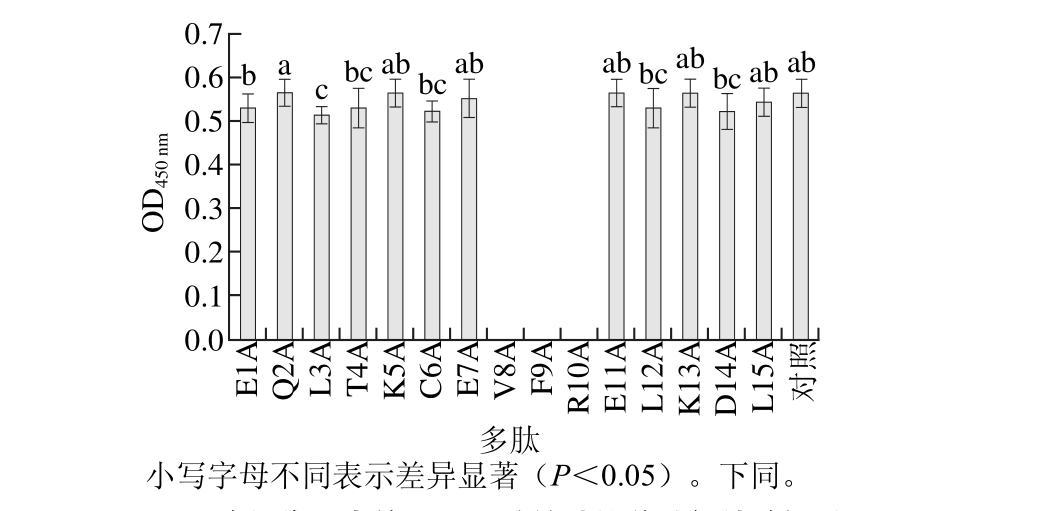
图2 降低作用表位aa1-15致敏性的关键氨基酸识别
Fig. 2 Single amino acid change in the peptide at aa1-15 resulted in the loss of IgE binding to this epitope
同样方法确定aa101-115肽段上关键氨基酸,结果见图3。第103位的酪氨酸,第105位的亮氨酸和第107位的组氨酸分别被丙氨酸取代后合成的多肽(Y103A、L105A、H107A)OD值为0,多肽致敏性消失。其他绝大多数氨基酸被丙氨酸取代后的多肽OD值与对照组(aa101-115)差异不显著。
基于以上实验,得出第8位的缬氨酸、第9位的苯丙氨酸、第10位的精氨酸、第103位的酪氨酸、第105位的亮氨酸和第107位的组氨酸是影响ALA致敏性的关键氨基酸,这些氨基酸残基的空间位置如图1所示。
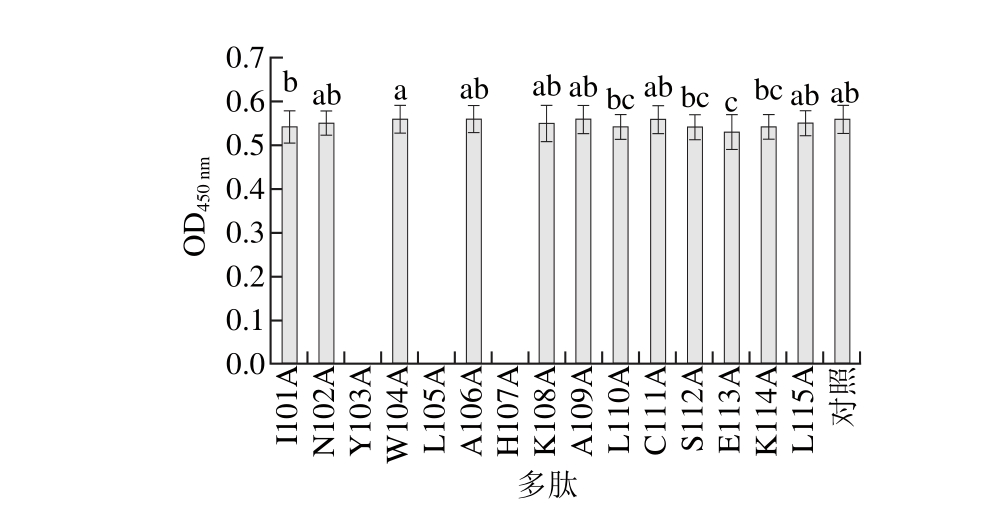
图3 降低作用表位aa101-115致敏性的关键氨基酸的识别
Fig. 3 Single amino acid change in the peptides at aa101-115 resulted in reduced IgE binding to this epitope
国外对牛乳过敏原作用表位识别的研究开展比较早,Järvinen等[14]收集了持续性CMA患者血清和暂时性CMA患者血清识别ALA作用表位,用持续性CMA患者识别到4 个IgE作用表位和3 个IgG作用表位,而暂时性CMA患者血清没有识别到作用表位。Hochwallner等[15]重组的ra-乳白蛋白能够被57.6%的CMA病人识别,可以用于诊断严重的CMA患者。同时对αs1-酪蛋白和BLG开展的研究也得出类似结论,即不同临床症状病人血清识别的作用表位不同[6,16-21],这为临床CMA的诊治提供了重要思路。本课题组在前期用对CMA的临床症状为湿疹的患者血清识别αs1-酪蛋白IgE和IgG作用表位,发现具有同一症状病人血清识别的IgE作用表位相同,而IgE和IgG作用表位不同,进而对BLG作用表位也行了研究,同样验证了上述结论[10-11]。
本研究目的在于建立ALA序列中抗原表位模型,探究降低过敏原致敏性的关键氨基酸,为ALA序列表位的识别模式与特异性皮炎临床症状之间的关系提供理论依据,并针对ALA的线性表位,对ALA构象表位的鉴定是该研究未来要做的工作。
尽管Adams等[22]以ALA的胰蛋白酶的裂解物为研究对象,而本研究基于ALA的序列合成不同的多肽,但是识别到了同一个作用表位aa1-15,另外,本研究还发现了IgE其他结合表位在aa101-115、aa6-20、aa46-60和aa71-85,识别到不同作用表位的原因可能与过敏患者遗传背景及血清学不同有关。那些没有被病人血清识别的多肽,推测其可能位于ALA核心部位的疏水区域。
Adams等[22]通过RAST实验证明了aa5-18肽段表现出较强的IgE结合能力,用胰蛋白酶消化ALA的另一项研究发现,aa17-58多肽序列更加容易被IgE识别。此外,多肽aa59-94、aa6-10、aa115-123和aa109-123与牛乳患者血清IgE较强的结合能力也被报道过[23]。基于以上结果,并结合本研究的实验结果,代表性的研究了aa1-15,aa101-115作用表位的关键氨基酸。
由于丙氨酸为分子质量相对较小的中性氨基酸,并且不会显著改变肽的电荷和溶解度,所以我们选用丙氨基作为取代氨基酸,这种方法已成功地用于其他过敏原关键氨基酸的分析[24-25]。IgE作用表位关键氨基酸的识别,可以为ALA的基因编码、降低过敏原的IgE结合能力提供重要的信息。本研究结果表明,修饰IgE结合表位aa1-15肽段上的第8位的缬氨酸、第9位的苯丙氨酸、第10位的精氨酸可以降低ALA的致敏性。另外,第103位的酪氨酸、第105位的亮氨酸和第107位的组氨酸也是影响作用表位aa101-115致敏性的关键氨基酸。并且,IgE结合表位主要由疏水性氨基酸构成,且至少包含一个赖氨酸[28-29],本研究中也得出了类似的结论。
通过“丙氨酸扫描技术”识别了降低ALA过敏原致敏性的关键氨基酸。第8位的缬氨酸,第9位的苯丙氨酸,第10位的精氨酸,第103位的酪氨酸,第105位的亮氨酸和第107位的组氨酸是IgE结合表位的关键氨基酸。本研究结论为蛋白质氨基酸定点修饰、基因工程技术重组过敏原蛋白以降低致敏性提供了重要的信息。此外,那些没有被IgE识别的氨基酸,也可以为过敏原的基因沉默研究提供思路。
参考文献:
[1] OSTERBALLE M, HANSEN T K, MORTZ C G, et al. The prevalence of food hypersensitivity in an unselected population of children and adults[J]. Pediatric Allergy and Immunology, 2005, 16(7): 567-573. DOI:10.1111/j.1399-3038.2005.00251.x.
[2] SAMPSON H A. Food allergy[J]. The Journal of Allergy and Clinical Immunology, 2003, 111(Suppl 2): 540-547. DOI:10.1067/mai.2003.134.
[3] WAL J M. Cow’s milk allergens[J]. Allergy, 1998, 53(11): 1013-1022. DOI:10.1111/j.1398-9995.1998.tb03811.x.
[4] CHEN F M, LEE J H, yANG y H, et al. Analysis of α-lactalbumin-, β-lactoglobulin-, and casein-specif i c IgE among children with atopic diseases in a tertiary medical center in northern Taiwan[J]. Journal of Microbiology, Immunology, and Infection, 2014, 47(2): 130-136. DOI:10.1016/j.jmii.2012.08.009.
[5] CERECEDO I, ZAMORA J, SHREFFLER W, et al. Mapping of the IgG and IgE sequential epitopes of milk allergens using a peptide microarray-based immunoassay[J]. The Journal of Allergy and Clinical Immunology, 2008, 122(3): 589-594. DOI:10.1016/j.jaci.2007.12.988.
[6] COCCO R R, JÄRVINEN K M, HAN N, et al. Mutational analysis of immunoglobulin E-binding epitopes of β-casein and β-lactoglobulin showed a heterogeneous pattern of critical amino acids between individual patients and pooled sera[J]. Clinical and Experimental Allergy, 2007, 37(6): 831-838. DOI:10.1111/j.1365-2222.2007.02712.x.
[7] BURKS A W, SHIN D, COCKRELL G, et al. Mapping and mutational analysis of the IgE-binding epitopes on Ara h 1, a legume vicilin protein and a major allergen in peanut hypersensitivity[J]. European Journal of Biochemistry, 1997, 245(2): 334-339. DOI:10.1111/j.1432-1033.1997.t01-1-00334.x.
[8] RABJOHN P, HELM E M, STANLEy J S, et al. Molecular cloning and epitope analysis of the peanut allergen Ara h 3[J]. Journal of Clinical Investigation, 1999, 103(4): 535-542. DOI:10.1172/JCI5349.
[9] STANLEy J S, KING N, BURKS A W, et al. Identification and mutational analysis of the immunodominant IgE binding epitopes of the major peanut allergen Ara h 2[J]. Archives of Biochemistry and Biophysics, 1997, 342(2): 244-253. DOI:10.1006/abbi.1997.9998.
[10] CONG y J, ZHOU S y, LI L F. Identification of the critical amino acid residues of immunoglobulin E and immunoglobulin G epitopes in β-lactoglobulin by alanine scanning analysis[J]. Journal of Dairy Science, 2012, 95(11): 6307-6312. DOI:10.1111/1750-3841.13425.
[11] CONG y J, yI H, QING y T, et al. Identif i cation of the critical amino acid residues of immunoglobulin E and immunoglobulin G epitopes on αs1-casein by alanine scanning analysis[J]. Journal of Dairy Science, 2013, 96(11): 6870-6876. DOI:10.3168/jds.2013-6880.
[12] RUITER B, TRÉGOAT V, M’RABET L, et al. Characterization of T cell epitopes in alphas1-casein in cow’s milk allergic, atopic and nonatopic children[J]. Clinical & Experimental Allergy, 2006, 36(3): 303-310. DOI:10.1111/j.1365-2222.2006.02436.x.
[13] COCCO R R, JÄRVINEN K M, SAMPSON H A, et al. Mutational analysis of major, sequential IgE-binding epitopes in alpha s1-casein, a major cow’s milk allergen[J]. The Journal of Allergy and Clinical Immunology, 2003, 112(2): 433-437. DOI:10.1067/mai.2003.1617.
[14] JÄRVINEN K M, CHATCHATEE P, BARDINA L, et al. IgE and IgG binding epitopes on alpha-lactalbumin and beta-lactoglobulin in cow’s milk allergy[J]. International Archives of Allergy and Immunology, 2001, 126(2): 111-118. DOI:10.1159/000049501.
[15] HOCHWALLNER H, SCHULMEISTER U, SWOBODA I, et al. Visualization of clustered IgE epitopes on alpha-lactalbumin[J]. The Journal of Allergy and Clinical Immunology, 2010, 125(6): 1279-1285. DOI:10.1016/j.jaci.2010.03.007.
[16] SÉLO I, NÉGRONI L, CRÉMINON C, et al. Allergy to bovine α-lactoglobulin: specificity of human IgE using cyanogen bromidederived peptides[J]. International Archives of Allergy and Immunology, 1998, 117(1): 20-28. DOI:10.1159/000023986.
[17] FRITSCHÉ R, ADEL-PATIENT K, BERNARD H, et al. IgE-mediated rat mast cell triggering with tryptic and synthetic peptides of bovine beta-lactoglobulin[J]. International Archives of Allergy and Immunology, 2006, 138(4): 291-297. DOI:10.1159/000088866.
[18] WILLIAMS S C, BADLEy R A, DAVIS P J, et al. Identif i cation of epitopes within beta-lactoglobulin recognized by polyclonal antibodies using phage display and PEPSCAN[J]. Journal of Immunological Methods, 1998, 213(1): 1-17. DOI:10.1016/S0022-1759(98)00022-2.
[19] CHATCHATEE P, JÄRVINEN K M, BARDINA L, et al. Identif i cation of IgE- and IgG-binding epitopes on alpha(s1)-casein: differences in patients with persistent and transient cow’s milk allergy[J]. The Journal of Allergy and Clinical Immunology, 2001, 107(2): 379-383. DOI:10.1067/mai.2001.112372.
[20] ELSAyED S, HILL D J, DO T V. Evaluation of the allergenicity and antigenicity of bovine-milk αs1-casein using extensively purif i ed synthetic peptides[J]. Scandinavian Journal of Immunology, 2004, 60(5): 486-493. DOI:10.1111/j.0300-9475.2004.01493.x.
[21] RUITER B, TRÉGOAT V, M’RABET L, et al. Characterization of T cell epitopes in alphas1-casein in cow’s milk allergic, atopic and nonatopic children[J]. Clinical & Experimental Allergy, 2006, 36(3): 303-310. DOI:10.1111/j.1365-2222.2006.02436.x.
[22] ADAMS S L, BARNETT D, WALSH B J, et al. Human IgE-binding synthetic peptides of bovine beta-lactoglobulin and alpha-lactalbumin. In vitro cross-reactivity of the allergens[J]. Immunology & Cell Biology, 1991, 69: 191-197. DOI:10.1038/icb.1991.28.
[23] MAyNARD F, JOST R, WAL J M. Human IgE binding capacity of tryptic peptides from bovine α-lactalbumin[J]. International Archives of Allergy and Immunology, 1997, 113(4): 478-488. DOI:10.1159/000237625.
[24] ELSAyED S, HILL D J, DO T V. Evaluation of the allergenicity and antigenicity of bovine-milk αs1-casein using extensively purif i ed synthetic peptides[J]. Scandinavian Journal of Immunology, 2004, 60(5): 486-493. DOI:10.1111/j.0300-9475.2004.01493.x.
[25] ROBOTHAM J M, TEUBER S S, SATHE S K, et al. Linear IgE epitope mapping of the English walnut (Juglans regia) major food allergen, Jug r 1[J]. The Journal of Allergy and Clinical Immunology, 2002, 109(1): 143-149. DOI:10.1067/mai.2002.120558.
[26] BØGH K L, NIELSEN H, MADSEN B C, et al. IgE epitopes of intact and digested Ara h1: A comparative study in human and rats[J]. Molecular Immunology, 2012, 51(3/4): 337-346. DOI:10.1016/ j.molimm.2012.04.002.
[27] MINE y, WEI Z J. Identif i cation and fi ne mapping of IgG and IgE epitopes in ovomucoid[J]. Biochemical & Biophysical Research Communications, 2002, 292(4): 1070-1074. DOI:10.1006/bbrc.2002.6725.
[28] TSAI L C, CHAO P L, HUNG M W, et al. Protein sequence analysis and mapping of IgE and IgG epitopes of an allergenic 98-kDa Dermatophagoides farinae, paramyosin, Der f 11[J]. Allergy, 2000, 55(2): 141-147. DOI:10.1034/j.1398-9995.2000.00315.x.
[29] CHAPMAN M D, SMITH A M, VAILES L D, et al. Recombinant allergens for diagnosis and therapy of allergic disease[J]. The Journal of Allergy and Clinical Immunology, 2000, 106(3): 409-418. DOI:10.1067/mai.2000.109832.
Identification of the Critical Amino Acids of IgE-Binding Epitopes in α-Lactalbumin
CONG Yanjun1, CHEN Shu1, LI Ye1, YU Xiaofeng1, LI Linfeng2
(1. Beijing Higher Institution Engineering Research Center of Food Additives and Ingredients, College of Food Science, Beijing Technology and Business University, Beijing 100048, China; 2. Department of Dermatology, Beijing Friendship Hospital, Beijing 100050, China)
Abstract:α-Lactalbumin represents one of the major allergens causing cow milk allergy. The identification of the epitopes of α-lactalbumin and the critical amino acids for its allergenicity is of great significance for understanding the mechanism of action of α-lactalbumin and developing hypoallergenic dairy products. In this study, a series of peptides were synthesized by a solid phase method for the characterization of immunological epitopes and critical amino acids. The immunoglobulin (Ig) E-binding epitopes were immunolabeled with individual sera from cow milk-allergic patients as probes by enzyme linked immunosorbent assay (ELISA). Alanine scanning of immunodominant epitopes was used to identify the critical amino acids (aa). The results showed that IgE-binding epitopes were located within the sequences of aa1-15, aa6-20, aa46-60, aa71-85 and aa101-115. Our initial data revealed that Val8, Phe9, Arg10, Tyr103, Leu105and His107were the critical amino acids for IgE-binding epitopes. This study will provide the necessary information to alter the cDNA to encode a protein capable of activating milk-specific T cells, but with reduced IgE-binding capacity.
Key words:cow milk allergy; α-lactalbumin; epitope; critical amino acid
DOI:10.7506/spkx1002-6630-201711001
中图分类号:TS252.1
文献标志码:A
文章编号:1002-6630(2017)11-0001-05
引文格式:
丛艳君, 陈澍, 李晔, 等. 牛乳α-乳白蛋白IgE线性表位的关键氨基酸识别[J]. 食品科学, 2017, 38(11): 1-5. DOI:10.7506/ spkx1002-6630-201711001. http://www.spkx.net.cn
CONG Yanjun, CHEN Shu, LI Ye, et al. Identification of the critical amino acids of ige-binding epitopes in α-lactalbumin[J]. Food Science, 2017, 38(11): 1-5. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201711001. http://www.spkx.net.cn
收稿日期:2016-10-08
基金项目:国家自然科学基金青年科学基金项目(31101236);北京市自然科学基金面上项目(6132004);北京市科技新星计划项目(Z131102000413005)
作者简介:丛艳君(1978—),女,副教授,博士,研究方向为乳品科学。E-mail:cyj_win@sina.com