
图1 酸汤子面团细菌扩增产物
Fig.1 Agarose gel electrophoresis of PCR amplified products from bacterial DNA from Suantangzi sourdough
乌日娜1,2,张 颖1,张红萧1,陶冬冰1,孙慧君1,3,岳媛媛1,武俊瑞1,*
(1.沈阳农业大学食品学院,辽宁 沈阳 110866;2.食品科学与技术国家重点实验室,江南大学食品学院,江苏 无锡 214122;3.辽宁省农业经济学校现代农业技术系,辽宁 锦州 121001)
摘 要:酸汤子是我国北方地区一种营养丰富、风味独特的民族传统食品,深受满族及东北人民的喜爱。多样的微生物在酸汤子玉米面团的营养品质形成过程中,发挥着极其重要的作用,然而到目前为止,对满族传统发酵食品酸汤子面团中的微生物菌群多样性,仍不明确。以不同地区采集的酸汤子玉米面团为研究对象,利用聚合酶链式反应结合变性梯度凝胶电泳技术探究了酸汤子中微生物菌群多样性。结果表明:在9 份酸汤子面团中共鉴定出14 种真菌,分别为Saccharomyces castellii、Geotrichun candidum、Simplicillium lanosoniveum、Rhizochaete sulphurosa、Guehomyces pullulans、Debaryomyces hansenii、Fusarium culmorum、Trichoderma brevicompactum、Oryza lonqistaminata、Geotrichun fraqrans、Galactomyces candidum、G. geotrichum、Geotrichum sp.和Galactomyces sp.。鉴于S. castellii在多数样品中被检测,推测其为酸汤样品中真菌的优势发酵菌种。鉴定出4 种细菌,分别是Bacillus pumilu、Lactobacillus tucceti、L. plantarum和Weissella paramesenteroides。由于W. paramesenteroides在多数样品中被检测出,推测其为酸汤样品细菌的优势菌群。
关键词:酸汤子;自然发酵;PCR-DGGE;微生物;多样性
酸汤子,又称汤子、馇子,是用玉米水磨发酵而成的一种粗面条样主食,具体做法是把新鲜嫩玉米脱粒放入盛有冷水的缸中浸泡,待米质松软后,磨成糊状,在适宜的温度进行自然发酵,等面飘出酸味时,烹调熟制后做成汤面或肉酱面即可食用[1]。酸汤子味道微酸,通脾健胃、消乏解渴,易消化又开胃,是一种老少皆宜的食品[2],但是其认知度也只局限于东北地区和少数人群。如今,城市里已经很难找到“酸汤子”了,很多身在外地的人们只有每当秋季回老家时,才能尝上一碗日思夜想的酸汤子。因此,将这种营养价值丰富且迎合大众口味的满族特色美食推广开来,丰富广大人民日常餐谱,推进产业化发展有着重要意义。
国内外对小麦、大麦和藜麦面粉发酵面团中的微生物研究较多,而对于玉米面发酵面团中微生物研究很少。对于小麦面粉发酵面团中微生物的研究,主要集中于面团中微生物之间的相互作用以及采样、分离和鉴定过程等一些外在因素,对不同地域生产的面粉所制得的酸面团中微生物组成的影响[3]。Harth等[4]发现不同处理的大麦粉发酵形成的酸面团在微生物群落组成方面存在着明显的差异;Rodríguez等[5]利用随机扩增多态性DNA标记(random amplification polymorphic DNA,RAPD)-聚合酶链反应(polymerase chain reaction,PCR)和PCR-变性梯度凝胶电泳(denatured gradient gel electrophoresis,DGGE)结合rRNA基因测序分析藜麦发酵面团中乳酸菌多样性,共鉴定出4 个菌属和9 个菌种。
PCR-DGGE技术不仅可以检测到自然界中无法培养或难以培养的微生物,且检测速率快,结果准确可靠,能够更好的反映酸汤子面团内细菌多样性的真实情况[6-7]。Muyzer等[8]证明DGGE技术在研究自然界微生物群落多样性及菌相差异方面具有明显的优越性,在发酵类食品以及自然环境中得到了很好的应用[9-13]。Ampe等[14]以墨西哥发酵玉米面团为材料,采用PCR-DGGE技术纯化DNA片段,鉴定乳杆菌;Ercolini等[15]用PCR-DGGE法在天然乳清培养基16S rDNA的V3区中鉴定出德氏乳杆菌和乳球菌等;Biddle等[16]运用宏基因组结合RT-PCR方法对秘鲁海域浅海水层生物圈中微生物群落进行多样性探究,结果表明秘鲁海域浅海水层的优势菌群未培养是Crenarchaeota菌;滕齐辉等[17]运用PCR-限制性内切酶片段长度多态性(restriction fragment length polymorphism, RFLP)结合DGGE技术对太湖地区土壤微生物多样性进行了研究,结果表明有机肥能提高土壤微生物的丰富度和多样性。
本实验主要通过PCR-DGGE分子生物学技术结合测序的方法,对传统自然发酵酸汤子面团中微生物多样性进行研究,为挖掘丰富的微生物资源,进一步实现传统酸汤子产品的现代化、规模化生产提供依据。
1.1 材料与试剂
酸汤样品分别来自7 个不同城市,其中地理位置与其对应的编号样品分别为鞍山1号、本溪2号、沈阳3号、丹东4号、丹东5号、抚顺6号、丹东7号、桓仁8号、通辽9号,共9 份酸汤样品,丹东样品分别随机采取3 户不同人家的酸汤样品。
去离子甲酰胺、四甲基二乙胺、双丙烯酰胺、过硫酸铵、灭菌蒸馏水、三蒸水、10×Easy Taq Buffer、High Pure dNTPs、F338-GC、R518、Easy Taq聚合酶、双蒸水沈阳力新生物技术有限公司。
1.2 仪器与设备
5417高速冷冻离心机 德国Eppendorf公司;ABI Veriti96 PCR扩增仪 美国ABI公司;水浴锅 上海精宏实验设备有限公司;DYCP-31BN型琼脂糖凝胶电泳仪深华生物技术有限公司;紫外凝胶成像及分析系统、DGGE仪 美国Bio-Rad公司。
1.3 方法
1.3.1 样品总DNA提取
从样品中间称取10 g酸汤子面团样品,加入15 mL灭菌磷酸盐缓冲液(phosphate buffered saline,PBS),用玻璃棒搅拌混匀,称取0.5 g混匀样品放入1.5 mL灭菌离心管中,其余实验步骤按照基因组DNA快速抽提试剂盒进行。以上操作均在无菌操作台上进行。
1.3.2 PCR扩增
1.3.2.1 细菌的PCR扩增
以提取的总D N A为模板,细菌16 S r D N A基因V 3区采用通用引物(上下游引物F3 3 8:5’-C C T A C G G G A G G C A G C A G-3’、R 5:5’-ATTACCGCGGCTGCTGG-3’、GC发卡:5’-(GC)nCCTACGGGAGGCAGCAG-3’)进行PCR扩增,扩增产物片段长约250 bp。PCR体系(50 µL):36 µL ddH2O、5 µL 10×Easy Taq Buffer、4 µL High Pure dNTPs、1.5 µL F338-GC、1.5 µL R518、0.5 µL Easy Taq聚合酶及适量的双蒸水补足50 µL。反应参数:95 ℃预变性4 min;95 ℃变性1 min,55 ℃退火45 s,72 ℃延伸1 min,30 个循环;72 ℃延伸7 min,4 ℃保存。PCR产物用质量分数为1%的琼脂糖凝胶电泳(100 V恒压电泳30 min)检测后-20 ℃保存备用。
1.3.2.2 真菌的PCR扩增
以提取的总DNA为模板,真菌采用26S rDNA D 1/D 2区具有特异性的引物(上游引物N L-1:5’-GCATATCAATAAGCGGAGGAAAAG-3’、下游引物NL-4:5’-GGTCCGTGTTTCAAGACGG-3’)进行26S rDNA的PCR扩增,扩增产物片段长约600 bp。反应体系(50 μL):3 μL基因组DNA模板、4 μL dNTP(2.5 mmol/L)、2条引物各1 μL、0.4 μL DNA Taq聚合酶、5 μL 10×PCR Buffer,补充ddH2O至50 µL。反应参数:94 ℃变性1 min,54 ℃退火1 min,72 ℃延伸1 min 30 s,36 个循环,最后72 ℃末端延伸10 min,PCR产物用质量分数为1%的琼脂糖凝胶电泳(100 V恒压电泳30 min)检测后-20 ℃保存备用。
1.3.2.3 DGGE与图谱分析
采用基因突变检测系统对PCR产物进行分析。使用梯度胶制备装置,制备变形质量分数35%~50%(100%变性剂、7 mol/L尿素、40%去离子甲酰胺的混合物)的8%的聚丙烯酰胺凝胶,其中变性剂质量分数从胶板由上到下依次递增。待胶完全凝固后,将胶板放入装有1×TAE电泳缓冲液的装置中,在每个加样孔中加入已和Marker混合好的PCR样品。细菌的变性剂质量分数范围35%~55%,在200 V电压下,60 ℃电泳5 h。真菌的变性剂质量分数范围30%~55%,在120 V电压下,60 ℃电泳5 h15 min。电泳完毕后,利用Gelred染色法将凝胶染色30 min,将染好的凝胶用扫描仪成像,得到PCR-DGGE图谱,对上述扫描后的图片进行分析,标记特异性条带,并回收条带,由上海桑尼生物技术有限公司测序,测序结果进行BLAST比对鉴定。
2.1 酸汤子面团样品的PCR扩增
由图1、2可知,所有样品细菌引物PCR扩增均获得一条约250 bp大小的特异性条带,所有样品真菌引物PCR扩增均获得一条约600 bp大小的特异性条带,它们分别与细菌16S rDNA的V3区和真菌26S rDNA的D1/D2区预期条带大小一致,可用于后续DGGE分析。

图1 酸汤子面团细菌扩增产物
Fig.1 Agarose gel electrophoresis of PCR amplified products from bacterial DNA from Suantangzi sourdough

图2 酸汤子面团真菌扩增产物
Fig.2 Agarose gel electrophoresis of PCR-amplified DNA fragments from fungi in Suantangzi sourdough
2.2 酸汤子面团样品中细菌多样性分析
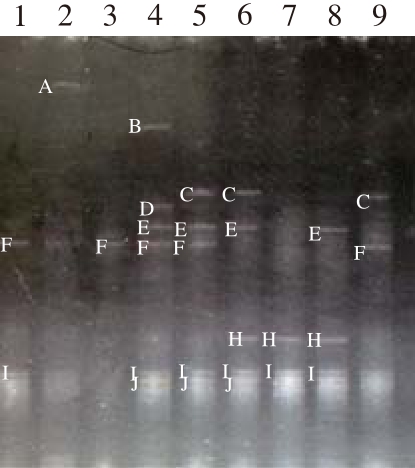
图3 酸汤子面团中细菌总DNA的PCR-DGGE图谱
Fig.3 DGGE profiles of PCR-amplified DNA from the total bacterial population in each sourdough
表1 不同发酵阶段酸汤子面团样品中细菌16S rDNA V3区PCR-DGGE特异性条带比对结果
Table1 Identification of the bands obtained by PCR-DGGE of bacterial populations based on 16S rDNA sequence
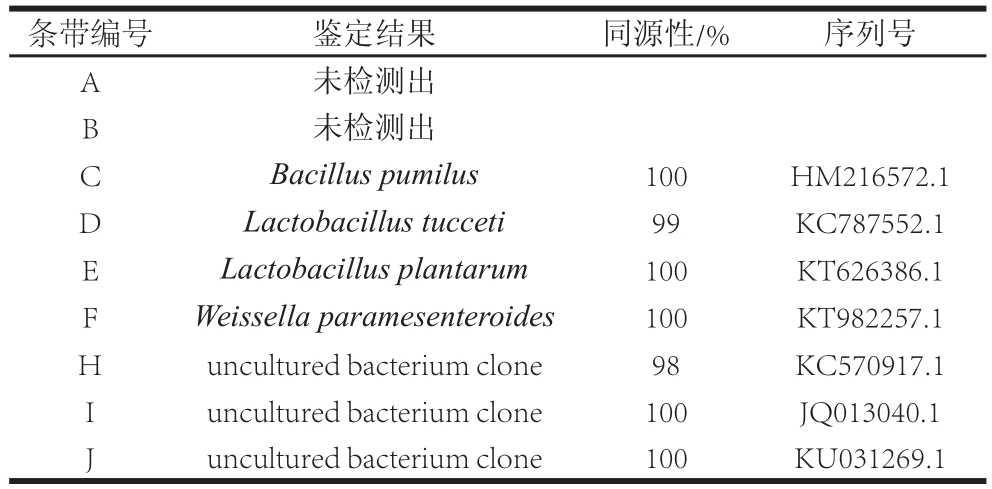
DGGE图谱条带的数目可以直观地反映样品中细菌菌落的遗传多样性[19]。由图3可知,酸汤子9 个样品的细菌PCR-DGGE图谱中共发现9 个特异性扩增条带,得到的条带数量和微生物分布情况均有明显不同,这说明酸汤样品中细菌分布有着较大的差异性。由图谱可以明显看出样品4~6中条带的数目与其他地区样品相比较多,说明丹东与抚顺地区酸汤子样品中细菌的种类最为丰富;样品2、3的条带数目最少,说明本溪与沈阳地区酸汤子样品中细菌数目最少。对9 个特征条带进行回收和测序,测序结果在NCBI数据库中进行BLAST同源性比对,比对结果如表1所示。
结合图3和表1可知,酸汤子9 个样品中共检测到了4 种细菌,它们分别属于3 个属。条带C在样品5、6、9中均检测出,且在样品5中条带较其他样品更亮,条带更宽,说明短小芽孢杆菌(Bacillus pumilus)在丹东地区酸汤子样品中数量最多;条带H、I、J的比对结果均为未培养细菌克隆,在少部分样品中被检测出。条带F、I、E在样品中出现的频率均在占总样品总数的44%以上,其中所占比例最高的是条带I未培养细菌克隆,为66.7%,其次为条带F,其鉴定结果为类肠膜魏斯氏菌(Weissella paramesenteroides),在5 份样品中均被检测出;条带E的比对结果分别为植物乳杆菌(Lactobacillus plantarum),在样品4、5、6、8中均被检测出;条带D(L. tucceti)在样品4中被检测出,为酸汤样品中的特殊菌群。综上,植物乳杆菌与类肠膜魏斯氏菌在酸汤子发酵面团样品中所占比例最多,可推测为酸汤子样品的优势细菌菌群;乳杆菌属为优势菌属。条带A、B均未检测出信号。
此外,由图3中条带分布结合鉴定结果可知,即使同一地区采集的酸汤子样品中的细菌构成也不尽相同。丹东地区3 个不同人家的酸汤样品的细菌菌群分布有着明显的差异性:样品4较样品5和7更为多样,样品4中有5 条清晰的条带,样品5中有4 条清晰的条带,而样品7中仅有2 条清晰的条带。李晓红利用传统分离鉴定方法,从自然发酵小麦面粉面团中分离纯化出2 株乳酸菌菌株,通过菌落形态、菌体形态观察和生理生化实验分别鉴定为植物乳杆菌和嗜热链球菌[18]。陶东娅等[19]采用DGGE结合基因测序技术得出在黑龙江黏豆包酸面团中乳杆菌属为样品的优势细菌菌属;Zhang Guohua等[20]利用Illumina Hiseq 2000系统测序结合贝塔多样性分析,主成分分析和聚类分析确定了明串珠菌属、乳酸杆菌和魏斯氏菌属为5 份中国传统小麦面粉发酵面团中重要细菌属。虽然上述结果均有所不同,但一致认为,乳杆菌属是酸面团中的优势菌属之一。然而,本研究中所采集的样品主要是以玉米为原料发酵而成的酸汤子面团,在细菌多样性方面也与小麦面粉发酵面团有所区别,尤其是本研究发现,除了乳杆菌属外,魏斯氏菌属细菌在大部分样品中均有检出,也可以初步推断其在酸汤子发酵中发挥着较大作用。
2.3 酸汤子面团样品中真菌多样性分析
由图4可知,酸汤子面团9 份样品的真菌PCR-DGGE图谱中共发现16 个特异性扩增条带,每个样品的扩增条带呈现出较大差异性,得到的条带数量、真菌分布情况以及清晰度均有明显不同,且与细菌图谱相比可明显看出酸汤子面团样品中真菌种类较细菌相比更为丰富。对16 个特征条带进行回收和测序,通过NCBI进行BLAST同源性比对,结果如表2所示。
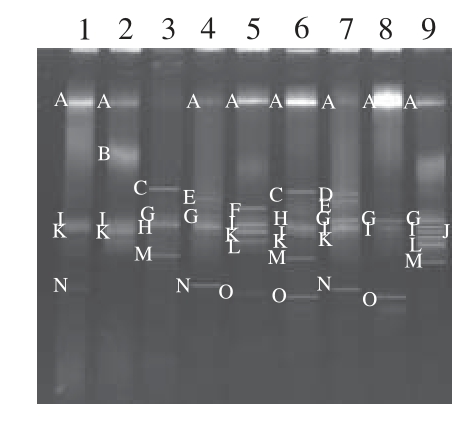
图4 酸汤子面团中真菌总DNA的PCR-DGGE图谱
Fig.4 DGGE profiles of PCR-amplified DNA from the total fungal population in each Suantangzi sourdough
表2 不同地区酸汤子面团样品中真菌26S D1/D2 rDNA区PCR-DGGE特异性条带比对结果
Table2 Identification of the bands obtained by PCR-DGGE of fungal populations based on 26S rDNA sequence
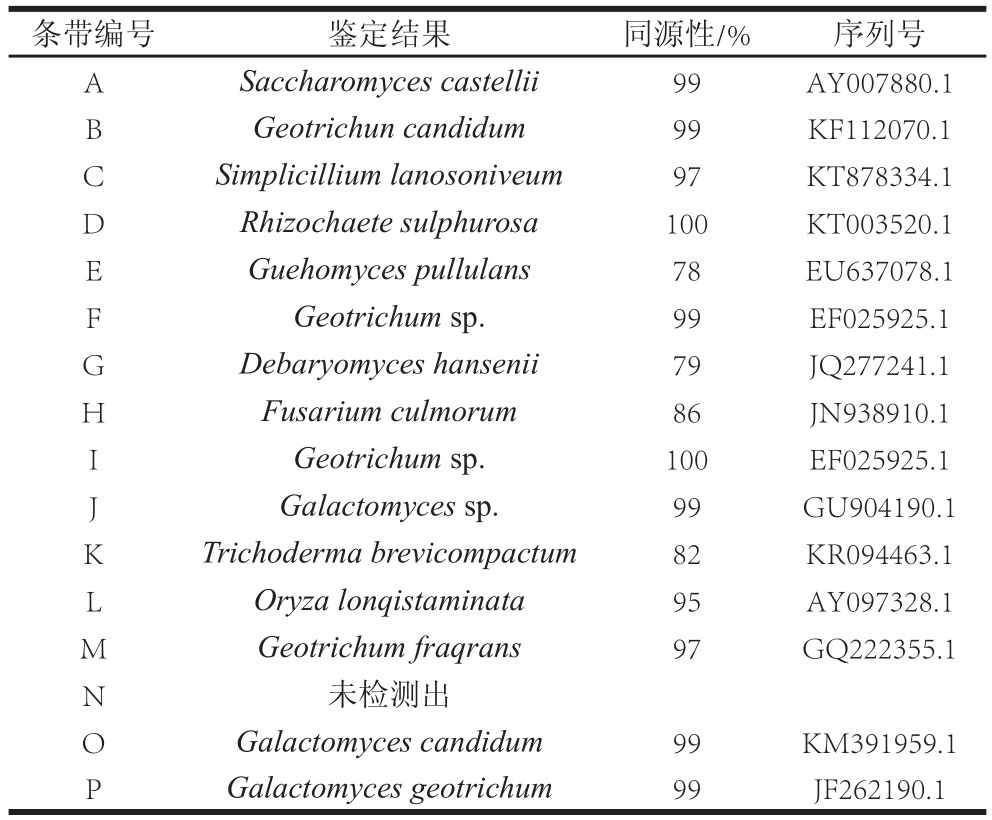
结合图4与表2分析可得,在获得的16 个特征条带中,共检测到14 种真菌,分别属于8 个菌属。其中,条带A、G、I、K条带出现的频率均占总样品总数的55.6%以上。其中条带A的鉴定结果为芽殖酵母菌(Saccharomyces castellii),出现的频率最高,占样品总数的88.9%;条带B、F、I、M的鉴定结果均属于地霉属(Geotrichun sp.),占总真菌种类的比例为25%,其中条带B、M经检测分别隶属于白地霉菌(G. candidum)和G. fraqrans;条带G、K的检测结果分别为汉逊德巴利酵母(Debaryomyces hansenii)和脐孢木霉菌(Trichoderma brevicompactum)。条带D、F、P和J只出现在个别样品中,为不同地区酸汤子样品的特征条带。由此可推断,酸汤子面团中的优势真菌菌属为酵母菌属和地霉菌属,其中芽殖酵母菌为7 个地区酸汤子面团样品中真菌的优势发酵菌种。
通过观察、分析可得出,不同地区的酸汤子面团样品中真菌分布存在着一定差异:样品9中的条带相对于其他地区样品最多、条带清晰、较亮,说明通辽地区酸汤子面团样品中真菌数目较其他地区样品比更多,其真菌菌群种类丰富,数目较多;相比之下,样品3中总条带数目较少,亮度较暗,说明沈阳地区酸汤子面团样品中真菌种类、数目较少。样品8中条带A与其他条带相比为最亮的菌带,说明芽殖酵母菌是桓仁地区酸汤子面团样品的优势菌群。同一地区不同区域酸汤样品中真菌的多样性有着共性,同时存在着明显的差异性。样品4、5、7均采自丹东地区的不同家庭,条带A在3 份样品中均被检测出,说明芽殖酵母菌与地霉属为丹东地区酸汤样品中真菌的优势发酵菌群。同时可以明显看出,条带A在样品5中与其余两家样品对应条带和同泳道其他条带相比均最亮,说明芽殖酵母菌在样品5中数量要比其他两个家庭的酸汤子面团样品多,为此样品中的优势菌群。而样品5中检测出的条带F、L和O,样品4和7中均没有检出。而样品7中检测出的条带D,样品4和5中没有检出。
本实验从酸汤子面团中鉴定出了普鲁兰久浩酵母(G. pullulans)与汉逊德巴利酵母。其中普鲁兰久浩酵母属于耐冷酵母,是一种能高产乳糖酶的菌株,乳糖酶是能将乳糖水解为葡萄糖和半乳糖的水解酶,在低温有高活性,在乳品加工和乳清引起的环境污染治理等方面,有着中温和高温乳糖酶无法达到的优越性,目前在医药、食品等领域应用广泛,在食品工业中有潜在的应用价值[21];汉逊德巴利酵母在体外实验扩散和挥发性化合物以及在无细胞上清液中表现出抗曲霉属显著抑制活性,其应用降低了生产胶孢镰刀菌(Fusarium subglutinans)伏马菌素的59.8%[22]。闫岩等[23]利用汉逊德巴利酵母不同浓度梯度的处理液对指状青霉体外和柑橘上进行生物防治实验,结果表明,汉逊德巴利酵母在适宜条件下能对青霉产生抑制,能够抑制霉菌孢子萌发和生长繁殖并在贮藏期间延缓柑橘青霉病的发生,明显降低腐烂指数,达到了最高的防治效率,在柑橘的整个贮藏期间保证了柑橘良好的贮藏品质。因此,汉逊德巴利酵母有很大的作生物防除剂的潜力。从本研究中发现的普鲁兰久浩酵母与汉逊德巴利酵母在之前酸汤子面团的研究中未分离出。同时值得注意,地霉属广泛分布在青贮饲料、泡菜、有机肥、各种乳制品和土壤等处,在本研究中酸汤子面团的样品中也广泛存在,在之前国内外酸面团的研究中从未鉴定出。白地霉现已广泛应用于酿造、医药、水处理及生物工程等多项产业,并在食品、香料、饲料、环境治理等领域有着巨大的开发价值[24-25],但也有着一定的致病性,其具体作用有待进一步研究。
本研究利用PCR-DGGE技术来分析酸汤子样品中菌群结构多样性,了解不同地区利用传统方法自然发酵的酸汤子面团中微生物的种类。结果发现酸汤子面团中真菌较细菌相比种类更为多样,魏斯氏菌和芽殖酵母菌是其中的优势真菌群,乳杆菌属为其优势细菌菌属。即不同地区样品存在共性,但同时同一地区不同样品之间也存在差异。该结果将为今后相关方面研究提供借鉴与参考,也为进一步开发质量安全、风味优良的酸汤子提供理论支持。
参考文献:
[1] 徐艳文. 满族的传统美食“酸汤子”[J]. 烹饪知识, 2013(8): 60.
[2] 何雨. 东北满族饮食中的面食文化研究[J]. 齐齐哈尔师范高等专科学校学报, 2014(2): 92-93. DOI:10.16322/j.cnki.23-1534/z.2014.02.036.
[3] de VUYST L, van KERREBROECK S, HARTH H, et al. Microbial ecology of sourdough fermentations: diverse or uniform?[J]. Food Microbiology, 2014, 37: 11-29. DOI:10.1016/j.fm.2013.06.002.
[4] HARTH H, van KERREBROECK S, de VUYST L. Community dynamics and metabolite target analysis of spontaneous, backslopped barley sourdough fermentations under laboratory and bakery conditions[J]. International Journal of Food Microbiology, 2016, 228: 22-32. DOI:10.1016/j.ijfoodmicro.2016.04.011.
[5] RODRÍGUEZ L R, PINGITORE E V, ROLLAN G, et al. Biodiversity and technological-functional potential of lactic acid bacteria isolated from spontaneously fermented quinoa sourdoughs[J]. Journal of Applied Microbiology, 2016, 120(5): 1289-1301. DOI:10.1111/jam.13104.
[6] 程晓燕. 酸面团乳酸菌优势菌群及发酵馒头品质与风味特性研究[D].无锡: 江南大学, 2015: 4-5.
[7] ZHANG J C, LIU W J, SUN Z H, et al. Diversity of lactic acid bacteria and yeasts in traditional sourdoughs collected from western region in Inner Mongolia of China[J]. Food Control, 2011, 22(5):767-774. DOI:10.1016/j.foodcont.2010.11.012.
[8] MUYZER G, de WAAL E C, UITTERLINDEN A G. Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J]. Applied and Environmental Microbiology, 1993, 59(3): 695-700.
[9] 周金明, 侯洁, 胡洋洋, 等. PCR-DGGE分析酸菜发酵液中菌群的动态变化[J]. 中国微生态学杂志, 2015, 27(10): 1124-1126. DOI:10.13381/j.cnki.cjm.201510003.
[10] 彭杨, 张奶英, 何利, 等. 基于PCR-DGGE技术的四川麸醋固态发酵过程中微生物群落分析[J]. 现代食品科技, 2016, 32(8): 171-177.
[11] 吴斯日古冷. 内蒙古西部地区酸面团中酵母菌和乳酸菌的分离鉴定及其生物多样性研究[D]. 呼和浩特: 内蒙古农业大学, 2011: 7-8.
[12] BOHANNON J. Ocean study yields a tidal wave of microbial DNA[J]. Science, 2007, 315: 1486-1487. DOI:10.1126/science.315.5818.1486.
[13] DANIEL R. The metagenomics of soil[J]. Nature Reviews Microbiology, 2005, 3(6): 470-478. DOI:10.1038/nrmicro1160.
[14] AMPE F, OMAR N B, MOIZAN C, et al. Polyphasic study of the spatial distribution of microorganisms in Mexican Pozol, a fermented maize dough, demonstrates the need for cultivation-independent methods to investigate traditional fermentations[J]. Applied and Environmental Microbiology, 1999, 65(12): 5464-5473.
[15] ERCOLINI D, HILL P J, DODD C E R. Development of a fluorescence in situ hybridisation method for cheese using a 16S rRNA probe[J]. Journal of Microbiological Methods, 2003, 52(2): 267-271. DOI:10.1016/ S0167-7012(02)00162-8.
[16] BIDDLE J F, FITZ-GIBBON S, SCHUSTER S C, et al. Metagenomic signatures of the Peru Margin subseafloor biosphere show a genetically distinct environment[J]. PNAS, 2008, 105(30): 10583-10588. DOI:10.1073/pnas.0709942105.
[17] 滕齐辉, 曹慧, 崔中利, 等. 太湖地区典型菜地土壤微生物16S rDNA的PCR-RFLP分析[J]. 生物多样性, 2006, 14(4): 345-351. DOI:10.3321/ j.issn:1005-0094.2006.04.010.
[18] 李晓红. 自然发酵面团中乳酸菌菌株的分离与鉴定[J]. 保鲜与加工, 2013, 13(4): 45-47. DOI:10.3969/j.issn.1009-6221.2013.04.012.
[19] 陶东娅, 金银旗. 不同地区粘豆包酸面团中微生物多样性分析[J]. 食品研究与开发, 2016, 37(13): 156-159. DOI:10.3969/ j.issn.1005-6521.2016.13.037.
[20] ZHANG G H, HE G Q. Predominant bacteria diversity in Chinese traditional sourdough[J]. Journal of Food Science, 2013, 78(8): 1218-1223. DOI:10.1111/1750-3841.12193.
[21] 宋春丽. 耐冷酵母Guehomyces pullulans 17-1菌株乳糖酶的研究[D].青岛: 中国海洋大学, 2010: 28-29.
[22] MEDINA-CÓRDOVA N, LÓPEZ-AGUILAR R, ASCENCIO F, et al. Biocontrol activity of the marine yeast Debaryomyces hansenii against phytopathogenic fungi and its ability to inhibit mycotoxins production in maize grain (Zea mays L. )[J]. Biological Control, 2016, 97: 70-79. DOI:10.1016/j.biocontrol.2016.03.006.
[23] 闫岩, 王明力, 李岑, 等. 汉逊德巴利酵母对采后柑橘青霉病的防治及贮藏品质的影响[J]. 食品与发酵工业, 2013, 39(8): 216-220. DOI:10.13995/j.cnki.11-1802/ts.2013.08.043.
[24] 田小海, 崔洪艳, 王莘. 白地霉发酵产物的应用研究进展[J].安徽农学通报, 2016, 22(9): 31-32. DOI:10.16377/j.cnki.issn1007-7731.2016.09.015.
[25] 孙丙升, 郑莉莉, 刘天明, 等. 白地霉的应用研究进展[J].食品研究与开发, 2008, 29(9): 163-166. DOI:10.3969/ j.issn.1005-6521.2008.09.049.
Microbial Diversity Analysis of Corn Sourdough for Suantangzi, a Traditional Northwestern Chinese Fermented Food Product
WU Rina1,2, ZHANG Ying1, ZHANG Hongxiao1, TAO Dongbing1, SUN Huijun1,3, YUE Yuanyuan1, WU Junrui1,*
(1. College of Food Science, Shenyang Agricultural University, Shenyang 110866, China; 2. State Key Laboratory of Food Science and Technology, School of Food Science and Technology, Jiangnan University, Wuxi 214122, China; 3. College of Morden Agricultural Technology, Liaoning Agricultural Economic School, Jinzhou 121001, China)
Abstract:The Suantangzi is a traditional ethnic food with good nutritional properties and unique flavor, which is popular among Manchu people and northeastern Chinese people. The diverse microbes in corn sourdough for Sutangzi play an extremely important role in the development of its nutritional quality. However, the microbial community diversity is still unclear today. This study aimed to study the diversity of the microflora in nine samples of Suantangzi sourdough collected from different regions by polymerase chain reaction-denaturing gradient gel electrophoresis (PCR-DGGE). The results showed that a total of14 species of fungi were identified from these samples, including Saccharomyces castellii, Geotrichun candidum, Simplicillium lanosoniveum, Rhizochaete sulphurosa, Guehomyces pullulans, Debaryomyces hansenii, Fusarium culmorum, Trichoderma brevicompactum, Oryza lonqistaminata, Geotrichun fraqrans, Galactomyces candidum, Galactomyces geotrichum,
Geotrichum sp. and Galactomyces sp. S. castellii was detected in most samples. Based on this observation, S. castellii was predicted to be the dominant fungal species in Suantangzi. Moreover,4 species of bacteria were also identified, including Bacillus pumilu, Lactobacillus tucceti, Lactobacillus plantarum and Weissella paramesenteroides. Weissella paramesenteroides was supposed to be the dominant species for the widest distribution in the samples.
Key words:Suantangzi; natural fermentation; polymerase chain reaction-denaturing gradient gel electrophoresis; microbe; diversity
DOI:10.7506/spkx1002-6630-201712004
中图分类号:TS264.2
文献标志码:A
文章编号:1002-6630(2017)12-0021-06
收稿日期:2016-06-15
基金项目:国家自然科学基金面上项目(31471713;31470538);中国博士后科学基金项目(2014M560395);辽宁省农业领域青年科技创新人才培养计划项目(2014048);辽宁省高等学校优秀人才支持计划项目(LR2015059;LJQ2015103);江苏省博士后科研资助计划项目(1402071C)
作者简介:乌日娜(1979—),女,副教授,博士,研究方向为食品生物技术。E-mail:wrn6956@163.com
*通信作者:武俊瑞(1977—),男,副教授,博士,研究方向为食品生物技术。E-mail:junruiwu@126.com
引文格式:乌日娜, 张颖, 张红萧, 等. 酸汤子玉米面团中微生物多样性分析[J]. 食品科学, 2017, 38(12): 21-26.
DOI:10.7506/ spkx1002-6630-201712004. http://www.spkx.net.cn
WU Rina, ZHANG Ying, ZHANG Hongxiao, et al. Microbial diversity analysis of corn sourdough for Suantangzi, a traditional northwestern Chinese fermented food product[J]. Food Science, 2017, 38(12): 21-26. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201712004. http://www.spkx.net.cn