表1 研究用探针和引物信息
Table1 Sequences of probes and primers used in this study
张佳玲1,潘 广2,章桂明2,程颖慧2,向才玉2,陈枝楠3,谢忠稳1,*,凌杏园2,*
(1.安徽农业大学茶与食品科技院,茶树生物学与资源利用国家重点实验室,安徽 合肥 230036;2.深圳出入境检验检疫局动植物检验检疫技术中心,广东 深圳 518045;3.深圳市检验检疫科学研究院,广东 深圳 518010)
摘 要:建立基于QX100微滴式数字聚合酶链式反应(polymerase chain reaction,PCR)平台的我国未批准转基因玉米品系VCO-01981-5的二重微滴式数字PCR定量检测方法。该方法选择基因组中单拷贝的玉米内源基因hmg和VCO-01981-5品系边界序列为定量靶序列,分别设计不同的PCR扩增引物和TaqMan探针,并对两种探针用不同的荧光进行标记,然后将上述探针和引物置于同一个PCR反应体系中以同时定量两个靶标序列。特异性实验结果显示该法只有VCO-01981-5品系的两个靶序列才都有扩增信号。灵敏度、线性和准确性实验结果显示在定量结果相对标准偏差不大于25%时,最低可稳定定量5 个拷贝的VCO-01981-5品系特异性序列分子和4 个拷贝的内源基因hmg分子;而在高达50 ng模板DNA以下范围内,PCR反应模板量与测定样品拷贝数之间呈高度正相关,相关系数达0.99以上;平均误差小于10%。结果表明本研究建立的该玉米品系定量方法特异性强,稳定性好,精确性、准确性以及灵敏度高,定量范围广,可用于进、出口农产品和食品中该转基因玉米品系成分的定量检测。此外,该法还可为其他转基因玉米品系及其他转基因作物品系建立类似定量检测方法提供参考。
关键词:转基因玉米;品系VCO-01981-5;微滴式数字PCR;定量检测
2015年是转基因作物商业化第20周年,全球转基因作物累计种植面积达到空前的20亿 公顷。20年来,农民收益超过了1 500亿 美元。然而转基因农作物在给人们带来巨大利益的同时也引发了公众对其安全性的担忧。同欧盟等国家一样,我国高度重视转基因农作物及食品的安全问题,不仅要求对转基因产品进行标识,还要求对未批准的转基因品系及其产品予以取缔,因此,必须对农产品进行定性和定量的转基因检测[1-3]。
目前,转基因检测的方法主要有基于核酸检测的聚合酶链式反应(polymerase chain reaction,PCR)技术[4]、核酸杂交技术[5]和基因芯片技术[6],基于蛋白质检测的Western Blot[7]、酶联免疫吸附测定[8]等。其中应用最广泛的是普通PCR方法和实时荧光PCR方法。如在定性检测方面,Matsuoka等[9]利用多重PCR同时检测玉米内源基因Ze1和5 种转基因品系,该方法特异性强、灵敏度较高,可以检出0.5%含量的转基因玉米品系。在定量检测方面,Kim等[10]用构建的含小麦内源基因acc和小麦品系MON71800特异性序列的标准质粒pGEM-M71800制作标准曲线,建立的普通PCR方法最低能检测10 个拷贝品系特异序列分子;而建立的实时PCR方法最低能检测5 个拷贝,定量限(limit of quantitation,LOQ)为10 个拷贝,其相对标准偏差在0.23%~1.12%范围内,表明实时荧光PCR方法灵敏度更高、定量结果重复性更好。Wu Gang等[11]同样采用实时荧光PCR方法建立了定量检测转基因油菜品系Topas 19/2的方法,该方法特异性强,只有品系Topas 19/2的内源基因HMG-I/Y和品系特异性序列两者均有扩增;品系特异性基因的检出限(limit of detection,LOD)和LOQ分别约为5 个和50 个拷贝数,相对标准偏差在0.260%~1.541%范围内,表明方法灵敏度和重复性较高。
不管是常规PCR还是实时荧光PCR定量检测,首先必须用已知拷贝数的标准DNA制定标准曲线,以确定PCR初始模板拷贝数与PCR产物量(普通PCR定量)或与Ct值(实时荧光PCR定量)的关系;再由PCR产物量或Ct值经由标准曲线公式计算得到样品目标序列的拷贝数。由于样品DNA与制作标准曲线的标准DNA在质量上(包括DNA降解程度、是否含有反应抑制物等)不可能完全一致,势必造成两者PCR反应扩增效率的差异,从而影响PCR反应的产物量或Ct值,进而影响定量结果的准确性。此外,常规PCR定量需要通过琼脂糖凝胶电泳获得目标条带产物量,不仅操作繁琐,且容易污染。实时荧光PCR尽管比普通PCR特异性强、污染少,但同样需要制作标准曲线,检测结果受扩增效率影响[12-13]。
微滴式数字PCR是最近兴起的一种核酸单分子扩增技术。其原理是:一个PCR反应管中90 μL的油水反应体系被微滴发生器制 备成近20 000 个油包水小微滴;当待测DNA模板分子数低于一定的数目,微滴只含有一个分子的DNA模板和足够量的用于荧光PCR反应所需的所有其他组分如dNTP、Taq DNA聚合酶以及探针和引物等。待所有微滴实时荧光PCR反应结束后,逐一检查每个微滴。只要微滴有荧光信号检出,就被认为有实时荧光PCR反应发生,记有一个分子的待测模板被检出;统计所有阳性微滴数,就可得出样品中待测DNA模板的分子数[14-18]。数字PCR技术的上述特点表明其对目标序列拷贝数的定量不依赖标准曲线,且定量结果不受PCR扩增效率影响,因此,具有比普通PCR和实时荧光PCR定量技术无法比拟的优越性[19-22]。
数字PCR在转基因定量检测方面也有应用[23-29]。Corbisier等[25]利用数字PCR检测转基因玉米MON810品系外源基因和内源参照基因的拷贝数比例,同时用实时PCR检测质粒DNA标准物质,并将两者的结果进行比较,获得了一致性,说明两种方法均能用于MON810品系拷贝数的分析测定。Demeke等[26]将外源基因和内源参照基因的探针引物混合到单个PCR反应中,利用二重ddPCR对转基因油菜OXY235品系和大豆DP305423品系进行绝对定量,结果发现含量为1%、0.1%及0.01%的标准品样品均能被ddPCR和实时PCR定量,而含量为0.001%的标准品样品只能被ddPCR检出。Fu Wei等[27]选取CaMV35s启动子和NOS终止子作为筛选转基因作物的通用元件,用数字PCR进行定量检测,并对MON810等9 种转基因品系进行特异性验证,结果获得LOD为0.1%,这一低于欧盟规定阈值的结果,表明该方法特异性好、灵敏度高,适于转基因成分含量的精确定量。
转基因玉米VCO-01981-5是有Genective S.A.公司开发的抗草甘膦除草剂的新品系,于2013和2014年分别在美国和加拿大获准商业化生产和利用。该品系未获我国农业部批准,国际、国内也未见有相应的品系特异性检测方法公开报道。本研究以采购的VCO-01981-5标准品为实验材料,根据文献公开的外源基因插入位点附件序列设计该品系特异性检测引物和探针,以Bio-Rad公司生产的QX100微滴式数字PCR为实验平台,研究建立了这一我国目前尚未批准进口的转基因玉米品系的精准定量方法,以实现对该转基因玉米成分的定量检测。
1.1 材料、试剂与仪器
本研究共选取26 份实验材料,分别为转基因玉米品系MIR162、MON89034、Bt176、Bt11、MON88017、MON810、MON863、T25、MIR604、ES3272、GA21、59122、MON87460、98140、DAS40278-9、MON87427、VCO-01981-5(100%质量百分含量),转基因油菜品系MS1、RF1、Topas19/2,转基因水稻品系KF6,转基因大豆品系A2704-12、MON89788、305423,转基因棉花品系GHB614,转基因马铃薯品系EH92-527-1,以上材料均于欧盟标准局和美国油脂化学协会。
ddPCR Super Mix、ddPCR Droplet Generation Oil、ddPCR Droplet Reader Oil、Droplet Generator DG8 Cartridge、Droplet Generator DG8 Gasket、plate holder和96孔板试剂和耗材 美国Bio-Rad公司;植物基因组DNA提取试剂盒DNeasy Plant Mini kit 德国Qiagen公司。
QX100TMDroplet Digital PCR系统(包括PCR仪、微滴生成仪和微滴读取仪和封膜仪四部分) 美国Bio-Rad公司;Mcrofuge®16型离心机 美国贝克曼库尔特公司;Vortex genie3型涡旋振荡仪 德国IKA公司;恒温水浴锅 北京市六一仪器厂;Nanodrop 2000c核酸蛋白分析仪 美国Thermo Scientif i c公司。
1.2 方法
1.2.1 DNA的提取
转基因玉米等供试材料的基因组DNA提取参照试剂盒说明书。
1.2.2 探针和引物
转基因玉米内源基因选用玉米高速泳动蛋白(high mobility group proteins,HMG)基因,HMG是细胞核中一种与染色质结合在一起的高度丰富的非组蛋白,其分子质量比较小,但在染色质的结构与功能中可能起着重要的作用。该基因在GenBank的检索号为AJ131373.1,在玉米基因组中仅一个拷贝。
转基因玉米VCO-01981-5品系特有序列选择外源基因插入位点5’端跨边界序列,该序列在玉米基因组中为单拷贝。
用于数字PCR检测转基因玉米内源基因hmg和品系VCO-01981-5特异序列的探针、引物序列及探针标记信息见表1。
表1 研究用探针和引物信息
Table1 Sequences of probes and primers used in this study

1.2.3 反应体系和反应条件
每个提取DNA样品检测时设置3 个平行PCR反应。
d d P C R反应体系共2 0 μ L,其中2×ddPCR Super Mix10 μL,品系特异性序列和内参照hmg基因正、反向引物各1 μL(6 μmol/L)、探针各1 μL(3.6 μmol/L),DNA模板(15 ng/μL)2 μL,补水至20 μL。分别将20 μL的反应体系和70 μL微滴生成油加在微滴生成卡槽相应的小室中,盖上胶垫后放入微滴生成仪中,由仪器自主振荡生成微滴。待振荡结束后将产生的40 μL左右的微滴转移到96 孔板中,并用热封膜仪对其进行封膜,最后置于PCR仪进行PCR反应。
反应条件:95 ℃预变性10 min;95 ℃变性15 s,56 ℃退火1 min,40 个循环,98 ℃、10 min酶热失活,产物4 ℃保存。
扩增结束后,将96 孔板置入微滴分析仪中读取信号,并使用QuantaSoft V1.3.2软件分析实验数据。样品中转基因品系含量以样品基因组DNA中品系特异序列拷贝数占内源基因拷贝数的百分比即拷贝数含量表示。
1.2.4 稳定性实验
以1 ng/μL转基因玉米品系VCO-01981-5(100%质量百分含量)基因组DNA作为模板DNA的工作质量浓度,再按照上述反应体系要求配制反应体系并进行PCR反应。稳定性实验设置3 个平行,要求3 个重复实验结果的相对标准偏差(relative standard deviation,RSD)不超过25%。
1.2.5 特异性实验
选用转基因玉米品系MIR162、MON89034、Bt176、Bt11、MON88017、MON810、MON863、T25、MIR604、ES3272、GA21、59122、MON87460、98140、DAS40278-9、MON87427、VCO-01981-5(1 0 0%含量),转基因油菜品系M S 1、R F 1、Topas19/2,转基因水稻品系KF6,转基因大豆品系A2704-12、MON89788、305423,转基因棉花品系GHB614,转基因马铃薯品系EH92-527-1为实验材料。以这26 种转基因样品基因组DNA为模板配制ddPCR反应体系,并进行PCR反应。其中VCO-01981-5品系作为阳性对照。
1.2.6 定量线性范围测定
转基因玉米品系VCO-01981-5(100%质量百分含量)基因组DNA分别用纯水稀释至25、5、1、0.2、0.04、0.008、0.0016 ng/μL共7 个工作质量浓度。以2 μL上述各稀释DNA作为模板进行ddPCR反应,每个质量浓度重复3 个反应。计算各质量浓度样品3 次重复实验结果的RSD,以起始样品用量对RSD不超过25%的各质量浓度样品的定量结果计算两者的线性关系和相关系数。LOQ定义为检测结果RSD不超过25%的最低样品用量或样品拷贝数;LOD定义为能够检测的样品最低拷贝数或样品用量。
1.2.7 LOQ验证
取定量性范围下限的DNA样品用量进行LOQ验证,共进行10 个平行反应,计算平行反应实验结果的平均值和RSD,如RSD不大于25%,则为该方法目标序列的LOQ。
1.3 准确性检测
本研究实验材料VCO-01981-5标准品(100%)是玉米种子粉末,种子由转基因父本和非转基因母本杂交产生(F1代)。这种质量百分含量100%的杂合型玉米标准品,其拷贝数含量(品系特异序列和内源基因拷贝数比值)理论上仅为39%左右[25,30]。这里取1.2.6节中定量线性范围测定中RSD不大于25%的实验数据,计算不同模板DNA用量测定拷贝数含量和RSD、平均拷贝数含量和RSD,定量结果准确性以误差百分数表示。
2.1 方法的建立和稳定性检测结果
反应体系参照Morisset[17]建立的MON810品系的二重数字PCR方法。本实验检测VCO-01981-5品系特异性序列探针采用5’-FAM—3’-BHQ1荧光标记,检测内源基因hmg探针采用5’-HEX—3’-BHQ1荧光标记,从而保证同一个反应体系中两种目标序列的扩增产物可以同时检出。反应酶采用Bio-Rad生产的与其平台相适应的预混液(Super Mix),由于该预混液对体系中各种成分抗干扰性和适应性广,探针和引物用量等同文献[17]用量。
由于针对不同目标序列设计的引物、探针序列不同,其退火温度不同,因而二重数字PCR反应条件中的退火和延伸温度特别重要。本研究应用合成的内、外源基因引物、探针和转基因玉米品系VCO-01981-5标准品基因组DNA按1.2.3节要求配制PCR反应体系并测试了7 种(65、63.8、62.0、59.1、55.7、52.9、51 ℃)不同的退火温度,结果发现55.7 ℃有利于两个目标序列的扩增(数据未提供)。
56 ℃反应条件下3 次重复实验结果如图1所示。图1A为FAM通道,是各微滴VCO-01981-5品系特异序列扩增反应结果;图1B图为HEX通道,是各微滴内源基因hmg扩增反应结果。如图1A中荧光扩增值介于10 000~14 000之间的圆点和图1B中荧光扩增值介于4 000~6 000之间的圆点分别代表发生PCR扩增反应的微滴,荧光分析仪判读为阳性信号;而位于本底荧光值附近的圆点代表未发生扩增的微滴,荧光分析仪判读为阴性信号。阳性微滴的荧光增量大、信号强,代表扩增产物量大,说明该微滴扩增效率高;阴性微滴的荧光增量少、信号弱,表明该微滴扩增效率低。对数字PCR来说不管以上阳性微滴荧光信号强弱,均表示一个分子目标序列检出,检测结果不受微滴扩增效率影响。阳性微滴与阴性微滴荧光信号差别大,图谱分得很开,表明在同一个PCR反应体系(微滴)中可以同时检测内、外源两个目标序列,且本研究所确立的探针、引物、反应体系和反应条件等适于该品系内源基因和外源序列的扩增反应。
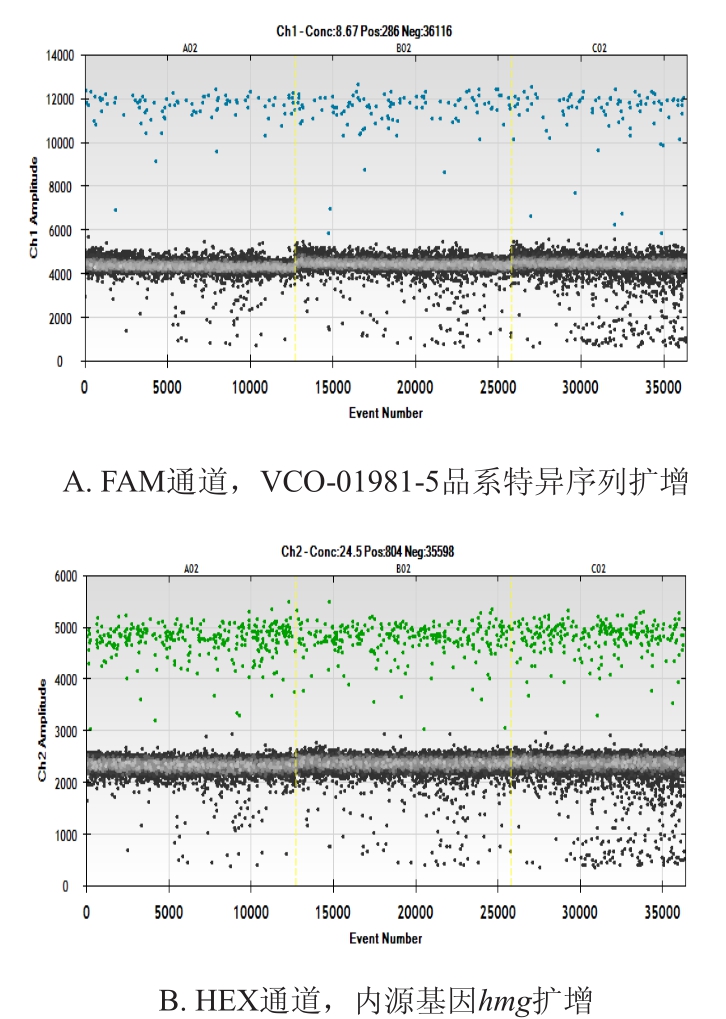
图1 各微滴品系标准品DNA内源基因和品系特异序列扩增反应情况
Fig.1 Amplification results of endogenous gene and event-specific boundary sequences of standard DNA in the droplets
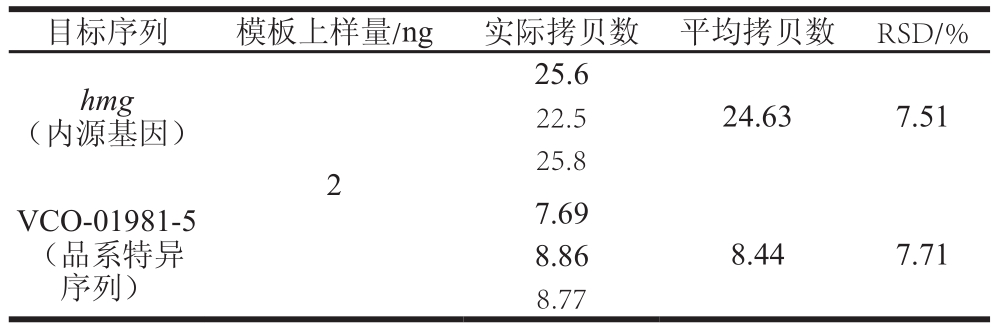
为考察研究建立方法的稳定性,本研究特地重复进行3 次实验,如图1和表2所示。内参基因3 次重复实验测的内源基因拷贝数分别为25.6、22.5和25.8,平均检测拷贝数为24.63,相对标准偏差(relative standard deviation,RSD)仅为7.51%;品系特异序列3 次扩增反应所的拷贝数分别为7.69、8.86和8.77,平均检测拷贝数为8.44,RSD仅为7.71%。可见,3 次重复实验所得实验结果非常一致,表明该新建立的定量检测方法具有很好的稳定性。
表2 玉米VCO-01981-5定量方法稳定性检测结果
Table2 Stability evaluation of the quantitative method for event
表2 玉米VCO-01981-5定量方法稳定性检测结果
Table2 Stability evaluation of the quantitative method for event VCO-01981-5 with droplet digital PCR
2.2 特异性检测结果
应用本研究建立数字PCR方法对品系VCO-01981-5和其他25 种转基因玉米、油菜、水稻、大豆、棉花、马铃薯品系基因组DNA进行扩增的结果如图2所示。17 个转基因玉米品系基因组DNA在HEX通道下都能检测到荧光信号,而转基因油菜、水稻、大豆、棉花、马铃薯品系基因组DNA均未检测到荧光信号,表明17 个品系的玉米内源基因hmg都发生了扩增反应。在FAM通道下,仅阳性对照样品VCO-01981-5品系DNA检测到荧光信号,其余品系的基因组DNA均未检出荧光信号,说明品系特异性边界序列发生了扩增反应。以上结果表明本研究所建立方法特异性强。图2显示阳性微滴很多且与阴性微滴区分不开,这是样品DNA用量太多的缘故。

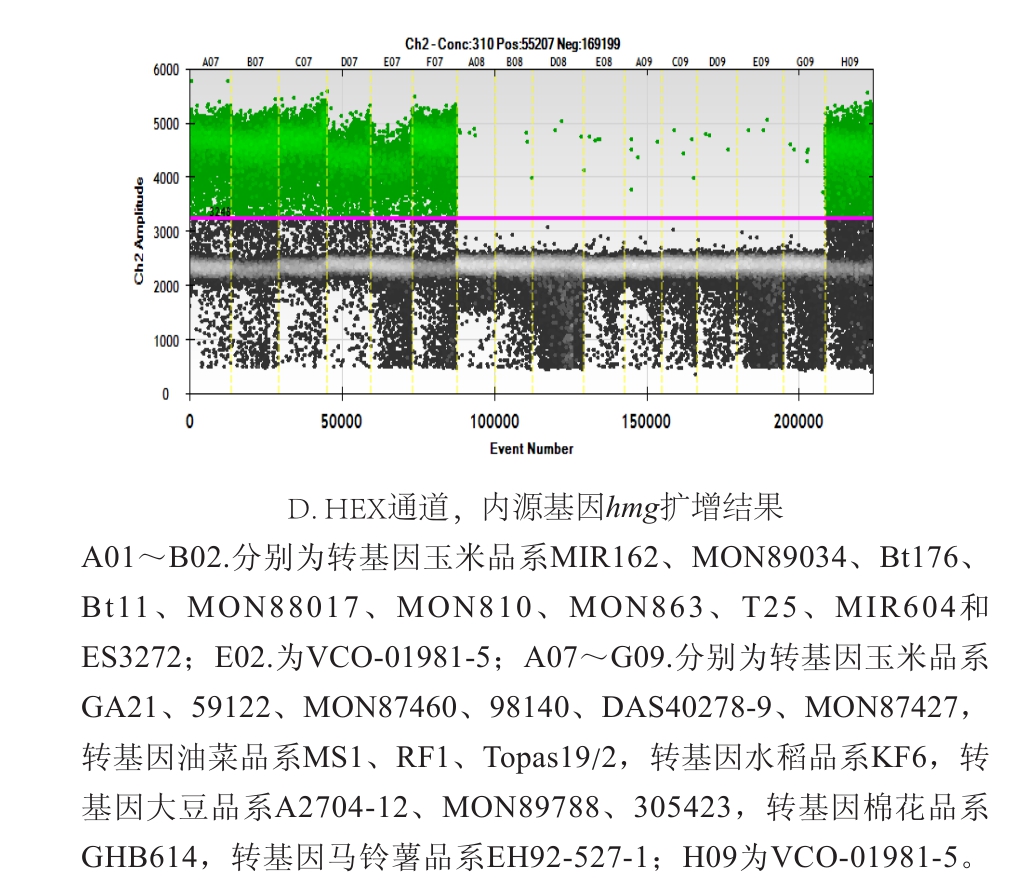
图2 VCO-01981-5品系特异性双重数字PCR定量方法特异性检测实验结果
Fig.2 Specificity evaluation of the quantitative method for event VCO-01981-5 event with duplex digital PCR
2.3 定量线性范围测定结果
表3 玉米VCO-01981-5数字PCR定量方法的线性范围测定结果
Table3 Linear range evaluation of the quantitative method for event VCO-01981-5 with droplet digital PCR

注:*. RSD不大于25%时玉米品系VCO-01981-5特异性序列每微升的最低拷贝数;#. RSD不大于25%时内源基因hmg每微升的最低拷贝数。表6同。
应用本研究建立二重数字PCR方法测定不同用量模板DNA目标序列拷贝数的结果如表3所示。单位是每微升反应体系(共20 μL)所含目标序列的拷贝数,每个测定3 次重复,以计算平均拷贝数和RSD。从表3可以看出,在RSD不大于25%的情况下,玉米内源基因hmg可以定量到3.8 拷贝(0.19×20),转基因玉米品系VCO-01981-5特异性序列可以定量到4.8 拷贝(0.24×20)。若以PCR模板DNA用量为横坐标,每微升目标序列拷贝数为纵坐标绘制曲线,发现两者呈线性关系。从样品DNA用量与所测品系特异序列拷贝数之间的关系图中发现在样品用量50~0.08 ng范围内,本研究方法测定品系特异序列的拷贝数与样品用量呈高度正相关,相关系数达0.99以上,其拟合曲线方程为 y=4.673x-0.631 3,表明本研究方法品系特异序列的定量线性范围在4.8~4 666.6拷贝之间,跨越了3 个数量级。从样品DNA用量与所测内源基因拷贝数之间的关系图中发现在样品用量50~0.016 ng范围内内参基因拷贝数与样品用量呈现出良好的线性关系,相关系数达0.99以上,其拟合曲线方程为y=13.669x-2.311 5,表明本研究方法内源基因序列的定量线性范围在3.8~13 646.6拷贝之间,跨越了4 个数量级。以上研究结果表明本研究建立的数字PCR方法定量范围广、定量精度高,结果重复性好。
2.4 LOQ验证结果
从表3可以看出,品系特异性序列定量线性范围下限的样品DNA用量为0.08 ng。取0.08 ng样品DNA进行10 次平行定量检测,结果如表4所示,10 次平行定量的品系特异性序列平均拷贝数为4.8 个,RSD为24%,符合要求(小于25%)。内源基因LOQ验证实验取用样品DNA量0.016 ng,10 次定量结果如表5所示,10 次平行定量的内源基因平均拷贝数为4,RSD为22.91%,符合要求(小于25%)。以上结果证实内源基因的LOQ为4 拷贝,品系特异性序列的LOQ为4.8 拷贝,表明本研究建立的转基因玉米VCO-01981-5品系特异性定量方法灵敏度高。
表4 玉米VCO-01981-5品系特异性序列LOQ验证结果
Table4 Limit of quantitative analysis for event-specific sequence of event VCO-01981-5 with droplet digital PCR
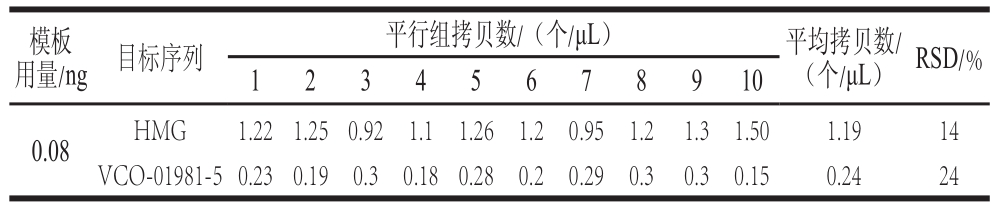
表5 玉米内源基因hmg LOQ验证结果
Table5 Limit of quantitative analysis for endogenous gene of event VCO-01981-5 with droplet digital PCR

2.5 定量准确性分析结果
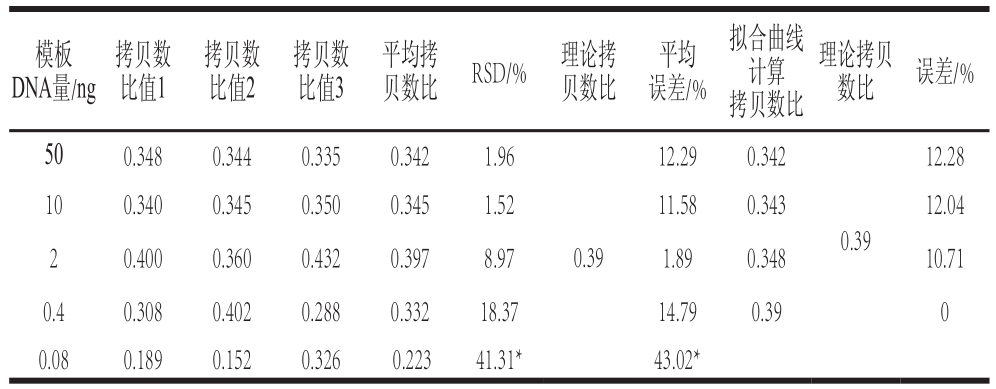
定量准确性的验证往往用已知准确含量的标准品或构建的质粒DNA作为实验材料。对于转基因玉米,如果供试标准品为叶片等除种子以外的组织,100%质量百分含量的转基因玉米标准品,如果是纯合子,则拷贝数含量理论上也为100%;如果是杂合子,则拷贝数含量理论上仅为50%。玉米种子由胚、果皮、种皮和胚乳4 部分构成,前3 部分拷贝数含量与质量百分含量的关系同叶片组织一样。玉米种子胚乳细胞是三倍体,如果胚乳细胞是杂合基因型,且其外源基因来源于父本,则胚乳细胞三倍体中只有一个拷贝的外源基因,这样的胚乳细胞拷贝数含量仅有质量百分含量的1/3(33.3%);如果外源基因来源于母本,则胚乳细胞三倍体中有两个拷贝的外源基因,这样的胚乳细胞拷贝数含量是质量百分含量的2/3(66.6%)。对于全部(100%质量百分含量)由转基因玉米整粒种子制成的粉末状标准品,如果胚乳细胞是杂合基因型,且其外源基因来源于父本,该标准品拷贝数含量取决于胚乳在整颗种子中所占的比例。如果胚乳占比越小或无,则拷贝数含量接近50%;如果胚乳占比越大,则拷贝数含量接近33%。因此,这种100%的外源基因源于父本的杂合型转基因玉米种子拷贝数含量在33%~50%之间,而根据Corbisier[20]和Zhang[30]等的研究,这样的玉米种子平均拷贝数含量为39%左右。
依标准品证书说明,本研究实验材料VCO-01981-5标准品(100%)是玉米种子粉末,种子由转基因父本和非转基因母本杂交产生(F1代),表明其拷贝数含量为39%左右。该标准品7 种模板DNA用量测定的内、外源基因拷贝数见表3,其中5 个用量测定数据的RSD均小于25%,表明精确性较高。本定量准确性验证采用同样的实验数据,计算拷贝数含量和定量检测误差,计算结果见表6。结果发现最低模板DNA用量(0.08 ng)因内、外源序列拷贝数测定的RSD值已近25%,拷贝数比值计算进一步放大了误差,比值计算结果的RSD超过25%;其他4 次定量的误差均在15%以内,4 次定量平均误差仅为9.23%。如果根据定量方法线性范围拟合曲线计算,这4 次定量结果的误差同样都小于15%,有的误差甚至为0%,平均误差只有8.76%。以上结果表明本研究建立VCO-01981-5品系数字PCR定量方法定量准确性高。
表6 玉米VCO-01981-5数字PCR定量方法定量检测准确性分析结果
Table6 Accuracy analysis for quantitative detection method of event VCO-01981-5 with droplet digital PCR
本实验利用微滴式数字PCR技术建立了针对我国未批准的转基因玉米品系VCO-01981-5特异性定量方法。数字PCR是一种近年发展起来的第3代先进定量检测手段,除具备不需要制作标准曲线、定量结果不受PCR扩增效率等实时荧光PCR定量不具有的优势外,本研究建立的二重ddPCR定量方法比单重微滴式数字PCR定量方法优势明显:其一,同一个微滴PCR反应体系在同时定量内源和外源两个靶序列,比一次反应只定量一个靶序列更经济和快速。其二,二重微滴式数字PCR定量结果不受样品DNA用量和质量如降解等的影响,因为同一DNA样品,由于降解是随机发生的,同一样品DNA不同用量之间内、外源基因的比例是不会改变的,所以定量结果更为精确和准确。二重微滴式数字PCR上述特点非常重要,不仅避免了单重ddPCR定量因二次取样所产生的样品内的误差,也可以消除不同样品DNA因取样不一致而造成的样品间的误差,因而在操作上使样品间的比较更为简单。针对本研究建立方法各项指标考察发现,该定量方法稳定性好、特异性强、定量范围广、定量检测灵敏度高,同时定量结果准确性高、重复性好,完全可以满足当前和今后对该品系精准定量的实际需要。
参考文献:
[1] JAMES C. 2015年全球生物技术/转基因作物商业化发展态势[J].中国生物工程杂志, 2016, 36(4): 1-11.
[2] 沈平, 武玉花, 梁晋刚, 等. 转基因作物发展及应用概述[J]. 中国生物工程杂志, 2017, 31(1): 119-128.
[3] 王荣谈, 姜羽, 韦娇君, 等. 转基因生物及其产品的标识与检测[J].植物生理学报, 2013, 49(7): 645-654.
[4] MIRAGLIA M, BERDAL K, BRERA C, et al. Detection and traceability of genetically modif i ed organisms in the food production chain[J]. Food Chemical Toxicology, 2004, 42: 1157-1180.
[5] SIDOROV V, GILBERTSON L, ADDAE P, et al. Agrobacteriummediated transformation of seedling-derived maize callus[J]. Plant Cell Reports, 2006, 25(4): 320-328.
[6] SHAO N, JIANG S, ZHANG M, et al. MACRO: a combined microchip-PCR and microarray system for high-throughput monitoring of genetically modif i ed organisms[J]. Analytical Chemistry, 2014, 86: 1269-1276.
[7] PAUL V, GURETLER P, WIEDEMANN S, et al. Degradation of Cry1Ab protein from genetically modified maize (MON810) in relation to total dietary feed proteins in dairy cow digestion[J]. Transgenic Research, 2010, 19: 683-689.
[8] KAMLE S, OJHA A, KUMAR A. Development of an enzyme linked immunosorbant assay for the detection of Cry2Ab protein in transgenic plants[J]. GM Crops, 2011, 2(2): 118-125.
[9] MATSUOKA T, KURIBARA H, AKIYAMA H, et al. A multiplex PCR method of detecting recombinant DNAs from fi ve lines of genetically modified maize[J]. Journal of the Food Hygienic Society of Japan, 2001, 42(1): 24-32.
[10] KIM J, PARK S, ROH H, et al. A simplif i ed and accurate detection of the genetically modified wheat MON71800 with one calibrator plasmid[J]. Food Chemistry, 2015, 176: 1-6.
[11] WU G, WU Y, XIAO L, et al. Event-specific qualitative and quantitative PCR detection of genetically modified rapeseed Topas 19/2[J]. Food Chemistry, 2009, 112: 232-238.
[12] JIANG Lingxi, YANG Litao, RAO Jun, et al. Development and inhouse validation of the event-specif i c qualitative and quantitative PCR detection methods for genetically modified cotton MON15985[J]. Journal of the Science of Food & Agriculture, 2010, 90(3): 402-408.
[13] KIM J, PARK S, ROH H, et al. Event-specific qualitative and quantitative detection of three genetically modified papaya events using a single standard reference molecule[J]. Food Control, 2015, 55:127-132.
[14] PINHEIRO L, COLEMAN V, HINDSON C, et al. Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantif i cation[J]. American Chemical Society, 2012, 84: 1003-1011.
[15] SANDERS R, HUGGETT J, BUSHELL C, et al. Evaluation of digital PCR for absolute DNA quantif i cation[J]. American Chemical Society, 2011, 83: 6474-6484.
[16] HINDSON B, NESS K, MASQUELIER D, et al. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number[J]. American Chemical Society, 2011, 83: 8604-8610.
[17] MORISSET D, STEBIH D, MILAVEC M, et al. Quantitative analysis of food and feed samples with droplet digital PCR[J]. PLoS ONE, 2013, 8(5): e62583.
[18] 苗丽, 张秀平, 陈静, 等. 微滴数字PCR法对肉制品中牛源和猪源成分的定量分析[J]. 食品科学, 2016, 37(8): 187-191. DOI:10.7506/ spkx1002-6630-201608033.
[19] HINDSON C, CHEVILLET J, BRIGGS H, et al. Absolute quantif i cation by droplet digital PCR versus analog real-time PCR[J]. Nature Methods, 2013, 10(10): 1003-1005.
[20] HAYDEN R, GU Z, INGERSOLL J, et al. Comparison of droplet digital PCR to real-time PCR for quantitative detection of cytomegalovirus[J]. Journal of Clinical Microbiology, 2013, 51(2):540-546.
[21] KELLETY K, COSMAN A, BELGRADER P, et al. Detection of methicillin-resistant staphylococcus aurous by a duplex droplet digital PCR assay[J]. Journal of Clinical Microbiology, 2013, 51(7):2033-2039.
[22] WHALE A, COWEN S, FOY C, et al. Methods for applying accurate digital PCR analysis on low copy DNA samples[J]. PLoS ONE, 2013, 8(3): e58177.
[23] 姜羽, 胡佳莹, 杨立桃. 利用微滴数字PCR分析转基因生物外源基因拷贝数[J]. 农业生物技术学报, 2014, 22(10): 1298-1305.
[24] BURNS M, BURRELL A, FOY C. The applicability of digital PCR for the assessment of detection limits in GMO analysis[J]. European Food Research Technology, 2010, 231: 353-362.
[25] CORBISIER P, BHAT S, PARTIS L. Absolute quantification of genetically modified MON810 maize (Zea mays L.) by digital polymerase chain reaction[J]. Analytical and Bioanalytical Chemistry, 2010, 396: 2143-2150.
[26] DEMEKE T, GRAFENHAN T, HOLIGROSKI M, et al. Assessment of droplet digital PCR for absolute quantification of genetically engineered OXY235 canola and DP305423 soybean samples[J]. Food Control, 2014, 46: 470-474.
[27] FU W, ZHU P, WANG C, et al. A highly sensitive and specif i c method for the screening detection of genetically modif i ed organisms based on digital PCR without pretreatment[J]. Scientif i c Reports, 2015, 5: 12715.
[28] DOBNIK D, STEBIH D, BLEJEC A, et al. Multiplex quantif i cation of four DNA targets in one reaction with Bio-Rad droplet digital PCR system for GMO detection[J]. Scientif i c Reports, 2016, 6: 35451.
[29] GERDES L, IWOBI A, BUSCH U, et al. Optimization of digital droplet polymerase chain reaction for quantification of genetically modified organisms[J]. Biomolecular Detection and Quantification, 2016, 7: 9-20. [30] ZHANG D, CORLET A, FOUILLOUX S. Impact of genetic structure on haploid genome-based quantification of genetically modified DNA:theoretical consideration, experimental data in MON810 maize kernels (Zea mays L.) and some practical applications[J]. Transgenic Research, 2008, 17: 393-402.
Quantitative Detection of Transgenic Maize Event VCO-01981-5 with Droplet Digital PCR
ZHANG Jialing1, PAN Guang2, ZHANG Guiming2, CHENG Yinghui2, XIANG Caiyu2, CHEN Zhinan3, XIE Zhongwen1,*, LING Xingyuan2,*
(1. State Key Laboratory of Tea Plant Biology and Utilization, College of Tea and Food Science and Technology, Anhui Agricultural University, Hefei 230036, China; 2. Animal and Plant Inspection and Quarantine Technology Center, Shenzhen Entry-Exit Inspection and Quarantine Bureau, Shenzhen 518045, China; 3. Shenzhen Academy of Inspection and Quarantine, Shenzhen5 18010, China)
Abstract:In this paper, a duplex droplet digital polymerase chain reaction (ddPCR) method was developed to detect the content of unauthorized genetically modified (GM) maize event VC O-01981-5 based on a QX100 ddPCR platform. Towards this goal, PCR primers and TaqMan probes were designed based on the maize endogenous gene hmg and VCO-01981-5 event specific boundary sequences, and the probes were labeled with different chromophores so that the two targets could be detected in one single droplet. Analysis of the specificity of the method showed that both endogenous gene hmg and foreign sequence could be detected only in event VCO-01981-5. Moreover, the sensitivity, linearity and accuracy were evaluated and it was found that when relative standard deviation (RSD) was 25% or less,5 copies of boundary sequences and4 copies of gene hmg could be quantitatively detected, and the quantitative results (target copy) were highly correlated with the amount of DNA template up to 50 ng (R2≥ 0.99), with an average error of less than 10%. All the above results indicated that the established method in this study was very specific, stable, accurate and sensitive, and had a wide quantitative range in quantitative analysis of GM maize VCO-01981-5. Therefore, this method can be applied practically in quantitatively determining this GM maize event in imported and exported agricultural product and foods. Besides, this reported method can be used as a reference to establish a quantitative method for the detection of other GM maize events and GM events of other plants.
Key words:transgenic maize; event VCO-01981-5; droplet digital PCR; quantitative detection
DOI:47,ebook=254
DOI:10.7506/spkx1002-6630-201712038
中图分类号:S513
文献标志码:A
文章编号:1002-6630(2017)12-0246-07
收稿日期:2016-07-26
基金项目:公益性行业(质检)科研专项(201410014-2);深圳市基础研究计划项目(JC201105190969A);国家质检总局科技司项目(2009IK257)
作者简介:张佳玲(1991—),女,硕士研究生,研究方向为转基因产品检测技术。E-mail:1030798546@qq.com
*通信作者:谢忠稳(1962—),男,教授,博士,研究方向为营养与代谢生物学。E-mail:zhongwenxie@ahau.edu.cn凌杏园(1964—),男,研究员,博士,研究方向为植物基因工程和分子生物学。E-mail:lxy6421@qq.com
引文格式:张佳玲, 潘广, 章桂明, 等. 微滴式数字PCR定量检测转基因玉米品系VCO-01981-5[J]. 食品科学, 2017, 38(12):246-252. DOI:10.7506/spkx1002-6630-201712038. http://www.spkx.net.cn
ZHANG Jialing, PAN Guang, ZHANG Guiming, et al. Quantitative detection of transgenic maize event VCO-01981-5 with droplet digital PCR[J]. Food Science, 2017, 38(12): 246-252. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201712038. http://www.spkx.net.cn