微滴式数字PCR技术定量检测发酵乳中金黄色葡萄球菌
周 巍1,李月华1,孙 勇2,李永波1,张 涛1,刘 琼1,张 岩1,*,王丽霞1,*
(1.河北省食品检验研究院,河北省食品安全重点实验室,河北 石家庄 050071;2.北京食品科学研究院,中国肉类食品综合研究中心,北京 100068)
摘 要:基于微滴式数字聚合酶链式反应(droplet digital polymerase chain reaction,ddPCR)技术,建立发酵乳中金黄色葡萄球菌定量检测方法。以金黄色葡萄球菌nuc基因为目的片段设计特异性的引物和探针,优化反应体系,通过活菌提取和ddPCR方法对靶标基因的检测特异性和灵敏度进行实验,并对定量结果进行分析。本研究建立了发酵乳中ddPCR技术定量检测金黄色葡萄球菌的方法,检测特异性良好,检测灵敏度为3.3×101CFU/g,定量的偏差率为+10.18%,证明了ddPCR用于绝对定量检测的可行性,为其他食品污染菌和致病菌标准化控制体系提供有益的示范作用。
关键词:微滴式数字聚合酶链式反应技术;发酵乳;定量检测;金黄色葡萄球菌
发酵乳中污染菌和致病菌的质量控制需要精准的检测技术体系作为支撑,发酵乳因其营养丰富的特性能够满足污染菌和致病菌的生长需求,单一的对终产品进行检测容易造成原辅料等生产资源的浪费,因而增加了生产成本[1-5]。因此,不同生产环节的污染菌和致病菌数量的精准检测是进行过程质控的关键[6-8]。
由林彩琴等[9]提出的数字聚合酶链式反应(digital polymerase chain reaction,dPCR)作为第3代PCR的技术代表[10],是把一个样本的反应体系均匀分配到大量反应单元中,每个反应单元中不包含或包含一个到多个目的核酸序列,独立地进行PCR扩增,检测每个反应单元的荧光信号,最终根据泊松分布以及荧光信号阳性的反应单元数量占所有反应单元的比例来计算目的核酸序列的拷贝数,dPCR中荧光信号的产生过程基本与实时定量PCR相同[11]。微滴式dPCR(droplet dPCR,ddPCR)技术是利用油包水原理完成微滴信号检测,具有应用范围广、操作简便等特点[12-15],目前,该技术已经在稀有突变检测、动植物源性成分检测、病原菌检测、基因拷贝数变异分析和复杂样本基因表达检测等方面广泛应用[16-25]。
本实验旨在将ddPCR技术应用到发酵乳中常见污染菌和致病菌的检测体系构建中,利用死细菌去除方法,结合ddPCR技术的精准定量,开发出发酵乳中污染菌和致病菌的检测方法,为发酵乳生产企业的过程控制提供必要的技术保障,也为ddPCR技术在其他污染菌和致病菌检测中推广应用提供理论支持。
1 材料与方法
1.1 材料与试剂
发酵乳 市购;实验采用菌株详见表1。所有菌株采购到货后首先用各自的指示培养基进行活化,在适宜的温度条件下培养,转接传代3 次后备用。
表1 实验用菌株
Table 1 Strains tested in this study
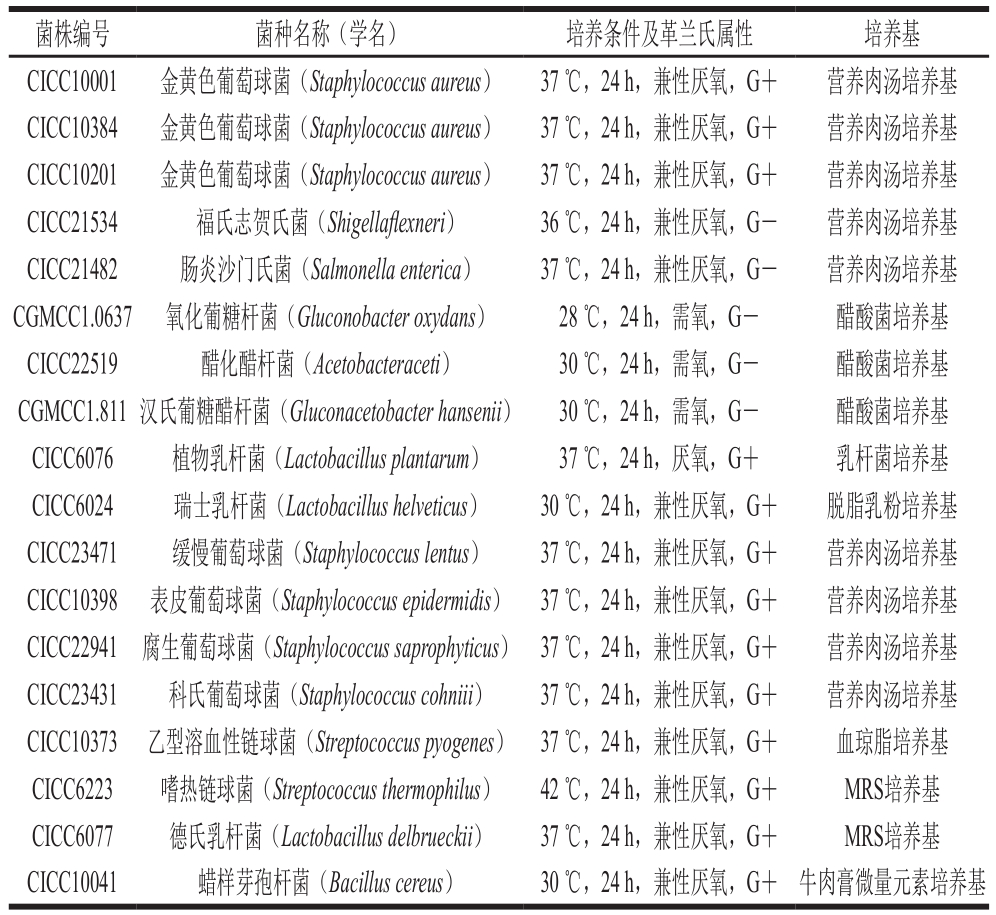
菌株编号菌种名称(学名)培养条件及革兰氏属性培养基CICC10001金黄色葡萄球菌(Staphylococcus aureus)37 ℃,24 h,兼性厌氧,G+营养肉汤培养基CICC10384金黄色葡萄球菌(Staphylococcus aureus)37 ℃,24 h,兼性厌氧,G+营养肉汤培养基CICC10201金黄色葡萄球菌(Staphylococcus aureus)37 ℃,24 h,兼性厌氧,G+营养肉汤培养基CICC21534福氏志贺氏菌(Shigellaflexneri)36 ℃,24 h,兼性厌氧,G-营养肉汤培养基CICC21482肠炎沙门氏菌(Salmonella enterica)37 ℃,24 h,兼性厌氧,G-营养肉汤培养基CGMCC1.0637氧化葡糖杆菌(Gluconobacter oxydans)28 ℃,24 h,需氧,G-醋酸菌培养基CICC22519醋化醋杆菌(Acetobacteraceti)30 ℃,24 h,需氧,G-醋酸菌培养基CGMCC1.811汉氏葡糖醋杆菌(Gluconacetobacter hansenii)30 ℃,24 h,需氧,G-醋酸菌培养基CICC6076植物乳杆菌(Lactobacillus plantarum)37 ℃,24 h,厌氧,G+乳杆菌培养基CICC6024瑞士乳杆菌(Lactobacillus helveticus)30 ℃,24 h,兼性厌氧,G+脱脂乳粉培养基CICC23471缓慢葡萄球菌(Staphylococcus lentus)37 ℃,24 h,兼性厌氧,G+营养肉汤培养基CICC10398表皮葡萄球菌(Staphylococcus epidermidis)37 ℃,24 h,兼性厌氧,G+营养肉汤培养基CICC22941腐生葡萄球菌(Staphylococcus saprophyticus)37 ℃,24 h,兼性厌氧,G+营养肉汤培养基CICC23431科氏葡萄球菌(Staphylococcus cohniii)37 ℃,24 h,兼性厌氧,G+营养肉汤培养基CICC10373乙型溶血性链球菌(Streptococcus pyogenes)37 ℃,24 h,兼性厌氧,G+血琼脂培养基CICC6223嗜热链球菌(Streptococcus thermophilus)42 ℃,24 h,兼性厌氧,G+MRS培养基CICC6077德氏乳杆菌(Lactobacillus delbrueckii)37 ℃,24 h,兼性厌氧,G+MRS培养基CICC10041蜡样芽孢杆菌(Bacillus cereus)30 ℃,24 h,兼性厌氧,G+牛肉膏微量元素培养基
2×探针法数字PCR预混液、微滴发生专用油、微滴分析专用油 德国Bio-Rad公司;细菌处理液(Reagent D)、磁珠法提取细菌DNA试剂盒 美国Biotecon Diagnostics公司;引物(正向引物,反向引物)、探针由上海生物工程公司合成。
1.2 仪器与设备
QX200 Droplet Reader、DG8 cartridge微滴生成卡、Holder微滴发生器、PCR基因扩增仪 伯乐生命医学产品有限公司;Nanodrop 2000超微量分光光度计、711全自动DNA提取仪 美国Thermo公司;1-14台式高速离心机美国Sigma公司。
1.3 方法
1.3.1 活菌基因组的提取
1.3.1.1 Reagent D处理细菌培养液
将传代3 次后的细菌培养液充分摇匀,吸取100 μL细菌培养液加入到2 mL的灭菌透明离心管中;将Reagent D从-20 ℃取出,室温25 ℃放置融化,取400 μL的Reagent D溶液加入到上述离心管中,室温条件下避光培养5 min;将离心管置于低温离心管架中,放在500 W卤素灯相距15~20 cm的正下方,曝光培养5 min后,8 000 r/min离心5 min;用移液器小心去除上清液,向离心管中加入100 μL无菌去离子水充分混匀,此菌悬液即可直接用于基因组的提取。
1.3.1.2 Food Proof磁珠提取法
1)取1 mL Reagent D处理后细菌培养液加入至2 mL试管中,8 000 r/min离心5 min,弃上清液,添加700 µL的裂解缓冲液以及80 µL的蛋白酶K,65 ℃振荡水浴30 min,12 000×g离心5 min,准备好上清液用于DNA提取。
2)准备仪器需要的加样板、反应结合板、洗涤板Ⅰ、洗涤板Ⅱ、洗涤板Ⅲ、洗脱板,并根据需要使用微量移液器或排式微量移液器将每种板对应的缓冲液添加至需要使用的样品孔。
3)其中反应结合板每个样品孔需添加250 µL的反应结合缓冲液和20 µL的磁珠颗粒且2 种液体需吹打混匀;洗涤板Ⅰ每个样品孔需添加600 µL的洗涤缓冲液Ⅰ;洗涤板Ⅱ每个样品孔需添加800 µL的洗涤缓冲液Ⅱ;洗脱板每个样品孔需添加80 µL的洗脱缓冲液。
4)分别移取第1步前处理离心完毕500 µL裂解液转移至已添加有反应结合缓冲液和磁珠颗粒的反应结合板相应样品孔。
5)打开仪器电源开关,选择所需的应用程序(细菌提取选择MPKⅣ程序)。
6)重复按Start按钮,根据屏幕指示分别将加样板、反应结合板B、洗涤板Ⅰ、洗涤板Ⅱ、洗涤板Ⅲ、洗脱板放置到指定位置。
7)当所有平板装载完毕后,点击Start按钮仪器开始运行核酸自动提取程序,大约需要35~40 min。
8)程序运行完毕后,根据仪器屏幕指示开始逐步卸载已经完成核酸提取任务的加样板、反应结合板、洗涤板Ⅰ、洗涤板Ⅱ、洗涤板Ⅲ、洗脱板,注意此时DNA已经溶解于洗脱板可用于后续PCR实验,及时将已经完成核酸提取任务的反应结合板、洗涤板Ⅰ、洗涤板Ⅱ、洗涤板Ⅲ相应样品孔的废液使用微量移液器移除。
9)使用微量移液器将洗脱板上提取的DNA分别转移至干净的离心管中定容至80 μL,可直接用于PCR实验或4~8 ℃保存几天,或-20 ℃长期保存。
1.3.2 引物设计
金黄色葡萄球菌特异性检测基因较少,选择文献报道较多的nuc基因为靶标基因[26-30],从GenBank数据库中公布的已知序列中选取特异性强且相对保守的序列进行引物设计,利用Primer Premier 6.0设计引物探针,并通过Oligo 7.37进行验证,再进行BLAST在线比对后,经实验验证,最终确定上述菌株的引物探针,如表2所示。
表2 引物探针序列
Table 2 Primer and probe sequences used in this study

靶标菌株引物序列5’→3’靶标基因上游引物:GCGATTGATGGTGATACGGTT金黄色葡萄球菌下游引物:AGCCAAGCCTTGACGAACTAAAGC探针:FAM-GGTGTAGAGAAATATGGTCCTGAAGCAAGTGCA-BHQI nuc
1.3.3 单一污染菌和致病菌ddPCR检测方法的建立
ddPCR检测方法:步骤1:配制ddPCR体系,体系为2×探针法数字PCR预混液10 µL,将引物稀释到10 µmol/L,取1.2 µL上下游引物和0.4 μL探针加入体系,加入4.4 µL的模板,最后用灭菌双蒸水补足20 µL。步骤2:将充分混匀的PCR体系转移到微滴发生卡中,并向微滴发生卡中加入70 μL微滴发生专用油,将微滴发生卡放到微滴发生器中进行反应。随后将生成的微滴转移到ddPCR的96 孔中,用封膜仪对96 孔板封膜,准备进行PCR。步骤3:ddPCR的反应程序:95 ℃预变性10 min;94 ℃变性1 min;56 ℃退火45 s;进行40 个循环,98 ℃延伸10 min。步骤4:ddPCR结束后将96 孔板放入QX200 Droplet Reader仪器中,依次录入样品信息,检测开始后仪器按顺序自动识别每个样品的微滴,在微滴读取油的配合下微滴依次通过双色检测器,根据微滴发出的荧光信号的强度判定阳性和阴性结果,并记录每个样品的阳性和阴性的微滴数。信号采集完毕后,采用Quantasoft软件计算出最终结果并以图像的形式给出。
1.3.4 ddPCR检测方法的温度优化
在设计ddPCR扩增的反应程序时,对ddPCR的退火温度进行优化。对金黄色葡萄球菌设置退火约为52~63 ℃的温度梯度ddPCR。根据微滴检测仪生成的结果筛选出最佳反应温度。
1.3.5 ddPCR检测特异性验证
分别提取表1中所列菌株的纯培养物过夜菌液的DNA,将提取的DNA分别稀释到浓度为103拷贝数,按照分别优化好的反应条件进行金黄色葡萄球菌的特异性验证实验。
1.3.6 ddPCR检测灵敏度
发酵乳分别按照国标法检测证实没有被金黄色葡萄球菌污染。将其人工添加到发酵乳中,保证样品中金黄色葡萄球菌的浓度依次为100~106CFU/g,随后按照1.3.1节方法完成污染物中细菌的基因组DNA的提取工作,并用ddPCR方法检测金黄色葡萄球菌污染酸奶的灵敏度。
1.3.7 ddPCR绝对定量
从NCBI上查找出金黄色葡萄球菌nuc相应的靶基因在该菌的全基因组中存在的理论拷贝数,根据ddPCR得出金黄色葡萄球菌的实测拷贝数的结果,计算出20 μL的体系中存在的拷贝数,最终的实测灵敏度和偏差率按公式(1)、(2)计算:
实测灵敏度=20 μL拷贝数×18.18
为了验证ddPCR检测方法的可行性,将不同浓度的金黄色葡萄球菌进行ddPCR检测,分析计数结果与ddPCR检测结果的线性关系。
2 结果与分析
2.1 金黄色葡萄球菌ddPCR法检测温度优化结果
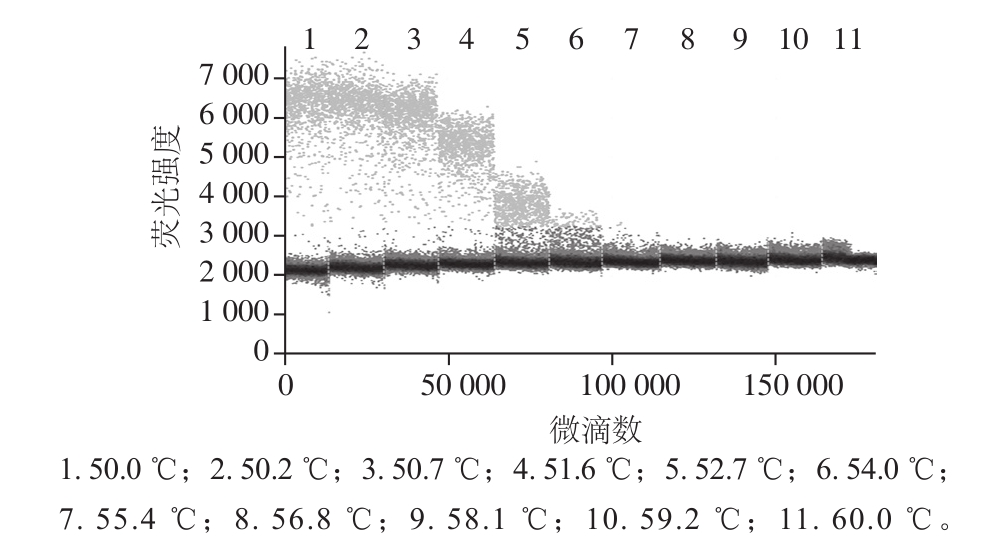
图1 金黄色葡萄球菌ddPCR法检测温度优化结果
Fig. 1 Optimization of ddPCR annealing temperature for detection of Staphyloccocus aureus
设置金黄色葡萄球菌的PCR程序的退火温度为55 ℃,梯度差异为5 ℃,扩增反应结束后,将96 孔板转移到微滴检测仪中进行检测,如图1所示,50.2 ℃时微滴数量和质量都较好,所以金黄色葡萄球菌ddPCR的较适退火温度为50.2 ℃。
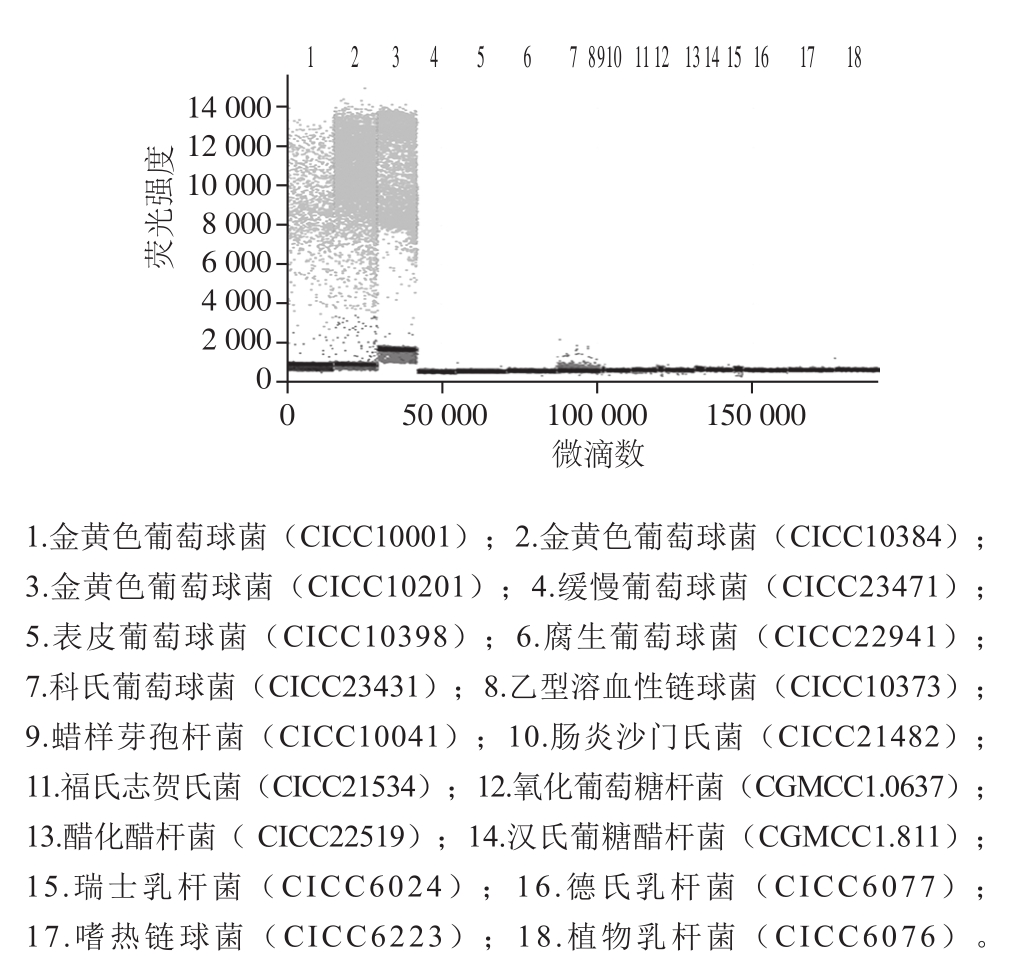
图2 金黄色葡萄球菌ddPCR法检测特异性结果
Fig. 2 Specificity of ddPCR for detection of Staphyloccocus aureus
2.2 金黄色葡萄球菌ddPCR法检测特异性结果将表1所列的供试菌株提取DNA后,用金黄色葡萄球菌的引物进行PCR扩增,随后用微滴检测仪进行检测,如图2所示,3 株金黄色葡萄球菌被检测为阳性微滴信号,而阴性和其他非金黄色葡萄球菌均未出现扩增,表明设计的金黄色葡萄球菌的引物特异性较强。
2.3 金黄色葡萄球菌ddPCR法检测灵敏度结果
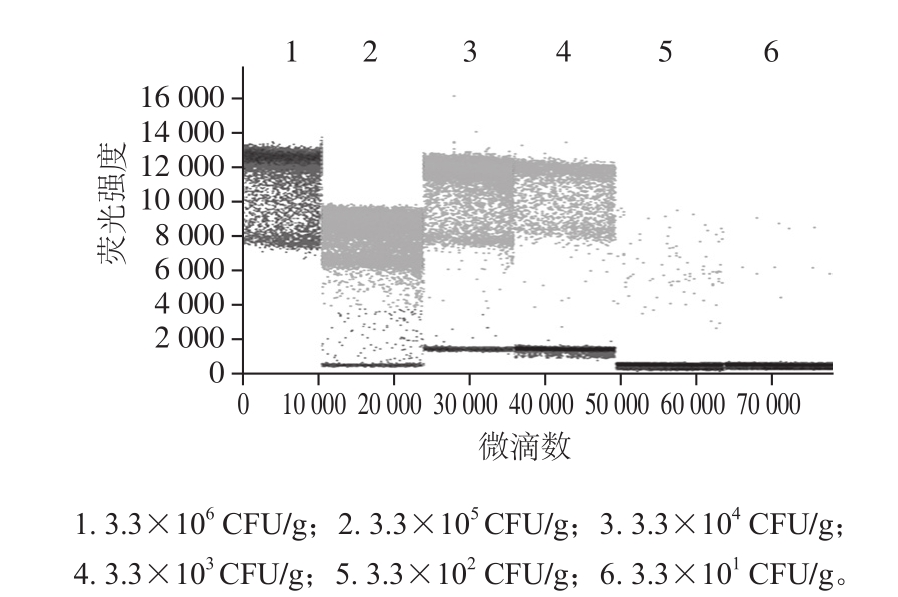
图3 金黄色葡萄球菌ddPCR法检测灵敏度结果
Fig. 3 Sensitivity of detection of Staphylococcus aureus by ddPCR
金黄色葡萄球菌的活菌培养物的平板计数浓度为3.3×106CFU/g,如图3所示,反应1的原始模板浓度太高,无阴性微滴,数据无效。当原始模板浓度为3.3×101CFU/g时,检测出的拷贝数为0.20 copies/μL,所以,ddPCR方法检测金黄色葡萄球菌的计数灵敏度为3.3×101CFU/g。
2.4 ddPCR绝对定量结果
根据金黄色葡萄球菌的理论拷贝数和实际检测结果拷贝数计算出实测灵敏度,如表3所示。比较实测灵敏度和计数灵敏度的差值,当实测灵敏度大于计数灵敏度时,偏差率为正偏差;当实测灵敏度小于计数灵敏度时,偏差率为负偏差。结果显示,金黄色葡萄球菌的实测灵敏度均略高于计数灵敏度,且经过偏差率计算,ddPCR的实测灵敏度与计数灵敏度偏差率均低于25%,保证了ddPCR进行绝对定量检测实际应用的可行性。
表3 拷贝数和实测灵敏度关系
Table 3 Relationship between copy number and measured sensitivity
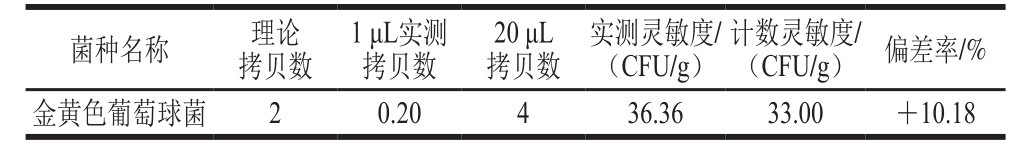
菌种名称理论拷贝数1 μL实测拷贝数20 μL拷贝数实测灵敏度/(CFU/g)计数灵敏度/(CFU/g)偏差率/%金黄色葡萄球菌20.20436.3633.00+10.18
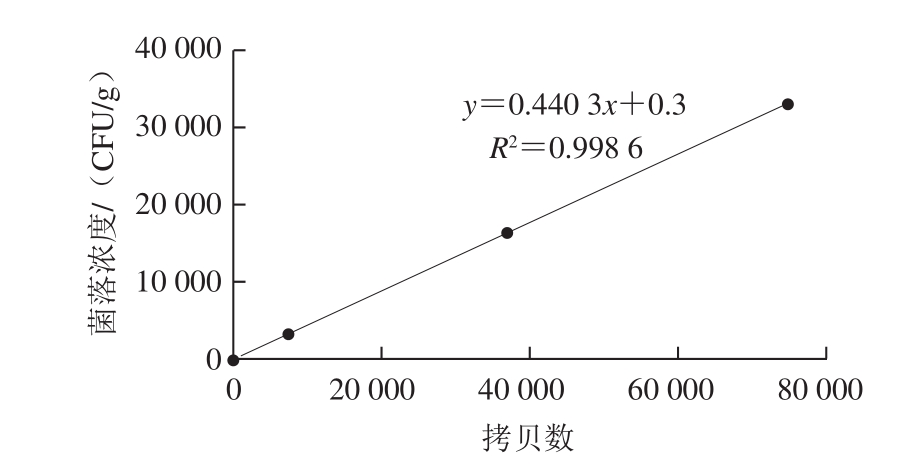
图4 金黄色葡萄球菌ddPCR法检测灵敏度的线性关系
Fig. 4 Standard curve for sensitivity of ddPCR detection of Staphylococcus aureus
为了验证ddPCR检测方法的可行性,将不同浓度的金黄色葡萄球菌进行ddPCR检测,分析计数结果与ddPCR检测结果的线性关系。如图4所示,根据金黄色葡萄球菌的拷贝数和菌落浓度的对应关系所生成的标准曲线显示R2大于0.99,说明ddPCR检测酸奶中污染菌的灵敏度线性关系良好,进一步为ddPCR在绝对定量检测中的实际应用范围提供了保障。
3 讨 论
Reagent D可以有选择性地渗透进入死菌的细胞中,在卤素灯的照射下使Reagent D的光敏基团转化为自由基,并能和DNA分子发生共价交联反应,从而阻断死菌的DNA分子发生PCR扩增。本研究中采用Reagent D处理细菌培养液,并与ddPCR技术相结合从一定程度上排除了死菌对检测方法的影响,实现了定量检测活菌的ddPCR检测。
ddPCR方法的结果判定是根据反应结束后微滴检测仪收集的发出荧光信号的微滴数量,阳性为荧光信号值高于阈值的反应,阴性为荧光信号值低于阈值的反应,然后将阳性微滴数和阴性微滴数用泊松分布公式计算出DNA分子的拷贝数。所以微滴检测仪最后收集到的荧光信号的强弱直接反映出ddPCR的准确性和PCR的扩增效率。影响PCR扩增效率的因素很多,例如引物和探针的浓度,引物的扩增效率,合适的退火温度等。本研究对这些影响因素都进行了优化,最终筛选出每一株菌的最佳反应条件。
微滴发生器将配制好的PCR混合液打散并被包裹在微滴发生专用油中形成微滴,进行PCR扩增。模板DNA的浓度对ddPCR的检测结果有很大的影响。如果模板中的拷贝数低于所生成的微滴数量,每一个微滴中有一个或没有含有目标模板的DNA分子,在PCR完成后,含有目标模板的微滴发生扩增被微滴检测仪检测为阳性,不含目标模板的微滴没有扩增被检测为阴性,根据这2 种微滴的比例通过泊松分布可以准确地实现绝对定量。如果模板中的拷贝数高于或者接近拷贝数,该浓度条件下所有的微滴处于饱和状态,无阴性微滴或者阴性微滴的数量太少,则不能满足泊松分布的条件,绝对定量的结果与真实值相差太大,则无法准确实现ddPCR的绝对定量。
金黄色葡萄球菌对其数值的过程控制显得尤为重要,通过分析计数结果与ddPCR检测结果的线性关系(标准曲线R2均大于0.99),证明了ddPCR用于绝对定量检测的可行性,进而为不同浓度污染菌对终产品造成危害的研究提供了保障,也为ddPCR技术在其他污染菌和致病菌的检测推广提供理论依据。
参考文献:
[1] MURAMALLA T, ARyANA K J. Some low homogenization pressures improve certain probiotic characteristics of yogurt culture bacteria and Lactobacillus acidophilus LA-K[J]. Journal of Dairy Science, 2011, 94(8): 3725-3738. DOI:10.3168/jds.2010-3737.
[2] 杜磊, 张光杰. 酸奶在储藏过程中酸度、pH值、细菌的变化[J]. 河南工业大学学报(自然科学版), 2011, 32(2): 36-40. DOI:10.16433/ j.cnki.issn1673-2383.2011.02.018.
[3] WORKU K F, TEFERA A T, TUJI F A. Comparative analysis of microbial load of commercially prepared and traditionally homemade yoghurt (Ergo) retailed in Addis Ababa[J]. Scientific and Academic, 2015, 5(3): 58-63. DOI:10.5923/j.als.20150503.02.
[4] NWAMAKA N T, CHIKE A. Bacteria population of some commercially prepared yoghurt sold in Enugu State, Eastern Nigeria[J]. African Journal Microbiology Research, 2010, 4(10): 984-988. DOI:10.1016/j.vetmic.2009.12.026.
[5] 曾娟, 赖熙梅, 吴正云. 酸奶制作中金黄色葡萄球菌污染的模拟实验研究[J]. 中国酿造, 2011, 30(9): 122-124. DOI:10.3969/ j.issn.0254-5071.2011.09.033.
[6] SALVATIERRA M, MOLINA A, GAMBOA M D, et al. Evaluation of the effect of probiotic cultures on two different yogurt brands over a known population of Staphylococcus aureus and the production of thermonuclease[J]. Archivos Latinoamericanos De Nutricion, 2004, 54(3): 298-302. DOI:10.9684/j.als.20040321.09.
[7] 陈倩, 骆海朋, 赵春玲, 等. 北京市食品中五种食源性致病菌污染状况调查研究[J]. 中国卫生检验杂志, 2003, 13(5): 570-571. DOI:10.3969/j.issn.1004-8685.2003.05.014.
[8] FARNWORTH E R, MAINVILLE I, DESJARDINS M P, et al. Growth of probiotic bacteria and bifidobacteria in a soy yogurt formulation[J]. International Journal of Food Microbiology, 2007, 116(1): 174-181. DOI:10.1016/j.ijfoodmicro.2006.12.015.
[9] 林彩琴, 姚波. 数字PCR技术进展[J]. 化学进展, 2012, 24(12): 2415-2423. DOI:10.11895/j.issn. 2415-2423.160120.
[10] VOGELSTEIN B, KINZLER K W. Digital PCR[J]. Proceedings of the National Academy Sciences of the United States of America, 1999, 96: 9236-9241. DOI:10.1073/pnas.96.16.9236.
[11] 李春勇. 数字PCR技术原理及应用[J]. 生物技术世界, 2014(11): 10-13. DOI:10.13470/j.cnki.cjpd.2014.41.013.
[12] KOCH H, JESCHKE A, BECKS L. Use of ddPCR in experimental evolution studies[J]. Methods in Ecology and Evolution, 2015, 9(6): 527-532. DOI:10.1111/2041-210X.12467.
[13] MOSER D A, BRAGA L, RASO A, et al. Transgene detection by digital droplet PCR[J]. PloS One, 2014, 9(11): e111781. DOI:10.1371/ journal.pone.0111781.
[14] MORISSET D, MILAVEC M, GRUDEN K, et al. Quantitative analysis of food and feed samples with droplet digital PCR[J]. PloS ONE, 2013, 8(5): e62583. DOI:10.1371/journal.pone.0062583.
[15] MCDERMOTT G P, DO D, LITTERST C M, et al. Multiplexed target detection using DNA-binding dye chemistry in droplet digital PCR[J]. Analytical Chemistry, 2013, 85(23): 11619-11627. DOI:10.1021/ ac403061n.
[16] MORRISON T, HURLEy J, GARCIA J, et al. Nanoliter high throughput quantitative PCR[J]. Nucleic Acids Research, 2006, 34(18): e123. DOI:10.1093/nar/gkl639.
[17] 詹成, 燕丽, 王琳, 等. 数字PCR技术的发展和应用[J]. 复旦学报, 2015, 42(6): 786-789. DOI:10.3969/j.issn.1672-8467.2015.06.017.
[18] HINDSON B J, NESS K D, MASQUELIER D A. High-throughput droplet digital PCR system for absolute quantitation of DNA copy number[J]. Analytical Chemistry, 2011, 83(22): 8604-8610. DOI:10.1021/ac202028g.
[19] CHEN X, WU X, GAN M, et al. Rapid detection of Staphylococcus aureus in dairy and meat foods by combination of capture with silicacoated magnetic nanoparticles and thermophilic helicase-dependent isothermal amplification[J]. Journal of Dairy Science, 2015, 98(3): 1563-1570. DOI:10.3168/jds.2014-8828.
[20] YANG R, PAPARINI A, MONIS P. Comparison of next-generation droplet digital PCR (ddPCR) with quantitative PCR (qPCR) for enumeration of Cryptosporidium oocysts in faecal samples[J]. International Journal for Parasitology, 2014, 44(14): 1105-1113. DOI:10.1016/j.ijpara.2014.08.004.
[21] CHEN W W, BALAJ L, LIAU L M. BEAMing and droplet digital PCR analysis of mutant IDH1 mRNA in glioma patient serum and cerebrospinal fluid extracellular vesicles[J]. Molecular Therapy Nucleic Acids, 2013, 2(7): e109. DOI:10.1038/mtna.2013.28.
[22] HAYDEN R T, GU Z, INGERSOLL J. Comparison of droplet digital PCR to real-time PCR for quantitative detection of cytomegalovirus[J]. Journal of Clinical Microbiology, 2012, 51(2): 540-546. DOI:10.1128/ JCM.02620-12.
[23] KELLEY K, COSMAN A, BELGRADER P, et al. Detection of methicillin-resistant Staphylococcus aureus by a duplex droplet digital PCR assay[J]. Journal of Clinical Microbiology, 2013, 51(7): 2033-2039. DOI:10.1128/JCM.00196-13.
[24] MIOTKE L, LAU B T, RUMMA R T, et al. Correction to high sensitivity detection and quantitationof DNA copy number and single nucleotide variants with single color droplet digital PCR[J]. Analytical Chemistry, 2014, 86(5): 2618-2624. DOI:10.1021/ac403843j.
[25] DREO T, PIRC M, RAMSAK Ž , et al. Optimising droplet digital PCR analysis approaches for detection and quantification of bacteria: a case study of fire blight and potato brown rot[J]. Nucleic Acids Research, 2006, 34(18): e123. DOI:10.1007/s00216-014-8084-1.
[26] VREMERA T, IANCU L S, LOGIGAN C, et al. Optimization of triplex real time PCR for detecting Staphylococcus aureus mecA, pvl and nuc genes[J]. Roumanian Archives of Microbiology and Immunology, 2011, 70(2): 69-73. DOI:10.1348/vr6819431.
[27] LEEUWEN W V, ROORDA L, HENDRIKS W, et al. A nuc-deficient meticillin-resistant Staphylococcus aureus strain[J]. Pathogens and Disease, 2008, 54(2): 157. DOI:10.1111/j.1574-695X.2008.00478.x.
[28] HOEGH S V, SKOV M N, BOYE K, et al. Variations in the Staphylococcus aureus-specific nuc gene can potentially lead to misidentification of meticillin-susceptible and -resistant S. aureus[J]. Journal of Medical Microbiology, 2014, 63(Pt 7): 1020-1022. DOI:10.1099/jmm.0.076638-0.
[29] KIM C H, KHAN M, MORIN D E, et al. Optimization of the PCR for detection of Staphylococcus aureus nuc gene in bovine milk[J]. Journal of Dairy Science, 2001, 84(1): 74-83. DOI:10.3168/jds.S0022-0302(01)74454-2.
[30] SAIFUL A J, MASTURA M, ZARIZAL S, et al. Detection of methicillin-resistant Staphylococcus aureus using mecA/nuc genes and antibiotic susceptibility profile of malaysian clinical isolates[J]. World Journal of Microbiology and Biotechnology, 2006, 22(12): 1289-1294. DOI:10.1007/s11274-006-9174-9.
Quantitative Detection of Staphylococcus aureus in Yogurt by Droplet Digital PCR Assay
ZHOU Wei1, LI Yuehua1, SUN Yong2, LI Yongbo1, ZHANG Tao1, LIU Qiong1, ZHANG Yan1,*, WANG Lixia1,*
(1. Hebei Food Safety Key Laboratory, Hebei Food Inspection and Research Institute, Shijiazhuang 050071, China; 2. Beijing Academy of Food Sciences, China Meat Research Center, Beijing 100068, China)
Abstract:This study aimed to establish a new accurate quantitative method to detect Staphylococcus aureus in yogurt using droplet digital PCR (ddPCR). Specific primers and probes targeting the nuc gene of Staphylococcus aureus were designed, and the PCR reaction system was optimized. The specificity and sensitivity of ddPCR for detecting the target gene were tested using the DNA extracted viable bacterial cells, and the quantitative results were analyzed. The ddPCR method could be useful to quantitatively detect S. aureus in yogurt with good specificity. The limit of detection (LOD) of the method was 3.3 × 101CFU/g, and the quantitative deviation was +10.18%, which proved the feasibility of using ddPCR for absolute quantitation of S. aureus. It will provide a useful demonstration for standardized control systems of other food-contaminating bacteria and foodborne pathogenic bacteria.
Key words:droplet digital PCR; yogurt; detection; Staphylococcus aureus
DOI:10.7506/spkx1002-6630-201716046
中图分类号:TS201.3
文献标志码:A
文章编号:1002-6630(2017)16-0287-05
引文格式:
周巍, 李月华, 孙勇, 等. 微滴式数字PCR技术定量检测发酵乳中金黄色葡萄球菌[J]. 食品科学, 2017, 38(16): 287-291. DOI:10.7506/spkx1002-6630-201716046. http://www.spkx.net.cn
ZHOU Wei, LI Yuehua, SUN Yong, et al. Quantitative detection of Staphylococcus aureus in yogurt by droplet digital PCR assay[J]. Food Science, 2017, 38(16): 287-291. (in Chinese with English abstract)
DOI:10.7506/spkx1002-6630-201716046. http://www.spkx.net.cn
收稿日期:2016-11-04
基金项目:科技部公益性行业科研专项(201510208-08);河北省科技计划项目(16275506D;17275507D);“十二五”国家科技支撑计划项目(2015BAK36B03)
作者简介:周巍(1983—),男,高级工程师,博士,研究方向为食品安全。E-mail:zhouwei0311@163.com
*通信作者:张岩(1979—),男,正高级工程师,博士,研究方向为食品安全。E-mail:snowwinglv@126.com王丽霞(1963—),女,正高级工程师,硕士,研究方向为食品安全检测与风险评估技术。E-mail:lisawang9078@vip.sina.com






