
图1 Unigene GC含量分布图
Fig. 1 GC content distribution of Unigene
聂文强1,吴天祥1,2,*,钟 敏1,芦红云1
(1.贵州大学酿酒与食品工程学院,贵州 贵阳 550025;2.贵州大学明德学院,贵州 贵阳 550025)
摘 要:灰树花多糖具有抗肿瘤、抗HIV、抗氧化等作 用,为探明灰树花多糖合成的遗传基础,采用Illumina高通量测序技术对灰树花转录组进行测序,获得74 575 910 个reads,10.4 G数据量;将测序数据进行de novo拼接后得到18 077 个Unigene。对所得Unigene进行不同数据库比 对,在Nr数据库中有11 651 个Unigene被注释到,其中灰树花转录组与变色栓菌相似序列最多(18.74%);有8 332 个Unigene在GO数据库中注释到,分为细胞组分、分子功能及生物学过程等3 大类59 个分支;与KEGG数据库比对,共有5 200 个Unigene被注释,分属376 条代谢通路。从18 077 个Unigene中共找到1 155 个简单重复序列(simple sequence repeat,SSR)位点,其中单核苷酸重复最高,六核苷酸重复最少,A/T出现频率最高。本研究中在KEGG数据库注释到115 个Unigene与灰树花多糖合成有关,这些Unigene及其注释信息为今后深入开展灰树花多糖代谢途径及相关功能基因等研究提供了依据。
关键词:灰树花;菌丝体;转录组测序;功能注释
Abstract: The exopolysaccharide of Grifola frondosa possesses anti-tumor, anti-HIV, and anti-oxidant effects. In order to investigate the genet ic basis for polysaccharide synthesis by G. frondosa, the transcriptome of G. frondosa was sequenced by high-throughput sequencing technology. A total of 74 575 910 reads and 10.4 G data were generated, which formed 18 077 Unigenes after de novo splicing. A total of 11 651 Unigenes we re annotated in the Nr database. Among them, G. frondosa transcriptome had the most similar sequence (18.74%) to M. tumefaciens. A total of 8 332 Unigenes were annotated in the GO database, which were divided into 3 major categories∶ molecular composition, molecular function and biological processes, including 59 branches. Compared with the KEGG database, 5 200 Unigenes were annotated which belonged to 376 metabolic pathways. A total of 1 155 simple sequence repeat (SSR) loci were found in 18 077 Unigenes, among which the highest repeat was single nucleotide whereas the repetition of hexanucleotide was the least, and the frequency of A/T was the highest. In this s tudy, 115 unigenes which have been annotated in the database of KEGG were related to the biosynthesis of polysaccharide by G. frondosa. These annotated Unigenes and the information about them can lay the foundation for further studies on polysaccharide metabolism pathways and related functional genes in G. frondosa.
Key words: Grifola frondosa; myce lium; transcriptome sequencing; functional annotation
DOI∶10.7506/spkx1002-6630-201720002
灰树花(Grifola frondosa)是一种营养丰富的药食两用真菌。多糖是灰树花的主要活性成分,具有抗肿瘤[1-4]、抗HIV[5]、抗氧化[6-8]、清除自由基[8]、降血糖[9]、促进胶原蛋白合成[10]、提高机体免疫力[11-12]等生物活性。
转录组测序是基于Illumina HiSeq等测序平台,对特定组织或细胞在某个时期转录出来的所有mRNA进行测序。近年来,随着转录组测序技术不断发展和完善,为研究功能基因、生物学性状的分子机制提供了全新的方法[13-14]。Yu Guojun等[15]对灵芝菌丝体进行转录组测序,获得了18 892 个Unigene,其中1 389 个Unigene被京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)注释到,分属16 类代谢途径;Huang Yating等[16]对药用真菌桑黄的菌丝体转录组进行测序分析,获得25 811 个Unigene,并发现23 个候选基因可能与甾醇的生物合成有关;Yang Fang等[17]对白蚁蘑菇进行转录组测序,获得6 494 个Unigene,并发现在白蚁蘑菇中许多已知的酶类和三萜皂苷的生物合成有关;Shu Shaohua等[18]对茯苓的菌丝体和菌核进行转录组测序,分别获得40 939 个Unigene和37 220 个Unigene,并发现在茯苓中萜类化合物的生物合成只能通过甲戊二羟酸(mevalonic acid pathway,MVA)途径;在草菇[19]、牛樟芝[20]、酵母[21]、紫背天葵[22]等多个物种的研究中都应用了转录测序技术。
本课题组前期研究结果表明,在灰树花液体发酵体系中添加天麻提取物或其主要成分均可显著促进灰树花多糖的合成[23-27]。基于天麻提取物等促进灰树花多糖合成的机理尚不明确,本研究以真菌灰树花菌丝体为材料,进行转录组测序,并对所有Unigene进行生物信息学分析,探明灰树花细胞生长过程中的相关基因,为研究天麻提取物促进灰树花多糖的合成的机理、进一步挖掘功能基因及开发分子标记提供科学方法的理论依据。
1.1 材料与试剂
灰树花菌株(Grifola frondosa,菌种编号:51616)中国农业微生物菌种保藏管理中心;Total RNA Extractor(Trizol) 生工生物工程(上海)股份有限公司;Qubit2.0 RNA检测试剂盒、Qubit2.0 DNA检测试剂盒美国Thermo Fisher公司。
1.2 仪器与设备
BXM-30R立式灭菌锅 上海博讯实业有限公司医疗设备厂;SW-CJ-1D净化工作台 苏州净化设备有限公司;TG2-16G低速离心机 上海安亭科学仪器厂;Qubit2.0荧光计 美国Thermo Fisher公司;微型旋涡混合仪 上海沪西分析仪器 厂有限公司;聚合酶链式反应仪美国Bio-Rad公司;电泳仪 北京市六一仪器厂。
1.3 方法
1.3.1 培养基配制
斜面培养基:马铃薯葡萄糖琼脂培养基。
液体种子培养基:葡萄糖30 g/L,酵母膏6 g/L,蛋白胨2 g/L,KH2PO40.5 g/L,MgSO4·7H2O 0.5 g/L;pH值自然。
发酵培养基:葡萄糖50 g/L,酵母膏10 g/L,蛋白胨5 g/L,KH2PO42 g/L,MgSO4·7H2O 2 g/L;pH值自然。
1.3.2 样品制备
于母种试管中挑取黄豆粒大小菌丝块接种于斜面中部,置于25 ℃恒温培养箱中培养至菌丝长满整个斜面。在培养好的斜面菌种切取蚕豆大小于液体培养基中,250 mL三角瓶装液量为100 mL,置于25 ℃、150 r/min摇床培养7d。按10%的接种量,用移液枪量取液体种子于发酵培养基中,250 mL三角瓶装液量为100 mL,置于25 ℃、150 r/min摇床培养12 d,过滤得灰树花菌丝体。
1.3.3 总RNA的提取
总RNA提取方法按照试剂盒说明书进行。利用Qubit 2.0 RNA试剂盒检测RNA浓度及琼脂糖凝胶检测RNA完整性以及基因组污染情况。检测合格的RNA用于后续测序。
1.3.4 转录组测序数据处理及分析
将测序得到的原始数据去除3’端测序接头序列,去除融合后的reads尾部质量在20以下的碱基,切除reads中含N部分序列并进行污染评估。利用Trinity软件(版本trinityrnaseq_rr20140717)通过序列之间的Overlap将序列延伸成Contig,再根据序列的Paired-end信息,将Contig连接成转录本序列;对拼接序列去重复,取长度大于200 bp的序列,通过组装的Component从潜在的可变剪接转录本选取组装最长的转录本作为该样品的Unigene序列,最后将Unigene序列进行简单重复序列(simple sequence repeat,SSR)分析并与Nr、Nt、SwissProt、TrEMBL、CDD、PFAM、GO(gene ontology)、KOG、KEGG数据库比对,取相似度大于30%且e小于10-5的注释,获得Unigene的注释信息。
2.1 Unigene拼接
利用Trinity软件对测序得到的原始reads数据进行拼接,获得的contig再次组装得到33 898 个转录本,序列信息达54 167 696 bp(51.66 M),平均长度1 597.96 bp,N50(判断灰树花菌丝体基因组拼接结果优劣的依据)为2 969 bp,N90为718 bp。所得转录本进一步拼接获得18 077 个Unigene,Unigene总长度为23 615 260 bp,平均组装长度为1 306.37 bp;最长Unigene为19 899 bp,最短Unigene为201 bp;N50为2 885 bp,N90为445 bp。其中不小于500 bp和不小于1 000 bp分别占总数的50.33%和36.38%。

图1 Unigene GC含量分布图
Fig. 1 GC content distribution of Unigene
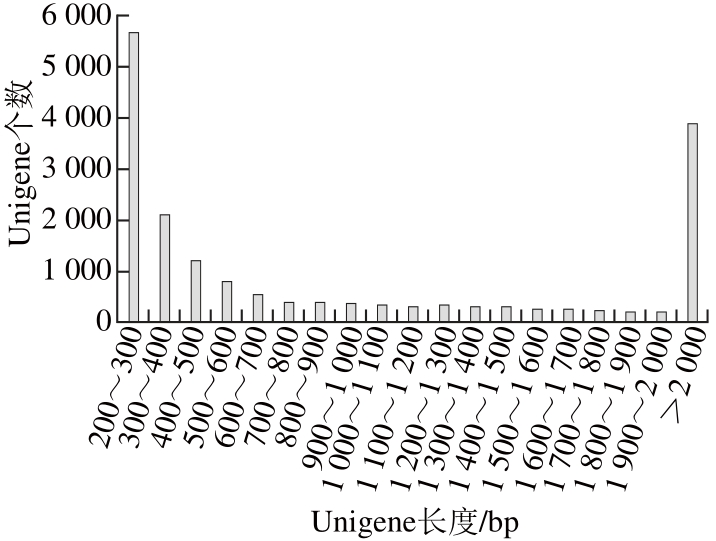
图2 Unigene长度分布图
Fig. 2 Length distribution of Unigene
由图1中Un igene GC含量分布可知,Unigene的GC含量主要分布在40~60 对之间。由图2可以看出,Unigene主要集中在200~300 bp之间和2 000 bp以上,其中在200~300 bp的最多为5 682 个,占总数的31.4%;大于2 000 bp为3 889 个,占总数的21.5%。
2.2 SSR分析
对灰树花菌丝体转录组进行SSR位点分析,筛选标准为:1~6 个核苷酸序列重复次数分别为大于等于10、6、5、5、5和5,两个SSR间最大间隔长度100 bp。从18 077 个Unigene中共找到1 155个SSR位点,占总Unigene的6.39%,其中单核苷酸重复重复最高为780 个,占总重复序列的67.53%,其次是三核苷酸序列,共249 个,占总重复序列的21.56%,最少为五核苷酸序列,只有2 个,仅占0.17%(表1)。在检出的SSR位点中,出现频率最高的是A/T(721 个),其次是C/G(59 个)、AGC/CTG(53 个)、ACG/CGT(51 个)。上述SSR特征分析,有助于开展灰树花及其同属物种的基因组差异分析、通用性标记开发和遗传图谱构建的研究。
表1 灰树花菌丝体SSR不同重复基序分布
Table 1 Distribution of different repeat motifs in SSR analysis

2.3 Unigene的功能注释
表2 Unigene 的功能注释在各数据库分布情况
Table 2 Database distribution of Unigene functional annotation

通过BLAST程序将Unigene序列分别与Nr、Nt、SwissProt、TrEMBL、CDD、PFAM、GO、KOG、KEGG数据库比对。由表2可以看出,18 077 个Unigene全部成功比对,其中12 025 个Unigene至少在一个数据库中被注释到,占总数的66.52%;1 766 个Unigene在所有的数据库中都被注释到,占总数的9.77%。在Nr和TrEMBL数据库中被注释到的Unigene最多,分别为11 651和11 507,占总数的64.45%和63.66%;在数据库Nt中被注释到的最少为3 643 个,仅占20.15%。
2.3.1 Unigene的Nr数据库比对分析
表3 灰树花Unigene的Nr数据库比对分析
Table 3 Blast results of maitake Unigene via Nr

注:*.经比对,Unigene与Nr数据库记录相同的物种。
通过BLAST程序,将 Unigene序列与Nr数据库比对,有11 651 个Unigene与Nr数据库中记录相同,占总Unigene的64.45%。灰树花转录组测序组装的Unigene与变色栓菌的相似性最多,为2 184 个,占18.74%;其次是木质素降解菌,比对上的Unigene为1 321 个,占11.34%。值得注意的是,有24.93%的Unigene属于其他序列,可能包含了灰树花菌丝体与大多数物种不同的、自身特有的基因序列(表3)。
2.3.2 Unigene的KOG数据库比对分析
将灰树花菌丝体转录组所有的Unigene与KOG数据库进行比对,有6 461 个Unigene与KOG数据库比对上,占总Unige ne的35.74%。根据功能划分,本研究中所获灰树花菌丝体转录组功能注释的Unigene可分25 类(图3);其中一般功能预测类基最多,有816 个,占12.63%;其次为翻译后修饰、蛋白质转换、伴侣和翻译,核糖体结构和生物合成相关的基因,分别有763 个和723个,占11.81%和11.19%。而与细胞运动有关只有5 个,与核结构有关的只有6 个。

图3 灰树花Unigene的KOG分类
Fig. 3 KOG functional classifi cation of maitake Unigene
2.3.3 Unigene的GO数据库注释分析
GO分类是国际标准化分类体系,适用于各个物种,能对基因进行限定和描述。对灰树花菌丝体Unigene进行GO功能分析,有8 332个Unigene被注释到,分为生物过程、细胞组成、分子功能3 大类59 个分支(图4)。统计每一类的基因数量发现,在细胞组成类的19 个分支中,涉及本转录组的Unigene最多,为26 962 个;生物过程类次之,为26 051 个;分子功能类最少,为11 573 个。其中代谢过程(5 272 个)、细胞过程(5 714 个)、细胞(6 016 个)、细胞器(4 532 个)、细胞组分(6 014 个)、催化活性(4 651 个)及连接(4 742 个)功能组中涉及的Unigene较多,病毒(3 个)、病毒组分(3 个)、通道调节活性(2 个)、金属伴侣活性(6 个)、受体调节活性(1 个)、蛋白标签(3 个)及转录调节因子活性(7 个)功能组涉及的Unigene较少(表4)。
表4 灰树花菌丝体转录组GO功能分类的Unigene分布
T
Taabbllee 44 MMaaiittaakkee UUnniiggeennee ddiissttrriibbuuttiioonn iinn ggeennee oonnttoollooggyy

2.3.4 Unigene的KEGG数据库注释分析
KEGG数据库能够系统分析基因产物在细胞中的代谢途径以及功能。将灰树花菌丝体的Unigene注释到KEGG数据库进行代谢分类分析,有5 200个Unigene被注释到,占总Unigene的28.77%。被注释到的通路有376 条,其中核糖体(400 个)、碳代谢(313 个)、氨基酸生物合成(245 个)通路涉及的Unigene较多(表5)。灰树花多糖具有抗肿瘤、抗HIV、抗氧化等作用,本研究中在KEGG代谢途径数据库注释到115 条Unigene与灰树花多糖合成有关。这些Unigene及其注释信息为今后深入开展灰树花多糖代谢途径及相关功能基因等研究提供了理论支持。
表5 KEGG注释Unigene最多的前20 位代谢通路
Table 5 Top 20 pathways with the largest number of annotated Unigenes in KEGG

2.4 Unigene的CDS预测

图4 灰树花转录组CDS长度分布图
Fig. 4 CDS length distribution for digital transcriptome of maitake
获取Nr数据库最佳比对结果,通过该结果确定Unigene的ORF的读码框,然后根据标准码子表确定其CDS及编码氨基酸序列,未比对上的Unigene通过OrfPredict软件预测其CDS序列。最终得到CDS序列片段18 040 个,其中长度为200 bp最多,为5 522 个;长度为100 bp的次之,为3 198个;长度为1 900 bp的最少,为155 个,序列长度分布见图4。
本研究采用高通量测序技术对灰树花菌丝体进行转录组测序。采用生物信息学软件对测序结果组装得到18 077 个Unigene,在所有的Unigene中共检测到1 155 个SSR位点,并对其进行了特征分析,这为灰树花及其同属物种的基因组差异分析、通用性标记开发和遗传图谱构建的研究提供了依据。
将Unigene与Nr数据库比对,与变色栓菌的相似序列最 多,为2 184 个,在Swissprot数据库中检测到蛋白同源序列有7 777 条;与KOG数据库比对,注释到的6 461 个Unigene在真核生物功能系统中分为25 类,其中一般功能预测类最多;利用GO数据库进行功能注释,可将注释到的8 332个Unigene分为生物过程、细胞组成、分子功能3 大类59 个分支;KEGG通路注释,注释到5 200个Unigene涉及376 条代谢通路,其中有115 个Unigene与灰树花多糖合成有关,这些数据为研究灰树花多糖合成途径提供了依据。
袁卫东等[28]2015年采用高通量测序技术对灰树花子实体进行转录组测序,获得了63 137个Unigene。将Unigene与Nr数据库比对,与变色栓菌的相似序列最多;在GO数据库中注释到3 大类35 个分支,其中最多的是生物过程分支;有27 472 个Unigene在KEGG数据库中被注释到239 条代谢途径。与上述研究结果相比本实验建立的灰树花菌丝体转录组数据库,获得的Unigene相对较少,但Unigene的GO功能分类有生物过程、细胞组成、分子功能3 大类59 个分支,KEGG通路注释,注释到376 条代谢通路。
本研究采用高通量测序技术对灰树花菌丝体进行转录组测序,获得了大量灰树花菌丝体转录组信息,并进行了生物信息学分析,为深入开展灰树花多糖合成机理、灰树花分子标记开发等研究提供了丰富的数据资源。
参考文献:
[1] LEE B C, BAE J T, PYO H B, et al. Biological activities of the polysaccharides produced from submerged culture of the edible basidiomycete Grifola frondosa[J]. Enzyme and Microbial Technology,2003, 32(5): 574-581. DOI:10.1016/S0141-0229(03)00026-7.
[2] MAO G H, REN Y, LI Q, et al. Anti-tumor and immunomodulatory activity of selenium(Se)-polysaccharide from Se-enriched Grifola frondosa[J]. International Journal of Biological Macromolecules, 2015,82∶ 607-613. DOI∶10.1016/j.ijbiomac.2015.10.083.
[3] 徐晓宝. 天麻对灰树花发酵代谢途径的影响机理研究[D]. 贵阳∶贵州大学, 2012∶ 61-71.
[4] 张勇. 麻组分对灰树花发酵的影响及其多糖抑瘤增效的研究[D].贵阳∶ 贵州大学, 2012∶ 58-64.
[5] HIROAKI N, NORIKO K, DOUGLAS S, et al. Effects of Maitake(Grifola frondosa) glucan in HIV-infected patients[J]. Mycoscience,2000, 41(4)∶ 293-295. DOI∶10.1007/BF02463941.
[6] LIN E. Production of exopolysaccharides by submerged mycelial culture of Grifola frondosa TFRI1073 and their antioxidant and antiproliferative activities[J]. World Journal of Microbiology and Biotechnology, 2011, 27(3)∶ 555-561. DOI∶10.1007/s11274-010-0498-1.
[7] CHEN G T, MA X M, LIU S T, et al. Isolation, purification and antioxidant activities of polysaccharides from Grifola frondosa[J].Carbohydrate Polymers, 2012, 89∶ 61-66. DOI∶10.1016/j.carbpol.2012.02.045.
[8] FAN Y N, WU X Y, ZHANG M, et al. Physical characteristics and antioxidant effect of polysaccharides extracted by boiling water and enzymolysis fro m Grifola frondosa[J]. International Journal of Biological Macromolecules, 2011, 48(5)∶ 798-803. DOI∶10.1016/j.ijbiomac.2011.03.013.
[9] TALPUR N A, ECHARD B W, FAN A Y, et al. Antihypertensive and metabolic effects of whole Maitake mushroom powder and its fractions in two rat strains[J]. Molecular & Cellular Biochemistry,2002, 237(1/2)∶ 129. DOI∶10.1023/A∶1016503804742.
[10] KIM J H, BAE J T, SONG M H, et al. Biological activities of fructus arctii fermented with the basidiomycete Grifola frondosa[J]. Archives of Pharmacal Research, 2010, 33(12)∶ 1943-1951. DOI∶10.1007/s12272-010-1209-y.
[11] NORIKO K, TADAHIRO K, HIROAKI N. Stimulation of the natural immune system in normal mice by polysaccharide from maitake mushroom[J]. Mycoscience, 2003, 44(3)∶ 257-261. DOI∶10.1007/s10267-003-0099-y.
[12] DENG G, LIN H, SEIDMAN A, et al. A phase I/II trial of a polysaccharide extract from Grifola frondosa (Maitake mushroom)in breast cancer patients∶ immunological effects[J]. Journal of Cancer Research and Clinical Oncology, 2009, 135(9)∶ 1215-1221.DOI∶10.1007/s00432-009-0562-z.
[13] MARIONI J C, MASON C E, MANE S M, et al. RNA-seq∶ An assessment of technical reproducibility and comparison with gene expression arrays[J]. Genome Research, 2008, 18∶ 1 509-1517.DOI∶10.1101/gr.079558.108.
[14] FULLWOOD M J, WEI C L, LIU E T, et al. Next-generation DNA sequencing of paired-end tags (PET) for transcriptome and genome analyses[J]. Genome Research, 2009, 19∶ 521-532. DOI∶10.1101/gr.074906.107.
[15] YU G J, WANG M, HUANG J, et al. Deep insight into the Ganoderma lucidum by comprehensive analysis of its transcriptome[J]. PLoS ONE, 2012, 7(8)∶ 1-13. DOI∶10.1371/journal.pone.0044031.
[16] HUNAG Y T, WU X Q, JIAN D, et al. De novo transcriptome analysis of a medicinal fungi Phellinus linteus and identification of SSR markers[J]. Biotechnology & Biotechnological Equipment, 2015,29(2)∶ 395-403. DOI∶10.1080/13102818.2015.1008228.
[17] YANG F, XU B, ZHAO S J, et al. De novo sequencing and analysis of the termite mushroom (Termitomyces albuminosus) transcriptome to discover putative genes involved in bioactive component biosynthesis[J]. Journal of Bioscience and Bioengineering, 2012,114(2)∶ 228-231. DOI∶10.1016/j.jbiosc.2012.03.009.
[18] SHU S H, CHEN B, ZHOU M C, et al. De novo sequencing and transcriptome analysis of Wolfiporia cocos to reveal genes related to biosynthesis of triterpenoids[J]. PLoS ONE, 2013, 8(8)∶ 1-10.DOI∶10.1371/journal.pone.0071350.
[19] BAO D P, GONG M, ZHENG H J, et al. Sequencing and comparative analysis of the straw mushroom (Volvariella volvacea) genome[J].PLoS ONE, 2013, 8(3)∶ 1-12. DOI∶10.1371/journal.pone.0058294.
[20] LU M Y, FAN W L, WANG W F, et al. Genomic and transcriptomic anal yses of the medicinal fungus Antrodia cinnamomea for its metabolite biosynthesis and sexual development[J]. PNAS, 2014,111(44)∶ E4743-E4752. DOI∶10.1073/pnas.1417570111.
[21] NAGALAKSHMI U, WANG Z, WAERN K, et al. The transcriptional landscape of the yeast genome defined by RNA sequencing[J].Science, 2008, 3 20∶ 1344-1349. DOI∶10. 1126/science.1158441.
[22] 张少平, 洪建基, 邱珊莲, 等. 紫背天葵高通量转录组测序分析[J]. 园艺学报, 2016, 43(5)∶ 935-946. DOI∶10.16420/j.issn.0513-353x.2016-0140.
[23] XU X B, WU T X, WANG F. The effect of exopolysaccharide biosynthesi s and related enzyme activities of Grifola frondosa by the addition of ethanol extracts from traditional Chinese medicine,Gastrodia tuber[J]. African Journal of Biotechnology, 2012, 11(15)∶3656-3662. DOI∶10.5897/AJB11.3744.
[24] WANG N, WU T X, ZHANG Y, et al. Experimental analysis on the effect of addition of Rhizoma gastrodiae on mycelia and exopolysaccharide productions by submerged culture of Grifola frondosa[J]. African Journal of Biotechnology, 2012, 11(20)∶ 4666-4672. DOI∶10.5897/AJB12.195.
[25] WU C Y, WU T X. Effect of the main ingredients of Rhizoma gastrodiae on mycelial biomass and exopolysaccharide productions by submerged culture of Grifola frondosa[J]. International Journal of Food Science and Technology, 2015, 50∶ 1726-1730. DOI∶10.1111/ijfs.12831.
[26] 吴彩云, 吴天祥, 朱俊杰, 等. 对羟基苯甲醛等3 种天麻成分对灰树花胞外多糖生物合成的影响[J]. 食品科学, 2016, 37(7)∶ 83-87.DOI∶10.7506/spkx1002-6630-201607016.
[27] 朱俊杰, 吴天祥, 吴彩云, 等. 对羟基苯甲醇对灰树花产胞外多糖的影响及其发酵动力学[J]. 食品科学, 2016, 37(19): 13-18.DOI∶10.7506/spkx1002-6630-201619021.
[28] 袁卫东, 陆娜, 陈青, 等. 灰树花子实体转录组测序和分析[J]. 复旦学报(自然科学版), 2015, 54(5)∶ 673-678. DOI∶10.15943/j.cnki.fdxbjns.2015.05.019.
Transcriptome Sequencing and Analysis of Grifola frondosa Mycelia
NIE Wenqiang1, WU Tianxiang1,2,*, ZHONG Min1, LU Hongyun1
(1. School of Liquor and Food Engineering, Guizhou University, Guiyang 550025, China;2. Mingde College of Guizhou University, Guiyang 550025, China)
中图分类号:Q939.99
文献标志码:A
文章编号:1002-6630(2017)20-0006-06
引文格式:
聂文强, 吴天祥, 钟敏, 等. 真菌灰树花菌丝体转录组测序及分析[J]. 食品科学, 2017, 38(20)∶ 6-11. DOI∶10.7506/spkx1002-6630-201720002. http∶//www.spkx.net.cn
NIE Wenqiang, WU Tianxiang, ZHONG Min, et al. T ranscriptome sequencing and analysis of Grifola frondosa mycelia[J].Food Science, 2017, 38(20)∶ 6-11. (in Chinese with English abstract) DOI∶10.7506/spkx1002-6630-201720002. http∶//www.spkx.net.cn
收稿日期:2016-10-24
基金项目:国家自然科学基金地区科学基金项目(31460537)
作者简介:聂文强(1993—),男,硕士研究生,研究方向为生物信息学。E-mail:648462055@qq.com
*通信作者:吴天祥(1965—),男,教授,博士,研究方向为发酵工程和生物转化。E-mail:txwu@gzu.edu.cn