表1 耐药基因、整合子酶基因和16S rRNA引物
Table 1 Primers of antibiotic resistance genes, integron gene and 16S rRNA gene

李 姝1,2,邵 毅2,*,周昌艳2,*,王 华3,4,邢增涛1,5,赵志辉2
(1.上海海洋大学食品学院,上海 201306;2.上海市农业科学院农产品质量标准与检测技术研究所,上海 201403;3.美国俄亥俄州立大学食品科学与技术系,美国 俄亥俄 哥伦布 43210;4.复旦大学生命科学院微生物系,上海 200433;5.上海市农业委员会,上海 200003)
摘 要:为评价抗生素耐药基因通过市售鸡肉产品进行迁移的风险,从上海5 个大型超市和综合市场采集的9 个鸡肉样品和4 个内脏样品中分离对磺胺甲噁唑/甲氧苄啶不敏感的细菌(Sul/Trir),研究其耐药特征,并用Southern杂交对耐药基因进行定位。结果表明,所有样品均分离到了Sul/Trir菌,菌落数为2.0×102~1.0×107CFU/g,占总可培养菌的1.42%~82.00%。297 株Sul/Trir菌的多重耐药现象普遍,耐药基因sul1、sul2、tetA、tetB、tetC、tetE、tetG、tetL、tetM和intI的检出率分别为11.33%、32.81%、6.64%、12.89%、2.73%、6.25%、9.38%、18.75%、39.84%和28.52%。耐药基因的寄主菌包括Escherichia sp.、Pedobacter sp.、Staphylococcus sp.、Enterococcus sp.、Sphingobacterium sp.和Acinetobacter sp.等,大多数的磺胺最小抑菌浓度大于512 μg/mL,其中一株葡萄球菌的质粒上有sul2基因。综上,耐药菌和耐药基因可能通过市售鸡肉及内脏产品进行迁移。
关键词:磺胺耐药菌;耐药基因;鸡肉;内脏
细菌中抗生素耐药性的快速攀升已成为威胁全球健康的一大问题[1],耐药基因通过食物链迁移至人类共生菌是其中一种重要的途径[2]。抗生素的不当使用使得养殖场成为耐药菌产生和增殖的重要场所[3-4],进而,肉产品中的耐药菌可能通过加工、零售等环节进一步污染、扩散[5]。因此,肉产品中残留微生物的抗生素耐药性问题尤其值得关注。在我国,多种禽源致病菌已具有多重耐药性[6-8],但耐药性的发生和迁移机制尚未揭示,亟需探明鸡肉及相关产品中耐药菌的污染特征和耐药基因的迁移风险。
现有的耐药性研究多集中于(条件性)致病菌,关于非致病菌的耐药数据十分有限。数量庞大的非致病菌是耐药基因的供体、受体和中间载体[9],甚至会促进耐药基因的水平迁移[10],在抗生素耐药性的迁移过程中扮演了关键角色。因此,在非致病菌范围内研究抗生素耐药性污染特征,是揭示耐药性发生和迁移机制,进而提出耐药性迁移的控制措施的基础和新途径。本研究以市售鸡肉和内脏产品为对象,研究了非致病磺胺耐药菌的污染水平、耐药特性、耐药基因携带情况及潜在的迁移风险,以期为肉鸡养殖中的抗生素管理、初加工过程的微生物控制提供理论依据。
1.1 材料与试剂
生鲜整鸡、鸡腿、鸡翅和鸡爪共9 份样品以及鸡胗和鸡肠共4 份样品购自上海市奉贤区和闵行区5 个大型超市和综合菜场。样品采集后用冰包保存,3 h内运回实验室进行耐药菌的分离。
脑心浸液肉汤(brain heart infusion broth,BHI)琼脂培养基 英国Oxoid公司;磺胺甲噁唑(sulfamethoxazole,Sul)、甲氧苄啶(trimethoprim,Tri)、四环素(tetracycline,Tet)、强力霉素(doxycycline,Dox)、头孢噻肟(cefotaxime,Ctx)、红霉素(erythromycin,Erm)、环丙沙星(ciprofloxacine,Cip) 美国Sigma公司;真菌抑制剂放线菌酮(cycloheximide,Cyc) 美国Amresco公司;聚合酶链式反应(polymerase chain reaction,PCR)试剂、耐药基因克隆试剂、质粒提取试剂盒 北京全式金生物技术有限公司;DIG DNA Labeling and Detection Kit瑞士罗氏公司。PCR引物由生工生物工程(上海)股份有限公司合成。
1.2 仪器与设备
生物安全柜 美国Thermo公司;恒温培养箱德国Friocell公司;PCR仪、核酸电泳系统 美国Bio-Rad公司;全自动凝胶成像系统 上海复日公司;匀浆机法国Interscience公司。
1.3 方法
1.3.1 磺胺耐药菌的分离、计数和多耐表型的初步分析
根据Wang Hua等[2]的方法从样品中分离对Sul不敏感的细菌并按标准方法计数:无菌条件下将样品剪碎,混匀,准确称量25 g于无菌袋中,用225 mL无菌生理盐水均质10 min,10 倍梯度稀释,每个稀释度取200 μL均匀涂布于含有152 μg/mL Sul、8 μg/mL Tri和100 μg/mL Cyc的BHI琼脂培养基上,或仅含有100 μg/mL Cyc的BHI琼脂培养基上,32 ℃培养48 h,分别进行磺胺甲噁唑/甲氧苄啶耐药菌(Sul/Trir)和总可培养菌的菌落计数[11]。
根据Huang Ying等[12]的方法对分离获得的Sul/Trir菌进行多耐表型的初步分析:每个样品选取多至25 个形态不同的Sul/Trir菌,共256 株,用灭菌牙签接种至含16 μg/mL Tet、10 μg/mL Erm、4 μg/mL Ctx、16 μg/mL Dox或4 μg/mL Cip的BHI平板上,32 ℃培养48 h,记录其在含各种抗生素的平板上的生长情况。
1.3.2 耐药基因的筛查及菌种鉴定
表1 耐药基因、整合子酶基因和16S rRNA引物
Table 1 Primers of antibiotic resistance genes, integron gene and 16S rRNA gene

用pEASY-T3载体克隆9 个耐药基因sul1、sul2、tetA、tetB、tetC、tetE、tetG、tetL、tetM和一类整合子酶基因intI,在256 株Sul/Trir菌中用PCR法筛查上述基因[4],随机选取10%的阳性PCR产物进行测序确认[12]。对含有至少3 个耐药基因的菌株进行后续药敏实验,并进行16S rRNA PCR后送生工生物工程(上海)股份有限公司测序,序列用NCBI BLAST比对初步鉴定菌种。PCR引物见表1。
1.3.3 Sul/Trir菌的Sul/Tri MIC的测定
用美国临床与实验室标准化研究所推荐的微量肉汤稀释法[19]对含有至少3 个耐药基因的Sul/Trir菌进行Sul/Tri的最小抑制浓度(minimum inhibitory concentration,MIC)测定。以BHI培养基替代肉汤培养基[12,17],以葡萄球菌Staphylococcus aureus ATCC 29213作为质控菌株。
1.3.4 耐药基因sul的Southern杂交及含有耐药基因的质粒的转化
含有耐药基因的菌株用碱裂解法提取质粒后,按照DIG DNA Labeling and Detection Kit的操作流程稍加改动后进行耐药基因的Southern杂交[20]。地高辛标记探针制备中,用本实验室构建并测序的sul1-pEASY-T3质粒和sul2-pEASY-T3质粒作为模板,区别于试剂盒操作步骤中的随机标记法。
提取用Southern杂交初步鉴定的含有耐药基因的质粒,用化学转化法转化至大肠杆菌DH5-α感受态细胞中[21],在含有152 μg/mL Sul和8 μg/mL Tri的LB培养基上筛选阳性菌株,并按1.3.3节的方法测定Sul/Tri的MIC。
2.1 磺胺耐药菌计数和多耐表型
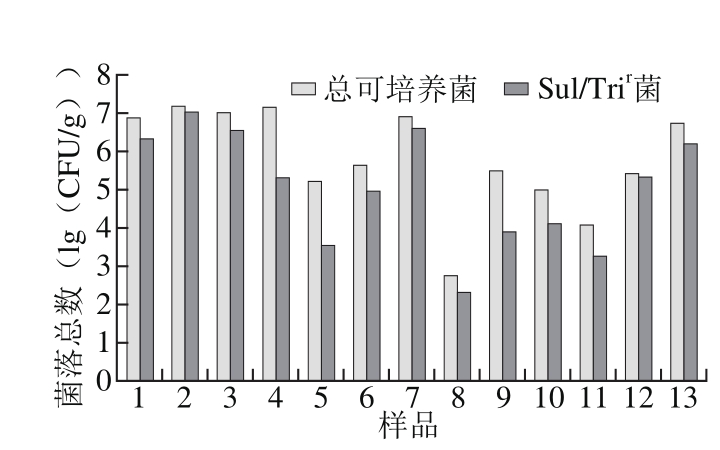
图1 市售鸡肉及内脏产品中可培养细菌总数和Sul/Trir菌总数
Fig. 1 Prevalence of total cultivable bacteria and Sul/Trirbacteria in chicken and giblet samples
1~3.鸡腿;4.鸡翅;5.鸡爪;6~9.生鲜整鸡;10~11.鸡胗;12~13.鸡肠。
13 个样品的总可培养菌数量差异较大,但均分离到了耐药菌Sul/Trir,菌落数为2.0×102~1.0×107CFU/g。69.23%的样品中Sul/Trir菌的数量高于104CFU/g,有3 个鸡腿样品(图1,1~3号样品)和1 个整鸡样品(图1,7号样品)的Sul/Trir菌数量甚至达到了106CFU/g的水平。9 个鸡肉样品中,Sul/Trir菌占总可培养菌的比例为1.42%~70.42%,4 个内脏样品的这一比例为13.74%~82.00%。因此,市售鸡肉及内脏样品中普遍存在Sul/Trir菌。
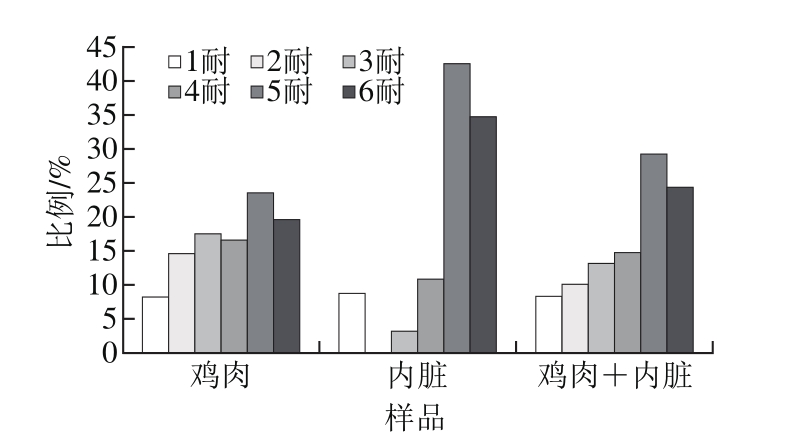
图2 多重耐药菌的比例
Fig. 2 Proportions of multi-drug resistant bacteria
分离到的256 株Sul/Trir菌中,多重耐药现象普遍:91.58%的菌至少对2 种抗生素不敏感,近1/4的菌对全部6 种抗生素均不敏感(图2)。内脏样品中分离的Sul/Trir菌的多重耐药性较鸡肉样品更为普遍,77.17%的菌至少对5 种抗生素不敏感。除Sul/Tri外,256 株菌对Tet、Dox、Erm、Ctx和Cip的耐药率分别为87.21%、68.69%、68.69%、64.31%和48.82%。
2.2 Sul/Trir菌的MIC、耐药基因和菌种鉴定
表2 部分耐药菌株的耐药基因、MIC和菌种
Table 2 MIC and species of Sul/Trirbacteria harboring at least three AR genes
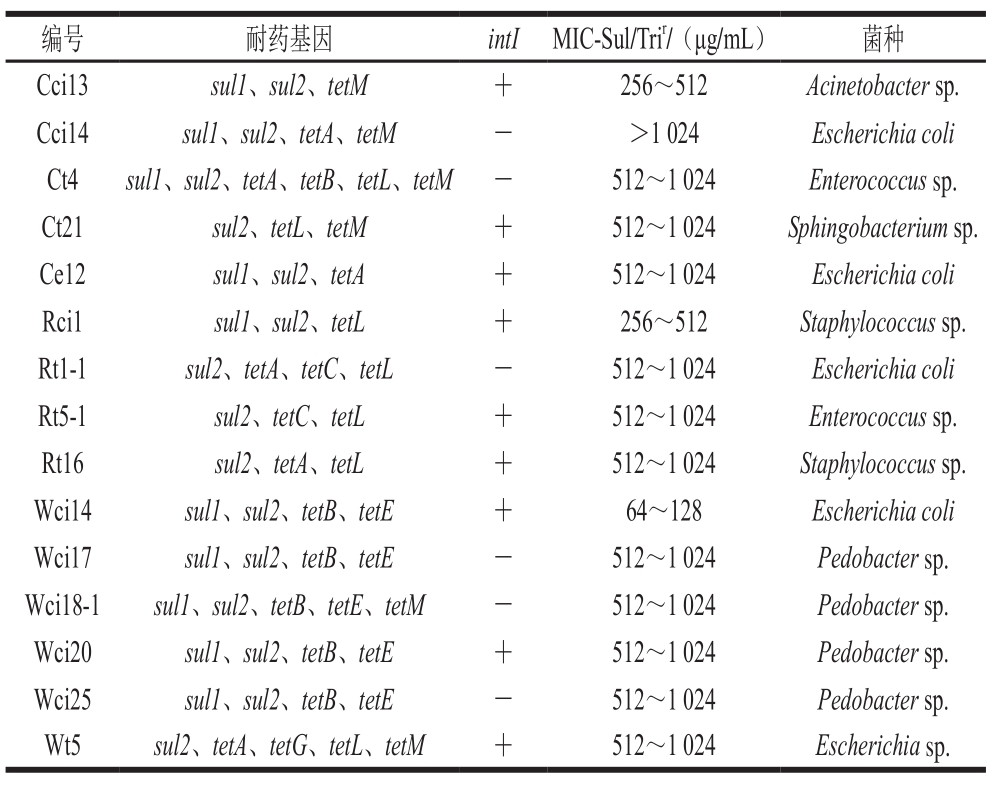
在256株Sul/Trir菌中筛查9 个耐药基因和一类整合子酶基因intI,结果表明,sul1、sul2、tetA、tetB、tetC、tetE、tetG、tetL、tetM和intI的检出率分别为11.33%、32.81%、6.64%、12.89%、2.73%、6.25%、9.38%、18.75%、39.84%和28.52%。同时含有至少3 个耐药基因的菌有15 株(表2),包括5 株埃希氏菌(Escherichia sp.,其中4 株为大肠杆菌)、4 株土地杆菌(Pedobacter sp.)、2 株葡萄球菌(Staphylococcus sp.)、2 株肠球菌(Enterococcus sp.)、1 株鞘氨醇杆菌(Sphingobacterium sp.)和1 株不动杆菌(Acinetobacter sp.)。这些菌株显示了对Sul/Tri较强的耐药性,12 株菌(80%)的磺胺MIC值大于512 μg/mL。Sul/Trir菌的耐药特性表明,市售鸡肉和内脏产品中的细菌包含了多种耐药基因,且对Sul/Tri的耐药程度较高。
2.3 耐药基因的定位
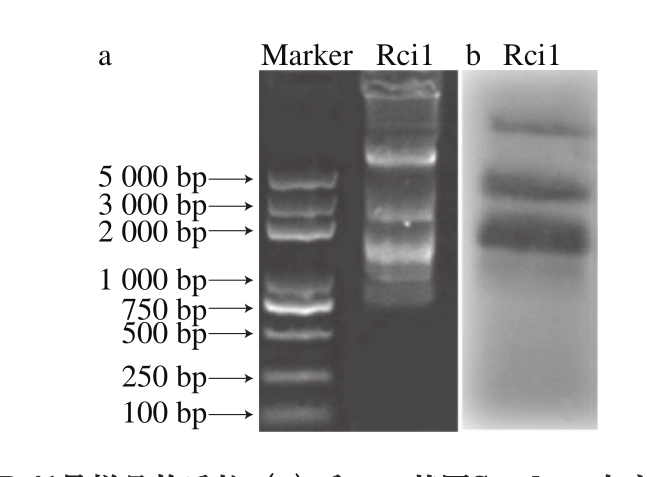
图3 Rci1号样品的质粒(a)和sul2基因Southern杂交结果(b)
Fig. 3 Plasmid of Rci1 (a) and Southern blot analysis of sul2 gene (b)
用Southern杂交研究耐药基因sul1和sul2的定位,结果表明,本研究中的Rci1号样品Sul/Trir菌的质粒上可能存在sul2基因(图3)。提取Rci1号样品Sul/Trir菌的质粒并转化至大肠杆菌DH5-α中,Sul/Tri对阳性菌株的MIC大于256 μg/mL,而对原始DH5-α的MIC小于4 μg/mL。因此认为Rci1号样品的Sul/Trir菌中,质粒上含有sul2基因,存在sul2基因随质粒在不同菌种间迁移扩散的可能性。所有菌株的质粒上未发现sul1基因(结果未显示)。
已有报道表明,我国的生鲜鸡肉中沙门氏菌等致病菌对磺胺类抗生素具有较高的耐药性[22],本研究则表明,生鲜鸡肉和内脏产品中的细菌包含了大量磺胺耐药基因sul1和sul2,具有磺胺耐药表型的细菌数量和占总菌的比例高于同类产品中的四环素耐药菌[17]或其他产品的Sul/Trir菌[20],且对Sul/Tri的耐药程度较高,可见肉鸡养殖和初加工链中细菌的磺胺耐药性普遍存在。尽管鸡肉和内脏产品往往需要高温烹饪才能食用,这一深加工过程能基本消除耐药菌的威胁,但从养殖、宰杀、零售到消费者初加工的整个过程中,耐药菌仍有可能通过直接接触[5]等方式进行扩散、威胁人类健康。因此探明Sul/Trir菌的产生和留存机制及耐药性向人类的迁移潜势,是确保鸡肉产品安全性的重要基础。本研究的鸡肉和内脏样品中Sul/Trir菌普遍存在,并从中筛查到了携带耐药基因的质粒,但16 种磺胺类抗生素[23]的检出率和残留量均很低(数据未显示),说明鸡肉和内脏中Sul/Trir菌的稳定存活并不依赖于抗生素筛选压,因此,限制肉鸡养殖中抗生素的使用并不一定减少肉鸡产品中微生物的耐药性[24],其耐药机制值得深入研究。除sul基因外,样品中还发现了多种tet基因,tetM和tetL的检出率较高,与前人研究结果相似[25-26],因此,鸡肉产品中四环素耐药菌的污染特征也需要全面地评估。
近年来,由接合性质粒、整合子和转座子等介导的水平基因转移(horizontal gene transmission,HGT)被多次证明是耐药基因迁移扩散的重要机制之一[27-28],HGT与耐药基因池的大小、耐药基因供体/受体的遗传背景是否相近等均有关系[29],因此,在数量上远超致病菌且遗传背景多样化的非致病菌中,耐药基因的含量和迁移频率可能更高。本研究分离获得的Sul/Trir菌中检出了大量耐药基因,说明市售鸡肉和内脏产品上的微生物曾经可能并且未来也可能进一步涉及耐药基因的水平迁移;在其中一株对Sul/Tri具有耐药性的葡萄球菌中,发现耐药基因sul2位于质粒上,且该质粒存在向其他细菌进行水平迁移的潜在风险。此外,有证据表明,肠球菌在HGT中的敏感性较高,在耐药基因在微生物系统内的迁移中扮演了重要的角色[30]。而本研究在市售鸡肉和鸡肠样品中均分离到了含有多个耐药基因的肠球菌,也指示了耐药基因发生迁移的风险性。
综上,市售鸡肉及内脏样品中Sul/Trir菌分布广泛,普遍具有多重耐药性,且含有大量耐药基因,存在耐药基因随质粒在细菌间水平迁移的潜在风险。需要深入研究鸡肉产品中Sul/Trir菌存在并维持的机制,评估磺胺耐药基因向人类共生菌迁移的风险。
参考文献:
[1] CENTNER T J. Efforts to slacken antibiotic resistance: labeling meat products from animals raised without antibiotics in the United States[J]. Science of the Total Environment, 2016, 563: 1088-1094.DOI:10.1016/j.scitotenv.2016.05.082.
[2] WANG Hua, MANUZON M, LEHMAN M, et al. Food commensal microbes as a potentially important avenue in transmitting antibiotic resistance genes[J]. FEMS Microbiology Letters, 2006, 254(2): 226-231. DOI:10.1111/j.1574-6968.2005.00030.x.
[3] ZHANG L, HUANG Y, ZHOU Y, et al. Antibiotic administration routes significantly influence the levels of antibiotic resistance in gut microbiota[J]. Antimicrobial Agents & Chemotherapy, 2013, 57(8):3659-3666. DOI:10.1128/AAC.00670-13.
[4] ZHU Y G, JOHNSON T A, SU J Q, et al. Diverse and abundant antibiotic resistance genes in Chinese swine farms[J]. Proceedings of the National Academy of Sciences, 2013, 110(9): 3435-3440.DOI:10.1073/pnas.1222743110.
[5] TAN S L, LEE H Y, MAHYUDIN N A. Antimicrobial resistance of Escherichia coli, and Staphylococcus aureus, isolated from food handler’s hands[J]. Food Control, 2014, 44(3): 203-207. DOI:10.1016/j.foodcont.2014.04.008.
[6] 赖海梅, 刘书亮, 邹立扣, 等. 肉鸡屠宰场多重耐药沙门氏菌Ⅰ类整合子与磺胺类耐药基因(sul1, sul2和sul3)的检测[J]. 食品科学, 2014,35(24): 178-183. DOI:10.7506/spkx1002-6630-201424034.
[7] 孙桂霞, 徐世文. 鸡源空肠弯曲菌调查与耐药性分析[J].东北农业大学学报, 2015, 46(9): 64-68. DOI:10.3969/j.issn.1005-9369.2015.09.010.
[8] ZHANG P, SHEN Z Q, ZHANG C P, et al. Surveillance of antimicrobial resistance among Escherichia coli, from chicken and swine, China, 2008—2015[J]. Veterinary Microbiology, 2017, 203: 49-55. DOI:10.1016/j.vetmic.2017.02.008.
[9] ANDREMONT A. Commensal flora may play key role in spreading antibiotic resistance[J]. ASM News, 2003, 69(2): 601-607.
[10] LUO H L, WAN K, WANG H H. High-frequency conjugation system facilitates biofilm formation and pAM 1 transmission by Lactococcus lactis[J]. Applied and Environmental Microbiology, 2005, 71(6): 2970-2978. DOI:10.1128/AEM.71.6.2970-2978.2005.
[11] 国家卫生和计划生育委员会, 国家食品药品监督管理总局. 食品微生物学检验 菌落总数测定: GB 4789.2—2016[S]. 北京: 中国标准出版社, 2016: 2-4.
[12] HUANG Ying, ZHANG Lu, TIU L, et al. Characterization of antibiotic resistance in commensal bacteria from an aquaculture ecosystem[J].Frontiers in Microbiology, 2015, 6: 914-919. DOI:10.3389/fmicb.2015.00914.
[13] KERRN M B, KLEMMENSEN T, FRIMODT M N, et al.Susceptibility of Danish Escherichia coli strains isolated from urinary tract infections and bacteraemia, and distribution of sul genes conferring sulphonamide resistance[J]. Journal of Antimicrobial Chemotherapy, 2002, 50(4): 513-516. DOI:10.1093/jac/dkf164.
[14] ZHANG L, KINKELAAR D, HUANG Y, et al. Acquired antibiotic resistance: are we born with it?[J]. Applied and Environmental Microbiology, 2011, 77(20): 7134-7141. DOI:10.1128/AEM.05087-11.
[15] CHEN S, ZHAO S H, MCDERMOTT P F, et al. A DNA microarray for identification of virulence and antimicrobial resistance genes in Salmonella serovars and Escherichia coli[J]. Molecular & Cellular Probes, 2005, 19(3): 195-201. DOI:10.1016/j.mcp.2004.11.008.
[16] 欧阳本. 动物源沙门氏菌耐药基因及毒力岛基因mgtC和sopB的检测[D]. 合肥: 安徽农业大学, 2013: 21-22.
[17] LI X J, WANG H H. Tetracycline resistance associated with commensal bacteria from representative ready-to-consume deli and restaurant foods[J]. Journal of Food Protection, 2010, 73(10): 1841-1848. DOI:10.4315/0362-028X-73.10.1841.
[18] LI X H, SHI L, YANG W Q, et al. New array of aacA4-catB3-dfrA1 gene cassettes and a noncoding cassette from a class-1-integronpositive clinical strain of Pseudomonas aeruginosa[J]. Antimicrobial Agents & Chemotherapy, 2006, 50(6): 2278-2279. DOI:10.1111/j.1365-2621.2006.01457.x.
[19] COCKERILL F R. Performance standards for antimicrobial susceptibility testing: twenty-third informational supplement[M].America: Clinical and Laboratory Standards Institute, 2013: 34-42.
[20] YE L, LU Z, LI X H, et al. Antibiotic-resistant bacteria associated with retail aquaculture products from Guangzhou, China[J]. Journal of Food Protection, 2013, 76(2): 295-301. DOI:10.4315/0362-028X.JFP-12-288.
[21] 叶蕾. 广州市水产养殖品中耐药共生菌分布及耐药基因传播机制的研究[D]. 广州: 华南理工大学, 2012: 70.
[22] LI K T, YE S Y, ALALI W Q, et al. Antimicrobial susceptibility,virulence gene and pulsed-field gel electrophoresis profiles of Salmonella enterica serovar Typhimurium recovered from retail raw chickens, China[J]. Food Control, 2017, 72: 36-42. DOI:10.1016/j.foodcont.2016.07.032.
[23] 许旭, 耿丹丹, 迟晓峰, 等. 高效液相色谱光化学在线衍生荧光法检测鸡肉中16 种磺胺类药物残留[J]. 食品科学, 2015, 36(22): 165-168. DOI:10.7506/spkx1002-6630-201522031.
[24] TOUTAIN P L, FERRAN A A, BOUSQUETMELOU A, et al.Veterinary medicine needs new green antimicrobial drugs[J]. Frontiers in Microbiology, 2016, 7(1): 20-35. DOI:10.3389/fmicb.2016.01196.
[25] RUZAUSKAS M, SIUGZDINIENE R, ZYMANTIENE J, et al.Prevalence and characterization of multi-resistant Staphylococcus spp.isolated from poultry liver[J]. Journal of Food Safety, 2016, 36(4):508-514. DOI:10.1111/jfs.12270.
[26] SEPUTIENE V, BOGDAITE A, RUZAUSKAS M, et al. Antibiotic resistance genes and virulence factors in Enterococcus faecium and Enterococcus faecalis from diseased farm animals: pigs, cattle and poultry[J]. Polish Journal of Veterinary Sciences, 2012, 15(3): 431-438.DOI:10.2478/v10181-012-0067-6.
[27] JAYKUS L A, WANG H H, SCHLESINGER L S, et al. Foodborn microbes: shaping the host ecosystems[M]. Washington D. C.: ASM Press, 2009: 267-281.
[28] WINTERSDORFF V C, PENDERS J, NIEKERK M V J, et al.Dissemination of antimicrobial resistance in microbial ecosystems through horizontal gene transfer[J]. Frontiers in Microbiology, 2016,7(110): 173. DOI:10.3389/fmicb.2016.00173.
[29] WANG H H, SCHAFFNER D W. Antibiotic resistance: how much do we know and where do we go from here?[J]. Applied and Environmental Microbiology, 2011, 77(20): 7093-7095. DOI:10.1128/AEM.06565-11.
[30] PALMER K L, KOS V N, GILMORE M S. Horizontal gene transfer and the genomics of enterococcal antibiotic resistance[J]. Current Opinion in Microbiology, 2010, 13(5): 632-639. DOI:10.1016/j.mib.2010.08.004.
Prevalence and Characteristics of Sulfamethoxazole Resistant Bacteria in Retail Chicken Meat and Giblets
LI Shu1,2, SHAO Yi2,*, ZHOU Changyan2,*, WANG Hua3,4, XING Zengtao1,5, ZHAO Zhihui2
(1. College of Food Science and Technology, Shanghai Ocean University, Shanghai 201306, China; 2. Institute for Agri-Food Standards and Testing Technology, Shanghai Academy of Agricultural Sciences, Shanghai 201403, China; 3. Department of Food Science and Technology, The Ohio State University, Columbus 43210, USA; 4. Department of Microbiology, School of Life Sciences, Fudan University, Shanghai 200433, China; 5. Shanghai Agriculture Committee, Shanghai 200003, China)
Abstract:In order to investigate the potential risk of retail chicken products in transmitting antibiotic resistance (AR)encoding genes, sulfamethoxazole/trimethoprim resistant (Sul/Trir) bacteria were isolated from 13 chicken products,including nine chicken meat samples and four giblet samples, purchased from five supermarkets and local markets. The Sul/Trirresistance genes were mapped by Southern blot analysis. Sul/Trirbacteria were detected in all chicken samples.The prevalence of Sul/Trirbacteria ranged from 2.0 × 102CFU/g to 1.0 × 107CFU/g, accounting for 1.42%-82.00% of the total cultivable bacteria. Multi-drug resistance was observed for all 297 Sul/Tririsolates, and the positive rates of AR genes including sul1, sul2, tetA, tetB, tetC, tetE, tetG, tetL, tetM and intI were 11.33%, 32.81%, 6.64%, 12.89%, 2.73%, 6.25%,9.38%, 18.75%, 39.84% and 28.52% respectively. Escherichia sp., Pedobacter sp., Staphylococcus sp., Enterococcus sp.,Sphingobacterium sp. and Acinetobacter sp. were identified as hosts for AR genes. The minimum inhibitory concentration(MIC) of Sul for most isolates was higher than 512 μg/mL. Furthermore, a plasmid harboring sul2 gene was found in one Sul/Tririsolate by Southern blot hybridization. In conclusion, retail chicken products can be a potential avenue transmitting antibiotic resistant bacteria and AR genes.
Key words:sulfamethoxazole/trimethoprim resistant bacteria; antibiotic resistance genes; chicken meat; giblets
DOI:10.7506/spkx1002-6630-201721027
中图分类号:TS201.3
文献标志码:A
文章编号:1002-6630(2017)21-0170-05
收稿日期:2017-03-13
基金项目:国家自然科学基金青年科学基金项目(31401599);上海市科技兴农重点攻关项目(应用基础类)(沪农科攻字(2014)第7-3-6号);上海市青年科技英才扬帆计划项目(14YF1413000);美国外交部国际教育协会美英联合全球创新倡议(GII)项目(GRT00033913)
作者简介:李姝(1990—),女,硕士研究生,研究方向为肉产品耐药菌。E-mail:slishu@163.com
*通信作者:邵毅(1982—),女,副研究员,博士,研究方向为农产品耐药菌。E-mail:shao_saas@163.com周昌艳(1969—),女,研究员,博士,研究方向为农产品质量安全。E-mail:changyanz@sina.com
引文格式:李姝, 邵毅, 周昌艳, 等. 市售鸡肉及内脏中磺胺耐药菌污染特征[J]. 食品科学, 2017, 38(21): 170-174.
DOI:10.7506/spkx1002-6630-201721027. http://www.spkx.net.cn
LI Shu, SHAO Yi, ZHOU Changyan, et al. Prevalence and characteristics of sulfamethoxazole resistant bacteria in retail chicken meat and giblets[J]. Food Science, 2017, 38(21): 170-174. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201721027. http://www.spkx.net.cn