番茄NF-Y转录因子生物信息学分析及其与RIN共同调控果实成熟研究
李 珊,许蕙金兰,朱本忠,罗云波*
(中国农业大学食品科学与营养工程学院,北京 100083)
摘 要:运用生物信息学分析方法,对番茄NF-Y家族59 个成员的结构域和进化关系进行分析,并比较番茄NF-Y基因在番茄野生型AC及rin突变体果实成熟过程中的表达量变化,结合转录因子RIN对NF-Y基因DNA的结合能力,分析其可能的互作关系。研究结果为阐述番茄NF-Y转录因子与RIN共同调控果实成熟的功能提供理论依据。
关键词:番茄果实;NF-Y;RIN;生物信息学分析
果实成熟是一个复杂的动态过程,涉及果实质构、色泽和风味物质等变化[1]。在这个过程中,许多因子通过调控成熟相关基因的表达,从而发挥了对果实成熟过程的重要调控作用。其中,相关转录因子就是其中很重要的一类调控因子[2]。目前,已经有许多家族的转录因子被研究发现参与了果实的成熟调控过程,如M ADS-box、M YB、AP2/ERF以及SBP/SPL等[2]。在众多转录因子家族中,MADS-box转录因子RIN是果实成熟的关键调控因子之一,因为在RIN转录因子功能缺失的番茄rin突变体中,许多成熟相关的途径受到影响,比如果实乙烯合成受到抑制、果实不能正常软化,以及色泽的形成受到影响,果实的成熟过程无法正常启动,最终导致果实严重不成熟的表型[3-4]。近些年的研究发现,转录因子RIN通过调控许多成熟相关的基因影响果实的成熟进程,比如成熟相关的转录因子基因,包括NOR、HB1、CNR和TDR4等[3];乙烯合成与代谢相关的基因,包括ACS2、ACS4等[5];果实细胞壁代谢相关的基因,包括PG2a和EXP1等[3];果实成熟色素积累相关的基因,比如PSY1等[3,6];以及果实成熟香气物质形成的相关基因,包括LoxC和ADH 2等[6-7]。此外,RIN转录因子能够与FUL1、FUL2、M ADS1和TAGL1形成异源四聚体,发挥对果实成熟过程的调控作用[3,8-10]。由此可见,RIN转录因子是果实成熟过程中的核心调控因子。
此外,番茄NF-Y转录因子是近年来被发现参与果实成熟的一类家族,较早的研究认为NF-Y主要参与植物生长发育、生物与非生物胁迫相关的途径[11]。研究发现,番茄中存在59 个NF-Y基因,通过表达分析,有13 个NF-Y基因在果实成熟过程中表达量较高,运用病毒诱导基因沉默技术[12],沉默其中5 个较高表达的NF-Y基因会影响果实成熟表型[13]。由此可见,NF-Y在番茄果实成熟过程中发挥了重要作用,那么NF-Y是否与果实成熟核心调控因子RIN协同调控果实的成熟过程,暂未发现有相关的研究。
综上,为明确番茄NF-Y与RIN转录因子在调控果实的成熟过程中是否存在联系,本研究利用数据库中番茄NF-Y的相关信息,通过生物信息学方法分析番茄NF-Y基因与果实成熟以及与RIN基因表达模式的相关性,并结合RIN转录因子对NF-Y基因DNA的结合分析,阐述NF-Y转录因子在果实成熟过程及其与RIN共同调控果实成熟过程中发挥的重要作用,为进一步研究NF-Y基因的生物学功能提供理论支持。
1 材料与方法
1.1 植物中NF-Y基因的鉴定
番茄(So la n um ly c o p e rs ic um)和拟南芥(Arabidopsis thaliala)的NF-Y基因信息来源于植物转录因子数据库PlantTFDB database v3.0(http://planttfdb.cbi.edu.cn/)[14]。番茄的基因注释信息来源于茄科基因组数据库Sol Genom ics Network(SGN,ftp://ftp.solgenom ics.net/genomes/Solanum_lycopersicum/annotation/ITAG2.4_release/)。
1.2 番茄NF-Y蛋白结构域与进化分析
番茄和拟南芥NF-Y氨基酸序列源于Plan tTFDB database v3.0(http://planttfdb.cbi.edu.cn/)[14]。运用Vector NTI Advance 14 11.5.1软件(Invitrogen)进行氨基酸序列分析,选用Clustal X 2.1和MEGA 4.0软件进行氨基酸序列聚类和进化分析,采用邻接法构建系统进化树,用1 000 次Bootstrap进行检验[15-16]。
1.3 番茄果实NF-Y基因的表达模式及受到RIN结合的分析
番茄NF-Y基因在果实成熟不同时期,包括花后17、39、42 d及52 d,以及野生型AC和突变体rin果实破色时期的表达量数据来自转录组数据[17]。所有的表达值中,剔除Reads Per Kilobase per M illion mapped reads(RPKM)值为零的基因,并运用R软件(http://www.r-project.org/)进行表达值热图的绘制。RIN转录因子对NF-Y基因DNA的结合数据来源于染色质免疫共沉淀-测序(ChIP-Seq)数据[17]。
2 结果与分析
2.1 番茄NF-Y因子的鉴定
通过植物转录因子数据库PlantTFDB database v3.0(http://p lanttfdb.cbi.edu.cn/)获得59 个番茄NF-Y基因的信息,根据其具有的亚基进行分类,包括10 个NF-YA、29 个NF-YB和20 个NF-YC,见表1。番茄NF-Y基因编码的氨基酸长度具有差异,如NF-YA基因编码的氨基酸长度为154~325个,NF-YB基因编码的氨基酸长度为86~357 个,而NF-YC基因编码的氨基酸长度为93~677 个。番茄NF-Y基因编码的蛋白理论等电点也具有明显的差异,如NF-YA蛋白预测等电点均大于7.0,为酸性氨基酸;NF-YB蛋白的预测等电点范围为4.18~9.61,包括25 个酸性氨基酸和4 个碱性氨基酸,其中编码NF-YB3的3 个不同基因编码的蛋白,酸碱性存在明显差异,具有NF-YB亚基的13 个NF-YB6蛋白中,有11 个为酸性氨基酸,另2 个为碱性氨基酸;而NF-YC蛋白的预测等电点范围为4.05~10.94,包括14 个酸性氨基酸和6 个碱性氨基酸,其中4 个NF-YC1和8 个NF-YC4具有酸碱性差异的氨基酸。通过等电点的分析,可以预知番茄不同NF-Y蛋白在不同酸碱环境中的稳定性,从而进行更进一步的功能分析。
表1 番茄NF-Y转录因子的分类及基本特征
Table 1 Classifi cation and characteristics of NF-Y genes in tomato

注:表中基因名称与茄科基因组数据库The So l Genom ics Netw ork(solgenomics.net)一致。
亚基 基因名称 氨基酸 等电点 亚基 基因名称 氨基酸 等电点长度/个 长度/个NF-YA1 Solyc01g008490 312 7.005 5 NF-YB3 Solyc04g054150 188 8.184 1 NF-YA1 Solyc11g065700 300 7.232 5 NF-YB3 Solyc07g065500 182 6.306 2 NF-YA3 Solyc03g121940 241 9.392 4 NF-YB3 Solyc12g006120 202 6.357 5 NF-YA3 Solyc12g009050 253 8.858 3 NF-YB5 Solyc01g067130 138 5.961 7 NF-YA7 Solyc02g069860 208 7.837 4 NF-YB5 Solyc01g099320 146 4.900 4 NF-YA7 Solyc10g079150 219 9.878 6 NF-YB5 Solyc06g009010 178 5.395 1 NF-YA8 Solyc08g062210 325 9.073 4 NF-YB5 Solyc09g074760 138 5.310 9 NF-YA9 Solyc01g087240 303 8.824 4 NF-YB6 Solyc02g032180 287 5.020 1 NF-YA10 Solyc01g006930 311 10.055 0 NF-YB6 Solyc02g032190 95 4.843 1 NF-YA10 Solyc10g081840 154 10.927 0 NF-YB6 Solyc04g015060 237 5.894 3 NF-YC1 Solyc03g110860 230 5.036 2 NF-YB6 Solyc05g005350 219 4.179 1 NF-YC1 Solyc03g111450 318 4.438 4 NF-YB6 Solyc05g005360 224 4.273 1 NF-YC1 Solyc03g111460 288 8.202 1 NF-YB6 Solyc05g005380 357 5.962 9 NF-YC1 Solyc06g072040 232 5.022 2 NF-YB6 Solyc05g005390 238 5.005 5 NF-YC3 Solyc08g007960 124 6.944 9 NF-YB6 Solyc05g005440 208 4.948 1 NF-YC4 Solyc00g107050 144 5.732 0 NF-YB6 Solyc05g015550 219 5.340 3 NF-YC4 Solyc02g021330 153 4.673 5 NF-YB6 Solyc07g065570 188 8.361 2 NF-YC4 Solyc02g091030 677 4.053 7 NF-YB6 Solyc07g065580 178 9.605 5 NF-YC4 Solyc03g110840 162 5.296 9 NF-YB6 Solyc10g009440 238 4.875 6 NF-YC4 Solyc03g110850 163 5.100 2 NF-YB6 Solyc11g012750 152 6.230 6 NF-YC4 Solyc03g111470 273 8.080 9 NF-YB7 Solyc12g027650 208 7.958 2 NF-YC4 Solyc11g016910 93 4.855 5 NF-YB8 Solyc04g009520 86 5.881 2 NF-YC4 Solyc11g016920 138 4.760 2 NF-YB8 Solyc04g049910 165 4.858 5 NF-YC9 Solyc01g079870 258 6.149 0 NF-YB10 Solyc09g007290 129 6.521 4 NF-YC10 Solyc06g016750 446 8.858 5 NF-YB11 Solyc11g068480 175 4.642 1 NF-YC11 Solyc01g096710 276 5.169 1 NF-YB13 Solyc03g114400 132 4.264 5 NF-YC13 Solyc05g015330 144 9.956 4 NF-YB13 Solyc06g069310 131 4.250 3 Histone H2A2 Solyc05g014800 135 10.937 0 Histone superfamily Solyc00g270510 184 6.598 6 Histone H2A2 Solyc05g014830 292 10.752 0 Histone superfamily Solyc05g005370 224 4.329 6 Histone superfamily Solyc11g072150 305 4.682 9
2.2 番茄NF-Y氨基酸序列比对及进化分析

图1 番茄和拟南芥NF-Y氨基酸序列比对
Fig. 1 Am ino acid sequence alignment of tomato and Arabidopsis NF-Y domains
由于拟南芥中NF-Y因子的功能研究得较为透彻,因此将番茄的59 个NF-Y因子与拟南芥的NF-Y家族进行多重序列比对进而进行功能域的分析(图1)。拟南芥NF-Y家族是由10 个A tNF-YA、13 个A tNF-YB和14 个A tNF-YC组成。根据NF-Y亚基的不同,包括NF-YA、NF-YB和NF-YC,分别进行多重氨基酸的序列比对,每个亚家族都有一个或多个核心区域,具有明显的同源结构域,据文献报道,这些保守的结构域与亚基之间的联系,以及对DNA的结合起到很重要的作用[18-20],见图1中加以强调标注的部分。此外,运用番茄和拟南芥NF-Y全长的氨基酸序列,基于不同的NF-Y亚基,构建了无根的进化树,通过进化分析对番茄中相应的NF-Y进行功能预测,见图2。
番茄和拟南芥的NF-YA蛋白都具有两个典型的保守结构域(图1),根据已有研究表明,在动物和酵母中,第一个结构域包含有20 个保守的氨基酸,该结构域能够与NF-YB或者NF-YC发生结合,另一个结构域包含21 个氨基酸,则能够特异性的识别靶基因DNA序列上的CCAAT元件[21]。而在保守的结构域以外,氨基酸的保守性很低。但是,NF-YA氨基酸序列富含谷氨酰胺和丝氨酸/苏氨酸残基,据研究其与增强转录激活作用紧密相关。
番茄和拟南芥NF-YB蛋白具有一段多于90 个氨基酸的保守域,该结构域能够与NF-YC以及NF-YA亚基发生作用,同时起到结合DNA的功能(图1)。在保守域以外番茄NF-YB因子的氨基酸长度以及氨基酸的种类差异较大。基于进化分析,可以预测进化树同一分支中蛋白的功能。比如拟南芥A tNF-YB,又名为LEC1-LIKE(L1L),是调控胚胎发育的关键因子,编码该蛋白的基因在发育的胚胎中具有明显的富集[22]。类似地,番茄中L1L的同源基因,包括L1L1~L1L13,在种子和发育的果实中,具有相类似的表达模式,因此可以预测其可能具有类似的功能[23](图2)。
番茄和拟南芥NF-YC亚家族具有一个近80 个氨基酸的保守域(图1),已有研究表明,该结构域参与靶基因DNA的结合,以及与其他NF-Y因子的互作中起关键作用。这种机制存在于植物拟南芥[24]、二穗短柄草[25]以及其他植物[26]中。此外,在酵母和真菌中发现,绝大多数NF-YC蛋白具有含量较高的谷氨酰胺残基,能够作用于增强转录激活[27-28]。
2.3 番茄NF-Y基因在果实成熟过程中的表达
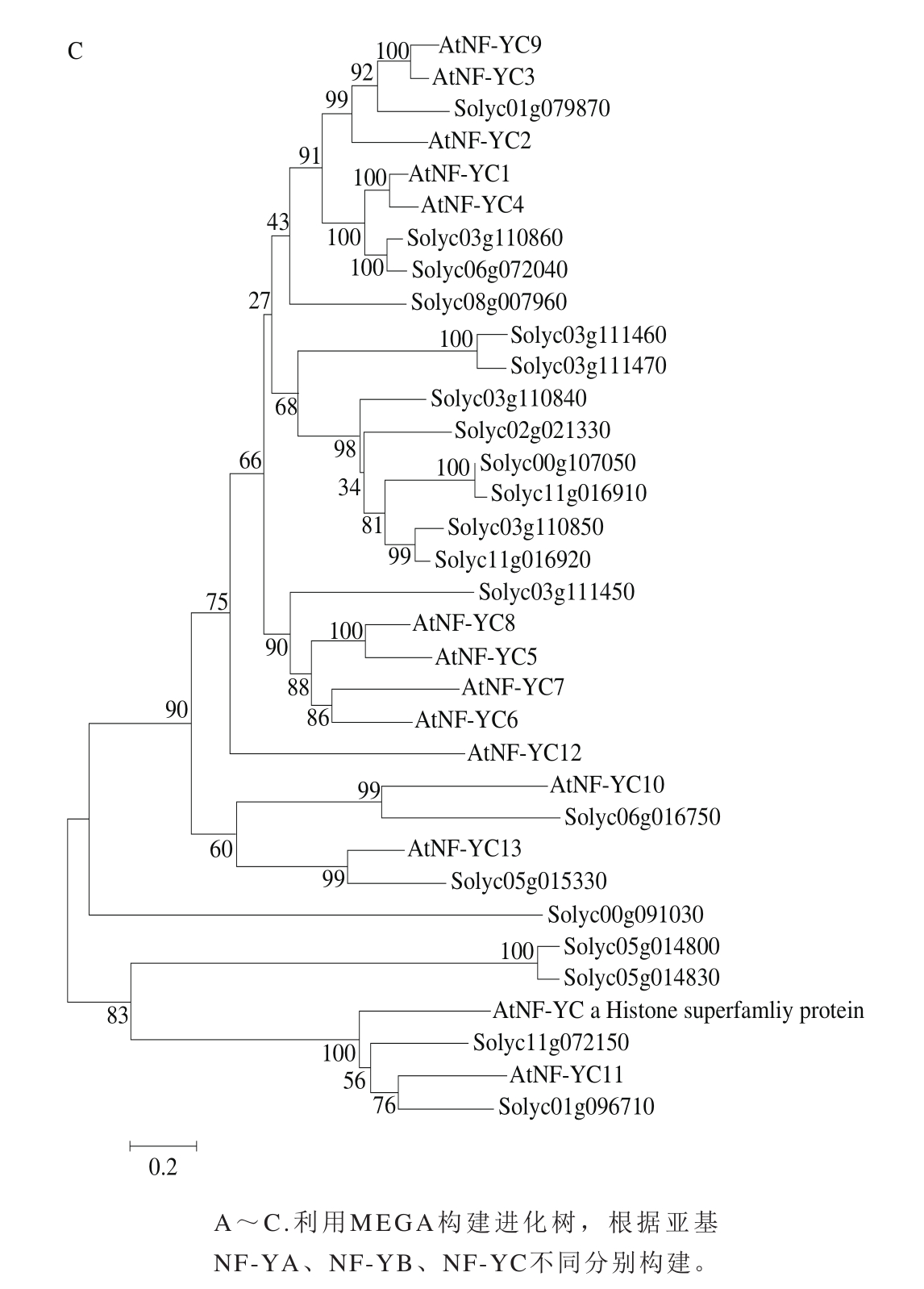
图2 番茄和拟南芥NF-Y进化树
Fig. 2 Phylogenetic relationships based on am ino acid sequence sim ilarity of NF-Y in tomato and Arabidopsis
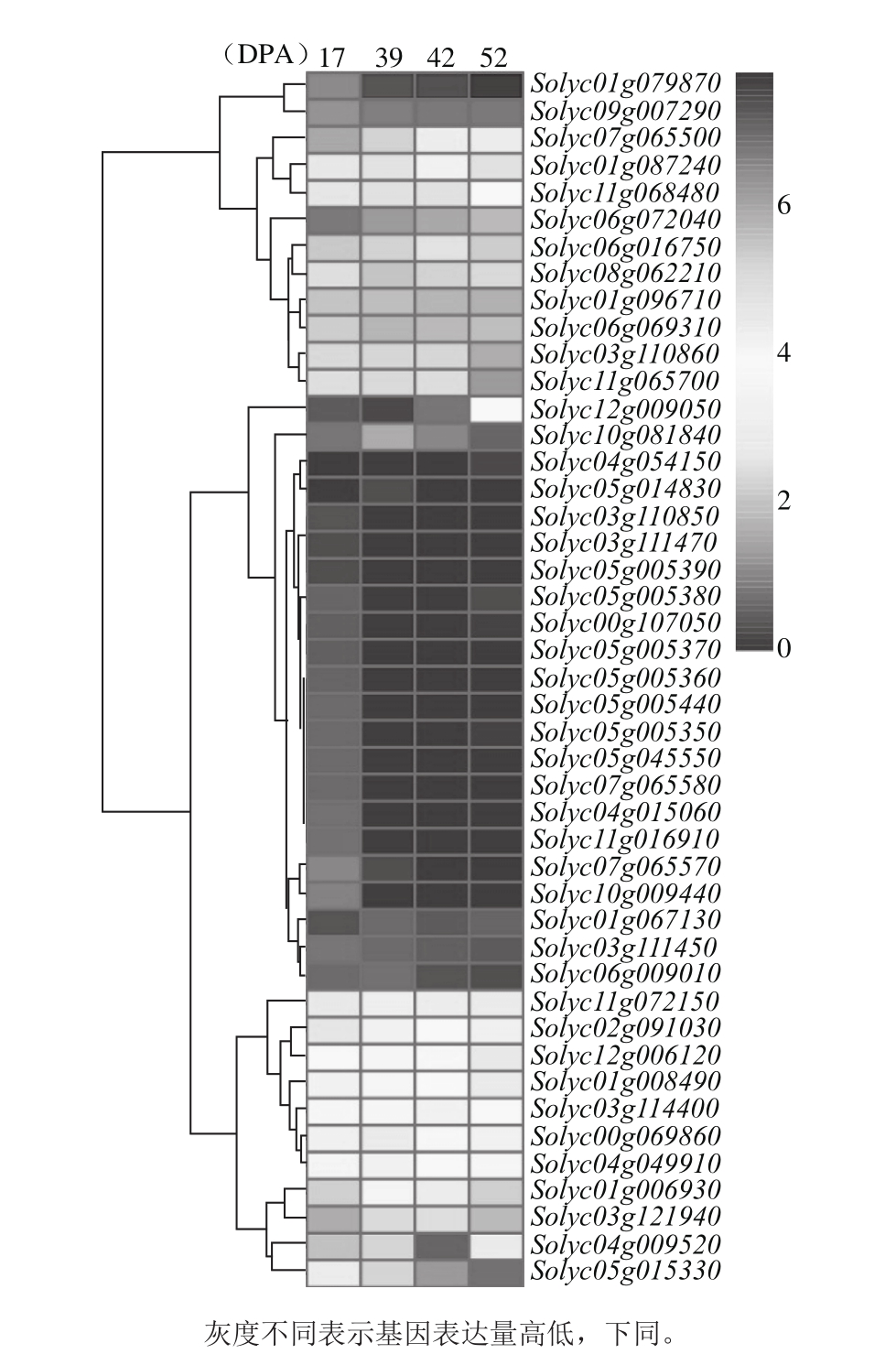
图3 番茄NF-Y基因在果实成熟不同时期的表达量热图
Fig. 3 Heat map of NF-Y expression in tomato during various ripening stages
番茄果实的转录组数据(RNA-Seq)已公开发表[17],本研究选用自花后17、39、42 d及52 d的番茄果实转录组数据,分别对应于果实未熟、绿熟、破色以及红熟期的基因表达情况,着重关注番茄NF-Y基因的表达模式,剔除基因表达RPKM值在4 个时期均为零的基因,并对其余45 个NF-Y基因进行表达量的可视化,并构建了表达模式聚类图,如图3所示。结果表明,有21 个NF-Y基因的表达与成熟过程相关,包括7 个NF-YA、7 个NF-YB以及7 个NF-YC。具体如下:
番茄NF-YA基因表达与成熟相关的7 个基因中,3 个基因在成熟过程具有明显的上调表达,包括Solyc12g009050、So lyc01g008490和So lyc11g065700,因此这3 个基因表达模式与成熟过程呈正相关;3 个基因在成熟过程中呈现波动趋势,包括Solyc01g006930、Solyc01g087240和Solyc08g062210,在果实未熟期表达相对较低,果实成熟过程开始时表达显著上调,之后会出现下调的趋势,因此,这3个基因可能只在成熟的不同时期起不同的作用;研究表明,沉默Solyc01g087240和So lyc08g062210、So lyc11g065700基因会导致果实呈现黄色,对果实的成熟起到抑制作用[13];此外,Solyc02g069860在观测的4 个时期表达量较高切呈现稳定趋势,表明该基因可能影响果实发育和成熟的整个过程。
番茄NF-YB基因表达与成熟相关的7 个基因中,只有1 个基因的表达模式与成熟过程呈现明显的正相关,即Solyc09g007290基因;有2 个基因,包括Solyc11g068480和Solyc07g065500的表达模式与成熟过程呈负相关,研究表明,沉默Solyc07g065500会促进果实变红,证实了其是果实成熟抑制子的作用模式[13];另有2个基因,包括So lyc03g114400和So lyc04g049910,在观测的4 个时期表达量较高且呈现稳定趋势,表明该基因可能具有果实发育和成熟的全局作用模式;而Solyc12g006120和Solyc06g069310基因,在果实成熟过程中表达情况呈现波动情况,研究表明沉默Solyc06g069310会抑制果实成熟,表明该基因至少在成熟的某个时期起促进作用。
番茄NF-YC基因表达与成熟相关的7 个基因中,有4 个基因的表达模式与成熟过程呈正相关,包括Solyc11g072150、So lyc03g110860、Solyc01g096710和So lyc01g079870;有2 个基因的表达模式与成熟过程呈负相关,包括So lyc02g091030和Solyc06g072040;而Solyc06g016750基因的表达则在成熟过程中呈现波动,可能在成熟不同时期具有不同的作用模式。
2.4 番茄NF-Y基因与成熟重要转录因子RIN的联系
通过上述分析发现,番茄NF-Y基因在果实的成熟过程中起到重要的作用[13]。众所周知,转录因子RIN是果实成熟的关键调控因子,因为RIN能够调控许多成熟相关的基因影响果实的成熟进程,包括成熟相关的转录因子基因、乙烯合成与代谢相关的基因、果实细胞壁代谢相关的基因、果实成熟色素积累相关的基因,以及果实成熟香气物质形成的相关基因等[3,5-7]。因此,本研究进一步对番茄NF-Y与RIN转录因子在调控果实的成熟过程中的联系进行了分析。
如图4所示,选用野生型AC和突变体rin果实破色时期的表达量数据[17],着重关注番茄NF-Y基因的表达模式,剔除基因表达RPKM值为零的基因,并对余下30 个NF-Y基因进行表达量的可视化,并构建了表达模式聚类图。结果表明,其中有6个NF-Y基因在野生型AC和突变体rin果实中RPKM值较高(RPKM>10),且表达倍数差异较高(1.5 倍)。这些差异基因包括4 个NF-YA基因,分别为Solyc02g069860、Solyc01g006930、Solyc08g062210和Solyc01g087240,这4 个基因在突变突变体rin果实中的表达高于野生型AC果实,表明其与RIN基因的表达情况呈负相关,此外So lyc08g062210和Solyc01g087240在成熟过程中表达趋势是波动的,并且分别沉默这2个基因,会抑制果实的成熟过程[13],因此可以推测,这2个NF-YA基因至少在花后42 d的时期,与转录因子RIN并不存在直接的调控关系。

图4 番茄NF-Y基因在野生型AC和突变体rin果实中的表达量热图
Fig. 4 Heat map of tomato NF-Y expression in wild-type AC and rin mutant fruits
在野生型AC和突变体rin果实中RPKM值较高(RPKM>10),且表达倍数差异较高(1.5 倍)的另两个基因分别为1 个NF-YB即Solyc11g068480、1 个NF-YC即Solyc02g091030基因,其中Solyc11g068480与RIN基因的表达情况呈正相关,而Solyc02g091030与RIN基因的表达情况呈负相关。
为进一步明确转录因子RIN与NF-Y在成熟过程中的联系,本研究分析了RIN转录因子对NF-Y基因DNA的结合数据[17],数据基于染色质免疫共沉淀-测序(Ch IPSeq)技术,挖掘的基因信息表明转录因子与靶DNA序列的物理结合情况。如表2所示,转录因子RIN对4 个NF-Y基因具有直接结合能力,包括3 个NF-YA,即Solyc10g079150、Solyc10g081840和Solyc12g009050,以及1 个NF-YC,即Solyc03g111460。其中,属于NF-YA家族的Solyc12g009050基因,其表达模式与成熟过程呈正相关(图3),并且与RIN基因表达模式呈正相关(图4),因此转录因子RIN可能调控该基因的表达。而RIN对其具体的调控模式,则可以通过反式调控能力分析获知,即将Solyc12g009050基因的启动子克隆到LUC(萤火虫荧光素酶)基因与35S启动子连REN(海肾荧光素酶)的双元荧光报告体系中,通过超表达RIN蛋白,获知其对靶基因的激活或抑制的调控模式,该体系已被广泛应用于转录因子的调控模式分析[29-30]。
表2 转录因子RIN对番茄NF-Y的结合位点富集分析
Table 2 Binding peaks of RIN to tomato NF-Y genes
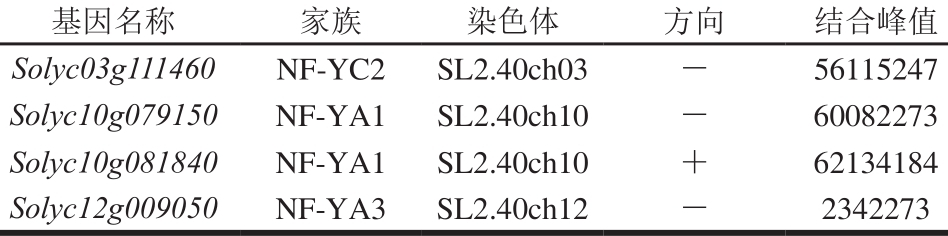
注:+.与染色体测序方向一致;-.与染色体测序方向不一致。
基因名称 家族 染色体 方向 结合峰值Solyc03g111460 NF-YC2 SL2.40ch03 - 56115247 Solyc10g079150 NF-YA1 SL2.40ch10 - 60082273 Solyc10g081840 NF-YA1 SL2.40ch10 + 62134184 Solyc12g009050 NF-YA3 SL2.40ch12 - 2342273
3 结 论
番茄中含有59 个NF-Y基因,包括10 个NF-YA、29 个NF-YB和20 个NF-YC。番茄NF-Y基因编码的氨基酸长度差异明显,但具有明显的保守结构域。通过与拟南芥NF-Y因子的进化分析,番茄NF-Y与之同源的因子具有相似的功能,因此保守性较强。根据番茄NF-Y基因在果实成熟过程中的表达模式分析,表明不同的NF-Y因子在成熟过程中的作用可能存在差异,这与文献报道的沉默番茄中不同的NF-Y基因会获得不同的表型是一致的[13],证实番茄NF-Y因子对果实成熟其重要的调控作用。通过分析番茄NF-Y基因与RIN基因的表达模式,以及转录因子RIN对NF-Y基因DNA的结合能力,结果表明转录因子RIN可能能够直接调控NF-YA家族Solyc12g009050基因的表达,但具体的反式调控模式则有待进一步的实验分析,而对其他NF-Y基因的影响也有待更进一步的明确。
参考文献:
[1] SEYMOUR G, OSTERGAARD L, CHAPMAN N, et al. Fruit development and ripening[J]. Annual Review of Plant Biology, 2013,64: 219-241. DOI:10.1146/annurev-arplant-050312-120057.
[2] KARLOVA R, CHAPMAN N, DAVID K, et al. Transcriptional control of fleshy fruit development and ripening[J]. Journal of Experimental Botany, 2014, 65(16): 4527-4541. DOI:10.1093/jxb/eru316.
[3] M ARTEL C, VREBALOV J, TAFELM EYER P, et al. The Tomato MADS-Box transcription factor RIPENING INHIBITOR interacts w ith promoters involved in numerous ripening processes in a COLORLESS NONRIPENING-dependent manner[J]. Plant Physiology, 2011, 157(3): 1568-1579. DOI:10.1104/pp.111.181107.
[4] VREBALOV J, RUEZINSKY D, PADMANABHAN V, et al. A MADS-Box gene necessary for fruit ripening at the tomato ripeninginhibitor (Rin) locus[J]. Science, 2002, 296(5566): 343-346.DOI:10.1126/science.1068181.
[5] FUJISAWA M, NAKANO T, SHIMA Y, et al. A large-scale identification of direct targets of the tomato MADS box transcription factor RIPENING INH IBITOR reveals the regulation of fruit ripening[J]. The Plant Cell, 2013, 25(2): 371-386. DOI:10.1105/tpc.112.108118.
[6] QIN G, WANG Y, CAO B, et al. Unraveling the regulatory network of the MADS box transcription factor RIN in fruit ripening[J]. The Plant Journal, 2012, 70(2): 243-255. DOI:10.1111/j.1365-313X.2011.04861.x.
[7] 黎卡, 李珊, 罗云波, 等. 番茄果实成熟关键调控因子MADS-RIN的研究进展[J]. 中国农学通报, 2015, 31(18): 126-131.
[8] FUJISAWA M, SHIMA Y, NAKAGAWA H, et al. Transcriptional regulation of fruit ripening by tomato FRUITFULL homologs and associated MADS box proteins[J]. The Plant Cell, 2014, 26(1): 89-101. DOI:10.1105/tpc.113.119453.
[9] DONG T, HU Z, DENG L, et al. A tomato MADS-Box transcription factor, SlMADS1, acts as a negative regulator of fruit ripening[J]. Plant Physiology, 2013, 163(2): 1026-1036. DOI:10.1104/pp.113.224436.
[10] SH IM A Y, K ITAGAWA M, FUJISAWA M, et al. Tom ato FRUITFULL homologues act in fruit ripening via form ing MADS-box transcription factor complexes w ith RIN[J]. Plant Molecular Biology,2013, 82(4/5): 427-438. DOI:10.1007/s11103-013-0071-y.
[11] LIANG M, YIN X, LIN Z, et al. Identification and characterization of NF-Y transcription factor fam ilies in canola (Brassica napus L.)[J].Planta, 2014, 239(1): 107-126. DOI:10.1007/s00425-013-1964-3.
[12] FU D, ZHU B, ZHU H, et al. Virus-induced gene silencing in tomato fruit[J]. The Plant Journal, 2005, 43(2): 299-308. DOI:10.1111/j.1365-313X.2005.02441.x
[13] LI S, LI K, JU Z, et al. Genome-w ide analysis of tomato NF-Y factors and their role in fruit ripening[J]. BMC Genom ics, 2016, 17(1).DOI:10.1186/s12864-015-2334-2.
[14] JIN J, ZHANG H, KONG L, et al. PlantTFDB 3.0: a portal for the functional and evolutionary study of plant transcription factors[J]. Nucleic Acids Research, 2013, 42(D1): 1182-1187. DOI:10.1093/nar/gkt1016.
[15] LARKIN M A, BLACKSHIELDS G, BROWN N P, et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics, 2007, 23(21): 2947-2948.DOI:10.1093/bioinformatics/btm404.
[16] TAMURA K, DUDLEY J, NEI M, et al. MEGA 4: M olecu lar evolutionary genetics analysis (MEGA) softw are version 4.0[J].M o lecu lar Bio logy and Evo lution, 2007, 24(8): 1596-1599.DOI:10.1093/molbev/msm092.
[17] ZHONG S, FEI Z, CHEN Y, et al. Single-base resolution methylomes o f tomato fruit developm ent reveal epigenom e modifications associated w ith ripening[J]. Nature Biotechnology, 2013, 31(2): 154-159. DOI:10.1038/nbt.2462.
[18] KIM I, SINHA S, DE CROMBRUGGHE B, et al. Determ ination of functional domains in the C subunit of the CCAAT-binding factor(CBF) necessary for formation of a CBF-DNA comp lex: CBF-B interacts simultaneously w ith both the CBF-A and CBF-C subunits to form a heterotrimeric CBF molecule[J]. M olecular and Cellular Biology, 1996, 16(8): 4003-4013. DOI:10.1128/MCB.16.8.4003.
[19] MCNABB D S, TSENG K A, GUARENTE L. The Saccharomyces cerevisiae Hap5p homolog from fission yeast reveals two conserved domains that are essential for assembly of heterotetrameric CCAAT-binding factor[J]. Molecular & Cellular Biology, 1997, 17(12): 7008-7018. DOI:10.1128/MCB.17.12.7008.
[20] SINHA S, KIM I S, SOHN K Y, et al. Three classes of mutations in the A subunit of the CCAAT-binding factor CBF delineate functional domains involved in the three-step assembly of the CBF-DNA comp lex[J]. Molecular & Cellular Biology, 1996, 16(1): 328-337.DOI:10.1128/MCB.16.1.328.
[21] QUACH T N, NGUYEN H T M, VALLIYODAN B, et al. Genomew ide expression analysis of soybean NF-Y genes reveals potential function in development and drought response[J]. Molecular Genetics and Genom ics, 2015, 290(3): 1095-1115. DOI:10.1007/s00438-014-0978-2.
[22] KWONG R, BUI A, LEE B, et al. LEAFY COTYLEDON1-LIKE defines a class of regulators essential for embryo development[J]. The Plant Cell, 2003, 15(1): 5-18. DOI:10. 1105/ tpc. 006973.
[23] HILIOTI Z, GANOPOULOS I, BOSSIS I, et al. LEC1-LIKE paralog transcrip tion factor: how to survive extinction and fit in NF-Y protein complex[J]. Gene, 2014, 543(2): 220-233. DOI:10.1016/j.gene.2014.04.019.
[24] SIEFERS N, DANG K K, KUM IMOTO R W, et al. Tissue-specifi c expression patterns of arabidopsis NF-Y transcription factors suggest potential for extensive combinatorial complexity[J]. Plant Physiology,2008, 149(2): 625-641. DOI:10.1104/pp.108.130591.
[25] CAO S, KUM IMOTO R, SIRIWARDANA C, et al. Identifi cation and characterization of NF-Y transcription factor fam ilies in the monocot model plant Brachypodium distachyon[J]. PLoS ONE, 2011, 6(6):e21805. DOI:10.1371/journal.pone.0021805.
[26] PETRONI K, KUM IMOTO R W, GNESUTTA N, et al. The prom iscuous life of plant NUCLEAR FACTORY transcription factors[J]. The Plant Cell, 2013, 24(12): 4777-4792. DOI:10.1105/tpc.112.105734.
[27] COUSTRY F, MAITY S N, SINHA S, et al. The transcriptional activity of the CCAAT-binding factor CBF is mediated by two distinct activation domains, one in the CBF-B subunit and the other in the CBF-C subunit[J]. Journal of Biological Chem istry, 1996, 271(24):14485-14491. DOI:10.1074/jbc.271.24.14485.
[28] DE S A, IMBRIANO C, MANTOVANI R. Dissection of the NF-Y transcriptional activation potential[J]. Nucleic Acids Research, 1999,27(13): 2578-2584.
[29] HELLENS R P, ALLAN A C, FRIEL E N, et al. Transient expression vectors for functional genom ics, quantification of promoter activity and RNA silencing in p lants[J]. Plant M ethods, 2005, 1: 13.DOI:10.1186/1746-4811-1-13.
[30] BA L, SHAN W, KUANG J, et al. The banana MaLBD (LATERAL ORGAN BOUNDARIES DOMAIN) transcription factors regulate EXPANSIN expression and are involved in fruit ripening[J]. Plant Molecular Biology Reporter, 2014, 32(6): 1103-1113. DOI:10.1007/s11105-014-0720-6.
Bioinformatics Analysis of Tomato Nuclear Factor Y (NF-Y) Transcription Factors and Their Relationship w ith Ripening Inhibitor (RIN) in Co-Regulating Fruit Ripening
LI Shan, XU Huijinlan, ZHU Benzhong, LUO Yunbo*
(College of Food Science and Nutritional Engineering, China Agricultural University, Beijing 100083, China)
Abstract:In this study, am ino acid motifs and evolutionary relationship of 59 tomato nuclear transcription factor Y (NF-Y)members were analyzed using bioinformatics approaches. Furthermore, changes in the expression pattern of tomato NF-Y genes in the w ild-type AC and ripening inhibitor (rin) mutant were investigated during fruit ripening. On the basis of the NF-Y gene binding capacity of the transcription factor RIN, their co-regulation on tomato fruit ripening was elucidated.
Key words:tomato fruit; NF-Y; RIN; bioinformatics analysis
DOI:10.7506/spkx1002-6630-201722001
中图分类号:TS255.3
文献标志码:A
文章编号:1002-6630(2017)22-0001-07
引文格式:
李珊, 许蕙金兰, 朱本忠, 等. 番茄NF-Y转录因子生物信息学分析及其与RIN共同调控果实成熟研究[J]. 食品科学,2017, 38(22): 1-7. DOI:10.7506/spkx1002-6630-201722001. http://www.spkx.net.cn
LI Shan, XU Huijinlan, ZHU Benzhong, et al. Bioinformatics analysis of tomato nuclear factor Y (NF-Y) transcription factors and their relationship w ith ripening inhibitor (RIN) in co-regulating fruit ripening[J]. Food Science, 2017, 38(22): 1-7.(in Chinese w ith English abstract)
DOI:10.7506/spkx1002-6630-201722001. http://www.spkx.net.cn
收稿日期:2017-04-11
基金项目:国家自然科学基金面上项目(31571894;31271959)
作者简介:李珊(1988—),女,博士研究生,研究方向为果蔬采后生理分子生物学。E-mail:419584434@qq.com
*通信作者:罗云波(1958—),男,教授,博士,研究方向为果蔬采后生理和分子生物学。E-mail:lyb@cau.edu.cn






