表1 样品含量及纯度
Table 1 Purity and concentration of extracted DNA in each sample
肖英平1,何祥祥2,戴宝玲1,桂国弘1,唐 标1,杨 华1,*
(1.浙江省农业科学院农产品质量标准研究所,浙江省植物有害生物防控省部共建国家重点实验室培育基地,浙江 杭州 310021;2.西北农林科技大学食品科学与工程学院,陕西 杨凌 712100)
摘 要:目的:比较涂抹法和冲洗法对冷鲜鸡表面细菌DNA提取及高通量测序结果的影响。方法:将4 只冷鲜鸡对半分开,分别采用涂抹法和冲洗法采集其表面微生物,提取基因组DNA,对细菌16S rRNA基因V3~V4区进行聚合酶链式反应扩增,通过Illumina Hiseq测序平台对扩增产物进行高通量测序,使用QIIME等软件对测序序列进行分析统计。结果:两种方法采集的样品在细菌基因组DNA提取、菌群丰富度、多样性及菌群结构等方面均无显著差异(P>0.05)。高通量测序结果表明,冷鲜鸡表面细菌菌群主要分布于7 个门,10 个属。变形菌门为优势菌门,占70%以上;希瓦氏菌属、假单胞菌属、不动杆菌属、嗜冷杆菌属和环丝菌属为优势菌属,分别占10%~20%。结论:两种采样方法对冷鲜鸡表面细菌DNA提取及高通量测序没有明显影响,但冲洗法比涂抹法简单易行,且不易发生二次污染,因此更具有实用性。本实验同时也为冷鲜鸡表面细菌的研究提供了数据资料。
关键词:冷鲜鸡;菌群结构;涂抹法;冲洗法;高通量测序
近年来,我国许多城市已禁止活禽交易,实施肉鸡统一屠宰,以冷鲜鸡或“杀白鸡”形式上市。所谓冷鲜鸡,是指在严格执行兽医检验检疫制度和无公害管理的前提下,使屠宰后的鸡肉胴体中心温度在1~2 h内降至0~4 ℃,并在后续加工、流通及销售过程中一直保持此温度的生鲜鸡肉[1-2]。与现宰活鸡相比,冷鲜鸡不仅更加安全卫生,而且由于温度迅速降低而使肌肉僵直程度减弱,又经过充分的解僵熟化和排酸过程,蛋白质分解成较多的肽类、氨基酸及具有鲜味的核苷酸,使肉质变得柔软有弹性、口感细腻、味道鲜美,并且营养物质容易被人体消化吸收。因此冷鲜鸡的口感和营养价值都高于现宰活鸡。自20世纪90年代起,冷鲜鸡就已在欧美等市场普及,逐渐取代了现宰活鸡和冷冻鸡[2]。
冷鲜鸡在屠宰、分割及其他加工过程中污染的微生物在肉表面繁殖造成其腐败,同时污染的一些致病菌也易导致食源性疾病[3-5]。冷鲜鸡污染的微生物主要生长在肉的表面,通常情况下肉深层无菌,因此研究肉品表面微生物群落特征对于保证冷鲜鸡的质量安全具有十分重要的意义。
近年来高通量测序技术应用于农产品、畜产品微生物污染研究得到了迅速发展。高通量测序技术在分析微生物群落结构时,有着独特的优势,已成为微生物群落研究中非常重要的工具[6-7]。国内已有研究者利用16S rRNA基因的高通量测序技术对肉制品和水产品中细菌群落多样性进行分析[8-10]。
应用高通量测序方法研究冷鲜鸡微生物污染的第一步就是采集样品表面的微生物并提取DNA。采样方法不同可能会影响DNA提取效果并进而影响后续的实验结果。因此,本实验采用涂抹和冲洗两种方法对冷鲜鸡表面微生物进行采集,提取细菌基因组DNA,进行基于16S rRNA基因的高通量测序分析,从而对两种采样方法进行比较,以找到较适合的整只冷鲜鸡的前处理方法,为冷鲜鸡微生物检测提供理论和实践依据。同时也为冷鲜鸡表面微生物的菌群结构研究提供了数据资料。
1.1 材料、试剂与仪器
在杭州4 家不同的超市购买4 只不同厂家生产的冷鲜鸡,将它们对半分开,装于无菌采样袋中,分别编号为A、B、C、D和a、b、c、d(A和a为来自同一只鸡对半分开的样品,其他3 只同),置于4 ℃的车载冰箱中带回实验室用于实验。
无菌采样袋 青岛海博生物技术有限公司;细菌基因组DNA提取试剂盒Fungal/Bacterial DNA MiniPrep™美国Zymo Research公司;琼脂糖 美国Sunshine公司;DL5000 DNA Marker 北京擎科新业生物技术有限公司。
5427 R台式高速离心机 德国Eppendorf公司;Nanodrop 2000超微量分光光度计 美国Thermo Fisher Scientific公司;水平电泳仪 北京百晶生物技术有限公司;SW-CF-IF超净工作台 苏州安泰充气技术有限公司。
1.2 方法
1.2.1 样品微生物采样方法
冷鲜鸡中微生物的采样通常运用组织切割法、皮肤切割法、涂抹法和冲洗法,前两者为破坏性采样方法,操作较为复杂且取样位置局限[11-13]。冷藏条件下,细菌主要分布于表面,因此比较分析涂抹法和冲洗法两种非破坏性采样方式对冷鲜鸡表面微生物采集效果的影响。
1.2.1.1 涂抹法
在洁净室中,编号为A、B、C、D的样品,采用2 块20 cm×20 cm的无菌纱布,涂抹1/2 只鸡的整个表面,然后置于与1/2 只鸡质量相等的灭菌生理盐水中,摇匀,静置10 min,取20 mL上清液,10 000×g离心10 min。
1.2.1.2 冲洗法
在盛放编号为a、b、c、d样品的无菌采样袋中加入与1/2只鸡质量相等的灭菌生理盐水,用手握住无菌袋激烈振荡1 min,取出鸡肉样品,静置10 min,取20 mL上清液,10 000×g离心10 min。
1.2.2 细菌DNA提取
使用ZR Fungal/Bacterial DNA MiniPrep™提取细菌DNA。将上述不同采集方法离心得到的沉淀,用200 μL无菌生理盐水重悬后转移至ZR BashingBeadTMLysis Tube,然后按照试剂盒使用说明进行操作。使用Nanodrop 2000测定提取的DNA纯度和含量,并采用1%琼脂糖凝胶进行电泳检测。
1.2.3 16S rRNA基因PCR扩增
以提取的冷鲜鸡表面细菌DNA为模板,使用引物338F(5’-ACTCCTACGGGAGGCAGCA-3’)和806R(5’-GGACTACHVGGGTWTCTAAT-3’)对细菌16S rRNA基因V3~V4区进行聚合酶链式反应(polymerase chain reaction,PCR)扩增。
1.2.4 高通量测序及数据分析
测序由北京诺禾致源生物信息科技有限公司完成。采用Illumina Hiseq 2500高通量测序平台对冷鲜鸡表面细菌16S rRNA基因的V3~V4区进行测序。通过QIIME软件(Quantitative Insights Into Microbial Ecology,V1.7.0,http://qiime.org/scripts/split_libraries_fastq.html)对获得的序列进行质控和过滤,剔除低质量的DNA序列[14-16]。使用QIIME的Ucluster方法根据相似性不低于97%原则将通过质控的有效序列聚类成为操作分类单元(operational taxonomic unit,OTU)[17]。通过绘制稀释曲线评价所测序量是否覆盖全部类群。采用QIIME默认参数计算各样品的Alpha多样性指数(ChaoⅠ、ACE、Simpson、Shannon和覆盖度指数)。使用Sliva和RPD数据库对所有OTU的代表性序列进行物种匹配,在门和属的水平上对样品的细菌组成进行统计,得到菌群分布结果,并绘制柱状图。使用GraphPad Prism 5对两种采样方法的数据进行成对t检验,P<0.05为显著性差异判定标准。根据各个样品OTU的种类及其丰度,计算样品间的遗传距离,用加权UniFrac距离表示,根据计算结果使用R软件(Version 2.15.3)中的FactoMineR软件包对不同采样方法所获得的样品表面细菌结构相似度进行主坐标分析(principal coordinate analysis,PCoA)。使用ggplot2软件包绘制散点图。
2.1 冷鲜鸡细菌基因组DNA提取
表1 样品含量及纯度
Table 1 Purity and concentration of extracted DNA in each sample

涂抹法(A、B、C、D样品)和冲洗法(a、b、c、d样品)提取细菌基因组DNA的纯度和含量见表1。1%的琼脂糖凝胶电泳结果显示,8 个样本基因组条带清晰,无降解,可满足后续实验要求(图1)。两种采样方法对细菌基因组DNA提取效果无明显影响。

图1 冷鲜鸡样品细菌基因组DNA提取
Fig. 1 Agarose gel electrophoresis of bacterial genomic DNA extracted from chilled chicken
2.2 样品微生物物种丰富度及多样性
经过质控、过滤等处理,本实验最终获得429 492 条有效序列,各样品间有效序列数差别不大,涂抹法样品平均(53 700±3 663)条,冲洗法样品平均(53 670±3 399)条,两组无显著性差异(P=0.994)。所获得的有效序列根据97%相似性水平进行OTU聚类分析,各样品最终获得89~157 个OTU,两组亦无显著差异(OUT分别为136±16和125±13,P=0.531)。各样品的覆盖度指数均为0.999~1,表明样本中序列没有被测出的概率极低,因此本次测序结果能够代表样本的真实多样性组成(表2)。各样品的稀释曲线均趋于平缓(图2),表明测序已趋于饱和,测序深度已基本覆盖样品中所有物种。

图2 各样品稀释曲线
Fig. 2 Rarefaction curves of 8 different samples
进一步根据97%相似性水平下OTU的信息,使用ChaoⅠ指数、ACE指数、Shannon指数及Simpson指数对样品微生物物种的丰富度和多样性进行评估。观察物种数量ChaoⅠ指数(151.4±17.16和131.8±11.79,P=0.432)和ACE指数(150.2±15.14和133.8±12.23,P=0.405)显示两种采样方法对于样品细菌群落丰富度无显著影响。Shannon指数(4.282±0.262和4.381±0.203,P=0.243)和Simpson指数(0.912±0.018和0.924±0.010,P=0.235)显示,两种采样方法所获样品的多样性指数也无显著性差异(表2)。
表2 样品测序概况
Table 2 Sequencing results for each sample
注:N/A.不适用。
2.3 冷鲜鸡表面细菌菌群结构

图3 各样品的细菌菌群结构
Fig. 3 Bacterial community structures of different samples
将8 个样品中获得的OTU分别在门和属水平上进行物种注释,具体结果见表2和图3。可以看出,本实验所得到的冷鲜鸡表面细菌样品的序列主要分属于7 个门,分别为变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、放线菌门(Actinobacteria)、梭杆菌门(Fusobacteria)、脱铁杆菌门(Deferribacteres)以及待定门TM7。其中变形菌门占70%以上,为优势门,其次为厚壁菌门和拟杆菌门,三者共同构成了冷鲜鸡菌群的主要结构。在属的水平,希瓦氏菌属(Shewanella)、假单胞菌属(Pseudomonas)、不动杆菌属(Acinetobacter)、嗜冷杆菌属(Psychrobacter)和环丝菌属(Brochothrix)为主要菌属,约占冷鲜鸡菌群的90%以上,丰度分布较均匀;此外,还有少量的黄杆菌属(Flavobacterium)、香味杆菌属(Myroides)、魏斯氏菌属(Weissella)、漫游球菌属(Vagococcus)以及肉杆菌属(Carnobacterium)。
希瓦氏菌属和假单胞菌属是典型的腐败菌,其低温环境适应能力强,是冷藏禽畜肉中的优势菌,具有很强的产生氨等腐败产物的能力[18-22]。此外,不动杆菌属、嗜冷杆菌属以及环丝菌属中的热死环丝菌(Brochothrix thermosphacta)也能在冷藏温度下快速生长,是冷鲜禽畜肉及水产品低温贮藏过程中的常见腐败菌[23-28]。这些腐败菌主要是在屠宰、加工、包装、运输及销售等过程中污染鸡肉,且在冷藏过程中大量繁殖,最终会导致冷鲜鸡腐败变质[29-30]。本研究显示以上菌属分别占冷鲜鸡表面菌群的约10%~20%,除环丝菌属外,两种采样方法没有显著性差异(表3)。
表3 不同分类水平上样品的细菌群落结构及其相对丰度
Table 3 Bacterial community structures and their relative abundances at different classification levels in the swabbing and rising samples

注:*.差异显著,P<0.05。
2.4 样品的聚类与主坐标分析
根据各样品OTU的种类及其丰度计算各样品间的加权UniFrac距离(图4A),并基于此对8 个样品进行PCoA(图4B)。结果表明,同一只冷鲜鸡的样品显示出明显聚集,而不同冷鲜鸡样品之间相距较远。这表明涂抹和冲洗两种采样方法对于冷鲜鸡表面微生物的菌群结构无明显影响。

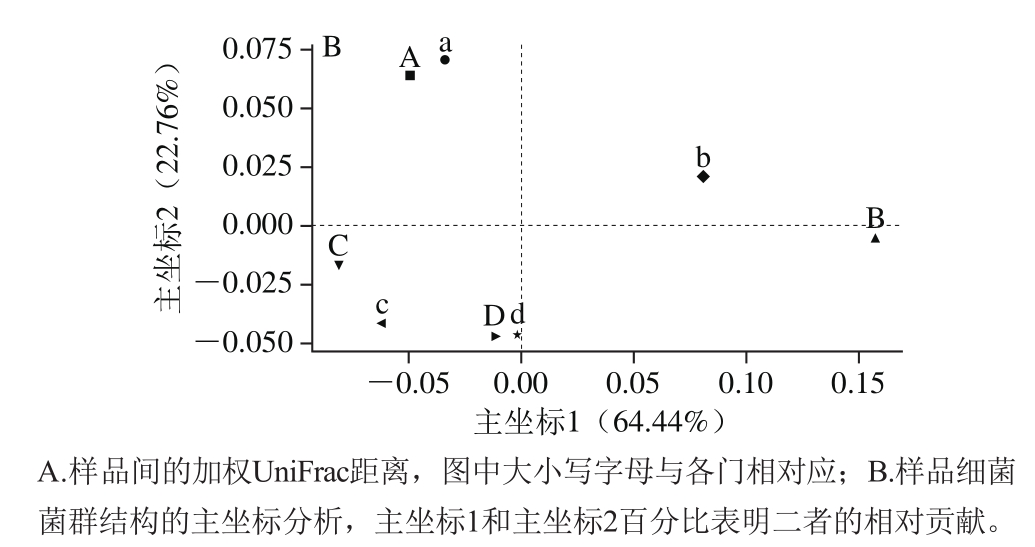
图4 各样品菌落结构的聚类和PCoA
Fig. 4 Cluster analysis and PCoA of bacterial community structure among the samples
本实验采用涂抹法和冲洗法对冷鲜鸡表面微生物进行采集,提取细菌基因组DNA,进行基于16S rRNA基因的高通量测序分析。结果表明:1)涂抹法和冲洗法对冷鲜鸡表面微生物DNA提取效果无显著差异;2)冷鲜鸡表面细菌菌群主要分布于7 个门,变形菌门为优势菌门,其中的希瓦氏菌属、假单胞菌属、不动杆菌属和嗜冷杆菌属为优势菌属,此外,厚壁菌门的环丝菌属丰度也较高,这些优势菌为典型的腐败菌[26,30];3)两种采样方法对冷鲜鸡表面细菌菌群的丰富度、多样性以及菌群结构均没有明显影响,但冲洗法比涂抹法简单易行,且不易发生二次污染,因此更具有实用性。
参考文献:
[1] 吴海虹, 刘朏, 刘芳, 等. 低温贮藏对冷鲜鸡腐败菌菌群变化的影响[J]. 现代食品科技, 2016, 32(4): 177-181. DOI:10.13982/j.mfst.1673-9078.2016.4.029.
[2] 易鹤. 现宰鸡、冷冻鸡和冷鲜鸡有何不同[J]. 农村新技术, 2014(6):57. DOI:10.3969/j.issn.1002-3542.2014.06.040.
[3] NYCHAS G J, SKANDAMIS P N, TASSOU C C, et al. Meat spoilage during distribution[J]. Meat Science, 2008, 78(1/2)∶ 77-89.DOI∶10.1016/j.meatsci.2007.06.020.
[4] 王虎虎, 徐幸莲. 冰鲜鸡肉中致病菌三重PCR检测方法的建立[J]. 中国农业科学, 2010, 43(17): 3608-3615. DOI:10.3864/j.issn.0578-1752.2010.17.015.
[5] HINTON J R, CASON J A, INGRAM K D. Tracking spoilage baeteria in commereial poultry processing and refrigerated storage of poultry carcasses[J]. Food Microbiology, 2004, 91(2): 155-165. DOI:10.1016/S0168-1605(03)00377-5.
[6] SEGATA N, BOERNIGEN D, TICKLE T L, et al. Computational meta’omics for microbial community studies[J]. Molecular Systems Biology, 2013, 9: 666. DOI:10.1038/msb.2013.22.
[7] DEGNAN P H, OCHMAN H. Illumina-based analysis of microbial community diversity[J]. The ISME Journal, 2012, 6(1): 183-194.DOI:10.1038/ismej.2011.74.
[8] 张赫宇, 杨波, 罗瑞明, 等. 高通量测序分析冷鲜滩羊肉储藏过程中的细菌群落多样性[J]. 食品工业科技, 2016, 37(13): 177-182.DOI:10.13386/j.issn1002-0306.2016.13.027.
[9] 范晓攀, 王娉, 陈颖, 等. 肉类调理食品中细菌多样性的分析[J]. 现代食品科技, 2017, 33(1): 237-242. DOI:10.13982/j.mfst.1673-9078.2017.1.036
[10] 江艳华, 姚琳, 李风铃, 等. 基于高通量测序的冷冻南极磷虾中细菌菌群结构分析[J]. 食品安全质量检测学报, 2016, 7(7): 2840-2845.
[11] COSSI M V, DE ALMEIDA M V, DIAS M R, et al. Comparison of destructive and nondestructive sampling techniques of retail chicken carcasses for enumeration of hygiene indicator microorganisms[J].Journal of Food Protection, 2012, 75(1): 29-33. DOI:10.4315/0362-028X.JFP-11-247.
[12] ZHANG Q Q, YE K P, XU X L, et al. Comparison of excision,swabbing and rinsing sampling methods to determine the microbiological quality of broiler carcass[J]. Journal of Food Safety,2012, 32(1): 134-139. DOI:10.1111/j.1745-4565.2011.00360.x.
[13] GIOMBELLI A, CAVANI R, GLORIA M B. Evaluation of three sampling methods for the microbiological analysis of broiler carcasses after immersion chilling[J]. Journal of Food Protection, 2013, 76(8)∶1330-1335. DOI∶10.4315/0362-028X.JFP-13-004.
[14] CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods, 2010, 7(5)∶ 335-336. DOI∶10.1038/nmeth.f.303.
[15] BOKULICH N A, SUBRAMANIAN S, FAITH J J, et al. Qualityfiltering vastly improves diversity estimates from Illumina amplicon sequencing[J]. Nature Methods, 2013, 10(1)∶ 57-59. DOI∶10.1038/nmeth.2276.
[16] MAGOČ T, SALZBERG S L. FLASH∶ fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics, 2011, 27(21)∶2957-2963. DOI∶10.1093/bioinformatics/btr507.
[17] EDGAR R C. UPARSE∶ highly accurate OTU sequences from microbial amplicon reads[J]. Nature Methods, 2013, 10(10)∶ 996-998.DOI∶10.1038/nmeth.2604.
[18] 商宝娣, 杨星, 李正友, 等. 希瓦氏菌的研究进展[J]. 福建农业,2015(7): 152-154.
[19] 高磊, 谢晶, 叶藻, 等. 冷鲜鸡腿肉中优势腐败菌的分离鉴定及腐败能力研究[J]. 食品与发酵工业, 2015, 41(8): 48-53. DOI:10.13995/j.cnki.11-1802/ts.201508009.
[20] OLOFSSON T C, AHRNE S, MOLIN G. Composition of the bacterial population of refrigerated beef, identified with direct 16S rRNA gene analysis and pure culture technique[J]. International Journal of Food Microbiology, 2007, 118(3): 233-240. DOI:10.1016/j.ijfoodmicro.2007.07.017.
[21] 曾晓房, 林惠珍, 邝智祥, 等. 冰鲜肉中腐败菌的研究现状[J]. 安徽农业科学, 2010, 38(34): 19550-19552. DOI:10.3969/j.issn.0517-6611.2010.34.124.
[22] SALAZAR J K, WANG Y, YU S, et al. Polymerase chain reactionbased serotyping of pathogenic bacteria in food[J]. Journal of Microbiological Methods, 2015, 110∶ 18-26. DOI∶10.1016/j.mimet.2015.01.009.
[23] 江芸, 高峰, 徐幸莲, 等. 冷却猪肉不同前处理对细菌DNA提取及PCR-DGGE的影响[J]. 食品科学, 2010, 31(6): 258-262.
[24] 梁荣蓉. 生鲜鸡肉调理制品菌群结构分析和产品货架期预测模型的研究[D]. 泰安: 山东农业大学, 2010: 11-15.
[25] 蓝蔚青, 谢晶, 周会, 等. 不同时期鲳鱼冷藏期间优势腐败菌的多样性变化[J]. 食品科学, 2015, 36(2): 226-231. DOI:10.7506/spkx1002-6630-201502044.
[26] 江芸. 托盘和真空包装冷却猪肉冷藏过程中菌相变化规律研究[D].南京: 南京农业大学, 2010.
[27] 倪萍, 纪文营, 黄洁洁, 等. 冷却猪肉中热死环丝菌的分离、鉴定及致腐能力[J]. 食品科学, 2013, 34(21): 140-144. DOI:10.7506/spkx1002-6630-201321029.
[28] PENNACCHIA C, ERCIKUBU D, VILLANI F. Development of a real-time PCR assay for the specific detection of Brochothrix thermosphacta in fresh and spoiled raw meat[J]. International Journal Food Microbiology, 2009, 134(3): 230-236. DOI:10.1016/j.ijfoodmicro.2009.07.005.
[29] 孙彦雨, 周光宏, 徐幸莲. 冰鲜鸡肉贮藏过程中微生物菌相变化分析[J]. 食品科学, 2011, 32(11): 146-151.
[30] 白芸, 吴亨, 林莹. 冷却鸡肉加工工艺微生物控制及保鲜防腐的研究[J]. 食品科技, 2015, 40(8): 122-127. DOI:10.13684/j.cnki.spkj.2015.08.026.
Effects of Different Sampling Methods on Microbial DNA Extraction from Chilled Chicken and the High-Throughput Sequencing of Amplification Products
XIAO Yingping1, HE Xiangxiang2, DAI Baoling1, GUI Guohong1, TANG Biao1, YANG Hua1,*
(1. Institute of Quality and Standard for Agro-Products, State Key Laboratory Breeding Base for Zhejiang Sustainable Pest and Disease Control, Zhejiang Academy of Agricultural Sciences, Hangzhou 310021, China;2. College of Food Science and Engineering, Northwest A&F University, Yangling 712100, China)
Abstract:Objective∶ To compare the swabbing and rinsing methods used for sampling chilled chicken by evaluating their effects on microbial DNA extraction and subsequent high-throughput sequencing. Methods∶ Four chilled chickens were divided into equal halves and sampled by superficial swabbing and rinsing methods for each half, respectively. Microbial genomic DNA was extracted from the collected samples and the V3-V4 region of the bacterial 16S rRNA gene was amplified by PCR. The PCR products were then subjected to high-throughput sequencing on an Illumina HiSeq sequencing platform. The obtained sequences were processed and analyzed using QIIME and other softwares. Results∶ There was no difference in bacterial genomic DNA extraction or bacterial community richness, diversity and structures between the samples collected by the two methods (P > 0.05). High-throughput sequencing showed that the bacterial community in chilled chicken samples consisted mainly of 7 phyla and 10 genera. Proteobacteria was the dominant phylum, accounting for more than 70% of the bacterial community; Shewanella, Pseudomonas, Acinetobacter, Psychrobacter, and Brochothrix were the dominant genera, each representing 10%-20% of the bacterial community. Conclusions∶ The two sampling methods exhibit no obvious difference in bacterial DNA extraction from chilled chicken or high-throughput sequencing. However,compared with swabbing, rinsing has the advantages of easy operation and non-secondary contamination, so it is more practical in microbiological analysis of chilled chicken. The experiment also provides useful data for the study of bacteria on chilled chicken.
Key words:chilled chicken; bacterial community; swabbing method; rinsing method; high-throughput sequencing
收稿日期:2016-12-11
基金项目:浙江省公益技术运用研究项目(2016C32073);
浙江省植物有害生物防控重点实验室——省部共建国家重点实验室培育基地项目(2010DS700124-ZM1608)
作者简介:肖英平(1984—),男,助理研究员,博士,研究方向为畜产品质量安全。E-mail:ypxiaozju@126.com
*通信作者:杨华(1972—),男,高级畜牧师,硕士,研究方向为畜产品质量安全。E-mail:yanghua806@hotmail.com
DOI∶10.7506/spkx1002-6630-201724042
中图分类号:TS251.55;TS207.4
文献标志码:A
文章编号:1002-6630(2017)24-0260-05
引文格式:
肖英平, 何祥祥, 戴宝玲, 等. 采样方法对冷鲜鸡表面细菌DNA提取及高通量测序结果的影响[J]. 食品科学, 2017,38(24): 260-264.
DOI:10.7506/spkx1002-6630-201724042. http://www.spkx.net.cn
XIAO Yingping, HE Xiangxiang, DAI Baoling, et al. Effects of different sampling methods on microbial DNA extraction from chilled chicken and the high-throughput sequencing of amplification products[J]. Food Science, 2017, 38(24)∶ 260-264.(in Chinese with English abstract) DOI∶10.7506/spkx1002-6630-201724042. http∶//www.spkx.net.cn