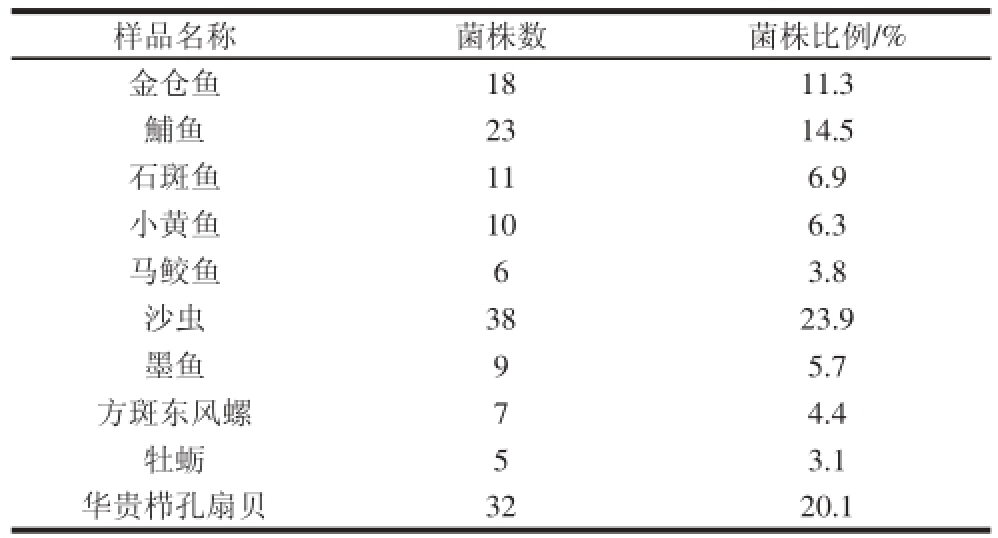
表1 不同样品中分离的菌株数
Table 1 The number of LAB strains isolated from different marine animals
焦冬冬1,任文彬2,赵鸭美3,侯小琴1,刘 颖1,*,孙力军1,王雅玲1
(1.广东海洋大学食品科技学院,广东省水产品加工与安全重点实验室,水产品深加工广东普通高等学校重点实验室,广东 湛江 524088;2.仲恺农业工程学院轻工食品学院,广东 广州 510230;3.湖州环境科技创新中心,浙江 湖州 313000)
摘 要:以湛江南海海域常见的10 种海洋动物为研究对象,采用倾注平板法分离肠道乳酸菌,以单核细胞增生李斯特氏菌(Listeria monocytogenes)等5 株有害菌为指示菌,采用双层牛津杯扩散法测定其抗菌活性,通过建 立16S rRNA基因序列的系统发育树对具有抗菌活性的乳酸菌进行多样性分析。结果表明:根据形态观察和部分生理生化实验去重复后,共分离出43 株有溶钙圈、革兰氏染色呈 阳性、过氧化氢酶反应为阴性的乳酸菌,并且均不同程度地具有抗菌活性。对其中的13 株抗菌能力强、抗菌谱广的乳酸菌进行16S rRNA分子生物学鉴定与分析,发现它们分属于厚壁菌门(Firmicutes)的4 个科6 个属,且除菌株LY-87与模式菌株Lactococcus lactis subsp. lactis的16S rRNA基因序列相似性为100%外,其他菌株与其系统发育关系最密切的典型菌株之间都存在着一定的遗传差异(相似性为95.7%~99.9%),其中菌株PQ-26与模式菌株Pediococcus pentosaceus的相似性仅为95.7%,可能是Pediococcus属中潜在的新种。研究表明湛江南海海域动物肠道中存在较为丰富的抗菌活性乳酸菌,并可能蕴藏着乳酸菌新种,其抗菌物质资源有待进一步研究与开发。
关键词:海洋动物肠道;抗菌活性;乳酸菌;16S rRNA基因;多样性
海洋作为地球上生命的发源地,占据了地球表面积的71%。海洋环境的复杂性造就了海洋动物的多样性,同时海洋环境的高压、高盐、低温、低光照、寡营养等特性也赋予了海洋动物的特殊性[1-2]。与海洋动物共附生的微生物同 样也具有复杂的多样性,尤其是肠道微生物,它们不仅对维持动物体肠道的稳态起着重要的作用,而且对一些鱼类的病原菌也有很好的抑制作用[3]。这些共附生的海洋微生物不仅种类丰富,而且在基因组成、产生的抗菌物质及抗菌机制方面也具有其自身的特殊性[4-5]。近年来,不断有科研工作者从海洋动物肠道中发现微生物新种[6],其 中乳酸菌居多,其代谢产物(如乳酸、乳酸菌素、过氧化氢等)也表现出良好的抗菌活性[7-8]。不同的代谢产物其抗菌机制也各不相同,乳酸主要是通过降低胞质内的pH值来影响细胞膜的稳定性和传递功能,乳酸菌素则是吸附在细胞膜上形成孔道,使得细胞膜的通透性增加从而引起细胞内各种离子的渗漏和能量物质的消耗,导致细胞解体死亡[9-11]。这些代谢产物为天然的食品防腐剂与药物资源的开发提供了新的线索。此外,大部分乳酸菌由于其公认的安全性[12]而被广泛应用于食品防腐保鲜领域,它不仅可以作为保护剂与潜在的病原菌或一些有害的微生物形成竞争从而增加食品的安全性,还可以利用其代谢产物的特性(如降低pH值、抗菌活性等)来抑制多种微生物的生长从而延长食品的货架期[13]。
南海作为中国最广阔的海域,属于热带海洋性季风气候,是海洋生物聚居的天堂。2013—2014年,刘颖等[14]对湛江南海硇洲岛海域生活的褐蓝子鱼肠道中的微生物进行了分离和鉴定,发现其肠道具有较丰富的细菌多样性,并分离到了2 株具有抗菌活性的乳酸菌。本研究对广东湛江南海海域东海岛的10 种海洋动物进行肠道乳酸菌的分离与抗菌活性评价,采用16S rRNA基因序列的系统发育分析对抗菌活性菌株进行多样性分析,该研究为新型天然食品防腐剂资源的开发与利用提供一定的理论基础与实践指导。
1.1 材料
1.1.1 样品
10 种海洋动物均采集于湛江东海岛(20°55′~21°55′N,110°11′~110°21′E),分别为5 种海洋脊椎动物(金仓鱼、鯆鱼、石斑鱼、小黄鱼、马鲛鱼)、2 种无脊椎动物(沙虫、墨鱼)和3 种贝类(方斑东风螺、牡蛎、华贵栉孔扇贝)。
1.1.2 指示菌
大肠埃希氏菌(Escherichia coli ATCC 25922)、副溶血性弧菌(Vibrio parahemolyticus ATCC 17802)、金黄色葡萄球菌(Staphylococcus aureus ATCC 6538)、单核细胞增生李斯特氏菌(Listeria monocytogenes ATCC 19111)、枯草芽孢杆菌(Bacillus subtilis ATCC 6633)。
1.1.3 试剂
分离培养基(ATP培养基、MRS固体培养基、Elliker琼脂培养基、D-MRS培养基、SL培养基、M17培养基)、发酵培养基(MRS液体培养基)、指示菌培养基(胰酪胨大豆酵母浸膏琼脂、3%氯化钠营养琼脂、营养琼脂),培养基配方详见文献[15]。
革兰氏染色试剂盒、生化鉴定管 北京陆桥技术有限责任公司;MightyAmp DNA 聚合酶Ver.2试剂盒 宝生物工程有限公司;16S rRNA细菌通用引物(27F:5-AGAGTTTGATCCTGGCTCAG-3,1492R:5-GGTTACCTTGTTACGACTT-3) 生工生物工程(上海)股份有限公司。
1.1.4 仪器与设备
2-16KL台式冷冻离心机 德国Sigma公司;MultiGeneTM梯度聚合酶链式反应(polymerase chain reaction,PCR)仪 美国Labnet公司;L-08无菌均质器上海皓庄仪器有限公司。
1.2 方法
1.2.1 样品处理
样品采集后立即用冰盒运回实验室,用无菌水将样品表面冲洗3 遍,沥干水分后快速取出样品的胃肠道,称取10 g并置于盛有90 mL无菌生理盐水的均质袋中于均质器中拍打1~2 min,得到样品匀浆悬浮液。
1.2.2 乳酸菌的分离及发酵上清液的制备
吸取0.1 mL适宜稀释度的菌悬液分别涂布于6 种不同的含0.05% CaCO3的分离培养基上,30 ℃培养。对有溶钙圈的菌落进行反复分离纯化,根据菌落形态及镜检结果去掉重复。获得的分离菌株进行革兰氏阳性和触酶实验,并挑取目标菌1~2 环接种到100 mL MRS液体培养基中30 ℃培养48 h,发酵液12 000 r/min离心10 min,收集上清液测定其酸度,并备用于抑菌实验。
1.2.3 抗菌活性测定
通过双层牛津杯扩散法测定发酵上清液的抑菌活性[16]。将指示菌S. aureus、B. subtilis与E. coli接种于营养琼脂培养基,L. monocytogenes接种于TSA-YE培养基,V. parahemolyticus接种于3%氯化钠营养琼脂培养基,均于37 ℃静置培养24 h。在灭菌培养皿中加入50~60 ℃条件下的2.0%素琼脂,冷却凝固后,将灭菌牛津杯均匀地置于素琼脂中,再倒入含108CFU/mL指示菌且冷却至60 ℃左右的相应培养基15 mL,待凝固后,用镊子拔出牛津杯。向牛津杯孔中加入200 μL分离的乳酸菌的发酵上清液,于4 ℃冰箱中扩散24 h后将其移至37 ℃培养24 h,观察是否有抑菌圈并测量其直径的大小。
1.2.4 活性菌株的生理生化鉴定
参考《伯杰氏细菌鉴定手册》[17]、《常见细菌系统鉴定手册》[18]和《乳酸细菌分类鉴定及实验方法》[19],对分离菌株进行部分生理生化实验初步鉴定其种属。
1.2.5 活性菌株的16S rRNA分子生物学鉴定与系统发育分析
分别以2 7 F、1 4 9 2 R为正、反向引物,使用M i g h t y A m p D N A聚合酶对活性菌株进行菌落PCR,PCR体系与反应条件按照Lu Qingfang等[20]的方法。P C R体系(3 0 μ L):以培养皿中的单菌落作为模板,2×M i g h t y A m p b u f f e r 15 μL,MightyAmp DNA聚合酶 0.75 μL,27F(10 μmol/μL)0.75 μL,1492R(10 μmol/μL)0.75 μL,ddH2O 12.75 μL。反应条件:98 ℃预变性2 min;98 ℃变性10 s,55 ℃退火15 s,68 ℃延伸1.5 min,40 个循环;10 ℃保存10 min。取3 μL PCR产物进行1.0%的琼脂糖凝胶电泳检测,以DL2000 DNA Marker为标样,电泳结束后在ZF-20D暗箱式紫外分析仪中观察条带并判断其分子质量。对扩增成功的PCR产物寄送生工生物工程(上海)股份有限公司进行测序。
根据测得的1 6 S r R N A基因序列,从N C B I(GenBank)和EzBioCloud(EzTaxon)数据库进行同源性搜索,下载相似性高的模式菌株的16S rRNA基因序列,用MEGA 5.03软件包采用邻接法(Neighbor-Joining method)进行聚类分析并构建16S rRNA基因系统发育树[21],系统进化树的拓扑结构稳定性评估参照文献[22]所用方法进行。
2.1 乳酸菌的分离
表1 不同样品中分离的菌株数
Table 1 The number of LAB strains isolated from different marine animals

根据对溶钙圈、菌落形态 的观察,从样品中共分离出159 株菌株。由表1可知,不同的海洋动物肠道中分离到的微生物数量存在一定的差异性,其中从无脊椎动物沙虫的肠道中分离的菌株最多(38 株),占总分离菌株数的23.9%,依次为华贵栉孔扇贝(32 株,20.1%)、鯆鱼(23 株,14.5%)、金仓鱼(18 株,11.3%)、石斑鱼(11 株,6.9%)、小黄鱼(10 株,6.3%)、墨鱼(9 株,5.7%)、方斑东风螺(7 株,4.4%)、马鲛鱼(6 株,3.8%)、牡蛎(5 株,3.1%)。按革兰氏染色为阳性、触酶反应为阴性及发酵液pH值呈酸性的乳酸菌筛选标准[23-24],鉴定其中的43 株为乳酸菌,占总分离菌株的27.0%。
2.2 抗菌活性测定结果
采用双层牛津杯扩散法对43 株乳酸菌进行抗菌活性测定(表2),结果发现43 株乳酸菌均表现出一定的抗菌活性,其中,对E. coli、V. parahaemolytics 2 种G-指示菌均有抗菌活性的菌株23 株,占总数的53.5%;对S. aureus、L. monocytogens、B. subtilis 3 种G+指示菌均有抗菌活性的菌株有8 株,占总数的18.6%。其中,菌株ZH-11、YM-12、ZH-39、ZH-48、ZH-54、ML-67、ML-70、LY-87对5 种指示菌有抗菌活性,占18.6%;菌株LY-45、YM-49、ZH-53、LY-81、ZH-103、YM-104、LY-121对4 种指示菌有抗菌活性,占16.3%;菌株PQ-26、ZH-44、LY-51、ZY-55、ZY-58、ZY-80、YM-84、LY-100、YM-117、LY-120、ZH-146对3 种指示菌有抗菌活性,占25.6%;菌株YM-6、YM-15、LY-95、YM-105、YM-114、YM-118、YM-138、LY-140、ZH-142、ZH-143、ZY-144、ZY-145、ZH-147对2 种指示菌有抗菌活性,占27.9%;仅菌株ZY-38、LY-97、ZY-133、ZH-141对一种指示菌有抗菌活性,占9.3%。因此,可以看出从海洋动物肠道分离的乳酸菌具有抗菌活性强、抗菌谱广的特点。
表2 43 株活性乳酸菌对不同指示菌的抑菌作用
Table 2 Antimicrobial activities of 43 strains of LAB against different indicator bacteria
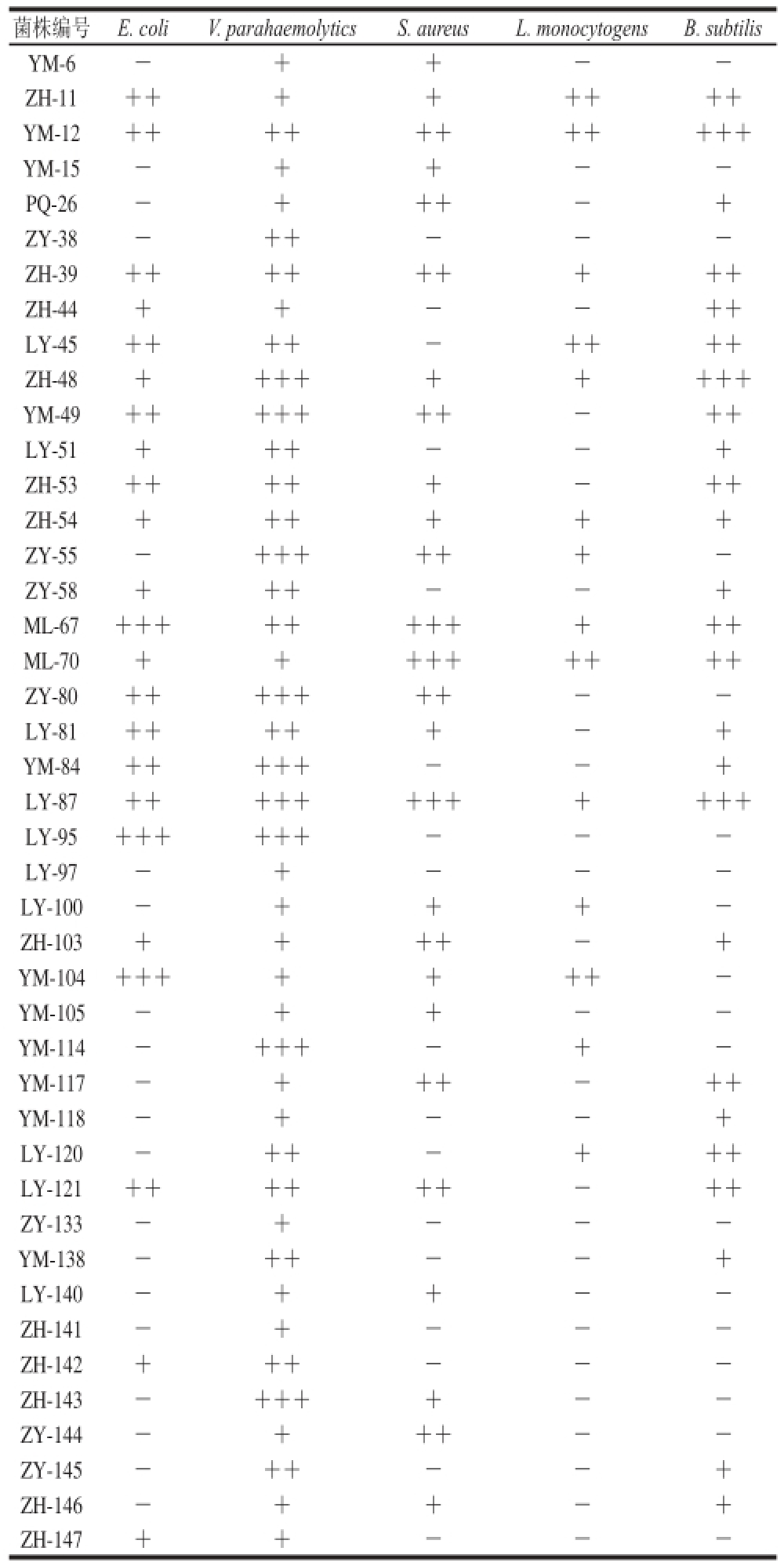
注:+++.抑菌圈直径>20 mm;++. 14 mm≤抑菌圈直径≤20 mm;+.抑菌圈直径<14 mm;-.无抑菌活性。
2.3 活性菌株的形态与生理生化鉴定
表3 43 株乳酸菌生理生化鉴定结果
Table 3 Identification of the 43 strains of LAB based on physiological and biochemical characteristics
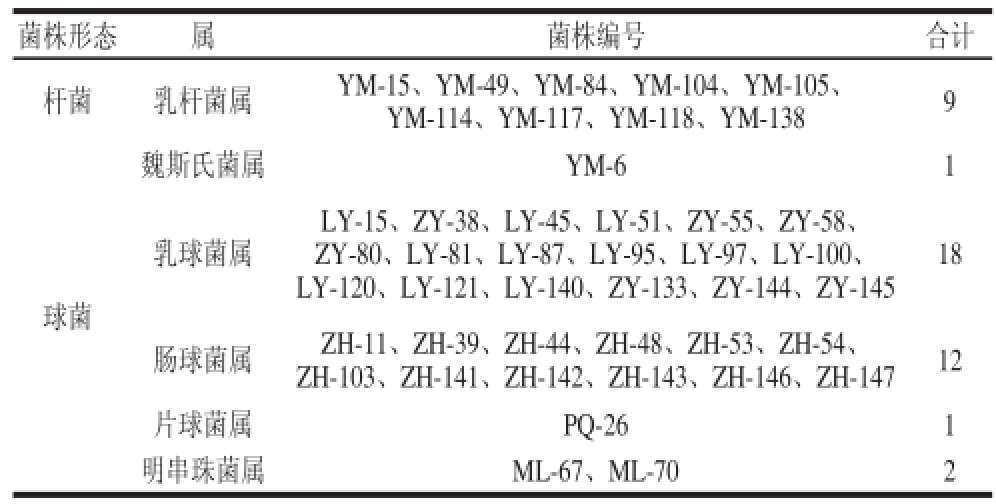
显微形态观察,43 株乳酸菌分为杆菌和球菌2 种。结合生化鉴定(表3),发现杆菌主要是乳杆菌属(9 株,20.9%);球菌包括乳球菌属(18 株,41.9%)、肠球菌属(12 株,27.9%)、明串珠菌属(2 株,4.7%)、片球菌属(1 株,2.3%)和魏斯氏菌属(1 株,2.3%)。其中,乳球菌属、肠球菌属和乳杆菌属3 个属占活性乳酸菌总数的90.7%,为该研究中乳酸菌的优势菌属。
2.4 抗菌活性菌株的16S rRNA分子生物学鉴定与分析
根据表2、3结果,选择其中13 株抗菌活性强、抗菌谱广的乳酸菌进行菌落16S rRNA扩增与测定。经生工生物工程(上海)股份有限公司测定,PCR产物的分子大小均在1 500 bp左右,属于典型的16S rRNA分子的特征,说明13 株乳酸菌的16S rRNA基因序列扩增成功。
2.5 抗菌活性菌株类群的多样性
从NCBI(GenBank)和EzBioCloud(EzTaxon)数据库对13 株抗菌活性乳酸菌的16S rRNA基因序列进行相似性搜索,调出相似性最高的模式菌株(表4)。结果表明,13 株乳酸菌均属于细菌界的厚壁菌门(Firmicutes),并分属于4 个科(Enterococcaceae、Streptococcaceae、Leuconostocaceae、Lactobacillaceae)中的6 个属(Enterococcus、Lactococcus、Weissella、Leuconostoc、Lactobacillus、Pediococcus),表现出较高的种属多样性。其中,菌株ZH-53、ZH-54属于肠球菌属(Enterococcus),占乳酸菌总数的15.4%;菌株LY-51、LY-87、ZY-55、ZY-58、ZY-144属于乳球菌属(Lactococcus),占38.5%;菌株YM-6属于魏斯氏菌属(Weissella),占7.7%;菌株ML-67、ML-70属于明串珠菌属(Leuconostoc),占15.4%;菌株YM-49、YM-104属于乳杆菌属(Lactobacillus),占15.4%;菌株PQ-26属于片球菌属(Pediococcus),占7.7%。
表4 13 株乳酸菌与其系统发育关系最密切的典型菌株间的系统发育关系
Table 4 Closest phylogenetic neighbors of 13 strains of LAB determined based on their partial 16S rRNA sequences

注:T.模式菌株。图1同。
2.6 物种与遗传差异性

图1 13 株乳酸菌的16S rRNA与相关菌种的系统发育进化树
Fig. 1 Phylogenetic tree based on the 16S rRNA of the 13 strains of LAB
将13 株乳酸菌的16S rRNA序列用软件MEGA 5.03按邻接(Neighbor-Joining)法构建系统发育树。由图1可知,13 株菌在系统发育树上没有聚集,且较分散分布于不同的分枝,再根据表4菌株相似性比对结果,发现除了菌株LY-87与相似性最高模式菌株Lactococcus lactis subsp. lactis JCM 5805T的16S rRNA基因序列100%相似外,其他乳酸菌与其对应的相似性最高模式菌株的16S rRNA基因序列相似性在95.7%~99.9%之间,说明大部分菌株与其系统发育关系最密切的相关菌株之间存在不同程度的遗传差异。其中,菌株PQ-26与Pediococcus pentosaceus DSM 20336T之间的16S rRNA基因序列相似性仅为95.7%,按16S rRNA基因序列相似性小于97%的菌株是属于不同种类的微生物的归类原则[25],再结合形态和生理生化实验结果,推测其可能为Pediococcus属中的新种,其确认需进一步研究。
本研究从南海东海岛10 种常见的海洋动物肠道中共分离出43 株乳酸菌,经测定它们均不同程度地具有抗菌活性,其中对E. coli、V. parahaemolytics、S. aureus、L. monocytogens、B. subtilis 5 种指示菌均有抗菌活性的菌株有8 株,占乳酸菌总数的18.6%。通过对其中13 株抗菌活性强、抗菌谱广的菌株进行16S rRNA分子生物学鉴定及系统发育分析得出,它们属于1 个大的系统发育类群(Firmicutes)的4 个科、6 个属(Enterococcus、Lactococcus、Weissella、Leuconostoc、Lactobacillus、Pediococcus),显示出较丰富的生物多样性,其中Lactococcus(38.5%)为优势菌属。崔美岩等[26]从青海湖裸鲤肠道中分离出47 株乳酸菌,有13 株(27.7%)对所选6 种指示菌均有抗菌活性,属于厚壁菌门(Firmicutes)的4 个科、5 个属(Lactobacillus、Leuconostoc、Weissella、Lactococcus、Enterococcus),Lactobacillus为优势菌属。任士菊等[27]从冷水鱼肠道分离出的24 株乳酸菌,具有明显抑菌活性的有9 株,占总分离菌株数的37.5%,属于Lactococcus(6 株)和Enterococcus(3 株)2 个属。
此外,大部分菌株与其系统发育关系最密切的模式菌株之间的基因序列存在一定的差异(相似性为95.7%~99.9%),原因可能是模式菌株为陆生而分离的菌株为水生,生存环境的特殊性造就了它们之间的差异性。从16S rRNA基因相似性比较以及系统发育树分析,菌株PQ-26可能为一株潜在的乳酸菌新种,有望为新型抗菌活性物质资源的开发提供较大的可能。
参考文献:
[1] 刘玉娟, 田新朋, 黄小芳, 等. 中国南海沉积环境可培养细菌多样性研究[J]. 微生物学通报, 2014, 41(4): 661-673. DOI:10.13344/ j.microbiol.china.130257.
[2] 李艳华, 张利平. 海洋微生物资源的开发与利用[J]. 微生物学通报, 2003, 30(3): 113-114. DOI:10.3969/j.issn.0253-2654.2003.03.028.
[3] LEE H I, KIM M H, KIM K Y, et al. Screening and selection of stress resistant Lactobacillus spp. isolated from the marine oyster (Crassostrea gigas)[J]. Anaerobe, 2010, 16(5): 522-526. DOI:10.1016/ j.anaerobe.2010.07.002.
[4] RONDON M R, AUGUST P R, BETTERMANN A D, et al. Cloning the soil metagenome: a strategy for accessing the genetic and functional diversity of uncultured microorganisms[J]. Applied and Environmental Microbiology, 2000, 66(6): 2541-2547. DOI:10.1128/ AEM.66.6.2541-2547.2000.
[5] HENRIQUEZ M, VERGARA K, NORAMBUENA J, et al. Diversity of cultivable fungi associated with antarctic marine sponges and screening for their antimicrobial, antitumoral and antioxidant potential[J]. World Journal of Microbiology and Biotechnology, 2014, 30(1): 65-76. DOI:10.1007/s11274-013-1418-x.
[6] DEWAPRIYA P, KIM S K. Marine microorganisms: an emerging avenue in modern nutraceuticals and functional foods[J]. Food Research International, 2014, 56(2): 115-125. DOI:10.1016/ j.foodres.2013.12.022.
[7] FRANÇOISE L. Occurrence and role of lactic acid bacteria in seafood products[J]. Food Microbiology, 2010, 27(6): 698-709. DOI:10.1016/ j.fm.2010.05.016.
[8] 李婷, 满朝新, 李琳瑶, 等. 乳酸菌在食品防腐中的应用[J]. 中国食物与营养, 2013, 19(7): 34-37. DOI:10.3969/j.issn.1006-9577.2013.07.009.
[9] 李铁军, 李爱云, 张晓峰. 乳酸菌抗菌机理研究进展[J]. 微生物学通报, 2002, 29(5): 81-85. DOI:10.3969/j.issn.0253-2654.2002.05.020.
[10] 刘翀, 杨洋. 安全的天然食品防腐剂细菌素[J]. 食品科学, 2005, 26(7): 251-255. DOI:10.3321/j.issn:1002-6630.2005.07.062.
[11] 赵娜, 刘鑫, 石和平, 等. 乳酸菌抗菌物质分类及作用机理[J].农产品加工, 2015(5): 58-60. DOI:10.3969/jissn.1671-9646(X).2015.05.053.
[12] SILVA J, CARVALHO A S, TEIXEIRA P, et al. Bacteriocin production by spray-dried lactic acid bacteria[J]. Letters in Applied Microbiology, 2002, 34(2): 77-81. DOI:10.1046/j.1472-765x.2002.01055.x.
[13] PILAR C M, SAMUEL A, KAROLA B, et al. Current applications and future trends of lactic acid bacteria and their bacteriocins for the biopreservation of aquatic food products[J]. Food and Bioprocess Technology, 2008, 1(1): 43-63. DOI:10.1007/s11947-007-0021-2.
[14] 刘颖, 时威, 安俊莹, 等. 硇洲岛褐蓝子鱼肠道抗菌活性菌株的分离及多样性[J]. 微生物学报, 2014, 54(5): 582-588. DOI:10.13343/ j.cnki.wsxb.2014.05.013.
[15] 赵鸭美. 产乳酸菌素乳酸菌的筛选及对对虾防腐效果的初步研究[D].湛江: 广东海洋大学, 2013: 11-12.
[16] 刘冬梅, 李理, 杨晓泉, 等. 用牛津杯法测定益生菌的抑菌活力[J]. 食品研究与开发, 2006, 27(3): 110-111. DOI:10.3969/ j.issn.1005-6521.2006.03.044.
[17] BUDANAN R E, GIBBONR N E. 伯杰细菌鉴定手册[M]. 8版. 北京:科学出版社, 1984: 136-357.
[18] 东秀珠, 蔡妙英. 常见细菌系统鉴定手册[M]. 北京: 科学出版社, 2001: 349-370. DOI:10.1093/clinids/24.1.41.
[19] 凌代文. 乳酸细菌分类鉴定及实验方法[M]. 北京: 中国轻工业出版社, 1999: 1-129.
[20] LU Q F, HU H Q, MO J J, et al. Enhanced amplifi cation of bacterial and fungal DNA using a new type of DNA polymerase[J]. Australasian Plant Pathology, 2012, 41(6): 661-663. DOI:10.1007/s13313-012-0144-4.
[21] ZHANG W, SUN Z R. Random local neighbor joining: a new method for reconstructing phylogenetic trees[J]. Molecular Phylogenetics and Evolution, 2008, 47(1): 117-128. DOI:10.1016/j.ympev.2008.01.019.
[22] FELSENSTEIN J. Confidence limits on phylogenies: an approach using the bootstap[J]. Evolution, 1985, 39(4): 783-791. DOI:10.2307/2408678.
[23] HWANHLEM N, BURADALENG S, WATTANACHANT S, et al. Isolation and screening of lactic acid bacteria from Thai traditional fermented fish (Plasom) and production of Plasom from selected strains[J]. Food Control, 2011, 22(3/4): 401-407. DOI:10.1016/ j.foodcont.2010.09.010.
[24] ONDA T, YANAGIDA F, UCHIMURA T, et al. Widespread distribution of the bacteriocin-producing lactic acid cocci in Misopaste products[J]. Applied Microbiology, 2002, 92(4): 695-705. DOI:10.1046/j.1365-2672.2002.01573.x.
[25] STACKEBRANDT E, GOEBEL B M. Taxonomic note: a place for DNA-DNA reassociation and 16S rRNA sequence analysis in the present species definition in bacteriology[J]. Systematic and Evolutionary Microbiology, 1994, 44(4): 846-849. DOI:10.1099/00207713-44-4-846.
[26] 崔美岩, 吕好新, 张志霞, 等. 青海湖裸鲤肠道乳酸菌多样性与抑菌活性[J]. 微生物学通报, 2016, 43(9): 1-14. DOI:10.13344/j.microbiol. china.150834.
[27] 任士菊, 王俊钢, 周红, 等. 冷水鱼肠道抑菌活性乳酸菌的筛选及其生理特性分析[J]. 食品工业科技, 2014, 35(14): 178-181; 185. DOI:10.13386/j.issn1002-0306.2014.14.031.
Separation and Diversity of Lactic Acid Bacteria Possessing Antimicrobial Activity Isolated from the Intestine of Marine Animals in South China Sea
JIAO Dongdong1, REN Wenbin2, ZHAO Yamei3, HOU Xiaoqin1, LIU Ying1,*, SUN Lijun1, WANG Yaling1
(1. Key Laboratory of Aquatic Product Processing and Safety of Guangdong, Key Laboratory of Advanced Processing of Aquatic Products of Guangdong Higher Education Institution, College of Food Science and Technology, Guangdong Ocean University, Zhanjiang 524088, China; 2. College of Food Science and Technology, Zhongkai University of Agriculture and Engineering, Guangzhou 510230, China; 3. Huzhou Environmental Science and Technology Innovation Center, Huzhou 313000, China)
Abstract:Lactic acid bacteria (LAB) were isolated from the intestine of 10 common kinds of marine animals collected from the South China Sea by the pour plate method. The antimicrobial activity of the LAB isolates was determined by Oxford cup diffusion method against fi ve pathogenic indicator strains including E. coli, V. parahaemolytics, S. aureus, B. subtilis, and L. monocytogenes. The diversity analysis of these LAB strains was conducted by establishing phylogenetic tree based on 16S rRNA gene sequences. The results showed that 43 isolates, which presented soluble calcium circle and were positive by Gram staining and negative in catalase reaction, were identifi ed as LAB according to morphological observation and partial physiological and biochemical experiments, and they all possessed antimicrobial activity in different degrees. Out of these, 13 strains were found to have stronger bacteriostatic ability and a wider antimicrobial spectrum, and they were further identifi ed based on phyogenetical analysis of 16S rRNA sequence. The results indicated that they belonged to 6 genera in 4 families of Firmicutes. Although strain LY-87 and the type strain of Lactococcus lactis subsp. lactis shared 100% 16S rRNA gene sequence sim ilarity, there were genetic differences between other LAB strains and the typical strains showing the closest phylogenetic relationship with them (similarity ranging from 95.7% to 99.9%). The similarity between strain PQ-26 and the type strain of Pediococcus pentosaceus was only 95.7% suggesting that it is likely to be a potential new speciesof Pediococcus. This research indicates that LAB with antimicrobial activity are diverse and there may exist potential new species in the intestine of animals from the South China Sea in Zhanjiang. The antimicrobial substances produced by these bacteria need to be further studied and developed.
Key words:marine animal intestine; antimicrobial activity; lactic acid bacteria; 16S rRNA gene; diversity
收稿日期:2016-05-31
基金项目:广东省科技计划项目(2014A020217018;2016A020222014);
广东省水产品加工与安全重点实验室开放基金课题(GDPKLAPPS1503);国家自然科学基金面上项目(31371777;31171634)
作者简介:焦冬冬(1991—),男,硕士研究生,研究方向为海洋生物活性物质研究与开发。E-mail:jddhxq@163.com
*通信作者:刘颖(1966—),女,教授,博士,研究方向为食品质量与安全。E-mail:liuyingxk@sina.com
DOI:10.7506/spkx1002-6630-201708013
中图分类号:Q938
文献标志码:A
文章编号:1002-6630(2017)08-0074-06
引文格式:
焦冬冬, 任文彬, 赵鸭美, 等. 南海海域动物肠道中抗菌活性乳酸菌的分离及多样性分析[J]. 食品科学, 2017, 38(8): 74-79. DOI:10.7506/spkx1002-6630-201708013. http://www.spk x.net.cn
JIAO Dongdong, REN Wenbin, ZHAO Yamei, et al. Separation and diversity of lactic acid bacteria possessing antimicrobial activity isolated from the intestine of marine animals in South China Sea[J]. Food Science, 2017, 38(8): 74-79. (in Chinese with English abstract)
DOI:10.7506/spkx1002-6630-201708013. http://www.spkx.net.cn