
图1 1996—2015年间以“Predictive Microbiology”为主题每年出版的文献数量
Fig.1 Annual number of published literature with “Predictive Microbiology”as the theme keyword from 1996 to 2015
张昭寰1,娄 阳1,杜苏萍1,潘迎捷1,2,3,赵 勇1,2,3,*
(1.上海海洋大学食品学院,上海 201306;2.上海水产品加工及贮藏工程 技术研究中心,上海 201306;3.农业部水产品贮藏保鲜质量安全风险评估实验室,上海 201306)
摘 要:预测微生物学是食品微生物学的重要组成部分,其本质在于利用数学模型描述特定环境条件下微生物的生长和死亡规律。预测微生物模型既能应用于预测食品的货架期、控制腐败菌的滋生,又有助于完善食品微生物风险评估体系,减少致病菌的患病风 险,对保障食品安全和改善公共卫生状况具有十分重要的意义。本文以综述的形式,概述预测微生物学的发展历史,并分析当前预测微生物学的研究热点。在此基础之上,着重介绍分子生物学技术在预测微生物学中应用的最新研究进展,阐述分子预测模型的概念和构建方法,并对其他分子生物学技术在预测微生物学中应用的可行性以及分子预测模型的应用前景进行展望,以期为全面推动预测微生物学这一学科的进步提供理论参考。
关键词:分子生物学技术;预测微生物学;风险评估;食品安全;货架期
食品安全问题是全世界密切关注的热点问题,而由微生物导致的食源性疾病和食品腐败变质问题则被视为食品安全问题的重中之重[1-2]。预测微生物学(predictive microbiology)是利用数学模型描述在特定环境条件下食源性微生物的生长、残存、死亡动态的一门科学。它结合了微生物学、化学、数学、统计学和应用计算机等交叉学科间的技术[3-5]。预测微生物模型既能用于预测食品的货架期、控制腐败微生物的数量,又有助于完善食品微生物风险评估体系,减少致病微生物的患病风险,对于保障食品质量安全和改善公共卫生状况具有十分重要的意义。
传统预测微生物模型的构建通常基于微生物培养的方法,被视为构建预测模型的“金标准”。然而,这种方法操作繁琐、费时费力,而且难以描述复杂食品基质条件下微生物的消长情况,往往只局限于单一微生物在纯培养或消除本底微生物食品中的消长情况,这明显不符合实际食品中真实微生物的动态变化规律[5]。随着科学技术的迅猛发展,研究人员开始将分子生物学技术应用于预测微生物学的研究之中:Reichert-Schwillinsky等[6]首先使用实时荧光定量聚合酶链反应(real-time polymerase chain reaction,Real-time PCR)技术描述了培养基中单增李斯特菌的生长情况,随后,Ye Keping[7-8]、孙文烁[9]、Zhang Zhaohuan[10]等分别沿用了这一方法,构建了多种食源性致病菌在食品中的预测模型;Liao Chao等[11]尝试将PCR结合变性梯度凝胶电泳(PCR-denaturing gradient gel e lectrophoresis,PCR-DGGE)技术应用于预测模型的构建;Parveen等[12]运用DNA探针的方法构建预测微生物模型;Korem等[13]利用宏基因组学测序技术分析人类肠道菌群的生长速率。这些技术不仅高效快速,而且能够提供传统培养方法无法量化的信息,弥补了传统预测微生物模型的不足。现阶段,以分子生物学技术所构建的预测微生物模型已受到国内外预测微生物学专家的认可,具有巨大的发展潜力和应用前景。
本文通过绘制预测微生物学发展史的时间轴,回顾了预测微生物学的发展历程,并总结了当前预测微生物学的三大研究热点。并以此为基础,着重介绍了分子生物学技术在预测微生物学中的应用,对其未来的发展做出了展望,以期提供全面系统的信息阐释分子预测模型的概念和构建方法,为预测微生物学的进一步发展提供良好的理论基础。
在信息检索平台Web of Science的“Web of ScienceTM核心合集”中,以“Predictive Microbiology”作为“主题”关键词进行检索,结果显示:截止至2015年12月31日,1996—2015年20 年间共出版了1 126 篇相关文献。以“文献出版年份”为X轴、“每年出版文献数量”为Y轴进行作图,结果如图1所示,最初4~6 年预测微生物学的发展较为缓慢,年文章数仅为30 篇左右。随后,预测微生物学进入蓬勃发展阶段,年文章数量以每年4 篇的速率逐年递增。在2011—2015年间达到稳定,平均每年有76 篇相关文献出版。

图1 1996—2015年间以“Predictive Microbiology”为主题每年出版的文献数量
Fig.1 Annual number of published literature with “Predictive Microbiology”as the theme keyword from 1996 to 2015

图2 预测微生物学发展史上的重要事件[55--3355]]
Fig.2 The important events in the history of predictive microbiology[5-35]
同时绘制了预测微生物学发展历程的时间轴,如图2所示,通过罗列预测微生物学发展史上的“大事件”,从历史的角度回顾了这一学科的“前世今生”,并将预测微生物学的发展史分为4 个阶段进行综述,分别为预测微生物学的萌芽、预测微生物学的开端、预测微生物学的发展以及预测微生物学当前的研究热点。
1.1 预测微生物学的萌芽
如图2所示,预测微生物学的萌芽最早源于20世纪初期。Esty等[14]应用数学方程研究了肉毒梭菌在热作用下的失活动力学。Scott[15]创新性地提出了预测微生物学的模糊概念:应当推行一种新的方法主动地提升微生物安全和食品质量,而不是通过琼脂培养的方法进行回顾性的评价。Ball出版有关食品杀菌技术原理的专著,首次定义了D值这一经典的杀菌动力学指标[16-17]。Frederickson等[18]创办Mathematical Biosciences杂志,刊登了许多有关微生物动力学的早期研究。
1.2 预测微生物学的开端
20世纪80年代初,计算机技术的日益成熟为预测微生物学的发展提供了先决条件。同时,由于细菌性食物中毒事件的频发,政府和公众都急需一种行之有效的措施用以控制食品中微生物污染,因此,预测微生物学应运而生。Roberts和Jarvis提出了“食品预测微生物学(food predictive microbiology)”这一名词作为学科名称[3],为长达60多年的“萌芽”阶段做出了形象生动、准确具体的总结。同年,一支由30 位微生物学家组成的食品小组,用计算机预测了食品的货架期,并建立了腐败菌生长的数据库,这一历史事件被视为预测微生物学研究的正式开始[19]。
1.3 预测微生物学的发展历程
随着预测微生物学的诞生,20世纪80—90年代间,预测微生物学专家们探究或发明了许多预测微生物模型,并随之出现了多种模型的分类方法,目前认可度最高的是Whiting等[20]在1993年提出的基于变量类型的分类方法,该方法将预测模型分为3 个层次:一级、二级和三级模型。本文结合这种分类方法与图2,对预测微生物学的发展历程进行简要的介绍。
1.3.1 一级模型
一级模型主要用于描述在特定的环境条件下,微生物数量与时间之间的函数关系[20-21]。由于微生物随时间变化通常呈现S型的生长趋势,因此,S型的函数最常用于拟合微生物的生长动力学。预测微生物学发展至今,涌现了大量S型的方程,主要包括:修正Gompertz方程、修正Logistic方程、Baranyi方程等。Gompertz模型是20世纪80年代末至90年代初应用最普遍的S型方程, Gibson等[22]首先将Gompertz方程应用于肉毒梭菌生长动力学的描述。Zwietering等[23]比较了Gompertz、Richards和Logistic等方程,得出修正的Gompertz方程能够更好地描述微生物的生长动力学,修正后的方程如下:
式中:Nt是在时间t时微生物数量;A是随时间无限减小时的渐进值(相当于初始菌数);C是随时间无限增加时菌的增量,即是初始菌数和最大菌数之间的差值;e为2.718;μmax为最大比生长速率;λ为微生物生长的延滞期。
另一个应用较为广泛的模型是Baranyi等[24]提出的Baranyi & Roberts模型(简称Baranyi模型),其表达式为:
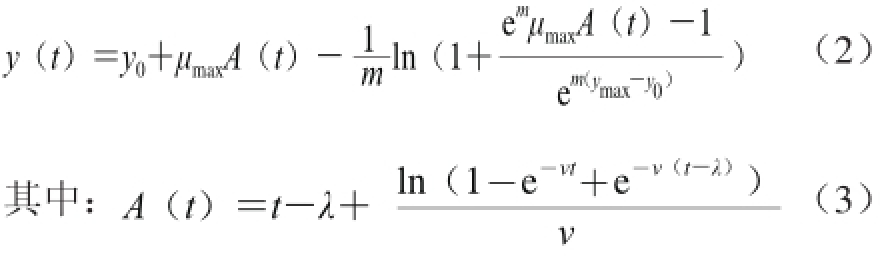
式中:y(t)是在时间t时微生物数量的对数值;y0是初始微生物浓度;μmax为最大比生长速率;λ为微生物生长的延滞期;v和m是模型的调节参数。
1.3.2 二级模型
二级模型主要用于描述不同环境(如温度、盐度、pH值、水分活度等)对初级模型的参数(如生长速率、延滞期等)的影响,目前,比较常用的二级模型主要包括:平方根方程、Arrhenius公式、响应面方程、主参数模型等[20-21]。平方根模型是现今最为常用的二级预测模型,由Ratkowsky等[25]在1982年提出,最初用于描述微生物的生长速率(或延滞期)随温度变化的情况,具体方程如下:
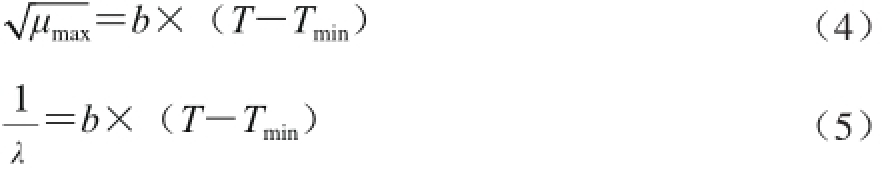
式中:μmax为生长速率;λ为微生物生长的延滞期;b为平方根模型的系数;T为微生物培养温度/℃;Tmin为微生物最低生长温度/℃。
最初的平方根模型只适用于描述微生物生长速率随单一参数(温度)变化而变化的情况[26],平方根模型的其他几位发明者McMeekin等[27]综合水分活度、pH值和温度等多个参数,提出了新的平方根模型,该模型的提出为今后多参数模型的构建提供了一个经典的范例,具体方程如下:
式中:μmax为生长速率;aw为水分活度;awmin为微生物生长的最小水分活度;pHmin为微生物生长的最小pH值;T为微生物培养温度/℃;Tmin为微生物最低生长温度/℃。
尽管经过多次修正和拓展,在拟合多种因素共同影响微生物生长时,平方根模型还是显现出一定的局限性。而另外一种常用的二级模型—响应面模型[28-29],被更多地应用于多种因素共同影响微生物生长的拟合。较平方根模型而言,响应面模型的形式更为复杂,但是能够较好地解决多因素存在条件下模型拟合准确性的问题,所以得到了较为广泛的应用[30-31]。
1.3.3 三级模型
三级模型是指将一级模型和二级模型整合在一起,并通过计算机软件的形式展现出来,即预测微生物软件[20-21,32]。目前已经开发出多款预测微生物软件,其中最为著名的是由英国、美国与澳大利亚共同支持和维护的预测微生物学平台:ComBase,其登录网址为www.combase.cc。国内外其他机构也构建了一些预测微生物软件[21]:如加拿大开发的微生物动态专家系统(microbial kinetics expert system,MKES);丹麦水产研究院开发的海产品食品货架期软件(seafood spoilage and safety predictor software,SSSP);中国水产科学研究院的鱼类货架期系统;南京农业大学的冷却猪肉微生物预测软件;上海海洋大学的猪肉货架期预测系统和副溶血性弧菌预测系统。
1.4 预测微生物学当前研究的热点
2006年ComBase正式成立并免费公开之后,研究人员具备了完善的预测微生物学理论和有效的模型构建工具,预测微生物学因此进入了蓬勃发展的阶段。如图1所示,2006—2015年10 年间共发表SCI论文709 篇,文章数量较之前的一个10 年(1996—2005年)多了将近1 倍。2006—2015年,预测微生物学展现出多元化的发展趋势,当前主要的研究热点分为以下3 个方面:1)定量微生物风险评估:van Gerwen等[35]在第84届国际食品、乳品和环境卫生协会(International Association of Milk, Food and Environmental Sanitarians,IAMFES)年会上提出微生物预测模型可以应用于定量微生物风险评估之中,创新性地启发了研究人员对于预测模型的新型应用。现阶段,预测微生物模型已被广泛应用于食物链各环节中微生物数量的计算,成为定量微生物风险评估中暴露评估环节必不可少的部分。2002—2013年期间,联合国粮农组织/世界卫生组织公布了19 个定量微生物风险评估报告,均使用了预测微生物模型作为描述致病菌生长和失活的工具[36]。2)随机模型(stochastic model)的研究:随机模型利用概率分布来代替具体的数值,相对于确定性模型(deterministic model)而言,随机模型具有更广泛的使用范围,更符合真实情况的拟合结果。当前关于微生物生长动力学随机模型的研究,主要分为基于菌株变异性(stain variability)的随机模型研究[37]和基于单细胞生长情况的随机模型研究[38-40]。3)分子生物学技术在预测微生物学中的引入:Baranyi等曾建议使用“食品微生物生态定量学(quantitative microbial ecology of food)”作为学科名称[17],但是普遍观点认为“预测微生物学”的概念更加深入人心。尽管“食品微生物生态定量学”最终没有成为本学科的名称,但是这一定义也给了研究人员深刻的启示:即“食品预测微生物学”的本质在于研究食品内部微生物的生态动力学变化。因此,从2009年开始,一些研究人员开始尝试将生态学研究中常使用的分子生物学技术应用于预测微生物学之中[6-13]。2013年,Ye Keping等[7]的研究对这类基于分子生物学方法构建的模型下了定义,称其为分子预测模型(molecular predictive model)。
相较于日趋成熟的随机模型和定量微生物风险评估体系,分子预测模型的研究尚处于起步阶段。但是,由于分子生物学技术能够提供传统模型无法提供的信息和数据,描述传统模型难以描述的生长趋势,已经成为现今预测微生物学最新的研究热点之一。
尽管预测微生物学在过去10 年间蓬勃发展,但是近几年已经基本趋于稳定,年文章数甚至还有下降的趋势(图1),而分子生物学技术的引入则为预测微生物学提供了“新的血液”。传统的预测模型构建方法常基于微生物培养技术,不仅耗时费力,而且由于其特异性较差等原因,往往只能描述单一微生物在纯培养或消除本底微生物食品中的生长情况,这明显不符合实际食品中微生物的动态变化规律。随着科学技术的不断发展,变性梯度凝胶电泳、荧光定量PCR、DNA杂交 探针和宏基因组学测序等分子生物学技术被应用于预测微生物学的研究之中,这些技术具有省力、快速、高效等优点,大大改善了传统方法的不足,已受到国内外预测微生物学专家的认可。基于以上分析,现将分子生物学技术在预测微生物学中应用的研究现状综述如下。
2.1 变性梯度凝胶电泳技术
DGGE是Fischer等[41]提出一项应用于DNA点突变检测的技术。该技术的分辨精度高于传统的琼脂糖凝胶电泳和聚丙烯酰胺凝胶电泳,可以分辨只有一个碱基差异的基因序列。Muyzer等[42]首次将该技术应用于分子微生物学领域,证实了其在揭示自然界微生物区系的遗传多样性和种群差异方面具有独特的优越性。
PCR结合DGGE技术(以下简称PCR-DGGE)是DGGE方法中最为常见的一种技术,该技术首先扩增微生物DNA片段的16S rRNA或18S rRNA中包含保守区和可变区的基因序列,然后利用含有变性剂(尿素和甲酰胺)的聚丙烯酰胺凝胶电泳对PCR扩增产物进行电泳,从而得到微生物群落结构信息。该技术无需培养过程,大幅度地缩短了样品分析时间,能显示样品中不可培养微生物的遗传信息,并对微生物种群动态性、重要基因定位、表达和调控进行评价分析,在土壤、海洋及食品等领域得到了广泛的应用。
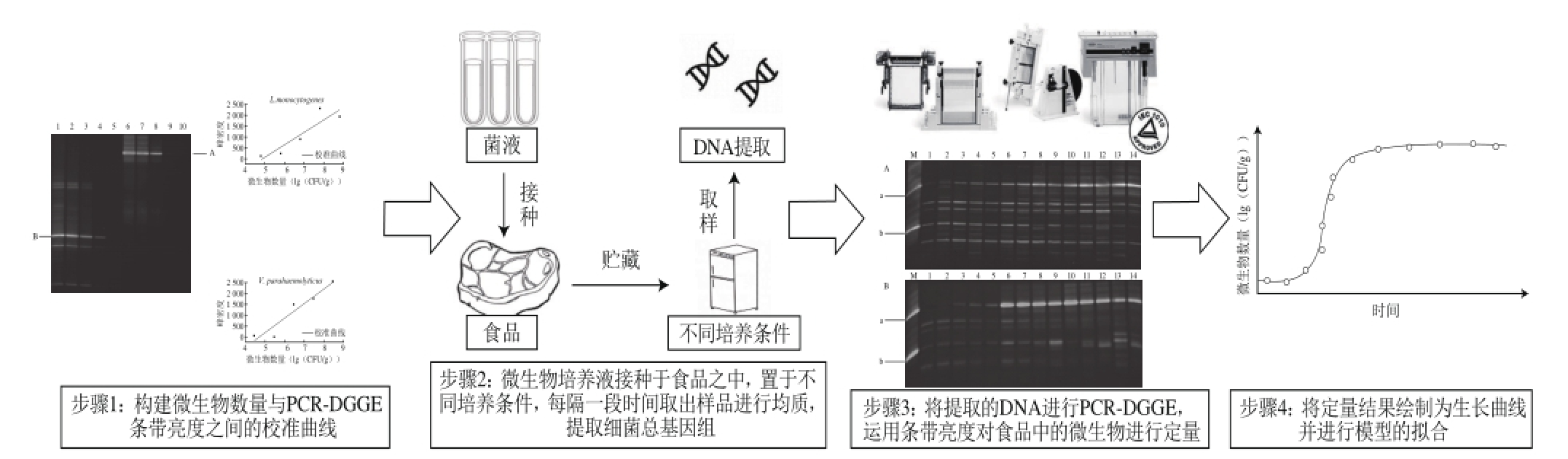
图3 应用PCR-DGGE技术构建预测模型的流程图[[88]]
Fig.3 Schematic representation of application of PCR-DGGE techniques in predictive modelling[8]
Liao Chao等[11]将PCR-DGGE技术应用于预测微生物模型的构建,具体的操作步骤如图3所示:首先,利用微生物数量和PCR-DGGE条带亮度之间的关系构建校准曲线;然后,依据传统的建模步骤,将单增李斯特菌和副溶血性弧菌的菌液接种于即食虾中,置于4 ℃和10 ℃条件下进行培养,每隔一段时间取出样品进行均质,提取细菌的总基因组DNA;接着,将提取的DNA进行PCR-DGGE分析,运用DGGE的条带亮度同时定量描述即食虾中的单增李斯特菌和副溶血性弧菌的存活情况;最后,将定量结果绘制为生长曲线,并用Baranyi模型进行拟合。实验结果表明:对比于传统涂布方法构建的预测模型,PCR-DGGE技术可以较好地描述单增李斯特菌的生长趋势,但是不适合描述副溶血性弧菌的失活趋势。Liao Chao等[11]分析导致这一现象的原因可能是:低温条件下,副溶血性弧菌处于失活状态,提取的细菌总基因组DNA中既包括活菌的DNA,也包括死菌的DNA,因此导致定量效果的失准,而单增李斯特菌在低温条件下处于生长状态,死菌DNA的因素对其干扰较小。
由此可见,PCR-DGGE技术应用于预测微生物学中具有高效快速的优点,可用于同时分析两种或两种以上微生物的生长动力学情况,但是在定量精度和失活模型的构建方面,该方法存在一定的缺陷,因此没有得到很好的推广。本文建议今后研究可对该方法进行一定程度的优化,使其能够更好地描述微生物的失活趋势,或者将该方法应用于复杂体系中微生物生长动力学的初探,为进一步的深入研究奠定基础。
2.2 Real-time PCR技术
PCR是Mullis等[43]发明的一种核酸体外扩增检测技术。目前,该技术已成功应用于食品中多种致病菌的检测,具有特异性强、灵敏度高等特点。但是,传统的PCR技术需要借助电泳等手段对扩增产物进行分析,不仅耗时费力、容易污染,而且无法对目标微生物进行准确地定量,使其应用受到限制。美国Applied Biosystems公司推出了Real-time PCR[44],该技术弥补了传统PCR的缺陷,实现了PCR从定性到定量的突破。该技术具有灵敏性高、特异性强、定量结果精准等优点,可直接收集PCR过程中的荧光信号,以获得扩增产物定量的结果,不需要后续的琼脂糖凝胶电泳检测,克服了传统PCR技术缺陷,达到实时检测扩增信号的目的。Real-time PCR方法分为荧光染料法、荧光探针法和分子信标法,其中最为常用的是SYBRGreen法[45]和TaqMan探针法[46],SYBRGreen法在特异性和准确性方面不如TaqMan探针,但其造价相对较低,所以使用频率也相对较高。
目前,在所有用于构建预测微生物模型的分子生物学技术中,Real-time PCR是最被研究人员认可,也是应用最为广泛的一项技术。表1总结了近年来Real-time PCR技术在预测微生物学中的应用情况。
表1 Real-time PCR技术在预测微生物学中的应用
Table1 Application of real-time PCR techniques in predictive microbiology

如表1所示,Reichert-Schwillinsky等[6]首先使用Realtime PCR技术描述了肉汤培养基中单增李斯特菌的生长情况,提出该技术在预测微生物学中的潜在应用价值。Ye Keping等[7]运用这项技术成功地构建了猪肉中单增李斯特菌的一级预测模型,证明了这种方法在食品中应用的可行性。孙文烁等[9]利用Real-time PCR技术构建了即食虾中副溶血性弧菌的预测模型,首次证明了这项技术不仅可应用于一级预测模型,也可用于二级模型的构建。同年,Ye Keping等[8]在Food Research International上发表了最新的研究成果:利用Real-time PCR技术描述了4 ℃条件下单增李斯特菌和乳酸菌的存活情况,并构建了这两种菌的竞争抑制模型。利用Real-time PCR技术构建预测模型的方法与PCR-DGGE技术相类似,如图4所示,通过构建微生物数量与Real-time PCR的循环阈值(cycle threshold,Ct值)之间的校准曲线,定量描述微生物在不同条件下的生长(或失活)情况。
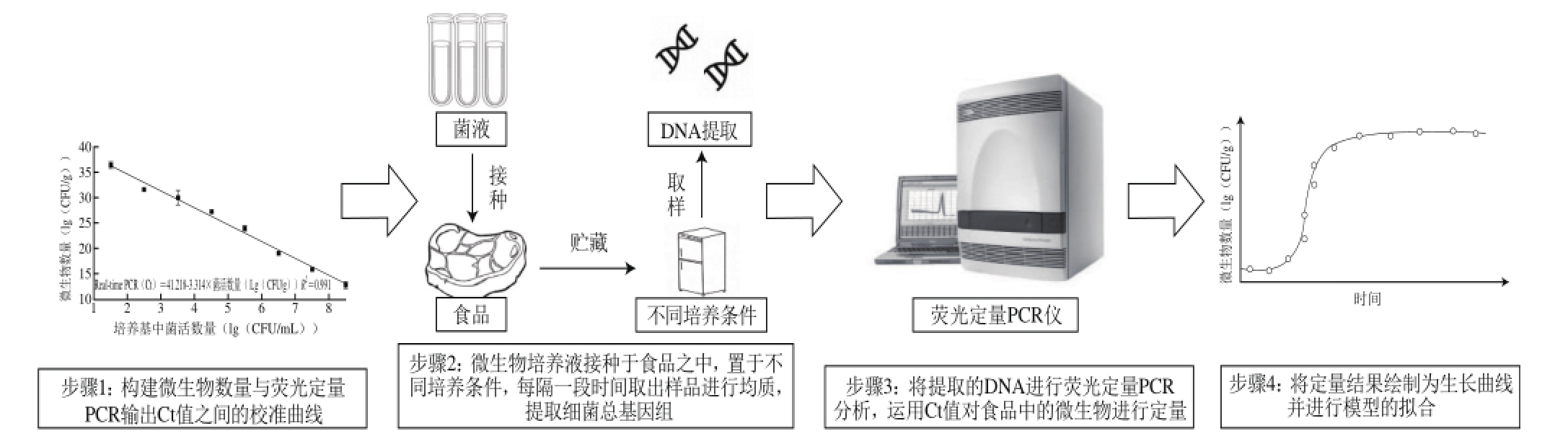
图4 应用Real-time PCR技术构建预测模型的流程图[[99]]
Fig.4 Schematic representation of application of real-time PCR techniques in predictive modelling[9]
尽管Real-time PCR已经被上述研究证明是一种可靠的预测模型构建方法,但是这类基于DNA扩增的定量方法存在一个极大的缺陷,即无法区分样品中死菌和活菌的问题,死菌DNA的存在导致传统Real-time PCR无法准确地描述微生物在失活情况下的动力学变化。叠氮溴化丙锭(propidium monoazide,PMA)是一种对核酸具有高度亲和力的光敏反应染料[47],能够进入具有不完整细胞壁或细胞膜的细菌死细胞中,选择性地交联死菌DNA,将其与DNA扩增检测技术相结合,可有效地抑制死菌细胞DNA的扩增。基于此,Zhang Zhaohuan等[10]将PMA与多重Real-time PCR技术相结合,用于定量描述4 ℃条件下生虾样品中 副溶血性弧菌和单增李斯特菌的行为变化。尽管这两种食源性致病菌在4 ℃条件下呈现迥异的存活行为,但是这种新颖的PCR技术能够精准并同时描述副溶血性弧菌的失活情况和单增李斯特菌的生长情况。PMA和Real-time PCR技术的有机结合,弥补了传统分子生物学定量方法无法区分死活菌的不足,使其能够用于细菌失活状况的描述。因此,这项技术可以作为一个有力的工具应用于今后的预测微生物学研究之中。
2.3 DNA杂交探针技术
DNA杂交探针技术(DNA hybridization probes)也称为核酸分子杂交技术,是兴起于20世纪70年代的一种重要的分子生物学技术[48]。其原理基于DNA分子碱基互补配对原则,通过设计合适的DNA探针,观测待测样品中是否存在能与DNA探针形成杂交的物质,用以判断样品中是否存在靶标微生物。DNA杂交探针技术具有强特异性、高敏感性、无放射性等优点,近年来,DNA探针技术已被广泛应用于各类食品中致病菌的定性或定量检测[49-51]。
Parveen等[12]运用碱性磷酸酶标记的DNA杂交探针技术构建了鲜活牡蛎中自然存在副溶血性弧菌的预测微生物模型。传统的预测模型常 基于人工接种的微生物和消除本底微生物的样品,而这项研究却首次描述了自然存在于鲜活牡蛎中的副溶血性弧菌的生长和失活情况,为今后预测微生物学的发展指出了一条道路,即应当发展此类基于真实样品中自然存在微生物的预测模型,而不是仅仅局限于实验室环境下人工接种模型的构建。这一预测模型的成功构建,得益于特异性极强的DNA杂交探针技术,该技术不受复杂背景菌群的干扰,专一地定量了鲜活样品中的副溶血性弧菌,从而进一步说明了分子生物学技术在预测微生物学中具有极为广阔的应用前景。
2.4 宏基因组学测序技术
宏基因组(metagenome),又名环境微生物基因组或元基因组,是指环境样品中全部微小生物的基因总和。宏基因组学是由Handelsman于1998年首次提出的一门应用学科[52],宏基因组学测序技术对环境样品DNA进行直接测序,克服了微生物难以培养的缺点,揭示了微生物基因序列特征,开启了微生物学研究新纪元。该技术还可用于揭示微生物之间、微生物与环境之间相互作用的规律,大大拓展了微生物学的研究思路 与方法,从群落结构的水平全面认识微生物的生态特征和功能[52-53]。
由于宏基因组测序技术只能用于确定样本中微生物的种类,而无法对其定量,所以其在预测微生物学中的应用常被人所忽视。但是,近期发表在国际顶级期刊Science上的一项最新研究中,Korem等[13]运用宏基因组学测序技术对人类肠道菌群进行了分析,发现通过分析起始DNA和末端DNA的相对含量,可以反映出肠道中每一株菌株的生长速率。研究人员对该方法进行了验证,结果显示,该方法预测的细菌生长速率与实验观察的细菌生长速率几乎保持一致。进一步分析表明,一些细菌生长速率的特定变化与Ⅱ型糖尿病相关,而另一些变化与炎症性肠道疾病有关。这一新方法的出现或可在未来应用于特殊样品(如肠道、粪便、复杂的食品基质等)中微生物的生长动力学描述,为预测微生物学在防治疾病和维护人类健康的研究中提供了可能性。
尽管已有上述研究作为铺垫,分子生物学技术在预测微生物学中的应用尚处于起步阶段,后续研究还需要进一步将各类分子生物学技术逐步应用于预测微生物学之中,以构建更加高效精准、更加符合实际情况的分子预测模型。如图5所示,本 文不仅综述了分子生物学技术在预测微生物学中的现有研究,还对其他生物学技术在预测微生物学中应用的进行了展望,分析了它们的主要优势以及在预测模型中应用的可行性。
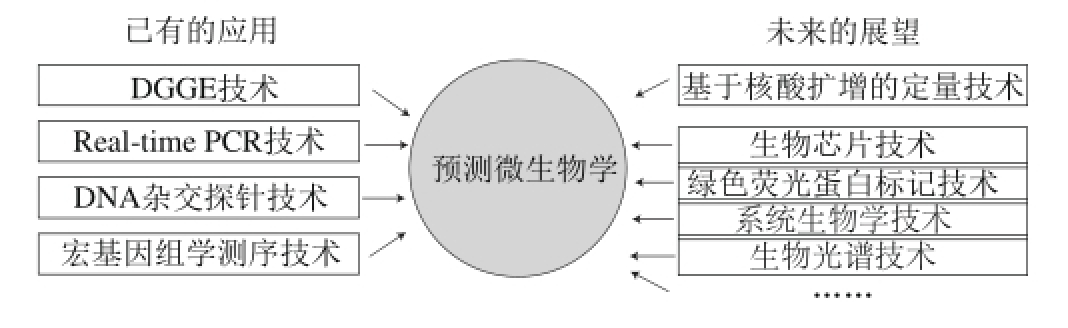
图5 生物学技术在预测微生物学中应用的展望
Fig.5 Prospects for the applications of biological techniques in predictive microbiology
3.1 基于核酸扩增的定量技术
基于核酸扩增的技术是分子生物学稳定发展的基础,除了上文提及的PCR和Real-time PCR技术,其他基于核酸扩增的技术也可用于微生物的定量检测,如环介导等温扩增技术(loop-mediated isothermal amplif i cation,LAMP)、荧光LAMP技术、赖解旋酶恒温基因扩增技术以及数字PCR技术[54]。其中,数字PCR技术被称为PCR和Real-time PCR技术之后的第三代PCR技术,是近年来迅速发展起来的一种定量分析技术[55],与传统PCR定量技术不同,数字PCR通常在微流控芯片上完成,不依赖于荧光 定量PCR仪器的循环阈值进行定量,不受到扩增效率的影响,也不必采用管家基因和标准曲线,具有很好的准确度和重现性,可以实现真正的绝对定量分析。
若将数字PCR技术应用于预测微生物学研究,具有以下几点优势:第一,快速。数字PCR技术应用于预测微生物学可大大缩短建模时间,传统的平板计数法构建预测微生物模型需要花费3~5 d的时间,而该方法仅需要几个小时;第二,高特异性。数字PCR技术具有极高的特异性,这一优势保证该方法应用于生鲜样品中细菌行为变化的研究时,不易受到复杂的食品基质和背景菌群的影响;第三,高通量。多重荧光定量PCR技术受限于PCR仪中报告基团的数量,往往最多只能同时定量检测3~5 种微生物,而数字PCR技术能够同时定量检测更多种类的微生物,只要芯片设计合理,该技术可用于同时构建复杂体系中所有微生物的预测模型。
3.2 生物芯片技术
生物芯片是20 世纪90 年代初发展起来的一种全新的微量分析技术,它综合了分子生物学、免疫学、机械学以及计算机等多项技术,其原理在于采用原位合成或微矩阵点样等方法,将大量生物大分子如核酸片段、多肽片段、细胞等样品有序地固定在硅胶片或聚丙烯酰胺凝胶等支持物的表面,组成密集的二维分子排列,然后与已标记的待测生物样品中的靶分子杂交,通过特定的仪器对杂交信号的强度进行快速、并行、高效的检测分析,判断样品中靶分子的数量,从而达到分析检测的目的[49-50,56]。
生物芯片技术具有高通量、高灵敏度、多参数同步分析等传统检测方法不可比拟的优点,因此,若将该技术应用于预测模型的构建,不仅能够准确地反映食品中微生物生长或衰亡的状况,还可以同时分析食品组分中的其他物质,从而判断影响微生物生长或衰亡真正原因,从机理的角度揭示微生物生长动力学的本质。
3.3 绿色荧光蛋白标记技术
绿色荧光蛋白(green fluorescent protein,GFP)最初为Morse等在研究西北太平洋海域的发光水母中发现的一种发光蛋白的伴侣蛋白。随后,Shimomura和War等对GFP的结构和功能进行了全面的研究,证明该蛋白是一种具有独特功能的蛋白质,编码238 个氨基酸残基,分子质量为2 688 D,在受到紫外或蓝光激发时,可以自身催化形成发色结构并发出绿色荧光,其吸收波长为395 nm,发射波长为509 nm[57]。近年来,利用GFP可以发出绿色荧光的这一特点,绿色荧光蛋白标记技术逐渐发展成最热门的一种基因标记技术。相比于常规的荧光标记和检测技术,该技术具有以下诸多优点:荧光特性稳定,不易受到干扰;检测方便;易于构建载体,且无种属特异性;分辨率高;对生物细胞无毒害作用;可进行活细胞定时定位观察。
GFP标记技术在预测微生物学中的应用,首先应对目标菌株进行GFP遗传标记,然后通过紫外线的照射,经GFP标记的菌株可以发出强烈的荧光,从而可以直接进行细胞的显微观察和计数,实时地监测GFP标记菌株的生长或存活情况。相较于传统培养方法仅能提供对数化的数值,GFP标记技术可利用连续变化的图片或动态的录像,直接描述微生物的存亡,输出的定量结果更为真实可靠,也能够更直观、更深入地反映微生物的生长动力学问题。
3.4 系统生物学技术(组学技术)
食品中的微生物群落本身是一个复杂系统, 传统的预测微生物学往往只局限于单个微生物的研究,现阶段对已经不能满足人类对于微生物了解的需要,解答食品组分中所有微生物生态生长动力学的关键在于利用系统的方法学[58]。系统生物学(systems biology)是研究一个生物系统中所有组成成分的构成,以及在特定条件下这些组分间的相互关系,并通过计算生物学建立数学模型来定量描述和预测生物功能表型和行为的学科[59]。系统生物学的技术平台称为组学(omics)[60],包括基因组学、转录组学、蛋白质组学、代谢组学、相互作用组学和表型组学等,它们分别构成了生命信息传递的几个层次,在蛋白质和代谢产物水平检测和鉴别各种分子并研究其功能。
宏基因组学在预测微生物学中的应用[13],只展现出组学技术强大功能的冰山一角,若将系统生物学技术应用于预测微生物学研究,可综合基因组、蛋白组、转录组及代谢物组等技术,全面地解读食品组分中所有微生物的生态和生长动力学变化,并分析产生这些变化背后的原因,从源头上遏制致病菌的风险和腐败菌的滋生,有助于推动预测微生物学这一学科的全面进步。
3.5 生物光谱技术
严格意义而言,光谱技术并非分子生物学技术。但是,近年来,光谱技术因具有快速、准确、安全、高效、非破坏性和便于在线侦检等特点,成为一种新型无创检测技术,目前已广泛应用于病原微生物的检测[61]。常用的光谱技术分为激光激发荧光光谱、红外光谱和拉曼光谱。人的指纹具有唯一性,常用于个体身份识别,科学家认为自然界的物质也有指纹,而光谱则被认为是指纹的表现形式之一。同一种物质,无论其来源与产生方式,都应具有相同的光谱。病毒主要由蛋白质和核酸组成,细菌还包括脂质糖类辅酶维生素等基本成分。针对不同微生物的分子组成含量和构成不同,找出对生物分子敏感的光谱技术,结合主成分分析分级聚类或人工神经网络等化学计量学方法,可对微生物进行无损定量分析。正是由于光谱技术具有无损检测这一独一无二的优势,使其在预测微生物学中具有很大的潜在应用价值。通过光谱技术可无损地定量食品中的微生物,直接描述食品中微生物的生长情况,避免了传统方法回顾性的评价方式,可使实验结果更加准确可靠。
为了区别于传统基于培养的方法构建的预测微生物模型,研究人员将利用分子生物学技术构建的预测微生物模型,称为分子预测模型。这一新兴模型的诞生,有助于克服传统研究方法与技术的局限,加速预测微生物学的发展,为微生物生长动力学的研究 提供了强有力的工具。但是,任何一种方法都有各自优点和局限性,分子预测模型的兴起,并不能完全取代传统的预测模型,而应该作为传统模型的一个良好的补充。在预测微生物学的研究中,应根据不同的情况选择不同的方法,将传统模型与分子模型有机地结合在一起,使各种方法技术之间得到相互补充、相互印证。现将分子预测模型未来可能的应用前景展望如下:1)有助于提升定量微生物风险评估的准确性。预测微生物模型是定量微生物风险评估核心基础之一,是风险评估有效实施的可靠保障[1,39]。分子预测模型可以提供一些传统预测模型难以提供的信息,若将其与传统预测模型有机地结合,共同服务于定量微生物风险评估体系,可大大提升定量微生物风险评估的准确性。此外,传统的风险评估常常止步于“从农田到餐桌”的过程,一味地把“入口概率”作为“致病概率”,而忽略了致病菌进入人体之后的存活情况。分子预测模型具有极高的特异性,能够探究许多特殊条件下(如肠道中)微生物的生长动力学,因此,可用于模拟致病菌“从摄入到致病”过程中微生物的存活情况,弥补这一环节研究上的空白,以构建更加精准的定量微生物风险评估模型。2)有助于揭示食品腐败变质的机制。食品中的微生物是导致食品腐败变质的主要原因,其生长繁殖状况直接影响产品的货架期[62]。近年来,国内外许多学者都致力于食品货架期的预测微生物模型研究,为此构建了大量的货架期预测模型,为更好地延长食品货架期,防止食品腐败变质提供了强有力的工具[63]。但是,这些模型往往仅揭示腐败微生物的消长情况,而忽视了食品腐败变质内在机制的揭示。分子预测模型可很好地弥补这一研究空白,通过结合系统生物学技术,能够全面宏观地再现食品腐败变质的全过程,准确地把握微生物消长与食品腐败间的内在联系,从而揭示导致食品腐败变质的真正原因。3)有助于构建更加完善的食品冷藏链体系。食品冷藏链是指易腐食品从产地收购或捕捞之后,在产品加工、贮藏、运输、分销和零售直到消费者手中各个环节始终处于产品所必需的低温环境下[64]。预测微生物学有助于冷藏链中食品货架期的预测、致病菌风险的估算,推动食品冷藏链的标准化、信息化、一体化发展。但是在低温条件下,食品中的微生物存活情况极为复杂,其消长的动力学变化极为不规律:一部分微生物处于生长状态,而另外一部分则缓慢失活,还有一些微生物甚至还会进入一种特殊的存活状态,即活的不可培养状态(viable but non-culturable state,VBNC)[65]。传统基于培养方法构建的预测模型,显然不能描述这些微生物的生长变化规律,因此需要分子预测模型的辅助,才能预测冷藏链中食品货架期和致病菌风险,以构建更加完善的食品冷藏链体系。
作为预测微生物学研究人员已经渡过了没有学科定位的“黑暗时代”,也经历了没有固定模型的“蛮荒时代”,还见证了以传统培养方法构建模型的“辉煌时代”。现阶段预测微生物学的发展已经进入瓶颈期,但是,分子生物学技术的蓬勃发展,为继续推动这一学科的进步提供了强有力的工具和绝佳的机会。21世纪是一个大数据的时代,如果总是将目光局限于一条条生长曲线,只关注纯培养、单一菌株和消除本底微生物的食品基质,预测微生物学将固步自封、裹足不前。应该从整个系统的角度分析微生物的消长以及其背后的原因,推动学科的发展,让其更好地服务于人类。
参考文献:
[1] DONG Q L, BARKER G C, GORRIS L G M, et al. Status and future of quantitative microbiological risk assessment in China[J]. Trends in Food Science & Technology, 2015, 42(1): 70-80. DOI:10.1016/ j.tifs.2014.12.003.
[2] 赵勇, 王敬敬, 唐晓阳, 等. 水产品中食源性致病微生物风险评估研究现状[J]. 上海海洋大学学报, 2012, 21(5): 899-905.
[3] ROSS T, MCMEEKIN T A. Predictive microbiology[J]. International Journal of Food Microbiology, 1994, 23(3/4): 241-264. DOI:10.1016/0168-1605(94)90155-4.
[4] McMEEKIN T A, OLLEY J N, ROSS T, et al. Predictive microbiology: theory and application[M]. Taynton: Research Studies Press, 1993: 419.
[5] McMEEKIN T, BOWMAN J, MCQUESTIN O, et al. The future of predictive microbiology: strategic research, innovative applications and great expectations[J]. International Journal of Food Microbiology, 2008, 128(1): 2-9. DOI:10.1016/j.ijfoodmicro.2008.06.026.
[6] REICHERT-SCHWILLINSKY F, PIN C, DZIECIOL M, et al. Stressand growth rate-related differences between plate count and Real-time PCR data during growth of Listeria monocytogenes[J]. Applied and Environmental Microbiology, 2009, 75(7): 2132-2138. DOI:10.1128/ AEM.01796-08.
[7] YE Keping, WANG Huhu, ZHANG Xinxiao, et al. Development and validation of a molecular predictive model to describe the growth of Listeria m onocytogenes i n vacuum-packaged chilled pork[J]. Food Control, 2015, 32(1): 246-254. DOI:10.1016/j.foodcont.2012.11.017.
[8] YE Keping, WANG Huhu, JIANG Yun, et al. Development of interspecific competition models for the growth of Listeria monocytogenes and Lactobacillus on vacuum-packaged chilled pork by quantitative real-time PCR[J]. Food Research International, 2014, 64: 626-633. DOI:10.1016/j.foodres.2014.07.017.
[9] 孙文烁, 靳梦曈, 王敬敬, 等. 运用Real-time PCR建立即食虾中副溶血性弧菌分子预测模型[J]. 现代食品科技, 2014, 30(7): 142-148.
[10] ZHANG Zhaohuan, LIU Haiquan, LOU Yang, et al. Quantifying viable Vibrio parahaemolyticus and Listeria monocytogenes simultaneously in raw shrimp[J]. Applied Microbiology and Biotechnology, 2015, 99(15): 6451-6462. DOI:10.1007/s00253-015-6715-x.
[11] LIAO Chao, PENG Zhiyun, LI Jibing, et al. Simultaneous construction of PCR-DGGE-based predictive models of Listeria monocytogenes and Vibrio parahaemolyticus on cooked shrimps[J]. Letters in Applied Microbiology, 2015, 60(3): 210-216. DOI:10.1111/lam.12376.
[12] PARVEEN S, DASILVA L, DEPAOLA A, et al. Development and validation of a predictive model for the growth of Vibrio parahaemolyticus in post-harvest shellstock oysters[J]. International Journal of Food Microbiology, 2013, 161(1): 1-6. DOI:10.1016/ j.ijfoodmicro.2012.11.010.
[13] KOREM T, ZEEVI D, SUEZ J, et al. Growth dynamics of gut microbiota in health and disease inferred from single metagenomic samples[J]. Science, 2015, 349: 1101-1106. DOI:10.1126/science. aac4812.
[14] ESTY J R, MEYER K F. The heat resistance of the spore of B. botulinus and allied anaerobes XI[J]. Journal of Infectious Diseases, 1922, 31(6): 650-663.
[15] SCOTT W J. The growth of microorganisms on ox muscle: II. The influence of temperature[J]. Australia Council for Scientific and Insustrial Research, 1937, 10(4): 338-350.
[16] BALL C O, OLSON F C W. Sterilization in food technology theory, practice and calculations[M]. New York: McGraw-Hill Book, 1957.
[17] 董庆利. 食品预测微生物学: 过去 现在 将来[J]. 农产品加工(学刊), 2009, 166(3): 38-46.
[18] FREDERICKSON A G, RAMKRISHNA D, TSUHIYA H M. Statistics and dynamics of prokaryotic cell populations[J]. Mathematical Biosciences, 1967, 1(3): 327-374.
[19] 张秋勤, 徐幸莲. 预测微生物学在禽肉质量安全控制中的应用[J].食品科学, 2010, 31(13): 292-296.
[20] WHITING R C, BUCHANAN R L. A classification of models for predictive microbiology[J]. Food Microbiology, 1993, 10(12): 175-177. DOI:10.1006/fmic.1993.1017.
[21] 姬华. 对虾中食源性弧菌预测模型建立及风险评估[D]. 无锡: 江南大学, 2012: 8-9.
[22] GIBSON A M, BRATCHELL N, ROBERTS T A. The effect of sodium chloride and temperature on the rate and extent of growth of Clostridium botulinum type A in pasteurized pork slurry[J]. Journal of Applied Microbiology, 1987, 62(6): 479-490. DOI:10.1111/j.1365-2672.1987.tb02680.x.
[23] ZWIETERING M H, JONGENBURGER L, ROMBOUTS F M, et al. Modeling of the bacterial growth curve[J]. Applied Environment Microbiology, 1990, 56(6): 1875-1881.
[24] BARANYI J, ROBERTS T A. A dynamic approach to predicting bacterial growth in food[J]. International Journal of Food Microbiology, 1994, 23(3/4): 277-294. DOI:10.1016/0168-1605(94)90157-0.
[25] RATKOWSKY D A, OLLEY J, MCMEEKIN T A, et al. Relat ionship between temperature and growth rate of bacterial cultures[J]. Journal of Bacteriology, 1982, 149(1): 1-5.
[26] RATKOWSKY D A, LOWRY R K, MCMEEKIN T A, et al. Model for bacterial culture growth rate throughout the entire biokin etic temperature range[J]. Journal of Bacteriology, 1983, 154(3): 1222-1226.
[27] McMEEKIN T A, ROSS T, OLLEY J N. Application of predictive microbiology to assure the quality and safety of fish and fish products[J]. International Journal of F ood Microbiology, 1992, 15(1/2): 13-32. DOI:10.1016/0168-1605(92)90132-M.
[28] SPENCER R, BAINES C R. The effect of temperature on the spoilage of wet white fi sh. I. Storage at constant temperature between -1 and 25 ℃[J]. Food Technology, 1964, 18(5): 769-773.
[29] ROBERT T A, GIBSON A M, ROBINSON A. Prediction of toxin production by Clostridium botulinum in pasteurized pork slurry[J]. International Journal of Food Science and Technology, 1981, 16(4): 337-355.
[30] HUDSON J A. Construction of and comparisons between response surface models for Aeromonas hydrophila ATCC 7966 and food isolate under aerobic conditions[J]. Journal of Food Protection, 1992, 55(12): 968-972.
[31] HUDSON J A. Comparison of response surface models for Listeria monocytogenes strains under aerobic conditions[J]. Food Research International, 1994, 27(1): 53-59. DOI:10.1016/0963-9969(94)90177-5.
[32] 李秋鹏. 食品预测微生物学三级模型的研究进展[J]. 农产品加工(学刊), 2013, 328(9): 54-56. DOI:10.3969/jissn.1671-9646(X).2013.09. 016.
[33] ROSS T. Indices for performance evaluation of predictive models in food microbiology[J]. Journal of Applied Bacteriology, 1996, 81(5): 501-508. DOI: 10.1111/j.1365-2672.1996.tb03539.x.
[34] BARANYI J, PIN C, ROSS T. Validating and comparing predictive models[J]. International Journal of Food Microbiology, 1999, 48(3): 159-166. DOI: 10.1016/S0168-1605(99)00035-5.
[35] van GERWEN S J C, ZWIETERING M H. Growth and inactivation models to be used in quantitative risk assessments[J]. Journal of Food Protection, 1998, 61(11): 1541-1549.
[36] Food and Agriculture Organization/World Health Organization. Microbiological risks publications [M/OL]. 2014 [2014-04-02]. http:// www.who.int/foodsafety/publications/micro/en/index.html.
[37] LIANOU A, KOUTSOUMANIS K P. Strain variability of the behavior of foodborne bacterial pathogens: a review[J]. International Journal of Food Microbiology, 2013, 167(3): 310-321. DOI:10.1016/ j.ijfoodmicro.2013.09.016.
[38] 董庆利, 刘阳泰, 苏亮, 等. 食源性致病菌单细胞观测与预测的研究进展[J]. 农业机械学报, 2015, 46(11): 221-229. DOI:10.6041/ j.issn.1000-1298.2015.11.030.
[39] RENSHAW E. Modelling biological population in space and time[M]. Cambridge: Cambridge University Press, 1991: 1-424.
[40] CUMMINS E, FRIAS J M, VALDRAMIDIS V P. Modelling at a single cell level[C]// Proceedings of the 7th International Conference on Predictive Modelling of Food Quality and Safety, Ireland: Dublin, 2011: 41-57.
[41] FISCHER S G, LERMAN L S. Length-independent separation of DNA restriction fragments in two-dimensional gel electrophoresis[J]. Cell, 1979, 16(1): 191-200. DOI:10.1016/0092-8674(79)90200-9.
[42] MUYZER G, WAAL E C, UITTERLINDEN A G. Prof i ling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplif i ed genes coding for 16S rRNA[J]. Applied Environmental Microbiology, 1993, 59(3): 695-700. [43] MULLIS K B, FALOONA F A. Specif i c synthesis of DNA in vitro via a polymerase-catalyzed chain reaction[J]. Methods in Enzymology, 1987, 155: 335-351.
[44] HEID C A, STEVENS J, LIVAK K J. Real time quantitative PCR[J]. Genome Research, 1996, 6(10): 986-994. DOI:10.1101/gr.6.10.986.
[45] YIN J L, SHACKEL N A, ZEKRY A, et al. Real-time reverse transcriptase-polymerase chain reaction (RT-PCR) for measurement of cytokine and growth factor mRNA expression with fl uorescent probes or SYBR Green I[J]. Immunology and Cell Biology, 2001, 79(3): 213-221. DOI:10.1046/j.1440-1711.2001.01002.x.
[46] LIVAK K J, FLOOD S J, MARMARO J, et al. Oligo nucleotides with fluorescent dyes at opposite ends provide a quenched probe system useful for detecting PCR product and nucleic acid hy bridization[J]. PCR Methods and Applications, 1995, 4(6): 357-362.
[47] NOCKER A, CHEUNG C Y, ANNE K, et al. Comparison of propidium monoazide with eth idium monoazide for differentiation of live vs. dead bacteria by selective removal of DNA from dead cells[J]. Journal of Microbiological Methods, 2006, 67(2): 310-320. DOI:10.1016/j.mimet.2006.0 4.015.
[48] TYAGI S, KRAMER F R. Molecular beacons: probes that fl uoresce upon hybridization[J]. Nature Biotechnology, 1996, 14(3): 303-308. DOI:10.1038/nbt0396-303.
[49] HONG B X, JIANG L F, HU Y S, et al. Application of oligonucleotide array technology for the rapid detection of pathogenic bacteria of foodborne infections[J]. Journal of Microbiological Methods, 2004, 58(3): 403-411. DOI:10.1016/j.mimet.2004.05.005.
[50] BODROSSY L, SESSITSCH A. Oligonucleotide microarrays in microbial diagnostics[J]. Curre nt Opinion in Microbiology, 2004, 7(3): 245-254. DOI:10.1016/j.mib.2004.04.005.
[51] CALL D R, BORUCKI M K, LOGE F J. Detection of bacterial pathogens in environmental samples using DNA microarrays[J]. Journal of Microbiol ogical Methods, 2003, 53(2): 235-243. DOI:10.1016/S0167-7012(03)00027-7.
[52] STREIT W R, SCHMITZ R A. Metagenomics-the key to the uncultured microbes[J]. Current Opinion in Microbiology, 2004, 7(5): 492-498. DOI:10.1016/j.mib.2004.08.002.
[53] DANILO E. High-throughput sequencing and metagenomics: moving forward in the culture-independent analysis of food microbial ecology[J]. Applied and Environmental Microbiology, 2013, 79(10): 3148-3155. DOI:10.1128/AEM.00256-13.
[54] 林彩琴, 姚波. 数字PCR技术进展[J]. 化学进展, 2012, 24(12): 2415-2423.
[55] OTTESEN E A, HONG J W, QUAKE S R. Microf l uidic digital PCR enables multigene analysis of individual environmental bacteria[J]. Science, 2006, 314: 1464-1467. DOI:10.1126/science.1131370.
[56] TEMPLIN M F, STOLL D, SCHRENK M. Protein microarray technology[J]. Trends in Biotechnology, 2002, 20(4): 160-166. DOI:10.1016/S0167-7799(01)01910-2.
[57] TSIEN R Y. The green fluorescent protein[J]. Annual Review of Biochemistry, 1998, 67(1): 509-544. DOI:10.1146/annurev. biochem.67.1.509.
[58] van IMPE J F, VERCAMMEN D, van DERLINDEN E. Toward a next generation of predictive models: a systems biology primer[J]. Food Control, 2013, 29(2): 336-342. DOI:10.1016/j.foodcont.2012.06.019.
[59] KITANO H. Systems biology: a brief overview[J]. Science, 2002, 295: 1662-1664. DOI:10.1126/science.1069492.
[60] GE H, WALHOUT A J M, VIDAL M. Integrating ‘omic’ information: a bridge between genomics and systems biology[J]. Trends in Genetics, 2003, 19(10): 551-560. DOI:10.1016/j.tig.2003.08.009.
[61] GOWEN A A, FENG Y, GASTON E. Recent applications of hyperspectral imaging in microbiology[J]. Talanta, 2015, 137: 43-54. DOI:10.1016/j.talanta.2015.01.012.
[62] GRAM L, RAVN L, RASCH M, et al. Food spoilage-interactions between food spoilage bacteria[J]. International Journal of Food Microbiology, 2002, 78(1): 79-97. DOI:10.1016/S0168-1605(02)00233-7.
[63] ROSS T, MCMEEKIN T A. Modeling microbial growth within food safety risk assessments[J]. Risk Analysis, 2003, 23(1): 179-197. DOI:10.1111/1539-6924.00299.
[64] 谢晶, 邱伟强. 我国食品冷藏链的现状及展望[J]. 中国食品学报, 2013, 13(3): 1-7.
[65] OLIVER J D. Recent fi ndings on the viable but nonculturable state in pathogenic bacteria[J]. FEMS Microbiology Reviews, 2010, 34(4): 415-425. DOI:10.1111/j.1574-6976.2009.00200.x.
Application and Prospects of Molecular Biology Techniques in Predictive Microbiology: A Review
ZHANG Zhaohuan1, LOU Yang1, DU Suping1, PAN Yingjie1,2,3, ZHAO Yong1,2,3,*
(1. College of Food Science and Technology, Shanghai Ocean University, Shanghai 201306, China; 2. Shanghai Engineering Research Centre of Aquatic-Product Processing & Preservation, Shanghai 201306, China; 3. Laboratory of Quality & Safety Risk Assessment for Aquatic Product on Storage and Preservation (Shanghai), Ministry of Agriculture, S hanghai 201306, China)
Abstract:Predictive microbiology is an important area of food microbiology, and its essence lies in the application of mathematical model to describe the growth and inactivation of microbiology under specific environmental conditions. Predictive microbiology model could not only predict the shelf life of foods to control spoilage organisms, but also contribute to food microbial risk assessment system to reduce the risk of pathogen infection. Therefore, it is of great importance for guaranteeing food safety and improving public health. This review overviews the history of predictive microbiology and analyzes the current research hotspots in predictive microbiology. The emphasis of this review is placed on the state of the art in the application of molecular biology techniques in predictive microbiology, and it also expounds the concept and methodological development of molecular predictive model. The feasibility of the application of other molecular biology techniques in predictive microbiology and the future pros pects for the application of molecular predictive model are also discussed. This review is expected to provide a firm theoretical basis for promoting the advancement of predictive microbiology.
Key words:molecular biology techniques; predictive microbiology; risk assessment; food safety; shelf life
DOI:10.7506/spkx1002-6630-201709040
中图分类号:TS201.3
文献标志码:A
文章编号:1002-6630(2017)09-0248-10
引文格式:
张昭寰, 娄阳, 杜苏萍, 等. 分子生物学技术在预测微生物学中的应用与展望[J]. 食品科学, 2017, 38(9): 248-257. DOI:10.7506/spkx1002-6630-201709040. http://www.spkx.net.cn
ZHANG Zhaohuan, LOU Yang, DU Suping, et al. Application and prospects of molecular biol ogy techniques in predictive micr obiology: a review[J]. Food Science, 2017, 38(9): 248-257. (in Chinese with English abstract)
DOI:10.7506/spkx1002-6630-201709040. http://www.spkx.net.cn
收稿日期:2016-04-27
基金项目:国家自然科学基金面上项目(31271870;31571917);上海市科委计划项目( 14DZ1205100;14320502100);上海市科技兴农重点攻关项目(沪农科攻字2014第3-5号、2015第4-8号);上海海洋大学研究生教育创新计划项目
作者简介:张昭寰(1989—),男,博士研究生,研究方向为食品安全风险评估。E-mail:gongziwuhen@126.com
*通信作者:赵勇(1975—),男,教授,博士,研究方向为食品安全与生物技术。E-mail:yzhao@shou.edu.cn.