邵碧英,陈文炳,刘正才,缪婷玉,彭 娟,杨 方
(福建出入境检验检疫局 福建省检验检疫技术研究重点实验室,福建 福州 350001)
摘 要:用DNA folding form在线软件分析河豚毒素DNA适配子A3、G10的二级结构,根据二级结构,分别合成适配子A3、G10的各4 个区域的5’端地高辛标记的序列。用丙烯酸-环氧乙烷珠子固定河豚毒素,测定抗地高辛碱性磷酸酶的适宜稀释倍数,用地高辛-抗地高辛碱性磷酸酶系统检测各区域与河豚毒素的亲和力,识别出适配子的核心区域。结果表明,抗地高辛碱性磷酸酶的适宜工作浓度为2×10 4倍稀释;合成的适配子A3的4 个区域中,A3-2区域与河豚毒素的亲和力最强,即适配子A3的核心区域为A3-2区域,实际上含有47 个核苷酸;合成的适配子G10的4 个区域中,G10-4区域与河豚毒素的亲和力最强,即适配子G10的核心区域为G10-4区域,实际上含有56 个核苷酸。
关键词:河豚毒素;适配子;核心区域;亲和力
适配子(也称适体)是通过指数富集配基的系统进化(systematic evolution of ligands by exponential enrichment,SELEX)技术从人工合成的DNA或RNA随机库中筛选出来的,与非核酸靶物质特异结合的高亲和性单链寡核苷酸序列。适配子在蛋白质 [1-5]、病毒 [6]、细菌 [7-9]、生物毒素 [10-16]、重金属 [17-18]、小分子物质 [19-23]等的筛选、制备及应用方面的报道已有不少。河豚毒素是毒性最强的非蛋白类神经毒素之一,是一种小分子物质,在采用SELEX技术筛选制备其DNA适配子的研究上已取得一定进展,应用SELEX技术、结合诱变聚合酶链式反应技术,通过克隆、测序、亲和力测定等手段,从人工合成的随机ssDNA文库中筛选、制备了2 条能特异结合河豚毒素且亲和力强的DNA适配子A3和G10,并进行了初步应用,在河豚毒素的分子生物学检测、化学分析中有较好的效果 [24-26]。但是,有文献报道,经过实验筛选获得的适配子在实际应用中起主要作用的只是其中的一小部分核苷酸,即所谓的核心区域 [3]。一旦识别出适配子的核心区域,剔除了不需要的核苷酸部分,既有利于适配子二级结构的形成使之更易与靶物质结合,又便于人工合成,减少成本。因此,本实验在前期研究成果 [25-26]基础上,以制备的河豚毒素DNA适配子为研究对象,分析其二级结构,根据二级结构合成不同区域的地高辛标记的序列,采用地高辛-抗地高辛碱性磷酸酶系统测定各区域与河豚毒素的亲和力,从而识别出适配子A3、G10的核心区域。
1.1 材料与试剂
河豚毒素(纯度≥99%,HPLC纯) 成都曼思特生物技术有限公司;丙烯酸-环氧乙烷珠子 美国Promega公司;抗地高辛碱性磷酸酶 艾美捷科技有限公司;对硝基苯磷酸二钠 厦门泰京生物技术有限公司;DNase/RNase-Free Deionized Water(ddH 2O)天根生化科技(北京)有限公司;结合缓冲液、磷酸钾缓冲液、洗涤缓冲液、封闭液、对硝基苯磷酸二钠溶液、酶终止液参照文献[24]配制;其他试剂均为分析纯。
1.2 仪器与设备
Mikro 120台式高速离心机 德国Hettich zentrifugen公司;SpectraMax M5多功能酶标仪 美国Thermo公司。
1.3 方法
1.3.1 适配子二级结构的分析及各区域的合成
将适配子A3、G10的序列用DNA folding form在线软件进行二级结构分析 [27]。根据二级结构,选择不同的区域,委托上海生工生物工程技术有限公司合成5’端地高辛标记的序列。加ddH 2O将序列稀释至50 μmol/L,-20 ℃保存备用。
1.3.2 河豚毒素的固定
加1 mL体积分数为1%的乙酸溶液,将1 mg河豚毒素干粉完全溶解,用0.1 mol/L磷酸钾缓冲液(pH 7.5)稀释至质量浓度为100 mg/L;称取250 mg丙烯酸-环氧乙烷珠子,加入1 mL稀释后的河豚毒素溶液,涡旋混匀,在室温条件下放置48 h;用0.01 mol/L 磷酸钾缓冲液(pH 7.5)悬浮珠子,在垂熔漏斗中用至少10 mL相同缓冲液漂洗,真空抽滤;加入1 mL含体积分数为5%的水合巯基乙醇的1 mol/L甘氨酸溶液(pH 7~8),室温条件下放置16 h;将珠子置于垂熔漏斗上,先用10 mL ddH 2O后用10 mL 0.01 mol/L磷酸钾缓冲液(pH 7.5)冲洗,真空抽滤;加入25 mL结合缓冲液,使河豚毒素-珠子的质量浓度为10 mg/mL。
1.3.3 抗地高辛碱性磷酸酶适宜稀释倍数的测定
加500 μL ddH 2O溶解抗地高辛碱性磷酸酶干粉,再加500 μL灭菌甘油,混匀,分装成100 μL/管,-20 ℃保存;用结合缓冲液将抗地高辛碱性磷酸酶分别进行5×10 3、1×10 4、2×10 4、4×10 4、8×10 4、1.6×10 5倍稀释;各取200 μL抗地高辛碱性磷酸酶溶液,加200 μL对硝基苯磷酸二钠溶液,显色20 min;加200 μL NaOH溶液(2 mol/L),终止显色反应。在30 min内,吸取200 μL反应液于酶标微孔中,于多功能酶标仪测定OD 405 nm值。
1.3.4 核心区域的识别
1.5 mL离心管先用封闭液于37 ℃封闭4 h,用洗涤缓冲液洗涤1 次,风干;用结合缓冲液将各区域序列稀释至1 μmol/L,95 ℃变性5 min,冷却至室温;在封闭管中加入100 μL河豚毒素-珠子和150 μL各区域序列,同时以不加序列的河豚毒素-珠子为空白对照,各重复3 管,室温结合30 min,期间不断涡旋;瞬离,弃上清液,加600 μL结合缓冲液漂洗,重复3 次;加入200 μL经1.3.3节测定的适宜稀释度的抗地高辛碱性磷酸酶,室温条件下放置20 min,期间不断涡旋;瞬离,弃上清液,漂洗3 次;加入200 μL对硝基苯磷酸二钠溶液,显色20 min;加200 μL NaOH(2 mol/L)溶液,中止显色;吸取200 μL于酶联板的微孔中,测定OD 405 nm值。
2.1 适配子二级结构的分析结果

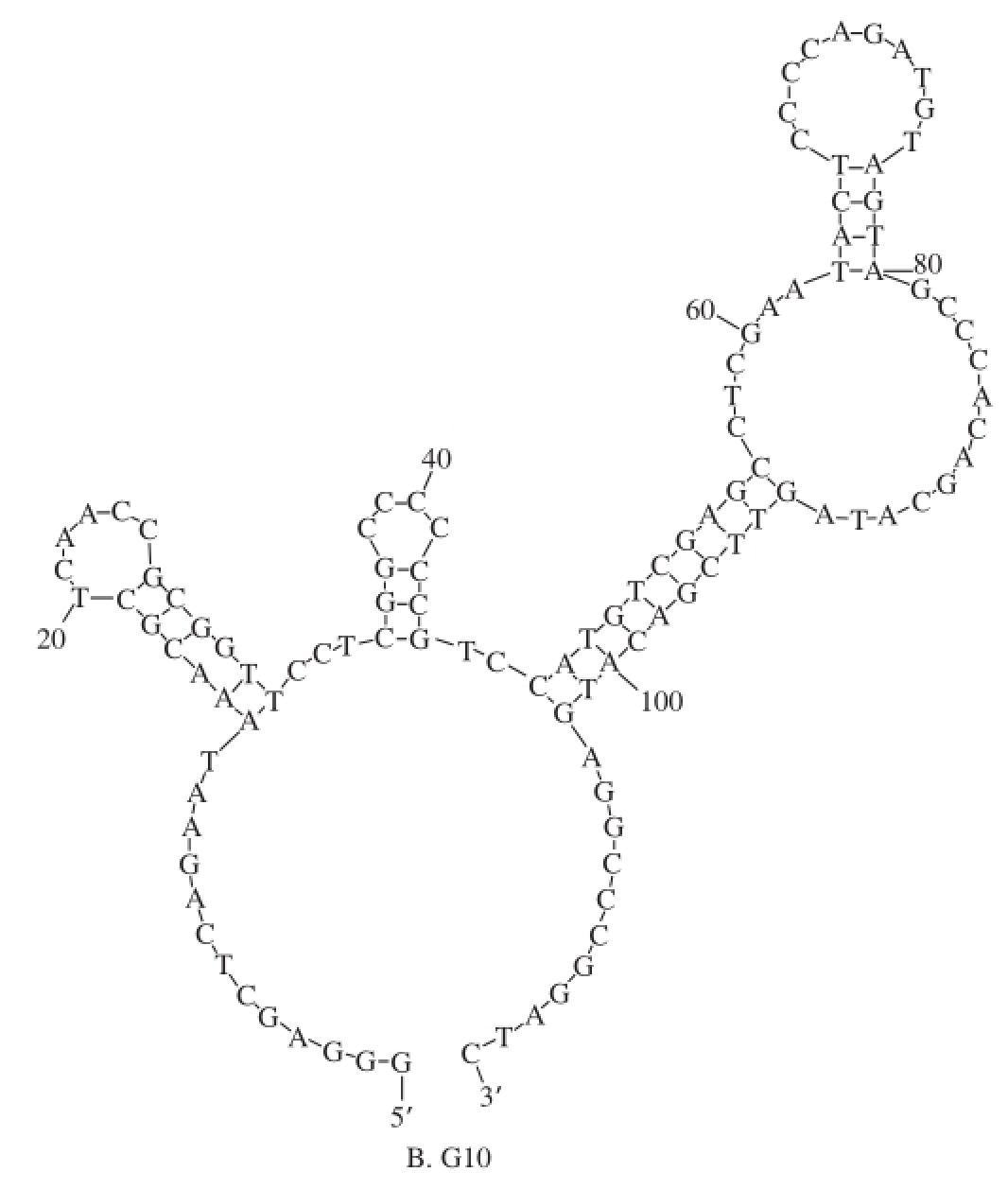
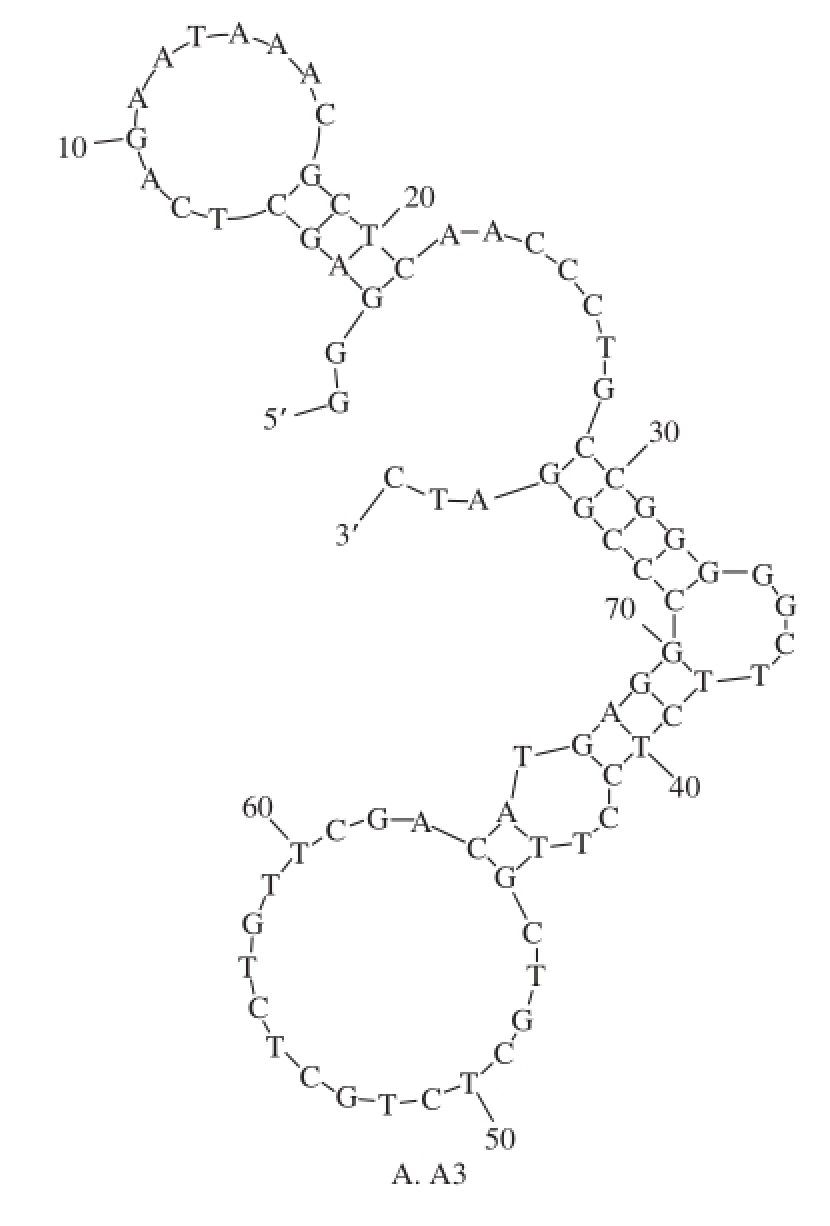
图1 适配子的二级结构
Fig.1 Secondary structures of DNA aptamers A3 and G10
适配子A3的序列为:5’-GGG AGC TCA GAA TAA ACG CTC AAC CCT GCC GGG GGC TTC TCC TTG CTG CTC TGC TCT GTT CGA CAT GAG GCC CGG ATC-3’,共78 个碱基。适配子G10的序列为:5’-GGG AGC TCA GAA TAA ACG CTC AAC CGC GGT TCC TCG GCC CCC CGT CCA TGT CGA GCC TCG AAT ACT CCC CAG ATG TAG TAG CCC ACA GCA TAG TTC GAC ATG AGG CCC GGA TC-3’,共113 个碱基。经DNA folding form在线软件分析,获得的适配子A3、G10的二级结构如图1所示。A3的二级茎环结构明显地被分为2 个部分,分别由5’-GAG CTC AGA ATA AAC GCT C-3’、5’-CCG GGG GCT TCT CCT TGC TGC TCT GCT CTG TTC GAC ATG AGG CCC GG-3’这2 段序列组成。G10的二级茎环结构明显地被分为3 个部分,分别由5’-AAA CGC TCA ACC GCG GTT-3’、5’-CGG CCC CCC G-3’、5’-CAT GTC GAG CCT CGA ATA CTC CCC AGA TGT AGT AGC CCA CAG CAT AGT TCG ACA TG-3’这3 段序列组成。
2.2 区域的选择结果
根据适配子二级结构的分析结果,又考虑到合成序列的5’端需标记地高辛,为了不影响完整的二级结构的形成,参考文献[2],在选取的各个区域的5’端多出3个碱基,在3’端多出1 个碱基。A3、G10各选取了4 个区域(表1),其中A3-3、A3-4是A3-2的部分区域,G10-3包括了G10-1和G10-2两个部分。用DNA folding form软件分析后,选取的各区域的二级结构不受增加的碱基的影响,且与在适配子A3、G10二级结构中的茎环结构完全一致。
表1 选择的区域及二级结构
Table1 Regions selected and their secondary structures

?
2.3 河豚毒素的固定
河豚毒素是一种小分子物质,在测定河豚毒素与各区域的亲和力时,需要固定河豚毒素。河豚毒素的分子式为C 11H 17O 8N 3,结构特征是有1 个碳环,1 个胍基,6 个羟基,在C-5和C-10位有一个半醛糖内酯连接着的分开的环(图2)。丙烯酸-环氧乙烷珠子具有化学稳定性高、极端的机械强度、非常良好的流动性能、良好的结合能力以及在pH值在0~12范围不受限制的适用性等优点,它通过环氧乙烷基团可以与蛋白质、硫醇、羟基和胺反应。鉴于河豚毒素和丙烯酸-环氧乙烷珠子的特性,因此完全可以用丙烯酸-环氧乙烷珠子来固定河豚毒素。
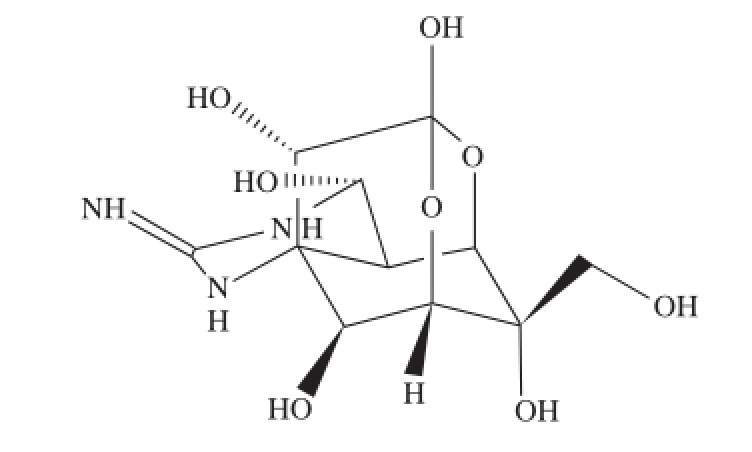
图2 河豚毒素的分子结构
Fig.2 Molecular structure of tetrodotoxin
2.4 抗地高辛碱性磷酸酶适宜稀释倍数的测定结果
随着抗地高辛碱性磷酸酶稀倍数的增加,加入底物后显色反应越来越弱,黄色越来越浅,稀释度为5×10 3、1×10 4、2×10 4时,颜色均很明亮(图3)。OD 405 nm值也随着稀释倍数的增加而减小,同一稀释度的平行3 管的读数接近,重复性好(表2)。当稀释度为1×10 4时,颜色过深超出了仪器的读数范围,而当稀释度为2×10 4时,OD 405 nm即为正常值,所以选择2×10 4为抗地高辛碱性磷酸酶的适宜稀释倍数。
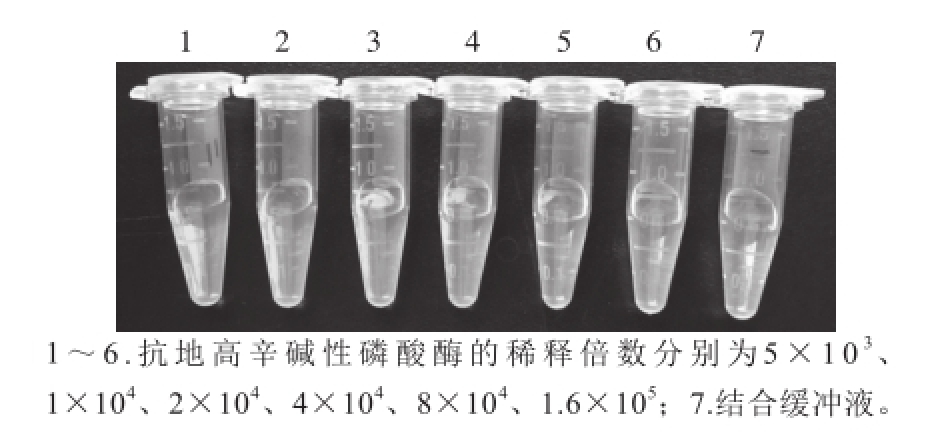
图3 不同稀释倍数的抗地高辛碱性磷酸酶的显色结果
Fig.3 Coloration results of anti-digoxin alkaline phosphatase diluted at different ratios
表2 不同稀释倍数的抗地高辛碱性磷酸酶的OD
405
nm测定值
Table2 OD
405
nmvalues of anti-digoxin alkaline phosphatase diluted at different ratios

注:4.000即为颜色过深,超出仪器测定范围。
?
2.5 核心区域的识别结果
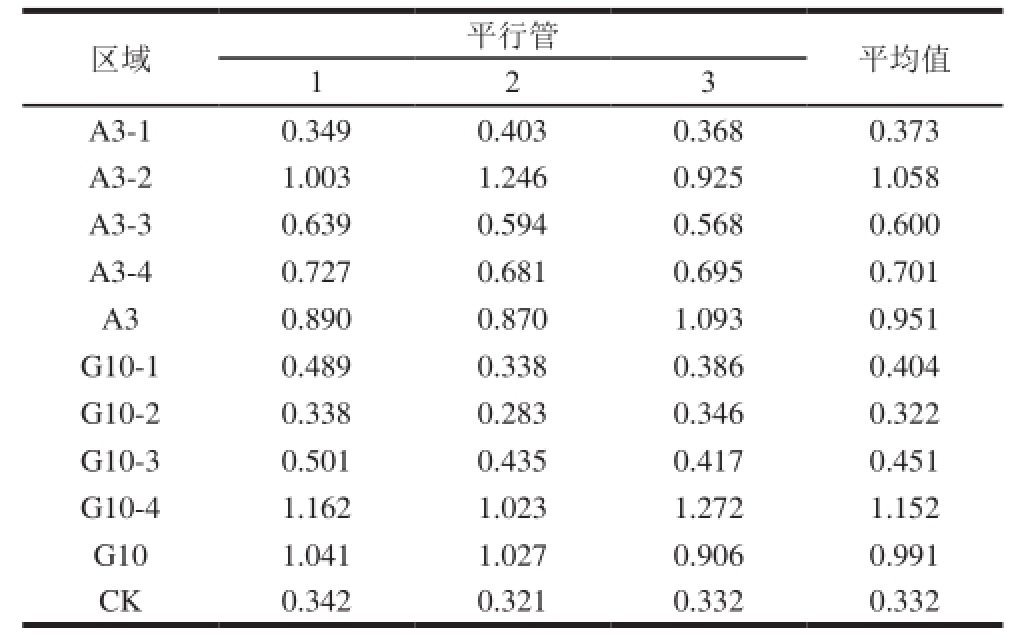
核心区域是通过测定各区域与河豚毒素的亲和力来进行识别的,而各区域与河豚毒素亲和力的强弱通过酶标仪测定的OD 405 nm值的大小来反映,数值越大亲和力越强,OD 405 nm值最大或与原适配子接近的区域即为核心区域。亲和力测定结果如表3所示。对于适配子A3,选定的4 个区域中,A3-1区域的OD 405 nm值与空白对照接近;A3-2区域的OD 405 nm值最大且稍大于适配子A3的OD 405 nm值;A3-3、A3-4区域的OD 405 nm值虽然大于A3-1区域,但都低于A3-2区域。对于适配子G10,选定的4 个区域中,G10-1、G10-2、G10-3区域的OD 405 nm值均低于G10,而G10-4区域的OD 405 nm值最大且稍大于适配子G10的OD 405 nm值。结果表明,原先含有78 个核苷酸的适配子A3的核心区域为A3-2区域,实际上含有47 个核苷酸;原先含有113 个核苷酸的适配子G10的核心区域为G10-4区域,实际上含有56 个核苷酸。
表3 各区域与河豚毒素亲和力的OD
405
nm测定结果
Table3 Results for the determination of aff i nity between each region with tetrodotoxin
?
河豚毒素是一种毒性很强的小分子物质,目前使用较普遍的检测方法是色谱法 [28]、色谱-质谱联用法 [29]、液相色谱-荧光检测法 [30]等,这些方法虽然特异性强、灵敏度高,但都需要昂贵的仪器且有很强的基质抑制效应。本实验成功筛选到2 条能与河豚毒素特异结合、亲和力高的DNA适配子A3、G10,建立了基于DNA适配子的河豚毒素分子生物学检测方法,无需昂贵的仪器和复杂的操作,具有操作简便、成本低廉、快速、灵敏等优点 [26],应用于色谱检测法中能很好地解决基质抑制效应 [24]。适配子的二级结构是适配子能与靶物质结合的结构基础,适配子的寡核苷酸之间可以通过碱基的相互作用,形成各种形状的空间结构,如发卡、茎-环、凸环、G-四分体、假结等,这些结构通过静电引力、分子间氢键、分子堆积及形状互补匹配等作用形式与靶物质特异性结合,形成稳定的靶物质-核酸适体复合物。在国内,虽然有多篇适配子(适体)的筛选及应用的研究报道,但是还未见识别适配子核心区域的研究报道。河豚毒素DNA适配子A3、G10均包括了两端固定序列和采用SELEX技术从随机序列库中筛选到的特定序列 [20-21],本实验在分析适配子二级结构的基础上对其核心区域进行了识别。
本实验先前是采用DNAMAN软件对适配子进行二级结构的分析 [24-26],根据分析得到的二级结构合成各区域的序列,但是亲和力测定结果都不满意。参考了国外的研究报道 [3,27],采用DNA folding form在线软件来分析适配子的二级结构,终于获得满意结果,识别出适配子的核心区域。将适配子A3由78 个碱基缩短为47 个碱基,将GA10从113 个碱基缩短为56 个碱基,降低了合成序列的成本。从亲和力测定结果看,2 个核心区域与河豚毒素的亲和力均大于原适配子,可能与序列缩短了、避免了额外无关序列的干扰更容易形成稳定的二级结构,从而更有利于与河豚毒素的结合有关。识别到的2 个核心区域均较长,其二级结构均较复杂,有可能是由于河豚毒素是小分子物质,越复杂的二级结构越有利于与河豚毒素的结合。河豚毒素DNA适配子A3的核心区域A3-2以及适配子G10的核心区域G10-4的应用效果如何尚待研究。
参考文献:
[1] WANG Xiaoli, LI Fang, SU Yanhua, et al. Ultrasensitive detection of protein using an aptamer-based exonuclease protection assay[J]. Analytical Chemistry, 2004, 76(19): 5605-5610.
[2] YOSHIHITO Y, KATSUNORI H, NOBUYA S, et al. Antibodyspeci fi c aptamer-based PCR analysis for sensitiveprotein detection[J]. Analytical and Bioanalytical Chemistry, 2009, 395(4): 1089-1096. DOI:10.1007/s00216-009-3041-0.
[3] LIN Junsheng, KENNETH P M. Aptamer-based regionally protected PCR for protein detection[J]. Clinical Chemistry, 2009, 55(9): 1686-1693. DOI:10.1373/clinchem.2009.127266.
[4] LI Lidong, ZHAO Hongtao, CHEN Zhengbo, et al. Aptamer-based electrochemical approach to the detectionof thrombin by modi fi cation of gold nanoparticles[J]. Analytical and Bioanalytical Chemistry, 2010, 398: 563-570.
[5] 江树勋, 黄新, 邵碧英, 等. 转基因玉米EPSPS蛋白适体的筛选与结构分析[J]. 热带作物学报, 2010, 31(4): 600-604.
[6] LI Pengfei, ZHOU Lingli, YU Yepin, et al. Characterization of DNA aptamers generated against the soft-shelled turtle iridovirus with antiviral effects[J]. BMC Veterinary Research, 2015, 30(11): 245-255. DOI:10.1186/s12917-015-0559-6.
[7] 陈敏, 江树勋, 邵碧英, 等. 单核增生李斯特氏菌适体的筛选与结构分析[J]. 食品科学, 2009, 30(19): 197-199.
[8] WANG K Y, ZENG Y L, YANG X Y, et al. Utility of aptamerfl uorescence in situ hybridizationfor rapid detection of Pseudomonas aeruginosa[J]. European Journal of Clinical Microbiology and Infectious Diseases, 2011, 30: 273-278. DOI:10.1007/s10096-010-1074-0.
[9] 郎春燕, 江树勋, 邵碧英, 等. 应用SELEX技术筛选沙门氏菌抗原的适配子[J]. 食品科学, 2011, 32(13): 203-206.
[10] 甘龙杰, 兰小鹏, 江丽, 等. 金黄色葡萄球菌外毒素B特异性适体的筛选及其应用[J]. 生物技术通讯, 2010, 21(2): 232-235. DOI:10.3969/jissn.1009-0002.2010.02.020.
[11] 段诺, 吴世嘉, 王周平. 核酸适配体识别荧光法检测赭曲霉素A[J].分析化学, 2011, 39(3): 300-304.
[12] 范婷, 张娟琨, 李敏, 等. 适配子生物传感器对青霉素的快速检测[J]. 生物加工过程, 2011, 9(3): 41-46. DOI:10.3969/ j.issn.1672-3678.2011.03.009.
[13] 杨锡辉, 孔维军, 杨美华, 等. 适配子识别技术在真菌毒素快速分析中的应用[J]. 分析化学, 2013, 41(2): 297-306.
[14] 周伟璐, 王宇婷, 孔维军, 等. 胶体金色谱技术在赭曲霉毒素A快检中的研究进展[J]. 中国中药杂志, 2015, 40(15): 2945-2951.
[15] 桂海娈, 金庆日, 张亚军, 等. 核酸适体检测赭曲霉毒素A荧光方法的建立[J]. 浙江畜牧兽医, 2015, 41(3): 1-5. DOI:10.3969/ j.issn.1005-7307.2015.03.001.
[16] 赵秋伶, 史素青. 酶联适体分析法检测葡萄酒中的赭曲霉素A[J]. 食品与生物技术学报, 2015, 34(4): 396-401.
[17] 雷兆静, 张存政, 胡秋辉. 核酸适体识别荧光法检测汞离子[J]. 分析化学, 2012, 40(12): 1827-1831.
[18] 刘庆业, 范燕燕, 凌绍明, 等. 适体修饰金纳米粒子光度法检测铅(Ⅱ)[J]. 冶金分析, 2010, 30(4): 58-61. DOI:10.3969/ j.issn.1000-7571.2010.04.012.
[19] CHEN Zhang, LI Guang, ZHANG Lan, et al. A new method for the detection of ATP using a quantum-dot-tagged aptamer[J]. Analytical and Bioanalytical Chemistry, 2008, 392(6): 1185-1188.
[20] 刘宾, 刘颖, 义志忠, 等. 适体在抗生素残留检测中的应用[J]. 食品科学, 2010, 31(17): 452-456.
[21] 丁晓莹, 李双, 王周平. 基于核酸适配子识别的荧光法检测可卡因[J]. 分析试验室, 2011, 30(4): 19-21. DOI:10.3969/ j.issn.1000-0720.2011.04.005.
[22] 高淑丽, 徐慧. 基于核酸适体的无标记氯化血红素荧光检测[J].鲁东大学学报(自然科学版), 2011, 27(2): 131-134. DOI:10.3969/ j.issn.1673-8020.2011.02.009.
[23] 魏泉德, 林毅雄, 沈韧, 等. 基于适配子及Dot-ELISA快速检测氯霉素残留[J]. 中国卫生检验杂志, 2013, 23(5): 1158-1160.
[24] 高兴. 河豚毒素特异性DNA适配子的筛选[D]. 福州: 福州大学, 2011.
[25] 邵碧英, 高兴, 杨方, 等. 河豚毒素DNA适配子的筛选与结构分析[J]. 中国食品学报, 2012, 12(2): 137-143. DOI:10.3969/ j.issn.1009-7848.2012.02.021.
[26] 邵碧英, 陈彬, 陈文炳, 等. 河豚毒素DNA适配子的制备及应用[J].食品科学, 2014, 35(24): 205-208. DOI:10.7506/spkx1002-6630-201424039.
[27] ZUKER M. Mfold Web server for nucleic acid folding and hybridization prediction[J]. Nucleic Acids Research, 2003, 31(13): 3406-3415. DOI:10.1093/nar/gkg595.
[28] 张虹, 柳正良, 黄蓓琳, 等. 反相离子对-高效液相色谱法测定河豚毒素[J]. 中国现代应用药学杂志, 2001, 8(3): 197-198.
[29] 阮丽萍, 蔡梅, 刘华良, 等. 高效液相色谱-串联质谱法测定烤鱼片中的河豚毒素[J]. 江苏预防医学, 2014, 25(2): 11-13.
[30] 杨方, 钱疆, 刘正才, 等. 水产品中河豚毒素的测定 液相色谱-荧光检测法: GB/T 23217—2008[S]. 北京: 中国标准出版社, 2008.
Identif i cation of Core Region of DNA Aptamers against Tetrodotoxin
SHAO Biying, CHEN Wenbing, LIU Zhengcai, MIAO Tingyu, PENG Juan, YANG Fang
(Fujian Provincial Key Laboratory of Inspection and Quarantine Technology Research, Fujian Entry-Exit Inspection and Quarantine Bureau, Fuzhou 350001, China)
Abstract:The secondary structures of DNA aptamers A3 and G10 against tetrodotoxin were analyzed using DNA folding form online software. Further, digoxin labeled 5’ end sequences of four regions in each aptamer were separately synthesized. Tetrodotoxin was fixed by acrylic acid-epoxy ethane beads. The core regions of these aptamers were identified using digoxin-anti digoxin alkaline phosphatase system to detect the affinity of these various regions with tetrodotoxin, after determining the appropriate dilution ratio of anti-digoxin alkaline phosphatase. The results showed that the suitable working concentration of anti-digoxin alkaline phosphatase was 2 × 10 4times dilution. The core region of aptamer A3 was A3-2 area which had the strongest aff i nity with tetrodotoxin among the four regions, including 47 nucleotides, while the core region of aptamer G10 was G10-4 area, including 56 nucleotides.
Key words:tetrodotoxin; aptamer; core region; aff i nity
DOI:10.7506/spkx1002-6630-201704042
中图分类号:Q78
文献标志码:A
文章编号:1002-6630(2017)04-0260-05
引文格式:
邵碧英, 陈文炳, 刘正才, 等. 河豚毒素DNA适配子核心区域的识别[J]. 食品科学, 2017, 38(4): 260-264. DOI:10.7506/ spkx1002-6630-201704042. http://www.spkx.net.cn
SHAO Biying, CHEN Wenbing, LIU Zhengcai, et al. Identif i cation of core region of DNA aptamers against tetrodotoxin[J]. Food Science, 2017, 38(4): 260-264. (in Chinese with English abstract)
DOI:10.7506/spkx1002-6630-201704042. http://www.spkx.net.cn
收稿日期:2016-06-30
基金项目:国家质检总局科技计划项目(2014IK104)
作者简介:邵碧英(1973—),女,研究员,博士,研究方向为食品生物学分子检测。E-mail:byshao@sohu.com