植物乳杆菌FS5-5在盐胁迫下的转录组学分析
宋雪飞
1,郭晶晶
1,姜 静
1,唐筱扬
1,张 颖
1,乌日娜
1,2,*
(1.沈阳农业大学食品学院,辽宁 沈阳 110866;2.江南大学食品学院,食品科学与技术国家重点实验室,江苏 无锡 214122)
摘 要:以分离自东北自然发酵大酱中的一株耐盐植物乳杆菌FS5-5(Lactobacillus plantarum FS5-5)为实验对象,在转录水平上对该菌株盐胁迫相关基因表达进行了研究。结果表明:在对数生长期,表达显著下调的共29 个基因,分别为参与碳水化合物转运和代谢的4 个基因,氨基酸转运和代谢的9 个基因,维生素代谢的3 个基因,核苷酸代谢的6 个基因,遗传信息翻译、核糖体结构和形成的7 个基因;表达显著上调的有参与碳水化合物转运和代谢的4 个基因。这些变化可能与L. plantarum FS5-5的盐胁迫机制密切相关。选取参与维生素代谢的3 个基因和核苷酸代谢的1 个基因,通过实时定量聚合酶链式反应对转录组学结果进行验证,结果表明两种方法中基因表达趋势一致。实验对L. plantarum FS5-5的盐胁迫反应进行了比较全面的分析,为提高工业生产中菌株的耐受性提供了理论依据。
关键词:植物乳杆菌;盐胁迫;实时定量聚合酶链式反应;转录组学
目前,越来越多的植物乳杆菌(L a c to b a c illu s plantarum)被证实为益生菌
[1]。植物乳杆菌代谢产物中,除氨基酸、短肽、乳酸外,还有多种有机酸、过氧化氢、细菌素、双乙酰等天然抑菌物质
[2]。L. plantarum具有诸多生理功能,包括改善胃肠道功能、抗肿瘤作用、增强机体免疫力、降低胆固醇、平衡泌尿生殖系统菌群
[3]。
L. plantarum在工业上的应用也引起了人们越来越多的关注。在食品工业中,L. p la ntarum主要用于发酵生产酱油
[4]、香肠
[5]、咸鱼
[6]、泡菜
[7]等,因此L. pla n taru m极易遭受盐胁迫。盐胁迫对菌体有很大的影响,例如盐胁迫所引起的环境渗透压的突然增加,使细胞内的水分外流,引起细胞膨压的损失,改变胞内溶质的浓度和细胞体积
[8]。研究表明,盐胁迫对益生乳杆菌如嗜酸乳杆菌(Lactobacillus a cidophilus)、德氏乳杆菌保加利亚亚种(Lactobacillus delbrueckii ssp. bulgaricus)都有一定的影响
[9-11]。因此,菌株的盐胁迫适应能力对其在工业应用中的存活和生长是极其重要的。
转录组学是研究特定细胞、组织或器官在特定生长发育阶段或某种生理状况下转录组的科学
[12]。转录组包括编码蛋白质的mRNA和非编码RNA(rRNA、tRNA和其他非编码RNA)
[13]。转录组并不是固定的,它会随着生长发育阶段、生理状态和外界环境的改变而变化。因此,转录组分析成为研究生物应激生理等作用机制的有力工具。高盐浓度下,L. p la n ta ru m运用许多策略来应对外界渗透压的变化。目前,虽然有一些关于盐胁迫反应的研究,但对L. p la n ta ru m的耐盐机制仍缺乏全面深入的了解。本实验以一株分离自东北自然发酵大酱样品中的具有耐盐特性的L. plan tarum FS5-5
[1
4]为实验菌株,并对其在NaCl含量分别为0.0、1.5、3.0、4.0、5.0、6.0 g/100 mL 6 个梯度下,生长至对数生长期的基因表达进行了研究,以期从转录水平上揭示L. plantarum的耐盐反应和渗透调节机制,并为进一步开发和改造耐盐性发酵菌种提供依据。
1 材料与方法
1.1 材料与试剂
L. plan tarum FS5-5为从东北自然发酵大酱中分离筛选出的一株耐盐菌株。
液体MRS(ma n,ro g o s a a n d sh a rp)培养基:牛肉浸膏0.8 g、蛋白胨1.0 g、酵母粉0.4 g、无水乙酸钠0.3 g、葡萄糖2.0 g、吐温-8 0 0.1 g、K
2HPO
40.2 g、Mg SO
4·7H
2O 0.058 g、柠檬酸钠0.2 g、MnSO
4·H
2O 0.019 g、蒸馏水100 mL,调节pH值为6.4,于121 ℃高温灭菌30 min。
RNAprep Pure培养细胞/细菌总RNA提取试剂盒、TIANScript cDNA第1链合成试剂盒、Real Master Mix(SYBR Green)实时定量聚合酶链式反应(quantitative real-time polymerase chain reaction,q RT-PCR)试剂盒天根生化科技(北京)有限公司;Eukaryote Total RNA试剂盒、High sensitivity DNA试剂盒、High sensitivity DNA试剂盒 安捷伦科技(中国)有限公司;Nex t Ultra RNA Library Prep Kit for Illumina 美国NEB公司;Axy Prep Mag PCR Clean-up试剂盒 美国Axygen公司;TruSeq PE Cluster Kit V4双端测序簇试剂盒、TruSeq SBS Kit v4-HS循环试剂盒 美国Illumina公司。
1.2 仪器与设备
DNP-9080型生化培养箱 上海振宇化工科技有限公司;CR-21G型高速冷冻离心机 日本日立公司;5418R小型台式冷冻离心机 上海研谨生物科技有限公司;LDZX-50KBS型立式压力蒸汽灭菌锅 河南兄弟仪器设备有限公司;2100生物分析仪 美国Agilent公司;cBoT簇生成系统、Hiseq2500测序仪 美国Illumina公司;ABI 7500 Fast实时荧光定量PCR仪 美国应用生物系统公司。
1.3 方法
1.3.1 菌体收集
配制MRS培养基,取5 mL于试管中,盖上试管塞,121 ℃条件下灭菌30 min。在无菌操作台上,于无菌条件下,将穿刺保存的菌接种到试管中,于37 ℃恒温箱中培养24 h,得到活化1代的菌液。吸取100 μL活化1代的菌液至装有5 mL灭菌后的MRS培养基的试管中,于37 ℃恒温箱中培养24 h,得到活化2代的菌液。
配制NaCl含量分别为0.0、1.5、3.0、4.0、5.0、6.0 g/100 mL的MRS培养基,分别取5 mL于若干试管中,盖上试管塞,121 ℃条件下灭菌30 min。待试管中培养基冷却到室温后,在无菌操作台上,于无菌条件下用移液枪分别吸取100 µL上述活化2代的菌液于各NaCl质量浓度试管中。于37 ℃恒温箱中培养,分别于接种后第5、6、7、8、10小时收集对数期菌体。重复以上操作3 次,将相应NaCl含量下的收集菌体均匀混合。
1.3.2 转录组测序
按照RNAp re p Pu re培养细胞/细菌总RNA提取试剂盒说明书提取菌体总RNA。然后运用生物分析仪和Eukaryote Total RNA试剂盒对RNA样品进行质量检测。运用Next Ultra RNA Library Prep Kit for Illumina进行文库构建。运用AxyPrep Mag PCR Clean-up试剂盒进行文库纯化。运用生物分析仪和High sensitivity DNA试剂盒进行文库检测。运用High sensitivity DNA试剂盒进行文库定量。运用TruSeq PE Cluster Kit V4双端测序簇试剂盒和cBoT簇生成系统产生适用于Hiseq测序仪的双端测序簇。最后运用TruSeq SBS Kit v4-HS循环试剂盒在Hiseq2500测序仪上进行高通量测序。
1.3.3 qRT-PCR对转录组结果的验证
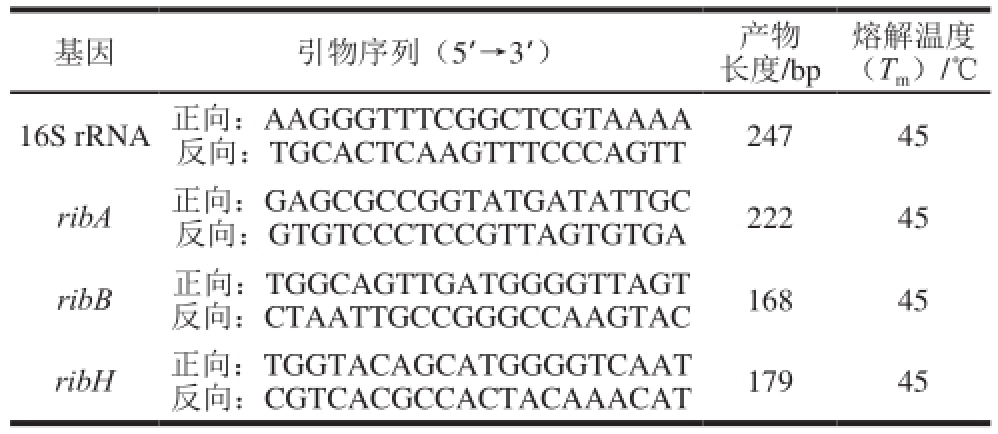
表1 实时荧光定量PCR引物
Table 1 Primers used for real-time fluorescent quantitative PCR
熔解温度(T
m)/℃16S rRNA正向:AAGGGTTTCGGCTCGTAAAA反向:TGCACTCAAGTTTCCCAGTT 247 45基因引物序列(5’→3’)产物长度/bp ribA正向:GAGCGCCGGTATGATATTGC反向:GTGTCCCTCCGTTAGTGTGA 222 45 ribB正向:TGGCAGTTGATGGGGTTAGT反向:CTAATTGCCGGGCCAAGTAC 168 45 ribH正向:TGGTACAGCATGGGGTCAAT反向:CGTCACGCCACTACAAACAT 179 45
按照TIANScript cDNA第1链合成试剂盒说明书合成cDNA第1链。qRT-PCR中所用引物列于表1中,本实验确定16S rRNA为内控基因。按照Real Master Mix(SYBR Green)试剂盒进行q RT-PCR。PCR体系(20 μL):荧光染料混合液9 μL、c DNA模板2 μL、正向引物2 μL、反向引物2 μL、超纯水5 μL。PCR扩增条件:95 ℃预变性2 min;95 ℃变性30 s,55 ℃退火33 s,68 ℃延伸34 s,循环40 次;熔解曲线:95 ℃ 30 s,60 ℃ 1 min,95 ℃ 15 s,60 ℃ 15 s。所有实验均设置3 次平行。
1.4 数据分析
1.4.1 基因表达分析
基因表达计算使用rsem软件(v1.2.6),该软件使用fragment per kilo bases per million reads(FPKM)方法计算基因表达量,FPKM计算公式如下
[15],本实验以FPKM数值0.1作为判断基因是否表达的阈值
[16]。
式中:C为唯一比对到某基因的片段数;N为唯一比对到全部基因的总片段数;L为基因的碱基长度/bp。
1.4.2 基因表达水平对比
通过所有基因的FPKM的分布图以及盒形图对不同实验条件下的基因表达水平进行比较。
1.4.3 基因差异表达分析
同实验条件下的基因差异表达分析使用Bioconductor软件包的DESeq(v1.12.1)。对DESeq检测的结果按照差异显著性标准(差异基因表达变化2 倍以上且FDR不大于0.05)进行筛选,统计基因显著性差异表达上下调情况。
1.4.4 基因差异表达聚类分析
以不同实验条件下的差异基因的FPKM值为表达水平,做层次聚类分析。
2 结果与分析
2.1 基因表达分析结果
表2 不同表达水平区间的基因数量统计
Table 2 The statistical gene numbers within different expression level ranges
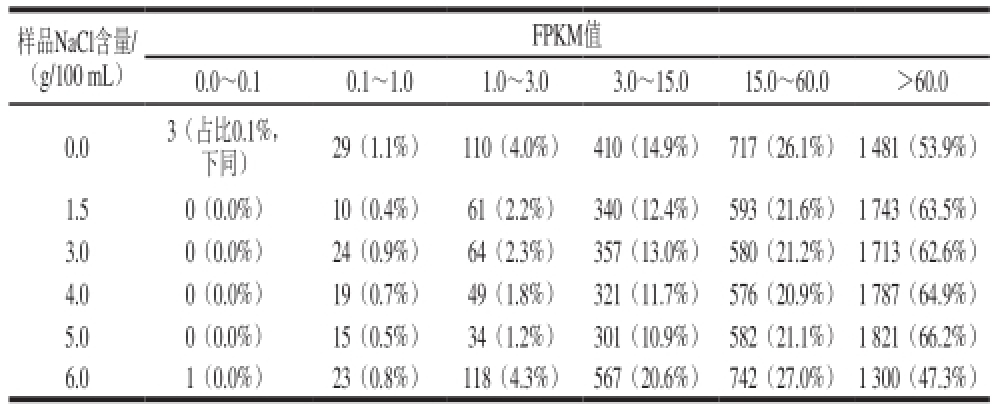
样品NaCl含量/(g/100 mL)FPKM值0.0~0.1 0.1~1.0 1.0~3.0 3.0~15.0 15.0~60.0>60.0 0.0 3(占比0.1%,下同)29(1.1%)110(4.0%)410(14.9%)717(26.1%)1 481(53.9%)1.5 0(0.0%)10(0.4%)61(2.2%)340(12.4%)593(21.6%)1 743(63.5%)3.0 0(0.0%)24(0.9%)64(2.3%)357(13.0%)580(21.2%)1 713(62.6%)4.0 0(0.0%)19(0.7%)49(1.8%)321(11.7%)576(20.9%)1 787(64.9%)5.0 0(0.0%)15(0.5%)34(1.2%)301(10.9%)582(21.1%)1 821(66.2%)6.0 1(0.0%)23(0.8%)118(4.3%)567(20.6%)742(27.0%)1 300(47.3%)
由表2可知,当样品NaCl含量为0.0、1.5、3.0、4.0、5.0、6.0 g/100 mL时,基因FPKM数值大于0.1的比率分别是99.9%、100%、100%、100%、100%和100%。因此不同NaCl含量下,基因FPKM数值几乎都大于0.1,几乎全部转录组测序得到的基因都进行了表达。
2.2 基因表达水平对比结果

图1 不同NaCl含量下基因表达水平比对图
Fig. 1 Comparison of gene expression levels at different NaCl concentrations
由图1A可知,当lgFPKM小于2时,代表NaCl含量为1.5、4.0、5.0 g/100 mL的3 条线在下侧,说明基因的密度较小;当lgFPKM大于2时,代表NaCl含量为1.5、4.0、5.0 g/100 mL的3条线在上侧,说明基因的密度较高。由图1B可知,在不同的NaCl含量下,数据的分散程度基本相同,且当NaCl含量为1.5、4.0、5.0 g/100 mL时中值较大。因此当NaCl含量为1.5、4.0、5.0 g/100 mL时,基因的整体表达量相对较高。
2.3 基因差异表达分析结果
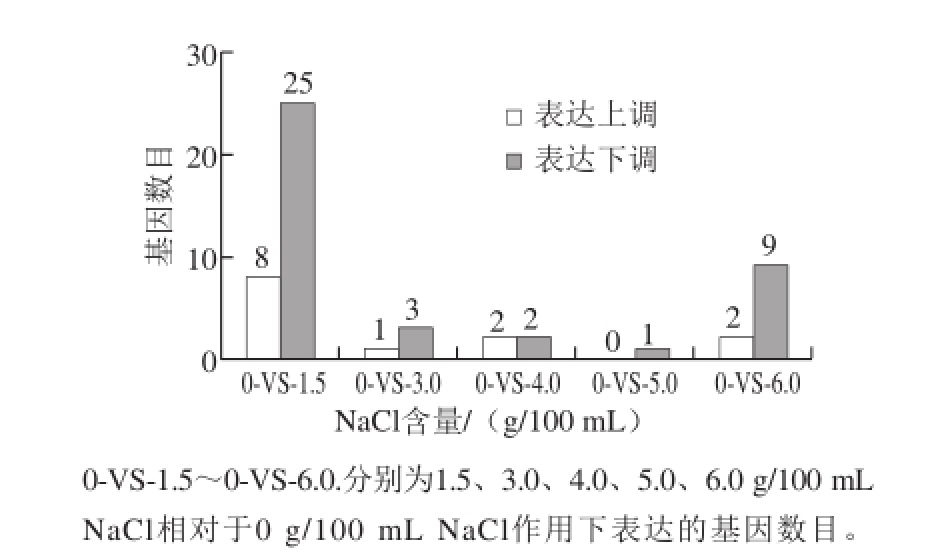
图2 不同NaCl含量条件下差异表达基因的上下调情况
Fig. 2 The up- and down-regulation of differentially expressed genes at different NaCl concentrations
由图2可知,以差异基因表达变化2 倍以上且FDR不大于0.0 5作为基因表达差异显著的标准,NaCl含量为1.5 g/100 mL相对于0 g/100 mL有8 个基因表达显著上调(IS P 2_2、m leS、lp_3 0 1 4、lp_0 3 0 4、IS P 1_1 0、ISP1_9、ISP 1_7、lp_036 1),25 个基因表达显著下调(lp_rRNA 0 8、lp_rR N A 04、lp_rR N A0 2、lp_rR N A 0 5、a sn A、lp_rR N A1 2、rib B、hisC、lp_rR N A 1 4、a sn S 1、lp_2 7 1 0、a d eC、rib A、rib H、lp_1 4 3 9、lp_1 2 8 9、lp_1746、metE、lp_rR NA1 5、lp_1374、h isA、lp_1290、hisB、hisE、lp_3679);NaCl含量为3.0 g/100 mL相对于0 g/100 mL有1 个基因表达显著上调(lp_3 01 4),3 个基因表达显著下调(srlM 2、pts38BC、p ts3 8C);NaCl含量为4.0 g/100 mL相对于0 g/100 mL有2 个基因表达显著上调(lp_3 0 14、lp_0 3 0 4),2 个基因表达显著下调(lp_rRN A 08、lp_rR NA 02);NaCl含量为5.0 g/100 mL相对于0 g/100 mL有0 个基因表达显著上调,1 个基因表达显著下调(lp_rRN A08);NaCl含量为6.0 g/100 mL相对于0 g/100 mL有2 个基因表达显著上调(lp_2 84 5、lp_3 0 1 4),9 个基因表达显著下调(p ts2 3 B、srlM2、p ts3 8C、lp_0438、glpF 3、p urQ、lp_2 9 77、p ts3 8 BC、mdxG)。
由图3可知,选取在转录组结果中显著下调的基因ribA、ribB、ribH和purQ进行qRT-PCR验证。在qRT-PCR实验中,基因的下调倍数与转录组结果不同,这有可能是以16S rRNA作为内控基因引起的。但在qRT-PCR实验与转录组分析实验中,基因的表达都是下调的,这很大程度地说明了qRT-PCR结果验证了转录组结果。
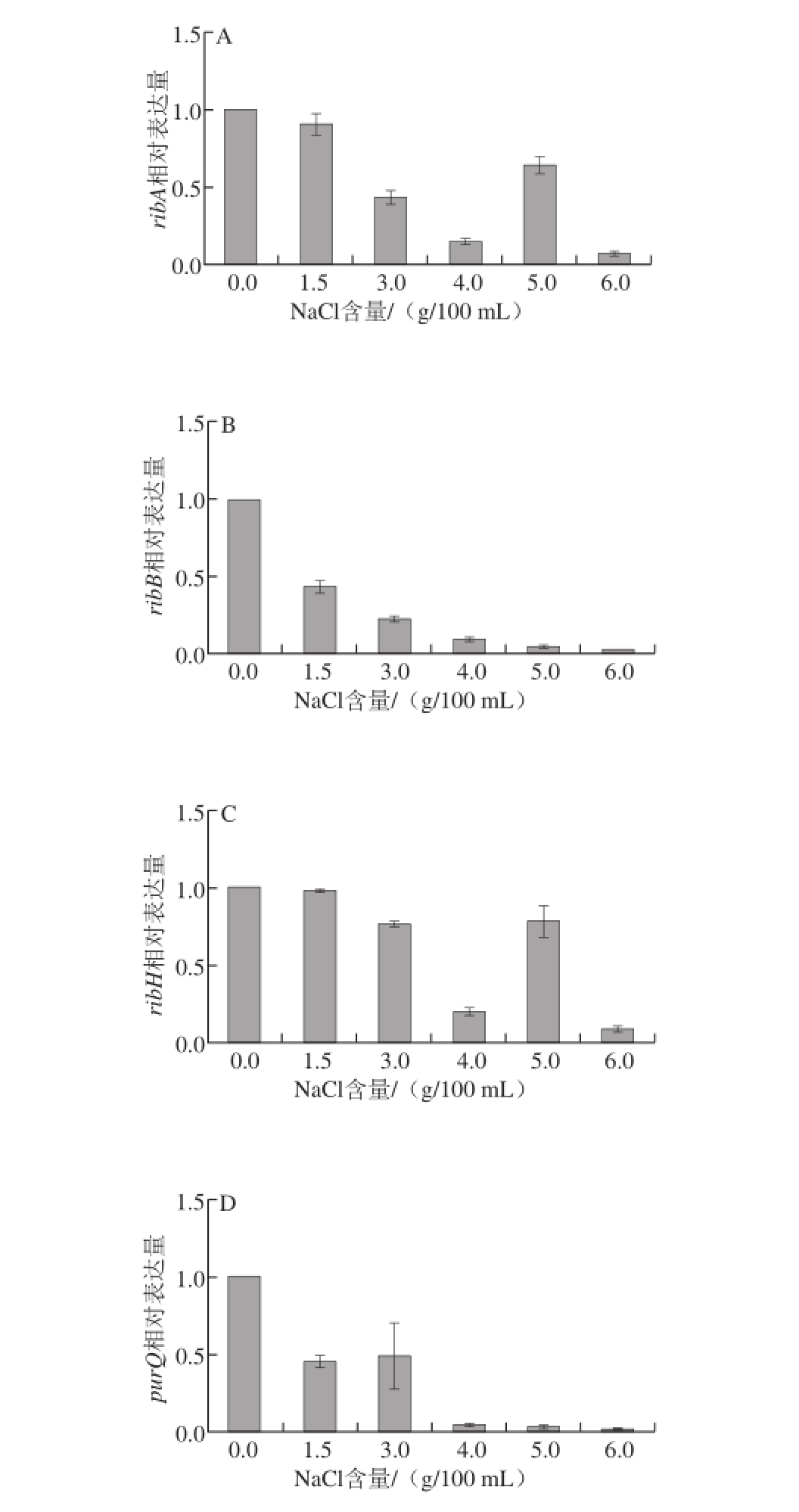
图3 不同NaCl含量条件下基因ribA(A)、ribB(B)、ribH(C)和purQ(D)的相对表达情况
Fig. 3 Expression levels of genes ribA(A),ribB(B),ribH (C) and purQ(D) at different NaCl concentrations
2.4 基因差异表达聚类分析结果
由图4可知,有6 类表达量相似的基因聚簇,每一类的基因表达模式相近,可能具有相似的功能或参与相同的生物学过程。这6 类基因聚簇所包含的具体基因列于表3。由表3可知,差异表达基因的基因本体(gene ontology,GO)富集分析表明,参与碳水化合物转运和代谢、氨基酸转运和代谢、维生素代谢、核苷酸代谢、翻译及核糖体结构和形成等多种细胞功能的基因进行了差异表达。
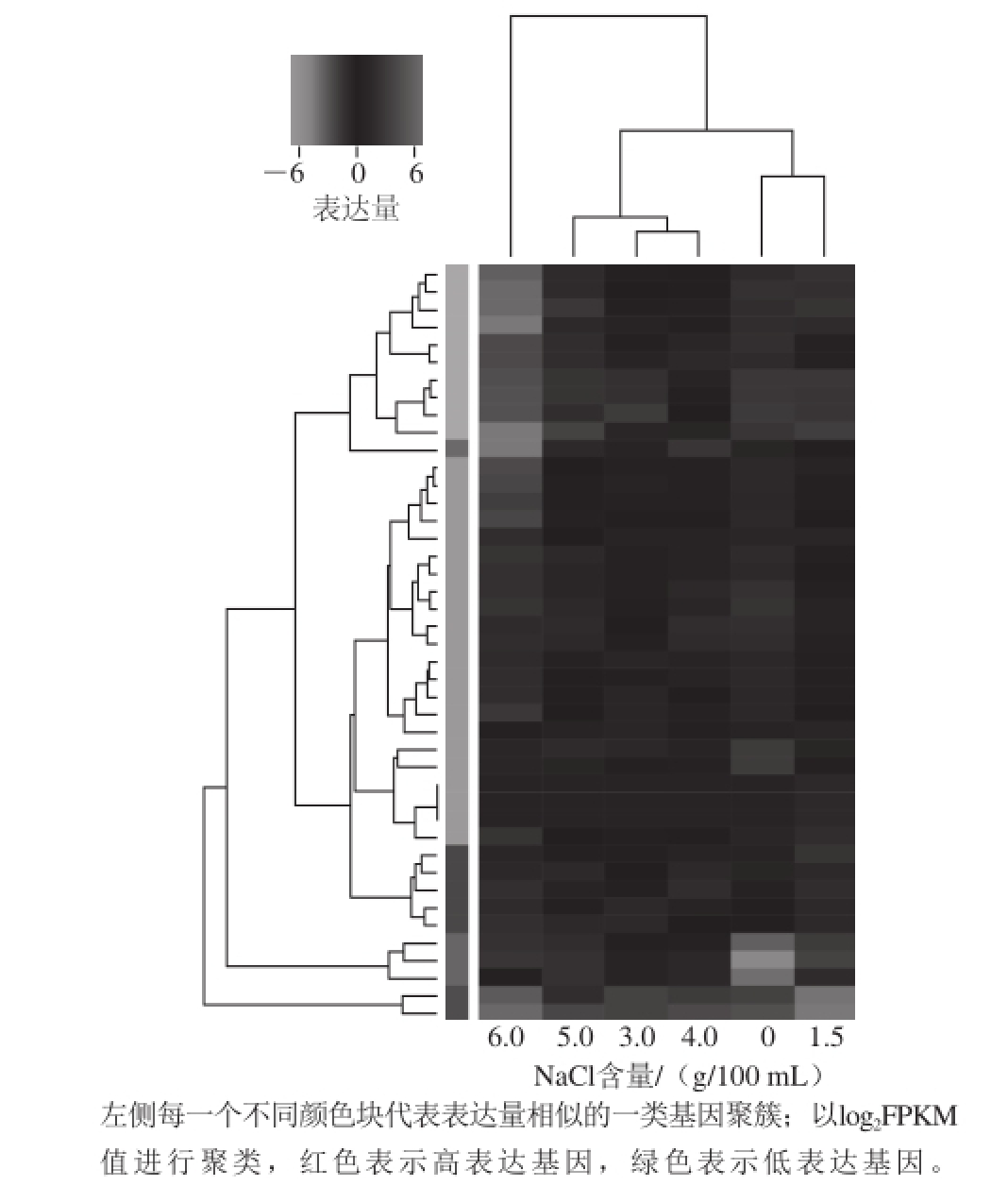
图4 不同NaCl含量条件下基因差异表达聚类图
Fig. 4 Clustering analysis of differentially expressed genes at different NaCl concentrations
2.4.1 NaCl对参与碳水化合物转运和代谢的基因表达的影响
由表3可知,mleS在NaCl含量为1.5、3.0、4.0、5.0、6.0 g/1 0 0 m L时表达均上调,且在Na Cl含量为1.5 g/1 0 0 m L时显著上调;细胞外的转糖苷酶基因lp_2 8 4 5、lp_3 0 1 4和lp_0 3 0 4在NaCl含量为1.5、3.0、4.0、5.0、6.0 g/10 0 mL时表达均上调,且lp_2 8 4 5在NaCl含量为6.0 g/100 mL时显著上调,lp_3 0 1 4在1.5、3.0、4.0、6.0 g/100 mL时显著上调,lp_03 0 4在NaCl含量为1.5、4.0 g/100 mL时显著上调;pts38 BC、pts3 8C、srlM2在NaCl含量为1.5、3.0、4.0、5.0、6.0 g/100 mL时表达均下调,且在Na Cl含量为3.0、6.0 g/10 0 mL时表达显著下调;p ts2 3 B在NaCl含量为1.5、3.0、4.0、5.0、6.0 g/1 0 0 m L时表达均下调,且在Na Cl含量为6.0 g/100 mL时表达显著下调。
表3 不同NaCl含量条件下部分表达有显著差异的基因
Table 3 Genes that showed significant differences in expression level at different NaCl concentrations
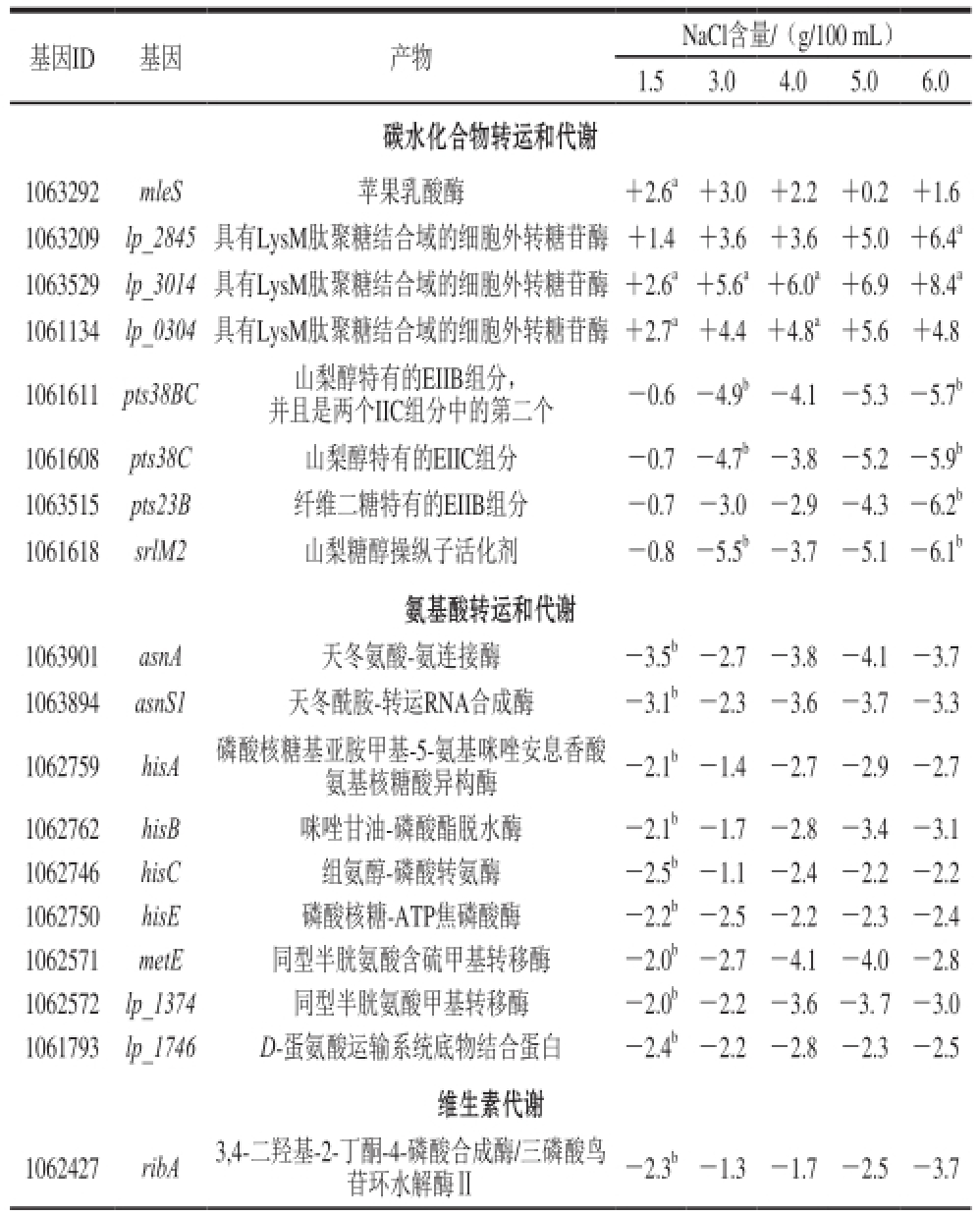
基因ID基因产物NaCl含量/(g/100 mL)1.5 3.0 4.0 5.0 6.0碳水化合物转运和代谢1063292 mleS苹果乳酸酶+2.6
a+3.0+2.2+0.2+1.6 1063209 lp_2845具有LysM肽聚糖结合域的细胞外转糖苷酶+1.4+3.6+3.6+5.0+6.4
a1063529 lp_3014具有LysM肽聚糖结合域的细胞外转糖苷酶+2.6
a+5.6
a+6.0
a+6.9+8.4
a1061134 lp_0304具有LysM肽聚糖结合域的细胞外转糖苷酶+2.7
a+4.4+4.8
a+5.6+4.8 1061611 pts38BC山梨醇特有的EIIB组分,并且是两个IIC组分中的第二个-0.6-4.9
b-4.1-5.3-5.7
b1061608 pts38C山梨醇特有的EIIC组分-0.7-4.7
b-3.8-5.2-5.9
b1063515 pts23B纤维二糖特有的EIIB组分-0.7-3.0-2.9-4.3-6.2
b1061618 srlM2山梨糖醇操纵子活化剂-0.8-5.5
b-3.7-5.1-6.1
b氨基酸转运和代谢1063901 asnA天冬氨酸-氨连接酶-3.5
b-2.7-3.8-4.1-3.7 1063894 asnS1天冬酰胺-转运RNA合成酶-3.1
b-2.3-3.6-3.7-3.3 1062759 hisA磷酸核糖基亚胺甲基-5-氨基咪唑安息香酸氨基核糖酸异构酶-2.1
b-1.4-2.7-2.9-2.7 1062762 hisB咪唑甘油-磷酸酯脱水酶-2.1
b-1.7-2.8-3.4-3.1 1062746 hisC组氨醇-磷酸转氨酶-2.5
b-1.1-2.4-2.2-2.2 1062750 hisE磷酸核糖-ATP焦磷酸酶-2.2
b-2.5-2.2-2.3-2.4 1062571 metE同型半胱氨酸含硫甲基转移酶-2.0
b-2.7-4.1-4.0-2.8 1062572 lp_1374同型半胱氨酸甲基转移酶-2.0
b-2.2-3.6-3. 7-3.0 1061793 lp_1746 D-蛋氨酸运输系统底物结合蛋白-2.4
b-2.2-2.8-2.3-2.5维生素代谢1062427 ribA 3,4-二羟基-2-丁酮-4-磷酸合成酶/三磷酸鸟苷环水解酶Ⅱ-2.3
b-1.3-1.7-2.5-3.7
续表3
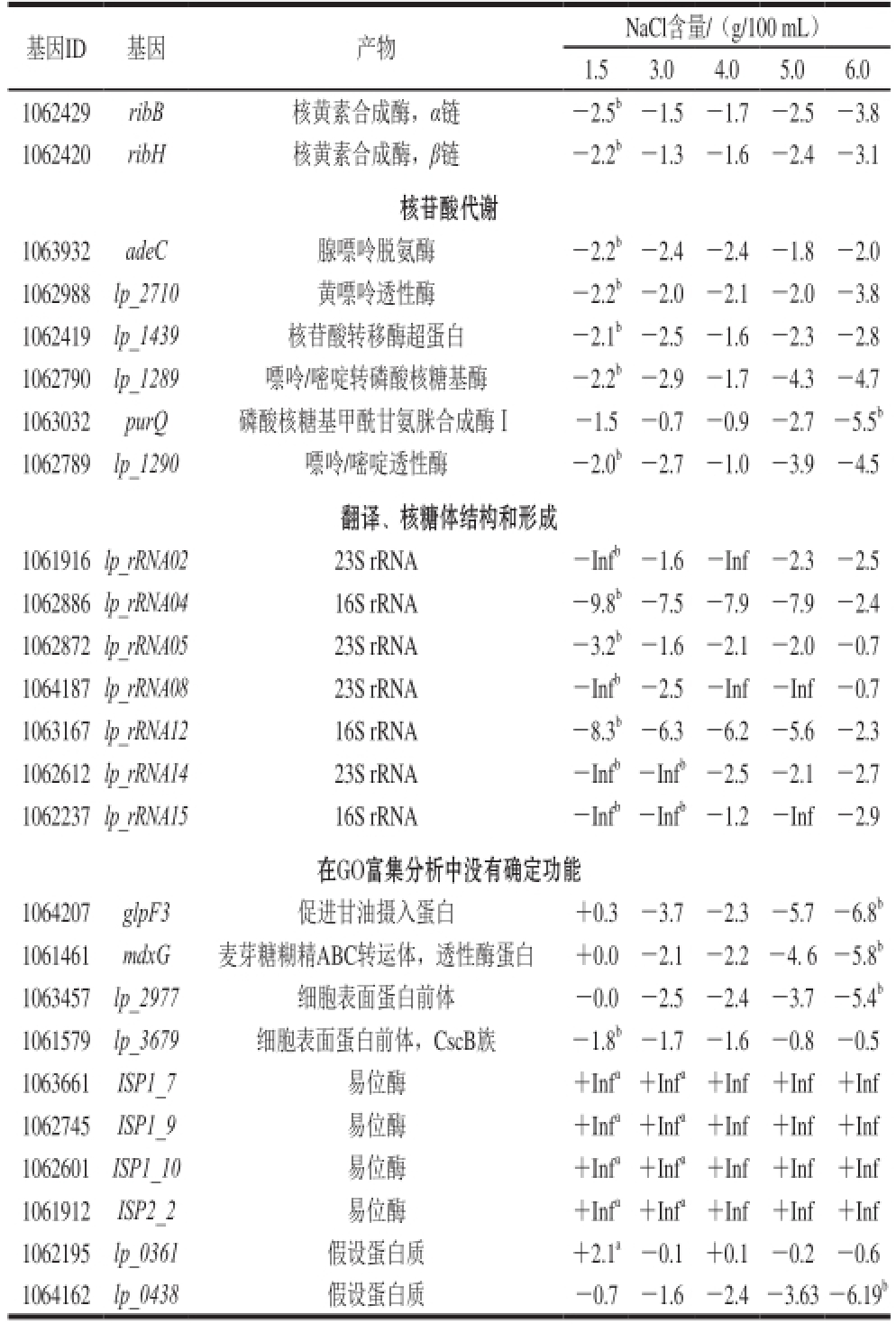
注:a.基因表达显著上调,表达上调倍数大于2 倍且FDR不大于0.05;+Inf.上调倍数远大于2;b.基因表达显著下调,表达下调倍数大于2 倍且FDR不大于0.05;-Inf.下调倍数远大于2。
基因ID基因产物NaCl含量/(g/100 mL)1.5 3.0 4.0 5.0 6.0 1062429 ribB核黄素合成酶,α链-2.5
b-1.5-1.7-2.5-3.8 1062420 ribH核黄素合成酶,β链-2.2
b-1.3-1.6-2.4-3.1核苷酸代谢1063932 adeC腺嘌呤脱氨酶-2.2
b-2.4-2.4-1.8-2.0 1062988 lp_2710黄嘌呤透性酶-2.2
b-2.0-2.1-2.0-3.8 1062419 lp_1439核苷酸转移酶超蛋白-2.1
b-2.5-1.6-2.3-2.8 1062790 lp_1289嘌呤/嘧啶转磷酸核糖基酶-2.2
b-2.9-1.7-4.3-4.7 1063032 purQ磷酸核糖基甲酰甘氨脒合成酶Ⅰ-1.5-0.7-0.9-2.7-5.5
b1062789 lp_1290嘌呤/嘧啶透性酶-2.0
b-2.7-1.0-3.9-4.5翻译、核糖体结构和形成1061916 lp_rRNA02 23S rRNA-Inf
b-1.6-Inf-2.3-2.5 1062886 lp_rRNA04 16S rRNA-9.8
b-7.5-7.9-7.9-2.4 1062872 lp_rRNA05 23S rRNA-3.2
b-1.6-2.1-2.0-0.7 1064187 lp_rRNA08 23S rRNA-Inf
b-2.5-Inf-Inf-0.7 1063167 lp_rRNA12 16S rRNA-8.3
b-6.3-6.2-5.6-2.3 1062612 lp_rRNA14 23S rRNA-Inf
b-Inf
b-2.5-2.1-2.7 1062237 lp_rRNA15 16S rRNA-Inf
b-Inf
b-1.2-Inf-2.9在GO富集分析中没有确定功能1064207 glpF3促进甘油摄入蛋白+0.3-3.7-2.3-5.7-6.8
b1061461 mdxG麦芽糖糊精ABC转运体,透性酶蛋白+0.0-2.1-2.2-4. 6-5.8
b1063457 lp_2977细胞表面蛋白前体-0.0-2.5-2.4-3.7-5.4
b1061579 lp_3679细胞表面蛋白前体,CscB族-1.8
b-1.7-1.6-0.8-0.5 1063661 ISP1_7易位酶+Inf
a+Inf
a+Inf+Inf+Inf 1062745 ISP1_9易位酶+Inf
a+Inf
a+Inf+Inf+Inf 1062601 ISP1_10易位酶+Inf
a+Inf
a+Inf+Inf+Inf 1061912 ISP2_2易位酶+Inf
a+Inf
a+Inf+Inf+Inf 1062195 lp_0361假设蛋白质+2.1
a-0.1+0.1-0.2-0.6 1064162 lp_0438假设蛋白质-0.7-1.6-2.4-3.63-6.19
b
以上变化说明碳水化合物代谢在L. p lantarum FS5-5的盐胁迫反应中的重要性。Belfiore等
[17]的研究表明,将10 g/100 mL NaCl和0.06 g/100 mL甘氨酸甜菜碱处理后的清酒乳杆菌CRL1756在含10 g/100 mL NaCl的培养基培养6 d后通过比较蛋白质组学进行分析,蛋白mleS的表达受到了诱导。Sheng Jiangyun等
[18]的研究表明,在苹果酸的发酵作用中,L-苹果酸分解为L-乳酸和二氧化碳并产生能量,能保护链球菌抵制酸胁迫、氧胁迫和饥饿胁迫。本实验中基因m leS的表达上调表明苹果乳酸酶mleS催化分解L-苹果酸也是L. p la ntarum FS5-5在盐胁迫下保护自己的一种方式。lp_2 845、lp_3 014和lp_0 304所编码的具有溶菌酶基序(lysin motif,LysM)肽聚糖结合位点的细胞外转糖苷酶是肽聚糖水解酶。LysM能帮助肽聚糖水解酶以非共价的方式结合到细胞壁肽聚糖上
[19]。Najjari等
[20]的研究表明,L. sake i肽聚糖水解酶的溶解活性在高盐浓度和酸性条件下依然很高。肽聚糖是细菌细胞壁的重要组成成分,决定着细胞形状并且能帮助细胞抵抗内部渗透压胁迫。本实验中具有肽聚糖LysM结合位点的细胞外转糖苷酶基因表达显著上调,说明细胞壁的溶解程度增加,L. pla ntaru m FS5-5通过改变细胞壁的特性来应对外界渗透压的变化。磷酸转移酶系统(phosphotransferase system,PTS)在糖磷酸化和转运过程中起重要作用
[21]。Zhao Shanshan等
[22]对L. plantarum ST-Ⅲ的研究表明,添加6.0 g/100 mL NaCl情况下,纤维二糖PTS、甘露糖PTS等均受到了抑制。本实验中的pts38BC、pts38C、srlM2和pts23B是PTS的关键酶
[23],在高盐含量下它们的表达显著下调,说明L. plan ta rum FS5-5糖的磷酸化和转运受到了抑制。这些结果表明在高渗条件下,L. plan tarum FS5-5的碳水化合物代谢是整体抑制的。
2.4.2 NaCl对参与氨基酸转运和代谢的基因表达的影响
由表3可知,基因asnA、a snS1、hisA、hisB、hisC、hisE、metE、lp_1374、lp_1 746在NaCl含量为1.5、3.0、4.0、5.0、6.0 g/100 mL时表达均下调,且在NaCl含量为1.5 g/100 mL时的表达显著下调。
asn A和asn S1是天冬氨酸代谢的关键酶,hisA、hisB、hisC和hisE是参与组氨酸代谢的关键酶,metE和lp_1374是参与半胱氨酸的关键酶,lp_1746是参与蛋氨酸代谢的关键酶。Pieterse
[2
4]通过转录组学对L. plantarum WCFS1的研究表明,当盐含量为4.7 g/100 mL时,除了调控丝氨酸代谢的基因,参与氨基酸合成的基因均表达下调,并且编码氨基酸转运蛋白的基因lp_3049的表达也显著下调。这与Hahne等
[2
5]对枯草杆菌(Bacillus sub tilis)的研究结果一致,他们将B. su b tilis培养在盐含量为6.0 g/100 mL的培养基中,分别于10、30、60、120 min进行转录组学分析,结果显示多个参与氨基酸合成的基因均表达下调。
2.4.3 NaCl对参与维生素代谢的基因表达的影响
由表3可以看出,基因ribA、ribB、rib H在NaCl含量为1.5、3.0、4.0、5.0、6.0 g/100 mL时表达均下调,且在NaCl含量为1.5 g/100 mL时的表达显著下调。
基因ribA编码的是核黄素(riboflavin,VB
2)生物合成途径中最初用到的两个酶。它们分别催化D-核酮糖-5-磷酸(D-ribulose-5-phosphate)转变为L-3,4-二羟基-2-丁酮-4-磷酸(L-3,4-dihydroxy-2-butanone-4-phosphate,DHBP)和甲酸盐,和催化三磷酸鸟苷(g u an o sin e triphosphate,GTP)转变为2,5-二氨基-6-核糖氨基-4(3氢)-嘧啶酮-5’-磷酸盐(2,5-diamino-6-ribosylamino-4(3 H)-pyrimidinone-5’-phosphate,DARP)
[26]。rib B和ribH分别在α链和β链促进核黄素的合成。核黄素是细胞生物化学过程的基本成分,是辅酶黄素单核苷酸(flavin mononucleotide,FMN)和黄素腺嘌呤二核苷酸(flavin adenine dinucleotide,FAD)合成的前体物质
[27]。基因rib A、rib B、rib H的表达受抑制,说明核黄素的合成减少。这可能是L. p lantaru m FS5-5为了存活和维持生长,减少了维生素的合成。
2.4.4 NaCl对参与核苷酸代谢的基因表达的影响
由表3可知,基因a d e C、lp_2 7 1 0、lp_1 4 3 9、lp_12 89、lp_1 29 0和pu rQ在Na Cl含量为1.5、3.0、4.0、5.0、6.0 g/100 mL时表达均下调,且a d eC、lp_2 7 1 0、lp_1 4 39、lp_128 9和lp_1290在盐含量为1.5 g/100 mL时的表达显著下调,purQ在盐含量为6.0 g/100 mL时的表达显著下调。
Ku ltz等
[28]的研究表明,高盐含量引起的高渗透压会导致DNA双键断裂概率的增加。Pieterse
[24]通过转录组学对L. p la n ta ru m WCFS1的研究表明,当NaCl含量为4.7 g/100 m L时,编码嘌呤合成的基因lp_2 7 1 9(p u rD)和lp_2 7 2 9(p u rE)、编码腺嘌呤脱氢酶的基因lp_3 3 3 4(a d eC)、编码腺嘌呤琥珀酸合酶的基因lp_3270(purA)、编码单磷酸鸟苷还原酶的基因lp_32 71(guaC)等的表达均显著下调,并且推测说核苷酸水平的降低可能使高渗透压对胞内含水量的降低有抵消作用。
2.4.5 NaCl对参与遗传信息翻译、核糖体结构和形成的基因表达的影响
由表3可知,基因lp_r R N A 0 2、lp_r R N A 0 4、lp_rR N A 0 5、lp_rR N A 0 8、lp_rR N A 1 2、lp_rR N A 1 4和lp_rRNA15在NaCl含量为1.5、3.0、4.0、5.0、6.0 g/100 mL时表达均下调,且在NaCl含量为1.5 g/100 mL时的表达显著下调。mRNA翻译的整体抑制是细胞适应环境压力的普遍方式。在压力条件下,菌体通过限制蛋白质合成的方式来保存能量和储备有限的资源,选择性翻译编码压力蛋白的mRNA
[29]。
3 结 论
碳水化合物转运和代谢基因(m le S、lp_2 8 4 5、lp_30 14和lp_03 04)受到NaCl胁迫时表达显著上调,碳水化合物转运和代谢基因(pts38 B C、p ts3 8C、srlM 2和p ts2 3 B)、氨基酸转运和代谢的基因(a sn A、a sn S1、hisA、h isB、hisC、h isE、metE、lp_1 374、lp_174 6)、维生素代谢基因(ribA、ribB、ribH)、核苷酸代谢基因(adeC、lp_2710、lp_1439、lp_1289、purQ和lp_1290)以及参与遗传信息翻译、核糖体结构和形成的基因(lp_rR N A0 2、lp_rR N A 0 4、lp_rR N A 05、lp_rRNA 0 8、lp_rR N A1 2、lp_rR N A1 4和lp_rRN A1 5)受到NaCl胁迫时表达显著下调,表明苹果乳酸酶mleS催化分解L-苹果酸、细胞壁特性的改变、糖磷酸化和转运过程的抑制、氨基酸转运和代谢的抑制、维生素合成的降低、核苷酸水平的降低及蛋白质合成的降低等都是L. p la n taru m FS5-5在NaCl胁迫下保护自己的方式。但上述基因表达显著上调或下调的程度与NaCl含量没有一定的规律。面对高含量NaCl带来的渗透压胁迫,基因结构本身或其表达的蛋白受到了破坏,不能全面发挥其作用;或者是它们的功能与L. plan ta rum FS5-5的耐盐性相关性不是很高,但具体机理还有待进一步的研究。
参考文献:
[1] WASSENAAR T M, KLEIN G. Safety aspects and implications of regulation of probiotic bacteria in food and food supplements[J]. Journal of Food Protection, 2008, 71(8): 1734-1741.
[2] 靳志强, 王延样. 植物乳杆菌在人体肠道的益生特性[J]. 中国乳品工业, 2007, 35(9): 30-34. DOI:10.3969/j.issn.1001-2230.2007.09.008.
[3] 乌日娜, 宋雪飞, 刘倩颖, 等. 植物乳杆菌分子伴侣蛋白基因在盐胁迫下的表达分析[J]. 食品科学, 2015, 36(11): 95-99. DOI:10.7506/ spkx1002-6630-201511019.
[4] TANAKA Y, WATANABE J, MOGI Y. Monitoring of the microbial communities involved in the soy sauce manufacturing process by PCR-denaturing gradient gel electrophoresis[J]. Food Microbiology, 2012, 31(1): 100-106. DOI:10.1016/j.fm.2012.02.005.
[5] TRAN K T M, MAY B K, SMOOKER P M, et al. Distribution and genetic diversity of lactic acid bacteria from traditional fermented sausage[J]. Food Research International, 2011, 44(1): 338-344. DOI:10.1016/j.foodres.2010.10.010.
[6] NOONPAKDEE W, JUMRIANGRIT P, WITTAYAKOM, et al. Two-peptide bacteriocin from Lactobacillus plantarum PMU 33 strain isolated from som-fak, a Thai low salt fermented fish product[J]. Asia Pacific Journal of Molecular Biology and Biotechnology, 2009, 17(1): 19-25.
[7] PÉREZDÍAZ I M, MCFEETERS R F. Preparation of a Lactobacillus p an ta ru m starter culture for cucumber fermentations that can meet kosher guidelines[J]. Journal of Food Science, 2011, 76(2): M120-M123. DOI:10.1111/j.1750-3841.2010.01981.x.
[8] ANGELIS M D, GOBBETTI M. Environmental stress responses in L actob acillus: a review[J]. Proteomics, 2004, 4(1): 106-122. DOI:10.1002/pmic.200300497.
[9] KIM W S, PERL L, PARK J H, et al. Assessment of stress response of the probiotic Lactobacillus acidophilus[J]. Current Microbiology, 2001, 43(5): 346-350. DOI:10.1007/s002840010314.
[10] GOUESBET G, JAN G, BOYAVAL P. Lactobacillus delbrueckii ssp. bulgaricus thermotolerance[J]. Dairy Science and Technology, 2001, 81(1/2): 301-309. DOI:10.1051/lait:2001133.
[11] DESMOND C, STANTON C, FITZGERALD G F, et al. Environmental adaptation of pro b iotic lactobacilli towards improvement of performance during spray drying[J]. International Dairy Journal, 2001, 11(10): 801-808. DOI:10.1016/S0958-6946(01)00121-2.
[12] WANG Z, GERSTEIN M, SNYDER M. RNA-Seq: a revolutionary tool for transcriptomics[J]. Nature Reviews Genetics, 2009, 10(1): 57-63. DOI:10.1038/nrg2484.
[13] LINDBERG J, LUNDEBERG J. The plasticity of the mammalian transcriptome[J]. Genomics, 2 010, 95(1): 1-6. DOI:10.1016/ j.ygeno.2009.08.010.
[14] 徐鑫, 王茜茜, 王晓蕊, 等. 传统农家大酱中耐盐性乳酸菌的分离与鉴定[J]. 食品与发酵工业, 2014, 40(11): 33-40. DOI:10.13995/ j.cnki.11-1802/ts.201411006.
[15] TRAPNELL C, ROBERTS A, GOFF L, et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks[J]. Nature Protocols, 2012, 7(3): 562-578. DOI:10.1038/ nprot.2012.016.
[16] FLEGEL C, MANTENIOTIS S, OSTHOLD S, et al. Expression p rofile o f ec to p ic o lfac to ry rec eptors determin ed by dee p sequencing[J]. PLoS ONE, 2013, 8(2): e55368. DOI:10.1371/journal. pone.0055368.
[17] BELFIORE C, FADDA S, RAYA R, et al. Molecular basis of the adaption of the anchovy isolate Lacto bacillus sa kei CRL1756 to salted environments through a proteomic approach[J]. Food Research International, 2013, 54(1): 1334-1341. DOI:10.1016/ j.foodres.2012.09.009.
[18] SHENG J Y, BALDECK J D, NGUYEN P T M, et al. Alkali production associated with malolactic fermentation by oral streptococci and protection against acid, oxidative, or starvation damage[J]. Canadian Journal of Microbiology, 2010, 56(7): 539-547. DOI:10.1139/w10-039.
[19] BUIST G, STEEN A, KOK J, et al. LysM, a widely distributed protein motif for binding to (peptido) glycans[J]. Molecular Microbiology, 2008, 68(4): 838-847. DOI:10.1111/j.1365-2958.2008.06211.x.
[20] NAJJARI A, AMAIRI H, CHAILLOU S, et al. Phenotypic and genotypic characterization of peptidoglycan hydrolases of Lac to b acillus sa kei[J]. Journal of Advanced Research, 2016, 7(1): 155-163. DOI:10.1016/j.jare.2015.04.004.
[21] JECKELMANN J M, HARDER D, MARI S A, et al. Structure and function of the glucose PTS transporter from E sc herichia coli[J]. Journal of Structural Biology, 2011, 176(3): 395-403. DOI:10.1016/ j.jsb.2011.09.012.
[22] ZHAO S S, ZHANG Q X, HAO G F, et al. The protective role of glycine betaine in L acto bacillus plan ta ru m ST-Ⅲ against salt stress[J]. Food Control, 2014, 44(1): 208-213. DOI:10.1016/ j.foodcont.2014.04.002.
[23] BOYD D A, THEVENOT T, GUMBMANN M, et al. Identification of the operon for the sorbitol (glucitol) phosphoenolpyruvate: sugar phosphotransferase system in Strep toco ccus mutans[J]. Infection and Immunity, 2000, 68(2): 925-930. DOI:10.1128/IAI.68.2.925-930.2000.
[24] PIETERSE B. Transcriptome analysis of the lactic acid and NaCl stress response of L a c to b a cillu s p la n ta r u m[D]. Wageningen: Wageningen University, 2006: 113-136.
[25] HAHNE H, MADER U, OTTO A, et al. A co mpreh ensiv e proteomics and transcriptomics analysis of B acillus su btilis salt stress adaptation[J]. Journal of Bacteriology, 2010, 192(3): 870-882. DOI:10.1128/JB.01106-09.
[26] SINGH M, KUMAR P, KARTHIKEYAN S. Structural basis for pH dependent monomer-dimer transition of 3,4-dihydroxy 2-butanone-4-phosphate synthase domain from M ycobacterium tuberculosis[J]. Journal of Structural Biology, 2011, 174(2): 374-384. DOI:10.1016/ j.jsb.2011.01.013.
[27] GRILL S, YAMAGUCHI H, WAGNER H, et al. Identification and characterization of two S trep tomy ce s d av awe nsis riboflavin biosynthesis gene clusters[J]. Archives of Microbiology, 2007, 188(4): 377-387. DOI:10.1007/s00203-007-0258-1.
[28] KULTZ D, CHAKRAVARTY D. Hyperosmolality in the form of elevated NaCl but not urea causes DNA damage in murine kidney cells[J]. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(4): 1999-2004. DOI:10.1073/ pnas.98.4.1999.
[29] MAJUMDER M, MITCHELL D, MERKULOV S, et al. Residues required for phosphorylation of translation initiation factor eIF2α under diverse stress conditions are divergent between yeast and human[J]. The International Journal of Biochemistry and Cell Biology, 2015, 59(1): 135-141. DOI:10.1016/j.biocel.2014.12.004.
Transcriptomic Analyses of Lactobacillus plantarum FS5-5 against Salt Stress
SONG Xuefei
1, GUO Jingjing
1, JIANG Jing
1, TANG Xiaoyang
1, ZHANG Ying
1, WU Rina
1,2,*
(1. College of Food Science, Shenyang Agricultural University, Shenyang 110866, China; 2. State Key Laboratory of Food Science and Technology, College of Food Science, Jiangnan University, Wuxi 214122, China)
Abstract:This investigation studied the expression of salt stress-related genes in L acto b acillus pla n taru m FS5-5, a salt-tolerance strain isolated from naturally fermented miso in northeastern China, at the level of transcription. Results showed that in the logarithmic growth phase, 29 genes were significantly down-regulated, including four ones involved in carbohydrate transport and metabolism, nine ones involved in amino acids transport and metabolism, three ones involved in vitamin metabolism, six ones involved in nucleotide metabolism and seven ones involved in genetic information translation, ribosomal structure and biogenesis. Four genes involved in carbohydrate transport and metabolism were signifi cantly upregulated, which might be closely associated with salt stress resistance of L. plantarum FS5-5. Three genes involved in vitamin metabolism and one gene involved in nucleotide metabolism were selected to be analyzed by real-time fluorescent quantitative polymerase chain reaction and the results showed that the expression of these genes exhibited the same trend in these two methods. The comprehensive analysis of the mechanism of action of L. plantarum FS5-5 to resist salt stress presented in this study which would provide a theoretical basis to improve the tolerance of strains in industrial production.
Key words:L a cto b a cillu s p la n ta ru m; salt stress; real-time fluorescent quantitative polymerase chain reaction; transcriptomics
DOI:10.7506/spkx1002-6630-201706004
中图分类号:Q939.9
文献标志码:A
文章编号:1002-6630(2017)06-0020-07
引文格式:
宋雪飞, 郭晶晶, 姜静, 等. 植物乳杆菌FS5-5在盐胁迫下的转录组学分析[J]. 食品科学, 2017, 38(6): 20-26. DOI:10.7506/ spkx1002-6630-201706004. http://www.spkx.net.cn
SONG Xuefei, GUO Jingjing, JIANG Jing, et al. Transcriptomic analyses of Lactob acillus plantarum FS5-5 against salt stress[J]. Food Science, 2017, 38(6): 20-26. (in Chinese with English abstract)
DOI:10.7506/spkx1002-6630-201706004. http://www.spkx.net.cn
收稿日期:2016-05-14
基金项目:国家自然科学基金青年科学基金项目(31000805);国家自然科学基金面上项目(31471713);
中国博士后科学基金项目(2014M560395);辽宁省农业领域青年科技创新人才培养计划项目(2014048);
辽宁省高等学校优秀人才支持计划项目(LR2015059);江苏省博士后科研资助计划项目(1402071C)
作者简介:宋雪飞(1988—),女,硕士研究生,研究方向为食品生物技术。E-mail:sxf5896@163.com
*通信作者:乌日娜(1979—),女,副教授,博士,研究方向为食品生物技术。E-mail:wrn6956@163.com







