多重PCR法检测鲜切哈密瓜中3 种食源性致病菌
冯 可
1,2,胡文忠
2,*,姜爱丽
2,萨仁高娃
1,2,徐永平
1,杨 柳
2,王 馨
2
(1.大连理工大学生命科学与技术学院,辽宁 大连 116024;2.大连民族大学生命科学学院,辽宁 大连 116600)
摘 要:建立可同时检测鲜切哈密瓜中的单增李斯特菌、鼠伤寒沙门氏菌和大肠杆菌O157:H7的多重聚合酶链式反应(polymerase chain reaction,PCR)检测方法。根据单增李斯特菌in lA基因、鼠伤寒沙门氏菌invA基因、大肠杆菌O157:H7的w zy基因设计3 对特异性引物。对多重PCR反应体系中的引物浓度、退火温度、Mg
2+浓度、dNTP浓度进行了优化,并确定适宜的多重PCR反应体系及反应条件。结果表明:25 μL反应体系,10×PCR buffer为2.5 μL,Mg Cl
2(25 mmol/L)为3.5 μL,d NTPs(2.5 mmol/L)为2 μL,inlA基因上下游引物(5 μmol/L)为1 μL,invA基因上下游引物(5 μmol/L)为1 μL,wzy基因上下游引物(5 μmol/L)为2 μL,单增李斯特菌DNA模板为1 μL,鼠伤寒沙门氏菌DNA模板为1 μL,大肠杆菌O157:H7 DNA模板为1 μL,ex Ta q DNA聚合酶(5 U/L)为0.3 μL,加ddH
2O补足25 μL。反应条件为95 ℃预变性3 min;94 ℃变性30 s,53.9 ℃退火30 s,72 ℃延伸30 s,32 个循环;72 ℃延伸10 min。鲜切哈密瓜中人工接种的目标菌的灵敏度为单增李斯特菌2.7×10
4CFU/g,大肠杆菌O157:H7 3.3×10
4CFU/g以及鼠伤寒沙门氏菌3.8×10
4CFU/g。该技术可为快速检测鲜切哈密瓜中病原菌污染度及其控制提供参考依据。
关键词:多重PCR;快速检测;食源性致病菌;鲜切哈密瓜
食源性疾病报告的漏报率在发达国家为90%,而在发展中国家约为95%~99%,全球每年发生40~60亿 例食源性腹泻病例,发展中国家每年有180万 人口死于食源性腹泻
[1]。2014年,全国共发生了食物中毒类公共卫生事件报告160 起,中毒5 657 人,死亡110 人。由微生物引起的食物中毒人数最多,分别占食物中毒事件总起数和中毒总人数的42.5%和67.7%,且均为由沙门氏菌、副溶血性弧菌、金黄色葡萄球菌及其肠毒素、蜡样芽胞杆菌、大肠埃希氏菌、肉毒毒素、椰毒假单胞菌、志贺氏菌、变形杆菌、弗氏柠檬酸杆菌等引起的细菌性食物中毒事件
[2]。2011年9月在美国爆发了大规模的甜瓜受单增李斯特菌污染的食物中毒事件,即刻引起专家学者的注意,该事件导致全美24 个州出现单增李斯特菌感染病例,30 人确认死亡
[3]。自1982年美国首次报告了由大肠杆菌O157:H7引起的出血性肠炎,随后在全球范围内出现了多起大肠杆菌O157:H7食物中毒事件,仅在日本,曾发生过9 000多位儿童集体感染的事件,1996—2004年间,每年用于大肠杆菌O157:H7感染的费用为4.05亿 美元
[4-5]。2011—2013年,甘肃省共报告食源性病原菌爆发时间约为81 起,发病1 089 起,死亡17 例,病死率为1.56%。因此,致病菌的检测在食品安全检测方面尤为重要
[6]。随着食品工业技术的发展,建立一种快速、准确、灵敏度高、特异性强的方法来检测食品中的致病菌是当今社会最需要的检测技术
[7]。
1 材料与方法
1.1 材料与试剂
鲜切哈密瓜 大连市购。
单核细胞增多性李斯特菌(ATCC 19115)、伊氏李斯特氏菌(ATCC 19119)、威氏李斯特氏菌(ATCC 35 897) 中国工业微生物菌种保藏管理中心;大肠埃希氏菌O1 5 7:H7(NCTC 12 9 00)、大肠埃希氏菌(ATCC 8739)、鼠伤寒沙门氏菌(ATCC 14028)、普通变形杆菌(CMCC 49027) 中国微生物菌种网。
胰酪胨大豆酵母浸膏琼脂培养基、胰酪胨大豆酵母浸膏肉汤、营养肉汤培养基 青岛海博生物技术有限公司;细菌基因组DNA提取试剂盒、Taq DNA聚合酶、10×PCR缓冲液、dNTPs、DNA Makrer、引物 大连宝生物有限公司;琼脂糖 西班牙Biowest公司;GelRed染料 上海拜力生物科技有限公司。
1.2 仪器与设备
Th ermo Ark tic重聚合酶链式反应(p o ly merase ch ain rea ctio n,PCR)仪、Mu ltisk an GO核酸测定仪 美国Thermo Fisher Scientific公司;DYY-11型电泳仪 北京市六一仪器厂;BioSpectrum 310凝胶成像系统 美国UVP公司;全自动高压灭菌锅 日本Sanyo公司。
1.3 方法
1.3.1 细菌培养与基因组DNA提取
将实验室保存的单增李斯特菌、鼠伤寒沙门氏菌和大肠杆菌O157:H7分别接种于LB固体培养基中,36 ℃培养24 h,分别挑取单菌落接种于5 mL营养肉汤培养基中,36 ℃静止培养18~24 h。取1 mL菌悬液(约为10
8CFU/mL)进行DNA提取。按照细菌DNA提取试剂盒的操作步骤分别对3 种致病菌的DNA进行提取。用核酸测定仪对DNA浓度及纯度进行测定。
1.3.2 引物设计
选取单增李斯特菌的in lA基因、沙门氏菌的in vA基因、大肠杆菌O157:H7的w zy基因为目的基因
[8-10]。利用Premier 5.0和Oligo 6.0软件对目的基因进行引物设计以及评价,选取出3 对不同片段大小及相近退火温度的特异性引物,经过BLAST分别将引物与目的基因同源性进行比对,确定引物序列见表1。
表1 多重PCR引物序列
Table 1 Primer pairs designed for the multiplex PCR

细菌引物序列(5’→3’)产物/bp单增李斯inlA-F:GAGCTAACCAAATAAGTAACA 285特菌inlA-R:AGGTCGCTAATTTGGTT A鼠伤寒invA-F:CTTTATGATGCATTCTACCAACGACTG 121沙门氏菌invA-R:CCGAATCACCACTGTTAGGA大肠杆菌wzy-F:GTTCCATATGTTGTTTCTGA 193 O157:H7wzy-R:CTGCTCCATACGTAGTAA
1.3.3 单重PCR反应体系的建立
分别以单增李斯特菌、鼠伤寒沙门氏菌和大肠杆菌O157:H7的DNA为模板进行PCR反应,其总体积为25 μL,其中10×PCR buffer 2.5 μL,MgCl
2(25 mmol/L)3 μL,dNTPs(2.5 mmol/L)2 μL,上下游引物(5 μmol/L)各1 μL,DNA模板 1 μL,Ta q酶(5U/L)0.3 μL,加dd H
2O 补足25 μL。单一PCR反应程序为:95 ℃预变性3 min;94 ℃变性30 s,退火温度分别设定为49、50.1、51.7、53.3、56.1、58.1、59.6、61 ℃ 8 个梯度,退火30 s,72 ℃延伸30 s,32 个循环;72 ℃延伸10 min。扩增产物检测:经质量浓度为3 g/100 mL的琼脂糖凝胶电泳后,采用凝胶成像分析系统观察结果。将扩增产物送到宝生物工程有限公司进行测序。
1.3.4 多重PCR反应条件建立及优化
[11-12]
1.3.4.1 引物终浓度的优化
将单增李斯特菌、鼠伤寒沙门氏菌和大肠杆菌O157:H7 3 种目标菌的DNA等比例混合后作为模板,其起始引物浓度均为5 μmol/L。将扩增体系中目标菌对应的3 种引物进行按照3 个梯度进行正交试验设计L
9(3
3),如表1所示,确定最佳引物浓度。
表2 三因素三水平L
9(3
3)正交试验设计
Table 2 Three levels of three factors used in orthogonal array design
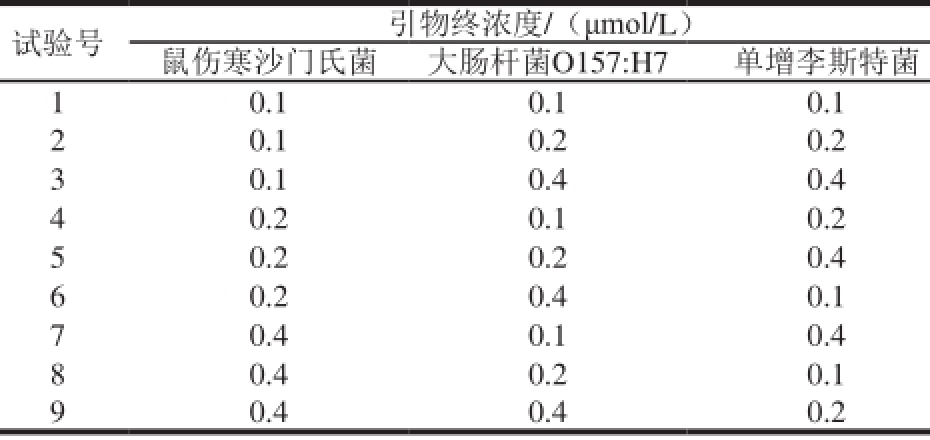
试验号引物终浓度/(μmol/L)鼠伤寒沙门氏菌大肠杆菌O157:H7单增李斯特菌1 0.1 0.1 0.1 2 0.1 0.2 0.2 3 0.1 0.4 0.4 4 0.2 0.1 0.2 5 0.2 0.2 0.4 6 0.2 0.4 0.1 7 0.4 0.1 0.4 8 0.4 0.2 0.1 9 0.4 0.4 0.2
1.3.4.2 退火温度优化
在引物浓度确定的情况下,对多重PCR反应中退火温度进行优化,依次为49、50.1、5 1.7、53.3、56.1、58.1、59.6、61、62.3、64 ℃。Mg
2+浓度为3.0 mmol/L,dNTPs浓度为0.20 mmol/L,经过多重PCR扩增,然后进行电泳分析以确定最适合的退火温度。
1.3.4.3 Mg
2+浓度的优化
在1.3.4.2节条件确定的情况下,对多重PCR反应条件中的Mg
2+浓度进行优化,调整加入的Mg
2+浓度梯度变化为1.0、1.5、2.0、2.5、3.0、3.5、4.0 mmol/L,dNTPs浓度为0.20 mmol/L,经过多重PCR扩增,然后进行电泳分析以确定最适合的Mg
2+浓度。
1.3.4.4 dNTPs浓度优化
在1.3.4.3节条件(退火温度、Mg
2+浓度)确定的情况下,对多重PCR反应条件中的d NTPs浓度进行优化,调整加入的d NTPs浓度梯度变化为0.05、0.10、0.15、0.20、0.25、0.30、0.35、0.40 mmol/L,经过多重PCR扩增,然后进行电泳分析以确定最适合的dNTPs浓度。
1.3.5 多重PCR反应特异性检测
1.3.5.1 引物间特异性验证
利用优化好的反应体系,将3 对引物同时对单一目标菌、随机两两组合的目标菌进行扩增。
1.3.5.2 菌间特异性
[13]
为验证多重PCR反应体系的特异性,将3 对引物分别对奇异变形杆菌、大肠埃希氏菌、威氏李斯特氏菌、普通变形杆菌、伊氏李斯特氏等非目的菌进行PCR扩增。采用3%的琼脂糖凝胶对扩增产物进行电泳,利用凝胶成像系统观察电泳结果,对多重PCR反应体系的特异性进行验证。
1.3.6 多重PCR灵敏度进行验证
通过平板计数法对经过培养后的菌液进行菌落计数,分别对已制备的3 种致病菌的菌液进行10倍稀释后进行DNA提取。按已优化的多重PCR反应体系及条件对其分别进行PCR扩增,以确定最低检出限
[14-15]。
1.3.7 人工接种鲜切哈密瓜中多重PCR灵敏度的验证
制备3 种致病菌菌悬液,哈密瓜清洗后切块呈0.5 c m×0.5 c m×0.5 c m,将1 m L菌悬液(约10
8CFU/mL)侵染至10 g鲜切哈密瓜表面,在安全柜中吹干1 h,将其分别浸泡于90 mL营养肉汤培养基中培养24 h、37 ℃。均质后取1 mL菌液进行10 倍梯度稀释并进行菌落计数。对其稀释管分别进行DNA提取。并采用多重PCR技术对其进行验证,以确定该技术在应用于鲜切哈密瓜时的最低检出限。
2 结果与分析
2.1 单重PCR扩增
利用单重PCR反应体系分别对单增李斯特菌、鼠伤寒沙门氏菌和大肠杆菌O157:H7的靶基因进行扩增,结果如图1~3所示。在不同退火温度的条件下,可分别扩增得到3 个与预计大小相符的目标条带:28 5、121 b p和193 bp处均有条带,且条带清晰、亮度适中。测序结果与Gen Bank的BLAST比对程序进行比对,序列相似性(98%),说明扩增的目的基因,引物特异性强。
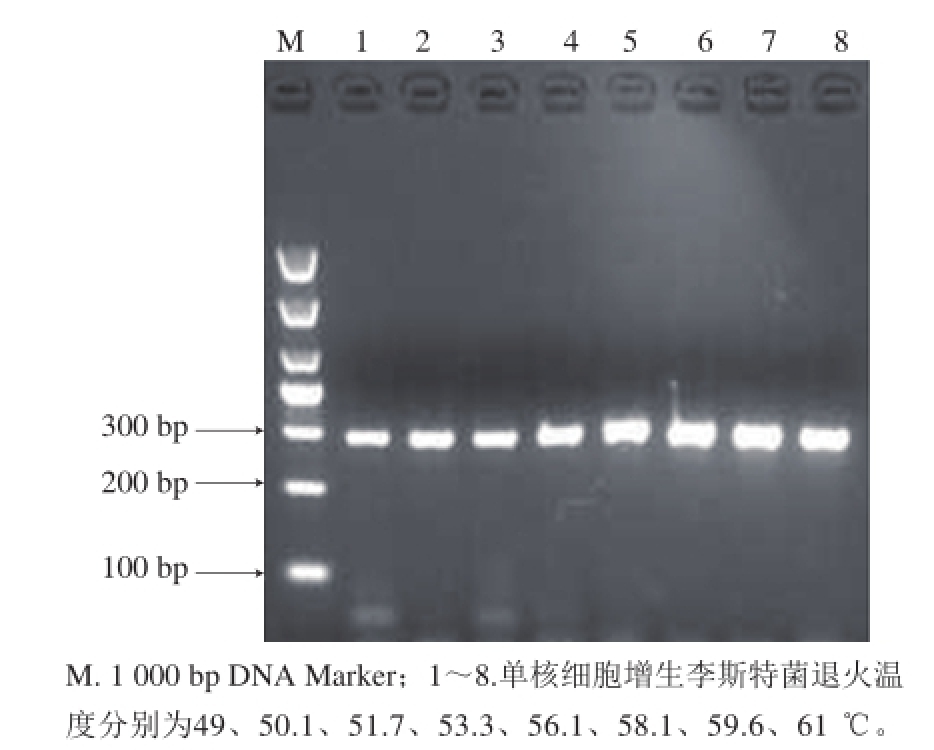
图1 单增李斯特菌PCR扩增退火温度优化
Fig. 1 Optimization of PCR annealing temperature for L. monocytogenes
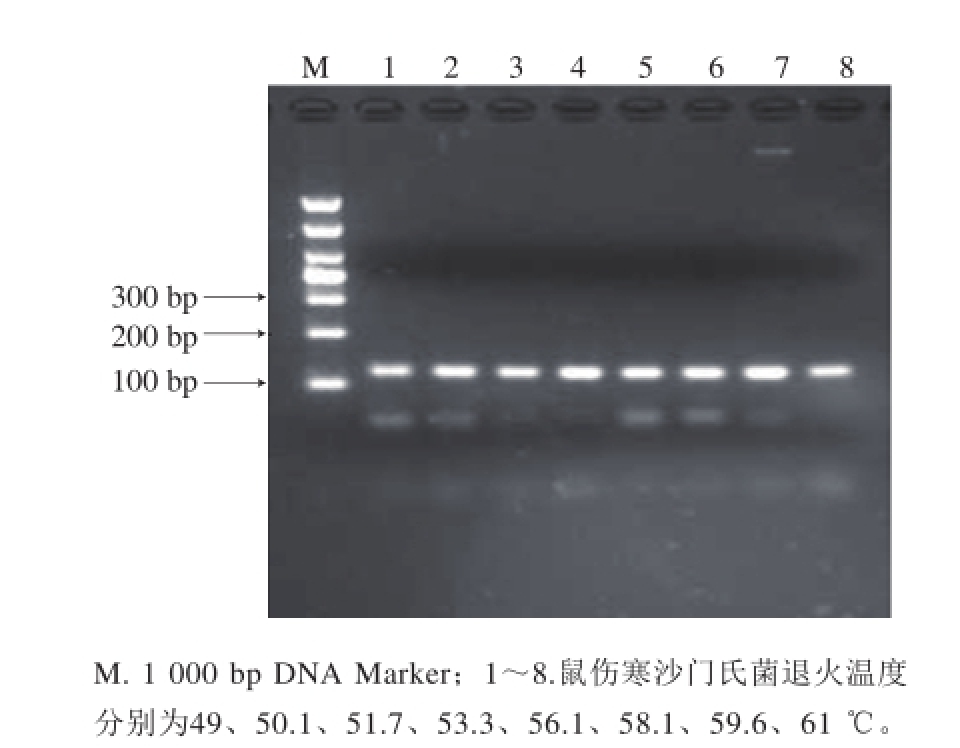
图2 鼠伤寒沙门氏菌PCR扩增退火温度优化
Fig. 2 Optimization of PCR annealing temperature for Salmonella typhimurium
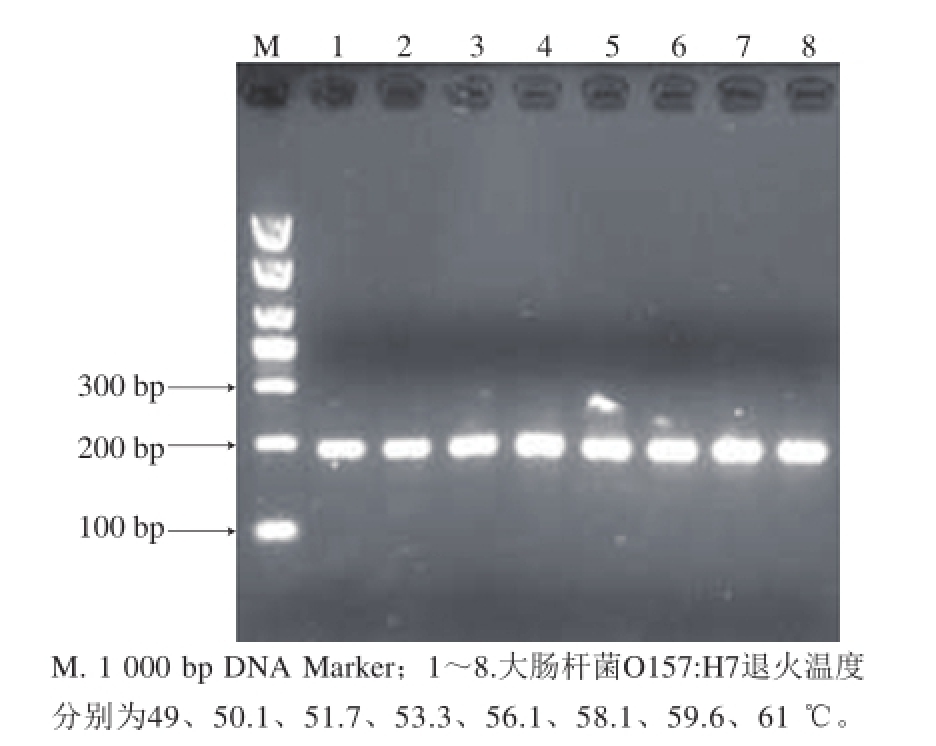
图3 大肠杆菌O157:H7 PCR扩增退火温度优化
Fig. 3 Optimization of PCR annealing temperature for E. coli O157:H7
2.2 多重PCR反应引物浓度优化

图4 多重PCR引物浓度优化结果
Fig. 4 Optimization of primer concentrations for multiplex PCR
如图4所示,不同比例的引物浓度扩增的结果有所不同,引物浓度用量较低时,会出现漏检目标条带的现象。而当引物用量过高时,会导致单一目标条带过于明亮且弥散。在泳道5中,当鼠伤寒沙门氏菌、大肠杆菌O157:H7、单核细胞增生李斯特菌引物终浓度分别为0.2、0.2、0.4 μmol/L时,能够扩增出3 条清晰、明亮且特异性良好的条带。故选择该组合为最终引物浓度配比。
2.3 多重PCR退火温度优化
在鼠伤寒沙门氏菌、大肠杆菌O157:H7、单核细胞增生李斯特菌引物终浓度分别为0.2、0.2、0.4 μmol/L时,对多重PCR退火温度进行优化,结果如图5所示,在49~61 ℃范围内扩增的目标条带明显不同,当退火温度较低时如泳道1、2,鼠伤寒沙门氏菌目标条带的亮度较暗,随着退火温度升高,如泳道6~10,扩增条带呈现弥散及隐约可见的现象,这是由于退火温度过高导致扩增效率显著降低的结果
[16]。而泳道3~5退火温度为51.7、53.3、56.1 ℃,则呈现出清晰、明亮的扩增条带。取均值后确定退火温度为53.9 ℃。
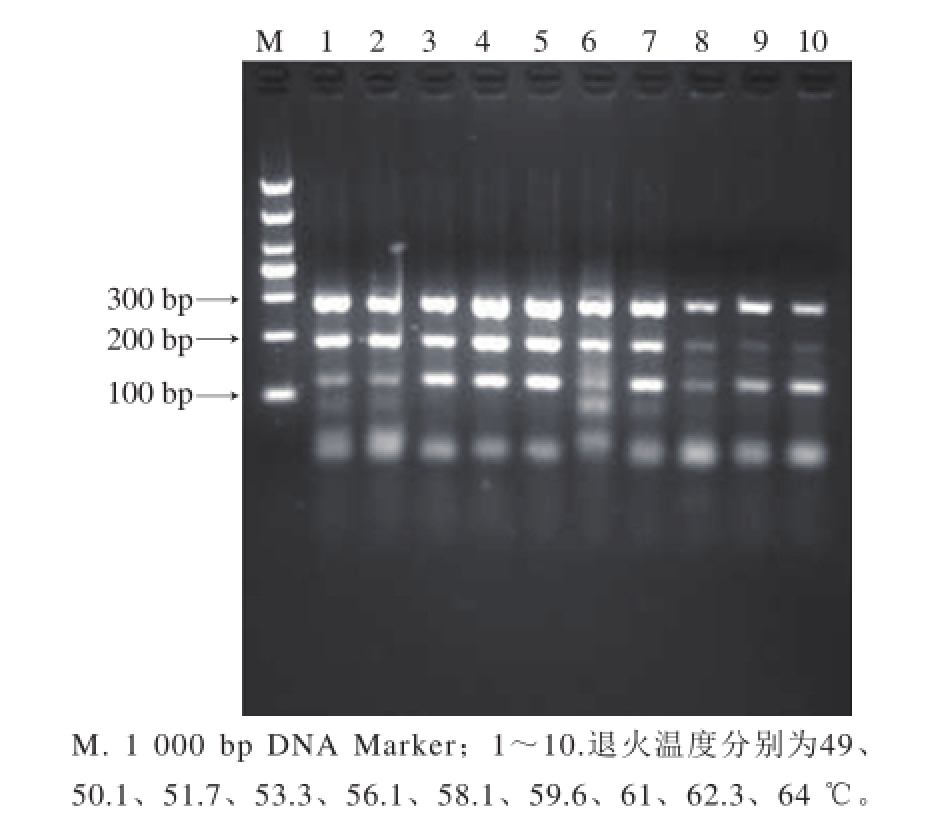
图5 退火温度优化结果
Fig. 5 Optimization of annealing temperature for multiplex PCR
2.4 多重PCR反应体系中Mg
2+浓度优化
在鼠伤寒沙门氏菌、大肠杆菌O157:H7、单核细胞增生李斯特菌引物终浓度分别为0.2、0.2、0.4 μmol/L时,退火温度为53.9 ℃条件下,对Mg
2+浓度进行优化,结果如图6所示,可见在泳道6中,当Mg
2+的浓度为3.5 mmol/L时,条带明亮、清晰。当Mg
2+浓度较低时,条带不整齐且会出现无扩增条带的现象。
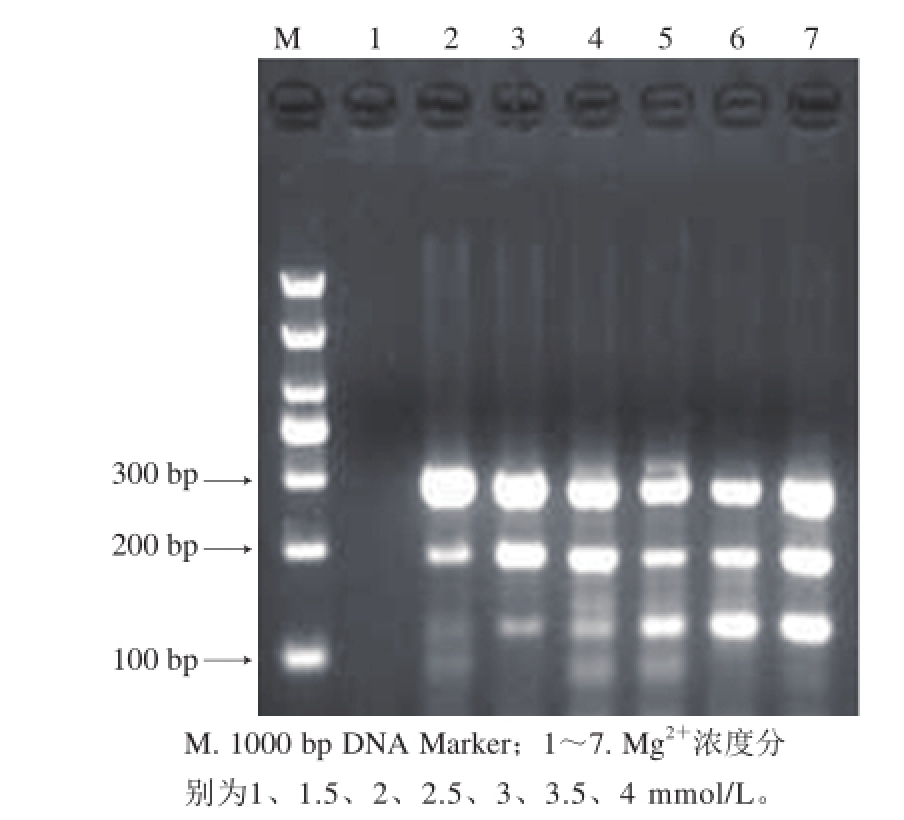
图6 Mg
2+浓度优化结果
Fig. 6 Optimization of Mg
2+concentration for multiplex PCR
2.5 多重PCR反应体系中dNTPs浓度优化
在鼠伤寒沙门氏菌、大肠杆菌O157:H7、单核细胞增生李斯特菌引物终浓度分别为0.2、0.2、0.4 μmol/L时,退火温度为53.9 ℃条件下,Mg
2+浓度为3.5 mmol/L时,对反应体系中d NTPs浓度进行优化,如图7所示,当dNTPs浓度为01~0.3 mmol/L时,能有效扩增出3 条特异性条带,且无拖带现象,当d NTPs浓度升高后,则出目标条带无法扩增的现象。因此择优选择0.2 mmol/L为dNTPs的确定终浓度。
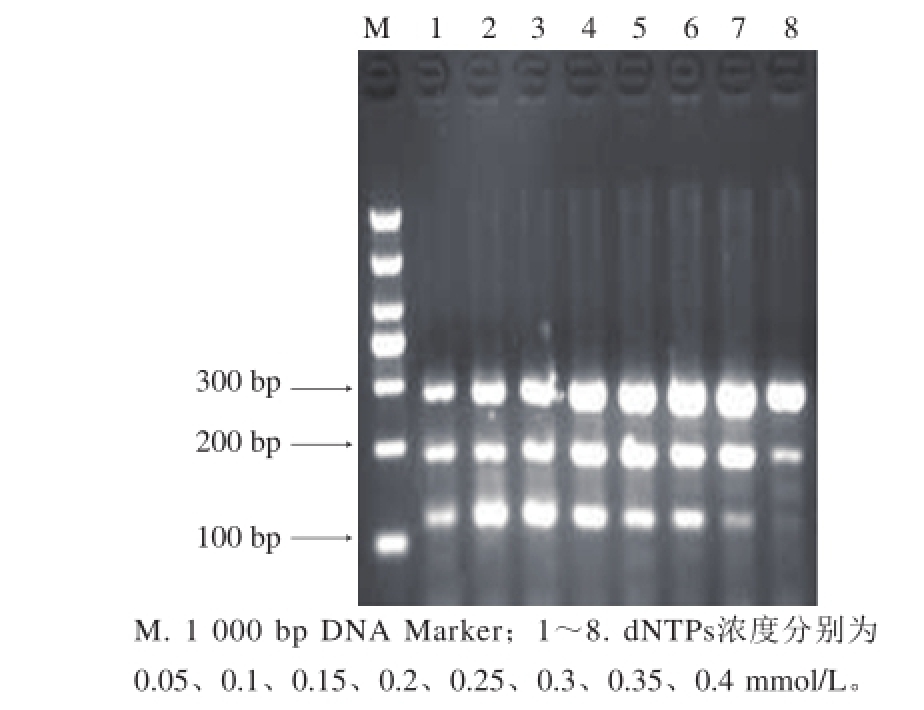
图7 dNTPs浓度优化结果
Fig. 7 Optimization of dNTPs concentration for multiplex PCR
2.6 多重PCR反应体系中引物间特异性检测

图8 引物间特异性检测结果
Fig. 8 Detection of primer specificity
利用已建立的多重PCR反应体系对目标菌进行引物间特异性的检测,结果如图8所示。泳道1中含有3 种目标菌,泳道2~4则可分别有效扩增出鼠伤寒沙门氏菌、大肠杆菌O157:H7和单增李斯特菌,泳道5~7则可效扩增出两两随机组合的目标病原菌。无非特异性扩增条带出现
[17]。
2.7 多重PCR反应体系中菌间特异性检测
按建立的PCR反应体系对靶基因进行扩增,检测结果见图9,在同一试管中可同时扩增得3 种目的基因片段,3%的琼脂糖凝胶电泳显示在285、193 bp和121 bp处均有条带出现,且没有引物二聚体形成,没有非特异性条带出现。泳道2~6为非目标菌扩增结果,与对照组一致无条带扩增,表明其特异性良好。
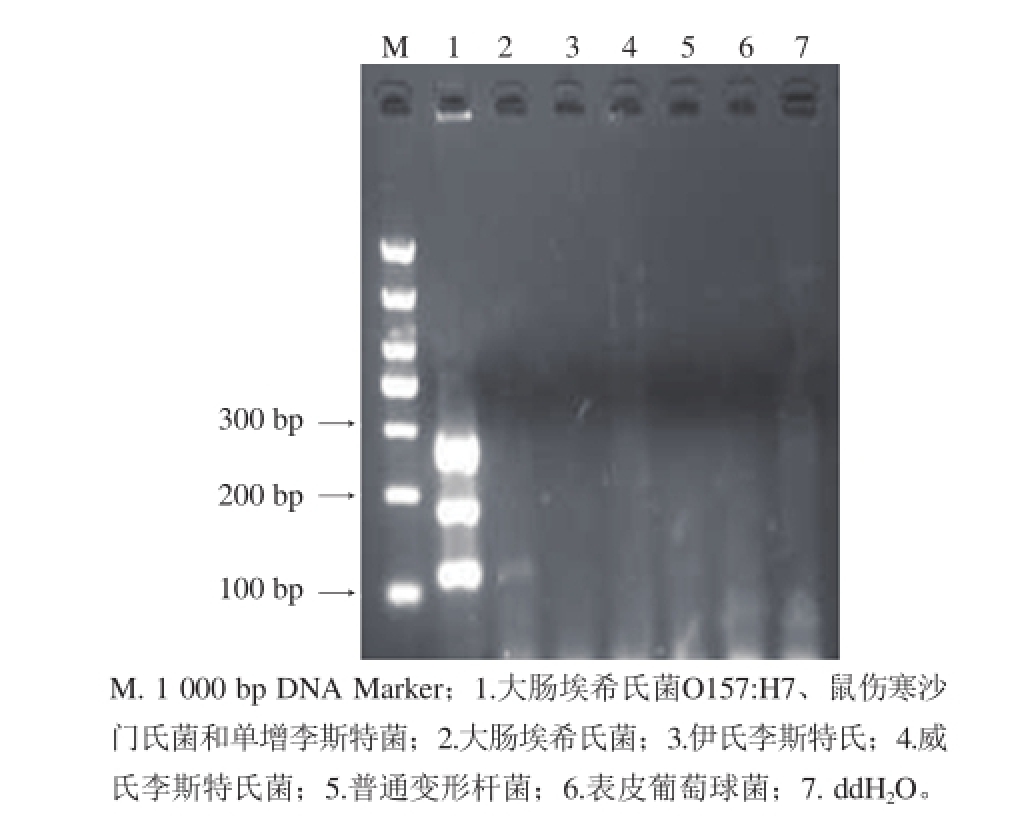
图9 多重PCR特异性的检测结果
Fig. 9 Specificity of multiplex PCR
2.8 多重PCR反应体系中灵敏度验证
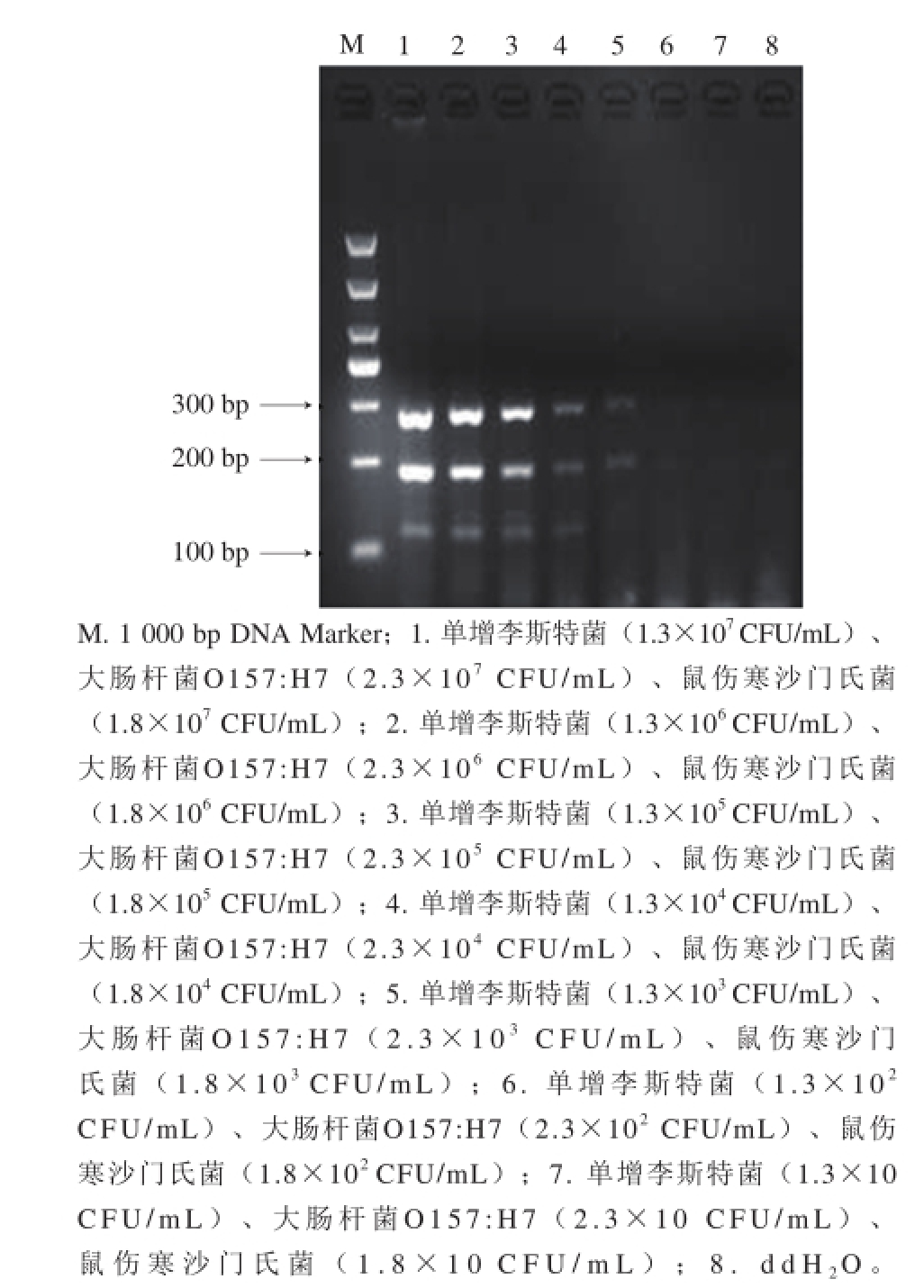
图10 多重PCR灵敏度验证
Fig. 10 Sensitivity of multiple PCR
将3 种致病菌的菌悬液进行梯度稀释后,进行菌落数确定,单增李斯特菌的菌落数为1.3×10
7CFU/mL,鼠伤寒沙门氏菌的菌落数为1.8×10
7CFU/mL,大肠杆菌O157:H7的菌落数为2.3×10
7CFU/mL。分别将稀释后提取的DNA等比例进行混合,用优化好的多重PCR反应体系进行检测验证,结果如图10所示,3 种目标菌的检测灵敏度分别为单增李斯特菌为1.3×10
3CFU/mL,大肠杆菌O157:H7为2.3×10
3CFU/mL以及鼠伤寒沙门氏菌为1.8×10
4CFU/mL。
2.9 人工污染鲜切哈密瓜中多重PCR灵敏度验证

图11 鲜切哈密瓜中多重PCR灵敏度
Fig. 11 Sensitivity of multiple PCR for fresh-cut cantaloupe
将3 种目标菌接种于鲜切哈密瓜后进行多重PCR灵敏度验证,结果如图1 1所示,在鲜切哈密瓜中的检测灵敏度分别为单增李斯特菌为2.7×10
4CFU/g,大肠杆菌O157:H7为3.3×10
4CFU/g以及鼠伤寒沙门氏菌为3.8×10
4CFU/g。灵敏度低于在纯菌中的检测结果,可能由于PCR反应受到了哈密瓜汁液的影响,降低了扩增效率。扩增的目标条带未发生交叉影响,从而证明了已建立的多重PCR检测体系较为稳定
[18-20]。
3 讨 论
传统的PCR检测手段无法同时检测食品样品中携带的多种病原菌。多重PCR作为一种可同时检测多种病原菌的快速且可靠的手段,已在疾病预防控制中心及第三方检测平台得到广泛应用
[21]。本研究利用多重PCR检测技术成功建立了可同时检测单增李斯特菌、鼠伤寒沙门氏菌和大肠杆菌O157:H7的方法,且适用于鲜切哈密瓜受这3 种目标菌污染后同时检测。
在多重PCR检测技术中,靶基因的选择直接决定着检测方法的准确性和可靠性。单增李斯特菌的内化素岛上含有一个多基因家族(in lA、in lB、in lC、in lC 2 D、inlD、inlE、in lF、in lG和inlH),其中InlA和InlB分别与钙黏蛋白和补体分子CIq受体结合,从而介导单增李斯特菌进入上皮细胞、肝细胞、成纤维细胞等
[22-24]。Liu Dongyou等
[25]利用多重PCR技术以单增李斯特菌中的inlA、in lC、inlJ基因为目的基因进行扩增均可得到相应的目标条带。鼠伤寒沙门氏菌携带的具有较强特异性的靶基因分别为pefA、sefA和invA
[26]。据报道,有2 000多种沙门氏菌血清型的菌株带有侵袭蛋白基因in vA,也有研究表明该基因序列为属间特有
[27]。大肠杆菌O157:H7的O抗原抗体由wzy基因编码,李睿等
[28]进一步证实了wzy基因作为靶基因可以特异性的区分出O157菌株与非O157菌株。wzy基因可以作为O157的特异性标志基因,检测食品中的大肠杆菌O157。因此,该技术选择单增李斯特菌的inlA基因、沙门氏菌的invA基因及大肠杆菌O157:H7的wzy基因作为检测的靶基因。
影响多重PCR有效扩增的因素较多,引物浓度配比、Mg
2+浓度、d NTPs浓度及其比例等因素的控制不当,都将导致产生非特异性产物甚至扩增失败。对多重PCR反应条件的优化有利于得到更明确、清晰且均匀的目标条带。研究表明,经优化后得到的单增李斯特菌和鼠伤寒沙门氏菌的引物浓度均为0.2 μmol/L,大肠杆菌O157:H7的引物浓度为0.4 μmol/L。钱志伟等
[29]利用多重PCR技术特异性扩增金黄色葡萄球菌的nuc基因、单增李斯特菌的prs基因、沙门氏菌的in vA基因,经优化后,其在检测水平为3.8、5.1、7.6 pg/μL,该技术可用于牛奶中这3 种菌的检测。Yang Youjun等
[30]建立了基于免疫磁珠分离技术基础上的多重PCR技术,其对人工污染鼠伤寒沙门氏菌、大肠杆菌O157:H7及单增李斯特菌于莴苣、西红柿和牛肉的灵敏度分别为5.1×10
3、7.5×10
3CFU/g和8.4×10
3CFU/g,可见,经免疫磁珠富集后,检出限稍有下降,而缺点在于检测成本高昂,对人工操作水平要求较高,无法实现规模化推广。而本研究建立的多重PCR快速检测技术,其对鲜切哈密瓜中单增李斯特菌、大肠杆菌O157:H7及鼠伤寒沙门氏菌检测灵敏度分别为2.7×10
4、3.3×10
4CFU/g和3.8×10
4CFU/g。与相关报道的检测结果相似,且操作简便,易实施平民化推广。
4 结 论
针对单增李斯特菌inlA基因、鼠伤寒沙门氏菌invA基因、大肠杆菌O157:H7 w zy基因设计及筛选出3 对引物,引物特异性强,互不干扰。
建立了一种能够同时检测单增李斯特氏菌、大肠杆菌O157:H7和鼠伤寒沙门氏菌3 种致病菌的多重PCR检测方法。其反应体系为:25 μL反应体系:10×PCR buffer为2.5 μL,Mg Cl
2(2 5 mmo l/L)为3.5 μL,d NTPs(2.5 mmol/L)为2 μL,in lA上下游引物(5 μmol/L)为1 μL,in vA上下游引物(5 μmol/L)为1 μL,w zy上下游引物(5 μmol/L)为2 μL,单增李斯特菌DNA模板为1 μL,鼠伤寒沙门氏菌DNA模板为1 μL,大肠杆菌O157:H7 DNA模板为1 μL,ex Taq DNA 聚合酶(5 U/L)为0.3 μL,加dd H
2O补足25 μL。多重PCR反应程序为:95 ℃预变性3 min;94 ℃变性30 s,53.9 ℃退火30 s,72 ℃延伸30 s,32 个循环;72 ℃延伸10 min。
多重PCR对单增李斯特菌,大肠杆菌O157:H7及鼠伤寒沙门氏菌的检测灵敏度分别为1.3×10
3、2.3×10
3CFU/mL和1.8×10
4CFU/mL。而对鲜切哈密瓜中人工接种的目标菌的灵敏度分别为单增李斯特菌2.7×1 0
4CFU/g,大肠杆菌O157:H7 3.3×10
4CFU/g以及鼠伤寒沙门氏菌3.8×10
4CFU/g。
本研究建立的检测鲜切哈密瓜中易受侵染的3 种病原菌的多重PCR检测方法具有很高的特异性和灵敏度,该方法的建立相较于传统的培养检测方法可节约大量的劳力、试剂、时间等,对于企业或分析检验中心大批量样品的监测具有指导意义。
参考文献:
[1] 侯君, 陈玉凤, 栾明春, 等. 2009—2012年大连市食品中食源性致病菌污染状况调查[J]. 医学动物防制, 2015(1): 7-9. DOI:10.7629/ yxdwfz201501003.
[2] QIAN Y, ZHAI L G, BIE X M, et al. Survey of five food-borne pathogens in commercial cold food dishes and their detection by multiplex PCR[J]. Food Control, 2016, 59: 862-869. DOI:10.1016/ j.foodcont.2015.06.027.
[3] 关棣锴, 胡文忠, 姜爱丽, 等. 鲜活农产品中单核细胞增多性李斯特菌快速检测技术的研究进展[J]. 食品工业科技, 2013, 34(11): 361-368. DOI:10.13386/j.issn1002-0306.2013.11.062.
[4] FRENZEN P D, DRAKE A, ANGULO F J, et al. Economic cost of illness due to Escherichia coli O157 infections in the United States[J]. Journal of Food Protection, 2005, 68(12): 2623-2630.
[5] 张雪寒, 张碧成, 栾晓婷, 等. 遗传标志性基因Z0372 PCR检测肠出血大肠杆菌O157:H7[J]. 西南农业学报, 2015(1): 409-413. DOI:10.16213/j.cnki.scjas.2015.01.076.
[6] 崔燕, 梁效成. 2011─2013年甘肃省食源性疾病暴发事件分析[J].疾病预防控制通报, 20 1 5(1): 7 6-78. DOI:1 0.1 32 15/j.cn ki. jbyfkztb.1408015.
[7] 栗建永, 赵琢, 贾晓川, 等. 食源性致病菌检测分析技术的研究进展[J]. 食品研究与开发, 2013, 34(18): 110-115. DOI:10.3969/ j.issn.1005-6521.2013.18.028.
[8] ENRICA O, GIULIA A, GIORGIO B, et al. A new platform for realtime PCR detection of Salmonella spp., Listeria monocytogenes and Escherichia coli O157 in milk[J]. Food Microbiology, 2009, 26(6): 615-622. DOI:10.1016/j.fm.2009.04.008.
[9] HIROHITO O, OSAMU T, TAKUYA H, et al. Multiplex PCR and multiplex RT-PCR for inclusive detection of major swine DNA and RNA viruses in pigs with multiple infections[J]. Journal of Virological Methods, 2009, 106(1/2): 210-214. DOI:10.1016/ j.jviromet.2009.05.010.
[10] ANDREA G, ANNALISA M, PAOLA C, et al. Simultaneous detection of Escherichia coli O157:H7, Salmonella spp., and Listeria monocytogenes by multiplex PCR[J]. Food Control, 2009, 20(8): 733-738. DOI:10.1016/j.talanta.2005.09.036.
[11] HU L, LIN X, NIE F, et al. Simultan eous typing of seven porcine pathogens by multiplex PCR with a GeXP analyser[J]. Journal of Virological Methods, 2016, 232: 21-28. DOI:10.1016/ j.jviromet.2015.12.004.
[12] 蔡军, 李慧, 欧静堃, 等. 3 种食源性致病菌多重PCR检测体系的建立[J]. 食品科技, 2015, 40(3): 324-329. DOI:10.13684/j.cnki. spkj.2015.03.076.
[13] ZHANG Z H, XIAO L L, LOU Y, et al. Development of a multiplex re al-time PCR method for simultane ous detection of Vib rio p ara h emo lyticu s, L isteria m o n ocytog enes and S alm on ella spp. in raw shrimp[J]. Food Control, 2015, 51: 31-36. DOI:10.1016/ j.foodcont.2014.11.007.
[14] GUAN Z P, JIANG Y, GAO F, et al. Rapid and simultaneous analysis of five foodborne pathogenic bacteria using multiplex PCR[J]. European Food Research and Technology, 2013, 237: 627-637. DOI 10.1007/s00217-013-2039-1.
[15] ALVES J, HIROOKA E Y, DE OLIVEIRA T. Development of a multiplex real-time PCR assay with an internal amplifi cation control for the detection of Ca m py lo ba cte r spp. and S almo n e lla spp. in chicken meat[J]. LWT-Food Science and Technology, 2016, 72: 175-181. DOI:10.1128/AEM.00460-07.
[16] YANG I, KIM Y, BYUN J, et al. Use of multiplex polymerase chain reactions to indicate the accuracy of the annealing temperature of thermal cycling[J]. Analytical Biochemistry, 2005, 338(2): 192-200. DOI:10.1016/j.ab.2004.09.035.
[17] XU Y, SUN L, WANG Y, et al. Simultaneous detection of Vib rio cho lerae, Vibrio algino lyticus, Vib rio parahaemolyticus and Vibrio v u lnific us in seafood using dual priming oligonucleotide (DPO) system-based multiplex PCR assay[J]. Food Control, 2017, 71: 64-70. DOI:10.1016/j.foodcont.2016.06.024.
[18] PUI C F, WONG W C, CHAI L C, et al. Simultaneous detection of Salmonella spp., Salmonella Typhi and Salmonella typhimurium in sliced fruits using multiplex PCR[J]. Food Control, 2011, 22(2): 337-342. DOI:10.1016/j.foodcont.2010.05.021.
[19] ELIZAQUÍVEL P, AZNAR R, A multiplex RTi-PCR reaction for simultaneous detection of Esch erichia co li O157:H7, Salm on ella spp. and S ta ph ylococcus a ureu s on fresh, minimally processed vegetables[J]. Fo od Micro biology, 20 08, 2 5(5): 70 5-713. DOI:10.1016/j.fm.2008.03.002.
[20] ROY A, FAYAD A, BARTHE G, et al. A multiplex polymerase chain reaction method for reliable, sensitive and simultaneous detection of multiple viruses in citrus trees[J]. Journal of Virological Methods, 2005, 129(1): 47-55. DOI:10.1016/j.jviromet.2005.05.008.
[21] CUI Z Q, OJAGHIAN M R, TAO Z Y, et al. Multiplex PCR assay for simultaneous detection of six major bacterial pathogens of rice[J]. Journal of Applied Microbiology, 2016, 120: 1357-1367. Doi:10.1111/jam.13094.
[22] DUSSURGET O, PIZARRO C J, COSSART P. Molecu lar determinants of L isteria m o no cyto g enes virulence[J]. Annual Review of Microbiology, 2004, 58: 587-610. DOI:10.1146/annurev. micro.57.030502.090934.
[23] GLASER P, FRANGEUL L, BUCHRIESER C, et al. Comparative genomics of L iste ria species[J]. Science, 2001, 294: 849-852. DOI:10.1126/science.1063447.
[24] 冯 可, 胡文忠, 姜爱丽, 等. 单核细胞增生性李斯特菌分子生物学检测技术的研究进展[J]. 食品工业科技, 2014, 35(4): 392-396. DOI:10.13386/j.issn1002-0306.2014.04.044.
[25] LIU D Y, MARK L L, FRANK W A. A multiplex PCR for speciesand virulence-specific determination of Listeria mo nocytogenes[J]. Journa l of Microbiological Methods, 2007, 71(2): 133-140. DOI:10.1016/j.mimet.2007.08.007.
[26] 杨柳, 胡文忠, 姜爱丽, 等. 分子生物学方法检测沙门氏菌的研究进展[J]. 食品工业科技, 2016, 37(9): 372-379. DOI:10.13386/ j.issn1002-0306.2016.09.065.
[27] D’SOUZA D H, CRITZER F J, GOLDEN D A. Real-time reversetranscriptase polymerase chain reaction for the rapid detection of Salmonella using invA primers[J]. Foodborne Pathogens and Disease, 2009, 6(9): 1097-1106. DOI:10.1089/fpd.2009.0322.
[28] 李睿, 张忠美, 戴诗皎, 等. 食品中大肠杆菌O157的PCR检测与wzy基因的测序分析[J]. 食品科学, 2010, 31(12): 193-196.
[29] 钱志伟, 孙新城. 食品中3 种致病菌多重PCR检测体系的建立及初步应用[J]. 食品科学, 2011, 32(16): 236-239.
[30] YANG Y J, XU F, XU H Y, et al. Magnetic nano-beads based separation combined with propidium monoazide treatment and multiplex PCR assay for simultaneous detection of viable Salmonella typhimurium, Escherichia coli O157:H7 and Listeria mo nocytogenes in food products[J]. Food Microbiology, 2013, 34(2): 418-424. DOI:10.1016/j.fm.2013.01.004.
Establishment of Multiplex PCR Detection Method for Three Foodborne Pathogens on Fresh-Cut Cantaloupe
FENG Ke
1,2, HU Wenzhong
2,*, JIANG Aili
2, Sarengaowa
1,2, XU Yongping
1, YANG Liu
2, WANG Xin
2
(1. School of Life Science and Biotechnology, Dalian University of Technology, Dalian 116024, China; 2. College of Life Science, Dalian Minzu University, Dalian 116600, China)
Abstract:The objective of this study was to establish a multiplex polymerase chain reaction (PCR) detection method for Listeria m ono cyto genes, S almon ella typhim urium and E. co li O157:H7 on fresh-cut cantaloupe. Three pairs of specific primers were designed according to the inlA gene of L. monocytogenes, the invA gene of Salmonella typhimurium, and the wzy gene of E. coli O157:H7. The primer concentration, annealing temperature, Mg
2+concentration and dNTP concentration were optimized. The results showed that the PCR reactions were performed in a total volume of 25 μL consisting of 2.5 μL of 10 × PCR buffer, 3.5 μL of 25 mmol/L MgCl
2, 2 μL of 2.5 mmol/L dNTPs, 1 μL of DNA template, 0.3 μL of ex Taq DNA polymerase, 1 μL of 5 μmol/L upstream and downstream primers for inlA and invA and 2 μL of 5 μmol/L upstream and downstream primers for wzy made up to 25 μL with ddH
2O. The reaction conditions were as follows: pre-degeneration at 95 ℃ for 3 min followed by 32 cycles of degeneration at 94 ℃ for 30 s, annealing at 53.9 ℃ for 30 s and extension at 72 ℃ for 30 s and a final extension step at 72 ℃ for 10 min. The sensitively of the multiplex PCR was 2.7 × 10
4CFU/g for L. monocytogenes, 3.3 × 10
4CFU/g (E. coli O157:H7) and 3.8 × 10
4CFU/g Salmonella typhimurium inoculated onto freshcut cantaloupe. This method can lay the basis to detect and control these pathogenic microorganisms on fresh-cut cantaloupe.
Key words:multiplex PCR; rapid detection; foodborne pathogen; fresh-cut cantaloupe
DOI:10.7506/spkx1002-6630-201706046
中图分类号:TS207.4
文献标志码:A
文章编号:1002-6630(2017)06-0295-08
引文格式:
冯可, 胡文忠, 姜爱丽, 等. 多重PCR法检测鲜切哈密瓜中3 种食源性致病菌[J]. 食品科学, 2017, 38(6): 295-302.
DOI:10.7506/spkx1002-6630-201706046. http://www.spkx.net.cn
FENG Ke, HU Wenzhong, JIANG Aili, et al. Establishment of multiplex PCR detection method for three foodborne pathogens on fresh-cut cantaloupe[J]. Food Science, 2017, 38(6): 295-302. (in Chinese with English abstract) DOI:10.7506/ spkx1002-6630-201706046. http://www.spkx.net.cn
收稿日期:2016-07-01
基金项目:“十三五”国家重点研发计划重点专项(2016YFD0400903);国家自然科学基金面上项目(31471923)
作者简介:冯可(1985—),男,博士研究生,研究方向为食品安全检测。E-mail:fengkessky@sina.com
*通信作者:胡文忠(1959—),男,教授,博士,研究方向为食品科学。E-mail:wenzhongh@sina.com












