乳杆菌噬菌体基因组学研究进展
刘 颖,习 羽,范梦茹,汪政煜,吴文茹,张和平,陈 霞*
(内蒙古农业大学 乳品生物技术与工程教育部重点实验室,内蒙古 呼和浩特 010018)
摘 要:随着乳杆菌在乳品工业中的广泛应用,噬菌体污染对终产品造成的严重威胁日益增加。目前,噬菌体相关研究已从最初的形态学进入到了分子水平。许多噬菌体的全基因组测序工作已经完成。基因组研究有助于揭示噬菌体侵染宿主的机制,能挖掘其关键功能基因。本文基于乳杆菌噬菌体的基因组信息对主要乳杆菌噬菌体基因组的特征、功能基因组及比较基因组进行了分析和概述,为进一步研究噬菌体提供理论依据。
关键词:乳杆菌;噬菌体;基因组
噬菌体是细菌病毒,由于细菌分布广泛,其噬菌体在自然界中也是一种普遍存在的生物实体。乳杆菌广泛分布于自然界中,按照伯杰氏细菌学手册中的生化分类法,乳杆菌的生理生化特性为:革兰氏染色阳性、无芽孢,能分解糖产生乳酸,作为益生菌的主要成员。常见的乳杆菌包括德氏乳杆菌、植物乳杆菌、干酪乳杆菌、鼠李糖乳杆菌、发酵乳杆菌和瑞士乳杆菌等
[1]。
众所周知,发酵乳制品的生产和感官特性依赖于发酵剂菌株的稳定性。发酵剂菌株一旦受到噬菌体侵染,则可能推迟发酵过程,改变产品质量,甚至导致发酵失败。因此,噬菌体侵染成为了乳品发酵业不可避免的问题。此外,随着益生菌市场份额的日益增加,噬菌体侵染益生菌也成为益生菌生产中重点关注的问题。另外,在乳酸菌的许多种属中,溶源现象普遍存在。目前,已有研究证明从溶源菌株中释放的噬菌体可能是乳品发酵中烈性噬菌体的主要来源之一
[2]。因此,了解相关噬菌体生态学、系统发育学和基因组学知识,有利于分析噬菌体与宿主的相互作用,选育抗性菌株,提高发酵稳定性。
1 乳杆菌噬菌体基因组研究概况
由于噬菌体基因组较小、易测序,噬菌体基因组序列信息近10 年呈大幅增长趋势。在乳酸菌噬菌体全基因组序列中乳球菌序列占绝大多数,而乳杆菌噬菌体全基因组序列信息较为有限。乳杆菌作为乳酸菌1 种,其噬菌体基因组均为双链线性DNA分子。目前,已完成全基因组测序的乳杆菌噬菌体增至30 个左右,其GC含量变化范围从37%(植物乳杆菌噬菌体LP65)至48%(植物乳杆菌噬菌体LL-H),接近寄主菌的GC含量(32.9%~49.7%)
[3],由此可以看出噬菌体与寄主间的关系密切。乳杆菌噬菌体基因组一般为30~50 kb,但个别较大。由表1可知,噬菌体Ld1基因组为74.81 kb;植物乳杆菌噬菌体LP65基因组为131.521 kb;副干酪乳杆菌噬菌体Lb338-1基因组为142.11 kb。
表1 乳杆菌噬菌体全基因组序列
Table1 Complete genomic sequences of Lactobaciilllluuss phaaggeess
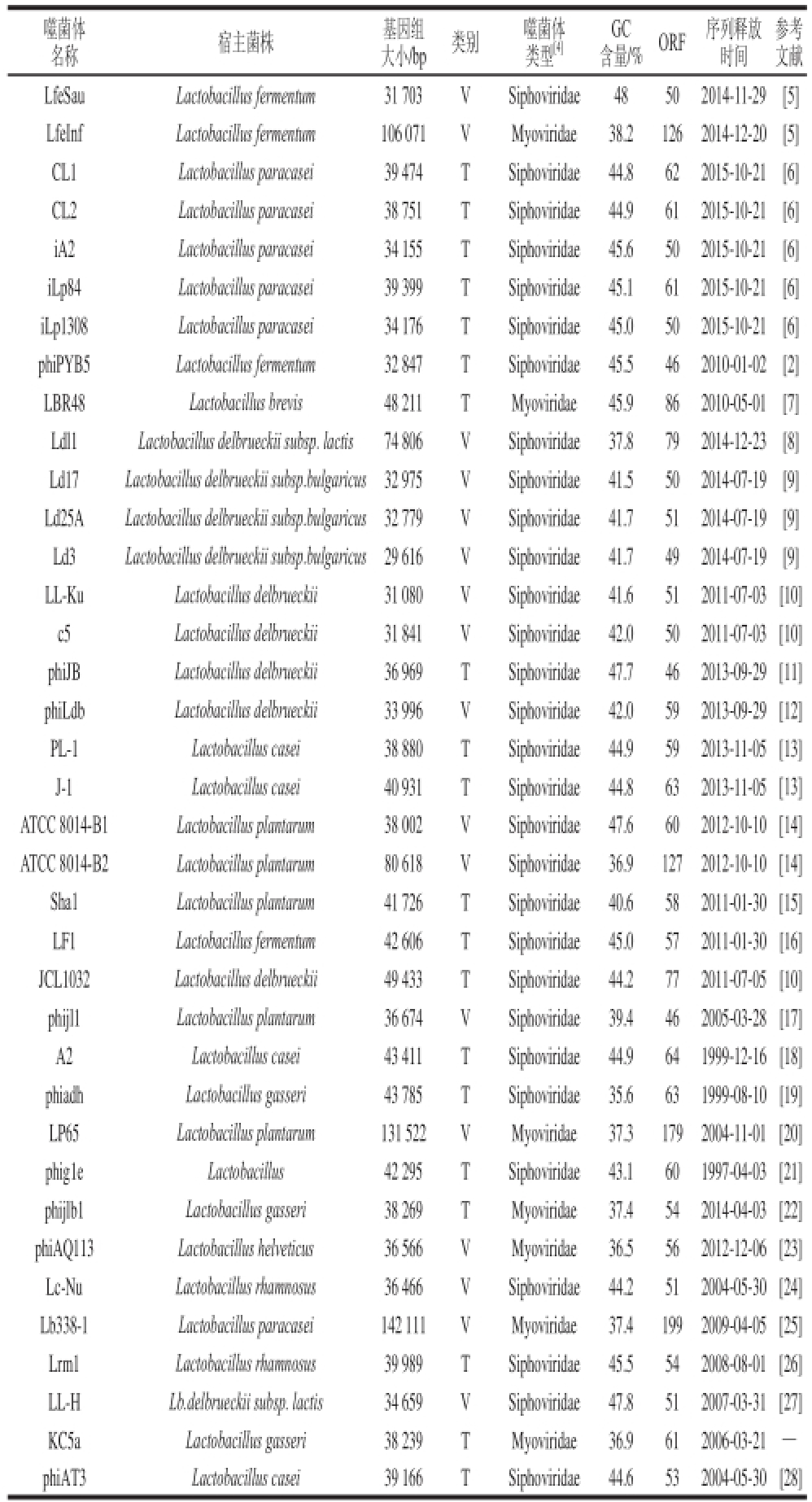
注:T.温和;V.烈性;GC含量为鸟嘌呤和胞嘧啶所占的比率;ORF.开放阅读框(open reading frame);—.出自GenBank数据库。
参考文献LfeSauLactobacillus fermentum31 703VSiphoviridae48502014-11-29 [5] LfeInfLactobacillus fermentum106 071VMyoviridae38.21262014-12-20 [5] CL1Lactobacillus paracasei39 474TSiphoviridae44.8622015-10-21 [6] CL2Lactobacillus paracasei38 751TSiphoviridae44.9612015-10-21 [6] iA2Lactobacillus paracasei34 155TSiphoviridae45.6502015-10-21 [6] iLp84Lactobacillus paracasei39 399TSiphoviridae45.1612015-10-21 [6] iLp1308Lactobacillus paracasei34 176TSiphoviridae45.0502015-10-21 [6] phiPYB5Lactobacillus fermentum32 847TSiphoviridae45.5462010-01-02 [2] LBR48Lactobacillus brevis48 211TMyoviridae45.9862010-05-01 [7] Ldl1Lactobacillus delbrueckii subsp. lactis74 806VSiphoviridae37.8792014-12-23 [8] Ld17Lactobacillus delbrueckii subsp.bulgaricus32 975VSiphoviridae41.5502014-07-19 [9] Ld25ALactobacillus delbrueckii subsp.bulgaricus32 779VSiphoviridae41.7512014-07-19 [9] Ld3Lactobacillus delbrueckii subsp.bulgaricus29 616VSiphoviridae41.7492014-07-19 [9] LL-KuLactobacillus delbrueckii31 080VSiphoviridae41.6512011-07-03 [10] c5Lactobacillus delbrueckii31 841VSiphoviridae42.0502011-07-03 [10] phiJBLactobacillus delbrueckii36 969TSiphoviridae47.7462013-09-29 [11] phiLdbLactobacillus delbrueckii33 996VSiphoviridae42.0592013-09-29 [12] PL-1Lactobacillus casei38 880TSiphoviridae44.9592013-11-05 [13] J-1Lactobacillus casei40 931TSiphoviridae44.8632013-11-05 [13] ATCC 8014-B1Lactobacillus plantarum38 002VSiphoviridae47.6602012-10-10 [14] ATCC 8014-B2Lactobacillus plantarum80 618VSiphoviridae36.91272012-10-10 [14] Sha1Lactobacillus plantarum41 726TSiphoviridae40.6582011-01-30 [15] LF1Lactobacillus fermentum42 606TSiphoviridae45.0572011-01-30 [16] JCL1032Lactobacillus delbrueckii49 433TSiphoviridae44.2772011-07-05 [10] phijl1Lactobacillus plantarum36 674VSiphoviridae39.4462005-03-28 [17] A2Lactobacillus casei43 411TSiphoviridae44.9641999-12-16 [18] phiadhLactobacillus gasseri43 785TSiphoviridae35.6631999-08-10 [19] LP65Lactobacillus plantarum131 522VMyoviridae37.31792004-11-01 [20] phig1eLactobacillus42 295TSiphoviridae43.1601997-04-03 [21] phijlb1Lactobacillus gasseri38 269TMyoviridae37.4542014-04-03 [22] phiAQ113Lactobacillus helveticus36 566VMyoviridae36.5562012-12-06 [23] Lc-NuLactobacillus rhamnosus36 466VSiphoviridae44.2512004-05-30 [24] Lb338-1Lactobacillus paracasei142 111VMyoviridae37.41992009-04-05 [25] Lrm1Lactobacillus rhamnosus39 989TSiphoviridae45.5542008-08-01 [26] LL-HLb.delbrueckii subsp. lactis34 659VSiphoviridae47.8512007-03-31 [27] KC5aLactobacillus gasseri38 239TMyoviridae36.9612006-03-21—phiAT3Lactobacillus casei39 166TSiphoviridae44.6532004-05-30 [28]噬菌体名称宿主菌株基因组大小/bp类别噬菌体类型
[4]含量/%ORF序列释放时间GC
由表1可知,已完成全基因组测序的乳杆菌噬菌体数量增加。CL1、CL2、iLp84、iLp1308和iA2等全基因组信息是在近几年公布的
[6]。上述噬菌体均分离自副干酪乳杆菌,对其最初的研究仅局限在生物学特性方面
[6,29]。2010年,Capra等
[29]对3 个副干酪乳杆菌温和噬菌体iA2、CL1和CL2的特征进行表述,未对其分子特性进行研究。直至2015年,Mercanti等
[6]才分析了5 个副干酪乳杆菌噬菌体(CL1、CL2、iLp84、iLp1308、iA2)的全基因组序列。研究表明这些噬菌体基因组大小为34 155(iA2)~39 474 bp(CL1),GC含量为44.8%~45.6%。此外,Mercanti等
[6]指出了副干酪乳杆菌噬菌体的多样性差异,提出噬菌体和其宿主间常常发生DNA交换。
2 主要乳杆菌噬菌体基因组学
2.1 德氏乳杆菌噬菌体基因组特征
德氏乳杆菌作为发酵剂普遍应用在发酵乳制品生产中以完成产品发酵,提高终产品感官特性
[30]。基于DNA同源性,通过基因杂交及比较基因组分析将德氏乳杆菌噬菌体分成5 个类群(a、b、c、d、e)
[8]。对a和c组德氏乳杆菌噬菌体研究最为广泛。a组噬菌体LL-H最初于1972年被分离
[31],其全基因组序列和2 个不同转录阶段的转录图谱已被确定
[27]。同样,扁平头部的温和噬菌体JCL1032是c组德氏乳杆菌噬菌体的代表成员,该噬菌体已经过系统的研究,包括基因组测序
[10]、基因的整合分析
[32]和作为其受体的磷壁酸的识别
[33]。
随着分离频率明显增加,近期研究主要集中在b组噬菌体,如结构蛋白已确定的噬菌体Ld3、Ld17、Ld25A
[9],而d组噬菌体仍处于被广泛研究阶段。Casey等
[8]对噬菌体Ldl1的基因组和蛋白质组特征论证后,发现该噬菌体不属于已确定的德氏乳杆菌噬菌体组中任何一类,通过创建蛋白质树,将其归为新组即e组中,成为迄今为止该组的唯一成员。Ld1基因组为74 806 bp,有79 个ORF,GC含量为37.8%,具有与植物乳杆菌噬菌体B2和Sha1较为密切的关系,表明Ldl1也许与B2、Sha1具有共同宿主。
分离于乳清中的保加利亚乳杆菌噬菌体Ld17基因组为32 975 bp,GC含量为41.97%,有50 个ORF,与Ld3和Ld25A有97%和95%的序列一致性,并与噬菌体c5、LL-Ku和phiLdb基因组有高度序列一致性
[12]。b组噬菌体大部分结构模块间是高度保守的,而Ld17在编码产物ORF19
Ld17和ORF20
Ld17所代表的区域表现出与同组其他噬菌体显著的差异。ORF19
Ld17编码产物,包含一个蛋白三联体重复单元(G-X-Y)
16和重复7 次的氨基酸三联体甘氨酸-天冬氨-赖氨酸。研究表明该重复序列可促进糖蛋白的结合
[34]。ORF20
Ld17编码ClpP蛋白酶。噬菌体头部蛋白比预计分子质量低,可能由ClpP蛋白酶翻译后的修饰作用引起的
[9]。
2.2 发酵乳杆菌噬菌体基因组特征
发酵乳杆菌是专性异型发酵乳酸菌,参与很多传统食品的自然发酵
[35]。大多数菌株能产生细菌素并利用一些难消化的糖类,发挥某些益生特性
[36]。
发酵乳杆菌YB5是分离自中国酸奶,产细菌素的耐酸菌株
[37]。Zhang Xiuhong等
[38]经丝裂霉素诱导获得其温和长尾噬菌体ФPYB5,有一个pac-型包装机制。该报道指出该噬菌体基因组为32 847 bp,GC含量为45.21%,其序列含有46 个ORFs其中25 个有确定功能。基因orf15~orf18编码产物分别为1 个细胞壁水解酶、2 个葡聚糖酶和1 个脂解蛋白,序列分析发现其与噬菌体—宿主相互作用是相关的,能在噬菌体吸附和侵入过程中发挥作用。噬菌体ФPYB5编码一个由穴蛋白和细胞溶解酶组成的独特裂解盒
[2]。通常情况下,宿主细胞裂解依赖穴蛋白和细胞溶解酶作用,穴蛋白在细菌细胞膜上引起小孔,通过该小孔细胞溶解酶传到胞壁质层。许多噬菌体典型穴蛋白有2 个或3 个跨膜转运区域
[39]。ФPYB5的穴蛋白只有一个跨膜区,并且在不存在穴蛋白情况下,细胞溶解酶的表达可能引起宿主细胞裂解
[12]。以上研究暗示了细胞溶解酶有着一个特殊的输出机制,但具体机理仍需进一步分析
[2]。
温和噬菌体LF1是从野生型发酵乳杆菌经丝裂霉素诱导获得的
[16],透射电子显微镜形态分析显示,LF1有等距的头部和不能收缩的尾部,属于长尾噬菌体科。Yoon等
[16]并对其基因组进行研究,测序结果表明LF1含有1 条42 606 bp的双链DNA,GC含量为45%,有57 个ORF。同源性分析表明其全基因组序列与噬菌体ФPYB5的核苷酸同源性为28%
[2,38]。
2.3 干酪乳杆菌噬菌体基因组特征
侵染干酪乳杆菌的第一个噬菌体分离于日本
[40]。最早研究的干酪乳杆菌噬菌体基因组是A2。这个长尾温和噬菌体能整合其基因组到干酪乳杆菌ATCC 27092中
[41]。在基因组初端转录区域,有2 个启动子已被确定,其中启动子P
L指导基因cI的表达(裂解循环阻遏物,lytic cycle repressor),启动子P
R调停裂解功能和cro溶原阻遏物基因的转录
[42-44]。噬菌体结合位点在裂解和溶原模块之间,接近编码整合酶基因orf20
[45]。该噬菌体基因组存在2 个-1核糖体移码翻译
[33,46],分别产生了主要衣壳蛋白和尾部蛋白
[47-48]。
噬菌体J-1是从益力多异常发酵产物中分离得到的,在抗噬菌体J-1的菌株用于饮料生产2 a后,噬菌体PL-1被分离
[49]。起初日本研究组对该噬菌体做了大量研究,包括噬菌体形态,失活,细胞吸附,DNA注入和分解酶特性等
[50-53]。Dieterle等
[13,54]对噬菌体J-1和PL-1基因组序列和功能进行了阐述,噬菌体J-1的DNA长40 931 bp,噬菌体PL-1的DNA长38 880 bp。GC含量分别是44.8%和44.9%。2 个噬菌体均有含10 个碱基的独特黏性末端,该黏性末端是单链的3’延长物(左端末尾为3’-CGGTCGGCCT),比之前Nakashima等
[55]所描述的序列GAACGGTCGGCCTC短4 个碱基对。J-1基因组中含有63 个ORF,不含有tRNA基因。J-1和PL-1的基因产物gp16识别糖结构,可结合到干酪乳杆菌细胞中,这暗示着gp16与宿主识别相关。此外,研究者还发现L-鼠李糖的加入可以抑制两者间的结合及噬菌体对细胞壁的吸附作用
[54]。
2.4 植物乳杆菌噬菌体基因组特征
目前,已有6 株植物乳杆菌噬菌体基因组完成测序
[14,15,20,21,56]。从植物原料中分离噬菌体phigle的基因组大小为42 259 bp,包含62 个ORF
[21]。分离自朝鲜泡菜的噬菌体Sha1基因组为大小为41 726 bp,GC含量为40.6%
[15]。分离自发酵黄瓜的噬菌体phiJL-1基因组大小为36 674 bp,GC含量为39.4%及52 个ORF
[17]。分离自发酵肉的噬菌体LP65的基因组较大,为13 1573 bp,其GC含量为37.7%,有165 个ORF
[20]。Marcó等
[14]对分离自青贮玉米和厌氧污泥中的2 个植物乳杆菌烈性长尾噬菌体ATCC 8014-B1(B1)和ATCC 8014-B2(B2)的基因组进行了研究,研究发现,pac-型噬菌体B1的线性双链DNA基因组有38 002 bp,GC含量为47.6%,含有60 个ORF,覆盖基因组长度的93%。噬菌体B1基因组和植物乳杆菌噬菌体JL-1有77%的同源性。cos-型噬菌体B2双链DNA基因组大小为80 618 bp,GC含量为36.9%,含有127 个ORF,覆盖基因组长度的87%。噬菌体B2的GC含量较低,与植物乳杆菌肌尾噬菌体LP65的GC含量相似
[20]。在噬菌体B2中发现6 个tRNAs,但在B1中没有。该6 个tRNAs位于B2基因组中的2 个区域(6 246~7 814 nt和42 308~42 522 nt),编码产生天冬酰胺、亮氨酸、蛋氨酸、甘氨酸和精氨酸。tRNA的存在与噬菌体较大基因组联系在一起的
[56]。噬菌体B1基因组中没有发现溶原相关基因,证实了其烈性本质。噬菌体B2中发现少数与前噬菌体相关的蛋白质(Orf39、Orf43和Orf105),但噬菌体B2表现出烈性噬菌体的生长特性。
2.5 瑞士乳杆菌噬菌体基因组特征
Zago等
[23]对瑞氏乳杆菌噬菌体ΦAQ113的基因组特性进行了研究。研究表明,该噬菌体的线型环状双链DNA基因组为36 566 bp,GC含量为37%。在瑞氏乳杆菌噬菌体ΦAQ113基因组中不存在黏性末端,DNA连接未能改变限制性图谱,并提出ΦAQ113可利用pac-DNA包装机制的假设。该噬菌体基因组大小与烈性长尾植物乳杆菌噬菌体ФphiJL-1、鼠李糖乳杆菌噬菌体ΦLc-Nu和温和肌尾格氏乳杆菌噬菌体ΦKC5a相似
[12]。GC含量类似于格氏乳杆菌噬菌体Φadh
[57]。56 个ORF中约有90%被确定具有假定功能(DNA复制/修饰、DNA包装、头部及尾部形态形成等)。与此同时该研究小组发现ΦAQ113基因组与加氏乳杆菌噬菌体KC5a和约氏乳杆菌噬菌体Lj771基因组关系密切,尤其在包装及尾部和头部形态发生的基因模块排列上与加氏乳杆菌和约氏乳杆菌噬菌体相似。ΦAQ113和这2 个噬菌体间的进化相似性证实了它们可能来自于一个共同的祖先
[23]。
3 乳杆菌噬菌体功能基因组学
继乳杆菌噬菌体基因组测序完成后,利用基因组结构所提供的信息及同源性比较,可预测ORF的功能
[58]。基因组研究已确定了大量重要噬菌体侵染相关基因。研究表明
[2,7.8,14,15,24,54],噬菌体基因组是高度模块化的,功能基因共同聚集在基因组结构中,并在协调中被开启和关闭,基因组可能包括以下功能模块:DNA包装、形态发生、细胞裂解与溶原模块及DNA复制模块,其中一些ORF编码蛋白在噬菌体侵染过程中至关重要,如溶菌酶、转录调控因子、抗阻遏物、受体结合蛋白、DNA甲基化酶、毒素组分、末端酶亚基、细胞壁水解酶和尾部卷曲蛋白等。噬菌体ΦAQ113基因组中,ORF1、3、4的基因产物是噬菌体末端酶的大小亚基,与加氏乳杆菌噬菌体ФKC5a中的相似度在66%~83%间。表明这3 个蛋白质参与噬菌体DNA包装。ORF35编码蛋白显示出属于裂解模块基因的相似基因
[23],与瑞士乳杆菌温和噬菌体Φ-0303的细胞内溶素Mur-LH具有高度一致性(88%)
[59],并在其他瑞士乳杆菌噬菌体中也被检测到,同源性达99%
[60]。温和噬菌体LBR48全基因组序列分析显示尾部蛋白质基因不是长的,与许多乳品噬菌体尾部形态形成模块不同,编码一个很长的多区域蛋白质
[61]。该尾丝是缺失的,与透射电子显微镜观察相一致
[7]。噬菌体Ldl1中ORF23
Ldl1编码的尾部卷曲蛋白(tape measure protein,TMP)包含结合肽聚糖的LysM(lysin motif)区域
[62]和催化肽聚糖水解的转糖苷酶区域,这2 个区域暗示出噬菌体侵染过程中TMP发挥重要及多功能作用并决定噬菌体尾部长度。Ldl1包含溶原性遗传因子和一个重组酶/整合酶基因,然而即使有溶原性ORF存在,Ldl1也未表现溶原特性,表明它也许是从溶原性噬菌体进化的
[8]。
噬菌体侵染周期的第一步是噬菌体吸附,吸附专一性取决于宿主菌株表面组分和噬菌体组分,受体和反受体。吸附第一阶段是受体结合蛋白识别受体结合部位,该结合是可逆的,因此不能保证成功的噬菌体侵染;在第二阶段,由于细菌与噬菌体表面蛋白质的不可逆结合,噬菌体牢固地附着在细菌细胞上
[63-64]。基因组研究揭示了噬菌体编码受体结合蛋白的基因,如噬菌体Ldl1基因组中存在2 个可能的宿主识别蛋白基因ORF26
Ldl1和ORF27
Ldl1
[8]。在革兰氏阳性菌中,肽聚糖、磷壁酸、脂磷壁酸和细胞膜相关蛋白已被报道为噬菌体受体分子
[33,65]。Munsch-Alatossava等
[66]提出并讨论了噬菌体LL-H侵染乳酸乳杆菌ATCC 15808过程中相关的胞外噬菌体-宿主相互作用模型,即在噬菌体侵染的胞外阶段,LL-H的反受体-乳酸乳杆菌ATCC 15808的脂磷壁酸相互作用模型并阐述德氏乳杆菌脂磷壁酸的结构和噬菌体受体性质,表明特有基因改变如影响噬菌体受体分子结构的自发突变可能阻止特定噬菌体吸附从而增加突变菌株对噬菌体的抗性。另外噬菌体与宿主菌的共同进化可促进突变发生,并且反受体基因突变可能改变这些突变噬菌体的宿主范围。对噬菌体建立紧密联系后,噬菌体将遗传物质注入细菌细胞质中,衣壳留在细胞外
[67]。烈性噬菌体通过改变宿主复制机制和代谢功能来复制自身的遗传物质并合成噬菌体编码蛋白质,产出丰富的子代噬菌体。然而某些温和噬菌体在宿主中以休眠状态进入溶原性生命周期
[63]。
4 比较基因组分析
部分乳杆菌噬菌体全基因组测序的完成,为不同乳杆菌噬菌体间全基因组比较提供了基础。通过不同乳杆菌噬菌体种间(或种内)的比较基因组学,发现不同种(或种内不同类型)乳杆菌噬菌体在基因组结构上的差异、获得噬菌体进化的相关信息
[68]。目前,比较基因组分析主要集中在长尾噬菌体,因为仅有7 个肌尾噬菌体的全基因组是可获得的
[5,7,20,22-23,25]。
Riipinen等
[10]对德氏乳杆菌噬菌体LL-Ku、c5、JCL1032的基因组序列进行了比较分析,得出同属于b组噬菌体LL-Ku和c5的基因组显示较高的核苷酸序列同源性。噬菌体LL-Ku和c5在时间和空间上是相隔孤立的,但基因组间是高度保守的。1950年早期,cos型乳酸乳杆菌噬菌体LL-Ku分离自芬兰奶酪厂的乳清样品中
[69]。酸奶中产生的cos型保加利亚乳杆菌烈性噬菌体c5于1963年在法国被分离得到
[70]。LL-Ku和c5蛋白质组和基因组结构几乎是相同的,除DNA包装模块(g1
c5/g2
c5和g4
LL-Ku)和复制模块(g34
c5)中3 个大小为859、678、470 bp的插入和缺失。比较序列分析表明插入、缺失和重组在LL-Ku和c5的多样性中发挥了重要作用。点突变和小的插入和缺失会进一步导致噬菌体的遗传变异。JCL1032作为c组中的温和噬菌体,其基因组包含功能性溶原模块、2组Ⅰ型内含子和尾部模块,基因组远大于噬菌体LL-Ku和c5,且与LL-Ku和c5显示出较低的DNA同源性
[10]。Dieterle等
[54]对分离自同一株干酪乳杆菌的噬菌体PL-1和J-1进行基因组比较分析,数据表明这两个噬菌体基因组几乎是相同的,但与J-1相比,PL-1在基因切换区域有一个与免疫相关的大小为1.9 kb基因缺失,编码特定尾部蛋白的基因也有所不同。和其他乳酸菌噬菌体核苷酸序列相比,噬菌体PL-1和J-1与噬菌体A2在包装和结构基因方面密切相关
[71]。根据BLAST分析,发酵乳杆菌噬菌体LF1和ФPYB5的核苷酸一致性为28%
[2,16],包装和结构模块的基因同源性较高。两者DNA包装区域的序列相似为93%,因此LF1和ФPYB5的基因组间有良好的基因镶嵌关系
[2]。
5 结 语
目前,研究领域对乳杆菌噬菌体研究取得了重大进步,但仍滞后于其他噬菌体的研究。且国内对乳杆菌噬菌体研究很少,这方面亟待加强。乳杆菌噬菌体分子水平上的研究揭示了其多样性和进化规律,根据基因组特点对乳杆菌噬菌体重新进行了分类及界定。随着益生乳杆菌产品的增加,噬菌体相关的发酵、生产困难在未来将会增加。对新型噬菌体的特性进行详细阐明,包括它们的起源、进化及与其他噬菌体的关系,都需以噬菌体全基因组为基础。乳杆菌噬菌体的全基因组测序,为深入挖掘功能基因明确基因表达特点及系统阐述噬菌体增殖机制提供一种有效的研究手段。另外采用现代生物技术对宿主菌株进行基因改造这可阻断噬菌体对宿主菌的裂解作用,从而选育出抗噬菌体菌株,对有效预防噬菌体污染具有重要意义。
参考文献:
[1] 何亮, 熊礼宽, 曾忠铭. 乳杆菌的分类与分子鉴定方法研究进展[J].中国微生态学杂志, 2007, 19(3): 312-313. DOI:10.3969/j.issn.1005-376X.2007.03.037.
[2] ZHANG X H, WANG S H, GUO T T, et al. Genome analysis of Lactobacillus fermentum temperate bacteriophage ФPYB5[J]. International Journal of Food Microbiology, 2011, 144(3): 400-405. DOI:10.1016/j.ijfoodmicro.2010.10.026.
[3] 崔艳华, 徐德昌, 曲晓军. 乳酸菌基因组学研究进展[J]. 生物信息学, 2008, 6(2): 85-89. DOI:10.3969/j.issn.1672-5565.2008.02.012.
[4] 冯烨, 刘军, 孙洋, 等. 噬菌体最新分类与命名[J]. 中国兽医学报, 2013, 33(12): 1954-1958.
[5] LIU M, BISCHOFF K M, GILL J J, et al. Bacteriophage application restores ethanol fermentation characteristics disrupted by Lactobacillus fermentum[J]. Biotechnology for Biofuels, 2015, 6(1): e20067. DOI:10.1186/s13068-015-0325-9.
[6] MERCANTI D J, ROUSSEAU G M, CAPRA M L, et al. Genomic diversity of phages infecting probiotic strains of Lactobacillus paracasei[J]. Applied and Environmental Microbiology, 2016, 82(1):95-105. DOI:10.1128/AEM.02723-15.
[7] JANG S H, YOON B H, CHANG H I. Complete nucleotide sequence of the temperate bacteriophage LBR48, a new member of the family Myoviridae[J]. Archives of Virology, 2011, 156(2): 319-322. DOI:10.1007/s00705-010-0841-7.
[8] CASEY E, MAHONY J, NEVE H, et al. Novel phage group infecting Lactobacillus delbrueckii subsp. lactis, as revealed by genomic and proteomic analysis of bacteriophage Ldl1[J]. Applied and Environmental Microbiology, 2015, 81(4): 1319-1326. DOI:10.1128/ AEM.03413-14.
[9] CASEY E, MAHONY J, BOTTACINI F, et al. Molecular characterization of three Lactobacillus delbrueckii subsp. bulgaricus phages[J]. Applied and Environmental Microbiology, 2014, 80(18):5623-5635. DOI:10.1128/AEM.01268-14.
[10] RIIPINEN K A, FORSMAN P, ALATOSSAVA T. The genomes and comparative genomics of Lactobacillus delbrueckii phages[J]. Archives of Virology, 2011, 156(7): 1217-1233. DOI:10.1007/s00705-011-0980-5.
[11] GUO T, ZHANG C, XIN Y, et al. A novel chimeric prophage vB_ LdeS-phiJB from commercial Lactobacillus delbrueckii subsp. bulgaricus[J]. Journal of Industrial Microbiology & Biotechnology, 2016, 43(5): 1-9. DOI:10.1007/s10295-016-1739-5.
[12] WANG S H, KONG J, GAO C, et al. Isolation and characterization of a novel virulent phage (phiLdb) of Lactobacillus delbrueckii[J]. International Journal of Food Microbiology, 2010, 137(1): 22-27. DOI:10.1016/j.ijfoodmicro.2009.10.024.15.
[13] DIETERLE M E, RUSSELL D, HATFULL G, et al. Complete genome sequences of Lactobacillus phages J-1 and PL-1[J]. Genome Announcements, 2014, 2(1): e00998-13. DOI:10.1128/genomeA.00998-13.
[14] MARCÓ M B, GARNEAU J E, TREMBLAY D, et al. Characterization of two virulent phages of Lactobacillus plantarum[J]. Applied and Environmental Microbiology, 2012, 78(24): 8719-8734. DOI:10.1128/ AEM.02565-12.
[15] YOON B H, JANG S H, CHANG H I. Sequence analysis of the Lactobacillus temperate phage Sha1[J]. Archives of Virology, 2011, 156(9): 1681-1684. DOI:10.1007/s00705-011-1048-2.
[16] YOON B H, CHANG H. Complete genomic sequence of the Lactobacillus temperate phage LF1[J]. Archives of Virology, 2011, 156(10): 1909-1912. DOI:10.1007/s00705-011-1082-0.
[17] LU Z, ALTERMANN E, BREIDT F, et al. Sequence analysis of the Lactobacillus plantarum bacteriophage phiJL-1[J]. Gene, 2005, 348(1): 45-54. DOI:10.1016/i.gene.2004.12.052.
[18] PROUX C, van SINDEREN D, SUÁREZ J, et al. The dilemma of phage taxonomy illustrated by comparative genomics of Sfi21-like Siphoviridae in lactic acid bacteria[J]. Journal of Bacteriology, 2002, 184(21): 6026-6036. DOI:10.1128/JB.184.21.6026-6036.2002.
[19] ALTERMANN E, KLEIN J R, HENRICH B. Primary structure and features of the genome of the Lactobacillus gasseri temperate bacteriophage (phi) adh[J]. Gene, 1999, 236(2): 333-346
[20] DILLMANN M L, BRUSSOW H, MARVIN-GUY L, et al. Lactobacillus plantarum Bacteriophage LP65: a new member of the spo1-like genus of the family myoviridae[J]. Journal of Bacteriology, 2004, 186(21): 7069-7083. DOI:10.1128/JB.186.21.7069-7083.2004.
[21] KODAIRA K I, OKI M, KAKIKAWA M, et al. Genome structure of the Lactobacillus temperate phage phi φg1e: the whole genome sequence and the putative promoter/repressor system[J]. Gene, 1997, 187(1): 45-53. DOI:10.1016/S0378-1119(96)00687-7.
[22] BAUGHER J L, DURMAZ E, KLAENHAMMER T R. Spontaneously induced prophages in Lactobacillus gasseri contribute to horizontal gene transfer[J]. Applied & Environmental Microbiology, 2014, 80(11): 3508-3517. DOI:10.1128/AEM.04092-13.
[23] ZAGO M, SCALTRITI E, ROSSETTI L, et al. Characterization of the genome of the dairy Lactobacillus helveticus bacteriophageФAQ113[J]. Applied and Environmental Microbiology, 2013, 79(15): 4712-4718. DOI:10.1128/AEM.00620-13.
[24] TUOHIMAA A, RIIPINEN K A, BRANDT K, et al. The genome of the virulent phage Lc-Nu of probiotic Lactobacillus rhamnosus, and comparative genomics with Lactobacillus casei phages[J]. Archives of Virology, 2006, 151(5): 947-965. DOI:10.1007/s00705-005-0672-0.
[25] ALEMAYEHU D, ROSS R P, O’SULLIVAN O, et al. Genome of a virulent bacteriophage Lb338-1 that lyses the probiotic Lactobacillus paracasei cheese strain[J]. Gene, 2009, 448(1): 29-39. DOI:10.1016/ j.gene.2009.08.008.
[26] DURMAZ E, MILLER M J, AZCARATE-PERIL M A, et al. Genome sequence and characteristics of Lrm1, a prophage from industrial Lactobacillus rhamnosus strain M1[J]. Applied & Environmental Microbiology, 2008, 74(15): 4601-4609. DOI:10.1128/AEM.00010-08.
[27] MIKKONEN M, RÄISÄNEN L, ALATOSSAVA T. The early gene region completes the nucleotide sequence of Lactobacillus delbrueckii subsp. lactis phage LL-H[J]. Gene, 1996, 175(1/2): 49-57. DOI:10.1016/0378-1119(96)00119-9.
[28] LO T C, SHIH T C, LIN C F, et al. Complete genomic sequence of the temperate bacteriophage phiAT3 isolated from Lactobacillus casei ATCC 393[J]. Virology, 2005, 339(1): 42-55. DOI:10.1016/ j.virol.2005.05.022.
[29] CAPRA M L, MERCANTI D J, REINHEIMER J A, et al. Characterisation of three temperate phages released from the same Lactobacillus paracasei commercial strain[J]. International Journal of Dairy Technology, 2010, 63(3): 369-405. DOI:10.1111/j.1471-0307.2010.00600.x.
[30] GUGLIELMOTTI D M, GOLOWCZYC M, REINHEIMER J A, et al. Probiotic potential of Lactobacillus delbrueckii strains and their phage resistant mutants[J]. International Dairy Journal, 2007, 17(8): 916-925. DOI:10.1016/j.idairyj.2006.11.004.
[31] RAVIN V, RÄISÄNEN L, ALATOSSAVA T. A conserved C-terminal region in gp71 of the small isometric-head phage LL-H and ORF474 of the prolate-head phage JCL1032 is implicated in specificity of adsorption of phage to its host, Lactobacillus delbrueckii[J]. Journal of Bacteriology, 2002, 184(9): 2455-2459. DOI:10.1128/JB.184.9.2455-2459.2002.
[32] RIIPINEN K A, RÄISÄNEN L, ALATOSSAVA T. Integration of the group c phage JCL1032 of Lactobacillus delbrueckii subsp. lactis and complex phage resistance of the host[J]. Journal of Applied Microbiology, 2007, 103(6): 2465-2475. DOI:10.1111/j.1365-2672.2007.03479.x.
[33] RÄISÄNEN L, SCHUBERT K, JAAKONSAARI T, et al. Characterization of lipoteichoic acids as Lactobacillus delbrueckii phage receptor components[J]. Journal of Bacteriology, 2004, 186(16):5529-5532. DOI:10.1128/JB.186.16.5529-5532.2004.
[34] SCARBOROUGH R M, NAUGHTON M A, TENG W, et al. Design of potent and specific integrin antagonists. peptide antagonists with high specificity for glycoprotein IIb-IIIa[J]. Journal of Biological Chemistry, 1993, 268(2): 1066-1073.
[35] CALDERON SANTOYO M, LOISEAU G, GUYOT J P, et al. Study of starch fermentation at low pH by Lactobacillus fermentum Ogi E1 reveals uncoupling between growth and alpha-amylase production at pH 4.0[J]. International Journal of Food Microbiology, 2003, 80(1):77-87. DOI:10.1016/S0168-1605(02)00142-X.
[36] SONGISEPP E, KALS J, KULLISAAR T, et al. Evaluation of the functional efficacy of an antioxidative probiotic in healthy volunteers[J]. Nutrition Journal, 2005, 4: 22. DOI:10.1186/1475-2891-4-22.
[37] 张秀红, 孔健, 于文娟, 等. 发酵乳杆菌YB5生理特征的研究[J]. 中国食品学报, 2008, 8(6): 33-38. DOI:10.3969/j.issn.1009-7848.2008.06.006.
[38] ZHANG X H, KONG J, QU Y. Isolation and characterization of a Lactobacillus fermentum temperate bacteriophage from Chinese yogurt[J]. Journal of Applied Microbiology, 2006, 101(4): 857-863. DOI:10.1111/j.1365-2672.2008.03953.x.
[39] YOUNG I, WANG I N, ROOF W D. Phages will out: strategies of host cell lysis[J]. Trends in Microbiology, 2000, 8(3): 120-128. DOI:10.1016/S0966-842X(00)01705-4.
[40] VILLION M, MOINEAU S. Bacteriophages of Lactobacillus[J]. Frontiers in Bioscience, 2009, 14: 1661-1683. DOI:10.2741/3332.
[41] HERRERO M, CASO J L, SUÁREZ J E, et al. Characterization of phi 393-A2, a bacteriophage that infects Lactobacillus casei[J]. Microbiology, 1994, 140(10): 2585-2590. DOI:10.1099/00221287-140-10-2585.
[42] GARCÍA P, LADERO V, ALONSO J C, et al. Cooperative interaction of CI protein regulates lysogeny of Lactobacillus casei by bacteriophage A2[J]. Journal of Virology, 1999, 73(5): 3920-3929.
[43] LADERO V, GARCÍA P, BASCARAN V, et al. Identif i cation of the repressor-encoding gene of the Lactobacillus bacteriophage A2[J]. Journal of Bacteriology, 1998, 180(13): 3474-3476.
[44] LADERO V, GARCÍA P, ALONSO J C, et al. A2 cro, the lysogenic cycle repressor, specifically binds to the genetic switch region of Lactobacillus casei bacteriophage A2[J]. Virology, 1999, 262(1):220-229. DOI:10.1006/viro.1999.9903.
[45] ALVAREZM A, HERRERO M, SUÁREZ J. The site-specific recombination system of the Lactobacillus species bacteriophage A2 integrates in gram-positive and gram-negative bacteria[J]. Virology, 1998, 250(1): 185-193. DOI:10.1006/viro.1998.9353.
[46] XU J, HENDRIX R W, DUDA R L. Conserved translational frameshift in dsDNA bacteriophage tail assembly genes[J]. Molecular Cell, 2004, 16(1): 11-21. DOI:10.1016/j.molcel.2004.09.006.
[47] GARCÍA P, RODRIGUEZ I, SUÁREZ J E. A-1 ribosomal frameshift in the transcript that encodes the major head protein of bacteriophage A2 mediates biosynthesis of a second essential component of the capsid[J]. Journal of Bacteriology, 2004, 186(6): 1714-1719. DOI:10.1128/JB.186.6.1714-1719.2004.
[48] RODRIGUEZ I, GARCÍA P, SUARE J E. A second case of -1 ribosomal frameshif affecting a major virion protein of Lactobacillus bacteriophage A2[J]. Journal of Bacteriology, 2005, 187(23): 8201-8204. DOI:10.1128/JB.187.23.8201-8204.2005.
[49] WATANABE K, TAKESUE S, JINNAI K, et al. Bacteriophage active against the lactic acid beverage-producing bacterium Lactobacillus casei[J]. Applied Microbiology, 1970, 20(3): 409-415.
[50] KAKITA Y, KASHIGE N, MURATA K, et al. Inactivation of Lactobacillus bacteriophage PL-1 by microwave irradiation[J]. Microbiology and Immunology, 1995, 39(8): 571-576. DOI:10.1111/ j.13 48-0421.1995.tb02244.x.
[51] KASHIGE N, KAKITA Y, NAKASHIMA Y, et al. Mechanism of the photocatalytic inactivation of Lactobacillus casei phage PL-1 by titania thin fi lm[J]. Current Microbiology, 2001, 42(3): 184-189. DOI:10.1007/s002840010201.
[52] KASHIGE N, NAKASHIMA Y, MIAKE F, et al. Cloning, sequence analysis, and expression of Lactobacillus casei phage PL-1 lysis genes[J]. Archives of Virology, 2000, 145(8): 1521-1534. DOI:10.1007/s00705007 0073.
[53] CAPRA M L, QUIBERONI A, REINHEIMER J. Phages of Lactobacillus casei/paracasei: response to environmental factors and interaction with collection and commercial strains[J]. Journal of Applied Microbiology, 2006, 100(2): 334-342. DOI:10.1111/j.1365-2672.2005.02767.x.
[54] DIETERLE M E, BOWMAN C, BATTHYANY C, et al. Exposing the secrets of two well-known Lactobacillus casei Phages, J-1 and PL-1, by genomic and structural analysis[J]. Applied and Environmental Microbiology, 2014, 80(22): 7107-7121. DOI:10.1128/AEM.02771-14.
[55] NAKASHIMA Y, IKEDA H, KAKITA Y, et al. Restriction map of the genomic DNA of Lactobacillus casei bacteriophage PL-1 and nucleotide sequence of its cohesive single-stranded ends[J]. Journal of General Virology, 1994, 75(9): 2537-2541. DOI:10.1099/0022-1317-75-9-2537.
[56] SAMSON J, MOINEAU S. Characterization of Lactococcus lactis phage 949 and comparison with other lactococcal phages[J]. Applied and Environmental Microbiology, 2010, 76(20): 6843-6852. DOI:10.1128/AEM.00796-10.
[57] ALTERMANN E, KLEIN J R, HENRICH B. Primary structure and features of the genome of the Lactobacillus gasseri temperate bacteriophage Φadh[J]. Gene, 1999, 236(2): 333-346. DOI:10.1016/ S0378-1119(99)00236-X.
[58] 赵晋平, 徐平丽, 孟静静, 等. 从结构基因组学到功能基因组学[J].生命科学研究, 2006(增刊1): 58-62.
[59] DEUTSCH S M, GUEZENEC S, PIOT M, et al. Mur-LH, the broad-spectrum endolysin of Lactobacillus helveticus temperate bacteriophage Ф-0303[J]. Applied and Environmental Microbiology, 2004, 70(1): 96-103. DOI:10.1128/AEM.70.1.96-103.2004.
[60] ZAGO M, ROSSETTI L, REINHEIMER J, et al. Detection and identif i cation of Lactobacillus helveticus bacteriophages by PCR[J]. Journal of Dairy Research, 2008, 75(2): 196-201. DOI:10.1017/ S0022029908003208.
[61] BRÜSSOW H. Phages of dairy bacteria[J]. Annual Review of Microbiology, 2001, 55(1): 283-303. DOI:10.1146/annurev. micro.55.1.283.
[62] GIRBE B, ANTON S, JAN K, et al. LysM, a widely distributed protein motif for binding to (peptido)glycans[J]. Molecular Microbiology, 2008, 68(4): 838-847. DOI:10.1111/j.1365-2958.2008.06211.x.
[63] GÓRECKI R, SZCZEPANKOWSKA A, BARDOWSKI J, et al. Lactic acid bacteria resistance to bacteriophage and prevention techniques to lower phage contamination in dairy fermentation[M]. In Tech Open:Mercelino Kongo, 2013: 23-72.
[64] RÄISÄNEN L V, KOLOMIETS E I, DEY E S, et al. Bacteriophage receptors, mechanisms of phage adsorption and penetration into host cell[J]. Polish Journal of Microbiology, 2010, 59(3): 145-155.
[65] RÄISÄNEN L, DRAING C, PFITZENMAIER M, et al. Molecular interaction between lipoteichoic acids and Lactobacillus delbrueckii phages depends on D-alanyl and α-glucose substitution of poly (glycerophosphate) backbones[J]. Journal of Bacteriology, 2007, 189(11): 4135-4140. DOI:10.1128/JB.00078-07.
[66] MUNSCH-ALATOSSAVA P, ALATOSSAVA T. The extracellular phage-host interactions involved in the bacteriophage LL-H infection of Lactobacillus delbrueckii ssp. lactis ATCC 15808[J]. Frontiers in Microbiology, 2013, 4: 1-5. DOI:10.3389/fmicb.2013.00408.
[67] 姜延龙, 霍贵成, 田波. 乳酸菌抗噬菌体机制研究进展[J]. 中国乳品工业, 2005, 33(8): 30-33. DOI:10.3969/j.issn.1001-2230.2005.08.009.
[68] DESIERE F, PRIDMORE R D, BRUSSOW H. Comparative genomics of the late gene cluster from Lactobacillus phages[J]. Virology, 2000, 275(2): 294-305. DOI:10.1006/viro.2000.0498.
[69] FORSMAN P, ALATOSSAVA T. Genetic variation of Lactobacillus delbrueckii subsp. lactis bacteriophages isolated from cheese processing plants in finland[J]. Applied & Environmental Microbiology, 1991, 57(6): 1805-1812.
[70] ACCOLAS J P, SPILLMANN H. Morphology of bacteriophages of Lactobacillus bulgaricus, L. lactis and L. helveticus[J]. Journal of Applied Microbiologyl, 1979, 47(2): 309-319. DOI:10.1111/j.1365-2672.1979.tb01760.x.
[71] GARCÍA P, LADERO V, SUÁREZ J E. Analysis of the morphogenetic cluster and genome of the temperate Lactobacillus casei bacteriophage A2[J]. Archives of Virology, 2003, 148(6): 1051-1070. DOI:10.1007/ s00705-003-0008-x.
Advances in Genomic Studies of Lactobacillus Phages
LIU Ying, XI Yu, FAN Mengru, WANG Zhengyu, WU Wenru, ZHANG Heping, CHEN Xia*
(Key Laboratory of Dairy Biotechnology and Engineering, Ministry of Education, Inner Mongolia Agricultural University, Hohhot 010018, China)
Abstract:With the extensive application of Lactobacilli for the dairy industry, phage contamination has posed an increasingly serious threat to the final products. Currently, phage research has evolved from the morphological to the molecular level. The full-length genomic sequences of many phages have been acquired. Genomic studies have contributed to revealing the infective mechanisms of phages and in turn exploring the key functional genes. In this paper, the genomic characteristics, functio nal genomics and comparative genomics of the major Lactobacillus phages published were analyzed, aiming to provide a theoretical basis for further exploration of phages.
Key words:Lactobacillus; phage; genomics
DOI:10.7506/spkx1002-6630-201703043
中图分类号:Q754
文献标志码:A
文章编号:1002-6630(2017)03-0271-07
引文格式:
刘颖, 习羽, 范梦茹, 等. 乳杆菌噬菌体基因组学研究进展[J]. 食品科学, 2017, 38(3): 271-277. DOI:10.7506/spkx1002-6630-201703043. http://www.spkx.net.cn
LIU Ying, XI Yu, FAN Mengru, et al. Advances in genomic studies of Lactobacillus phages[J]. Food Science, 2017, 38(3):271-277. (in Chinese with English abstract)
DOI:10.7506/spkx1002-6630-201703043. http://www.spkx.net.cn
收稿日期:2016-03-18
基金项目:国家自然科学基金青年科学基金项目(31301517);内蒙古自治区自然科学基金面上项目(2013MS1207)
作者简介:刘颖(1993—),女,硕士研究生,研究方向为乳品生物技术。E-mail:932845217@qq.com
*通信作者:陈霞(1982—),女,副教授,博士,研究方向为乳品生物技术。E-mail:chenxia8280@163.com
