图1 ECL示意图 [5,36]
Fig. 1 Schematic illustration of ECL [5,36]
魏 麟 1 ,朱方莉 1 ,周 洋 2 ,Khairy Morsy MOHAMED 3 ,袁 超 4 ,董星星 1 ,王小红 1 ,李锦铨 1,2, *
关键词: 噬菌体;食源性病原菌;检测方法;基因工程
食源性疾病是人体摄入被污染的食品而引起感染或产生中毒性症状的疾病,可能由细菌、病毒、寄生虫、毒素等引起,其中细菌是导致食源性疾病的主要原因。常见的食源性致病菌有沙门氏菌(Salmonella)、空肠弯曲菌(Campylobacter jejuni)、单核细胞增生李斯特氏菌(Listeria moncytogenes)、大肠杆菌O157:H7(Escherichia coli O157:H7)、志贺氏菌(Shigella)和金黄色葡萄球菌(Staphylococcus aureus)等 [1] 。据统计,在发达国家,每年约有1/3的人群感染食源性疾病,全世界每年有220万~1 000万 人因患食源性疾病而丧生。美国食品工业中每年有约25%的食品由于被病原菌污染而损失 [2] 。为了降低食源性疾病的发生率和减轻由此造成的经济损失,世界卫生组织正在推动食品监管体系建立的进程,以保证从农场到餐桌整条食品链上的食品安全 [3] 。在此背景下,寻找快速高效、准确便捷的食源性病原菌检测方法逐渐成为研究人员关注的热点。
目前,除了传统的微生物学检测方法、生理生化鉴定外,现代的食源性病原菌快速检测方法还包括免疫学方法(如酶联免疫吸附测定法、免疫扩散法、胶体金技术)、分子生物学方法(如实时定量聚合酶链式反应(polymerase chain reaction,PCR)技术、多重PCR技术、环介导等温扩增技术、基因芯片技术、蛋白质芯片技术等)、仪器分析方法(如质谱法、流式细胞技术等)。上述方法在一定程度上缩短了检测时间,提高了检测效率,甚至能够同时检测多种病原菌。但已有的检测方法也面临着一些挑战,例如上述方法均不能有效区分死细菌、活细菌和处于存活非可培养(viable but nonculturable,VBNC)状态的细菌,免疫学方法中存在抗体生产成本高、批次不稳定等问题,仪器分析法存在设备昂贵、样品制备工作量大等问题。
噬菌体不仅具备结构简单、特异性强、价格低廉等特性,而且具有能够区分活细菌和死细菌的能力,并容易与其他传统检测方法相结合,因此,噬菌体在致病菌快速检测方面的应用被广大学者和研究人员所重视 [4] ,美国食品药品监督管理局宣布支持将一些噬菌体作为基础的检测方法应用到病原菌的检测中 [3] 。一些发达国家也已出现具备商业化能力的噬菌体相关检测产品,例如检测领域的国际知名公司——法国生物梅里埃公司现已研发出利用重组噬菌体蛋白检测食品和环境样本中沙门氏菌的技术,并取得国际标准化组织(International Organization for Standardization,ISO)和美国分析化学家协会(Association of Official Analytical Chemists,AOAC)的国际认证。噬菌体及其产物应用于食源性病原菌检测方法具有良好的发展前景。本文就噬菌体在快速检测食源性病原菌中的应用进行综述。
细菌噬菌体是一类感染细菌的病毒,具有病毒的一般特性:个体微小、不具有完整细胞结构,只含有单一核酸;其特别之处是专以细菌为宿主,且通常具有特异性。凡是有细菌的地方,就可能有相应的噬菌体存在。噬菌体主要由核酸和蛋白质外壳组成,目前已知的绝大多数噬菌体为有尾部结构的二十面体,其主要结构可分为头部、尾部与基部,二十面体的头部中包裹着核酸,中空针状结构的尾部包括尾领、尾鞘与尾髓,基部由尾板、尾刺和尾丝组成,尾部和基部在噬菌体与细菌接触的过程中起吸附等重要作用。
如今已有很多噬菌体快速检测食源性致病菌的方法,这些检测方法的原理涉及到噬菌体与细菌相互作用的全过程,有的建立在噬菌体与宿主细胞最初的识别与吸附过程上,另一些则依赖于侵染过程中噬菌体释放核酸进入宿主细胞体内,如噬菌体基因在宿主细胞内表达,或后代噬菌体裂解释放到胞外 [5] 。
2.1.1 噬菌体用于细菌分型
噬菌体分型是一种常用的传统细菌分型方法,用于区分细菌的不同亚型。噬菌体分型与常规生化实验法的检出率在统计学上无显著差异,且可缩短时间 [1] 。早在20世纪60年代,用噬菌体对鼠伤寒沙门氏菌进行分型的方法就已被建立,研究人员首次用11 种噬菌体区分鉴别了12 种鼠伤寒沙门氏菌 [5] 。迄今为止,对于绝大多数较普遍的食源性病原菌如沙门氏菌、弯曲杆菌、大肠杆菌和李斯特菌等,噬菌体分型体系已经较为成熟 [6] 。
研究表明,单独使用某种方法分型难以达到很高的准确度,而两种或多种分型技术的联合使用可使分型结果更加准确 [1] 。因此,尽管近年来基于分子生物学发展起来的分子分型技术迅猛发展,但噬菌体分型因其分辨率高、廉价、快速等特点作为一种联合分型方法仍然发挥着不可替代的重要作用。例如,在对O157:H7的分型进行研究时,研究人员指出,由于O157:H7的高度保守性,对该菌的流行病学研究必须采用不同的分型技术,在不同分型方法比较的实验中,噬菌体分型技术能够将O157:H7分成66 种详细类别,分辨率高,是重要的分型方法之一 [7] 。
2.1.2 噬菌体用于鉴别细菌是否处于存活状态
研究发现,包括沙门氏菌、副溶血弧菌、大肠杆菌等重要食源性病原菌在内的60多种细菌,在物理因素(温度、湿度、氧气含量、光照强度等)、化学因素(营养成分、有害化学物质等)及生物因素的诱导作用下,会进入VBNC状态 [8] 。当细菌处于VBNC状态时,其仍具有代谢活性并保持着致病的潜力,在适宜的环境中即可恢复。但是传统的微生物学方法或生化方法无法检测出该种状态细菌的存在,分子生物学方法、免疫学方法和仪器分析也很难将其与已经死亡的无害细胞进行区分,这对传统的微生态学和食品安全提出了新的挑战。而噬菌体只与活细胞识别、结合的选择特异性将出现假阳性与假阴性结果的可能性降到了最小 [3] 。
Fernandes等 [3] 对比了抗体与噬菌体在识别不同生理状态细胞方面的差异,不同样品中混有不同比例的VBNC状态和死亡细胞(包括热力杀死的细胞),抗体不加识别地结合捕获了处于不同生理状态下的所有细菌而造成假阳性现象,噬菌体则能够有效区分不同生理状态的细胞。
2.1.3 噬菌体扩增法
烈性噬菌体感染宿主菌后短时间内会引起细胞的裂解而在双层平板中形成明显的噬菌斑 [1] ,细胞裂解、后代噬菌体释放,即标志着特异性宿主细胞的存在 [5] ;因此根据噬菌斑的数目可以检测样品中病原菌的含量。噬菌体扩增法简单、成本较低 [1] ,其显著优点是不需要基因工程或化学修饰等复杂技术 [5] ,因此得以在一些规模较小的食品加工厂推广应用 [9] 。Stewart等 [10] 使用Felix O-1噬菌体检测沙门氏菌,用NCIMB 10116和NCIMB 10884噬菌体检测铜绿假单胞菌,在纯培养实验中,检测时间为4 h,检测限分别为600 CFU/mL和40 CFU/mL。此外,Rees [11-12] 、Siqueira [13] 等采用噬菌体扩增法已成功地检测出结核杆菌、沙门氏菌、李斯特菌、大肠杆菌、绿脓假单胞菌和弯曲杆菌等。
噬菌体扩增法还可与光密度法结合,光密度值的减小表示信号放大细胞由于被噬菌体感染而裂解,总数下降;光密度值增大则表示信号放大细胞没有被噬菌体感染,自身生长繁殖而总数上升。这种方法已经成功被应用于检测人工染菌的脱脂奶粉、鸡肉和碎牛肉中,平均检测限为3 CFU/g或3 CFU/mL,总检测时间为20 h(包括前增菌时间) [14] 。
基于噬菌体的微生物学检测方法的缺点在于其对噬菌体总量、感染时间等条件有一定要求,且微生物学检测方法通常耗时相对较长,对操作人员的操作技术要求高。
近年来,噬菌体与免疫学、分子生物学和纳米科学等其他方法的联合使用推动了基于噬菌体的食源性病原菌快速检测技术的迅速发展,催生了一系列操作简单、耗时短、灵敏度高的检测方法,下文将进行分述。
报告噬菌体是经过基因工程方法改造过的噬菌体,内含能够编码介导荧光发色的基因/基因簇 [15] ,或含有其他由噬菌体特异性编码的可表达被检测标记物的基因,在识别特异性宿主细胞(目标菌)后,噬菌体侵染细胞并将其DNA注射进入宿主细胞内 [16] ,随着报告基因的表达,目标菌体便可快速得到检测。
2.2.1 gfp基因
绿色荧光蛋白(green fluorescent protein,gfp)是一种生物发光蛋白,其内源荧光基因在受到紫外光或蓝光激发时可高效发射出清晰可见的绿光 [1] ,gfp基因的优势是无需再加任何底物和辅助因子即可构成荧光检测系统。
Tanji等 [17] 用T4噬菌体构建了一个大肠杆菌gfp报告噬菌体,T4噬菌体具有裂解性,其优点是目标病原菌在被检测到的同时可被杀死,缺点在于荧光基因表达后细菌裂解,这不利于待测产物荧光的检测。因此,研究人员改造出了一种不裂解细菌的T4e - /gfp噬菌体,并成功用于大肠杆菌的检测,检测时间缩短至1 h。Miyanaga等 [18] 也成功应用T4e - /gfp噬菌体检测出了污水中的大肠杆菌K12。相似地,Oda等 [19] 应用gfp标记T2类噬菌体,构建重组噬菌体PP01/gfp检测大肠杆菌O157∶H7,该法能够检测到处于VBNC状态下的大肠杆菌。
2.2.2 荧光素酶基因(lux和luc)
噬菌体自身无法合成发光反应所必需的荧光素酶、底物和能量,因此在细胞外不发光,当其侵染宿主细菌后,发光基因表达,细菌代谢活动可产生发光反应所必需的物质,噬菌体便在宿主细胞内发光 [1] 。lux基因操纵子包括luxCDE和luxAB,luxCDE负责编码脂肪酸还原酶体系(还原酶、合成酶和转移酶),用于促进作为反应底物的脂肪醇的生物合成。luxAB则编码荧光素酶α和β亚基,与底物一起用于发光反应 [20] 。因此,单独使用luxAB需要添加额外的物质如癸醛作为辅助 [5] ,使用复合的luxCDABE则无需添加。
Ripp [5] 利用能够感染近95%李斯特菌血清型的A511噬菌体构建了luxAB报告噬菌体,用于检测单核细胞增生李斯特菌。也有研究人员在鼠伤寒沙门氏菌P22噬菌体中引入luxAB基因,于16 h内检测出样品中的鼠伤寒沙门氏菌 [1] 。Ripp [5] 进一步在luxAB的基础上首次构建了luxCDABE报告噬菌体。随后,在此基础上,一种新的鼠伤寒沙门氏菌报告噬菌体SPC32H-CDABE被成功构建,并用于检测食物样品中的沙门氏菌,其在卷心莴苣、猪肉和牛奶中的检测限分别为22、37 CFU/g和700 CFU/g [21] 。
luc可编码真核生物荧光素酶,该酶可以催化D-荧光素氧化成氧化荧光素,氧化过程中会发出生物荧光,并可在560 nm波长处检测到。luc与luxAB的使用类似,也需要添加额外的辅助物质 [5] 。Bardarov等 [22] 使用luc报告噬菌体phAE142检测唾液样品中的结合分歧杆菌,检测时间为7 d,与传统的标准分歧杆菌生长指示管法相比缩短了检测时间,但缺点在于其检测限高于传统方法。
2.2.3 lacZ基因
β-半乳糖苷酶基因(lacZ)编码β-半乳糖苷酶,该酶可催化β-半乳糖苷的水解反应,常作为报告基因分子与荧光素酶联用 [1] ,或作为化学发光底物。Goodridge [23] 利用lacZ报告噬菌体成功检测出样品中的O157:H7,检测限为10 2 CFU/mL。与gfp、lux和luc相比,lacZ报告噬菌体使用较少 [3] 。
2.2.4 inaW基因
冰核蛋白基因(inaW)能够使没有成冰核能力的细菌具有成冰核能力,其与通常的报告基因不同之处在于它所介导的是一种物理过程,即水的液-固状态的改变 [1] 。Pawlowska等 [24] 将其与免疫磁珠分离法联用检测样品中的沙门氏菌,将检出限降低到5 CFU/mL。
2.2.5 碱性磷酸酶
将编码碱性磷酸酶的基因通过基因工程方法导入噬菌体基因组中,噬菌体侵染合适的宿主菌,在宿主菌体内表达该基因产生碱性磷酸酶,细菌裂解后碱性磷酸酶释放,通过检测该特殊蛋白质即可完成对宿主菌的检测。将噬菌体特异性编码的碱性磷酸酶作为检测标记物,与将细菌裂解后产生的自身的内容物作为待测物质相比,该方法有效地避免了由于细菌自然裂解而产生的检测误差,从而大大提高了准确度。Neufeld等 [25] 使用该方法在3 h内检测到了单个的大肠杆菌。
报告噬菌体法用时短、耗能少、准确性较好,随着转基因技术的不断进步与革新,有很好的应用前景,但由于噬菌体的专一性,该类方法无法同时检测一个细菌中的多种血清型 [1] 。
免疫学方法是依据抗原与抗体特异性反应的检测方法,如常见的酶联免疫吸附法。免疫学方法的核心是抗体和抗原的互作反应,而抗体的生产存在批次之间不稳定、分子质量大、保存条件要求高、纯化成本高、交叉反应和动物福利等问题,采用噬菌体及其结构成分替代抗体可以有效解决以上问题,极大地降低了检测成本。法国生物梅里埃公司已经利用噬菌体重组蛋白替代抗体,研发出用于检测沙门氏菌的商业化产品,且将检测时间由原来的3 d缩短至19 h [26] 。
Stambach等 [27] 以免疫层析法为基础,使用A511噬菌体代替原有第一抗体,结合拉曼活性染料,构建了SERS-LFI(surface enhanced Raman spectroscopy-lateral flow immunochromatography)法对李斯特菌进行检测,将检测时间由原来的8 h缩短至2 h。此外,研究人员指出,传统的噬菌体空斑检测方法需要长达24 h的时间,而SERS-LFI法有望将检测时间缩短到30 min。
近年来对噬菌体的研究表明,噬菌体的结构成分尾丝蛋白(tailspike proteins,TSP)对宿主细菌也具有高亲和性。Schmidt等 [28] 使用沙门氏菌噬菌体9NA和噬菌体P22的TSP替代抗体,构建ELITA(enzyme linked immunosorbent assay like tailspike adsorption)方法,对44 种沙门氏菌菌株进行分析,并成功将血清型为O1的菌株从非葡萄糖基化菌株中区分离出来。同时,噬菌体细胞壁结合区域(cell wall binding domain,CBD)也具有应用于酶联免疫吸附测定的潜在价值 [29] 。噬菌体结构成分TSP、CBD等亲和性、操作性强,生产应用简便,与传统免疫学方法结合使用具有很好的应用前景。
2.4.1 生物发光法
2.4.1.1 ATP生物发光法
ATP能够在真核生物luc基因编码的萤火虫荧光素酶的作用下发光。利用噬菌体裂解细菌释放出胞内物质ATP,使用荧光素酶使ATP释放出能量,其产生荧光的强度与ATP含量成正比,据此可推断出菌落总数 [1] 。Miyanaga等 [18] 根据腺苷酸激酶(adenylate kinase,AK)可催化ADP转化成ATP的原理将此法改进,噬菌体裂解细菌后释放出来的AK将加入样品中的ADP转化成ATP,提高了样品的发光强度,能够在1 h之内检测出低于10 3 CFU/mL的大肠杆菌。
2.4.1.2 NADH生物发光法
特定代谢时期的微生物细胞中的NADH(nicotinamide adenine dinucleotide)含量相对稳定,且其含量与食品中微生物的数量成正相关。细菌死亡后,NADH则在胞内酶作用下被分解。利用烈性噬菌体裂解宿主细菌,释放出胞内的NADH,其在氧化还原酶和荧光素酶作用下发光,通过测定荧光强度即可推算出菌落总数 [1] 。Roach等 [30] 利用噬菌体介导的NADH生物发光法成功检测出克雷伯氏菌。
生物发光法操作简便、灵敏度较高,也可用于食品生产设备及环境中的病原微生物检测。美国Sample6公司已研发出一种基于噬菌体和生物荧光素酶结合的检测系统 [31] ,即使是极少量细菌的存在也可被该系统检测。
2.4.2 化学发光试剂(荧光标记噬菌体法)
一些化学发光试剂可通过化学共价结合或物理吸附到细胞的某些基团上,从而表现其发光特性,检测宿主菌。随着科技的发展,YOYO-1、SYBR、SYTO等商业化荧光试剂逐步被开发出来 [1] ,由于其与核酸的高亲和性,该类荧光试剂在一定程度上取代了传统的荧光染料。Goodridge [23] 使用YOYO-1荧光标记的LG1噬菌体检测碎牛肉和牛奶中的大肠杆菌O157:H7,检测时间分别为12 h和7 h,检测限为100 CFU/mL。Mosier-Boss等 [32] 成功使用SYBR gold染料标记P22噬菌体以检测沙门氏菌。
2.4.3 物理发光(量子点)
纳米微粒半导体量子点是一种具有较高发光效率、巯基羧酸修饰的纳米微粒荧光探针,其具有很高的量子产率和光稳定性 [5] ,CdSe、CdTe等半导体量子点已成为科学界的研究热点 [1] 。
Edgar等 [33] 利用量子点与T7噬菌体结合检测大肠杆菌,其原理是通过改造噬菌体的衣壳蛋白,使其具有一段生物素酰化的肽段,噬菌体侵染宿主菌后,在宿主菌内合成的后代噬菌体就成为被生物素酰化的噬菌体,链霉亲和素功能化的量子点只结合并标记被生物素酰化的噬菌体 [5] 。使用该法检测河水样品中的大肠杆菌,检测时间为1 h,可检测到20 CFU/mL大肠杆菌。
然而,因为T7噬菌体对宿主菌持续的裂解使得噬菌体-细胞结合物的定量分析复杂化(目标细胞减少而碎片增多),量子点与噬菌体结合率低,且阻碍后续检测中量子点噬菌体与细菌细胞的相互作用,其应用方法还有待改进 [34] 。Yim等 [34] 使用改造过的温和型Lambda噬菌体规避了这些问题,实验结果证明Lambda噬菌体结合的平均量子点数量多于T7噬菌体。
生物传感器通常含有一种生物感应成分,如抗体、酶或受体配对成分。这类生物样成分被结合到一个理想的传感器上,传感器可以将不同的生物信号,如光信号、电化学信号、热信号等转换成可测量的数码读数。
2.5.1 电化学原理传感器
电化学原理传感器将待检测物质与生物活性材料发生生物学反应产生的信号,由换能器转换成可定量处理的电信号等其他信号,经二次仪表放大输出,获得待检物数量的信息 [1] 。
2.5.1.1 以噬菌体与宿主细胞吸附、互作过程为基础的检测
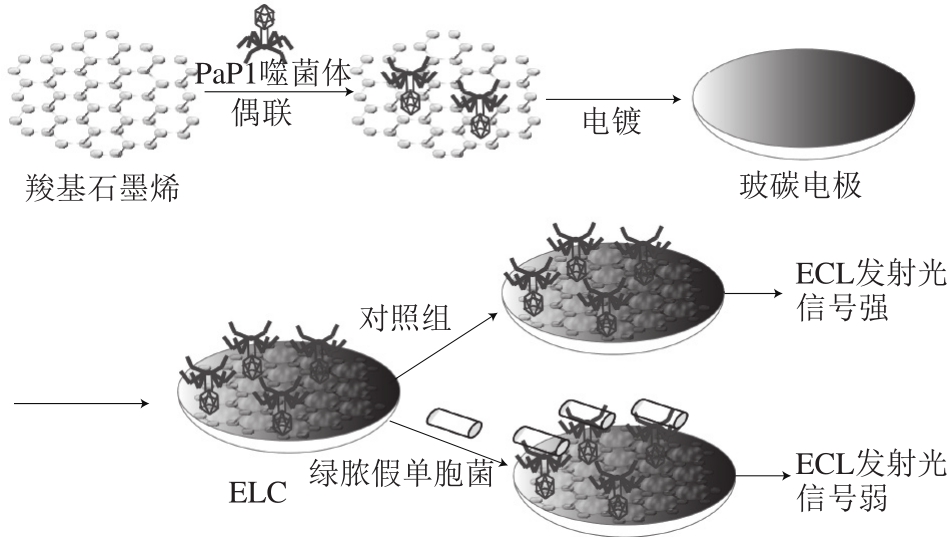
噬菌体对宿主细胞吸附、侵染的过程往往会产生电信号的变化,通过构建合适的电化学原理传感器以监测不同形式电信号的变化,经过一定的信号转换和定量计算,即可完成对目标菌的检测。主要包括以下几类:1)离子级联反应。噬菌体触发的离子级联反应使用电化学方法检测噬菌体与宿主菌吸附后的微小电压波动,噬菌体吸附后将其核酸注入宿主菌,宿主菌立刻释放近10 8 数量级的离子到周围的环境介质中,其离子通量可以通过两个金属薄膜微电极检测 [5] 。Dobozi-King等 [35] 以大肠杆菌作为目标检测菌,构建了一个nanowell钛电极电容器检测离子通量,并提出该方法具有灵敏度达到1 cell/μL的潜力。2)电化学发光生物传感器(electrochemiluminescent biosensor,ECL)。PaP1噬菌体是从医院污水中分离出来的一种烈性噬菌体,它对绿脓假单胞菌具有很强的吸附性和特异性,Yue Huan等 [36] 将分离出来的PaP1噬菌体与羧基石墨烯结合,并电镀到玻碳电极上,构建出了一种新的ECL(图1)。当噬菌体特异性吸附绿脓假单胞菌时,由于形成的绝缘生物复合物阻碍了表面电子转移和ECL活性分子的光散射,ECL光信号下降。当绿脓假单胞菌总数在1.4×10 2 ~1.4×10 6 CFU/mL范围时,ECL发射光信号随其数量上升而线性下降,检测限仅为56 CFU/mL,整个检测过程仅需30 min [36] 。3)表面等离子体共振(surface plasmon resonance,SPR)。SPR检测病原菌的原理是通过检测由抗体与病原菌的特异性结合导致的折射率变化,从而得出病原菌的数量 [5] 。与抗体相比,噬菌体同样具有与病原菌特异性结合的能力,且通常灵敏度更高,成本更低。Balasubramanian等 [37] 将SPR法与噬菌体结合,通过检测噬菌体与金黄色葡萄球菌特异性结合导致的折射率变化,完成了对金黄色葡萄球菌的定量检测,检测限为10 4 mL -1 。虽然SPR仪器比较昂贵,但是有望研发成为一种高通量的检测技术。

图1 ECL示意图 [5,36]
Fig. 1 Schematic illustration of ECL [5,36]
2.5.1.2 以噬菌体裂解宿主细胞产生内溶物为基础的检测
内溶物变化导致阻抗变化。细菌在培养基内生长繁殖的过程中,大分子电惰性物质(碳水化合物、蛋白质、脂类等)转化成具有电活性的小分子物质(乳酸盐、醋酸盐等),这些离子态物质能增加培养基的导电性,使培养基的阻抗发生变化 [1] ;因此可以通过检测培养基的阻抗变化情况来判定细菌的生长繁殖特性。Chang等 [38] 利用此法,将专一性噬菌体AR1作用于E. coil O157:H7,根据阻抗变化时间成功检测出大肠杆菌。
内溶物与外加底物发生反应产生信号变化。Lambda噬菌体侵染大肠杆菌后,最终会导致细胞裂解,释放出β-半乳糖苷酶,β-半乳糖苷酶可作用于外加底物对氨基苯-β-D-半乳吡喃糖苷,并在碳阳极产生氧化产物对氨基苯酚,通过安培计检测电流变化 [5] 。Yemini等 [39] 使用类似方法,通过噬菌体B1-7064引发细胞释放α-葡萄糖苷酶,从而实现了对蜡状芽孢杆菌的检测。
2.5.2 磁传感器
一些软磁铁非晶态合金可参与构建灵敏的磁传感器,用于食源性病原菌的检测 [40] 。Lakshmanan等 [41] 使用磁性传感器与丝状噬菌体结合检测鼠伤寒沙门氏菌,其原理是任何非磁弹性的物质接触传感器表面,就会抑制机械振荡,从而引起共振频率降低(图2)。使用丝状噬菌体作为生物探针,与磁传感器结合(物理吸附),然后暴露于数量范围在5×10~5×10 8 CFU/mL的鼠伤寒沙门式菌中,当鼠伤寒沙门氏菌特异性结合噬菌体时,通过磁场变化引起电信号转变,最终导致共振频率的改变,而共振频率与结合菌的数量有关。

图2 磁传感器示意图 [41-42]
Fig. 2 Schematic illustration of magnetoelastic sensors [41-42]
Chen等 [42] 采用与上述方法类似的原理,使用噬菌体C4-22与磁传感器结合,检测鸡胸肉样品中的鼠伤寒沙门氏菌,除了检测鸡胸肉表面的带菌情况,他们还将磁传感器放置在鸡肉内部,即距离表面分别为0.1、0.5 cm和1.0 cm的位置进行检测,这也是第一个使用传感器直接检测鸡肉表面和内部病原菌污染的成功实例 [42] 。结果表明,使用噬菌体C4-22与普通对照组(无噬菌体)相比,其与沙门氏菌的结合亲和度提高了12 倍,且在鸡肉表面样品中的检测限仅为7.86×10 3 CFU/mm 2 。
与传感器联用的噬菌体检测技术操作简单、分析速度快,可发展为便携式检测设备,但其抗干扰能力有待提高。
Davesiakwei等 [43] 将噬菌体与质谱法结合检测食品样品中的大肠杆菌,其原理为T7噬菌体可专一感染大肠杆菌,并在1~2 h后扩增100 倍,后用液相色谱-多级反应监测串联质谱法定量检测专一性噬菌体肽段。实验结果表明,肉汤培养基、椰子水和苹果汁中的检测限分别为3.0×10 3 、4.1×10 4 、1.9×10 3 CFU/mL。相似地,Madonna等 [44] 使用基质辅助激光/电离质谱法通过检测噬菌体衣壳蛋白的分子质量从而定量检测大肠杆菌,检测限为10 4 CFU/mL,检测时间为2 h。Martelet等 [45] 则进一步把噬菌体扩增与免疫磁珠分离法结合,用液相色谱与质谱联合检测噬菌体标记蛋白,完成对大肠杆菌的定量检测。以上结果表明噬菌体与质谱法联用不仅可以对大肠杆菌进行定量检测,而且能够提高检测灵敏度。
除噬菌体的整体应用外,噬菌体的某些结构成分或受体结合蛋白也可单独使用,由于其对细菌具有特殊的亲和力,且易于操作,因而被广泛应用于病原菌的快速检测中,如TSP和噬菌体CBD等。其中已有一些技术开始逐步迈向商业化,如法国生物梅里埃公司利用噬菌体重组蛋白开发出的用于检测沙门氏菌的Vidas ® up重组噬菌体技术已获得国际认证;Schmelcher等 [46] 研发出的由不同血清型李斯特菌噬菌体编码的CBD和荧光蛋白组成的综合性分型试剂盒,可同时区分和检测多种血清型的李斯特菌。下文以CBD为代表展开论述。
噬菌体能够编码一种细胞内溶素,其本质是肽聚糖水解酶,该酶包含两个结构域,肽聚糖水解酶活性区域也称N端裂解区域,CBD也称C端结合区域 [47-48] ,前者决定了内溶素酶对宿主的裂解活性,后者则负责结合宿主细胞壁上的蛋白质。其中CBD具有与宿主以高度特异性、亲和性结合的特点以及易于修饰的特性,因此其在病原菌快速检测领域中的应用成为了新的焦点,甚至比全噬菌体的应用得到了更为广泛的关注 [49] 。
抗体-免疫磁珠法已经广泛应用于病原菌快速检测中的分离、富集过程,但是抗体由于其成本、批次稳定性等问题还存在一定的改善空间。而CBD与特定细菌的结合具有很高的亲和性和特异性 [20] ,且CBD比抗体的体积小很多;研究表明,1 个细胞表面至少有10 7 数量级的CBD结合位点 [50] ,同时,CBD来源于噬菌体,成本较低。CBD所具有的这些特性使其成为快速检测病原菌方法中抗体的理想替代品 [20,51] 。
已有许多研究人员将CBD与免疫磁珠法结合完成了对病原菌的高效富集。Yu Junping等 [50] 提出细胞内溶素plyV12可通过免疫磁珠分离方法用来富集宿主细胞,实验结果表明利用CBD和磁珠组合物进行富集,可以检测到牛奶样品中的金黄色葡萄球菌,检测限为400 CFU/mL,检测时间仅为1.5 h。Schmelcher等 [46] 通过实验证实CBD能够特异性识别李斯特菌细胞壁肽聚糖上特有的碳水化合物组分,且具有很高的结合活性和亲和力。在此基础上,Kretzer等 [29] 证明了特定噬菌体编码的CBD能够分别识别其对应宿主蜡状芽孢杆菌和产气荚膜梭菌,从而推得CBD免疫磁珠方法的普遍适用性。
荧光蛋白标记的CBD可用于分型、检测多种特定的食源性病原菌 [46] 。Kretzer等 [29] 将构建CBD-GFP的重组蛋白成功用于李斯特菌的检测。
若用不同颜色的荧光蛋白对CBD进行标记,构建一种鸡尾酒CBD检测工具,则可同时识别食物样品中的多种病原菌 [20] 。例如,Schmelcher等 [46] 研制了一种由不同血清型李斯特菌噬菌体编码的CBD和荧光蛋白组成的综合性分型试剂盒。他们首先建立了一个分类系统,将来源于所有已知的李斯特菌噬菌体产生的内溶素酶CBD分类,并且根据CBD的结构特性、结合特异性与亲和性筛选出每一类中最具代表性的CBD,然后用不同荧光蛋白标记,构建成为报告CBD,并用该综合试剂盒在牛奶和奶酪等食物样品中进行了实验,分别用红星(RedStar)和GFP标记的CBD-P35和CBD500成功实现了同时区分和检测多种血清型的李斯特菌。
3.4.1 CBD-SPR
Kong等 [52] 将宿主为蜡状芽孢杆菌的噬菌体产生的CBD与SPR检测方法结合,这种CBD改良的SPR芯片可用于检测数量范围在10 5 ~10 8 CFU/mL的蜡状芽孢杆菌(图3)。在熟米饭样品中,10 3 CFU/mL的蜡状芽孢杆菌被检出。此外,实验结果证实CBD修饰的芯片所介导的SPR反应强度是抗体修饰的芯片的两倍,表明CBD与宿主结合特性强于抗体。这也是第一个用SPR与CBD结合检测蜡状芽孢杆菌成功的实例 [52] 。

图3 CBD与SPR结合检测方法示意图 [6,52]
Fig. 3 Schematic illustration for the detection method using a CBD-modifed SPR chip [6,52]
3.4.2 CBD-电化学
Tolba等 [48] 将李斯特菌噬菌体产生的内溶素酶与电化学检测方法相结合,CBD与李斯特菌细胞的特异性吸附导致了电子转移电阻的显著变化,当细胞数量范围在10 4 ~10 8 CFU/mL时,电子转移电阻与细胞数量呈线性关系。实验证明在纯培养液和牛奶样品中,李斯特菌的检测限分别为1.1×10 4 、1.1×10 5 CFU/mL。
食源性病原菌的检测涉及从食品原料到成品的每一个环节,无论是大型食品企业还是小型食品工厂或是专门检测机构,在引进一种新的检测技术或设备时必然要考虑其安全性能、准确程度、经济成本、时间成本以及对于技术人员的操作要求等因素。因此,这就需要针对食源性病原菌的快速检测技术不断完善,逐步迈向商业化的道路。
目前,一些基于噬菌体检测的方法已经较为成熟,国外已有进入研发实验阶段甚至投入市场推广应用的先例。例如,法国生物梅里埃公司开发出一种Vidas ® up重组噬菌体技术用于检测沙门氏菌,利用噬菌体重组蛋白替代抗体,大大提高了检测灵敏度,将检测时间缩短至19 h,而现有其他参考方法往往需要3 d,并可验证375 g的样品,该技术目前已得到ISO(AFNOR BIO-12/32-10/11)和AOAC(Official Method 2013.01)的权威认证。
美国Sample6公司已研发出一种基于噬菌体和荧光素酶结合的检测系统 [31] ,为了达到更好的灵敏度,该公司修饰了由噬菌体注入病原菌的生物荧光酶,使得即使只有极少数细菌存在也能产生很亮的荧光,且该方法可仅检测活细胞。该检测系统称为生物发光平台,工厂工人只需使用取样装置(如海绵或棉签)擦拭一下样品,然后将样液经过处理、离心,并与荧光素酶混合,等待噬菌体的作用效果即可,之后通过Sample6 Luminometer检测发射光,并将结果输入公司的软件中,该软件跟踪溯源被污染的产品,从而可以提供一些分析信息,例如污染是否与特定的时间、人员或供应商相关。该方法具有极高的特异性和灵敏性,无需经过其他大多数方法所必须的扩增或培养过程,是世界上第一个具有实用价值的“自我富集病原体诊断系统”。
美国Micro Phage公司研发出一种针对金黄色葡萄球菌的噬菌体扩增检测技术,用FAST Plaque-Response试剂盒能够区分对甲氧西林有抗性的金黄色葡萄球菌和无抗性的菌株,该公司也针对其他重要的细菌(包括不同的抗生素抗性)研发了噬菌体扩增检测的方法,这些产品已在欧盟、北美上市 [53] 。随着科技的发展,由试剂公司开发的商业化荧光试剂如YOYO、SYTO、SYBR Green I等也在很大程度上代替了传统的荧光染料 [1] 。此外,商业荧光素Nano-Glo也因其产生的生物发光信号强烈而具有应用价值 [54] 。
虽然如今还是存在许多阻碍噬菌体检测方法商业化的因素,但不可否认的是,基于噬菌体的检测方法因其具有的明显优势而必然成为食源性病原菌快速检测方法新的发展方向之一。尤其是随着基因工程、基因组测序 [55-56] 和纳米技术等在噬菌体应用领域的逐渐深入,将进一步加速其成为商业化快速检测方法的进程 [20] 。
国内已有少数实验室研究开发出了有关噬菌体检测和控制食源性病原菌的技术方法 [57] ,展示出较好的应用前景,如何突破应用瓶颈、提高其实用性,走出实验室,走向市场,成为研究人员面临的一大挑战。
随着经济的发展,食品安全问题受到了越来越广泛的关注,食源性病原菌则是影响食品安全的主要因素之一。传统的检测手段已经很难满足现代食品工业对于食品安全快速检测的要求,食源性病原菌的检测技术面临着向省时简便、准确高效的现代检测技术转型的问题。噬菌体由于具有结构简单、特异性强、价格低廉等特点,加之其作为现有的极少数能够鉴别区分活细菌和死细菌的检测方法之一,渐渐成为研究的热点。基于噬菌体延伸出来的检测方法多种多样,每种方法都有一定的长处与不足,但往往可以在现有技术中提供一个关键优势,如敏感度高、检测限低或检测成本低等。同时,研究人员也倾向于把噬菌体与免疫学方法、分子生物学方法、传感器技术等检测方法联合使用,将其作为必要的补充技术以获得更好的检测效果。此外,随着人们对噬菌体认识和研究的逐步深入,噬菌体编码的内溶素酶、CBD、TSP等因其所具有的高亲和性和易操作性引起了关注,并已经在一定程度上替代抗体等应用于食源性病原菌的检测。近年来,国外已涌现出一批开发噬菌体相关检测产品的公司,如法国生物梅里埃公司、美国Sample6公司、美国Micro Phage公司等,并已取得显著进展,陆续有相关产品初步投入市场。伴随着生物领域相关技术的发展,以及对于现有应用瓶颈的突破,噬菌体及其相关产物必然将在食品安全检测中得到越来越广泛的应用,逐步走向市场化和常规化。
参考文献:
[1] 李萌, 王静雪, 林洪. 噬菌体检测食源性致病菌的研究进展[J]. 食品科学, 2010, 31(23): 439-446.
[2] SARHAN W A, AZZAZY H M. Phage approved in food, why not as a therapeutic[J]. Expert Review of Anti-Infective Therapy, 2014, 13(1):91-101. DOI:10.1586/14787210.2015.99038.
[3] FERNANDES E, MARTINS V C, NÓBREGA C, et al. A bacteriophage detection tool for viability assessment of Salmonella cells[J]. Biosensors & Bioelectronics, 2014, 52: 239-246.DOI:10.1016/j.bios.2013.08.053.
[4] SCHMELCHER M, LOESSNER M J. Application of bacteriophages for detection of foodborne pathogens[J]. Bacteriophage, 2014, 4(2):1-14. DOI:10.4161/bact.28137.
[5] RIPP S. Bacteriophage-based pathogen detection[J]. Advances in Biochemical Engineering/Biotechnology, 2010, 118: 65-84.DOI:10.1007/10_2009_7.
[6] HAGENS S, LOESSNER M J. Application of bacteriophages for detection and control of foodborne pathogens[J]. Applied Microbiology & Biotechnology, 2007, 76(3): 513-519. DOI:10.1007/s00253-007-1031-8.
[7] 王晓萌. 肠出血性大肠埃希菌O157:H7不同分型方法比较[J]. 中国公共卫生, 2005, 21(4): 451-452.
[8] 丁林贤, 苏晓梅, 横田明, 等. 活的但非可培养(VBNC)状态菌的研究进展[J]. 微生物学报, 2011, 51(7): 858-862.
[9] JUNG L S, AHN J. Evaluation of bacteriophage amplification assay for rapid detection of Shigella boydii in food systems[J]. Microbiology,2016, 66(2): 883-888. DOI:10.1007/s13213-015-1178-y.
[10] STEWART G, JASSIM S A, DENYER S P, et al. The specific and sensitive detection of bacterial pathogens with 4 h using bacteriophage amplification[J]. Journal of Applied Microbiology, 1998, 84(5): 777-783. DOI:10.1046/j.1365-2672.1998.00408.x.
[11] REES C, LOESSNER M J. Phage for the detection of pathogenic bacteria[J]. Bacteriophages: Biology and Applications, 2005: 267-285.DOI:10.1201/9780203491751.ch9.
[12] REES C E D, DODD C E R. Phage for rapid detection and control of bacterial pathogens in food[J]. Advances in Applied Microbiology,2006, 59: 159-186. DOI:10.1016/S0065-2164(06)59006-9.
[13] SIQUEIRA, DEDODD R S, REES C, et al. Phage amplification assay as rapid method for Salmonella detection[J]. Brazilian Journal of Microbiology, 2003, 34(12): 2840-2852. DOI:10.1590/S1517-83822003000500040.
[14] FAVRIN S J, JASSIM S A, GRIFFITHS M W. Application of a novel immunomagnetic separation-bacteriophage assay for the detection of Salmonella enteritidis and Escherichia coli O157:H7 in food[J].International Journal of Food Microbiology, 2003, 85(1/2): 63-71.DOI:10.1016/S0168-1605(02)00483-X.
[15] SMARTT A E, XU T T, JEGIER P, et al. Pathogen detection using engineered bacteriophages[J]. Analytical and Bioanalytical Chemistry,2012, 402(10): 3127-3146. DOI:10.1007/s00216-011-5555-5.
[16] MING H X, ZHU L, ZHANG Y. Rapid quantification of infectious enterovirus from surface water in Bohai Bay, China using an integrated cell culture-qPCR assay[J]. Marine Pollution Bulletin, 2011, 62(10):2047-2054. DOI:10.1016/j.marpolbul.2011.07.024.
[17] TANJI Y, FURUKAWA C, NA S H. et al. Escherichia coli detection by GFP-labeled lysozyme-inactivated T4 bacteriophage[J].Journal of Biotechnology, 2004, 114(1/2): 11-20. DOI:10.1016/j.jbiotec.2004.05.011.
[18] MIYANAGA K, HIJIKATA T, FURUKAWA C, et al. Detection of Escherichia coli in the sewage influent by fluorescent labeled T4 phage[J]. Biochemical Engineering Journal, 2006, 29(1/2): 119-124.DOI:10.1016/j.bej.2005.03.016.
[19] ODA M, MORITA M, UNNO H, et al. Rapid detection of Escherichia coli O157:H7 by using green fluorescent protein-labeled PP01 bacteriophage[J]. Applied of Environment Microbiology, 2004, 70(1):527-534. DOI:10.1128/AEM.70.1.527-534.2004.
[20] BAI J, RYU S, LEE J H. Biocontrol and rapid detection of foodborne pathogens using bacteriophages and endolysins[J]. Frontiers in Microbiology, 2016, 200(7): 1-15. DOI:10.3389/fmicb.2016.00474.
[21] KIM S, KIM M, RYU S. Development of an engineered bioluminescent reporter phage for the sensitive detection of viable Salmonella typhimurium[J]. Analytical Chemistry, 2014, 86(12): 5858-5864. DOI:10.1021/ac500645c.
[22] BARDAROV S, DOU H, EISENACH K, et al. Detection and drugsusceptibility testing of M. tuberculosis from sputum samples using luciferase reporter phage: comparison with the mycobacteria growth indicator tube (MGIT) system[J]. Diagnostic Microbiology& Infectious Disease, 2003, 45(1): 53-61. DOI:10.1016/S0732-8893(02)00478-9.
[23] GOODRIDGE L. Template reporter bacteriophage platform and multiple bacterial detection assays based thereon: US, 20060210968[P/OL].(2006-09-21) [2017-03-15]. https://ntrs.nasa.gov/search.jsp?R=20080009466 2018-06-03T05:00:10+00:00Z.
[24] PAWLOWSKA A M, ZANNINI E, COFFEY A, et al. “Green preservatives”: combating fungi in the food and feed industry by applying antifungal lactic acid bacteria[J]. Advances in Food and Nutrition Research, 2012, 66: 217-238. DOI:10.1016/B978-0-12-394597-6.00005-7.
[25] NEUFELD T, MITTELMAN A S. BUCHNER V, et al. Electrochemical phagemid assay for the specific detection of bacteria using Escherichia coli TG-1 and the M13KO7 phagemid in a model system[J]. Analytical Chemistry, 2005, 77(2): 652-657. DOI:10.1021/ac0488053.
[26] ZADERNOWSKA A, CHAJECKA-WIERZCHOWSKA W,KLEBUKOWSKA L. Vidas up-enzyme-linked fluorescent immunoassay based on recombinant phage protein and fluorescence in situ hybridization as alternative methods for detection of Salmonella enterica serovars in meat[J]. Foodborne Pathogens and Disease, 2014,11(9): 747-752. DOI:10.1089/fpd.2014.1738.
[27] STAMBACH N R, CARR S A, COX C R, et al. Rapid detection of Listeria by bacteriophage amplification and SERS-lateral flow immunochromatography[J]. Viruses, 2015, 7(12): 6631-6641.DOI:10.3390/v7122962.
[28] SCHMIDT A, RABSCH W, BROEKER N K, et al. Bacteriophage tailspike protein based assay to monitor phase variable glucosylations in Salmonella O-antigens[J]. BMC Microbiology, 2016, 16(1): 207.DOI:10.1186/s12866-016-0826-0.
[29] KRETZER J W, LOESSNER M J, SCHMELCHER M, et al. Use of high-affinity cell wall-binding domains of bacteriophage endolysins for immobilization and separation of bacterial cells[J]. Applied &Environmental Microbiology, 2007, 73(6): 1992-2000. DOI:10.1128/AEM.02402-06.
[30] ROACH D R, DONOVAN D M. Antimicrobial bacteriophage-derived proteins and therapeutic applications[J]. Bacteriophage, 2015, 5(3):e1062590. DOI:10.1080/21597081.2015.1062590.
[31] CAPPILLINO M, SHIVERS R P, BROWNELL D R, et al.Sample6 DETECT/L: an in-plant, in-shift, enrichment-free Listeria environmental assay[J]. Journal of AOAC International, 2015, 98(2):1-16. DOI:10.5740/jaoacint.14-213.
[32] MOSIER-BOSS P A, LIEBERMAN S H, ANDREWS J M, et al. Use of fluorescently labeled phage in the detection and identification of bacterial species[J]. Applied Spectroscopy, 2003, 57(9): 1138-1144.DOI:10.1366/00037020360696008.
[33] EDGAR R, MCKINSTRY M, HWANG J, et al. High-sensitivity bacterial detection using biotin-tagged phage and quantumdot nanocomplexes[J]. Proceedings of the National Academy of Science of the United States of America, 2006, 103(13): 4841-4845.DOI:10.1073/pnas.0601211103.
[34] YIM P B, CLARKE M L, MCKINSTRY M, et al. Quantitative characterization of quantum dot-labeled lambda phage for Escherichia coli detection[J]. Biotechnology & Bioengineering, 2009, 104(6):1059-1067. DOI:10.1002/bit.22488.
[35] DOBOZI-KING M, SEO S, KIM J U, et al. Rapid detection and identification of bacteria: sensing of phage-triggered ion cascade(SEPTIC)[J]. Journal of Biological Physics and Chemistry, 2002, 5(1):3-7. DOI:10.1063/1.2138624.
[36] YUE Huan, HE Yong, FAN Encui, et al. Label-free electrochemiluminescent biosensor for rapid and sensitive detection of Pseudomonas aeruginosa using phage as highly specific recognition agent[J]. Biosensors and Bioelectronics, 2017, 94: 429-432.DOI:10.1016/j.bios.2017.03.033.
[37] BALASUBRAMANIAN S, SOROKULOVA I B, VODYANOY V J, et al. Lytic phage as a specific and selective probe for detection of Staphylococcus aureus: a surface plasmon resonance spectroscopic study[J]. Biosensors and Bioelectronics, 2007, 22(6): 948-955.DOI:10.1016/j.bios.2006.04.003.
[38] CHANG T C, DING H C, CHEN S A. conductance method for the identification of Escherichia coli O157:H7 using bacteriophage AR1[J]. Journal of Food Protection, 2002, 65(1): 12-17.DOI:10.4315/0362-028X-65.1.12.
[39] YEMINI M, LEVI Y, YAGIL E, et al. Specific electrochemical phage sensing for Bacillus cereus and Mycobacterium smegmatis[J].Bioelectrochemistry, 2007, 70(1): 180-184. DOI:10.1016/j.bioelechem.2006.03.014.
[40] RUAN C, ZENG K, VARGHESE O K, et al. Magnetoelastic immunosensors: amplified mass immunosorbent assay for detection of Escherichia coli O157:H7[J]. Analytical Chemistry, 2003, 75(23):6494-6498. DOI:10.1021/ac034562n.
[41] LAKSHMANAN R S, GUNTUPALLI R, HU J, et al. Phage immobilized magnetoelastic sensor for the detection of Salmonella typhimurium[J]. Journal of Microbiological Methods, 2007, 71(1):55-60. DOI:10.1016/j.mimet.2007.07.012.
[42] CHEN I H, HORIKAWA S, CHIN B A, et al. Bacterial assessment of phage magnetoelastic sensors for Salmonella enterica Typhimurium detection in chicken meat[J]. Food Control, 2017, 71: 273-278.DOI:10.1016/j.foodcont.2016.07.003.
[43] DAVESIAKWEI O, BNAU M, NG D, et al. Rapid quantification of Escherichia coli in food and media using bacteriophage T7 amplification and liquid chromatography-multiple reaction monitoring tandem mass spectrometry[J]. Journal of Biotechnology, 2014, 192:50-58. DOI:10.1016/j.jbiotec.2014.10.017.
[44] MADONNA A J, VAN C S, VOORHEES K J. Detection of Escherichia coli using immunomagnetic separation and bacteriophage amplification coupled with matrix-assisted laser desorption/ionization time-of-flight mass spectrometry[J]. Rapid Communications in Mass Spectrometry, 2003, 17(3): 257-263. DOI:10.1002/rcm.900.
[45] MARTELET A, HOSTIS G L, NEVERS M C, et al. Phage amplification and immunomagnetic separation combined with targeted mass spectrometry for sensitive detection of viable bacteria in complex food matrices[J]. Analytical Chemistry, 2015, 87(11): 5553-5560.DOI:10.1021/ac504508a.
[46] SCHMELCHER M, SHABAROVA T, EUGSTER M R, et al. Rapid multiplex detection and differentiation of Listeria cells by use of fluorescent phage endolysin cell wall binding domains[J]. Applied &Environmental Microbiology, 2010, 76(17): 5745-5756. DOI:10.1128/AEM.00801-10.
[47] NELSON D C, SCHMELCHER M, RODRIGUEZ-RUBIO L, et al.Endolysins as antimicrobials[J]. Advances in Virus Research, 2012,83: 299-365. DOI:10.1016/B978-0-12-394438-2.00007-4.
[48] TOLBA M, AHMED M U, TLILI C, et al. A bacteriophage endolysinbased electrochemical impedance biosensor for the rapid detection of Listeria cells[J]. Analyst, 2012, 137(24): 5749-5756. DOI:10.1039/c2an35988j.
[49] BRZOZOWSKA E, ŚMIETANA M, KOBA M, et al. Recognition of bacterial lipopolysaccharide using bacteriophage-adhesin-coated longperiod gratings[J]. Biosensors and Bioelectronics, 2015, 67: 93-99.DOI:10.1016/j.bios.2014.07.027.
[50] YU Junping, ZHANG Yun, Zhang Yun, et al. Sensitive and rapid detection of Staphylococcus aureus in milk via cell binding domain of lysin[J]. Biosensors and Bioelectronics, 2016, 77: 366-371.DOI:10.1016/j.bios.2015.09.058.
[51] ROTHFUSS A, O’DONOVAN M, DE BOECK M, et al. Collaborative study on fifteen compounds in the rat-liver Comet assay integrated into 2- and 4- week repeat-dose studies[J]. Mutation Research, 2010,702(1): 40-69. DOI:10.1016/j.mrgentox.2010.07.006.
[52] KONG M, SIM J, KANG T, et al. A novel and highly specific phage endolysin cell wall binding domain for detection of Bacillus cereus[J].European Biophysics, 2015, 44: 437-446. DOI:10.1007/s00249-015-1044-7.
[53] 徐嘉良, 刘童, 黄海燕, 等. 噬菌体在食品安全领域的研究进展[J].食品科技, 2015, 40(8): 296-300. DOI:10.13684/j.cnki.spkj.2015.08.062.
[54] ZHANG D, CORONELAGUILERA C P, ROMERO P L, et al. The use of a novel nanoLuc-based reporter phage for the detection of Escherichia coli O157:H7[J]. Scientific Reports, 2016, 6: 33235.DOI:10.1038/srep33235.
[55] LU T K, COLLINS J J. Dispersing biofilms with engineered enzymatic bacteriophage[J]. Proceedings of the National Academy of Sciences,2007, 104(27): 11197-11202. DOI:10.1073pnas.0704624104.
[56] DONG Xingxing, HUANG Chenxi, MORSY M K, et al. Complete genome sequence of Salmonella enterica lytic bacteriophage LPST10,isolated in China[J]. Genome Announcement, 2017, 5(35): 7-17.DOI:10.1128/genomeA.00427-17.
[57] 李锦铨, 黄晨曦, 王小红, 等. 沙门氏菌噬菌体和噬菌体抗菌组合物及其应用: 201610924016.0[P/OL]. 2016-10-29[2017-03-15]. http://www2.soopat.com/Patent/201610924016.
Progress in the Application of Bacteriophage in the Detection of Foodborne Pathogenic Bacteria
WEI Lin 1 , ZHU Fangli 1 , ZHOU Yang 2 , Khairy Morsy MOHAMED 3 , YUAN Chao 4 , DONG Xingxing 1 , WANG Xiaohong 1 , LI Jinquan 1,2, *
(1. College of Food Science and Technology, Huazhong Agricultural University, Wuhan 430070, China;2. State Key Laboratory of Agricultural Microbiology, Wuhan 430070, China;3. Department of Food Science, Faculty of Agriculture, Benha University, Benha 11435, Egypt;4. College of Science, Huazhong Agricultural University, Wuhan 430070, China)
Abstract: Foodborne pathogenic bacteria are recognized as one of the major causes of food-borne diseases. With the fast development of modern food industry, higher requirements have been placed on the rapid detection of pathogens in foods.Considered as the most abundant organism on Earth, bacteriophage can infect bacterial hosts naturally. Current research has documented that phage not only has the characteristics of simple structure, high specificity and low price, but also has the ability to distinguish between living and dead bacteria and is easy to combine with traditional methods for detecting bacterial pathogens in foods. Phages and phage-encoded products can provide a new tool for the detection of foodborne pathogens.More recently, many rapid phage-based detection methods have been developed, especially the application of phage in conjunction with immunology, molecular biology and nanoscale science. In this paper, the principles and applications of phage-based methods for the detection of foodborne pathogens are reviewed and analyzed.
Keywords: bacteriophage; foodborne pathogenic bacteria; detection method; genetic engineering
WEI Lin, ZHU Fangli, ZHOU Yang, et al. Progress in the application of bacteriophage in the detection of foodborne pathogenic bacteria[J]. Food Science, 2018, 39(17): 314-322. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201817049. http://www.spkx.net.cn
文章编号: 1002-6630(2018)17-0314-09
引文格式: 魏麟, 朱方莉, 周洋, 等. 噬菌体在检测食源性病原菌中的应用研究进展[J]. 食品科学, 2018, 39(17): 314-322.DOI:10.7506/spkx1002-6630-201817049. http://www.spkx.net.cn
中图分类号: TS252.1
文献标志码: A
*通信作者简介: 李锦铨(1986—),男,副教授,博士,研究方向为食品安全。E-mail:lijinquan@mail.hzau.edu.cn
DOI: 10.7506/spkx1002-6630-201817049
基金项目: 中央高校基本科研业务费专项资金项目(2662017JC040;201710504081);国家自然科学基金青年科学基金项目(31502080);湖北省自然科学基金项目(2015CKC901)
第一作者简介: 魏麟(1995—),女,本科生,研究方向为食品安全。E-mail:377651396@qq.com
(1.华中农业大学食品科学技术学院,湖北 武汉 430070;2.农业微生物学国家重点实验室,湖北 武汉 430070;3.本哈大学农学院食品科学系,埃及 本哈 11435;4.华中农业大学理学院,湖北 武汉 430070)
收稿日期: 2017-06-04
摘 要: 食源性病原菌是导致食源性疾病的主要原因之一,现代食品工业的迅猛发展对食品中病原菌的快速检测提出了更高的要求。噬菌体作为地球上种类最丰富的微生物之一,能够侵染细菌。研究表明,噬菌体不仅具备结构简单、特异性强、价格低廉等特性,而且具有能够区分活细菌和死细菌的能力,以及容易与其他传统检测方法相结合等优势,噬菌体及其产物为食源性病原菌的检测提供了新的思路。近年来,噬菌体与免疫学、分子生物学和纳米科学等学科结合形成的新型快速检测方法已成为国际研究热点。本文就噬菌体检测食源性病原菌的原理及应用进行分类综述和分析。