
图1 梯棱羊肚菌成熟子实体
Fig. 1 Mature fruiting body of Morchella importuna
王金秋,叶鸿亮,梁道崴,刘达玉,耿 放,李 翔*
(成都大学药学与生物工程学院,四川 成都 610106)
摘 要: 对羊肚菌成熟子实体的转录组进行高通量测序,并对得到的unigene进行基因本体论富集分析、真核生物直系同源聚类分析和代谢通路分析。经组装,共得到14 637 条unigene,其中8 495 个unigene得到了功能注释;利用基因本体论富集分析、真核生物直系同源功能分类将注释unigene划分分别为58 个和25 个功能类别,涉及物质合成与代谢、呼吸与能量代谢、信号传导等诸多生理生化过程。在此基础之上,通过对羊肚菌采后生理涉及的unigene进行深入分析,发现大量参与呼吸代谢、碳水化合物代谢、组织软化、氧化褐变等相关途径的关键基因。研究结果为羊肚菌采后品质变化机制的研究、贮藏保鲜技术的开发提供了一定的理论基础。
关键词: 羊肚菌;转录组测序;功能注释;营养代谢;贮藏保鲜
羊肚菌(Morchella)隶属于盘菌目(Pezizales)羊肚菌科(Morohellaceae) [1] ,是一种全世界广泛分布的药食两用真菌 [2] 。羊肚菌子实体质地脆嫩,广受消费者欢迎 [3-4] ,其氨基酸含量丰富、种类齐全,是优质的植物蛋白资源,高比例鲜味氨基酸更是赋予其鲜美的滋味 [5-6] 。羊肚菌中不饱和脂肪酸比例高,以亚油酸为主,对人体健康具有积极作用 [7-8] 。羊肚菌中的碳水化合物主要为膳食纤维、甘露醇、海藻糖等,具有抗氧化、抗肿瘤、调控血糖、提高免疫力等功效 [9-11] 。此外,其他生物活性物质如甾醇、酚类等也具有多种功效 [12-13] 。
长期以来,羊肚菌都以野生栽培为主,因此产量较少、价格昂贵。近年来,羊肚菌人工栽培得已实现,性状稳定,产量逐年上升,产业快速发展 [14-16] 。但新鲜羊肚菌子实体质地脆嫩,不耐贮藏,且采摘后的呼吸活动剧烈,营养物质消耗快,易出现萎蔫、褐化、腐败等现象 [17] ,其采后贮藏保鲜问题限制了产业的发展。因此,通过遗传选育、采后分子调控等措施延长羊肚菌保藏时间,将具有重要意义。然而,目前羊肚菌尚鲜见高质量的遗传信息数据,限制了相关研究的开展。
转录组测序是一种高通量获取遗传信息的技术手段,已广泛应用于食用菌的研究中 [18-27] 。本研究利用高通量测序技术,以四川地区广泛的栽培种梯棱羊肚菌(Morchella importuna)为材料,对羊肚菌成熟子实体转录组进行深度测序,通过从头组装拼接获得unigene,并对其功能进行注释和分类;在此基础上,对影响羊肚菌采后食用品质的相关代谢路径中关键基因进行挖掘,以期为羊肚菌贮藏保鲜技术的开发提供实验参考。
梯棱羊肚菌(Morchella importuna)成熟子实体采自于四川省成都市金堂县(图1)。采摘后挑选新鲜、完整、无腐烂病害的子实体5 株,每株沿子实体轴线纵切,取约2 mm厚的组织,立即液氮速冻。

图1 梯棱羊肚菌成熟子实体
Fig. 1 Mature fruiting body of Morchella importuna
总RNA提取试剂盒(TRIzol ® Reagent)、Dynabeads TM mRNA纯化试剂盒 美国Thermo Fisher Scientific公司;文库建立试剂盒(Truseq TM RNA Sample Prep Kit)、桥式扩增试剂盒(HiSeq 4000 PE Cluster Kit)美国Illumina公司;文库回收试剂(Certified Low Range Ultra Agarose) 美国Bio-Rad公司。
DYY-12型电泳仪、DYCZ-24DN型水平电泳槽北京六一仪器厂;3-30K离心机 德国Sigma Laborzentrifugen公司;磁力架、NanoDrop 2000型紫外-可见分光光度计 美国Thermo Fisher Scientific公司;涡旋仪 上海安亭科学仪器厂。
1.3.1 羊肚菌子实体总RNA提取
将5 株羊肚菌子实体样品混合后研磨,使用总RNA提取试剂盒(TRIzol ® Reagent)按照说明书提取总RNA,利用紫外光吸收法检测RNA的浓度和纯度,琼脂糖凝胶电泳检测RNA完整性。
1.3.2 羊肚菌子实体RNA测序
向得到的总RNA中加入磁珠,通过碱基配对,进一步富集mRNA。随后加入片段化缓冲液,将mRNA随机断裂成200 bp左右的小片段。以这些小片段为模板,利用随机引物和逆转录酶合成cDNA第1链,随后进行cDNA第2链合成,得到黏性末端的双链cDNA。随后加入末端修复试剂,将其补成平末端,并在3’末端加上一个A碱基并连接测序接头。分别利用文库建立试剂盒和桥式扩增试剂盒进行cDNA文库构建和扩增,然后进行上机测序(HiSeq 4000测序系统)。本研究中的cDNA构建和测序服务由上海美吉生物医药科技有限公司提供。
1.3.3 转录组测序数据处理
利用SeqPrep(https://github.com/jstjohn/SeqPrep)软件,对以上得到的原始读段进行过滤,去除接头序列、低质量的末端序列(质量值小于20)和含N比例超过10%的序列,得到高质量的有效读段。羊肚菌无参考基因组,因此利用Trinity(http://trinityrnaseq.sourceforge.net/)软件将上述有效读段进行从头组装,得到高质量的转录本,取最长的转录本作为该样品的unigene进行后续分析。
1.3.4 unigene功能注释
得到1.3.3节序列后,利用Trinity软件提供的基于开放阅读框的预测流程,对组装得到的所有转录本序列进行基因预测,得到unigene。利用unigene序列信息分别与Nr、String、Swissprot、KEGG和Pfam数据库进行比对,获得unigene的注释信息。根据与String数据库比对结果,进行真核生物直系同源(euKaryotic of orthologous groups,KOG)功能归类。通过BLAST2GO(http://www.blast2go.com/b2ghome)对unigene进行基因本体论(gene ontology,GO)注释,并利用WEGO软件进行GO分类。在上述注释分析过程中,以期望值小于10 -5 为成功注释的阈值。通过HMMER3程序,以上述得到的unigene对应的蛋白序列,比对蛋白结构域数据库和dbCAN数据库(http://csbl.bmb.uga.edu/dbCAN/07/15/2016),进行蛋白家族的注释和相关基因功能的挖掘。
通过对梯棱羊肚菌成熟子实体样品转录组进行测序,共获得66 562 198 条原始读段,过滤掉接头、低质量和含N比例超过10%的序列后,得到63 939 094 条有效读段,总测序碱基数为9 282 996 323。碱基质量值(Q30,99.9%碱基正确率)为91.21%,表明测序数据质量较高,可满足后续分析要求(表1)。
表1 梯棱羊肚菌转录组测序质量检测统计
Table 1 Statistics and quality estimation of M. importuna transcriptome
表2 转录本和unigenes从头组装结果统计表
Table 2 Summary of de novo assembly of transcripts and unigenes
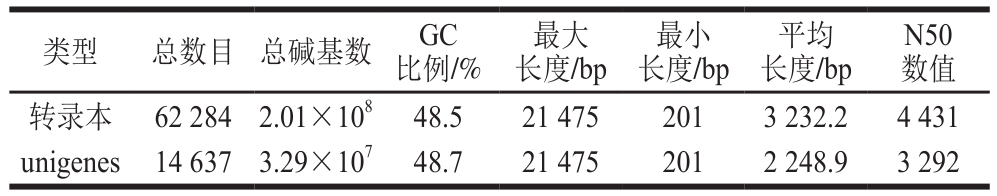
羊肚菌子实体目前无参考基因组,利用Trinity软件对转录本进行从头组装拼接,将所得有效读段进行从头拼接和去冗余,共生成62 284 条转录本,平均长度为3 232.2 bp。取最长的转录本作为unigene,得到14 637 条unigene,平均长度为2 248.9 bp(表2)。羊肚菌unigenes的转录本组装居中长度(N50)数值为3 292,远长于多种已测序食用菌,如毛木耳 [19] 、茶树菇 [20] 、茯苓 [21] 、香菇 [22-23] 、灵芝 [24] 、裂蹄木层孔菌 [25] 和白灵菇 [26] 。N50数值是无参考序列评估测度的重要指标,一定程度上反映拼接所得序列的完整性。物种差异、测序平台和拼接算法都可能会影响该值。在先前的食用菌转录组测序研究中,多采用Illumina HiSeq 2000或者454测序平台,本研究所采用的测序平台为较新的Illumina HiSeq 2500平台,读长可达150 bp。因此,在保证较高测序质量(Q30>90%)的前提下,可得到更长的测序长度和更高的覆盖度。
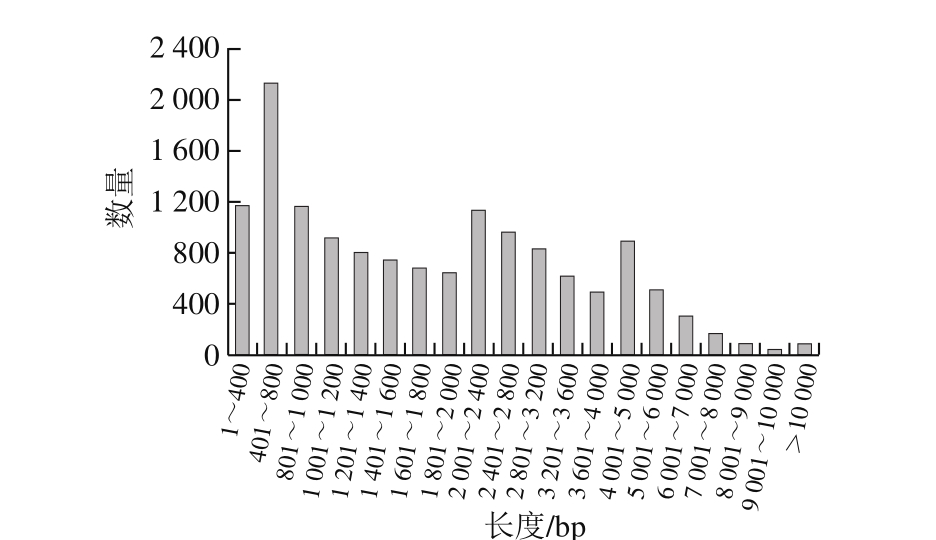
图2 梯棱羊肚菌unigene长度分布图
Fig. 2 Unigene length distribution profile of Morchella importuna
进一步分析显示,羊肚菌unigene长度范围在201~21 475 bp之间,长度在1~400、401~800、801~1 000、2 001~2 400 bp和2 401~2 800 bp之间的unigene数目最多(图2)。以上数据表明,本研究中转录组组装质量较高,为后续羊肚菌基因功能的分析提供了较为全面和可靠的序列信息。
通过BLAST程序将unigene序列分别与Nr、String、Swissprot、KEGG和Pfam数据库进行比对,各数据库分别注释到8 340、2 528、5 693、3 856、5 537 个unigene。有功能或代谢通路注释的unigene总数为8 495 个,占全部unigene的58%,其中,1 545 个unigene在所有的数据库中都被注释,占总数的10.6%(图3)。与大多数已经测序的其他食用菌相比,羊肚菌转录组中得到注释的unigene比例较低 [19-27] 。这可能由于羊肚菌属于子囊菌亚门,而目前已测序和注释的大多食用菌属于担子菌亚门,亲缘关系相对较远,参考信息较少。
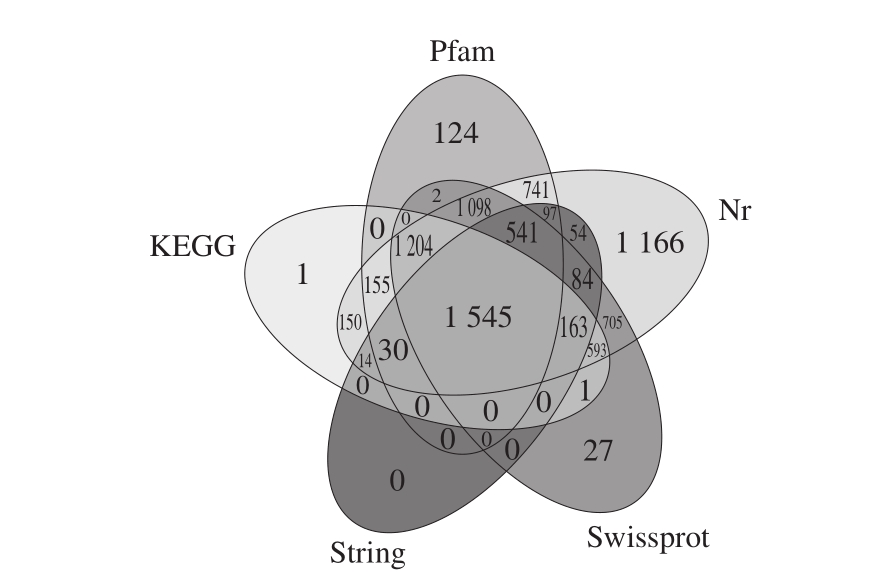
图3 各数据库注释到的unigene统计图
Fig. 3 Statistical results of annotated unigenes in various databases
2.3.1 同源物种比对分析
Nr数据库比对结果可以查看物种同源度情况,以及unigenes的功能信息。近缘物种比对结果表明,羊肚菌子实体unigene与黑松露(Tuber melanosporum)相似序列最多,为3 286 个,占据全部unigenes的39.3%,表明两者同源度较高。其次为烧土火丝菌(Pyronema omphalodes),相似序列为1 202 个,占14.4%。以上2 个物种均属于盘菌目,表明在已有遗传数据的食用菌中,羊肚菌与之的亲缘关系相对较近。
2.3.2 GO功能分类
GO数据库对关于基因和基因产物的生物学术语进行标准化,通过该数据库的比对,可对unigene的功能和分类进行统一的描述。分析结果显示,羊肚菌成熟子实体中共有3 994 个unigene得到GO功能注释,归类于生物学过程、细胞组分和分子功能(图4),较全面地对注释基因的功能进行了分类。生物学过程类别中,参与代谢过程、细胞内过程和单组织过程的unigene所占比例最高,分别为66.9%(2 671 个)、59.8%(2 387 个)和47.9 %(1 912 个)。细胞组分类别中,参与细胞、细胞部分和细胞器构成的unigene数量较多,前两者均为43.8%(1 749 个),后者为32.4%(1 295 个)。分子功能类别中,催化活性、结合活性和转运活性的基因最多,分别占47.2%(2 153 个)、54.0%(1 884 个)和6.4%(256 个)。梯棱羊肚菌中的主要生物学过程与其他食用菌如毛木耳 [19] 、茶树菇 [20] 、香菇 [22] 等相似,表明不同食用菌子实体的主要生理过程具有一定的相似性。
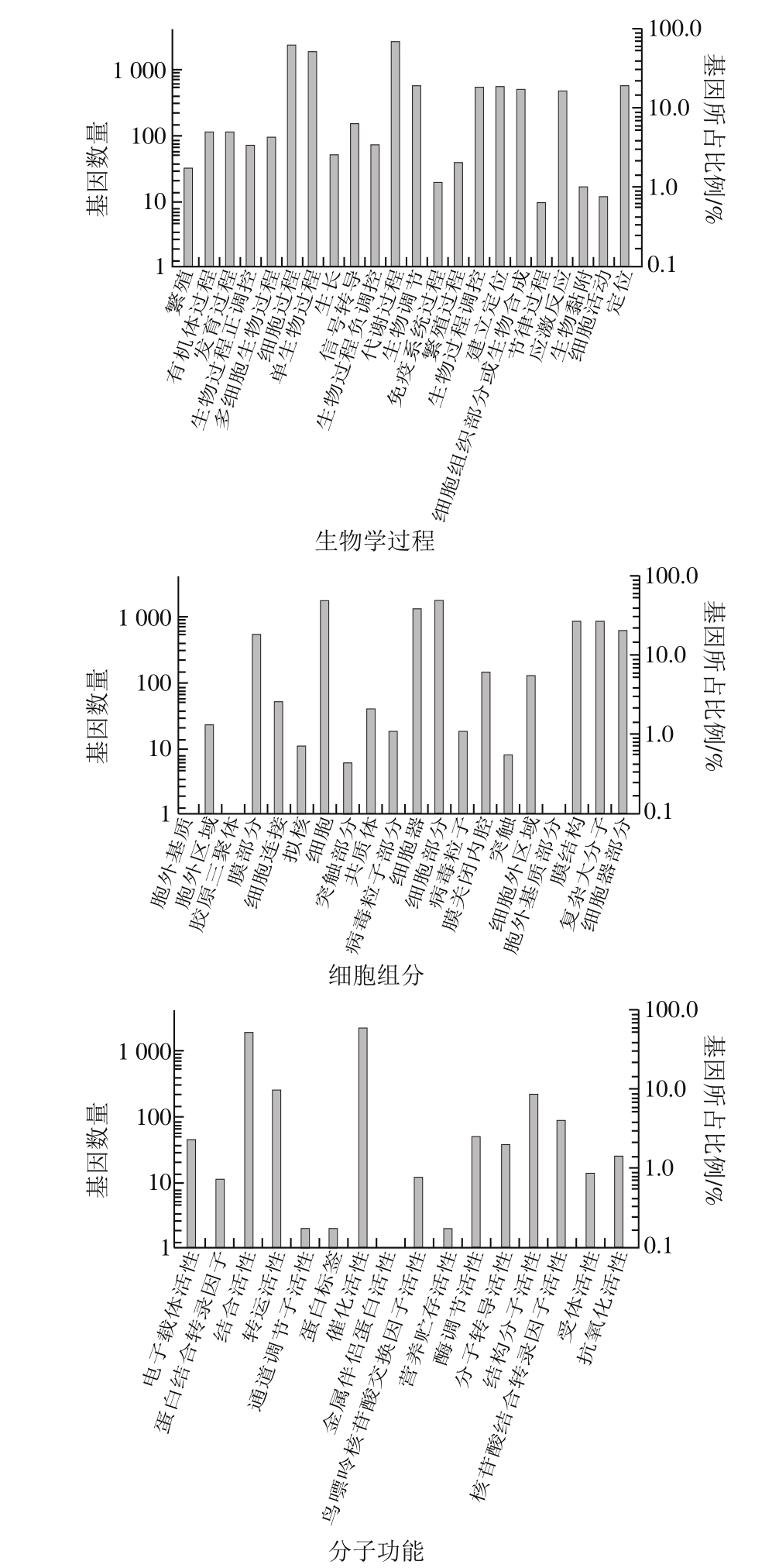
图4 梯棱羊肚菌unigene的GO功能分类
Fig. 4 GO classification of Morchella importuna unigenes
2.3.3 KOG功能分类
KOG数据库为蛋白直系同源簇数据库,是基于蛋白质序列的进化关系构建而成。该数据库的比对结果显示,羊肚菌子实体转录组中,共匹配上2 152 个unigene,根据蛋白质功能划分,归属于25 个家族(图5)。其中,最主要的5 类依次为:翻译、核糖体结构与合成(J,250 个,11.6%),翻译后修饰、蛋白质折叠和分子伴侣(O,218 个,10.1%),一般功能预测(R,168 个,7.8%),信号转导机制(T,153 个,7.1%),能量生产和转化(C,152 个,7.1%)。另外,与营养物质代谢路径相关的unigene较多,如碳水化合物转运和代谢、氨基酸转运和代谢、脂质转运和代谢,分别为116、112 个和88 个。
GO和KOG功能分类分析结果表明,羊肚菌成熟子实体中具有旺盛的生理活动,包括信号转导、转录调控、能量代谢、营养物质代谢和转运等。结合数据库相关同源基因的注释信息,可为羊肚菌采后生理生化过程、相关生物活性物质的合成等提供重要的信息,为研究羊肚菌采后品质变化规律和贮藏保鲜技术提供参考。
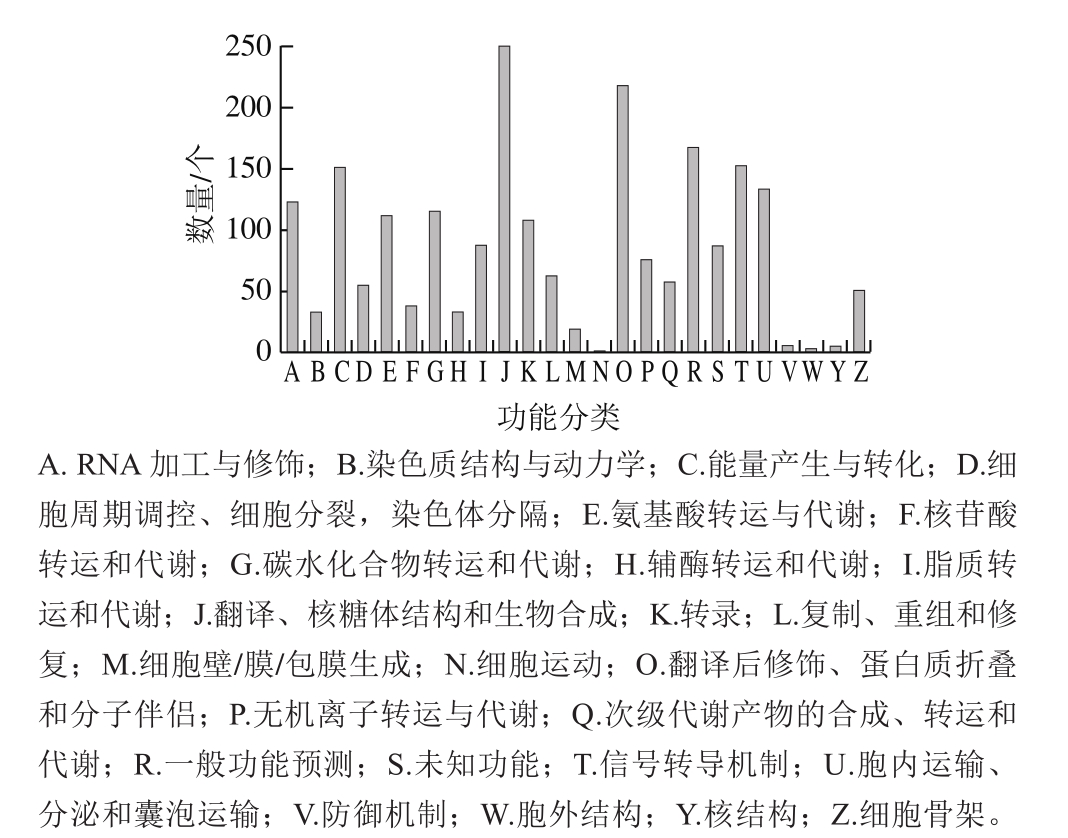
图5 梯棱羊肚菌unigenes的KOG功能分类
Fig. 5 KOG classification of Morchella importuna unigenes
2.3.4 KEGG通路注释

图6 梯棱羊肚菌unigenes的Top 10 KEGG代谢通路
Fig. 6 Top 10 KEGG categories of Morchella importuna unigenes
为了分析羊肚菌unigene参与的代谢路径,进一步通过KEGG通路注释分析,共注释到了3 856 个unigene,分布于376 个代谢通路。其中,羊肚菌unigene参与最多的10 个代谢通路依次为:核糖体(199 个)、碳代谢(174 个)、氨基酸合成(152 个)、内吞作用(120 个)、RNA运输(106 个)、肌动蛋白细胞骨架调节(96 个)、嘌呤代谢(95 个)、淀粉和蔗糖代谢(91 个)、细胞周期(90 个)、剪切体(90 个)(图6)。此外,在能量代谢、糖异生/糖酵解、脂质代谢、辅助因子和维生素代谢等通路中也富集了较多的unigene。这些结果进一步表明,羊肚菌成熟子实体具有旺盛的营养物质代谢和能量代谢,预示着在采后贮藏过程中,羊肚菌子实体营养组成和食用品质可能会发生较大的变化。此外,羊肚菌unigene参与的主要代谢路径与其他食用菌,如灰树花 [18] 、茶树菇 [20] 、灵芝 [24] 等存在一定的差异,这可能是物种差异、发育阶段等因素引起的。
在采后贮藏过程中,呼吸代谢与营养风味成分变化、多糖代谢与组织软化、氧化还原酶系与褐变等对羊肚菌子实体的食用品质具有重要影响,因此,本研究对这些过程中涉及到的关键基因进行挖掘和分析。
2.4.1 呼吸作用相关基因分析
呼吸作用主要包括磷酸戊糖途径、糖酵解、三羧酸(tricarboxylic acid,TCA)循环及氧化磷酸化等途径,这些途径的关键酶决定了呼吸作用的强度,且是各类营养素(糖类、脂类、蛋白质、核苷酸、维生素)代谢通路相互联系的枢纽,亦是产生滋味和风味物质的重要途径。
糖酵解是生物体内葡萄糖代谢所必须经过的共同阶段。在羊肚菌成熟子实体转录组中,共鉴定到了79 个参与糖酵解的unigene,涵盖了糖酵解几乎所有关键酶,涉及该过程中的31 个催化反应。TCA循环是糖类、脂类和氨基酸的最终代谢通路,亦是产生风味物质和有机酸等的主要产生途径,与羊肚菌子实体的风味和滋味密切相关。在羊肚菌转录组中,共发现了30 个unigene参与TCA循环,包括该途径中的所有关键酶,涉及16 个催化反应。磷酸戊糖途径是葡萄糖氧化降解的另一途径,羊肚菌转录组中共鉴定到了24 个unigene参与该途径,涉及12 个催化反应,包括了氧化阶段和非氧化阶段的几乎所有关键酶。氧化磷酸化发生在真核生物的线粒体中,是通过呼吸链将能量转化为腺嘌呤核苷三磷酸(ATP)的过程。在羊肚菌的转录组中,共发现79 个unigene参与该代谢路径,其编码的蛋白质是羊肚菌呼吸链的重要组成。以上结果表明,羊肚菌呼吸代谢所需的关键代谢酶齐全,在遗传水平阐释了羊肚菌子实体呼吸代谢旺盛的原因。
2.4.2 碳水化合物活性酶与羊肚菌子实体的质地及多糖合成
碳水化合物活性酶(carbohydrate active enzyme,CAZymes)是参与寡聚糖或多聚糖合成、水解和修饰的一类酶。CAZymes包括糖苷水解酶(glycosyl hydrolase,GH)、多糖裂解酶(pectate lyase,PL)、碳水化合物酯酶(carbohydrate esterase,CE)、辅助酶类(auxiliary activitie,AA)和糖苷转移酶(glycosyl transferase,GT)。
羊肚菌子实体质地脆嫩是其口感特点之一,但在采后贮藏过程中,子实体发生明显的软化现象,导致其食用品质下降。软化主要与子实体细胞壁的降解相关,主要涉及葡聚糖、几丁质、纤维素、半纤维素、果胶等的降解。因此,相关基因的挖掘对羊肚菌采后质地的保持具有重要意义。GH可催化糖苷键水解,其主要底物为纤维素、半纤维素和果胶。在羊肚菌子实体转录组中,共有157 个unigene注释到GH家族,分布于41 个亚家族;其中,GH61和GH5亚家族最多,分别为19 个和16 个,这两个亚家族均主要为纤维素降解酶,在其他食用菌中的丰度也较高;其次为GH3(12 个)和GH43(11 个)亚家族成员,主要编码纤维素酶和半纤维素酶。CE催化糖酯键水解,羊肚菌子实体转录组中共有36 个unigene注释到CE,其中,CE1亚家族最多(20 个),占据了整个家族的55.6%,其编码的酶类主要为纤维素酶和半纤维素酶。PL催化裂解含有糖醛酸的多聚糖链,共有22 个unigene注释到PL,其中最多的是PL1亚家族(8 个),其次为PL3亚家族(7 个),这些基因编码的酶类主要为果胶酶。AA主要包括木质素降解酶和溶解性多糖单加氧酶,羊肚菌转录组中有43 个unigene注释到AA,其中AA1亚家族18 个,AA3和AA5分别为15、10 个。
对上述催化多糖水解的CAZymes按照底物进行分类,主要为几丁质酶、葡聚糖酶、果胶酶、纤维素酶等。其中,几丁质相关降解酶总计14 个,包括10 个几丁质酶、2 个内切壳聚糖酶、1 个几丁质脱乙酰酶和1 个β-N-乙酰葡萄糖胺糖苷酶。葡聚糖酶总计7 个,分别为6 个β-1,3-葡聚糖酶和1 个β-1,6-葡聚糖酶。果胶降解相关酶总计26 个,主要包括:3 个多聚半乳糖醛酸酶、2 个果胶甲酯酶、12 个果胶裂解酶、6 个鼠李半乳糖醛酸聚糖酶、2 个不饱和鼠李半乳糖醛酸聚糖水解酶和1 个鼠李半乳糖醛酸聚糖乙酰酯酶。此外,共鉴定到了33 个纤维素酶和5 个纤维二糖水解酶。这些酶的底物包括了羊肚菌子实体中的主要多糖类型,在碳源吸收与利用、子实体形态发育、采后组织软化等过程中发挥着重要作用。
此外,单糖是碳水化合物降解的末端产物,亦是多糖合成的基本单元,主要包括木糖、甘露糖、阿拉伯糖、葡萄糖和半乳糖等。单糖的不同组合形成不同的糖苷键,需要不同的水解酶,因此半纤维素水解酶种类丰富。羊肚菌转录组中,鉴定到的木糖降解相关酶最多,为6 类38 个,主要包括:13 个木糖聚合物水解酶、3 个木聚糖酶、3 个乙酰木聚糖酯酶、2 个α-木糖苷酶等。甘露聚糖相关降解酶种类也较多,共4 类11 个,具体为5 个β-1,4-甘露聚糖酶切酶、3 个β-1,6-甘露聚糖酶、2 个甘露聚糖酶和1 个甘露糖基寡糖类葡糖苷酶。阿拉伯聚糖的相关降解酶鉴定到了5 类11 个,分别为4 个α-L-阿拉伯葡聚糖酶、3 个α-1,5-L-阿拉伯聚糖内切酶、2 个α-N-阿拉伯葡聚糖酶、1 个阿拉伯糖苷酶和1 个β-L-阿拉伯葡聚糖酶。此外,鉴定到了3 个以半乳糖聚合物为底物的降解酶,分别为2 个外切β-1,3-半乳聚糖酶和1 个内切β-1,4-半乳聚糖酶。除了以上种类的半纤维素酶,还鉴定到了1 个细胞壁酸性海藻糖酶。
与降解相对应,GT催化糖基转移,主要参与多糖合成。羊肚菌多糖具有抗氧化、调节免疫等多种功效,是其影响羊肚菌食用品质和功能活性的重要组分。羊肚菌转录组中注释到GT的unigene共有51 个,分布在9 个亚家族中,其中GT2亚家族成员最多,为13 个。发现的这些GT酶对调控羊肚菌多糖合成具有重要意义。
2.4.3 酶促氧化褐变基因分析
酶促氧化褐变是羊肚菌采后贮藏及加工过程中影响其食用品质和商品价值的另一重要因素。目前,羊肚菌的酶促褐变机制还不明确,本研究对褐变相关的unigene进行了分析和挖掘。先前的研究表明,影响褐变的酶主要有多酚氧化酶(polyphenol oxidase,PPO)、过氧化物酶(peroxidase,POD)和脂氧合酶(lipoxygenase,LOX) [28-29] 。同时,活性氧清除酶类对褐变具有抑制作用,主要包括过氧化氢酶(catalase,CAT)和超氧化酶歧化酶(superoxide dismutase,SOD)。
PPO是引起其酶促褐变的主要酶类,其催化酚类物质氧化成醌,进而生成黑色素导致褐变。在微生物中,PPO主要包括酪氨酸酶、漆酶 [30] 。本研究在羊肚菌转录组unigene中鉴定出了7 个酪氨酸酶和3 个漆酶。POD通过催化酚类物质的氧化和聚合,导致褐变 [29] ,羊肚菌转录组中共鉴定到了11 个POD和2 个谷胱甘肽过氧化物酶。LOX在特定条件下,能氧化膜脂进而破坏细胞膜结构,使得PPO与酚类物质接触而发生酶促褐变,羊肚菌转录组中该类酶鉴定到了2 个,分别是亚油酸脂氧合酶和亚油酸双加氧酶。上述氧化酶类可能是导致羊肚菌子实体采后酶促氧化褐变的关键酶。
与数量较多的氧化褐变酶类相比,活性氧清除酶类数量较少,SOD和CAT仅分别鉴定到了7 个和5 个。以上结果表明,羊肚菌成熟子实体中PPO和POD种类和数量均较多,为后期深入研究羊肚菌的酶促褐变机制提供了分子依据。
本研究采用Illumina高通量测序技术对羊肚菌成熟子实体的转录组进行深度测序,并对数据进行组装、注释和分析。结果显示,羊肚菌转录组包含14 637 条unigene,其中8 495 条unigene得到注释。注释基因的GO和KOG功能分类、以及KEGG通路注释结果,为系统和深入理解羊肚菌主要生理生化过程提供了重要线索。此外,分析和挖掘了与呼吸作用、碳水化合物合成与代谢、多酚氧化还原等相关的unigene,这些基因所编码的酶类在羊肚菌营养成分合成与代谢、风味与滋味物质产生、多糖代谢与组织软化、酶促氧化褐变等过程中具有重要作用。该研究为探究羊肚菌生理、生化过程相关基因功能及调控等提供了重要信息,亦为深入研究羊肚菌遗传育种、栽培技术、采后生理、贮藏保鲜等相关技术提供了基础。
参考文献:
[1] 刘伟, 张亚, 何培新. 羊肚菌生物学与栽培技术[M]. 长春: 吉林科学技术出版社, 2017: 28-29.
[2] 刘伟, 蔡英丽, 何培新. 粗柄羊肚菌转录组的SSR分布和序列特征分析[J]. 轻工学报, 2017, 32(2): 33-39. DOI:10.3969/j.issn.2096-1553.2017.2.006.
[3] 才晓玲, 何伟, 安福全, 等. 羊肚菌生物活性研究进展[J]. 中国食用菌, 2013, 32(5): 7-8. DOI:10.13629/j.cnki.53-1054.2013.05.008.
[4] TIETEL Z, MASAPHY S. True morels (Morchella)-nutritional and phytochemical composition, health benefits and flavor: a review[J].Critical Reviews in Food Science & Nutrition, 2017, 58(11): 1-14.DOI:10.1080/10408398.2017.1285269.
[5] TSAI S Y, WENG C C, HUANG S J, et al. Nonvolatile taste components of Grifolafrondosa, Morchella esculenta and Termitomyces albuminosus mycelia[J]. LWT-Food Science and Technology, 2006, 39(10): 1066-1071. DOI:10.1016/j.lwt.2005.07.017.
[6] ROTZOLL N, DUNKEL A, HOFMANN T. Quantitative studies, taste reconstitution and omission experiments on the key taste compounds in morel mushrooms (Morchella deliciosa Fr.)[J]. Journal of Agricultural &Food Chemistry, 2006, 54(7): 2705-2711. DOI:10.1021/jf053131y.
[7] HELENO S A, STOJKOVIĆ D, BARROS L, et al. A comparative study of chemical composition, antioxidant and antimicrobial properties of Morchella esculenta (L.) Pers. from Portugal and Serbia[J]. Food Research International, 2013, 51(1): 236-243.DOI:10.1016/j.foodres.2012.12.020.
[8] VIEIRA V, FERNANDES Â, BARROS L, et al. Wild Morchella conica Pers. from different origins: a comparative study of nutritional and bioactive properties[J]. Journal of the Science of Food &Agriculture, 2016, 96(1): 90-98. DOI:10.1002/jsfa.7063.
[9] 明建. 云南特有食药用菌活性多糖研究[D]. 重庆: 西南大学, 2008:92-95.
[10] 黄玲玲. 羊肚菌多糖的提取及体外抗氧化活性的研究[D]. 大连: 大连工业大学, 2015: 3-6
[11] YAO L, YUAN Y, LIN L, et al. Carboxymethylation of polysaccharide from Morchella angusticepes peck enhances its cholesterol-lowering activity in rats[J]. Carbohydrate Polymers, 2017, 172: 85-92.DOI:10.1016/j.carbpol.2017.05.033.
[12] GURSOY N, SARIKURKCU C, CENGIZ M, et al. Antioxidant activities, metal contents, total phenolics and flavonoids of seven Morchella species[J]. Food & Chemical Toxicology, 2009, 47(9):2381-2388. DOI:10.1016/j.fct.2009.06.032.
[13] KIM J A, LAU E. Antioxidant and NF-κB inhibitory constituents isolated from Morchella esculenta[J]. Natural Product Research, 2011,25(15): 1412-1417. DOI:10.1080/14786410802425746.
[14] 朱斗锡. 羊肚菌人工栽培研究进展[J]. 中国食用菌, 2008, 27(4): 3-5.DOI:10.3969/j.issn.1003-8310.2008.04.001.
[15] 谭方河. 羊肚菌人工栽培技术的历史、现状及前景[J]. 食药用菌,2016(3): 140-144.
[16] 赵琪, 康平德, 戚淑威, 等. 羊肚菌资源现状及可持续利用对策[J]. 西南农业学报, 2010, 23(1): 266-269. DOI:10.3969/j.issn.1001-4829.2010.01.061.
[17] GARCIAPASCUAL P, SANJUAN N, MELIS R, et al. Morchella esculenta (morel) rehydration process modelling[J]. Journal of Food Engineering, 2006, 72(4): 346-353. DOI:10.1016/j.jfoodeng.2004.12.014.
[18] 聂文强, 吴天祥, 钟敏, 等. 真菌灰树花菌丝体转录组测序及分析[J].食品科学, 2017, 38(20): 6-11. DOI:10.7506/spkx1002-6630-201720002.
[19] ZHOU Y, CHEN L, FAN X, et al. De novo assembly of Auricularia polytricha transcriptome using illumina sequencing for gene discovery and SSR marker identification[J]. PLoS ONE, 2014, 9(3): e91740.DOI:10.1371/journal.pone.0091740.
[20] WANG M, GU B, HUANG J, et al. Transcriptome and proteome exploration to provide a resource for the study of Agrocybe aegerita[J].PLoS ONE, 2013, 8(2): e56686. DOI:10.1371/journal.pone.0056686.
[21] SHU S, CHEN B, ZHOU M, et al. De novo sequencing and transcriptome analysis of Wolfiporia cocos to reveal genes related to biosynthesis of triterpenoids[J]. PLoS ONE, 2013, 8(8): e71350.DOI:10.1371/journal.pone.0071350.
[22] 易琳琳. 香菇转录组测序及细胞壁降解相关基因筛选的研究[D].杭州: 浙江大学, 2015: 9-24.
[23] ZHONG M, LIU B, WANG X, et al. De novo characterization of Lentinula edodes C 91-3 transcriptome by deep Solexa sequencing[J].Biochemical & Biophysical Research Communications, 2013, 431(1):111-115. DOI:10.1016/j.bbrc.2012.12.065.
[24] YU G J, WANG M, HUANG J, et al. Deep insight into the Ganoderma lucidum by comprehensive analysis of its transcriptome[J]. PLoS ONE, 2012, 7(8): e44031. DOI:10.1371/journal.pone.0044031.
[25] HUANG Y, WU X, DUAN J, et al. De novo transcriptome analysis of a medicinal fungi Phellinus linteus and identification of SSR markers[J].Biotechnology & Biotechnological Equipment, 2015, 29(2): 395-403.DOI:10.1080/13102818.2015.1008228.
[26] FU Y, DAI Y, YANG C, et al. Comparative transcriptome analysis identified candidate genes related to bailinggu mushroom formation and genetic markers for genetic analyses and breeding[J]. Scientific Reports, 2017, 7(1): 9266. DOI:10.1038/s41598-017-08049-z.
[27] YANG F, XU B, ZHAO S, et al. De novo sequencing and analysis of the termite mushroom (Termitomyces albuminosus) transcriptome to discover putative genes involved in bioactive component biosynthesis[J]. Journal of Bioscience & Bioengineering, 2012, 114(2):228-231. DOI:10.1016/j.jbiosc.2012.03.009.
[28] 张丽. 黑脉羊肚菌中PPO、POD、SOD三种酶的性质研究[D].昆明: 昆明理工大学, 2014: 8-14.
[29] 刘吟. 双孢蘑菇采后褐变的相关生理生化变化及其保鲜技术研究[D].武汉: 华中农业大学, 2010: 1-3.
[30] 吴瑜凡, 卢亚萍, 汪瑾. 植物和真菌多酚氧化酶的分子生物学研究进展[J]. 现代农业科技, 2008(18): 14-17. DOI:10.3969/j.issn.1007-5739.2008.18.005.
De Novo Sequencing and Transcriptome Analysis of Morchella importuna Fruiting Body
WANG Jinqiu, YE Hongliang, LIANG Daowei, LIU Dayu, GENG Fang, LI Xiang*
(College of Pharmacy and Biological Engineering, Chengdu University, Chengdu 610106, China)
Abstract: In the present work, high throughput de novo sequencing of the Morchella importuna transcriptome was carried out, and the annotated sequences were classified using gene ontology (GO), eukaryotic orthologous groups (KOG), and Kyoto Encyclopedia of Genes and Genomes (KEGG) metabolic pathways. After assembly, a total of 14 637 unigenes were formed, 8 495 of which were annotated. By functional classification using GO and KOG, the annotated unigenes were divides into 58 and 25 categories, respectively, involved in many physiological and biochemical processes including synthesis and metabolism of biomolecules, respiration and energy metabolism and signal transduction. Furthermore, a large number of key genes involved in energy metabolism, the synthesis and hydrolysis of nutrients, the softening of texture,oxidation of polyphenols and browning were identified through analysis of the unigenes related to postharvest physiology.The data obtained in this study can provide important information for the understanding of the molecular mechanism of postharvest quality changes in Morchella importuna and for the development of new preservation technologies.
Keywords: Morchella; transcriptome sequencing; functional annotation; nutritional metabolism; storage and preservation
WANG Jinqiu, YE Hongliang, LIANG Daowei, et al. De novo sequencing and transcriptome analysis of Morchella importuna fruiting body[J]. Food Science, 2018, 39(18): 81-87. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201818013. http://www.spkx.net.cn
王金秋, 叶鸿亮, 梁道崴, 等. 梯棱羊肚菌子实体转录组测序及解析[J]. 食品科学, 2018, 39(18): 81-87. DOI:10.7506/spkx1002-6630-201818013. http://www.spkx.net.cn
文章编号: 1002-6630(2018)18-0081-07
引文格式:
中图分类号: TS255.2
文献标志码: A
*通信作者简介: 李翔(1974—),女,教授,博士,研究方向为食品加工与安全。E-mail:lixiang@cdu.edu.cn
DOI: 10.7506/spkx1002-6630-201818013
基金项目: 国家自然科学基金青年科学基金项目(31701655);四川省教育厅重点项目(17ZA0080);四川省教育厅创新团队项目(16TD0036)
第一作者简介: 王金秋(1986—),女,讲师,博士,研究方向为农产品加工与贮藏。E-mail:shiyuejq023@163.com
收稿日期: 2017-10-16