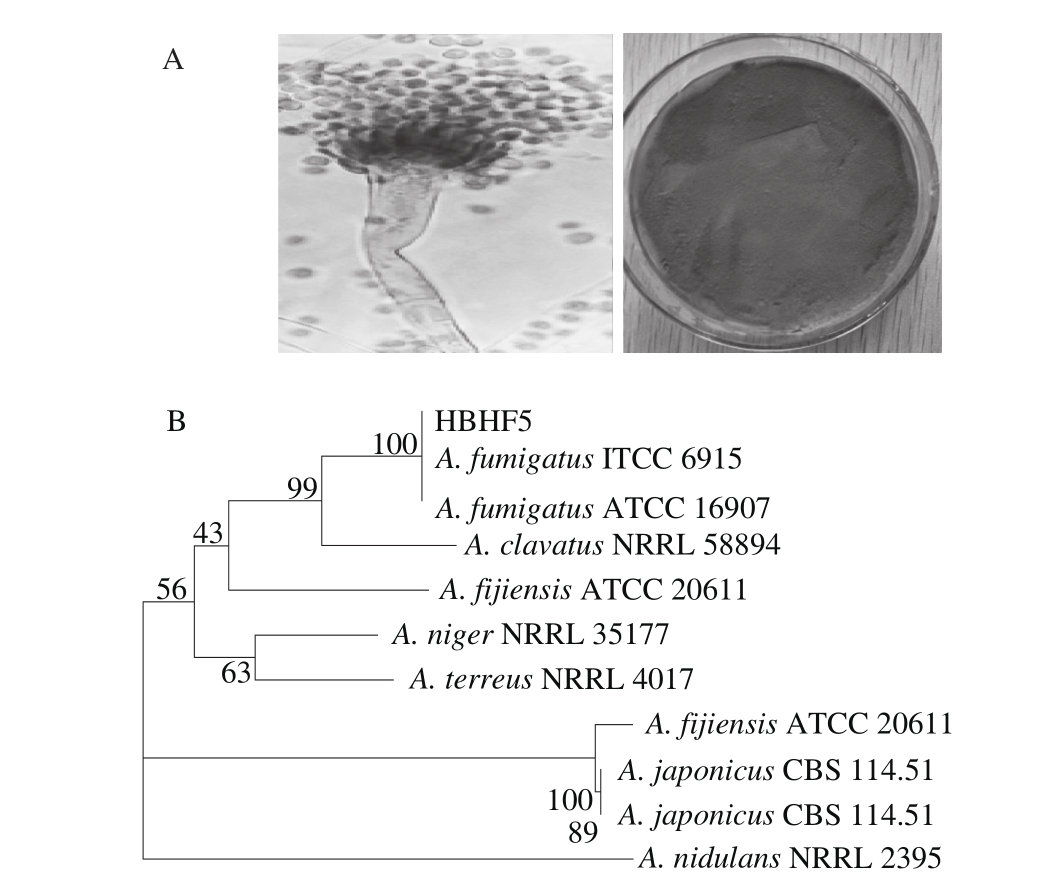
图1 HBHF5菌株菌落特征(A)及系统发育树(B)
Fig. 1 Cultural characteristics (A) and phylogenetic tree (B) of strain HBHF5
谷新晰 1,2 ,卢海强 1 ,刘亚娟 3 ,陈赛娟 3 ,谷子林 4 ,孟祥晨 2 ,陈宝江 4, *
(1.河北农业大学食品科技学院,河北 保定 071000;2.东北农业大学 乳品科学教育部重点实验室,黑龙江 哈尔滨 150030;3.河北农业大学山区研究所,河北 保定 071000;4.河北农业大学动物科技学院,河北 保定 071000)
摘 要: 对产单宁酶的1 株嗜热真菌HBHF5进行鉴定,开展单宁酶酶学性质的分析及碳水化合物活性酶(carbohydrate-active enzymes,CAZymes)的转录组学研究,探究嗜热真菌HBHF5在食品酶制剂开发中的潜力。经对菌株的菌落、孢子形态观察及ITS序列比对分析,最终鉴定嗜热真菌HBHF5为烟曲霉(Aspergillus fumigatus)。经分析,菌株HBHF5在固态发酵培养时不产单宁酶,而液态诱导培养时,菌株HBHF5胞内和胞外均检测到单宁酶活性,且以胞外酶为主(94%),酶活力最高达136 U/mL。单宁酶最适反应温度为60 ℃,在60 ℃处理30 min,能够维持酶原活力的90%以上。该酶最适反应pH值为6.0,在pH 5.0~9.0范围内,能够维持60%以上的酶活力。不同金属离子对酶活力的影响存在差异,Cu 2+ 、Fe 3+ 、Mn 2+ 和Zn 2+ 对该单宁酶活性抑制较强。经转录组学分析,该菌以麸皮为唯一碳源时,共有淀粉酶、纤维素酶和果胶酶等239 个CAZymes基因表达,其中糖苷水解酶类最为丰富,约占CAZymes表达总数的70%。A. fumigatus HBHF5是1 株优良产酶菌株,为具有食品酶制剂开发潜力的嗜热真菌。
关键词: 单宁酶;嗜热酶;脱涩;转录组;碳水化合物活性酶
单宁酶(EC 3.1.1.20)是一类降解单宁中酯键和缩酚酸键的水解酶,广泛分布在植物和微生物中 [1-2] 。其底物单宁作为一种天然大分子物质存在于植物的茎、根和果实当中。因作为食品加工原料的果实含有单宁,往往具有苦涩味,这使得柿子、刺梨等高含量单宁水果生产加工时需要进行脱涩处理 [3-4] 。采用乙醇脱涩、乙烯脱涩及加热脱涩等传统方法进行脱涩处理,达到了一定的效果,但是产品在加工处理过程中会出现返涩现象 [5-6] 。利用单宁酶脱涩法不仅可以避免上述现象的发生,同时还可以提高产品的稳定性 [7-9] 。单宁酶较高的价格限制了其在食品领域应用的范围,丰富单宁酶资源具有较强的现实意义。
嗜热真菌是一类能够在45 ℃以上正常生长,而在19 ℃以下无法生长的真核微生物 [10] 。嗜热真菌所分泌酶类大部分为嗜热酶 [11] ,因其具有较高的催化反应温度及良好的稳定性能,在食品、医药及化工等领域方面应用前景广阔 [12-13] 。目前,嗜热真菌来源的单宁酶研究鲜有报道。碳水化合物活性酶(carbohydrate-active enzymes,CAZymes)是一类降碳解水化合物酶类的总称 [14] ,大多数食品原料含有淀粉、纤维素、果胶类等物质,这使得降解这些物质酶在食品工业中发挥着越来越重要的作用。因此,CAZymes酶谱分析是对菌株应用潜力的重要指标,传统的酶谱分析策略无法全面对菌株酶谱分析。随着现代转录组学技术的不断发展,利用转录组学测序技术开展菌株产酶谱的研究成为新的策略。
本课题组前期从大曲中分离、筛选获得1 株产单宁酶的嗜热真菌HBHF5,本研究旨在对菌株HBHF5进行鉴定及酶学特性的相关研究;利用转录组测序技术对该菌株糖苷水解酶谱进行分析,掌握菌株的产酶性能,为寻找新型单宁酶及其他食品酶的开发提供理论指导。
菌株HBHF5筛选自河北保定刘伶醉酿酒股份有限公司大曲粉,并保存于本实验室。
牛肉膏、蛋白胨等均为国产分析纯;Trizol、氯仿-异戊醇(24∶1,V/V) 北京索莱宝科技有限公司;单宁酸 美国Sigma公司;真菌DNA提取试剂盒、聚合酶链式反应(polymerase chain reaction,PCR)相关试剂北京全式金生物技术有限公司。
基础盐溶液:NH 4 NO 3 11 g,MgSO 4 ·7H 2 O 1 g,NaCl 0.1 g,溶于100 mL水,用于固态发酵培养基和液态发酵培养基的制备。固态发酵培养基:麸皮5 g,单宁酸10 mg,基础盐溶液5 mL,放置于250 mL锥形瓶中,混匀,自然pH值,121 ℃灭菌20 min。液态发酵培养基:麸皮1.5 g,单宁酸150 mg,基础盐溶液150 mL,放置于250 mL锥形瓶中,混匀,自然pH值,121 ℃灭菌20 min。
CX31光学显微镜 日本Colmpus公司;GL-2M离心机上海市离心机械研究所;ABI 9700 PCR仪 美国Applied Biosystems公司;凝胶成像仪分析系统 美国Bio-Rad公司;立式高速低温离心机 日本Hitachi公司。
1.3.1 菌株的鉴定
1.3.1.1 菌落形态特征观察
将菌株HBHF5点植在PDA平板中,45 ℃倒置培养,观察并记录菌落形状、大小、生长速率、是否产色素等,对照真菌鉴定手册 [15] ,初步确定菌株的种属地位。
1.3.1.2 ITS序列分子鉴定
菌株HBHF5基因组DNA提取采用CTAB法;ITS序列PCR扩增参数及体系参照文献[16]进行,纯化后的PCR扩增产物由华大基因科技服务有限公司进行测序;将获得的测序结果提交NCBI数据库(http://www.ncbi.nlm.gov/blast/)进行BLAST同源性分析,采用MEGA 5.0软件构建系统进化树并分析。
1.3.2 生长曲线的测定
采用菌体干重法测定菌株生长曲线。菌株孢子悬浮液接种于50 mL PDB培养基中,45 ℃摇床培养,分别在12、24、36、48、60 h和72 h取样,过滤获得菌丝体,在105 ℃条件下烘干至质量恒定,测定菌体质量,绘制菌株的生长曲线。
1.3.3 单宁酶活力的测定
粗酶液的制备:菌株HBHF5种子接种于液体发酵培养基,在45 ℃条件下摇床培养72 h后,发酵液离心处理(12 000 r/min,10 min),收集上清液,即为粗酶液,4 ℃备用。
单宁酶活力测定参考Sharma等 [17] 方法。取100 μL适当稀释酶液与400 μL没食子酸丙酯溶液(1.25 mmol/L,pH 5.0)混匀,60 ℃反应10 min后,加入300 μL罗丹宁终止反应,并加入200 μL KOH(0.5 mmol/L)溶液进行显色,在520 nm波长处测定其吸光度,并计算酶活力。以每分钟水解没食子酸丙酯底物产成1 μmol没食子酸所需酶量定义为1 个酶活力单位(U)。
1.3.4 固态培养和液态培养对菌株HBHF5产酶的影响
将菌株HBHF5种子接种于液体或固态产酶培养基中,45 ℃诱导培养4 d后收集液体发酵培养液,离心处理收获上清液为样品1(SSF1),菌体进行超声波破碎为样品2(SSF2);固态发酵浸出液为样品3(SmF1),固态发酵菌体超声波破碎液为样品4(SmF2),分别对4 种样品进行单宁酶活力的测定,探究该菌单宁酶的在固液两种培养方式对产酶的影响。
1.3.5 酶学性质分析
1.3.5.1 最适反应pH值的测定
将酶液在不同pH值(2、3、4、5、6、7、8、9)条件下与底物反应,按照1.3.3节方法测定酶活力,以最高酶活力为100%,测定酶最适反应pH值。
1.3.5.2 最适反应温度的测定
在最适pH值条件下,将酶液在不同温度(20、30、40、50、60、70、80 ℃)与底物进行反应,按照标准方法测定酶活力,以最高酶活力为100%,测定酶最适反应温度。
1.3.5.3 热稳定性的测定
在最适pH值条件下,将酶液分别在50、60 ℃和70 ℃处理不同时间(0、2、5、10、20、30 min和60 min),以最高酶活力为100%,测定剩余酶活力,研究单宁酶的热稳定性。
1.3.5.4 金属离子对酶活力的影响
通过在酶反应体系中加入终浓度为1、5 mmol/L的金属离子(Cu 2+ 、Fe 3+ 、Mn 2+ 、Ca 2+ 、Mg 2+ 、Li + 、K + 、Na + 和Zn 2+ )研究其对酶活力的影响。以未经过添加任何金属离子的反应为对照组,按照标准酶活力测定方法测定其剩余酶活力。
1.3.6 CAZymes基因表达分析
菌株HBHF5接入液态诱导培养基中,45 ℃诱导培养72 h后离心收集菌体。采用Trizol方法提取总RNA,并使用Agilent 2100 Bioanalyzer和Agilent RNA 6000 Nano Kit对RNA浓度与完整性进行测定分析,纯度采用凯奥K5500微量分光光度计来测定。取3 μg总RNA样品,利用Ribo-Zero-magnetic-kit对样品mRNA进行富集,通过Fragmentation buffer试剂对mRNA进行片段化处理,并以此为模板构建表达文库。
采用Hiseq TM 2500测序平台进行双端测序,获得100 bp左右测序reads。对获得的原始测序序列进行分析,去除只含有接头或过短的序列数据。利用软件TOPHAT(v2.0.6)将预处理后的数据与参考Ensembl真菌数据库Aspergillus fumigatus.CADRE.33基因组序列进行比对获得基因的注释信息。依据CAZymes数据库(http://www.cazy.org/)分类标准,对测序数据进行统计分析。
每组实验均设3 次重复,取平均值,运用Microsoft Excel 2010和SPSS 19.0数据处理软件对试验数据结果进行处理和方差分析。

图1 HBHF5菌株菌落特征(A)及系统发育树(B)
Fig. 1 Cultural characteristics (A) and phylogenetic tree (B) of strain HBHF5
如图1A所示,菌株HBHF5在45 ℃条件下生长旺盛,培养3 d菌落即可铺满整个平皿,在 PDA平板中的菌落紧密,表面粗糙,菌落中心突起,边缘为辐射状褶痕;菌株生长初期时,菌落颜色为白色,随后菌落逐渐成为蓝绿色,在生长后期,菌落颜色为浅灰色,呈粉末状。分生孢子头顶囊呈烧瓶状,分生孢子梗单层,成木栅状排列状布满顶囊,孢子成球形,绿色。
如图1B所示,菌株HBHF5 ITS序列经BLAST比对分析,菌株HBHF5与烟曲霉(Aspergillus fumigatus AF293)一致性为99%,且在进化树中位于同一个分支中,即具有较近的亲缘关系。
综上所述,菌株HBHF5经形态观察和分子鉴定结果,该菌属于曲霉属,烟曲霉种,最终命名为烟曲霉HBHF5(A. fumigatus HBHF5)。
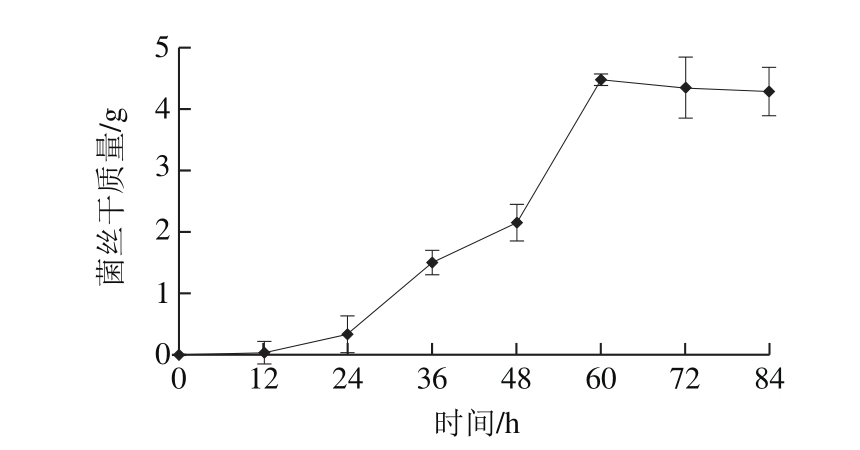
图2 HBHF5菌株生长曲线
Fig. 2 Growth curve of strain HBHF5
A. fumigatus HBHF5生长变化情况如图2所示,该菌以体积分数1%接菌培养时延滞期时间为12 h,菌株培养24 h后进入对数生长期,大约持续生长36 h,即在培养60 h后达到生长稳定期。同时通过对单宁酶活力测定分析,发现该菌在对数培养时期即开始进行产酶,随着菌株自身的生长,产酶量也随之增加,直至对数生长周期,单宁酶产酶量达到最大值,其酶活力为136 U/mL。以上结果表明A. fumigatus HBHF5菌株在45 ℃条件下生长迅速,能够短周期发酵生产即可获得单宁酶产物。
通过对比分析研究固态和液态发酵方式下A. fumigatus HBHF5产单宁酶差异,发现培养方式对该菌的产酶影响较大。A. fumigatus HBHF5在固态培养条件下几乎不合成单宁酶,而在液态发酵培养时可产生大量的单宁酶,且以胞外分泌形式为主(94%)(图3)。这与已报道大多数生产单宁酶菌株的产酶方式不同,该特性使得A. fumigatus HBHF5可以采用液态深层发酵方式生产单宁酶。
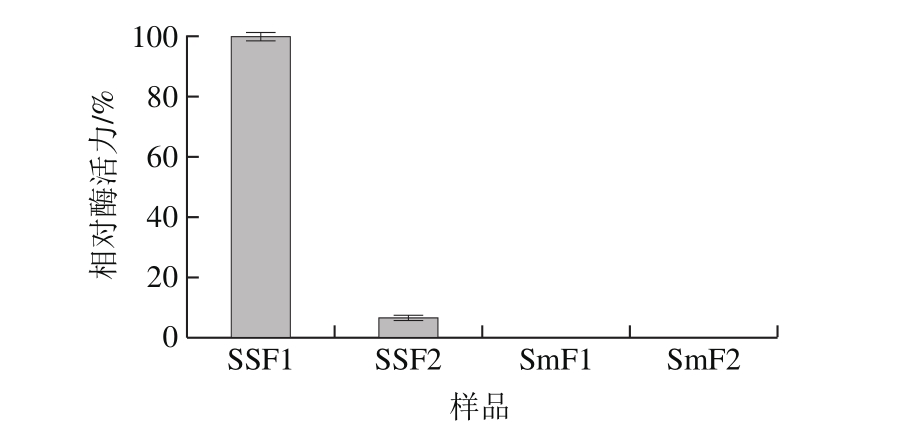
图3 培养方式对A. fumigatus HBHF5产单宁酶的影响
Fig. 3 Effect of culture systems on tannase production of strain HBHF5
经测定,该单宁酶在pH值为6.0时,酶活力最高。在pH 5.0~9.0范围内,能够维持60%以上的酶活力(图4A),具有一定的pH值适应性,能够满足多种原料加工生产中的需求。该单宁酶最适反应温度为60 ℃,在70 ℃时依然具有50%以上的酶活力,在20 ℃反应时,该单宁酶能维持大约57%的酶活力(图4B),该酶属于嗜热酶。
在酶热稳定性的研究中发现,该单宁酶在50 ℃热处理1 h,其剩余酶活力基本未发生改变;60 ℃处理60 min,相对酶活力在80%以上;当该酶在70 ℃处理30 min,能够保持80%的酶活力,且在处理60 min后,依然能够维持大约50%的酶活力,以上结果充分表明HBHF5菌株所产单宁酶具有极好的热稳定性(图4C),该性能能够保证该单宁酶在生产及贮存过程中保持较强活力,有利于酶制剂产品品质的稳定。
在低离子浓度(1 mmol/L)存在的条件下,除 K + 、Li + 和Na + 未对酶活力造成抑制之外,金属离子Cu 2+ 对酶活力抑制最为明显,其次是Zn 2+ 对酶活性的抑制明显(94%),其他活性抑制率结果为:Fe 3+ (69%),Mn 2+ (61%)和Mg 2+ (22%)。以上的金属离子在高离子浓度(5 mmol/L)下,对酶的抑制率不同程度增高,其中以Fe 3+ 和Na + 变化最为显著,其抑制率分别为88%和31%(图4D)。

图4 A. fumigatus HBHF5的单宁酶酶学性质分析
Fig. 4 Characterization of tannase from strain HBHF5

图5 A. fumigatus HBHF5碳水化合物活性酶基因表达分析
Fig. 5 Distribution of CAZymes gene expression in A. fumigatus HBHF5
通过对菌株HBHF5的转录组测序分析,共获得该菌的23 927 186 条reads,数据量为3 589 077 900 bp,GC含量为53.41%,Q 20 值为98.12%,Q 30 值为95.10%,Percentage值为92.73%。通过对比对到参照基因组(Aspergillus fumigatus.CADRE.33)上的reads分布统计发现,reads分布在Exon区域值最高为87.98%,其次是定位在Integenic区域(11.8%),只有0.22%的reads定位在Intron区域,以上数据说明测序结果符合要求,可以进行后续分析。
菌株所产酶的种类决定着其在酶制剂开发中的潜力的大小,经对A. fumigatus HBHF5的转录组分析,当该菌以麸皮作为唯一碳源时,共有2 151 个酶基因表达,其中氧化还原酶579 个、转移酶644 个、水解酶522 个、裂合酶167 个、异构酶71 个和合成酶168 个,水解酶类数量大约占总酶的24%左右。
CAZymes是一类具有降解、修饰及生成糖苷键的功能酶类的总称,由糖酯酶类(carbohydrate esterase,CEs)、糖苷水解酶类(glycoside hydrolase,GHs)、糖基转移酶类(glycosyl transferase,GTs)和多糖裂解酶类(polysaccharide lyase,PLs)组成 [18] 。以麸皮为碳源时,菌株HBHF5中CAZymes基因表达结果如图5所示。经分析,CEs有12 个基因表达,GHs共有56 个基因表达,PLs共有2个基因表达,其中GHs表达最为丰富,约占CAZymes酶类表达总数的70%(167 个基因),约占总GHs家族的14.5%(21种),其主要种类如下:聚半乳糖醛酸酶、几丁质酶、半乳糖苷酶、甘露糖苷酶、葡萄糖淀粉酶、纤维素酶、淀粉酶、木聚糖酶、甘露聚糖酶、海藻糖酶等。
以上结果表明,菌株A. fumigatus HBHF5酶系较丰富,是1 株优良的产酶菌株。
大曲中的微生物种类丰富,是一类亟待挖掘的我国特有微生物资源 [19] 。到目前为止,已从大曲中获得了产嗜热淀粉酶和半乳糖苷酶的芽孢杆菌 [20] ,产糖化酶的棒曲霉和黑曲霉 [21-22] 及产酯酶的少根根霉等 [23] ,而从大曲中获得产单宁酶的嗜热真菌鲜有报道。Penicillium sp.、Aspergillus sp.、Polyporus sp.和Trametes sp.等属均筛选获得了产单宁酶的真菌 [24] ,而Aspergillus sp.来源的微生物是最具有商业开发价值的单宁酶生产菌种属。本课题组前期从浓香大曲中筛选获得了1 株产单宁酶的嗜热真菌,经鉴定该菌属于烟曲霉种,命名为A. fumigatus HBHF5,该菌株在高温培养时生长良好。
大多数单宁酶最适反应温度范围在30~40 ℃之间,如A. niger N5-5单宁酶的最适温度为45 ℃ [25] ,而A. fumigatus HBHF5所产单宁酶最适反应温度为60 ℃左右,在70 ℃时依然具有50%以上的酶活力,属于嗜热酶,且该酶在50 ℃条件下酶活性稳定,这有利于该酶制剂的贮存加工。通过9 种金属离子对单宁酶活力的影响研究结果,几乎未发现有激活酶活性的金属离子,有6 种金属离子对该酶的活性有不同程度的抑制,这与Rajakumar等 [26] 的研究结果相似,却与Sena等 [27] 的研究存在一定的差异,这可能是由于不同来源的单宁酶性质存在一定的差异所致。
目前,生产所用菌株(黑曲霉)在液态发酵生产单宁酶时主要以胞内单宁酶形式存在,这使得在提取单宁酶时,往往需要破壁处理,该操作不仅增加了生产成本,同时还使得在破壁处理时大量细胞内蛋白溢出,加剧了单宁酶的分离纯化难度 [28] ,因此,获得在液体培养生产胞外单宁酶的菌株,具有极其重要的现实意义。研究发现,A. fumigatus HBHF5在液态发酵培养时,所产单宁酶主要以胞外酶的形式存在,这对于单宁酶的生产极具经济效应。固态发酵和液态发酵体系在含水量、含氧量、基质浓度及能量传递等方面存在较大差异。到目前为止,影响其胞外分泌机制仍不清楚,需进行进一步探究。
食品酶类主要被用在碳水化合物原料的加工处理,因此分析菌株CAZymes的丰富程度,直接影响到菌株在食品酶制研发中的潜力。研究常通过对菌株不同酶活性的检测来探究其产酶谱,虽然该策略具有一定的效果,但是酶种类数量往往较少,一般只有数种,因此不能客观的对菌株的产酶潜力进行评价 [29] 。随着组学时代的到来,转录组学技术的成熟和成本的降低为开展产酶谱的研究提供了有利的支撑 [30] 。A. fumigatus HBHF5在麸皮诱导时,经转录组表达分析,共有2 151 个酶基因表达,有239 个CAZymes类基因表达,涉及到淀粉降解相关酶,纤维素降解相关酶和半纤维素降解相关酶,其酶总量远高于传统的酶谱检测数量。因此,利用组学技术能够客观真实的评价菌株产酶谱。综上所述,A. fumigatus HBHF5是1 株具有巨大开发潜力的酶制剂生产菌株。
参考文献:
[1] YAO J, GUO G S, REN G H, et al. Production, characterization and applications of tannase[J]. Journal of Molecular Catalysis B:Enzymatic, 2014, 101: 137-147. DOI:10.1016/j.molcatb.2013.11.018.
[2] AOKI K, SHINKE R, NISHIRA H. Purfication and some properties of yeast tannase[J]. Agricultural and Biological Chemistry, 1976, 1(40):79-85. DOI:10.1080/00021369.1976.10861997.
[3] SIM H H, CHOI O J. Study on optimization of persimmon Kochujang Jangachi using response surface methodology[J]. Journal of the Society of Food Science and Nutrition, 2015, 44(9): 1364-1373.DOI:10.3746/jkfn.2015.44.9.1364.
[4] HWANG K A, HWANG Y J, HWANG I G, et al. Cholesterollowering effect of astringent persimmon fruits (Diospyros kaki Thunb.)extracts[J]. Food Science and Biotechnology, 2017, 26(1): 229-235.DOI:10.1007/s10068-017-0031-4.
[5] 余先纯, 韩大良, 吴瑛, 等. 微波和复合酶法提取野柿子树叶单宁的比较研究[J]. 应用化工, 2013, 42(4): 673-676. DOI:10.16581/j.cnki.issn1671-3206.2013.04.046.
[6] NOBUKI G, AKIO K, NASTUMI H, et al. Persimmon fruit tanninrich fiber reduces cholesterol levels in humans[J]. Annals of Nutrition and Metabolism, 2013, 62(1): 1-6. DOI:10.1159/000343787.
[7] TASKIN M. Co-production of tannase and pectinase by free and immobilized cells of the yeast Rhodotorula glutinis MP-10 isolated from tannin-rich persimmon (Diospyros kaki L.) fruits[J]. Bioprocess and Biosystems Engineering, 2013, 36(2): 165-172. DOI:10.1007/s00449-012-0771-8.
[8] 罗昱, 梁芳, 李小鑫, 等. 单宁酶对刺梨果汁单宁的脱除作用[J].食品科学, 2013, 34(18): 41-44. DOI:10.7506/spkx1002-6630-201318009.
[9] GOVINDARAJAN R K, REVATHI S, RAESHKUMAR N, et al.Microbial tannase: current perspectives and biotechnological advances[J]. Biocatalysis and Agricultural Biotechnology, 2016, 6:168-175. DOI:10.1016/j.bcab.2016.03.011.
[10] MAHESHWARI R, BHARADWAJ G, BHAT M K. Thermophilic fungi: their physiology and enzymes[J]. Microbiology and Molecular Biology Reviews, 2000, 64(3): 461-488. DOI:10.1128/MMBR.64.3.461-488.2000.
[11] VAN DEN BURG B. Extremophiles as a source for novel enzymes[J].Current Opinion in Microbiology, 2003, 6(3): 213-218. DOI:10.1016/S1369-5274(03)00060-2.
[12] GUMISIRIZA R, HAWUMBA J F, OKURE M, et al. Biomass waste-to-energy valorisation technologies: a review case for banana processing in Uganda[J]. Biotechnology for Biofuels, 2017, 10(1):1-14. DOI:10.1186/s13068-016-0689-5.
[13] 张耿崚, 韩业钜, 陈细妹, 等. 产纤维素酶嗜热地芽孢杆菌HTA426的筛选鉴定、酶学性质分析及其应用[J]. 环境科学学报, 2017,37(4): 1444-1453. DOI:10.13671/j.hjkxxb.2016.0392.
[14] KUNATH B J, BREMGES A, WEIMANN A, et al. Metagenomics and CAZyme discovery[J]. Protein-Carbohydrate Interactions:Methods and Protocols, 2017: 255-277. DOI:10.1007/978-1-4939-6899-2_20.
[15] 魏景超. 真菌鉴定手册[M]. 上海: 上海科学技术出版社, 1979.
[16] 李颖, 徐英春. 评价ITS、BenA和CaM序列分析在曲霉菌种鉴定方面的应用[J]. 中国真菌学杂志, 2017, 12(2): 74-77. DOI:10.3969/j.issn.1673-3827.2017.02.003.
[17] SHARMA S, BHAT T K, DAWRA R K. A spectrophotometric method for assay of tannase using rhodanine[J]. Analytical Biochemistry, 2000, 279(1): 85-89. DOI:10.1006/abio.1999.4405.
[18] PARK B H, KARPINETS T V, SYED M H, et al. CAZymes analysis toolkit (CAT): web service for searching and analyzing carbohydrateactive enzymes in a newly sequenced organism using CAZy database[J]. Glycobiology, 2010, 20(12): 1574-1584. DOI:10.1093/glycob/cwq106.
[19] ZHENG X W, TABRIZI M R, NOUT M J, et al. Daqu-a traditional Chinese liquor fermentation starter[J]. Journal of the Institute of Brewing,2011, 117(1): 82-90. DOI:10.1111/j.1365-2621.2011.02660.x.
[20] JIN F, LI Y, ZHANG C, et al. Thermostable α-amylase and α-galactosidase production from the thermophilic and aerobic Bacillus sp. JF strain[J]. Process Biochemistry, 2001, 36(6): 559-564.DOI:10.1016/S0032-9592(00)00247-8.
[21] 杨跃寰, 叶光斌, 边名鸿, 等. 泸曲中两株高产糖化酶菌的分离鉴定与初步应用[J]. 酿酒科技, 2013(10): 65-68. DOI:10.13746/j.njkj.2013.10.048.
[22] 张帅, 曹庸, 梁晓莹, 等. 黑曲霉N5-5单宁酶的纯化及酶学性质测定[J].食品科学, 2017, 38(6): 142-146. DOI:10.7506/spkx1002-6630-201706022.
[23] 黄丹, 储玉龙, 尚志超, 等. 大曲酯化酶根霉菌的分离及产酶条件研究[J]. 食品与发酵科技, 2010, 46(3): 30-32. DOI:10.3969/j.issn.1674-506X.2010.03-008.
[24] BELMARES R, CONTRERAS-ESQUIVEL J C, RODRIGUEZHERRERA R, et al. Microbial production of tannase: an enzyme with potential use in food industry[J]. LWT-Food Science and Technology,2004, 37(8): 857-864. DOI:10.1016/j.lwt.2004.04.002.
[25] ZHANG S, CUI F C, CAO Y, et al. Sequence identification, structure prediction and validation of tannase from Aspergillus niger N5-5[J].Chinese Chemical Letters, 2016, 27(7): 1087-1090. DOI:10.1016/j.cclet.2016.04.013.
[26] RAJAKUMAR G S, NANDY S C. Isolation, purification, and some properties of Penicillium chrysogenum Tannase[J]. Applied &Environmental Microbiology, 1983, 46(2): 525-527. DOI:0099-2240/83/080525-0302.00/0.
[27] SENA A R D, SANTOS A C D B D, GOUVEIA M J, et al.Production, characterization and application of a thermostable tannase from Pestalotiopsis guepinii URM 7114[J]. Food Technology and Biotechnology, 2014, 52(4): 459-467. DOI:10.17113/ftb.52.04.14.3743.
[28] LEKHA P K, LONSANE B K. Production and application of tannin acyl hydrolase: state of the art[J]. Advances in Applied Microbiology,1997, 44: 216-260. DOI:10.1016/S0065-2164(08)70463-5.
[29] 范光森, 严烨, 杨绍青, 等. 一株产木聚糖酶嗜热真菌樟绒枝霉的鉴定及其产纤维质降解酶系分析[J]. 应用与环境生物学报, 2013,19(1): 48-53. DOI:10.3724/SP.J.1145.2013.00048.
[30] WANG T Y, CHEN H L, LU M Y J, et al. Functional characterization of cellulases identified from the cow rumen fungus Neocallimastix patriciarum W5 by transcriptomic and secretomic analyses[J]. Biotechnology for Biofuels,2011, 4(1): 24. DOI:10.1186/1754-6834-4-24.
Identification, Properties and Carbohydrate-Active Eenzymes (CAZymes) Expression Analysis of a Thermophilic Fungal Strain Producing Tannase
GU Xinxi 1,2 , LU Haiqiang 1 , LIU Yajuan 3 , CHEN Saijuan 3 , GU Zilin 4 , MENG Xiangchen 2 , CHEN Baojiang 4, *
(1. College of Food Science and Technology, Hebei Agricultural University, Baoding 071000, China;2. Key Laboratory of Dairy Science, Ministry of Education, Northeast Agricultural University, Harbin 150030, China;3. Mountain Area Research Institute, Hebei Agricultural University, Baoding 071000, China;4. College of Animal Science and Technology, Hebei Agricultural University, Baoding 071000, China)
Abstract: In this study, a thermophilic fungal strain (HBHF5) producing tannase was identified. Also, the enzymatic properties of tannase from strain HBHF5 were evaluated and transcriptomic analysis of its carbohydrate-active enzymes(CAZymes) was carried out. We aimed to explore the potential of strain HBHF5 for use in the development of food enzyme preparations. Based on its morphology and ITS sequence alignment analysis, strain HBHF5 was identified as Aspergillus fumigatus. A. fumigatus HBHF5 did not produce tannase under solid-state fermentation (SSF), but it produced both extracellular and intracellular tannase when cultured under submerged fermentation conditions. The extracellular enzyme was found to be dominant (94%) and its activity was as high as 136 U/mL. Tannase activity was optimal at pH 6.0 and 60 ℃. After treatment at 60 ℃ for 30 min, more than 90% of the initial activity was retained, and the residual activity was over 60% in the pH range of 5.0 to 9.0. The tannase activity was inhibited by some metal ions, such as Cu 2+ ,Fe 3+ , Mn 2+ and Zn 2+ . Transcriptomic analysis showed that 239 CAZymes genes including those encoding amylase, cellulase and pectinase were expressed when wheat bran was used as the sole carbon source. Among these the most abundant was glycoside hydrolase, accounting for about 70% of the total CAZymes. It has been demonstrated that A. fumigatus HBHF5 is a good producer of tannase and has the potential to be used to develop food enzyme preparations.
Keywords: tannase; thermophilic enzyme; astringency removal; transcriptomics; carbohydrate-active enzymes
GU Xinxi, LU Haiqiang, LIU Yajuan, et al. Identification, properties and carbohydrate-active eenzymes (CAZymes)expression analysis of a thermophilic fungal strain producing tannase[J]. Food Science, 2018, 39(18): 88-93. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201818014. http://www.spkx.net.cn
谷新晰, 卢海强, 刘亚娟, 等. 1 株产单宁酶嗜热真菌的鉴定、酶学性质分析及碳水化合物活性酶表达[J]. 食品科学,2018, 39(18): 88-93. DOI:10.7506/spkx1002-6630-201818014. http://www.spkx.net.cn
文章编号: 1002-6630(2018)18-0088-06
引文格式:
中图分类号: Q939.97
文献标志码: A
*通信作者简介: 陈宝江(1971—),男,教授,博士,研究方向为分子营养。E-mail:chenbaojiang@vip.sina.com
DOI: 10.7506/spkx1002-6630-201818014
基金项目: 国家自然科学基金青年科学基金项目(31501417);河北省食品科学与工程学科“双一流”建设资金项目(2016SPGCA18)
第一作者简介: 谷新晰(1982—),女,讲师,硕士,研究方向为食品微生物资源的开发与应用。E-mail:helloguxinxi@163.com
收稿日期: 2017-07-28