
图1 细菌多样性
Fig. 1 Bacterial diversity of fermentation broth
张大力,冯艳鹤,刘炳利,潘 聪,封 玲,郑明珠*
(吉林农业大学食品科学与工程学院,小麦和玉米深加工国家工程实验室,吉林 长春 130118)
摘 要: 目的:研究自然发酵糯玉米中细菌的菌群组成,以及其中乳酸菌对糯玉米粉加工特性的影响。方法:以地方特色的糯玉米为原料,采用MiSeq技术对自然发酵菌液中细菌16S rDNA的V3~V4区进行测序分析,获得自然发酵液中的细菌多样性。对其中的1 株优势菌株进行筛选鉴定,比较纯菌种发酵和自然发酵对糯玉米粉的糊化特性和质构特性的影响。结果:自然发酵液菌中乳杆菌(Lactobacillus)为优势菌,经形态学特征、生理生化特征及16S rDNA基因序列分析鉴定其为发酵乳杆菌(Lactobacillus fermentum)。经过纯种发酵后糯玉米粉的峰值黏度、衰减值和最终黏度显著提高,回生值显著降低;经过蒸制后硬度、黏附性、胶着性和咀嚼性显著增高从而改善糯玉米粉质特性。
关键词: 糯玉米;多态性分析;16S rDNA;糊化特性;质构特性
糯玉米起源于中国,其含有人体所需的多种营养成分,蛋白质质量分数约为10.6%,淀粉质量分数为65%~75%,脂肪质量分数为4%~5.5%,还原糖质量分数达到1.6%。又因其分子质量比普通玉米小10 倍多,食用消化率高出20%。营养价值和消化率都超过玉米,所以以糯玉米为主的糯性谷物食品已成为当下餐桌热点 [1] 。我国北方冬天具有特色的传统食品之一的黏豆包正是以糯玉米粉为主要原料,通过自然发酵的方式制作而成的冷冻食品 [2] 。但糯玉米碴自然发酵周期长、微生物菌群复杂,在自然发酵过程中可能存在腐败菌甚至致病菌污染的情况,发酵过程受很多因素影响。
发酵是微生物活动的一种体现,自然发酵过程中环境条件、地域差异的不同,对产品的安全、质构、色泽、风味起着至关重要的作用 [3-4] 。利用微生物发酵技术调控和提升产品品质的关键在于要了解自然发酵食品中微生物群落结构,高通量测序方法不仅不需要对微生物进行分离培养,就能够检测到较低丰度的微生物,还能更加全面而准确地展示样品微生物的群落结构 [5-6] 。
因此,本研究拟对自然发酵糯玉米碴中的细菌多样性进行分析,揭示其中的优势菌群,然后对优势菌株进行分离鉴定,获得优势菌株后进行糯玉米粉的微生物改性处理,比较纯种发酵和自然发酵对糯玉米粉加工特性的影响。为今后工业化生产质量稳定、健康安全的发酵糯玉米粉为主要原料的产品提供基本理论和实践依据。
糯玉米碴由小麦和玉米深加工国家工程实验室提供。
MRS培养基 美国BD公司;新型微生物微量生化鉴定管 青岛宾得生物技术有限公司;E.Z.N.A.Soil DNA Ki美国Omega公司;Qubit2.0 DNA检测试剂盒 美国Life Tech公司;Ep0406 Taq DNA Polymerase 美国Thermo Fisher公司;Agencourt AMPure XP核酸纯化试剂盒美国Beckman公司;溶菌酶、蛋白酶K、酚-氯仿-异戊醇溶液、氯仿-异戊醇溶液、Tris、乙二胺四乙酸二钠、TE缓冲液、十二烷基硫酸钠、NaCl、琼脂糖 北京索莱宝科技有限公司。
DSX-280B型手提式压力蒸汽灭菌器 上海申安医疗器械厂;GL-88B旋涡混合器 海门市其林贝尔仪器制造有限公司;FW-100高速万能粉碎机 北京市永光明医疗仪器有限公司;HZQ-F160型全温振荡培养箱哈尔滨市东联电子技术开发有限公司;Allegra X-30型离心机 美国Beckman公司;电泳仪 北京市六一仪器厂;凝胶成像系统 美国GE公司;聚合酶链式反应(polymerase chain reaction,PCR)仪 德国Eppendorf公司;RVA-TecMaster TM 型快速黏度分析仪 澳大利亚Perten公司;TA-XT plus物性测定仪 英国Stable Micro System公司;Phenom pro型扫描电子显微镜 荷兰Phenom-world公司。
1.3.1 样品制备
将糯玉米碴用灭菌的蒸馏水浸泡,使蒸馏水没过样品,室温浸泡96 h后取4 mL液体样品,分多次加入灭菌的2 mL离心管中,10 000×g离心3 min,弃置上层液体,将离心管倒置直至没有液体流出。
1.3.2 DNA的提取与PCR扩增
根据CTAB法进行总DNA的快速提取 [7] ,以提取的总DNA为模板,采用16S rDNA V3~V4区引物,341F引物:5’-CCCTACACGACGCTCTTCCGATCTG-3’、(barcode)5’-CCTACGGGNGGCWGCAG-3’,805R引物:5’-GACTGGAGTTCCTTGGCACCCGAGAATT CCA-3’、5’-GACTACHVGGGTATCTAATCC-3′。PCR体系:2×Taq Master Mix 15 μL;Bar-PCR Primer F(10 μmol/L)1 μL;Primer R(10 μmol/L)1 μL;Genomic DNA 10~20 ng;H 2 O 30 μL。PCR扩增条件:94 ℃预变性3 min;94 ℃变性30 s,45 ℃退火20 s,65 ℃延伸30 s,5 个循环,94 ℃变性20 s,55 ℃退火20 s,72 ℃延伸30 s,20 个循环,72 ℃延伸5 min。PCR结束后进行第2轮扩增,反应体系:2×Taq Master Mix 15 μL;Primer F(10 μmol/L)1 μL;Primer R(10 μmol/L)1 μL;Genomic DNA 20 ng, H 2 O 30 μL。反应条件:95 ℃预变性3 min;94 ℃变性20 s,55 ℃退火20 s,72 ℃延伸30 s,5 个循环,72 ℃延伸5 min。PCR结束后,取PCR产物进行琼脂糖凝胶电泳检测。
1.3.3 发酵液中细菌多样性分析
自然发酵糯玉米碴中细菌多样性分析采用MiSeq技术 [8-13] ,测序由上海生工生物有限公司完成。
1.3.4 发酵液中乳酸菌分离、纯化与初步筛选
乳酸菌的分离采用稀释涂布平板法:取1 mL发酵菌悬液,置于9 mL灭菌生理盐水中,混匀后取1 mL加入另一管9 mL灭菌生理盐水中,以此类推,将发酵液稀释至原液的10 -9[14] 。取各稀释度的菌悬液各1 mL分别涂布于平板中,乳酸菌的纯化采用划线分离法:选取在MRS琼脂培养基中生长并产溶钙圈的单菌落,37 ℃厌氧条件下培养48 h后。选取产溶钙圈的单菌落进行革兰氏染色及过氧化氢酶检测 [15] ,符合革兰氏染色阳性且过氧化氢酶阴性特征即可以初步确定为乳酸菌。
1.3.5 乳酸菌的复筛与16S rDNA鉴定
复筛通过菌株的产酸能力、繁殖能力进行筛选 [16] 。确定一株产酸能力最快、繁殖能力最佳的菌株命名为FL-0616。将筛选得到的FL-0616菌株提取DNA经测序鉴定。采用SK8255(细菌)基因组提取试剂盒提取FL-0616菌株的基因组DNA [17] ,以该DNA为模板,27F引物:5’-AGTTTGATCMTGGCTCAG-3’;1492R引物:5’-GGTTACCTTGTTACGACTT-3’。分别为正向和反向引物,进行PCR扩增 [18] ,反应体系见1.3.2节,反应条件:94 ℃预变性4 min;94 ℃变性45 s,55 ℃退火45 s,72 ℃延伸1 min,30个循环,72 ℃延伸10 min。扩增产物经1%琼脂糖凝胶电泳检测后由生工生物工程(上海)有限公司测序。将测序基因序列通过NCBI的BLAST检索系统(http:∥www.ncbi.nlm1nih.gov /Blast.cgi/)进行序列同源性分析,采用邻接法(Neighbour-Joining)构建系统发育树,并通过1 000 次自举分析(Boostrap)进行置信度检测。
1.3.6 纯菌种发酵、自然发酵、未发酵样品的预处理
乳酸菌悬液的配制:将鉴定后的乳酸菌进行活化增殖培养,接种于MRS液体培养基上,置于37 ℃恒温培养箱中培养24 h,直到乳酸菌浓度达到5.21×10 8 CFU/mL。
纯菌种发酵:加入已灭菌的蒸馏水,糯玉米碴与蒸馏水的比例为1∶3(g/mL)装入1 000 mL锥形瓶中,将其混匀后加入分离得到的发酵乳杆菌菌悬液,菌悬液的接种量为体积分数5%,接种后置于37 ℃的恒温培养箱中静置发酵48 h。自然发酵:菌悬液用蒸馏水代替,其他条件与纯种发酵一致。未发酵:取纯种发酵质量相同的糯玉米碴置于37 ℃培养箱中,从烘箱中取出3 种状态下的糯玉米碴用高速万能粉碎机进行干磨制粉,磨粉后的糯玉米粉过80 目筛保存,样品预处理完成。
1.3.7 糊化特性的测定
向快速黏度分析仪测量筒中加入25 mL水,分别取3 种方式下的糯玉米粉3.00 g,用搅拌桨搅动20 s,搅拌均匀即可开始测量。在测量时起始温度设定为50 ℃,维持1 min;在3.9 min内升温至96 ℃;保持2 min;然后在3.5 min内降温至50 ℃,并保持1.5 min [19] 。
1.3.8 熟面团质构特性的测定
采用质构仪测定样品质构,取3 种方式下的糯玉米粉20 g,加入80%的蒸溜水分别制成厚度约为0.5 cm生面团,蒸熟后进行测定。物性仪选用的探头为P/36R,测定的条件为 [20] :测量前速率1.00 mm/s;测量速率2.00 mm/s;测量后速率2.00 mm/s;压缩率50.00%;压缩程度70%;两次压缩间隔5 s;强制触发力5.0 g,触发距离2.00 mm。
1.3.9 扫描电子显微镜观察
样品黏在样品台上,经真空喷金后,用扫描电子显微镜观察并采集图像(×5 000)。

图1 细菌多样性
Fig. 1 Bacterial diversity of fermentation broth
如图1所示,自然发酵的菌液中菌群组成为:乳杆菌属(Lactobacillus)65.14%、肠杆菌属(Enterobacter)23.85%、魏斯氏菌属(Weissella)7.09%、明串珠菌属(Leuconostoc)0.9%、未分类(unclassified)0.96%、克雷伯菌属(Klebsiella)0.45%、微小杆菌属(Exiguobacterium)0.42%、勒克菌属(Leclercia)0.23%、不动杆菌属(Acinetobacter)0.19%、肠球菌属(Enterococcus)0.14%、铜绿假单胞菌属(Pseudomonas)0.14%、泛菌属(Pantoera)0.14%、葡萄球菌属(Staphylococcus)0.14%。研究结果表明,乳杆菌属是自然发酵菌液中的优势菌属。
2.2.1 细菌生理生化鉴定
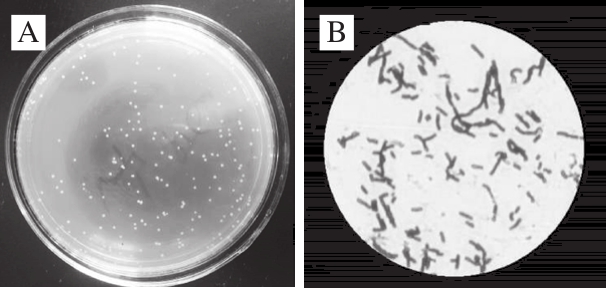
图2 分离株(A)及其革兰氏染色(B)(×400)
Fig. 2 Isolates (A) and Gram staining (B) (× 400)
如图2所示,菌落颜色为乳白色、凸起不透明、光滑湿润、边缘整齐、无芽孢杆状、直径在1.0~2.0 mm、革兰氏染色呈阳性,排列方式为单个或多个短链。
2.2.2 菌株液体培养基的最终pH值与生长量测定结果


图3 各菌株培养过程中菌液pH值与菌株生长量的变化
Fig. 3 Change in pH value and growth rate of six selected isolates
对筛选得到的6株符合革兰氏染色阳性过氧化氢酶阴性的菌株测定其pH值和生长曲线。由图3可知,菌株的pH值在24 h内下降均比较明显,菌体生长速率在18 h左右达到最大值。但菌株FL-0616的pH值最低3.87,OD 600 nm 为2.155,更加适合做发酵剂,因此对其进行16S rDNA鉴定。
表1 FL-0616细菌生理生化鉴定结果
Table 1 Physiological and biochemical identification of FL-0616
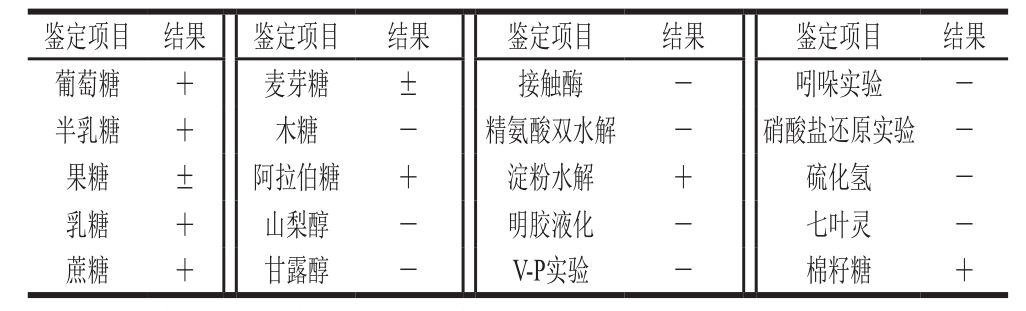
注:+.阳性;-.阴性;±.弱阳性。
由表1可知,经过筛选得到的菌株FL-0616能够发酵葡萄糖、半乳糖、乳糖、蔗糖及阿拉伯糖,对果糖与麦芽糖呈弱阳性反应。在明胶液化、V-P实验、吲哚实验、硝酸酸盐还原实验、硫化氢等实验中均呈阴性反应。通过参照文献[21-22]初步认定菌株FL-0616为发酵乳杆菌。
2.2.3 分子生物学鉴定

图4 菌株FL-0616片段DNA的PCR扩增电泳图
Fig. 4 PCR amplification of DNA fragment from strain FL-0616
如图4所示,菌株FL-0616扩增产物序列长度为1 435 bp,与预期结果一致。根据FL-0616的16S rDNA序列与NCBI数据库上Lactobacillus fermentum(MF3551691.1)、L. ingluviei(AB911500.1)、L. equigenerosi(NR041566.1)等不同种乳杆菌构建系统发育树,图5结果显示FL-0616与发酵乳杆菌(L. fermentum)自然聚为一支,且自展值为100,综合形态学观察,生理生化特征及分子生物学序列分析,最终确定菌株FL-0616为发酵乳杆菌。

图5 FL-0616系统发育树
Fig. 5 Phylogenetic tree of FL-0616
表2 不同发酵方式糯玉米粉糊化特性
Table 2 Pasting characteristics of waxy corn flour fermented with different strains
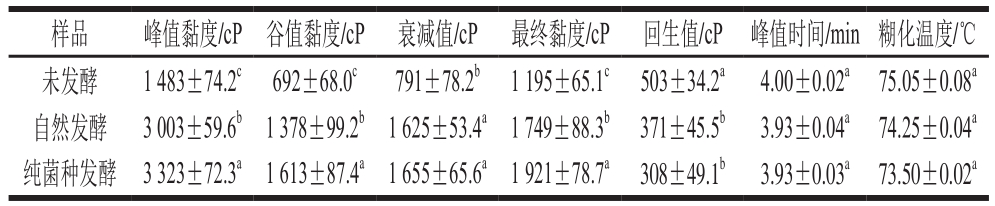
注:同列小写字母不同表示差异显著(P<0.05),下同。
由表2可见,两种方式均可显著提高糯玉米粉的峰值黏度,自然发酵和纯菌种发酵分别提高1 480 cP和1 565 cP。峰值黏度反映淀粉颗粒的膨胀性能,而影响淀粉膨胀性能的因素为支链淀粉的分子结构、淀粉的亲水性,乳酸菌纯菌种发酵使支链淀粉的含量降低,而中间及短链的支链淀粉含量相对增加,且发酵使蛋白质、脂肪明显减少,淀粉颗粒在糊化的过程中更易吸水膨胀至更大的体积,故峰值黏度增加 [23] 。自然发酵和纯菌种发酵对糯玉米粉的衰减值分别显著提高了834 cP和864 cP,衰减值可以评价淀粉热糊的稳定性,衰减值越大,表明淀粉颗粒越不稳定,其颗粒内部分子间结合力减弱,这使得颗粒更容易在剪切作用下发生破裂和瓦解 [24] 。自然发酵和纯菌种发酵对糯玉米粉的回生值分别显著降低了132 cP和195 cP,回生值可以评价淀粉的抗老化性能,回生值小,抗老化性能提高 [25] 。由于糯玉米的胚乳中100%的淀粉是支链淀粉,支链淀粉的平均外支链长度对糯玉米粉回生速率有重要影响,随着发酵的进行,支链淀粉的分支被适度降解,使回生速率降低 [26-28] 。综上所述,纯菌种发酵和自然发酵都能够对糯玉米粉起到一定的改性作用。与自然发酵相比,乳酸菌发酵显著提高糯玉米粉峰值黏度,降低回生值,改性效果较好。
表3 不同发酵方式糯玉米粉质构特性
Table 3 Texture characteristics of waxy corn flour fermented with different strains

自然发酵和乳酸菌纯菌种发酵对糯玉米粉的硬度、黏附性、胶着性等质构特性均产生了明显的影响。由表3可知,乳酸菌纯菌种发酵将糯玉米粉的硬度提高1 070 g,而自然发酵可提高275 g。硬度是描述与食品变形或穿透产品所需的力有关机械质地特性,是食品保持形状的内部结合力 [29] ,发酵能够提高糯玉米粉熟面团的硬度可能是由于自然发酵液中乳酸菌生长代谢利用了糯玉米粉的营养物质,降低蛋白含量并促进支链淀粉短链的水解从而导致淀粉凝胶网络结构加强 [30] 。乳酸菌发酵将糯玉米粉的黏附性提高332、胶着性提高600,黏附性和胶着性的增大表明面团更有韧性,在一定程度上补充了糯玉米粉类食品相对缺乏黏弹性的缺点。咀嚼性在数值上等于硬度、弹性、内聚性的乘积,原则上咀嚼性的变化受到3个因素的影响,但表中弹性和内聚性差异不显著,因此咀嚼性与硬度呈正相关。纯菌种发酵改性的糯玉米粉熟面团的胶着性和咀嚼性显著(P<0.05)高于未发酵及自然发酵。乳酸菌对糯玉米粉起到改性作用可能是因为糯玉米淀粉中含有利于乳酸菌生长代谢的营养物质,乳酸菌代谢产生的二氧化碳增多致使糯玉米粉熟面团弹性和回复性的增加 [31] ,人工添加的乳酸菌含量要比自然发酵产生的乳酸菌含量多,因此改性效果优于自然发酵。

图6 发酵前后扫描电子显微镜图片(×5 000)
Fig. 6 Scanning electron microscope pictures of waxy corn flour before and after fermentation (× 5 000)
由图6可见,发酵前后糯玉米粉的微观结构发生了变化。原糯玉米粉微观结构较为完整,自然发酵对糯玉米粉的侵蚀效果较轻,而纯菌种发酵对其结构的破坏程度较自然发酵要更加明显。自然发酵和纯种发酵过程中乳酸菌利用了糯玉米粉里淀粉、蛋白质及脂肪等物质,生长代谢过程中产酸产酶影响其分子结构对糯玉米粉起到了改性的作用。
本研究采用MiSeq技术对自然发酵菌液中细菌
16S rDNA的V3~V4区进行测序分析,分析结果显示自然发酵糯玉米碴中的细菌以乳杆菌属为主,占65.14%,与其他人研究相符。通过稀释涂布平板筛选出1株产酸能力最快、繁殖能力最佳的菌株进行鉴定,经测序比对鉴定其为发酵乳杆菌(L. fermentum),同时符合细菌多样性分析结果。通过对糊化特性和质构特性的测定,结果证明纯种发酵会显著提高峰值黏度、谷值黏度和最终黏度,显著降低回生值;蒸制前后对比显著增加硬度、黏附性、胶着性和咀嚼性从而改善糯玉米粉的粉质特性。为制作一种以发酵糯玉米粉为主要原料的食品且能够缩短自然发酵周期的发酵剂提供理论依据。此研究仅证明了乳酸菌发酵对糯玉米粉及糯玉米熟面团的改性作用,而对自然发酵过程中酵母菌对其加工特性的影响、以及发酵后糯玉米的熟面团进一步应用还有待研究。
参考文献:
[1] 郑洪建, 王义发, 沈雪芳, 等. 甜玉米糯玉米的营养价值及综合利用[J]. 上海蔬菜, 2003(4): 50-51. DOI:10.3969/j.issn.1002-1469.2003.04.045.
[2] ZHANG G, SADIQ F A, ZHU L, et al. Investigation of microbial communities of Chinese sourdoughs using culture-dependent and DGGE approaches[J]. Journal of Food Science, 2015, 80(11): M2535-M2542.DOI:10.1111/1750-3841.13093.
[3] 曹凝. 玉米食品自然发酵工艺调控与机理研究[D]. 沈阳: 沈阳农业大学, 2009.
[4] FUGL A, BERHE T, KIRAN A, et al. Characterisation of lactic acid bacteria in spontaneously fermented camel milk and selection of strains for fermentation of camel milk[J]. International Dairy Journal,2017, 73: 19-24. DOI:10.1016/j.idairyj.2017.04.007.
[5] 王凡华, 马务迢, 谭庆云, 等. 银耳干品的DNA提取及随机扩增多态性分析[J]. 食品科学, 2015, 36(4): 172-175. DOI:10.7506/spkx1002-6630-201504033.
[6] ERCOLINI D. High-throughput sequencing and metagenomics:moving forward in the culture-independent analysis of food microbial ecology[J]. Applied and Environmental Microbiology, 2013, 79: 3148-3155. DOI:10.1128/AEM.00256-13.
[7] 宋艳波, 吴国良, 牛洪斌. 改良CTAB法在核桃叶片基因组DNA提取中的应用研究[J]. 山西农业大学学报(自然科学版), 2011, 31(2):109-112. DOI:10.3969/j.issn.1671-8151.2011.02.003.
[8] FADROSH D W, MA B, GAJER P, et al. An improved dual-indexing approach for multiplexed 16S r RNA gene sequencing on the Illumina Mi Seq platform[J]. Microbiome, 2014, 2: 6-12. DOI:10.1186/2049-2618-2-6.
[9] SHAUFI M A M, SIEO C C, CHONG C W, et al. Deciphering chicken gut microbial dynamics based on high-throughput 16S rRNA metagenomics analyses[J]. Gut Pathogens, 2015, 7: 4-15.DOI:10.1186/s13099-015-0051-7.
[10] CAPORASO J G, LAUBER C L, WALTERS W A, et al. Ultra-highthroughput microbial community analysis on the Illumina Hi Seq and Mi Seq platforms[J]. The International Society for Microbial Ecology Journal, 2012, 6: 1621-1624. DOI:10.1038/ismej.2012.8.
[11] KOZICH J J, WESTCOTT S L, BAXTER N T, et al. Development of a dual-index sequencing strategy and curation pipeline for analyzing amplicon sequence data on the Mi Seq Illumina sequencing platform[J]. Applied and Environmental Microbiology, 2013, 79:5112-5120. DOI:10.1128/AEM.01043-13.
[12] LANGILLE M G I, ZANEVELD J, CAPORASO J G, et al. Predictive functional profiling of microbialcommunities using 16S rRNA marker gene sequences[J]. Nature Biotechnology, 2013, 31(9): 814-821.DOI:10.1007/s10532-015-9731-8.
[13] CAPORASO J G, BITTINGER K, BUSHMAN F D, et al. PyNAST:a flexible tool for aligning sequences to a template alignment[J].Bioinformatics, 2010, 26(2): 266-267. DOI:10.1038/nbt.2676.
[14] 张小美, 楼秀玉, 顾青. 1 株产细菌素乳酸菌的鉴定和细菌素的分离纯化[J]. 中国食品学报, 2013, 13(12): 181-186. DOI:10.16429/j.1009-7848.2013.12.039.
[15] 沈萍, 李秀荣, 李广武. 微生物学实验[M]. 北京: 高等教育出版社,2001: 69-77.
[16] GRECO M, MAZZETTE R. Evolution and identification of lactic acid bacteria isolated during the ripening of sar-dinian sausage[J].Meat Science, 2005, 69: 733-739. DOI:10.4028/www.scientific.net/AMR.361-363.701.
[17] 冯广达, 陈美标, 羊宋贞, 等. 用于PCR扩增的细菌DNA提取方法比较[J]. 华南农业大学学报, 2013, 34(3): 439-442. DOI:10.7671/j.issn.1001-411X.2013.03.031.
[18] 曲丰发, 蔡畅, 郑献进, 等. 16S rDNA建立种特异性PCR快速检测鸭疫里默氏菌[J]. 微生物学报, 2006, 46(1): 13-17. DOI:10.3321/j.issn:0001-6209.2006.01.004.
[19] 裴斐, 仲磊, 杨文建, 等. 蛹虫草复合谷物杂粮膨化产品的工艺优化及糊化特性[J]. 食品科学, 2016, 37(24): 47-54. DOI:10.7506/spkx1002-6630-201624007.
[20] 何财安, 张珍, 刘航, 等. 苦荞多酚对苦荞淀粉和小麦淀粉理化性质的影响[J]. 食品科学, 2017, 38(9): 66-71. DOI:10.7506/spkx1002-6630-201709011.
[21] 凌代文. 乳酸细菌分类鉴定及试验方法[M]. 北京: 中国轻工业出版社, 1999: 5-7.
[22] 布坎南R E, 吉本斯N E. 伯杰细菌鉴定手册[M]. 8版. 北京: 科学出版社, 1984.
[23] CHANG Y H, LIN C L, CHEN J C. Characteristics of mung bean starch isolated by using lactic acid fermentation solution as the steeping liquor[J]. Food Chemistry, 2006, 99(4): 794-802.DOI:10.1016/j.foodchem.2005.07.060.
[24] 赵云霞, 熊柳, 孙庆杰. 自然发酵对糯米糊化性质的影响[J]. 食品科学, 2012, 33(11): 237-240. DOI:10.7506/spkx1002-6630-201211033.
[25] PHOTHISET S, CHAROENREIN S. Morphology and physicochemical changes in rice flour during rice paper production[J].Food Research International, 2007, 40(2): 266-272. DOI:10.1016/j.foodres.2006.06.002.
[26] 王帅, 程建军, 倪春蕾, 等. 乳酸菌对玉米加工特性的影响及基于均匀设计优化玉米发酵条件[J]. 食品工业科技, 2016, 37(13): 183-187;191. DOI:10.13386/j.issn1002-0306.2016.13.028.
[27] 许梅. 复合方法提高糯玉米粉品质特性的研究[D]. 大庆: 黑龙江八一农垦大学, 2009.
[28] 寇芳, 康丽君, 宁冬雪, 等. 生物发酵处理对小米淀粉分子结构及糊化特性的影响[J]. 食品科学, 2017, 38(12): 36-42. DOI:10.7506/spkx1002-6630-201712006.
[29] 周显青, 胡育铭, 张玉荣, 等. 汤圆粉团制作方法比较及其对蒸煮品质的影响[J]. 粮食与饲料工业, 2014, 12(4): 30-33; 37. DOI:10.7633/j.issn.1003-6202.2014.04.009.
[30] ABDEL-RAHMAN M A, TASHIEO Y, SONOMOTO K. Lactic acid production from lignocellulose-derived sugars using lactic acid bacteria: overview and limits[J]. Journal of Biotechnology, 2011,156(4): 286-301. DOI:10.1016/j.jbiotec.2011.06.017.
[31] 朱津津, 潘治利, 谢新华, 等. 汤圆TPA质构特性测试条件的优化[J]. 食品科学, 2013, 34(6): 171-174. DOI:10.7506/spkx1002-6630-201306038.
Analysis of Bacterial Diversity in Naturally Fermented Waxy Corn and Effects of Pure Fermentation on Its Processing Characteristics
ZHANG Dali, FENG Yanhe, LIU Bingli, PAN Cong, FENG Ling, ZHENG Mingzhu*
(National Engineering Laboratory for Wheat and Corn Further Processing, College of Food Science and Engineering,Jilin Agricultural University, Changchun 130118, China)
Abstract: Objective: To study the bacterial diversity of waxy corn during natural fermentation and the influence of lactic acid bacterial fermentation on processing properties of waxy corn meal. Methods: The V3–V4 variable region of bacterial 16S rDNA genes from naturally fermented waxy corn was sequenced using MiSeq platform and the bacterial diversity was evaluated. One dominant strain was screened out and identified. Additionally, the effects of pure strain and natural fermentations on the pasting and textural properties of waxy corn meal were compared as well. Results: Lactobacillus fermentum was identified as the dominant bacteria by morphological, physiological and biochemical characteristics and 16S rDNA gene sequence analysis. Pure fermentation significantly increased the peak viscosity, final viscosity and breakdown value and significantly reduced setback value; furthermore, steaming significantly increased hardness, adhesiveness, and chewiness, thereby improving the farinographic properties of waxy corn meal.
Keywords: waxy corn; polymorphism analysis; 16S rDNA; pasting properties; texture characteristics
ZHANG Dali, FENG Yanhe, LIU Bingli, et al. Analysis of bacterial diversity in naturally fermented waxy corn and effects of pure fermentation on its processing characteristics[J]. Food Science, 2018, 39(18): 109-114. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201818017. http://www.spkx.net.cn
文章编号: 1002-6630(2018)18-0109-06引文格式:
张大力, 冯艳鹤, 刘炳利, 等. 自然发酵糯玉米中细菌多样性分析及纯菌种对其加工特性的影响[J]. 食品科学, 2018,39(18): 109-114. DOI:10.7506/spkx1002-6630-201818017. http://www.spkx.net.cn
中图分类号: TS213.4
文献标志码: A
*通信作者简介: 郑明珠(1979—),女,副教授,博士,研究方向为谷物食品开发。E-mail:zhengmzhu@163.com
DOI: 10.7506/spkx1002-6630-201818017
基金项目: “十三五”国家重点研发计划重点专项(2016YFD0400401)
第一作者简介: 张大力(1968—),男,副教授,硕士,研究方向为多谷物食品研究与开发。 E-mail:spzdl@126.com
收稿日期: 2017-07-14