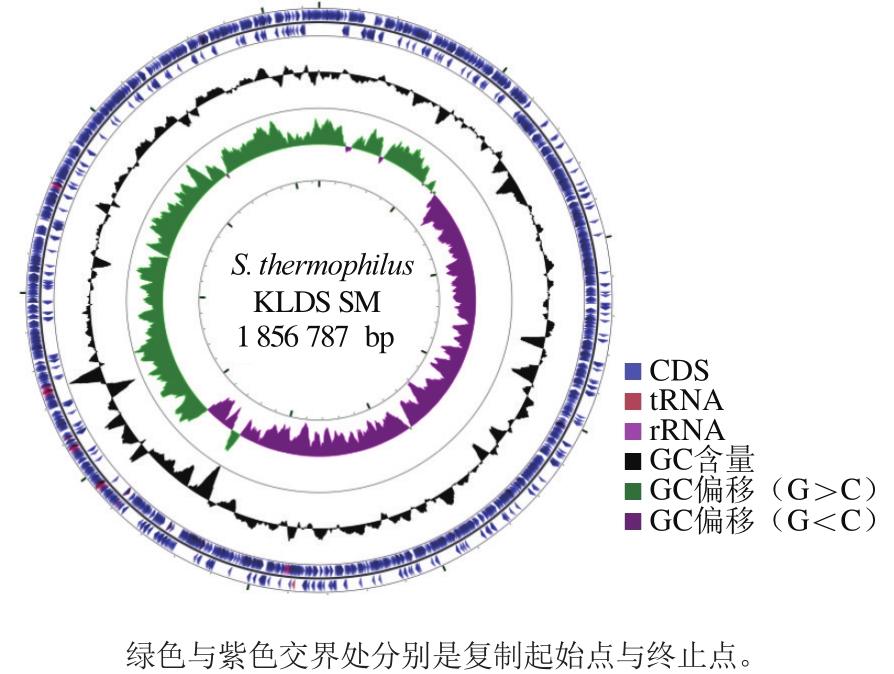
图1 菌株KLDS SM基因组圈图
Fig. 1 Circular genome map of strain KLDS SM
李柏良 1 ,丁秀云 1,2 ,靳 妲 1 ,刘 飞 1 ,蒙月月 1 ,李 娜 1 ,赵 莉 1 ,霍贵成 1, *
(1.东北农业大学 乳品科学教育部重点实验室,黑龙江 哈尔滨 150030;2.广州基迪奥生物科技有限公司,广东 广州 510000)
摘 要: 为从遗传水平上探究嗜热链球菌KLDS SM蛋白质水解和氨基酸合成的能力,首先基于Illumina Hiseq 2500与Pacbio RSII测序平台对嗜热链球菌KLDS SM进行全基因组测序并绘制基因组图谱;随后从胞外蛋白酶、转运系统、胞内肽酶以及氨基酸合成等方面所涉及的基因进行生物信息学分析;最后对15 株已完成全基因组测序的嗜热链球菌的氨基酸合成能力进行比较基因组学研究。结果表明:菌株KLDS SM的基因组由一个1 856 787 bp环状染色体组成,GC含量为39.08%,含有1 732 个蛋白质编码基因;菌株KLDS SM具有完整的蛋白水解系统和8 种氨基酸的合成能力;15 株嗜热链球菌在氨基酸合成方面上相对保守,仅在组氨酸合成途径存在较大的差异。本研究为该菌株氮代谢能力的挖掘提供了理论依据,并在将其开发为发酵剂方面上具有一定指导意义。
关键词: 嗜热链球菌;基因组;生物信息分析;蛋白质水解;氨基酸合成
嗜热链球菌是“公认安全性(GRAS)”菌株,广泛应用于发酵乳制品的工业生产中,是第二重要的工业用乳酸菌菌种,市场价值约400亿 美元 [1] 。嗜热链球菌具有快速产酸的能力,可以缩短发酵乳制品的凝乳时间,改善质地,同时提高营养价值 [2-4] 。
随着测序技术逐渐成熟和价格降低,微生物基因组测序基本普遍。2004年首次完成了嗜热链球菌LMG 18311与嗜热链球菌CNRZ1066的全基因组测序工作 [1] 。截至2017年2月,已有15 株嗜热链球菌的基因组已经完成测序。基因组序列分析可以更加深入研究一些代谢途径的遗传结构,如氨基酸合成 [5] 、蛋白水解系统 [5] 、抗噬菌体 [6] 、叶酸的生物合成及生物膜的形成 [7] 。
乳酸菌因缺乏必要的氨基酸代谢途径,不能从头合成生长所需的某些氨基酸。因此,乳酸菌需要从外界环境中获取相应的活性物质。乳酸菌可以利用蛋白水解系统水解牛乳中的酪蛋白供应生理代谢需要的肽类与氨基酸,同时部分氨基酸的代谢可进一步转化为醛类、醇类等风味物质,对发酵乳制品的风味物质形成有重要作用 [8] 。
酪蛋白水解过程分为3 个阶段。首先,胞外蛋白酶将乳制品中的酪蛋白水解成肽类物质;其次,通过ABC型寡肽转运系统(Opp)将肽类物质转运至胞内;最后,在胞内丰富的肽酶,如二肽酶、羧肽酶及内肽酶等的作用下形成游离的氨基酸,可以进入代谢途径或者用于合成蛋白质以供菌体需求。其中Opp系统是蛋白水解系统的重要组成部分,由1 个负责膜连接寡肽结合蛋白(OppA),2个负责转运的跨膜蛋白(OppB、OppC)和2个ATP结合蛋白(OppD、OppF)构成 [8-9] 。
嗜热链球菌KLDS SM是本实验室从内蒙古牧民家庭以传统方法自制的酸奶中分离鉴定得到的。前期实验研究发现该菌株具有快速产酸和高产黏的特性。为更加深入分析该菌株的性能,本研究采用二代与三代测序结合的策略对该菌株进行全基因组测序,基于生物信息学分析该菌株蛋白质水解系统关键控制基因及氨基酸合成途径的基因分布情况,同时利用比较基因组学分析该菌株与其他菌株在氨基酸合成方面上的差异。为该菌株后续更加合理的应用提供了理论依据,具有一定指导意义。
M17肉汤培养基 青岛高科园海博生物技术有限公司;细菌基因组提取试剂盒 北京天根生物技术有限公司;其他试剂均为国产分析纯。
嗜热链球菌KLDS SM由东北农业大学乳品科学教育部重点实验室工业微生物菌种保藏中心(KLDSDICC)提供,且通过16S rRNA测序鉴定。选择已完成测序的14 株嗜热链球菌进行比较基因组学分析,序列从NCBI(https://www.ncbi.nlm.nih.gov/genome/genomes/420?)下载。14 株菌株信息:菌株LMG 18311(CP000023)、菌株CNRZ1066(CP000024)、菌株LMD-9(CP000419、CP000420、CP000421)、菌株ND03(CP002340)、菌株MN-ZLW-002(CP003499)、菌株ASCC 1275(CP006819)、菌株MN-BM-A02(CP010999)、菌株SMQ-301(CP011217)、菌株MNBM-A01(CP012588)、菌株S9(CP013939)、菌株JIM 8232(FR875178)、菌株CS8(CP016439)、菌株KLDS 3.1003(CP016877)与菌株ND07(CP016394) [1,10-18] 。
LDZF-50KB-II立式蒸汽灭菌器 上海申安医疗器械厂;CJ-2D超净工作台 天津泰斯特仪器有限公司;DHP-927型电热恒温培养箱 上海一恒科技有限公司;GL-20G-II离心机 上海安亭科学仪器厂;DYY-10C电泳仪 北京六一仪器厂;PL2002电子天平 梅特勒-托利多仪器(上海)有限公司。
1.3.1 菌株的活化及基因组提取
将甘油保藏的菌株KLDS SM以2%的体积分数接种于M17液体培养基,42 ℃培养24 h,转接2 次,16 h后用于基因组提取。
菌株KLDS SM基因组提取按照细菌基因组提取试剂盒说明书进行。取5 µL基因组DNA样品进行1%琼脂糖凝胶电泳,检测提取的质量及完整性。
1.3.2 全基因组测序及组装
采用二代Illumina Hiseq 2500(500 bp,PE125)平台与三代Pacbio RSII(20 K)平台联合测序。过滤Hiseq数据除去低质量、去除接头、N含量过高的reads;过滤Pacbio的polymerase reads数据除去低质量reads、接头序列,并将polymerase reads转换为subreads。对每个ZMW(zero-mode waveguides)中的subreads去冗余等处理得到CCS(circular consensus sequences)序列,对CCS做自纠正并组装序列。使用SMRT Analysis v2.3.0流程中的RS_HGAP_Assembly3 [19] 软件将PacBio序列组装成完整的连续的contig,根据contig两端是否已经有Overlap判断基因组是否成环。基于Illumina Hiseq 2500数据利用两轮分析方法对contig进行单碱基纠错,即采用GATK分析流程对Contig进行第1轮纠错分析,采用软件SOAPsnp v1.05 [20] 与SOAPindel v1.08 [21] 对第1轮的纠错结果进行第2轮纠错分析。
1.3.3 基因组注释
组装成环的基因组序列提交到NCBI。采用NCBI原核基因组注释流程PGAP [22] 及RAST Server [23] 进行全基因组注释。注释结果及序列从NCBI(ftp://ftp.ncbi.nlm.nih.gov/genomes/all/GCA/001/663/795/GCA_001663795.1_ASM166379v1/)下载。基因组中蛋白序列通过WebMGA网站 [24] 进行COG(cluster of orthologous group)注释(RPSBLAST,e-value<1e -5 );在KAAS网站 [25] ,采用BBH(bi-directional best hit)方法对基因组中的蛋白编码基因进行KEGG(Kyoto encyclopedia of genes and genomes)在线注释,生物选择“eco,bsu,sau,lmo,lla,spy,spn,ste,lpl,lpj,ljo,ljf,lac,lsa,lsl,ldb,lbu,lbr,lca,lcb,lga,lre,lrf,lhe,lfe,lrh,lrl,stc,stl” [26] 。
1.3.4 生物信息分析
采用CGView Server [27] 绘制基因组圈图;蛋白质水解与氨基酸合成代谢途径参照KEGG通路数据库(http://www.genome.jp/kegg/pathway.html)进行挖掘;蛋白质序列从Uniprot数据库 [28] 下载;使用本地BLASTP(2.2.31+)确认各基因在该基因组中存在情况,取最优比对结果。
2.1.1 组装结果
经Illumina Hiseq 2500(500 bp,PE125)平台测序得到467 Mb原始数据,过滤后获得401 Mb Clean Data。Pacbio RSII(20 K)平台测序得到38 403 条polymerase reads,平均长度14 976 bp,共643 231 812 bp;过滤后获得63 855 条subreads,平均长度8 972 bp,共572 949 237 bp。经组装、纠错、成环判断等过程获得一条长为1 856 787 bp完整、连续且成环的contig。
2.1.2 基因组的基本特征

图1 菌株KLDS SM基因组圈图
Fig. 1 Circular genome map of strain KLDS SM
菌株KLDS SM的全基因组序列已提交到GenBank数据库,登录号为:CP016026。基因组图谱可以直观体现基因组功能注释结果和GC偏移情况,图1为菌株KLDS SM全基因组圈图,说明菌株KLDS SM全基因组测序已经达到完成图水平,且该基因组为双链环状分子。
表1 嗜热链球菌基因组基本信息比较
Table 1 Comparison of genomic features of Streptococcus thermophilus
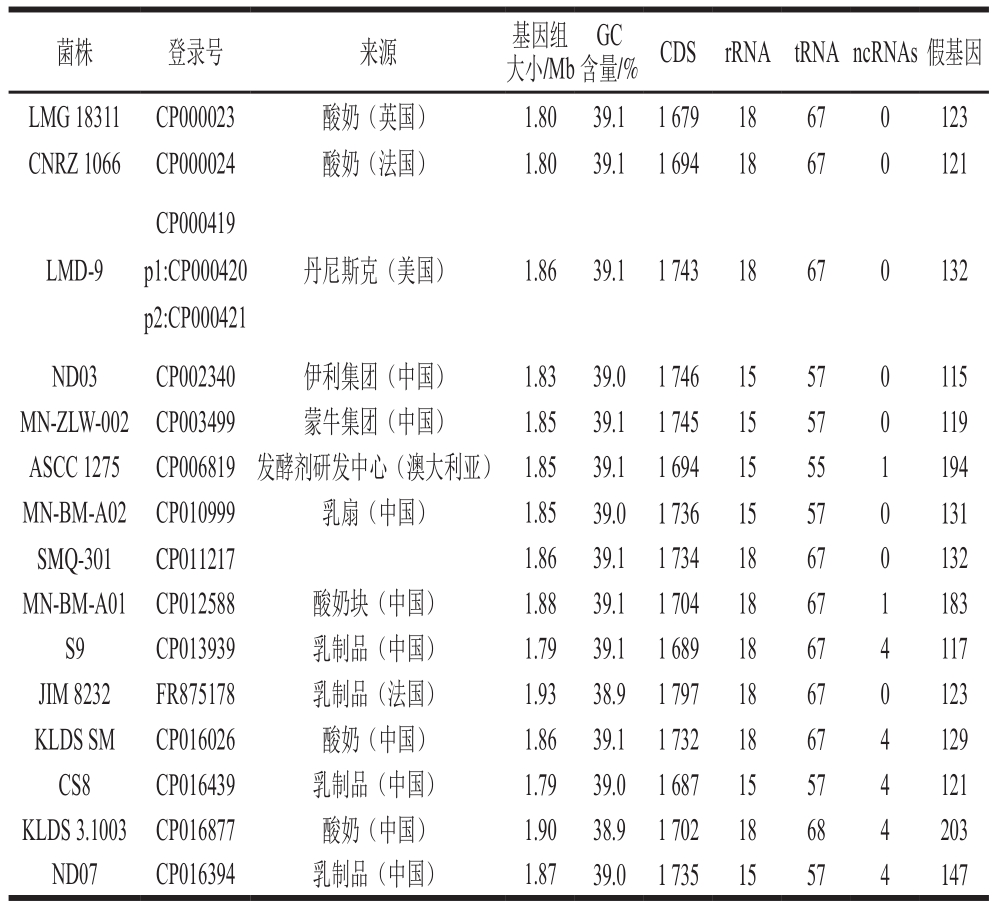
如表1所示,同绝大多数已测序的嗜热链球菌一样,菌株KLDS SM基因组中不存在质粒,仅由一条环状的染色体组成,全长1 856 787 bp,平均GC含量为39.08%。在基因组中共预测1 732 个蛋白质编码基因(protein coding genes,CDS),其中129 个(6.9%)基因发生突变为假基因,CDS总长1 559 226 bp,占全基因组序列的83.97%,基因的平均长度为838 bp。此外,基因组包含6 个完整的rRNA基因操纵元,其中一个操纵元与DNA复制的方向相反,67 个tRNA基因及4 个ncRNA。比较发现,菌株KLDS SM有较多的rRNA基因操纵子、tRNA基因及ncRNA,较少的假基因。
2.1.3 COG注释
通过WebMGA网站对菌株KLDS SM基因组中具有生物学功能的蛋白编码基因进行COG注释。结果表明共有1 400 个蛋白编码基因注释到COG数据库。如图2所示,分别有41、16、181、73、81、56、31、142、89、154、79、6、49、82、10、175、130、47、23、43 个基因注释到分类C~V。其中注释到氨基酸转运和代谢(12.9%),翻译、核糖体结构和生物合成(10.1%),复制、重组和修复(11%),一般功能(12.5%)及具有潜在功能的假定蛋白(9.3%)5 种分类的基因数较多。另有332 个(19.2%)具有潜在生物学功能的基因未被注释到数据库中。

图2 菌株KLDS SM基因组蛋白编码基因的COG功能分类
Fig. 2 COG functional classification of protein-encoding genes in strain KLDS SM genome
2.2.1 胞外蛋白水解
牛乳中的氮源多以酪蛋白的形式存在,很少存在游离的氨基酸。菌株KLDS SM基因组中存在一个完整的编码锚定细胞壁的丝氨酸蛋白酶PrtS的基因A9497_00420。PrtS是降解牛乳中酪蛋白最重要的蛋白酶,由1 618 个氨基酸组成且高度保守,与猪链球菌编码的PrtS的氨基酸序列有96%的一致性。如图3所示,该蛋白从N-末端至C-末端依次为YSIRK_signal、Peptidases_S8_C5a_Peptidase、PA_C5a_like、fn3_5、FIVAR与Gram_pos_anchor结构域,其中N-末端35 个氨基酸为信号肽序列。
图3 菌株KLDS SM的 PrtS结构
Fig. 3 PrtS architecture of strain KLDS SM
2.2.2 转运系统
如表2所示,菌株KLDS SM基因组中存在两个Opp系统,且二者结构上有所不同。其中一个Opp长约10 kb,由6 个基因组成(A9497_03140~A9497_03160,A9497_03170),由2 个oppA与oppB、oppC、oppD、oppF各1 个组成,且在oppA与oppB之间相反链上有一编码转座酶的基因。序列比对发现2 个oppA仅有86%的一致性。另一个Opp长约4 kb,同样以oppA、oppB、oppC、oppD、oppF顺序依次排列,仅有一个oppA,但该系统每个基因都发生了不同程度的突变,如oppA与oppB序列截短,oppC与oppD序列发生多处移码,oppF则因突变提前终止。因此这个Opp不具有转运功能。
表2 菌株KLDS SM的肽与氨基酸转运系统
Table 2 Peptide and amino acid transport systems of strain KLDS SM

同时,基因组中还存在许多编码转运氨基酸的基因,以供菌体生理代谢的需求。L-谷氨酰胺ABC型转运系统由1 个谷氨酰胺转运系统底物结合蛋白(GlnH)、1 个渗透酶蛋白(GlnP)及1 个ATP结合蛋白(GlnQ)组成,菌株KLDS SM基因组上存在完整的编码转运L-谷氨酰胺的ABC型转运系统的基因,并且每个基因都有2~3 个拷贝。支链氨基酸为亮氨酸、异亮氨酸与缬氨酸的总称,其ABC型转运系统由1 个支链氨基酸转运系统底物结合蛋白(LivK)及相应的2 个渗透酶蛋白(LivH、LivM)、2 个ATP结合蛋白(LivG、LivF)组成,该菌基因组中同样具有一个完整的该转运系统的编码基因。该菌可以通过蛋氨酸ABC型转运系统转运蛋氨酸,该系统同样由3 个蛋白构成,即蛋氨酸ABC型转运系统底物结合蛋白(MetQ)与相应的渗透酶蛋白(MetI)、ATP结合蛋白(MetN)。菌株可以转运亚精胺/腐胺,该转运系统由1 个亚精胺/腐胺ABC型转运系统底物结合蛋白(PotD)、2 个渗透酶蛋白(PotB、PotC)与ATP结合蛋白(PotA)构成,其中编码PotD的基因有两个拷贝。基因组中缺失编码赖氨酸ABC型转运系统的渗透酶蛋白(LysX2),因此该菌无法转运赖氨酸。除此之外,该菌株可以编码一些渗透酶及电化学势驱动转运体转运相应的氨基酸供菌体使用。
2.2.3 胞内肽酶
表3 菌株KLDS SM的肽酶
Table 3 Peptidases of strain KLDS SM

如表3所示,在菌株KLDS SM基因组中共预测出21 个肽酶编码基因,其中3 个基因编码胞外肽酶参与细胞壁的形成,剩余的18 个基因均编码胞内肽酶。这些胞内肽酶包括8 个氨肽酶、1 个羧肽酶、3 个二肽酶与4 个内肽酶,且仅有一个编码二肽酶PepD的基因因突变失去功能,其余的肽酶均具有生物学功能。
菌株KLDS SM的20 种氨基酸生物合成途径注释结果如图4所示,菌株KLDS SM有完整的组氨酸、色氨酸、丝氨酸、半胱氨酸、甘氨酸、缬氨酸、亮氨酸与丙氨酸生物合成途径,因此该菌具有合成这8 种氨基酸的能力。此外,基因组中缺失编码芳香族氨基酸转氨酶的基因,无法将谷氨酸的氨基转移到苯丙酮酸及4-羟基苯丙酮酸上,即无法合成酪氨酸与苯丙氨酸。缺失编码丙酮酸羧化酶PC(EC:6.4.1.1)的基因,不能将丙酮酸合成草酰乙酸,缺失编码TCA循环中的多种酶的基因不能为天冬氨酸的合成提供草酰乙酸,并且缺失编码天冬氨酸氨基转移酶(EC:2.6.1.1)的基因,无法转氨合成天冬氨酸。具有编码将游离的  结合到天冬氨酸形成天冬酰胺的天冬氨酸-氨连接酶的基因A9497_08040(asnA,EC:6.3.1.1)。具有催化天冬氨酸转化成天冬氨酸-β-半醛,合成苏氨酸、异亮氨酸主链、甲硫氨酸及二氨基庚二酸主链及甲硫氨酸的酶的编码基因,但因缺失编码D-柠苹酸合成酶的基因无法为异亮氨酸、二氨基庚二酸的合成提供相应的侧链,因此不能合成异亮氨酸、赖氨酸。该菌具有编码由草酰乙酸合成α-酮戊二酸以及由α-酮戊二酸合成谷氨酸、谷氨酰胺、精氨酸与脯氨酸的一系列酶的编码基因。
结合到天冬氨酸形成天冬酰胺的天冬氨酸-氨连接酶的基因A9497_08040(asnA,EC:6.3.1.1)。具有催化天冬氨酸转化成天冬氨酸-β-半醛,合成苏氨酸、异亮氨酸主链、甲硫氨酸及二氨基庚二酸主链及甲硫氨酸的酶的编码基因,但因缺失编码D-柠苹酸合成酶的基因无法为异亮氨酸、二氨基庚二酸的合成提供相应的侧链,因此不能合成异亮氨酸、赖氨酸。该菌具有编码由草酰乙酸合成α-酮戊二酸以及由α-酮戊二酸合成谷氨酸、谷氨酰胺、精氨酸与脯氨酸的一系列酶的编码基因。
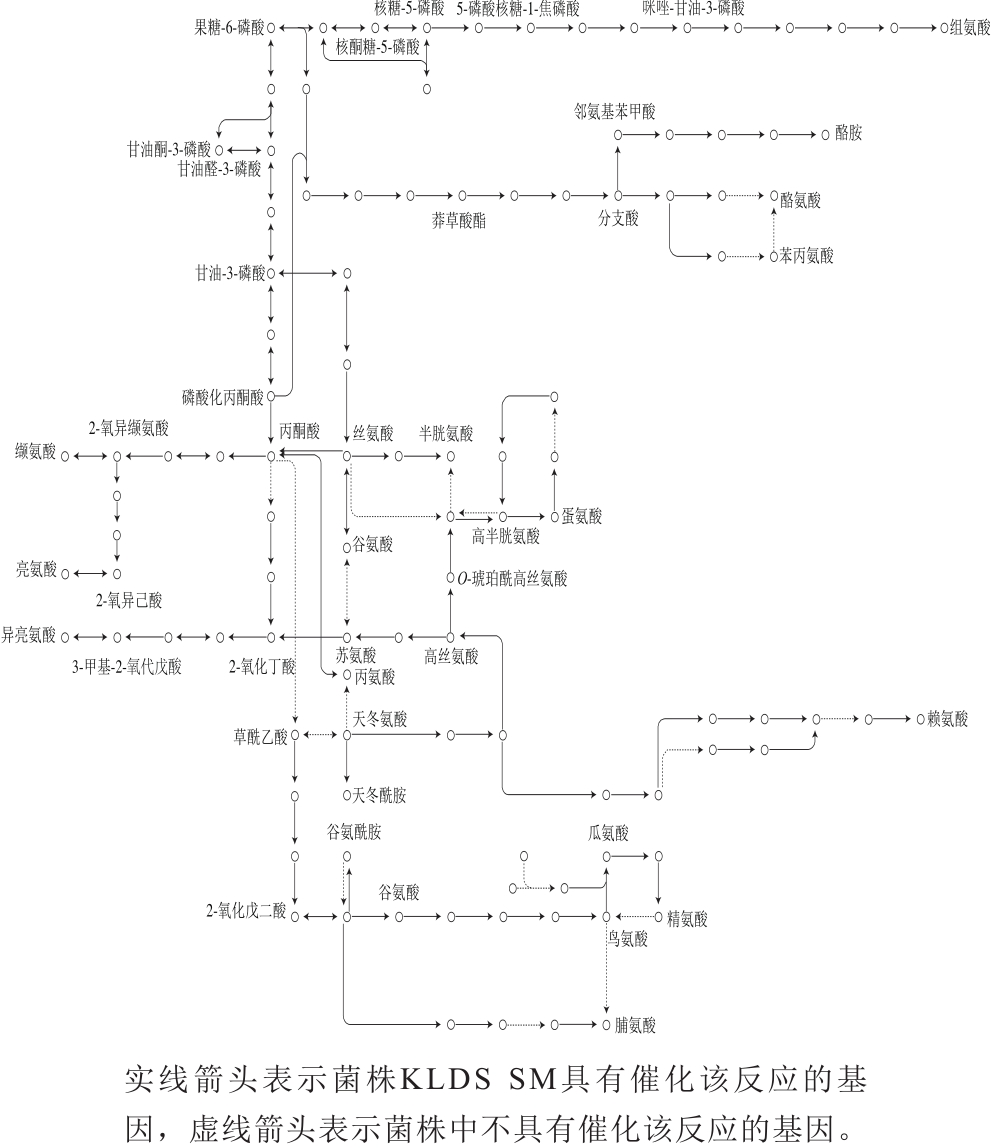
图4 菌株KLDS SM的氨基酸生物合成
Fig. 4 Amino acid biosynthesis in strain KLDS SM
比较基因组分析发现,15 株嗜热链球菌中氨基酸合成情况相对保守。除了菌株LMG 18311、菌株CNRZ1066、菌株S9与菌株CS8因缺失编码组氨酸途径的多种酶的基因而无法合成组氨酸外,15 株菌株合成氨基酸的能力相似。组氨酸的酶促合成有9 种酶参与反应,即由5-磷酸核糖-1-焦磷酸作为底物,在酶ATP磷酸核糖转移酶、焦磷酸水解酶、磷酸核糖-AMP环化水解酶、磷酸核糖亚氨甲基-5-氨基咪唑-4-羧酰胺核苷酸同分异构酶、谷氨酰胺氨基转移酶、咪唑甘油磷酸脱水酶、L-组氨醇磷酸氨基转移酶、L-组氨醇磷酸磷酸酶及组氨醇脱氢酶的作用下合成L-组氨酸。而这9 种酶的编码基因在菌株KLDS SM(A9497_02145~A9497_02190,GC含量42.6%)及另外10 株菌的基因组中常常成簇存在。
嗜热链球菌常与保加利亚乳杆菌一起作为发酵剂,广泛应用于酸奶、奶酪和其他乳制品的工业生产中 [29] 。嗜热链球菌具蛋白水解酶活性,快速增长,产生胞外多糖、细菌素、风味物质及抗噬菌体等特点,直接或间接影响着发酵乳制品的质量 [2-4] 。其中蛋白水解酶活性与风味物质的产生能力是筛选生产菌株的关键特性。风味是决定乳制品的可接受性的关键因素,嗜热链球菌蛋白水解系统降解酪蛋白是风味物质的重要前体,并且水解活性与产酸能力紧密相关 [3] 。
由于分子生物学研究手段的限制,传统的乳酸菌研究多集中于根据生理生化实验及常规的分子生物学实验探究乳酸菌的特性,只能以单个基因和途径为目标,很难对多基因及代谢网络进行深入研究 [30] 。随着基因组测序技术的发展,研究人员可以利用生物信息学的手段充分挖掘乳酸菌基因组信息,2004年,Bolotin等 [1] 通过比较基因组分析,证明了由于嗜热链球菌长期在乳生态位中生长,其有害基因已经失活或丢失;2009年,Pastink等 [31] 基于全基因组构建了嗜热链球菌LMG18311的代谢模型;2013年,Flahaut等 [32] 构建了乳酸乳球菌MG1363的代谢模型,并应用于风味形成途径分析;2017年,Veronica等 [33] 对8 株已完成基因组测序的嗜热链球菌进行了比较基因组学和生理学研究。基于此,本研究从遗传水平上分析了菌株KLDS SM在蛋白质水解和氨基酸生物合成2 个方面相关的一系列基因。菌株KLDS SM基因组中有2 个转运寡肽的Opp系统,其中一个完整的Opp系统,而且该系统有两个oppA,这或许与更加有效地捕捉底物有关,但另一个Opp系统中的每个基因发生了不同程度的缺失、移码突变及无义突变,说明该转运系统较早开始退化。除此之外,该菌株还可以通过ABC型转运系统转运L-谷氨酰胺(每个基因具有2 个拷贝)、支链氨基酸、蛋氨酸、亚精胺/腐胺(底物结合蛋白2 个拷贝),以及一些渗透酶及电化学势驱动转运体转运相应的氨基酸供菌体使用。
本研究共预测了18 个编码胞内肽酶基因,包括氨肽酶、羧肽酶、二肽酶与内肽酶,且仅有2 个基因发生突变。与大多数乳酸菌不同的是,嗜热链球菌基因组中编码肽酶的基因并不形成操纵子,而且也不位于编码氨基酸/肽类转运的基因的附近,这点与Goh等 [34] 分析的结果一致。此外,该菌株基因组中具有编码合成8 种氨基酸组氨酸、色氨酸、丝氨酸、半胱氨酸、甘氨酸、缬氨酸、亮氨酸和丙氨酸所需酶的基因,其中缬氨酸、亮氨酸和色氨酸为人体必需氨基酸。而在某些氨基酸合成路径中,如酪氨酸、苯丙氨酸、异亮氨酸与赖氨酸,仅因基因组中缺失某个催化酶的编码基因而无法合成。比较基因组分析发现,15 株嗜热链球菌中氨基酸合成情况相对保守,仅在组氨酸合成途径存在较大的差异。
菌株KLDS SM的基因组由一个1 856 787 bp环状染色体组成,GC含量为39.08%,含有1 732 个CDS。从基因组水平分析,菌株KLDS SM具有完整的蛋白水解系统,并可以合成组氨酸、色氨酸、丝氨酸、半胱氨酸、甘氨酸、缬氨酸、亮氨酸和丙氨酸8 种氨基酸。比较基因组分析发现,不同嗜热链球菌菌株间的氨基酸合成能力较为保守,仅在组氨酸合成途径存在较大的差异。
参考文献:
[1] BOLOTIN A, QUINQUIS B I T, RENAULT P, et al. Complete sequence and comparative genome analysis of the dairy bacterium Streptococcus thermophilus[J]. Nature Biotechnology, 2004, 22(12):1554. DOI:10.1038/nbt 1034.
[2] DE VUYST L, WECKX S, RAVYTS F E D E, et al. New insights into the exopolysaccharide production of Streptococcus thermophilus[J].International Dairy Journal, 2011, 21(9): 586-591. DOI:10.1016/j.idairyj.2011.03.016.
[3] GALIA W, PERRIN C, GENAY M, et al. Variability and molecular typing of Streptococcus thermophilus strains displaying different proteolytic and acidifying properties[J]. International Dairy Journal,2009, 19(2): 89-95. DOI:10.1016/j.idairyj.2008.08.004.
[4] ZOTTA T, RICCIARDI A, CIOCIA F, et al. Diversity of stress responses in dairy thermophilic streptococci[J]. International Journal of Food Microbiology, 2008, 124(1): 34-42. DOI:10.1016/j.ijfoodmicro.2008.02.024.
[5] HOLS P, HANCY F E D E, FONTAINE L, et al. New insights in the molecular biology and physiology of Streptococcus thermophilus revealed by comparative genomics[J]. FEMS Microbiology Reviews,2005, 29(3): 435-463. DOI:10.1016/j.femsre.2005.04.008.
[6] LI W, BIAN X, EVIVIE S E, et al. Comparative analysis of clustered regularly interspaced short palindromic repeats (CRISPR) of Streptococcus thermophilus St-I and its bacteriophage-insensitive mutants (BIM) derivatives[J]. Current Microbiology, 2016, 73(3): 393-400. DOI:10.1007/s00284-016-1076-y.
[7] COUVIGNY B, THÉRIAL C, GAUTIER C, et al. Streptococcus thermophilus biofilm formation: a remnant trait of ancestral commensal life?[J]. PLoS ONE, 2015, 10(6): e128099. DOI:10.1371/journal.pone.0128099.
[8] 白风翎, 张柏林, 赵宏飞. 乳酸菌蛋白代谢研究进展[J]. 食品科学,2010, 31(19): 381-384.
[9] 郑华军. 保加利亚乳酸杆菌工业生产菌株2038的基因组学分析[D].上海: 复旦大学, 2010.
[10] MAKAROVA K, SLESAREV A, WOLF Y, et al. Comparative genomics of the lactic acid bacteria[J]. Proceedings of the National Academy of Sciences of the United States of America, 2007, 189(4):1199-1208. DOI:10.1073/pnas.0607117103.
[11] SUN Z, CHEN X, WANG J, et al. Complete genome sequence of Streptococcus thermophilus strain ND03[J]. Journal of Bacteriology,2011, 193(3): 793. DOI:10.1128/JB.01374-10.
[12] KANG X, LING N, SUN G, et al. Complete genome sequence of Streptococcus thermophilus Strain MN-ZLW-002[J]. Journal of Bacteriology, 2012, 194(16): 4428-4429. DOI:10.1128/JB.00740-12.
[13] WU Q, TUN H M, LEUNG C C, et al. Genomic insights into high exopolysaccharide-producing dairy starter bacterium Streptococcus thermophilus ASCC 1275[J]. Scientific Reports, 2014, 4(7500): 4974.DOI:10.1038/srep04974.
[14] SHI Y, CHEN Y, LI Z, et al. Complete genome sequence of Streptococcus thermophilus MN-BM-A02, a rare strain with a high acid-producing rate and low post-acidification ability[J].Genome Announcements, 2015, 3(5): 2090-2094. DOI:10.1128/genomeA.00979-15.
[15] LABRIE S J, TREMBLAY D M, PLANTE P L, et al. Complete genome sequence of Streptococcus thermophilus SMQ-301, a model strain for phage-host interactions[J]. Genome Announcements, 2015,3(3): 1-2. DOI:10.1128/genomeA.00480-15.
[16] BAI Y, SUNA E, SHI Y, et al. Complete genome sequence of Streptococcus thermophilus MN-BM-A01, a strain with high exopolysaccharides production[J]. Journal of Biotechnology, 2016,224: 45-46. DOI:10.1016/j.jbiotec.2016.03.003.
[17] DELORME C, BARTHOLINI C, LURASCHI M, et al. Complete genome sequence of the pigmented Streptococcus thermophilus strain JIM8232[J]. Journal of Bacteriology, 2011, 193(19): 5581.DOI:10.1128/JB.05404-11.
[18] EVIVIE S E, LI B, DING X, et al. Complete genome sequence of Streptococcus thermophilus KLDS 3.1003, a strain with high antimicrobial potential against foodborne and vaginal pathogens[J]. Frontiers in Microbiology, 2017, 8: 1-4. DOI:10.3389/fmicb.2017.01238.
[19] CHIN C, ALEXANDER D H, MARKS P, et al. Nonhybrid, finished microbial genome assemblies from long-read SMRT sequencing data[J]. Nature Methods, 2013, 10(6): 563-569. DOI:10.1038/nmeth.2474.
[20] LI R, LI Y, FANG X, et al. SNP detection for massively parallel whole-genome resequencing[J]. Genome Research, 2009, 19(6): 1124-1132. DOI:10.1101/gr.088013.108.
[21] LI S, LI R, LI H, et al. SOAPindel: efficient identification of indels from short paired reads[J]. Genome Research, 2013, 23(1): 195-200.DOI:10.1101/gr.132480.111.
[22] TATUSOVA T, DICUCCIO M, BADRETDIN A, et al. NCBI prokaryotic genome annotation pipeline[J]. Nucleic Acids Research,2016, 44(14): 6614-6624. DOI:10.1093/nar/gkw569.
[23] AZIZ R K, BARTELS D, BEST A A, et al. The RAST Server: rapid annotations using subsystems technology[J]. BMC Genomics, 2008,9(1): 75. DOI:10.1186/1471-2164-9-75.
[24] WU S, ZHU Z, FU L, et al. WebMGA: a customizable web server for fast metagenomic sequence analysis[J]. BMC Genomics, 2011, 12(1):444. DOI:10.1186/1471-2164-12-444.
[25] MORIYA Y, ITOH M, OKUDA S, et al. KAAS: an automatic genome annotation and pathway reconstruction server[J]. Nucleic Acids Research, 2007, 35(Suppl 2): W182-W185. DOI:10.1093/nar/gkm321.
[26] EL KAFSI H, BINESSE J, LOUX V, et al. Lactobacillus delbrueckii ssp. lactis and ssp. bulgaricus: a chronicle of evolution in action[J].BMC Genomics, 2014, 15(1): 407. DOI:10.1186/1471-2164-15-407.
[27] STOTHARD P, WISHART D S. Circular genome visualization and exploration using CGView[J]. Bioinformatics, 2004, 21(4): 537-539.DOI:10.1093/bioinformatics/bti054.
[28] APWEILER R, BAIROCH A, WU C H, et al. UniProt: the universal protein knowledge base[J]. Nucleic Acids Research, 2004, 32(Suppl 1):D115-D119. DOI:10.1093/nar/gkh131.
[29] 刘文俊. 嗜热链球菌和保加利亚乳杆菌产酸, 风味特性及其功能基因分型和表达研究[D]. 呼和浩特: 内蒙古农业大学, 2014.
[30] 张文羿, 孟和, 张和平. 乳酸菌基因组学研究进展[J]. 微生物学报,2008, 48(9): 1270-1275. DOI:10.3321/j.issn:0001-6209.2008.09.023.
[31] PASTINK M I, TEUSINK B, HOLS P, et al. Genome-scale model of Streptococcus thermophilus LMG18311 for metabolic comparison of lactic acid bacteria[J]. Applied and Environmental Microbiology, 2009,75(11): 3627-3633. DOI:10.1128/AEM.00138-09.
[32] FLAHAUT N A, WIERSMA A, VAN DE BUNT B, et al.Genome-scale metabolic model for Lactococcus lactis MG1363 and its application to the analysis of flavor formation[J]. Applied Microbiology and Biotechnology, 2013, 97(19): 8729-8739.DOI:10.1007/s00253-013-5140-2.
[33] VENDRAMIN V, TREU L, CAMPANARO S, et al. Genome comparison and physiological characterization of eight Streptococcus thermophilus strains isolated from Italian dairy products[J]. Food Microbiology, 2017, 63: 47-57. DOI:10.1016/j.fm.2016.11.002.
[34] GOH Y J, GOIN C, O’FLAHERTY S, et al. Specialized adaptation of a lactic acid bacterium to the milk environment: the comparative genomics of Streptococcus thermophilus LMD-9[J]. Microbial Cell Factories, 2011, 10(1): S22. DOI:10.1186/1475-2859-10-S1-S22.
Genomic Studies of Proteolysis System and Amino Acid Biosynthesis Pathway in Streptococcus thermophilus KLDS SM
LI Bailiang 1 , DING Xiuyun 1,2 , JIN Da 1 , LIU Fei 1 , MENG Yueyue 1 , LI Na 1 , ZHAO Li 1 , HUO Guicheng 1, *
(1. Key Laboratory of Dairy Science, Ministry of Education, Northeast Agricultural University, Harbin 150030, China;2. Guangzhou Genedenovo Biotechnology Co. Ltd., Guangzhou 510000, China)
Abstract: The purpose of this study was to gain genomic insights into the proteolysis system and amino acid biosynthesis in Streptococcus thermophilus KLDS SM. Firstly, whole genome sequencing was performed using combination of Illumina Hiseq 2500 sequencing and Pacific Biosciences RSII sequencing and the circular genomic map was constructed; subsequently,in silico bioinformatics analysis was carried out with respect to extracellular proteinase, peptide transport system, intracellular peptidase and amino acid biosynthesis; finally, comparative genomics of amino acid biosynthesis between strain KLDS SM and 14 other S. thermophilus strains, all of which have had the whole genome sequenced, was performed. The results showed that the genome of S. thermophilus KLDS SM consisted of a circular chromosome (1 856 787 bp) with GC content of 39.08%. A total of 1 732 protein-encoding genes were predicted. S. thermophilus KLDS SM had a complete proteolytic system and could biosynthesize eight amino acids. Furthermore, comparative genomics analysis showed that the amino acid biosynthesis abilities of 15 strains were relatively conservative except for a large difference in histidine biosynthesis among different strains. The results of this study can provide a theoretical basis for better understanding of nitrogen metabolism in S. thermophilus KLDS SM and are of important significance to exploiting it as a starter culture.
Keywords: Streptococcus thermophilus; genome; bioinformatic analysis; proteolysis; amino acid biosynthesis
LI Bailiang, DING Xiuyun, JIN Da, et al. Genomic studies of proteolysis system and amino acid biosynthesis pathway in Streptococcus thermophilus KLDS SM[J]. Food Science, 2018, 39(18): 120-126. (in Chinese with English abstract)DOI:10.7506/spkx1002-6630-201818019. http://www.spkx.net.cn
李柏良, 丁秀云, 靳妲, 等. 基于基因组学分析嗜热链球菌KLDS SM的蛋白质水解系统和氨基酸合成途径[J]. 食品科学,2018, 39(18): 120-126. DOI:10.7506/spkx1002-6630-201818019. http://www.spkx.net.cn
文章编号: 1002-6630(2018)18-0120-07
引文格式:
中图分类号: R151.1
文献标志码: A
*通信作者简介: 霍贵成(1958—),男,教授,博士,研究方向为食品微生物与生物技术。E-mail:guichenghuo@126.com
DOI: 10.7506/spkx1002-6630-201818019
基金项目: “十三五”国家重点研发计划重点专项(2017YFD0400303);国家自然科学基金青年科学基金项目(31401512)
第一作者简介: 李柏良(1989—),男,博士研究生,研究方向为食品科学。E-mail:15846092362@163.com
收稿日期: 2017-08-17