1 株水开菲尔粒中植物乳杆菌的鉴定及产细菌素基因分析
梁新红 1 ,冉军舰 1, *,孙华迪 2 ,赵瑞香 1 ,焦凌霞 1 ,卢艳清 1
(1.河南科技学院食品学院,河南省高校重点实验室培育基地,新乡市农产品加工工程技术研究中心,河南 新乡 453003;2.河南科技学院新科学院,河南 新乡 453003)
摘 要: 目的:对从水开菲尔粒中分离得到1 株能产生抑菌物质的菌株QF01进行菌种鉴定,并对其产细菌素进行实验、鉴定和基因序列分析。方法:通过牛津杯法测定抑菌能力,采用聚合酶链式反应(polymerase chain reaction,PCR)扩增16S rDNA和细菌素相关基因并测序,利用ProParam tool、TMHMM 2.0、InterProScan、SOPM和SWISSMODEL在线软件对基因编码产物进行分析。结果:该菌株产的细菌素对大肠杆菌(Escherichia coli JM109)有明显的抑制作用,通过16S rDNA序列分析初步确定该菌株属于植物乳杆菌(Lactobacillus plantarum)。该菌株含有plnD、plnEF、plnV、plnR四种基因,其中plnV基因编码产物有跨膜螺旋结构,其结构存在信号肽特征。此外,从三级结构的模型预测得到4 种基因的编码产物均存在α-螺旋、β-转角、伸展链和无规则卷曲。结论:从水开菲尔粒分离得到的L. plantarum QF01菌株,含有plnD、plnEF、plnV、plnR细菌素基因,对大肠杆菌有很好的抑制作用。
关键词: 植物乳杆菌;细菌素;抑菌活性;基因克隆;生物信息学
食品安全是影响食品质量的重要方面,其中食源性微生物引发的疾病在发达和发展中国家均广泛存在,严重威胁公共健康。经过多年研究,与危害人类健康相关的食品病原微生物已经得到鉴定,比如肠炎沙门菌(Salmonella enteritidis)和大肠杆菌(Eschericia coli),这两种菌引起75%的食源性疾病 [1] 。通过抗生素可以抑制病原菌的生长,但是抗生素的滥用使病原菌产生耐药性,严重威胁人类的健康,所以天然抑菌物质得到越来越多的重视。细菌素是由乳酸菌产生的具有抑菌活性肽类或蛋白类物质,可用于食品的防腐。产生细菌素的菌种来源于食品、动物、植物和临床样品 [2-8] ,而且不同的菌种产生的细菌素种类不尽相同,如Nisin(Lactococcus sp.)、Pediocin(Pediococcus sp.)、Lactocin(Lactobacillus sp.)、Enterocin(Enterococcus sp.),革兰氏阳性菌比革兰氏阴性菌产生更多种类的细菌素 [9-12] 。细菌素因具有独特的抑菌机制且无毒、无残留、无抗药性的特点受到了越来越多的关注。目前已有多种细菌素已经被分离鉴定,比如Subtilin、Cerein、Thuricin、Plantaricin [13] 。但是到目前为止只有Nisin(Lactococcus lactis)、Pediocin(Pediococcus acidilactici)等少量的细菌素作为商业生产,其他细菌素仍处于实验阶段,所以细菌素作为添加剂或者化学抗菌剂的替代品具有很大潜力,可以广泛的应用在食品行业。水开菲尔是一种自然发酵的饮料,具有改善人体胃肠道蠕动、增强身体免疫力的作用,在全球范围广泛饮用 [14-18] 。将水果、水、糖和开菲尔粒混合无氧发酵2~4 d即可获得水开菲尔 [19-20] ,在发酵后期,水开菲尔粒在水开菲尔中形成,通过过滤将水开菲尔粒分离重复利用。水开菲尔粒易碎,由胞外多糖组成,并含有多种微生物 [21-23] 。近些年来,有研究关注于水开菲尔粒发酵过程的微生物菌落动力学、生物多样性和代谢组学,通过研究证明在水开菲尔发酵过程中起主要作用的是乳酸菌,如副干酪乳杆菌(Lactobacillus paracasei)、希氏乳杆菌(L.hilgardii)和植物乳杆菌(L. plantarum),以及酵母,如酿酒酵母(Saccharomyces cerevisiae)等 [24] 。
本研究从水开菲尔粒中分离出1 株具有抑菌活性的菌株,以该菌为研究对象,对该菌株所属菌种、所产细菌素相关基因进行鉴定,并对其编码基因进行分析。研究结果为该菌株和其细菌素的应用提供理论支持。
1 材料与方法
1.1 材料与试剂
菌株QF01从水开菲尔分离并于实验室保藏;大肠杆菌(Escherichia coli JM109)为实验室保藏菌种。
MRS液体培养基:蛋白胨10 g、葡萄糖20 g、乙酸钠5 g、柠檬酸氢二铵2 g、Mg 2 SO 4 ·7H 2 O 0.58 g、K 2 HPO 4 2 g、硫酸锰0.25 g、牛肉膏10 g、酵母膏5 g、吐温80 1 mL、蒸馏水1 000 mL,微调pH 6.5;MRS固体培养基:固体培养基加琼脂15 g/L,用于QF01菌的培养。
LB液体培养基:蛋白胨10 g/L,酵母浸膏5 g/L,NaCl 10 g/L,pH 7.0;LB固体培养基:固体培养基加琼脂15 g/L,用于E. coli JM109的培养及乳酸菌细菌素活性测定。
1.2 仪器与设备
ZHWY-211C恒温振荡培养箱 上海智城分析仪器制造有限公司;DZKW-4电子恒温水浴锅 北京中兴伟业仪器有限公司;JB-CJ-800超净台 苏州佳宝净化工程设备有限公司;JY600电泳仪 北京君意东方电泳设备有限公司;GelX1650凝胶成像分析系统 上海欧翔科学仪器有限公司;Labcycler Standard Plus聚合酶链式反应(polymerase chain reaction,PCR)仪 美国Bio-Rad公司。
1.3 方法
1.3.1 菌种的活化
在超净工作台中活化低温保藏的菌种QF01,将菌种划线于MRS固体培养基,在37 ℃条件下培养过夜,挑平板上的单菌落置于10 mL MRS液体培养基中,在培养温度37 ℃、摇床转速180 r/min条件下培养12 h。
1.3.2 抑菌活性测定
采用牛津杯法测定:取指示菌液100 μL均匀涂布于LB固体培养基上,放置牛津杯,在杯中注入细菌素样品50 μL,菌株QF01产生的细菌素由硫酸铵沉淀法获得 [25] 。在4 ℃条件下静置过夜,待细菌素充分扩散后放入37 ℃培养箱培养12~16 h,观察抑菌圈大小,以磷酸缓冲液(pH 6.8)为对照每个实验重复3 次。
1.3.3 引物设计
各细菌素基因PCR合成引物参考文献[26],扩增的各基因引物序列如表1所示,设计的引物在上海生工生物工程有限公司合成。
表1 11 对引物序列
Table 1 Sequences of 11 pairs of primers used for PCR

基因 引物名称 引物序列5’-3’ 片段大小/bp plnA plnA-F GTACAGTACTAATGGGAG 450 plnA-R CTTACGCCAATCTATACG plnB alba17-F GCTTCTTATTTAAGTAGAGGATTTCTG 927 alba19-R GCCACGATTACTACCCTTAG plnC plnC-F AGCAGATGAAATTCGGCAG 108 plnC-R ATAATCCAACGGTGCAATCC plnD plnD-F TGAGGACAAACAGACTGGAC 414 plnD-R GCATCGGAAAAATTGCGGATAC plnEF plnEF-F GGCATAGTTAAAATTCCCCCC 428 plnEF-R CAGGTTGCCGCAAAAAAAG plnR plnR-F CCCAGCAGTCCCATCACTAA 236 alba1-R TTACGGAGCGGCATCTATGTC plnK plnK-F CTGTAAGCATTGCTAACCAATC 246 plnK-R ACTGCTGACGCTGAAAAG plnN plnN-F ATTGCCGGGTTAGGTATCG 146 plnN-R CCTAAACCATGCCATGCAC plnV my41-F CAGTTTATTGGCAGCAATCG 724 my41-R ATCCACTCCATCCAAACAATC plnc8 plnc8-F GGTCTGCGTATAAGCATCGC 208 plnc8-R AAATTGAACATATGGGTGCTTTAAATTCC nisin nisin-F GGATAGTATCCATGTCTG 1 000左右nisin-R CAATAGGTTCGTTCGAAG
1.3.4 PCR扩增
用菌株QF01的DNA作为PCR的模板,对细菌素相关基因进行PCR检测。反应体系为25 μL,包括DNA模板1 μL,10×PCR缓冲液2.5 μL,dNTP 2 μL,Mg 2+ 1.5 μL,上游引物1 μL,下游引物1 μL,rTaq 0.125 μL,ddH 2 O 16 μL。细菌素相关基因的PCR扩增条件为:预热温度95 ℃,时间5 min;解链温度95 ℃、时间30 s,退火温度53 ℃、时间30 s,延伸温度72 ℃、时间90 s,循环30 次;温度72 ℃再延伸5 min。扩增结束后取5 μL扩增产物在1%的琼脂糖凝胶、1×TAE缓冲液中进行电泳,在130 V电压下,电泳30 min后,取出胶在EB染色池中染色15 min,然后通过紫外凝胶荧光扫描仪检测。将PCR产物纯化后连接到pMD-19T克隆载体上,转入E. coli DH-5α感受态细胞。在氨苄抗生素的LB平板上涂布培养,挑单菌落PCR鉴定,将获得的阳性克隆送上海生工生物工程有限公司进行测序。
1.3.5 序列分析
利用NCBI网站BLAST程序对产物序列进行同源性搜索,分析其保守序列并利用MEGA 5.1构建系统发育树;使用ProParam tool(http://www.expasy.ch/tools/protparam.html)对其基因编码产物的理化性质进行分析;利用TMHMM 2.0在线软件(http://www.cbs.dtu.dk/services/TMHMM/)对QF01细菌素合成必需基因编码产物的氨基酸序列进行跨膜螺旋信号分析;利用在线软件(http://www.ebi.ac.uk)中的InterProScan工具对QF01细菌素合成必需基因编码产物的氨基酸序列结构域预测;利用在线软件(http://npsa-pbil.ibcp.fr/)中的SOPM工具进行蛋白质二级结构预测;通过ExPASy(http://ca.expasy.org/)提供的SWISS-MODEL在线工具对蛋白质进行同源建模,查看空间模型 [27] 。
2 结果与分析
2.1 抑菌活性
菌株QF01发酵上清液中的细菌素作用于大肠杆菌,如图1所示,通过与对照组相比较,发酵上清液具有抑菌作用。
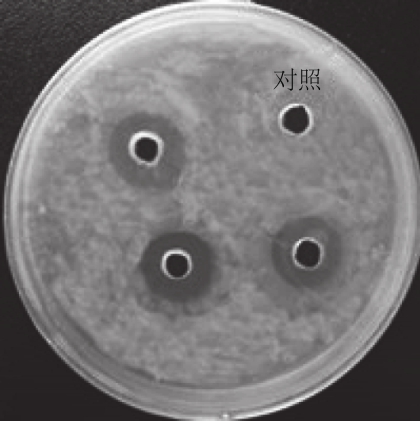
图1 菌株QF01产细菌素抑制大肠杆菌效果
Fig. 1 Inhibitory effect of bacteriocin produced by strain QF01 against E. coli
2.2 菌株QF01基因组DNA提取分析与菌种鉴定
提取菌株QF01总DNA通过琼脂糖凝胶电泳,提取的QF01总DNA条带清晰,没有杂条带,片段大小在20 000 bp左右,以此基因组DNA作为16S rDNA的PCR扩增模板,得到菌株QF01的16S rDNA片段。
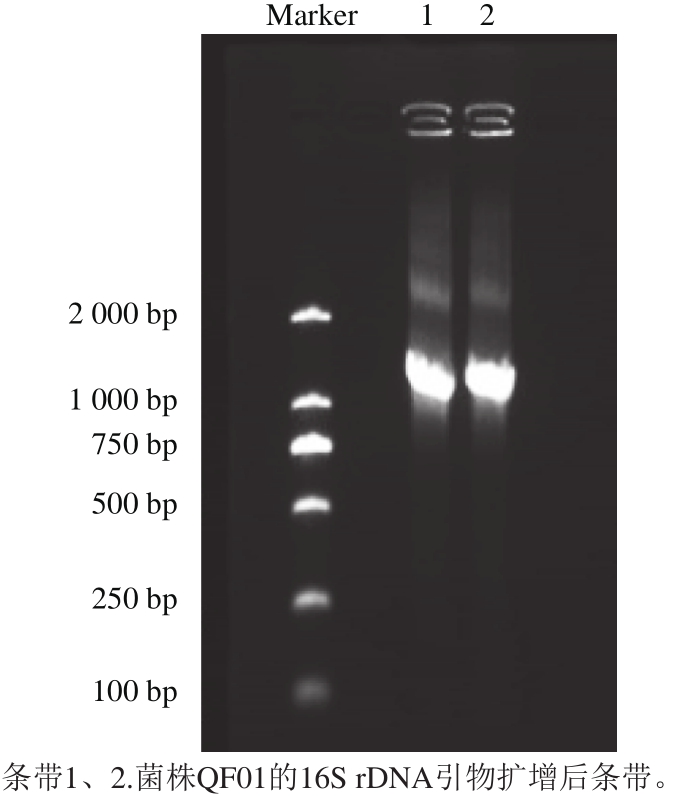
图2 菌株QF01 16S rDNA PCR扩增琼脂糖凝胶电泳分析
Fig. 2 Agarose gel electrophoresis of PCR amplification product of 16S rDNA from strain QF01
如图2所示,16S rDNA引物扩增后的条带在1 400 bp左右,将PCR产物送往测序公司进行测序。测定的菌株QF01的16S rDNA序列结果提交至NCBI中的BLAST进行对比,选取部分乳杆菌属标准菌株的16S rDNA序列共8 个,用软件MEGA 5.1构建菌种系统发育树。从图3可看出,该菌与L. planrarum strain Z55同源性最高,可初步确定该菌株为植物乳杆菌。

图3 菌株QF01 16S rDNA的系统发育树
Fig. 3 Phylogenetic tree of strain QF01 based on 16S rDNA sequence
2.3 细菌素合成基因鉴定
利用设计的11 对引物进行PCR扩增结果见图4。通过观察,发现只有plnD、plnEF、plnR、plnV扩增出现产物条带,将PCR产物回收测序,进行DNA序列的生物信息学分析。其他基因没有扩增出条带,可能是因为该菌不含有这些细菌素的基因。

图4 L. planrarum QF01细菌素相关基因PCR扩增电泳图
Fig. 4 Agarose gel electrophoresis of PCR amplification products of the bacteriocin-related genes of L. planrarum QF01
2.4 氨基酸序列比对分析
利用NCBI网站中的BLAST对植物乳杆菌QF01细菌素合成基因(plnD、plnEF、plnR、plnV)进行比对,发现PlnD、plnEF、plnR、plnV与植物乳杆菌相关基因核苷酸序列一致性达到96%~100%,这表明植物乳杆菌中plnD、plnEF、plnR、plnV基因的核苷酸序列有高度保守性。采用邻位相接法(Neighbor-Joining)构建系统发育树如图5所示,图中数字为Bootstrap 1 000 次的置信度,4 个pln系列基因在各自的发育树中均与植物乳杆菌聚类,表明4 个基因的氨基酸序列在相同物种亲缘关系较近,存在着共同进化模式,可为乳杆菌各物种的分类提供依据。

图5 植物乳杆菌QF01四种细菌素基因氨基酸序列系统发育树
Fig. 5 Phylogenetic analysis of amino acid sequences of four bacteriocin genes of L. plantarum QF01
2.5 相关基因编码产物理化性质分析
在线软件蛋白预测结果显示,plnD基因的编码产物蛋白质的分子质量为23 743.20 ku,氨基酸数为206,理论pI值为5.17,脂肪族氨基酸指数101.80,总平均亲水性-0.270,不稳定系数28.50,根据不稳定系数大于40则该蛋白结构不稳定的标准 [28] ,说明该蛋白质是稳定的。plnEF基因的编码产物蛋白质的分子质量为6 960.90 ku,氨基酸数为63,理论pI值为9.99,脂肪族氨基酸指数85.08,总平均亲水性-0.192,不稳定系数15.82,说明该蛋白质是稳定的。plnR基因的编码产物蛋白质的分子质量为3 288.62 ku,氨基酸数为234,理论pI值为5.75,脂肪族氨基酸指数56.15,总平均亲水性-0.692,不稳定系数49.76,说明该蛋白质不稳定。plnV基因的编码产物蛋白质的分子质量为25 022.97 ku,氨基酸数为226,理论pI值为9.65,脂肪族氨基酸指数132.29,总平均亲水性0.951,不稳定系数20.88,说明该蛋白质是稳定的。上述分析是软件预测结果,需要进一步表达纯化出细菌素进行验证。
2.6 跨膜螺旋信号分析
由表2可知,只有plnV基因编码产物的跨膜螺旋中的氨基酸残基数大于18 [29] ,前60 个氨基酸跨膜螺旋中的残基数44 个(图6),说明plnV基因编码产物可能存在跨膜序列,且蛋白的N端存在信号肽。在PDB(Protein Data Bank)数据库中只有1%左右的跨膜蛋白结构 [30] ,膜蛋白在细胞和外界环境、细胞与细胞之间的信息识别、物质交流及运转中起着不可替代的作用,所以plnV基因在细菌素合成过程中起着非常重要的作用,可深入研究。plnD、plnEF和plnR基因编码产物均无跨膜区域和信号肽,说明这3 种基因编码产物合成后不进行蛋白转运,保留在细胞基质,易于提取分离。
表2 L. planrarum QF01细菌素相关基因编码产物的氨基酸跨膜螺旋数据
Table 2 Analysis of amino acid trans- membrane helix of products encoded by bacteriocin genes in L. planrarum QF01
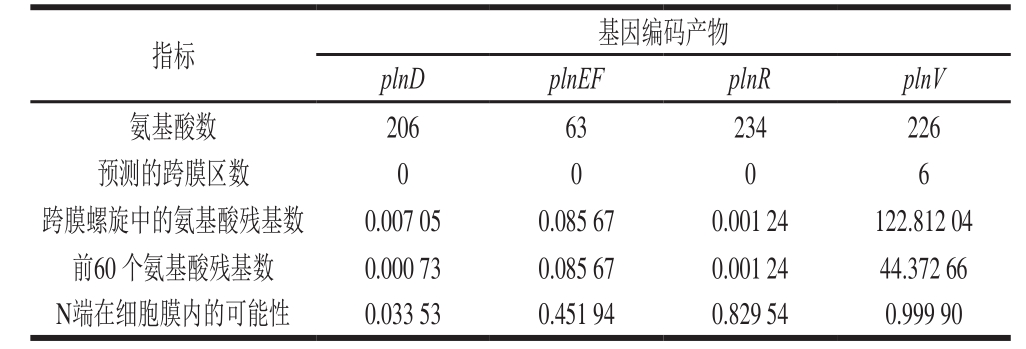
指标 基因编码产物plnD plnEF plnR plnV氨基酸数 206 63 234 226预测的跨膜区数 0 0 0 6跨膜螺旋中的氨基酸残基数 0.007 05 0.085 67 0.001 24 122.812 04前60 个氨基酸残基数 0.000 73 0.085 67 0.001 24 44.372 66 N端在细胞膜内的可能性 0.033 53 0.451 94 0.829 54 0.999 90
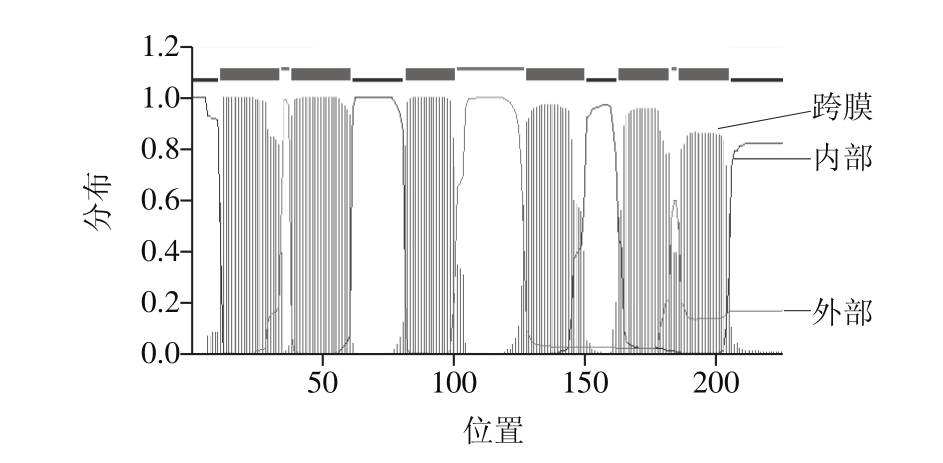
图6 plnV基因编码产物蛋白质序列跨膜螺旋预测分析
Fig. 6 Prediction of the transmembrane helixes of plnV encoding product
2.7 结构域分析
通过在线软件分析可知,在plnD基因编码产物序列中有信号传导响应调节接收器特征结构域,位于第2~129个氨基酸之间(图7),该结构域是群体效应中调控细菌素合成的反应调节器特征结构,对调节基因启动子有重要的作用;在plnEF、plnR、plnV基因编码产物序列中均没有家族结构特征。
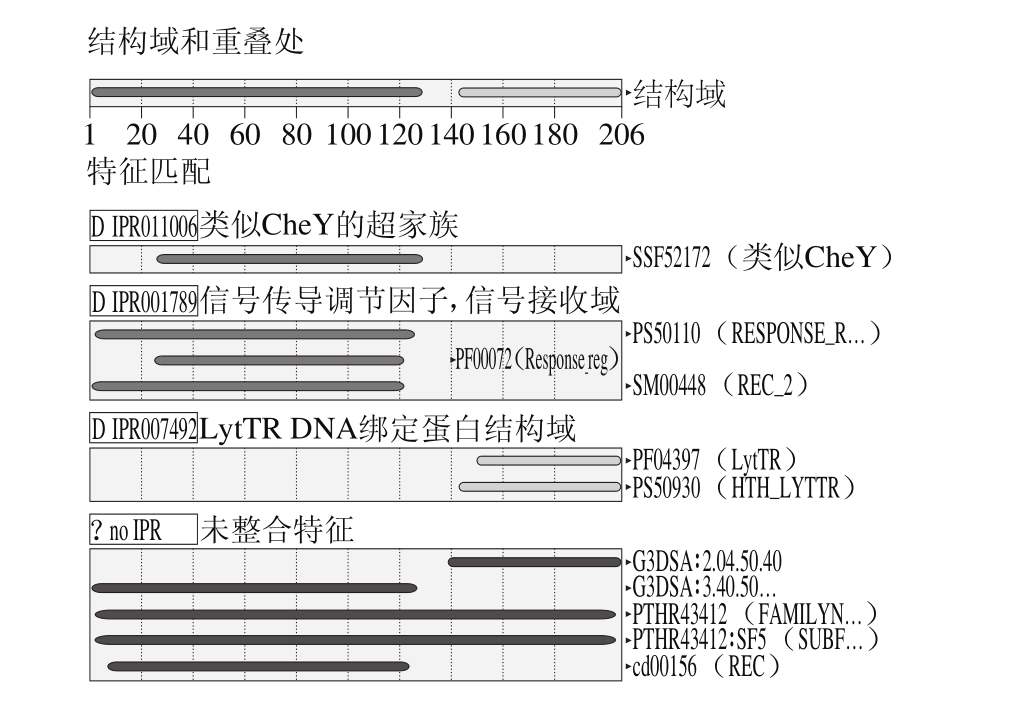
图7 L. plantarum QF01 plnD编码产物预测结构域
Fig. 7 Predicted domain of plnD-encoding product
2.8 二级结构和三级结构分析
表3 L. plantarum QF01细菌素合成相关基因编码产物的二级结构分析
Table 3 Analysis of secondary structures of proteins encoded by the bacteriocin-related genesof L. plantarum QF01
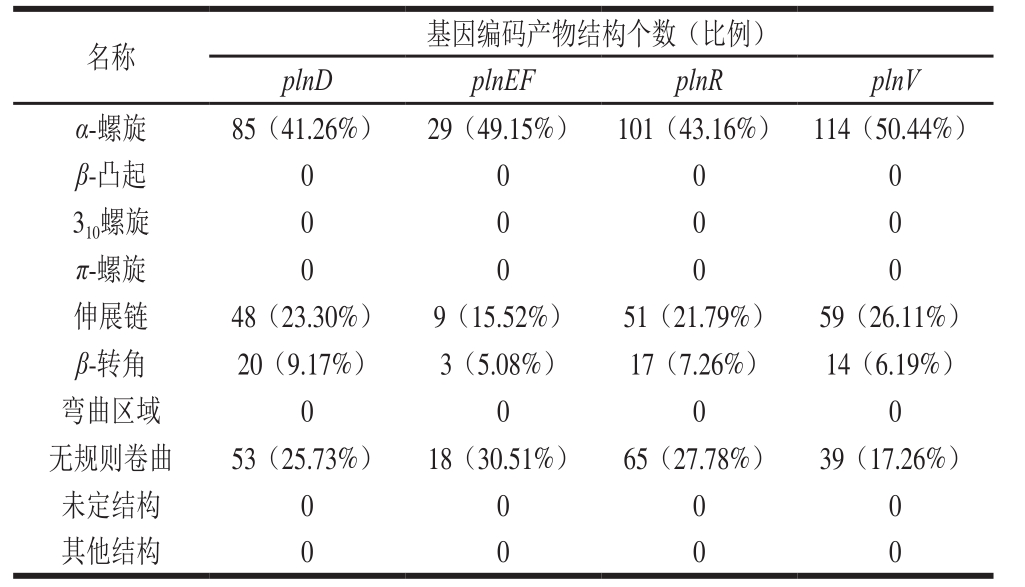
名称 基因编码产物结构个数(比例)plnD plnEF plnR plnV α-螺旋 85(41.26%) 29(49.15%) 101(43.16%) 114(50.44%)β-凸起 0 0 0 0 3 10 螺旋 0 0 0 0 π-螺旋 0 0 0 0伸展链 48(23.30%) 9(15.52%) 51(21.79%) 59(26.11%)β-转角 20(9.17%) 3(5.08%) 17(7.26%) 14(6.19%)弯曲区域 0 0 0 0无规则卷曲 53(25.73%) 18(30.51%) 65(27.78%) 39(17.26%)未定结构 0 0 0 0其他结构 0 0 0 0
如表3所示,plnD、plnEF、plnR和plnV基因的编码产物中均存在α-螺旋、伸展链、β-转角和无规则卷曲,其中α-螺旋在4 种基因编码产物中所占比例最高。plnD基因编码产物中有85 个氨基酸形成α-螺旋,占所有氨基酸的41.26%,plnEF基因编码产物中有29 个氨基酸形成α-螺旋,占所有氨基酸的49.15%,plnR基因编码产物中有101 个氨基酸形成α-螺旋,占所有氨基酸的43.16%,plnV基因编码产物中有114 个氨基酸形成α-螺旋,占所有氨基酸的50.44%。
将4 种氨基酸序列进行同源建模,结果如图8所示。plnD编码产物模型与DNA结合的反应调节器蛋白模型同源性最高(22.5%),该蛋白是细菌素合成的调节因子;plnEF编码产物模型与细菌素PlnF蛋白模型同源性为100%,plnE起到调控乳酸菌产生细菌素的作用;plnR编码产物模型与甲酸通道蛋白模型同源性为10.89%;plnV编码产物模型与Ras转化酶1蛋白模型同源性为22.73%,该蛋白是一种膜内蛋白酶,能阻断Ras通路传导信号。其中plnEF编码产物同源性最高,另外3 种模型同源性较低。在图8中能明显看出蛋白二级结构α-螺旋所占比例较大,对比空间结构的元件及分布,证明二级结构和三级结构结果一致,表明模型的可信度较高。
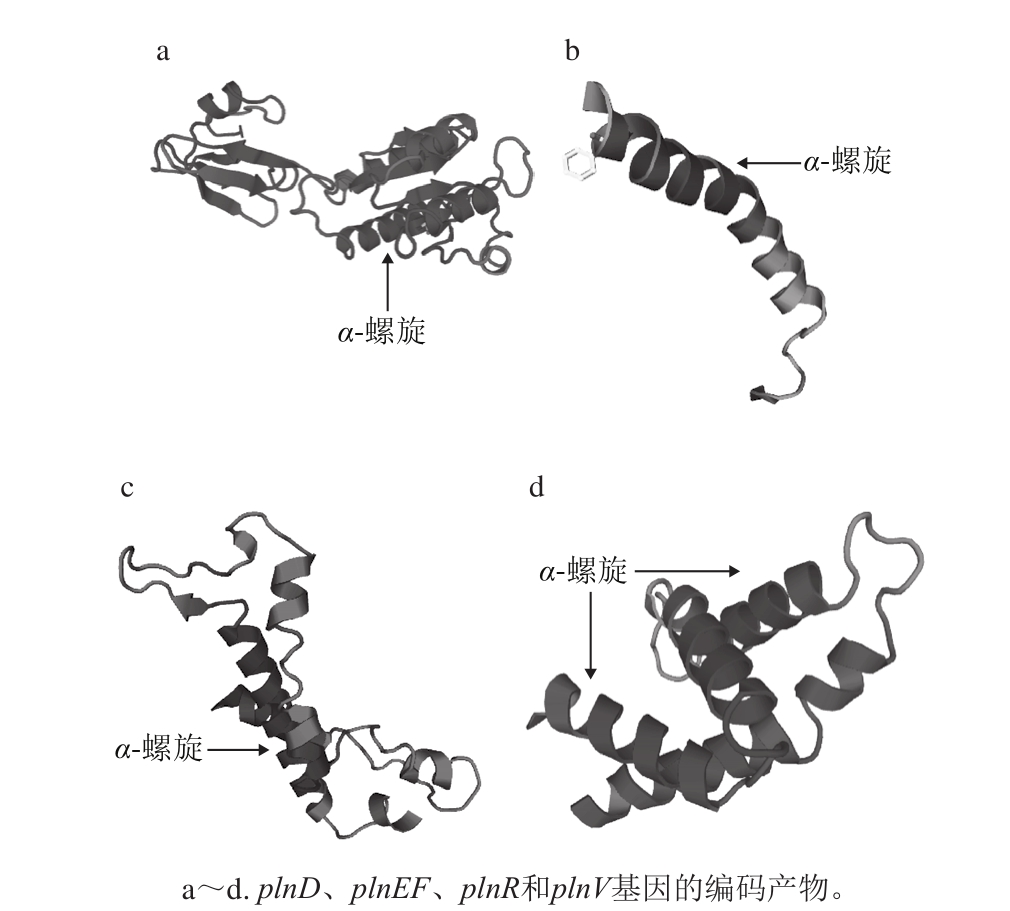
图8 L. plantarum QF01相关基因编码产物的三级结构
Fig. 8 Tertiary structures of proteins encoded by the bacteriocinrelated genes of L. plantarum QF01
3 结 论
本研究从水开菲尔粒中分离鉴定出1 株具有抑菌效果的植物乳杆菌QF01,利用已报道细菌素相关基因的11 对特异性引物对植物乳杆菌QF01全基因组DNA进行PCR扩增、测序,发现该菌基因组中存在plnD、plnEF、plnR和plnV基因,并获得了4 种细菌素基因序列,通过生物信息学分析得知4 种细菌素氨基酸序列,确定plnR基因编码产物结构不稳定,plnD、plnEF和plnV基因编码产物结构稳定。跨膜螺旋信号分析得知plnV基因编码产物可能存在跨膜序列,且蛋白的N端可能存在信号肽,由于跨膜蛋白在生命体内有着重要的作用,所以plnV基因编码产物可进行深入研究。因此,本研究利用分子技术鉴定细菌素的方法可以为食品安全提供材料,而且为细菌素外源表达提供理论支持。
参考文献:
[1] BALI V, PANESAR P S, BERA M B, et al. Bacteriocins: recent trends and potential applications[J]. Critical Reviews in Food Science &Nutrition, 2016, 56(5): 817-834. DOI:10.1080/10408398.2012.729231.
[2] BELGACEM Z B, ABRIOUEL H, OMAR N B, et al. Antimicrobial activity, safety aspects, and some technological properties of bacteriocinogenic Enterococcus faecium, from artisanal Tunisian fermented meat[J]. Food Control, 2010, 21(4): 462-470. DOI:10.1016/j.foodcont.2009.07.007.
[3] KUMAR M, TIWARI S K, SRIVASTAVA S. Purification and characterization of Enterocin LR/6, a bacteriocin from Enterococcus faecium, LR/6[J]. Applied Biochemistry & Biotechnology, 2010,160(1): 40-49. DOI:10.1007/s12010-009-8586-z.
[4] RASHID H U, TOGO K, UEDA M, et al. Characterization of bacteriocin produced by Streptococcus bovis, J2 40-2 isolated from traditional fermented milk ‘Dahi’[J]. Animal Science Journal, 2009,80(1): 70-78. DOI:10.1111/j.1740-0929.2008.00596.x.
[5] SHARMA N, KAPOOR G, NEOPANEY B. Characterization of a new bacteriocin produced from a novel isolated strain of Bacillus lentus, NG121[J]. Antonie Van Leeuwenhoek, 2006, 89(3/4): 337-343.DOI:10.1007/s10482-005-9036-8.
[6] SILKIN L, HAMZA S, KAUFMAN S, et al. Spermicidal bacteriocins:lacticin 3147 and subtilosin A[J]. Bioorganic & Medicinal Chemistry Letters, 2008, 18(10): 3103-3106. DOI:10.1016/j.bmcl.2007.11.024.
[7] SHIN M S, HAN S K, RYU J S, et al. Isolation and partial characterization of a bacteriocin produced by Pediococcus pentosaceus K23-2 isolated from Kimchi[J]. Journal of Applied Microbiology,2008, 105(2): 331-339. DOI:10.1111/j.1365-2672.2008.03770.x.
[8] ZHENG B, TOMITA H, INOUE T, et al. Isolation of VanB-type Enterococcus faecalis strains from nosocomial infections: first report of the isolation and identification of the pheromone-responsive plasmids pMG2200, encoding VanB-type vancomycin resistance and a Bac41-type bacteriocin, and pMG22 encoding erythromycin resistance and cytolysin (Hly/Bac)[J]. Antimicrobial Agents & Chemotherapy,2009, 53(2): 735-747. DOI:10.1128/AAC.00754-08.
[9] JACK R, BIERBAUM G, HEIDRICH C, et al. The genetics of lantibiotic biosynthesis[J]. Bioessays, 1995, 17(9): 793-802.DOI:10.1002/bies.950170909.
[10] SCHNÜRER J, MAGNUSSON J. Antifungal lactic acid bacteria as biopreservatives[J]. Trends in Food Science & Technology, 2005,16(1): 70-78. DOI:10.1016/j.tifs.2004.02.014.
[11] STOIANOVA L G, EGOROV N S, FEDOROVA G B, et al. A comparison of the properties of bacteriocins formed by Lactococcus lactis subsp. lactis strains of diverse origin[J]. Prikladnaâ Biohimiâ I Mikrobiologiâ, 2007, 43(6): 604-610.
[12] AHMED Z, WANG Y P, CHENG Q L, et al. Lactobacillus acidophilus bacteriocin, from production to their application: an overview[J]. African Journal of Biotechnology, 2010, 9(20): 2843-2850.
[13] AUNPAD R, NA-BANGCHANG K. Pumilicin 4, a novel bacteriocin with anti-MRSA and anti-VRE activity produced by newly isolated bacteria Bacillus pumilus strain WAPB4[J]. Current Microbiology,2007, 55(4): 308-313. DOI:10.1007/s00284-006-0632-2.
[14] MARSH A J, O’SULLIVAN O, HILL C, et al. Sequence-based analysis of the microbial composition of water kefir from multiple sources[J].FEMS Microbiology Letters, 2013, 348(1): 79-85. DOI:10.1111/1574-6968.12248.
[15] DAVIDOVIC S, MILJKOVIC M, ANTONOVIC D, et al. Water kefir grain as a source of potent dextran producing lactic acid bacteria[J]. Hemijska Industrija, 2015, 69: 83-83. DOI:10.2298/HEMIND140925083D.
[16] LAUREYS D, JEAN A V, DUMONT J, et al. Investigation of the instability and low water kefir grain growth during an industrial water kefir fermentation process[J]. Applied Microbiology & Biotechnology,2017, 101(7): 1-9. DOI:10.1007/s00253-016-8084-5.
[17] RANDAZZO W, CORONA O, GUARCELLO R, et al. Development of new non-dairy beverages from mediterranean fruit juices fermented with water kefir microorganisms[J]. Food Microbiology, 2016, 54:40-51. DOI:10.1016/j.fm.2015.10.018.
[18] POTHAKOS V, ILLEGHEMS K, LAUREYS D, et al. Acetic acid bacteria in fermented food and beverage ecosystems[M]//Acetic Acid Bacteria. Japan: Springer, 2016: 73-100.
[19] GULITZ A, STADIE J, WENNING M, et al. The microbial diversity of water kefir[J]. International Journal of Food Microbiology, 2011,151(3): 284-288. DOI:10.1016/j.ijfoodmicro.2011.09.016.
[20] LAUREYS D, DE V L. Microbial species diversity, community dynamics, and metabolite kinetics of water kefir fermentation[J].Applied & Environmental Microbiology, 2014, 80(8): 2564-2572.DOI:10.1128/AEM.03978-13.
[21] MARSH A J, HILL C, ROSS R P, et al. Fermented beverages with health-promoting potential: past and future perspectives[J]. Trends in Food Science & Technology, 2014, 38(2): 113-124. DOI:10.1016/j.tifs.2014.05.002.
[22] HERMANN M, KRONSEDER K, SORGEND J, et al. Functional properties of water kefir and its use as a hydrocolloid in baking[J].European Food Research & Technology, 2016, 242(3): 337-344.DOI:10.1007/s00217-015-2543-6.
[23] TANG H, WANG H, YU H, et al. Galactomyces geotrichum isolated from water kefir: interaction with Lactobacillus kefir[J]. Italian Journal of Food Science, 2016, 28(2): 287-297.DOI:10.14674/1120-1770/ijfs.v390.
[24] JANSSENS M, MYTER N, VUYST L D, et al. Species diversity and metabolic impact of the microbiota are low in spontaneously acidified Belgian sausages with an added starter culture of Staphylococcus carnosus[J]. Food Microbiology, 2012, 29(2): 167-177. DOI:10.1016/j.fm.2011.07.005.
[25] 冉军舰, 焦凌霞, 梁新红, 等. 嗜酸乳杆菌产细菌素的提取及其生物特性研究[J]. 食品工业科技, 2017, 38(13): 112-118. DOI:10.13386/j.issn1002-0306.2017.13.021.
[26] EVANGELIN Y, VENKATESWARULU T C, BABU D J, et al.Bacteriocins from lactic acid bacteria and its potential applications[J].International Journal of Pharmaceutical Sciences Review & Research,2015, 32(1): 306-309.
[27] 冉军舰, 徐剑宏, 赫丹, 等. 小麦赤霉病原菌拮抗菌Bacillus amyloliquefaciens 7M1产抗菌素的研究[J]. 微生物学通报, 2016,43(11): 2437-2447. DOI:10.13344/j.microbiol.china.151013.
[28] 张雨良, 张智俊, 杨峰山, 等. 新疆盐生植物车前PmNHX1基因的克隆及生物信息学分析[J]. 中国生物工程, 2009, 29(1): 27-33.DOI:10.13523/j.cb.20090106.
[29] GE J, SUN Y, XIN X, et al. Purification and partial characterization of a novel bacteriocin synthesized by Lactobacillus paracasei HD1-7 isolated from Chinese sauerkraut juice[J]. Scientific Reports, 2016, 6:19366. DOI:10.1038/srep19366.
[30] 杨洪乾, 郭城, 方慧生. α-螺旋跨膜蛋白结构预测的研究进展[J]. 药物生物技术, 2010(5): 466-470.
Identification of a Lactobacillus plantarum Strain Isolated from Water Kefir Grains and Sequence Analysis of Its Bacteriocin Genes
LIANG Xinhong 1 , RAN Junjian 1, *, SUN Huadi 2 , ZHAO Ruixiang 1 , JIAO Lingxia 1 , LU Yanqing 1
(1. Xinxiang Engineering Technology Research Center for Agricultural Products Processing, Key Lab of Breeding Base of College of Henan Province, School of Food Science, Henan Institute of Science and Technology, Xinxiang 453003, China;2. Xinke College, Henan Institute of Science and Technology, Xinxiang 453003, China)
Abstract: Objective: The bacteriocin-producing strain QF01, isolated from water kefir grains, was identified and the antibacterial activity of bacteriocin from the isolate was evaluated. The gene sequences encoding bacteriocin were analyzed as well. Methods: The antibacterial activity of bacteriocin was determined by the Oxford cup method. The bacteriocinrelated genes were amplified by PCR, 16S rDNA analysis was used for bacterial identification and bacteriocin gene analysis was carried out by using online software (ProParam tool, TMHMM 2.0, InterProScan, SOPM and SWISS-MODEL).Results: Bacteriocin produced by strain QF01 demonstrated a strong inhibitory activity against Escherichia coli JM109.This strain was identified as Lactobacillus plantarum based on its 16S rDNA sequence. The four bacteriocin-encoding genes plnD, plnEF, plnV and plnR were found in Lactobacillus plantarum QF01. The plnV gene encoding product had a helical transmembrane structure with signal peptide characteristics at its N-terminal. In addition, each of the four gene encoding products contained α-helix, β-turn, extended chain and random coil structures, as predicted from the tertiary structure model.Conclusion: L. plantarum QF01 contains the bacteriocin-encoding genes plnD, plnEF, plnV and plnR, and has a strong inhibitory activity against E. coli.
Keywords: Lactobacillus plantarum; bacteriocin; antibacterial activity; gene cloning; bioinformatics
LIANG Xinhong, RAN Junjian, SUN Huadi, et al. Identification of a Lactobacillus plantarum strain isolated from water kefir grains and sequence analysis of its bacteriocin genes[J]. Food Science, 2018, 39(18): 145-151. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201818023. http://www.spkx.net.cn
梁新红, 冉军舰, 孙华迪, 等. 1 株水开菲尔粒中植物乳杆菌的鉴定及产细菌素基因分析[J]. 食品科学, 2018, 39(18):145-151. DOI:10.7506/spkx1002-6630-201818023. http://www.spkx.net.cn
文章编号: 1002-6630(2018)18-0145-07
引文格式:
中图分类号: TS201.3
文献标志码: A
*通信作者简介: 冉军舰(1981—),男,讲师,博士,研究方向为食品生物技术。E-mail:ranjunjian@126.com
DOI: 10.7506/spkx1002-6630-201818023
基金项目: 河南省科技计划项目(182102110109);河南省科技厅科技攻关项目(182102410098);河南省高校重点科研项目(18A550006 );河南省高校科技创新人才支持计划项目(17HASTIT037)
第一作者简介: 梁新红(1971—),女,副教授,博士,研究方向为发酵食品。E-mail:liangxinhong2005@163.com
收稿日期: 2017-07-20










