表1 克隆目的基因gad和荧光定量PCR引物
Table 1 Primers used for cloning of target genes and real-time PCR
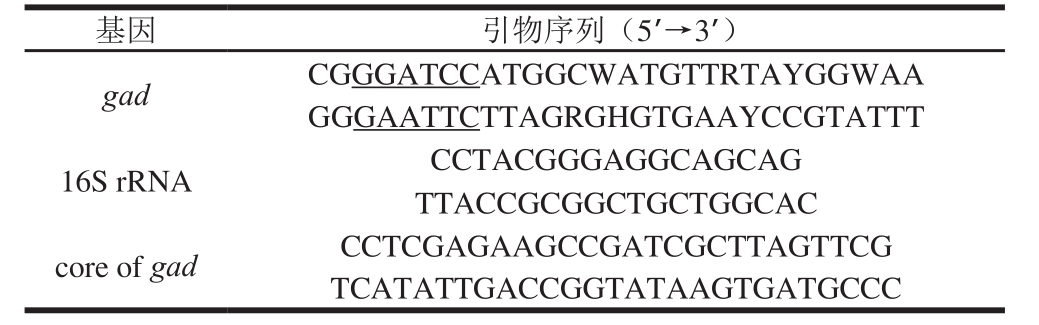
注:下划线为酶切位点。
谭 霄 1,2 ,孙 擎 1,2 ,曾 林 1,2 ,赵婷婷 1,2 ,谭金龙 1 ,张 庆 1,2, *,向文良 1
(1.西华大学食品与生物工程学院,四川省食品生物技术重点实验室,四川 成都 610039;2.西华大学古法发酵(酿造)生物技术研究所,四川 成都 610039)
摘 要: 以1 株产γ-氨基丁酸(γ-aminobutyric acid,GABA)植物乳杆菌(Lactobacillus plantarum)BC114谷氨酸脱羧酶为研究对象,通过聚合酶链式反应(polymerase chain reaction,PCR)技术获得该酶基因,对其进行生物信息学分析,并转入大肠杆菌BL21(DE3)中实现异源表达。采用实时荧光定量PCR、聚丙烯酰胺凝胶电泳和高效液相色谱分别测定重组菌不同发酵时间点谷氨酸脱羧酶基因的表达量、蛋白表达量以及产GABA能力。结果表明:植物乳杆菌中谷氨酸脱羧酶基因大小为1 410 bp,编码469 个氨基酸,蛋白二级结构由α-螺旋(32.2%)、β-折叠(11.5%)和无规则卷曲(56.3%)构成,完成了该酶的三维结构同源建模;成功构建了重组谷氨酸脱羧酶大肠杆菌,谷氨酸脱羧酶基因在诱导6 h相对表达量最大,而蛋白表达量较基因在转录水平表达量有一定滞后,在8 h达最大值,此时GABA产量也达最高,为2 387 mg/L。
关键词: 植物乳杆菌BC114;γ-氨基丁酸;谷氨酸脱羧酶;克隆;表达;生物信息学
γ-氨基丁酸(γ-aminobutyric acid,GABA)是广泛存在于植物、动物和微生物中的一种非蛋白质氨基酸 [1] 。在哺乳动物脑组织中,GABA是一种重要的抑制性递质,具有降血压、镇静神经、利尿、改善脑机能、增进脑活力等功能 [2-3] 。此外,GABA还具有抗热应激和提高饲料的利用率等作用 [4] 。因此GABA被广泛应用到食品、医药、保健品及农业等行业 [5] 。目前,合成GABA主要有化学合成法、植物富集法和微生物合成法,相比而言,微生物合成法具有条件温和、安全性好、耗能低等优点 [6-10] ,因此,近年来微生物合成法制备GABA受到国内外广泛关注,其中对红曲霉、酿酒酵母、乳酸菌等 [11] 研究较为深入,特别是具有保健作用的乳酸菌用于合成GABA受到人们的青睐。
乳酸菌合成GABA是通过谷氨酸脱羧酶(glutamate decarboxylase,GAD)专一性、不可逆地催化L-谷氨酸或钠盐α-脱羧而成 [12] 。GAD在野生乳酸菌中受到菌体自身代谢水平的控制,导致GAD的表达量不高 [13] 。同时,乳酸菌是兼性厌氧微生物,发酵周期较长,培养条件较难控制,而大肠杆菌(Escherichia coli)生长迅速、易于高密度发酵,易借助基因工程手段实现GABA的高效率生产 [14-16] 。目前,Park [17] 、李杰 [18] 及Somasundaram [19] 等分别将短杆菌、酿酒酵母、嗜热古菌的GAD转入到E. coli BL21(DE3)实现了异源表达;Mazzoli等 [20] 研究了短杆菌发酵产GABA转录组和蛋白质组的相互关系;李理等 [21] 研究植物乳杆菌(Lactobacillus plantarum)发酵不同时间点GABA产量和GAD相对表达量之间的关系;齐育平等 [22] 对短乳杆菌GAD的基因进行了生物信息学分析。而对L. plantarum GAD进行原核表达,并对基因工程菌的GAD相对表达量和GABA产量相互关系的探讨及生物信息学的综合分析鲜有报道。
本实验以1 株从四川泡菜中筛选的产GABA的L. plantarum BC114 [3] 为出发菌株,通过克隆其GAD基因gad,生物信息学分析,并将gad转入E. coli BL21(DE3)进行诱导表达,同时对gad表达量、蛋白表达量和GABA产量进行评估,探究gad在转录、翻译和表达上的相互关联,为gad改造和发酵提高GABA产量提供一定的理论依据。
E. coli BL21(DE3)、E. coli JM109、pGEM-T Simple Vector 普洛麦格(北京)生物技术有限公司;L. plantarum BC114、pET-28a(+)由本实验室保存。
限制性内切酶EcoRI和BamHI 美国Thermo公司;质粒小量抽提试剂盒、胶回收试剂盒、细菌总RNA提取试剂盒 天根生化科技有限公司;T4 DNA连接酶、Pfu DNA聚合酶 Promega(北京)生物技术有限公司;RT Easy TM I(For first-strand cDNA synthesis)试剂盒 福际生物技术(成都)有限公司。
Biometra聚合酶链式反应(polymerase chain reaction,PCR)仪、qTOWER2 2.2荧光定量PCR德国耶拿分析仪器有限公司;2695高效液相色谱(high performance liquid chromatography,HPLC)仪(配有Waters2998紫外检测器) 美国Waters公司;凝胶成像系统、DYCP-31琼脂糖水平电泳系统、垂直电泳系统美国Bio-Rad公司;BHC-1300A2冷冻离心机 美国贝克曼库尔特有限公司。
1.3.1 gad的克隆及测序
将L. plantarum BC114在MRS液体培养基过夜培养,利用细菌DNA提取试剂盒提取其基因组DNA,以此为模板进行PCR扩增gad基因,引物 [23] 见表1,下划线为EcoRI和BamHI限制性内切酶位点。PCR体系(50 μL):40.5 μL ddH 2 O,5 μL 10×Buffer with MgSO 4 ,1 μL dNTP(10 mmol/L),1 μL upstream primer(20 μmol/L),1 μL downstream primer(20 μmol/L),0.5 μL Pfu DNA Polymerase,1 μL DNA template(100 ng/μL);反应条件如下:95 ℃预变性5 min,94 ℃变性1 min,58 ℃退火1 min,72 ℃延伸2 min,循环30 次,最后72 ℃ 10 min。
表1 克隆目的基因gad和荧光定量PCR引物
Table 1 Primers used for cloning of target genes and real-time PCR

注:下划线为酶切位点。
以1%琼脂糖凝胶电泳检测PCR产物是否正确,并进行胶回收。目的片段与pGEM-T载体通过T4 DNA连接酶,25 ℃过夜连接,热激转入E. coli JM109感受态细胞中。在含100 μg/mL氨苄青霉素(ampicillin,Amp)平板上培养过夜,挑取单克隆在含100 μg/mL Amp液体LB培养基中培养8~10 h,抽提质粒得到pGEM-T-gad,并采用酶切电泳和测序鉴定,序列测定由成都擎科梓熙生物技术有限公司完成。
1.3.2 GAD的生物信息学分析
使用ExPASY的Protparam工具对gad编码的蛋白质的理化性质进行预测和分析;应用TMHMM Server v. 2.0在线工具预测基因编码蛋白的跨膜结构域,同时,利用PSIPED V3.3在线分析工具分析蛋白质的二级结构;利用MEGA 5.0软件构建蛋白的系统进化树;应用Swiss-Model在线工具进行同源建模,并用PROCHECK软件进行模型评估。
1.3.3 重组菌的构建
将克隆载体pGEM-T-gad经EcoRI和BamHI双酶切,产物进行琼脂糖核酸电泳,胶回收获得目的片段,并连接至同样经过EcoRI和BamHI双酶切的表达载体pET-28a(+),热激转入到感受态细胞E. coli BL21(DE3)中,经菌落和酶切鉴定,获得E. coli BL21(DE3)/pET-28a-gad。
1.3.4 重组蛋白在E. coli BL21(DE3)中的诱导表达
将E. coli BL21(DE3)/pET-28a-gad转入到卡那平板上,通过菌落PCR筛选出阳性克隆子,并将其接种至含50 μg/mL卡那霉素的10 mL LB培养基中37 ℃、150 r/min活化8~10 h,以2%接种量转到含50 mg/L卡那霉素的100 mL LB培养基,37 ℃、200 r/min培养到OD 600 nm 为0.6后加入1 mmol/L IPTG和0.05 mmol/L磷酸吡哆醛(pyridoxal phosphate,PLP),降温至28 ℃,180 r/min进行蛋白诱导表达,在诱导期间取2、4、6、8、10 h的发酵液于后续分析。
1.3.5 基因表达水平的测定
1.3.5.1 重组菌发酵过程中gad相对表达量的测定
取不同时间菌液,并按RNA提取试剂盒(天根)操作说明书步骤进行,并加入DNaseI酶(37 ℃,15 min;65 ℃,10 min)以去除RNA样品中残留的基因组DNA。按照试剂盒说明书(福际)进行反转录成cDNA第1链。以反转录成的cDNA为模板进行实时荧光定量PCR。利用软件Primer 5.0对目的基因core of gad和内参基因16S rRNA设计特异性引物(表1)。
PCR体系(20 μL):荧光混合染料10 μL、正反向引物各0.4 μL、cDNA模板0.5 μL,加无菌双蒸水至20 μL。反应条件:95 ℃ 2 min;95 ℃ 15 s,60 ℃ 34 s,循环40 次;溶解曲线分析:95 ℃ 15 s,60 ℃ 34 s,95 ℃ 15 s。
基因表达量的定量以16S rRNA为内参基因,发酵时间10 h的gad表达量为参比样本,以无菌双蒸水代替cDNA模板作为阳性对照,分析各基因Ct值(每个样品设置3次平行实验)。利用2 -ΔΔCt 法对GAD表达量进行相对定量(其中,Ct是扩增基因达到阈值时的循环数,ΔCt是目的基因与内参基因Ct值的差值,ΔΔCt是实验各时间点与实验结束时间点ΔCt值的差值) [24] 。
1.3.5.2 重组菌发酵过程中十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDS-PAGE)分析
将菌液离心收集菌体,并用生理盐水进行洗涤,加入2×样品缓冲液煮沸10 min,然后13 000 r/min离心10 min,取上清液进行10%的SDS-PAGE分析。
1.3.5.3 重组菌产GABA表达能力评估
根据陈琳等 [11] 报道,以5 mL质量浓度为5 g/L的L-谷氨酸钠为底物,以不同发酵时间的菌体4 mg,乙酸-乙酸钠缓冲液(含2.5 mmol/L Ca 2+ 、3.5 mmol/L Mg 2+ )调pH值为4.5,45 ℃条件下反应4 h。参照并结合Diana等 [25] 利用DNS-Cl衍生化的方法测定重组菌不同时间发酵液的GABA产量,HPLC分析条件:色谱柱为岛津-GL INERTSIL ODS-3(4.6 mm×150 mm,5 µm);紫外检测波长254 nm;柱温30 ℃;进样量20 μL;流动相A为甲醇,流动相B为醋酸钠(pH 6.2)-甲醇-四氢呋喃(84∶25∶1,V/V);流速1 mL/min;梯度洗脱时,流动相B比例:0~6 min,维持80%;6~20 min,80%~20%;20~27 min,20%~0%;27~30 min,返回80%。

图1 重组质粒pGEM-T-gad的酶切结果
Fig. 1 Identification of recombinant vector pGEM-T-gad by restriction enzyme digestion
以L. plantarum BC114基因组DNA为模板,经PCR扩增得到目的片段,胶回收该片段与pGEM-T Simple Vector连接,并转入E.coli JM109,筛选阳性克隆子,重组质粒酶切结果见图1。由EcoRI酶切结果可知,酶切后得到2 条明显分离的条带,其中1 条在大约3 000 bp处,另1 条在大约1 400 bp处,片段大小与李杰 [18] 报道的一致,表明该菌株可能有gad的存在,随后测序并对序列进行分析。
通过ExPASY的protparam在线工具结果表明:L. plantarum GAD蛋白由469 个氨基酸编码而成(GenBank登录号MF418590),其中带正电荷氨基酸残基(Arg+Lys)共40 个,带负电荷的氨基酸残基(Asp+Glu)总数为60 个,蛋白质理论分子等电点为5.58,分子式为C 2412 H 3652 N 648 O 693 S 2 ,脂溶性指数为84.65。GAD蛋白在溶液中不稳定指数为30.53(<40),说明该蛋白在溶液中性质稳定。克隆的GAD所编码的蛋白的二级结构由α-螺旋、β-折叠、无规则卷曲构成,分别占32.2%、11.5%、56.3%。同时,采用TMHMM Server v. 2.0预测显示,GAD蛋白中无跨膜结构域,为非跨膜蛋白。

图2 GAD氨基酸序列系统发育树
Fig. 2 Phylogenetic analysis of amino acid sequence of GAD
Grone等 [26] 分析了脊椎动物中3 种GAD的差异,并用氨基酸序列构建系统发育树分析亲缘关系,表明GAD3与GAD1和GAD2具有同源性,但是对于L. plantarum的GAD的亲缘关系鲜见报道。本研究基于GAD氨基酸序列,采用邻位相接法(Neighbor-Joining)对其进行系统发育树的构建,结果见图2。L. plantarum BC114的GAD聚类在同一类群,同时,短乳杆菌的GAD也聚为一类,而其他Enterococcus gallinarum和Lactococcus lactis不同属的聚为一类,Lactobacillus spicheri和Lactobacillus senmaizukei不同种也聚为一类。因此,GAD在细菌中既有物种之内的保守性,又有物种之间相对变异性,较真实地反映了不同物种间的自然演化过程 [19] 。

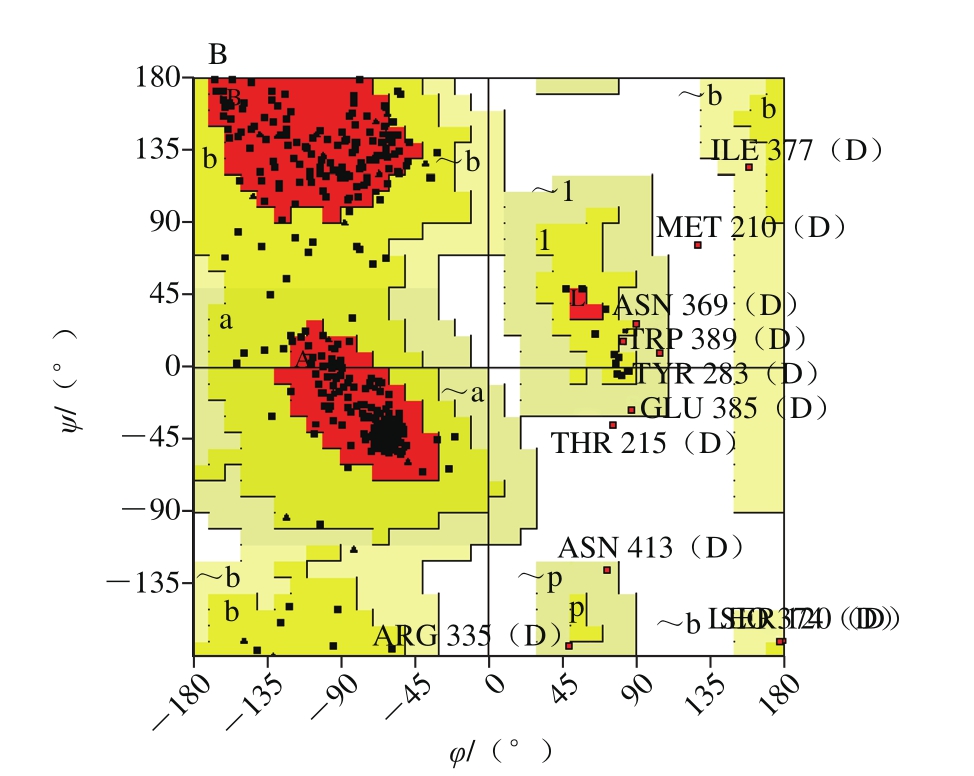
图3 GAD的三级结构(A)和拉氏构象图(B)
Fig. 3 Predicted tertiary structure (A) and Ramachandran plot (B) of GAD
采用Swiss-Model在线预测工具进行同源建模构建未知蛋白质三维结构,对于序列相似性大于30%的蛋白序列能够建立准确性较高的模型。对L. plantarum BC114的GAD进行同源建模,选择与PDB数据库中GAD蛋白同源最高的蛋白E. coli gad(3fz6.1),相似度45.50%,模拟GAD蛋白的三级结构,结果如图3A所示,GAD的折叠方式符合二级结构的分析结果。
采用PROCHECK工具对预测结果进行评估,结果见图3B。拉氏图中的每个区域的颜色由深到浅排列的次序依次为最佳合理区、额外合理区、一般合理区以及不合理区。结果显示,有85.9%处于最佳合理区,说明这些氨基酸残基之间不存在重叠和不合理接触,而额外合理区、一般不合理和不合理区分别占11.4%、2.2%、0.5%,此评估结果与郁凯 [27] 报道的大体一致。
随机挑取平板上菌落进行PCR,扩增产物经琼脂糖凝胶电泳分析在约1 400 bp出现条带,与预期的条带大小一致,结果见图4A,挑取该菌落培养并抽提质粒,进行EcoRI和BamHI双酶切,酶切结果见图4B。在5 000 bp处为pET-28a(+)质粒(5 369 bp),1 400 bp左右为gad片段。表明目的基因已成功连接到载体上,随后的测序结果准确无误,可用于后续的异源表达。
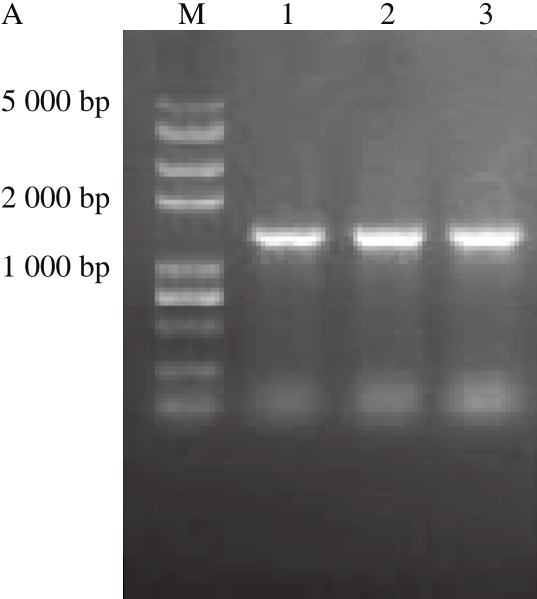

图4 菌落PCR(A)和重组质粒pET-28a-gad的双酶切验证(B)
Fig. 4 PCR analysis of positive monoclonal colonies (A) identification of recombinant vector pET-28a-gad by double restriction enzyme digestion (B)
2.4.1 重组菌gad相对表达量的测定结果
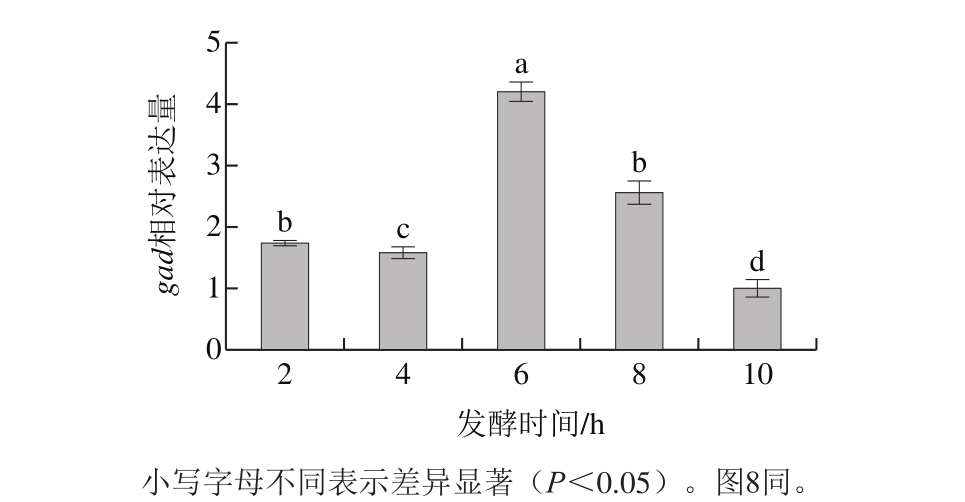
图5 不同发酵时间点gad相对表达量
Fig. 5 Relative expression levels of gad at different fermentation times
实时荧光定量PCR应用荧光信号的强弱与扩增物的量呈正比来实现定量,已广泛应用于基因突变检测、微生物学检测、基因表达的研究等领域 [28-29] 。图5结果表明,在不同发酵时间均检测到gad的mRNA,表明转入的gad在转录水平上已表达。gad表达量随着诱导时间的延长大体呈先升高后降低的趋势,与发酵相同时间点GABA产量趋势总体上一致(图5和图8)。发酵2~4 h,GABA产量已有显著增加,而此时的GAD基因gad相对表达量变化并不显著;6 h相对表达量达到最高值,为10 h的4.23倍;8 h相对表达量较6 h有所降低,而GABA产量有所推迟并在8 h达到最大值。本实验与基因表达相比,相关产物的合成具有一定的滞后性,但又不是单纯的滞后关系,Mazzoli等 [20] 报道GABA产量在基因或者转录水平上与GAD编码基因的关系不是非常紧密,可能受到其他基因的调控。
2.4.2 重组菌GAD表达蛋白SDS-PAGE分析

图6 pET-28a-gad表 达蛋白SDS-PAGE图
Fig. 6 SDS-PAGE analysis of expressed product from pET-28a-gad
将重组的pET-28a-gad转入表达型E. coli BL21(DE3)加入IPTG和PLP进行诱导表达,经过垂直电泳并染色再脱色,结果见图6。与E. coli BL21(DE3)相比,重组菌均在54 kDa附近出现特异性条带,与Park等 [17] 报道的GAD大小一致。随着诱导时间的延长,蛋白表达量相应的增多,在诱导8 h时蛋白表达量达到最大,随后有所减低。这与田灵芝 [29] 的研究结果相同,即用E. coli BL21(DE3)作为宿主细胞时,宿主菌本身的蛋白酶能降解异源蛋白,在诱导末期时较为明显,因此最佳诱导表达时间为8 h。
2.4.3 重组菌产GABA表达能力评估

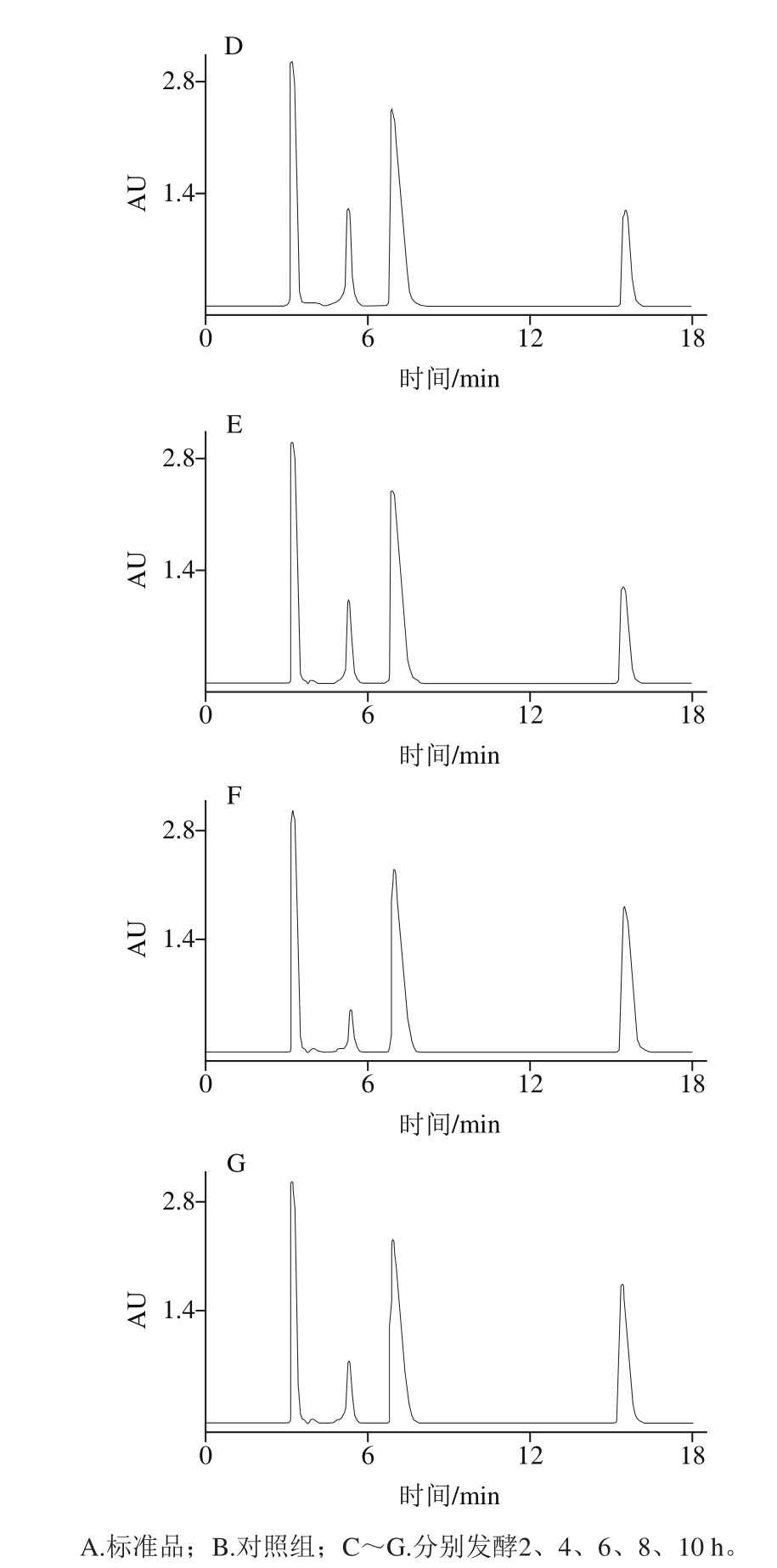
图7 不同发酵时间点GABA色谱图
Fig. 7 Chromatograms of GABA produced at different fermentation times
利用HPLC对不同诱导发酵时间重组菌产GABA能力进行评估,其标准曲线为Y=10 6 X-246 533(R 2 =0.999 9),可用于发酵液中GABA定量分析,结果见图7、8。结果表明,重组菌产GABA的最大值为2 387 mg/L,相比原始菌株L. plantarum BC114 GABA产量1 720 mg/L [3] ,提高了38.7%。GABA产量随着发酵时间的延长呈现增加趋势,随后有所降低,与对应的发酵时间蛋白表达量趋势变化一致。GABA产量在2~4 h显著增加,而此刻对应的蛋白量也增加,两者都在发酵8 h达到最大值,在发酵合成GABA主要受到GAD的影响 [30] ,因此酶含量越高更利于将L-谷氨酸钠转化为GABA。随着发酵时间的继续延长酶含量有所降低,可能是因为E. coli BL21(DE3)本身的蛋白酶能降解异源GAD从而导致GABA含量降低。

图8 不同发酵时间点GABA产量
Fig. 8 GABA accumulation levels at different time points of fermentation
本研究利用前期实验室筛选的L. plantarum BC114 GAD进行克隆,并对其进行生物信息学分析,GAD核酸序列长度1 410 bp,编码469 个氨基酸,等电点为5.58,蛋白的二级结构由α-螺旋、β-折叠、无规则卷曲构成,分别占32.2%、11.5%、56.3%,实现酶蛋白三级结构的建模预测;成功构建了基因工程菌E.coli BL21(DE3)/pET-28a-gad,在IPTG和PLP的诱导下,该重组菌表达了一个特异性的分子质量约为54 kDa的融合蛋白。重组菌生物合成GABA量最高达到2 387 mg/L,相比L. plantarum BC114提高了38.7%。GABA代谢物的生成与gad的转录、表达不是同步进行,而是具有一定滞后性。本实验为进一步进行GAD分子改造和提高GABA产量提高提供一定的理论依据。
参考文献:
[1] DIANA M, QUILEZ J, RAFECAS M. Gamma-aminobutyric acid as a bioactive compound in foods: a review[J]. Journal of Functional Foods, 2014, 10(3): 407-420. DOI:10.1016/j.jff.2014.07.004.
[2] 林亲录, 王婧, 陈海军. γ-氨基丁酸的研究进展[J]. 现代食品科技,2008, 24(5): 496-500. DOI:10.3969/j.issn.1673-9078.2008.05.028.
[3] 曾林, 刘波, 许小艳, 等. 四川泡菜中产γ-氨基丁酸微生物的系统发育与表达能力评估[J]. 食品科学, 2017, 38(2): 87-91. DOI:10.7506/spkx1002-6630-201702014.
[4] SANCHART C, RATTANAPOM O, HALTRICH D, et al.Technological and safety properties of newly isolated GABA-producing Lactobacillus futsaii strains[J]. Journal of Applied Microbiology, 2016, 121(3): 734-745. DOI:10.1111/jam.13168.
[5] VO T D L, KIM T W, HONG S H. Effects of glutamate decarboxylase and gamma-aminobutyric acid (GABA) transporter on the bioconversion of GABA in engineered Escherichia coli[J]. Bioprocess and Biosystems Engineering, 2012, 35(4): 645-648. DOI:10.1007/s00449-011-0634-8.
[6] BANCHUEN J, THAMMANRUTWASIK P, OORAIKUL B, et al.Increasing the bio-active compounds contents by optimizing the germination conditions of Southern Thai Brown Rice[J]. Songklanakarin Journal of Science & Technology, 2010, 32(3): 174-176.
[7] BHANWAR S, BAMNIA M, GHOSH M, et al. Use of Lactococcus lactis to enrich sourdough bread with γ-aminobutyric acid[J].International Journal of Food Sciences & Nutrition, 2013, 64(1):77-81. DOI:10.3109/09637486.2012.700919.
[8] CHO Y R, CHANG J Y, CHANG H C. Production of gammaaminobutyric acid (GABA) by Lactobacillus buchneri isolated from kimchi and its neuroprotective effect on neuronal cells[J]. Journal of Microbiology & Biotechnology, 2007, 17(1): 104.
[9] 田灵芝, 徐美娟, 饶志明. 一株重组大肠杆菌/pET-28a-lpgad的构建及其高效生产γ-氨基丁酸转化条件的优化[J]. 生物工程学报, 2012,28(1): 65-75.
[10] 乌云达来, 张博润, 郭雪娜, 等. 生物合成γ-氨基丁酸酿酒酵母谷氨酸脱羧酶基因的克隆与表达[J]. 食品科学, 2015, 36(13): 131-136.DOI:10.7506/spkx1002-6630-201513025.
[11] 陈琳, 张充, 吕凤霞, 等. 重组谷氨酸脱羧酶大肠杆菌合成γ-氨基丁酸条件的优化[J]. 食品科学, 2015, 36(1): 158-163. DOI:10.7506/spkx1002-6630-201501030.
[12] 黄燕, 宿玲恰, 吴敬, 等. 重组谷氨酸脱羧酶制备γ-氨基丁酸的工艺条件优化[J]. 生物技术通报, 2016, 32(6): 199-204. DOI:10.13560/j.cnki.biotech.bull.1985.2016.06.029.
[13] SHI F, LI Y. Synthesis of γ-aminobutyric acid by expressing Lactobacillus brevis-derived glutamate decarboxylase in the Corynebacterium glutamicum strain ATCC 13032[J]. Biotechnology Letters, 2011, 33(12): 2469-2474. DOI:10.1007/s10529-011-0723-4.
[14] 张天萌. 谷氨酸脱羧酶的克隆表达及酶学性质研究[D]. 无锡: 江南大学, 2012.
[15] KOMATSUZAKI N, NAKAMURA T, KIMURA T, et al.Characterization of glutamate decarboxylase from a high gammaaminobutyric acid (GABA)-producer Lactobacillus paracasei[J].Bioscience Biotechnology & Biochemistry, 2008, 72(2): 278.DOI:10.1271/bbb.70163.
[16] HUANG J, MEI L, SHENG Q, et al. Purification and characterization of glutamate decarboxylase of Lactobacillus brevis CGMCC 1306 isolated from fresh milk[J]. Chinese Journal of Chemical Engineering,2007, 15(2): 157-161. DOI:10.1016/S1004-9541(07)60051-2.
[17] PARK K, OH S. Cloning, sequencing and expression of a novel glutamate decarboxylase gene from a newly isolated lactic acid bacterium, Lactobacillus brevis OPK-3[J]. Bioresource Technology,2007, 98(2): 312. DOI:10.1016/j.biortech.2006.01.004.
[18] 李杰. 生物合成γ-氨基丁酸酵母菌谷氨酸脱羧酶基因的克隆及表达[D].金华: 浙江师范大学, 2009.
[19] SOMASUNDARAM S, TRAN K N T, RAVIKUMAR S, et al.Introduction of synthetic protein complex between Pyrococcus horikoshii glutamate decarboxylase and Escherichia coli GABA transporter for the improved production of GABA[J]. Biochemical Engineering Journal, 2017, 120: 1-6. DOI:10.1016/j.bej.2016.12.020.
[20] MAZZOLI R, PESSIONE E, DUFOUR M, et al. Glutamate-induced metabolic changes in Lactococcus lactis NCDO 2118 during GABA production: combined transcriptomic and proteomic analysis[J]. Amino Acids, 2010, 39(3): 727-737. DOI:10.1007/s00726-010-0507-5.
[21] 李理, 满朝新, 刘少敏, 等. 植物乳杆菌(Lactobacillus plantarum)NDC75017产γ-氨基丁酸的影响因素及其代谢机制[J]. 食品科学,2015, 36(7): 84-89. DOI:10.7506/spkx1002-6630-201507016.
[22] 齐育平, 孙蕾, 刘琴英, 等. 短乳杆菌谷氨酸脱羧酶的生物信息学分析[J]. 微生物学杂志, 2014, 34(1): 5-11. DOI:10.3969/j.issn.1005-7021.2014.01.002.
[23] FAN E, HUANG J, HU S, et al. Cloning, sequencing and expression of a glutamate decarboxylase gene from the GABA producing strain Lactobacillus brevis CGMCC 1306[J]. Annals of Microbiology, 2012,62(2): 689-698. DOI:10.1007/s13213-011-0307-5.
[24] 张祥, 方卉, 谢东芳, 等. 低H + -ATPase活性植物乳杆菌突变菌筛选及基因表达的相对定量分析[J]. 微生物学报, 2017, 57(2): 293-303.DOI:10.13343/j.cnki.wsxb.20160304.
[25] DIANA M, TRES A, QUILEZ J, et al. Spanish cheese screening and selection of lactic acid bacteria with high gamma-aminobutyric acid production[J]. LWT-Food Science and Technology, 2014, 56(2):351-355. DOI:10.1016/j.lwt.2013.11.027.
[26] GRONE B P, MARUSKA K P. Three distinct glutamate decarboxylase genes in vertebrates[J]. Scientific Reports, 2016, 6:30507. DOI:10.1038/srep30507.
[27] 郁凯. 以计算机辅助分子设计方法改造谷氨酸脱羧酶的研究[D].杭州: 浙江大学, 2013.
[28] NOLAN T, HANDS R E, BUSTIN S A. Quantification of mRNA using real-time RT-PCR[J]. Nature Protocols, 2006, 1(3): 1559-1562.DOI:10.1038/nprot.2006.236.
[29] 田灵芝. 高效转化生产γ-氨基丁酸重组大肠杆菌构建及其条件优化[D].无锡: 江南大学, 2012.
[30] LI H, CAO Y. Lactic acid bacterial cell factories for gammaaminobutyric acid[J]. Amino Acids, 2010, 39(5): 1107-1111.DOI:10.1007/s10035-005-0215-0.
Cloning and Expressing of Glutamate Decarboxylase Gene from Lactobacillus plantarum Producing γ-Aminobutyric Acid
TAN Xiao 1,2 , SUN Qing 1,2 , ZENG Lin 1,2 , ZHAO Tingting 1,2 , TAN Jinlong 1 , ZHANG Qing 1,2, *, XIANG Wenliang 1
(1. Provincial Key Laboratory of Food Biotechnology of Sichuan, College of Food and Bioengineering, Xihua University,Chengdu 610039, China; 2. Biotechnology Institute of Ancient Brewing, Xihua University, Chengdu 610039, China)
Abstract: The gene encoding glutamate decarboxylase (GAD), a key enzyme for the biosynthesis of γ-aminobutyric acid(GABA), from GABA-producing Lactobacillus plantarum BC114 was amplified by using PCR. Bioinformatics analysis of this gene was performed. Then, the target gene was subcloned into the expression vector pGEM-T-gad and the recombinant plasmids were transformed into competent Escherichia coli BL21 (DE3) for heterogeneous expression. Real-time reverse transcription PCR (RT-PCR), polyacrylamide gel electrophoresis (PAGE) and high performance liquid chromatography(HPLC) were used to measure the mRNA and protein expression of GAD and GABA production, respectively. Results showed that the size of the GAD gene from L. plantarum BC114, encoding 469 amino acids, was 1 410 bp. The predicted secondary structure of the GAD protein contained 32.2% α-helix, 11.5% β-sheet and 56.3% random coil. Meanwhile, the three dimensional structure was also predicted by using homologous modeling method. The recombinant E. coli harboring the gad gene was obtained successfully. The highest expression level of the gad gene was achieved after induction for 6 h,while the maximum protein expression level appeared 2 h later and the highest production of GABA of 2 387 mg/L was also obtained at this time.
Keywords: Lactobacillus plantarum BC114; γ-aminobutyric acid; glutamate decarboxylase; cloning; expression;bioinformatics
TAN Xiao, SUN Qing, ZENG Lin, et al. Cloning and expressing of glutamate decarboxylase gene from Lactobacillus plantarum producing γ-aminobutyric acid[J]. Food Science, 2018, 39(18): 159-165. (in Chinese with English abstract)DOI:10.7506/spkx1002-6630-201818025. http://www.spkx.net.cn
谭霄, 孙擎, 曾林, 等. 1 株产γ-氨基丁酸植物乳杆菌谷氨酸脱羧酶基因的克隆与表达[J]. 食品科学, 2018, 39(18):159-165. DOI:10.7506/spkx1002-6630-201818025. http://www.spkx.net.cn
文章编号: 1002-6630(2018)18-0159-07
引文格式:
中图分类号: TQ920.6
文献标志码: A
*通信作者简介: 张庆(1979—),男,副教授,博士,研究方向为食品微生物技术。E-mail:biozhangq@163.com
DOI: 10.7506/spkx1002-6630-201818025
基金项目: 四川省应用基础项目( 2016JY0253);四川省科技成果转化示范项目(2016CC0074);四川省食品生物技术重点实验室项目(szjj2016-019);西华大学大健康管理发展中心重点项目(szjj2017-037)
第一作者简介: 谭霄(1992—),女,硕士研究生,研究方向为食品微生物技术。E-mail:tx18200133971@163.com
收稿日期: 2017-07-12