表 1 单因素方差分析鲐鱼和大黄鱼冷藏期间体表细菌群落多样性
Table 1 One-way ANOVA analysis for the diversity of bacterial communities on mackerel and large yellow croaker during refrigerated storage
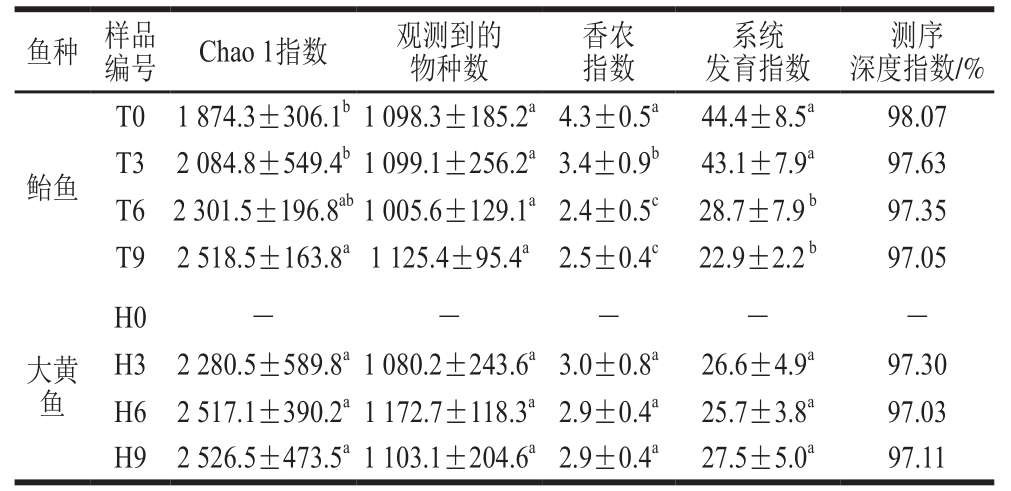
注:—.未测出,下同;对于同种鱼,同列肩标不同字母表示差异显著(P<0.05)。
程三红1,汤海青2,欧昌荣1,*,张梦思1,昝春兰1,李亚敏1
(1.宁波大学海洋学院,浙江 宁波 315211;2.浙江医药高等专科学校食品学院,浙江 宁波 315100)
摘 要:为研究鲐鱼和大黄鱼冷藏期间体表细菌群落和代谢功能的差异,采用Illumina MiSeq测序技术和PICRUSt工具对鲐鱼和大黄鱼冷藏期间体表细菌群落和代谢功能进行比较分析。结果表明:鲐鱼体表细菌群落的丰富度随冷藏时间的延长而增加,多样性却下降;大黄鱼体表细菌群落的丰富度和多样性变化均不显著。嗜冷杆菌属(Psychrobacter)和希瓦氏菌属(Shewanella)分别是鲐鱼和大黄鱼体表的优势菌,且其相对丰度分别与鲐鱼(r=0.709,P<0.001)和大黄鱼(r=0.600,P=0.008)挥发性盐基氮含量显著正相关;因此,它们可能分别是鲐鱼和大黄鱼体表的特定腐败菌。鲐鱼体表细菌的蛋氨酸、酪氨酸和组氨酸等参与氨基酸代谢的相关基因的相对丰度显著高于同一冷藏时期的大黄鱼(P<0.05),这在一定程度上从细菌代谢水平解释了鲐鱼比大黄鱼更易腐败的原因。研究结果可为不同水产品采取针对性的贮藏保鲜策略提供参考。
关键词:鲐鱼;大黄鱼;体表细菌群落;代谢功能;Illumina MiSeq测序;PICRUSt
鱼类由于高营养、高水分及接近中性的pH值特点使其在流通运输中极易受微生物活动和代谢的影响而腐败变质[1]。鲜活健康的鱼类肌肉组织内部无菌,但在其体表、腮和消化道内存在大量微生物,鱼类死亡后,这些微生物逐渐侵入鱼体进行繁殖和代谢,产生氨、胺类、硫化物、酯、酮、醛和挥发性脂肪酸等物质,引起鱼体腐败[2]。据文献报道,对鱼类腐败起主导作用的微生物通常只有一种或几种,即特定腐败菌[3]。目前,鱼类特定腐败菌的研究多集中在肌肉微生物方面[4-5],关于其体表微生物的研究则相对较少。然而,研究表明,鱼类体表微生物对鱼体腐败起着至关重要的作用[6]。因此,分析鱼类体表微生物组成及其代谢功能的变化对控制鱼类腐败具有重要意义。
随着科技的进步,以Illumina、Roche 454和Ion Torrent等测序系统为代表的高通量测序技术快速发展[7],已成为微生物群落研究的重要工具。部分学者将其应用于熏鲑鱼[8]、黄鳍金枪鱼[9]、牡蛎[10]和虾[11]等水产食品微生物群落的研究,以揭示水产食品的优势腐败微生物。但国内外利用该技术对鱼类体表微生物进行的研究却比较少见。
此外,现有研究主要关注鱼类腐败细菌群落组成,缺乏其代谢相关的功能信息。PICRUSt(Phylogenetic Investigation of Communities by Reconstruction of Unobserved States)是一项基于16S序列预测细菌群落功能的软件[12]。Stellato等[13]采用PICRUSt对肉类及其相关的环境样本细菌群落进行功能预测,发现细菌群落和预测的代谢功能之间具有相关性。Ferrocino等[14]对不同包装的牛肉汉堡微生物进行功能预测,发现采用乳酸链球菌素抗菌包装能够降低样品中腐败相关的代谢功能基因的丰度,延长产品的货架期。由此可见,采用PICRUSt功能预测可以将细菌群落与其代谢功能联系起来,更好地阐述食品加工贮藏过程中细菌群落组成、代谢功能和腐败变质之间的关系。
鲐鱼(Pneumatophorus japonica)和大黄鱼(Pseudosciaena crocea)是我国重要的海水经济鱼类,营养丰富,深受消费者喜爱,在水产市场占有较大份额。然而,鲐鱼属于红肉鱼类,大黄鱼属于白肉鱼类,红肉鱼为适应持续的洄游运动,常含有较多的蛋白质、脂质、糖和酶等。因此,同一贮藏条件下,鲐鱼往往比大黄鱼更容易受生理生化和微生物等因素影响而腐败变质[15]。鉴于此,本实验选取鲐鱼和大黄鱼为研究对象,采用Illumina MiSeq测序技术和PICRUSt功能预测来比较分析鲐鱼和大黄鱼冷藏期间体表细菌群落和代谢功能的差异,揭示两种鱼的特定腐败微生物,并探讨鲐鱼更容易腐败的原因,以期为靶向抑制水产品的腐败提供参考。
鲐鱼和大黄鱼由宁波梦婕食品有限公司提供。其中,鲐鱼(体质量250~300 g,体长25~30 cm)捕获于中国东海海域,大黄鱼(体质量400~450 g,体长30~35 cm)为宁波象山港海域深水网箱养殖鱼。
细菌基因组DNA快速提取试剂盒、λ DNA/Hind Ⅲ北京天根公司;Gelview核酸染料 北京百泰克公司;6×Loading Buffer、琼脂糖 北京全式金公司;溶菌酶、50×TAE 北京索莱宝公司。
H1850R高速冷冻离心机 湖南湘仪实验室仪器开发有限公司;凝胶成像系统、新JY300C电泳仪、JY-SPCT电泳槽 北京君意东方电泳设备有限公司;ND-1000分光光度计 美国Thermo Scientific公司;MiSeq测序仪 美国Illumina公司。
1.3.1 原料处理
鲐鱼和大黄鱼捕获后,在船上用无菌袋单独包装,层冰层鱼置于冰盒中12 h内运送至实验室,分别随机分成4 组,于4 ℃冰箱冷藏。取样测定时间分别为第0、3、6、9天(鲐鱼和大黄鱼分别记为:T0、T3、T6、T9和H0、H3、H6、H9),每个取样点各取6 尾鲐鱼和大黄鱼进行平行实验。
1.3.2 TVB-N含量的测定
参照国家水产行业标准SC/T 3032—2007《水产品中挥发性盐基氮的测定》[16]测定挥发性盐基氮(total volatile basic nitrogen,TVB-N)的含量,取鱼体背部肌肉进行测定,每个样品做3 个平行。
1.3.3 基因组DNA提取
鱼类体表微生物收集参考Ozaktas等[17]的方法进行。用无菌棉球沿鱼体背部中线长度约8 cm的表皮进行擦拭,两侧擦拭面积共约30 cm2,同时避开腹部和腮部以免肠道和腮部微生物污染。将棉球置于10 mL无菌离心管中,加入2 mL 8.5 g/L无菌生理盐水,高速漩涡2 min,吸出菌悬液置于2 mL离心管中,4 ℃、10 000×g离心20 min,收集沉淀。用细菌基因组DNA提取试剂盒按照说明书进行DNA提取。质量分数0.7%琼脂糖凝胶电泳检测DNA质量,ND-1000分光光度计测定DNA浓度和纯度。DNA样本于-80 ℃保存备用。
1.3.4 PCR扩增和测序
使用1 6 S r R N A基因引物3 4 1 F(5’-CCTACGGGNGGCWGCAG-3’)和805R(5’-GACTACHVGGGTATCTAATCC-3’)对细菌16S rRNA基因V3~V4高变区进行聚合酶链式反应(polymerase chain reaction,PCR)扩增。
PCR条件如下:95 ℃预变性3 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s,33 个循环;72 ℃延伸5 min。每个样品设计3 个平行进行PCR扩增以减少扩增造成的偏差。经凝胶电泳检测,PCR产物片段大小符合预期结果,且没有非特异性条带,用PCR片段纯化试剂盒纯化。使用ND-1000分光光度计测定浓度,每个样品取等量的PCR产物混合,用Illumina MiSeq测序平台测序。
1.3.5 测序数据处理
运用FLASH软件(version1.2.7,http://ccb.jhu.edu/software/FLASH/)对原始数据进行拼接;使用QIIME(version 1.9.0,http://qiime.org/)进行质控和去除嵌合体序列(Chimera sequence)[18],用Uclust在97%相似性水平上将序列聚类成操作分类单元(operational taxonomic units,OTUs)[19];选取丰度和覆盖度最高的序列作为每个OTU的代表序列,用PyNAST在Greengenes数据库中比对进行物种注释[20];去除古菌、叶绿体和非细菌序列及仅有一条序列的OTUs[21]。为消除样品间由于测序深度不同造成的偏差,对每个样品随机选取23 330 条(最低测序深度)序列用于后续分析。利用PICRUSt (http://picrust.github.io/picrust/)工具将OTU表标准化并与KEGG(Kyoto Encyclopedia of Genes and Genomes)数据库比对进行功能预测分析[12]。
用QIIME软件计算Chao 1和系统发育指数;用R软件(v.3.1.1)中vegan软件包计算细菌群落香农指数;参考Andersson等[22]计算测序深度指数;使用SPSS 17.0软件进行单因素方差分析(analysis of variance,ANOVA)和独立样本t检验;利用PAST软件基于Bray-Curtis距离的主坐标分析(principal coordinates analysis,PCoA)评估各样本细菌群落的差异性;利用PAST软件的群落相似性分析检测两种鱼体表菌群结构的差异及差异显著性[23]。利用Pearson相关性分析筛选与TVB-N含量相关的细菌属。
对48 个样本DNA进行PCR扩增、测序。其中,H0样品经PCR后未能扩增出目标条带,可能是由于冷藏初期大黄鱼体表细菌较少,导致提取的DNA浓度过低或者未抽提出;因此在后续数据处理中去除了对H0样品的分析。其他42 个样品经拼接、过滤处理后,共得到1 308 492 条序列,平均每个样本得到(31 154±2 760)条序列。每个样品随机选取23 330 条序列计算鲐鱼和大黄鱼冷藏期间体表细菌群落丰富度和多样性(表1)。Chao1指数表征物种丰富度,Chao1指数越大,物种丰富度越高;观察到的物种数表征样品中含有的物种数目,观察到的物种数值越大,物种丰富度越高;香农指数和系统发育指数则表征菌群多样性,其值越大,群落多样性就越高。结果表明,鲐鱼体表细菌群落的Chao1指数和观测到的物种数总体上均随冷藏时间的延长而上升,但其细菌群落的香农指数和系统发育指数则随冷藏时间的延长而显著下降(P<0.05);从冷藏第3~9天,大黄鱼体表细菌群落的Chao1指数、观测到的物种数、香农指数和系统发育指数变化均不显著(P>0.05)。本研究中,所有样本测序深度指数均在97%以上,说明测序结果能够反映样品中绝大多数微生物信息。
表 1 单因素方差分析鲐鱼和大黄鱼冷藏期间体表细菌群落多样性
Table 1 One-way ANOVA analysis for the diversity of bacterial communities on mackerel and large yellow croaker during refrigerated storage

注:—.未测出,下同;对于同种鱼,同列肩标不同字母表示差异显著(P<0.05)。
在门水平上,鲐鱼和大黄鱼体表细菌主要隶属于6 个门(图1a,平均相对丰度均大于1%)。其中,变形菌门(Proteobacteria)在两种鱼体表均占优势地位,其次是厚壁菌门(Firmicute)。变形菌门相对丰度在鲐鱼体表随冷藏时间的延长而上升,冷藏末期达到99.8%,但在大黄鱼体表却呈下降趋势;与之相反,厚壁菌门相对丰度在鲐鱼冷藏前期处于较高水平,后期则急剧下降,但在大黄鱼体表却随冷藏时间的延长呈上升趋势。
在属水平上,鲐鱼和大黄鱼体表菌相差异较大(图1b,平均相对丰度均大于1%)。冷藏初期,鲐鱼体表菌相以嗜冷杆菌属(Psychrobacter)和葡萄球菌属(Staphylococcus)为主。冷藏过程中,鲐鱼和大黄鱼体表优势细菌分别以嗜冷杆菌属和希瓦氏菌属(Shewanella)为主。嗜冷杆菌属在鲐鱼体表始终处于绝对优势地位,并随冷藏时间的延长而上升;希瓦氏菌属在大黄鱼冷藏第3~6天大幅上升,其后略微下降。鲐鱼体表次要微生物为葡萄球菌属和不动杆菌属(Acinetobacter)等。葡萄球菌属和不动杆菌属在鲐鱼冷藏前期有较高分布,后期则处于较低水平。大黄鱼体表次要细菌为发光杆菌属(Photobacterium),仅在大黄鱼冷藏第3天有较高分布,后期逐渐下降。其他细菌属在鲐鱼和大黄鱼冷藏期间相对丰度始终处于较低水平。

图 1 鲐鱼和大黄鱼冷藏期间体表细菌在门(a)和属(b)水平上的平均相对丰度
Fig. 1 Average relative abundance of bacterial communities on mackerel and large yellow croaker at the phylum (a) and genus (b) levels during refrigerated storage
利用PCoA分析鲐鱼和大黄鱼体表细菌群落结构差异,图中各样品之间的相对距离能够反映其细菌群落的相似程度,距离越近,相似性越高,结果显示不同鱼种之间的体表菌群差异性总体上随冷藏时间的延长而逐渐增加(图2a)。此外,同种鱼的6 个平行样本之间的体表菌群差异性却随冷藏时间的延长逐渐减小(图2b、c)。细菌群落相似性检验表明鲐鱼和大黄鱼之间的体表细菌群落组成具有高度显著差异(P<0.001);冷藏期间,除鲐鱼样品在0 d和3 d之间以及大黄鱼在6 d和9 d之间差异不显著外,同种鱼的其他样品之间均具有极显著差异(P<0.01)(表2)。

图 2 鲐鱼和大黄鱼冷藏期间体表细菌群落主坐标分析
Fig. 2 Principal coordinate analysis for bacterial communities on mackerel and large yellow croaker during refrigerated storage
表 2 基于Bray-Curtis距离的鲐鱼和大黄鱼冷藏期间体表细菌群落差异性分析
Table 2 Community dissimilarity based on the analysis of similarity using Bray-Curtis distance of bacterial communities on mackerel and large yellow croaker during refrigerated storage

注:对于同种鱼,上三角为样品间细菌群落差异性值R,下三角为样品间细菌群落差异性P值。

图 3 鲐鱼和大黄鱼冷藏期间TVB-N含量的变化
Fig. 3 Change in TVB-N contents of mackerel and large yellow croaker during refrigerated storage
如图3所示,整个冷藏期间鲐鱼和大黄鱼TVB-N含量均随冷藏时间的延长呈上升趋势,且鲐鱼的TVB-N含量极显著高于同一冷藏时期的大黄鱼(P<0.001)。冷藏末期鲐鱼TVB-N含量已超出GB 2733—2015《食品安全国家标准 鲜、冻动物性水产品》中规定的海水鱼的最高限值(30 mg/100 g)[24],而大黄鱼仍处于限值范围内。可见,同一冷藏条件下鲐鱼比大黄鱼更易分解产生TVB-N,腐败速度更快。
表 3 鲐鱼和大黄鱼冷藏期间体表细菌群落相对丰度 与TVB-N含量相关性
Table 3 Correlation between relative abundance and TVB-N content on mackerel and large yellow croaker during refrigerated storage

在属水平利用Pearson相关性分别检验鲐鱼和大黄鱼体表细菌群落相对丰度与TVB-N含量的相关性,挑出相对丰度>1%、r>0.5且P<0.05的TVB-N相关细菌,结果见表3。鲐鱼体表筛选到与TVB-N含量显著相关的细菌共10 种,其中与TVB-N含量呈正相关的有嗜冷杆菌属和弧菌属。大黄鱼体表筛选到与TVB-N含量显著相关的细菌共2 种,其中与TVB-N含量呈正相关的主要是希瓦氏菌属。
将鲐鱼和大黄鱼体表细菌群落进行PICRUSt功能预测,共获得296 种预测功能基因,其中与代谢相关的基因主要是氨基酸代谢、碳水化合物代谢、能量代谢、辅酶因子及维生素代谢、脂质代谢和核酸代谢相关基因等。线性回归分析显示,鲐鱼(r=0.809,P<0.001)和大黄鱼(r=0.771,P<0.001)的体表细菌群落相似性与功能相似性之间均显著正相关(图4)。将氨基酸、脂质和碳水化合物代谢相关基因的相对丰度分布特征绘制成热图(图5)(平均相对丰度>0.1%),发现鲐鱼和大黄鱼体表细菌代谢相关基因基本能够按照不同鱼种聚成两类,但鲐鱼在冷藏0 d时与大黄鱼聚成一类,这表明冷藏初期鲐鱼体表细菌代谢相关基因的相对丰度与大黄鱼类似,但中后期则差异较大。冷藏期间,鲐鱼和大黄鱼体表均有氨基酸、脂质和碳水化合物代谢相关基因的分布,且氨基酸代谢相关基因的相对丰度占据比例最大。鲐鱼体表细菌的半胱氨酸、蛋氨酸、组氨酸、色氨酸、酪氨酸等参与氨基酸代谢的基因相对丰度总体上随冷藏时间的延长而上升,而大黄鱼则表现出下降趋势。此外,鲐鱼体表细菌的蛋氨酸、酪氨酸、组氨酸等绝大部分参与氨基酸代谢相关基因的相对丰度显著高于同一冷藏时期的大黄鱼(P<0.05)。
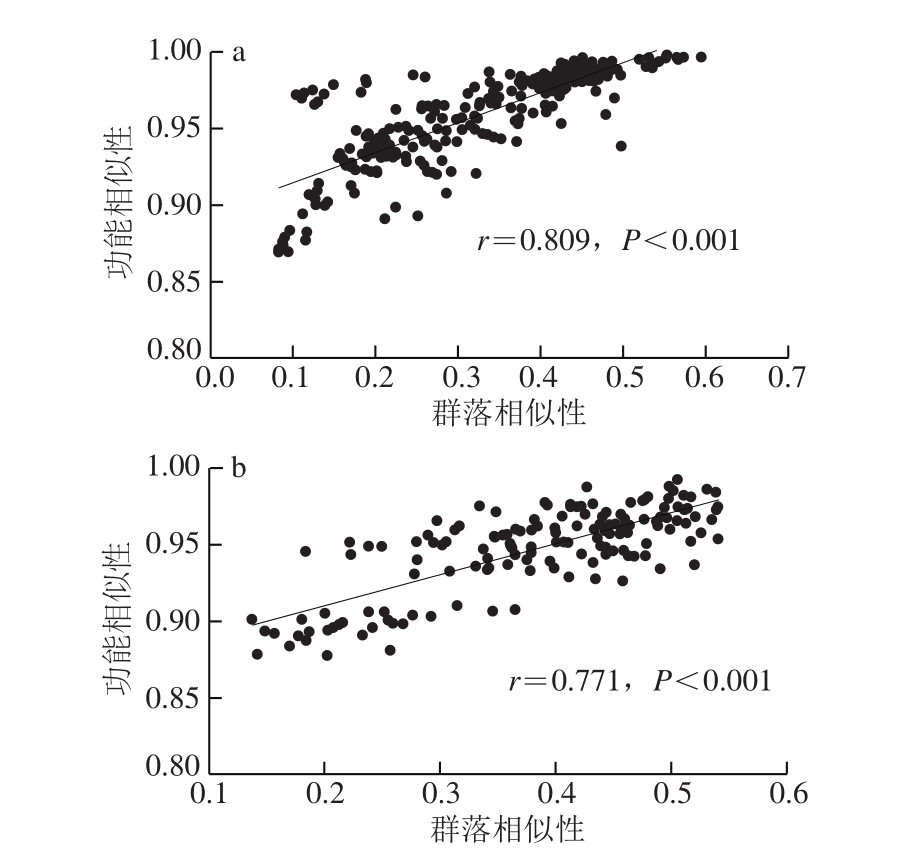
图 4 鲐鱼(a)和大黄鱼(b)冷藏期间体表细菌群落相似性与功能相似性的拟合相关性
Fig. 4 Correlation between composition and functional similarity of bacterial communities on mackerel (a) and large yellow croaker (b)during refrigerated storage

图 5 鲐鱼和大黄鱼冷藏期间体表细菌群落的氨基酸、脂质和碳水化合物代谢相关基因丰度分布热图
Fig. 5 Heatmap of the abundance of genes related to amino acid metabolism, lipid metabolism, and carbohydrate metabolism in bacterial communities on mackerel and large yellow croaker during refrigerated storage
高通量测序技术能够更真实、更全面地反映样本中微生物群落信息,本研究采用Illumina测序技术和PICRUSt功能预测比较分析了鲐鱼和大黄鱼冷藏期间体表细菌群落组成和代谢功能的差异。冷藏期间鲐鱼体表细菌群落的Chao1指数和观测到的物种数均随冷藏时间的延长而增加,香农指数和系统发育指数却下降,说明随冷藏时间的延长,鲐鱼体表细菌群落丰富度上升,多样性却下降,表面细菌主要由优势菌构成,这可能是菌群间的竞争和抑制作用所致[25]。大黄鱼在冷藏第0天未能抽提到DNA,可能是因为海水中微生物浓度较低,同时其体表黏液中含有凝集素、溶菌酶和抗菌肽等抗菌成分[26]。大黄鱼从冷藏第3~9天体表细菌群落的Chao1指数、观测到的物种数、香农指数和系统发育指数变化均不显著,说明其体表菌群丰富度和多样性在该时期相对稳定。
在门水平上,鲐鱼和大黄鱼体表主要优势菌为变形菌门。类似的,Larsen等[27]对6 种墨西哥湾鱼类体表微生物进行研究,也发现变形菌门是其体表优势菌。变形菌门广泛分布于海洋环境中,而鱼类体表细菌主要来自其周围的水生环境[28]。本研究中,大黄鱼采用深水网箱模式进行养殖,其生存环境高度模拟海水环境。因此,在细菌门水平,其体表菌相和鲐鱼等海水鱼类相似。
在属水平上,两种鱼体表菌相差异较大。冷藏期间鲐鱼体表优势菌始终以嗜冷杆菌属为主。嗜冷杆菌属是典型的耐寒性细菌,在多种水产品中均有检出[29]。本研究中,嗜冷杆菌属的相对丰度与鲐鱼TVB-N含量显著正相关。张雯[30]通过对无菌鱼肉进行接种研究,发现嗜冷杆菌属中的静止嗜冷杆菌(Psychrobacter immobilis)具有产三甲胺(trimethylamine,TMA)和TVB-N的能力。由此推测,嗜冷杆菌属可能是鲐鱼体表的特定腐败菌。此外,鲐鱼体表的弧菌属也与其TVB-N含量显著正相关,弧菌属种类较多,分解蛋白能力较强[10],但弧菌属多属于嗜温菌,低温能够抑制嗜温菌的生长。因此,弧菌属在冷藏期间相对丰度始终处于较低水平[31]。根据这一结果,在鲐鱼加工贮藏过程中,可采用抑制嗜冷杆菌属繁殖的方法对其进行处理,从而延长鲐鱼货架期。例如:张雯等[32]通过对峙实验和扩散抑菌实验发现,枯草芽孢杆菌BS08对分离自鱼体的嗜冷杆菌属具有强烈抑制作用。因此,可采用该益生菌对鲐鱼进行处理来延缓鱼体腐败。
从冷藏第3~9天,大黄鱼体表主要优势细菌为希瓦氏菌属,这可能与希瓦氏菌属具有较强的低温耐受性有关[33]。希瓦氏菌属是海水鱼类常见的腐败菌[34]。大量研究表明,希瓦氏菌属能还原氧化三甲胺(trimetlylamine oxide,TMAO)为TMA,并能生成H2S,使鱼体出现酸臭味[3]。本研究中,希瓦氏菌属的相对丰度亦与大黄鱼TVB-N含量呈显著正相关;因此,希瓦氏菌属可能是大黄鱼体表的特定腐败菌。值得注意的是,本研究中,嗜冷杆菌属与鲐鱼TVB-N含量表现出正相关性,却与大黄鱼TVB-N含量表现出负相关性,这可能是因为鲐鱼体表嗜冷杆菌属具有较强的环境适应能力,因而其相对丰度随冷藏时间的延长逐渐上升;相反的,大黄鱼体表嗜冷杆菌属在冷藏中后期可能受到乳酸菌分泌的过氧化氢、乳酸和细菌素等抗菌物质的抑制[35],其相对丰度随冷藏时间的延长而下降。为延长大黄鱼的货架期,可针对希瓦氏菌属采取一些抑菌措施。例如:He Mu等[36]采用10 g/L壳聚糖和6 g/L Nisin结合的保鲜剂对大黄鱼鱼肉进行浸泡处理,发现该保鲜剂能有效抑制鱼肉中希瓦氏菌的生长和生物膜的形成,从而减少鱼肉氨基酸的分解及腐败产物的生成,达到保鲜效果。
鱼类腐败细菌的繁殖常伴随着大量代谢产物的生成;因此,鱼类菌群结构在鱼体代谢产物形成方面起着重要作用。某些微生物在功能上有一定的相似性,当其中一种或几种微生物被去除后,细菌群落功能能基本保持不变,即微生物群落存在功能冗余[37]。本研究中,两种鱼体表细菌群落相似性与功能相似性之间均显著正相关,表明菌群功能冗余度相对较低[38]。鲐鱼体表细菌的氨基酸代谢相关基因的相对丰度随冷藏时间的延长而增加,大黄鱼却呈现出相反趋势,这可能与两种鱼体表细菌代谢能力随冷藏时间的延长而有不同的变化有关。冷藏初期,鲐鱼处于僵硬期,鱼肉pH值下降,酸性环境不适宜细菌的繁殖代谢,中后期随着氨和胺类碱性物质的累积,pH值上升,细菌的分解代谢能力逐渐增强[15];而大黄鱼体表细菌在冷藏中后期可能受到乳酸菌的抑制,分解代谢能力逐渐减弱[35]。同时这也解释了冷藏第0天鲐鱼体表细菌代谢相关基因的相对丰度与大黄鱼类似,中后期则差异较大的原因。
鱼肉的腐败主要与蛋白质、氨基酸等的分解有关。在细菌作用下,蛋氨酸和酪氨酸分解易生成含氮物质,使鱼体TVB-N含量升高[39];组氨酸分解则生成组胺,影响鱼类食用安全[5];色氨酸分解生成吲哚、甲基吲哚等,使鱼体产生异味[40]。一方面,鲐鱼体表细菌的蛋氨酸、组氨酸、色氨酸、酪氨酸等绝大部分参与氨基酸代谢的相关基因的相对丰度显著高于同一冷藏时期的大黄鱼;另一方面,鲐鱼皮肤细薄、易破肚,体表细菌极易侵入,且肌肉松软,富含组氨酸等游离氨基酸[15]。因此,当鲐鱼体表细菌侵入内部后,其氨基酸更易分解代谢产生TVB-N和组胺等不良物质,使鱼体腐败。研究表明,鲐鱼属于典型的高组胺鱼类[5],本研究中鲐鱼体表组氨酸代谢相关基因的相对丰度显著高于大黄鱼,这与鲐鱼更易产生组胺的特性一致。说明PICRUSt功能预测软件在组胺相关菌的研究上表现出良好的应用前景,可为组胺相关菌的研究提供新思路,相关研究值得进一步探讨。此外,鲐鱼体表的脂质和碳水化合物代谢相关基因的相对丰度也较高。在相关微生物作用下,鲐鱼比大黄鱼更易进行脂质和碳水化合物代谢产生醛、酮和酸等物质[41],使鱼体产生腥臭味,品质劣化。
冷藏期间,鲐鱼体表细菌群落的丰富度随冷藏时间的延长而增加,多样性却下降;大黄鱼体表细菌群落的丰富度和多样性变化均不显著。鲐鱼和大黄鱼体表的特定腐败菌可能分别是嗜冷杆菌属和希瓦氏菌属。与大黄鱼相比,鲐鱼体表细菌具有更高丰度的蛋氨酸、酪氨酸和组氨酸等参与氨基酸代谢的相关基因,这在一定程度上从微生物代谢水平解释了鲐鱼比大黄鱼更易腐败的原因。本研究可为鱼类针对性的贮藏保鲜技术的开发提供参考,为水产品加工贮藏过程中微生物组成及代谢功能变化的研究提供新思路。
参考文献:
[1] TAOUKIS P S, KOUTSOUMANIS K, NYCHAS G J E. Use of timetemperature integrators and predictive modelling for shelf life control of chilled fish under dynamic storage conditions[J]. International Journal of Food Microbiology, 1999, 53(1): 21-31. DOI:10.1016/S0168-1605(99)00142-7.
[2] GRAM L, HUSS H H. Microbiological spoilage of fish and fish products[J]. International Journal of Food Microbiology, 1996, 33(1):121-137. DOI:10.1016/0168-1605(96)01134-8.
[3] DALGAARD P. Qualitative and quantitative characterization of spoilage bacteria from packed fish[J]. International Journal of Food Microbiology, 1995, 26(3): 319-333. DOI:10.1016/0168-1605(94)00137-U.
[4] LI T T, LI J R, HU W Z. Changes in microbiological, physicochemical and muscle proteins of post mortem large yellow croaker(Pseudosciaena crocea)[J]. Food Control, 2013, 34(2): 514-520.DOI:10.1016/j.foodcont.2013.05.028.
[5] JIANG Q Q, DAI Z Y, ZHOU T, et al. Histamine production and bacterial growth in mackerel (Pneumatophorus Japonicus) during storage[J]. Journal of Food Biochemistry, 2013, 37(2): 246-253.DOI:10.1111/jfbc.12021.
[6] GENNARI M, TOMASELLI S. Changes in aerobic microflora of skin and gills of Mediterranean sardines (Sardina pilchardus) during storage in ice[J]. International Journal of Food Microbiology, 1988,6(4): 341-347. DOI:10.1016/0168-1605(88)90028-1.
[7] MARDIS E R. The impact of next-generation sequencing technology on genetics[J]. Trends in Genetics, 2008, 24(3): 133-141.DOI:10.1016/j.tig.2007.12.007.
[8] LEROI F, CORNET J, CHEVALIER F, et al. Selection of bioprotective cultures for preventing cold-smoked salmon spoilage[J].International Journal of Food Microbiology, 2015, 213: 79-87.DOI:10.1016/j.ijfoodmicro.2015.05.005.
[9] SILBANDE A, ADENET S, SMITH-RAVIN J, et al. Quality assessment of ice-stored tropical yellowf i n tuna (Thunnus albacares)and inf l uence of vacuum and modif i ed atmosphere packaging[J]. Food Microbiology, 2016, 60: 62-72. DOI:10.1016/j.fm.2016.06.016.
[10] 曹荣, 张井, 孟辉辉, 等. 高通量测序与传统纯培养方法在牡蛎微生物群落分析中的应用对比[J]. 食品科学, 2016, 37(24): 137-141.DOI:10.7506/spkx1002-6630-201624021.
[11] CHAILLOU S, CHAULOT-TALMON A, CAEKEBEKE H, et al.Origin and ecological selection of core and food-specific bacterial communities associated with meat and seafood spoilage[J]. The ISME Journal, 2015, 9(5): 1105-1108. DOI:10.1038/ismej.2014.202.
[12] LANGILLE M G I, ZANEVELD J, CAPORASO J G, et al. Predictive functional prof i ling of microbial communities using 16S rRNA marker gene sequences[J]. Nature Biotechnology, 2013, 31(9): 814-821.DOI:10.1038/nbt.2676.
[13] STELLATO G, LA STORIA A L, DE FILIPPIS F, et al. Overlap of spoilage-associated microbiota between meat and the meat processing environment in small-scale and large-scale retail distributions[J].Applied Environmental Microbiology, 2016, 82(13): 4045-4054.DOI:10.1128/AEM.00793-16.
[14] FERROCINO I, GREPPI A, LA STORIA A, et al. Impact of nisinactivated packaging on microbiota of beef burgers during storage[J].Applied Environmental Microbiology, 2015, 82(2): 549-559.DOI:10.1128/AEM.00793-16.
[15] 何敏元. 多获性红肉鱼类的处理及加工技术[J]. 国外水产, 1988(1):29-33.
[16] 农业部. 水产品中挥发性盐基氮的测定: SC/T 3032-2007[S]. 北京:中国农业出版社, 2007: 1-2.
[17] OZAKTAS T, TASKIN B, GOZEN A G. High level multiple antibiotic resistance among fish surface associated bacterial populations in nonaquaculture freshwater environment[J]. Water Research, 2012, 46(19):6382-6390. DOI:10.1016/j.watres.2012.09.010.
[18] CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, et al. QIIME allows analysis of high-throughput community sequencing data[J].Nature Methods, 2010, 7(5): 335-336. DOI:10.1038/nmeth.f.303.
[19] CAPORASO J G, BITTINGER K, BUSHMAN F D, et al. PyNAST:a flexible tool for aligning sequences to a template alignment[J].Bioinformatics, 2010, 26(2): 266-267. DOI:10.1093/bioinformatics/btp636.
[20] DESANTIS T Z , HUGENHOLTZ P, LARSEN N, et al. Greengenes, a chimera-checked 16S rRNA gene database and workbench compatible with ARB[J]. Applied and Environmental Microbiology, 2006, 72(7):5069-5072. DOI:10.1128/AEM.03006-05.
[21] XIONG J B, DAI W F, ZHU J Y, et al. The Underlying ecological processes of gut microbiota among cohabitating retarded, overgrown and normal shrimps[J]. Microbial Ecology, 2016, 73(4): 998-999.DOI:10.1007/s00248-016-0910-x.
[22] ANDERSSON A F, LINDBERG M, JAKOBSSON H, et al.Comparative analysis of human gut microbiota by barcoded pyrosequencing[J]. PLoS ONE, 2008, 3(7): e2836. DOI:10.1371/journal.pone.0002836.
[23] DAI W F, YU W N, ZHANG J J, et al. The gut eukaryotic microbiota influences the growth performance among cohabitating shrimp[J].Applied Microbiology and Biotechnology, 2017, 101(16): 6447-6457.DOI:10.1007/s00253-017-8388-0.
[24] 国家卫生和计划生育委员会. 食品安全国家标准 鲜、冻动物性水产品: GB 2733—2015[S]. 北京: 中国标准出版社, 2015: 1.
[25] ZHAO F, ZHOU G H, YE K P, et al. Microbial changes in vacuumpacked chilled pork during storage[J]. Meat Science, 2015, 100: 145-149. DOI:10.1016/j.meatsci.2014.10.004.
[26] SHEPHARD K L. Functions for fish mucus[J]. Reviews in Fish Biology and Fisheries, 1994, 4(4): 401-429. DOI:10.1007/BF00042888.
[27] LARSEN A, TAO Z, BULLARD S A, et al. Diversity of the skin microbiota of fishes: evidence for host species specificity[J]. FEMS Microbiology Ecology, 2013, 85(3): 483-494. DOI:10.1111/1574-6941.12136.
[28] YANG G M, BAO B L, PEATMAN E, et al. Analysis of the composition of the bacterial community in puffer fish Takifugu obscurus[J]. Aquaculture, 2007, 262(2/3/4): 183-191. DOI:10.1016/j.aquaculture.2006.11.031.
[29] BJØRKEVOLL I, OLSEN R L, SKJERDAL O T. Origin and spoilage potential of the microbiota dominating genus Psychrobacter in sterile rehydrated salt-cured and dried salt-cured cod (Gadus morhua)[J].International Journal of Food Microbiology, 2003, 84(2): 175-187.DOI:10.1016/S0168-1605(02)00418-X.
[30] 张雯. 冰鲜大黄鱼腐败菌的分析与生物保鲜剂的开发[D]. 福州: 福州大学, 2014: 38-84.
[31] 蒋倩倩, 戴志远, 赵巧灵, 等. 冰藏鲐鱼中组胺的产生及腐败微生物的变化[J]. 中国食品学报, 2013, 13(8): 219-224. DOI:10.16429/j.1009-7848.2013.08.025.
[32] 张雯, 倪莉, 陈雅静, 等. 枯草芽孢杆菌BS08对大黄鱼腐败菌的抑制及培养基优化[J]. 中国食品学报, 2013, 13(8): 102-109.DOI:10.16429/j.1009-7848.2013.08.039.
[33] VOGEL B F, VENKATESWARAN K, SATOMI M, et al.Identification of Shewanella baltica as the most important H2S-producing species during iced storage of Danish marine fish[J].Applied and Environmental Microbiology, 2005, 71(11): 6689-6697.DOI:10.1128/AEM.71.11.6689-6697.2005.
[34] GRAM L, DALGAARD P. Fish spoilage bacteria: problems and solutions[J]. Environmental Biotechnology, 2002, 13: 262-266.DOI:10.1016/S0958-1669(02)00309-9.
[35] TIAN X J, WU W, YU Q Q, et al. Bacterial diversity analysis of pork longissimus lumborum following long term ohmic cooking and water bath cooking by amplicon sequencing of 16S rRNA gene[J]. Meat Science, 2017, 123: 97-104. DOI:10.1016/j.meatsci.2016.09.007.
[36] HE Mu, GUO Quanyou, SONG Wei, et al. Inhibitory effects of chitosan combined with nisin on Shewanella spp. isolated from Pseudosciaena crocea[J]. Food Control, 2017, 79: 349-355.DOI:10.1016/j.foodcont.2017.04.012.
[37] WOHL D L, ARORA S, GLADSTONE J R. Functional redundancy supports biodiversity and ecosystem function in a closed and constant environment[J]. Ecology, 2004, 85(6): 1534-1540. DOI:10.1890/03-3050.
[38] ZHU J Y, DAI W F, QIU Q F, et al. Contrasting ecological processes and functional compositions between intestinal bacterial community in healthy and diseased shrimp[J]. Microbial Ecology, 2016, 72(4): 975-985. DOI:10.1007/s00248-016-0831-8.
[39] 何立超, 马素敏, 李成梁, 等. 辐照处理提高猪肉火腿肠保鲜效果[J]. 农业工程学报, 2016, 32(22): 296-302. DOI:10.11975/j.issn.1002-6819.2016.22.041.
[40] CHANG O, CHEUK W L, NICKELSON R, et al. Indole in shrimp: effect of fresh storage temperature, freezing and boiling[J]. Journal of Food Science, 1983, 48(3): 813-816. DOI:10.1111/j.1365-2621.1983.tb14906.x.
[41] CASABURI A, PIOMBINO P, NYCHAS G J, et al. Bacterial populations and the volatilome associated to meat spoilage[J]. Food Microbiology, 2015, 45: 83-102. DOI:10.1016/j.fm.2014.02.002.
Comparative Analysis of Compositions and Metabolic Functions of Bacterial Communities on the Surface of Mackerel and Large Yellow Croaker during Refrigerated Storage
CHENG Sanhong1, TANG Haiqing2, OU Changrong1,*, ZHANG Mengsi1, ZAN Chunlan1, LI Yamin1
(1. School of Marine Sciences, Ningbo University, Ningbo 315211, China;2. Faculty of Food Science, Zhejiang Pharmaceutical College, Ningbo 315100, China)
Abstract:The aims of this study were to compare the compositions and metabolic functions of the bacterial communities on mackerel and large yellow croaker during refrigerated storage by Illumina MiSeq sequencing and phylogenetic investigation of communities by reconstruction of unobserved states (PICRUSt). The results showed that the richness of bacteria on mackerel surface was increased while the bacterial diversity was decreased during prolonged storage; however, no signif i cant change was observed for large yellow croaker. Psychrobacter and Shewanella were the dominant bacteria on mackerel and large yellow croaker, respectively, and their relative abundance was positively correlated with total volatile basic nitrogen(TVB-N) contents (r = 0.709, P < 0.001 for mackerel; r = 0.600, P = 0.008 for large yellow croaker), respectively. This may suggest that Psychrobacter is the specif i c spoilage organism (SSO) on mackerel, while Shewanella is the SSO on large yellow croaker. The relative abundance of genes related to the metabolism of amino acids such as methionine, tyrosine and histidine on mackerel was higher than that on large yellow croaker at the same storage period (P < 0.05). This may partially explain why mackerel is more perishable than large yellow croaker based on the metabolic levels of the bacterial communities. This study provides a basis for adopting targeted strategy to the preservation of different aquatic products.
Keywords:mackerel; large yellow croaker; surface bacterial community; metabolic functions; Illumina MiSeq sequencing; PICRUSt
CHENG Sanhong, TANG Haiqing, OU Changrong, et al. Comparative analysis of compositions and metabolic functions of bacterial communities on the surface of mackerel and large yellow croaker during refrigerated storage[J]. Food Science, 2018,39(19): 218-225. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201819034. http://www.spkx.net.cn
程三红, 汤海青, 欧昌荣, 等. 鲐鱼和大黄鱼冷藏期间体表细菌群落组成和代谢功能的比较分析[J]. 食品科学, 2018,39(19): 218-225. DOI:10.7506/spkx1002-6630-201819034. http://www.spkx.net.cn
引文格式:
文章编号:1002-6630(2018)19-0218-08
文献标志码:A
中图分类号:TS254.4
DOI:10.7506/spkx1002-6630-201819034
*通信作者简介:欧昌荣(1974—),男,教授,博士,研究方向为水产品保鲜加工、食品风味化学。E-mail:ouchangrong@nbu.edu.cn
第一作者简介:程三红(1992—),女,硕士研究生,研究方向为水产品加工与贮藏。E-mail:chengsanhong@163.com
宁波大学科研基金项目(xkzsc1524)
宁波市市科学技术局农业重大择优委托项目(2012C10024);宁波市科技局富民项目(2017C10029);
浙江省公益研究项目(GN18C200033);“水产”浙江省重中之重学科开放基金项目(xkzsc1428);
基金项目:国家自然科学基金面上项目(31271890);国家自然科学基金青年科学基金项目(31501519);
收稿日期:2017-07-18