表1 通用PCR引物与自主设计的引物序列、退火温度及PCR产物
Table 1 Primer sequences for PCR amplif i cation of 16S rDNA, Cyt b,and COⅠ genes, annealing temperatures and PCR product size
陈文炳,邵碧英,缪婷玉,彭 娟,陈 彬,张志灯,江树勋
(福建出入境检验检疫局检验检疫技术中心,福建省检验检疫技术研究重点实验室,福建 福州 350003)
摘 要:建立6 种鳗鱼的物种多基因DNA条形码精准鉴定方法。以鳗鱼DNA为模板,采用3 对通用引物对6 种鳗鱼的3 个基因(16S rRNA、Cyt b、COⅠ)部分DNA片段进行聚合酶链式反应扩增、测序,结果6 种鳗鱼各获得3 条16S rDNA(638~643 bp)、Cyt b(464~466 bp)、COⅠ(705~707 bp)基因部分DNA序列,从中选取各物种序列同源的片段设计3 对新引物对6 种鳗鱼的16S rRNA、Cyt b、COⅠ基因部分DNA片段进行PCR扩增,其产物大小分别为504~507、400、609 bp,再从各片段中筛选出具有6 种鳗鱼物种特异性强的、碱基数分别为262、280、300 bp的3 条DNA片段序列,作为6 种鳗鱼物种的3 个基因的标准DNA条形码,应用DNAMAN V6软件进行同源性分析,并通过GenBank数据库的比对验证,制定了供检测实践用的同源率判别指标,建立鳗鱼物种的多基因条形码检测方法。应用该方法对30 个待检鳗鱼样品进行检测,结果表明,各样品基于3 个基因DNA条形码的比对,符合同源率指标,物种判别结果互相吻合,从而精准判别样品的所属物种。该方法稳定、精准、易于操作,可应用于6 种鳗鱼的物种鉴定,值得推广。
关键词:16S rRNA基因;Cyt b基因;COⅠ基因;多基因DNA条形码;鳗鱼物种鉴定
我国是鳗鱼养殖与加工出口大国,每年烤鳗与活鳗出口量达数万吨,货值达数十亿美元,主要出口国有日本、美国、俄罗斯、德国、韩国等。由于鳗鱼加工成熟食以后及进口鳗苗体积细小,难以从形态上辨别其种类。一方面不同种类的鳗鱼产品与鳗苗价格不同,另一方面,欧洲鳗(Anguilla anguilla)被自然保护国际联盟组织列为极度濒危物种[1-2],所有的欧洲鳗国际贸易都需要获得濒危物种贸易许可证。我国鳗鱼产品出口时,被进口国要求提供鳗鱼物种鉴定报告,如果产品物种属于欧洲鳗,又未能提供贸易许可证,将被视为非法贸易而被强行销毁处理。另外,近年来时有发现不良商家用在我国气候条件下无法正常生长成成品鳗的莫桑比克鳗苗冒充“南美鳗”,给养殖企业造成巨大损失。为了维护企业与消费者利益以及正常市场秩序,应企业与执法部门要求,建立鳗鱼种类的精准鉴定方法,对我国鳗鱼养殖业与加工出口外贸易具有重要的现实意义。
聚合酶链式反应(polymerase chain reaction,PCR)扩增技术在食品中鱼类物种成分的鉴别已有较多研究与应用[3-7],在鳗鱼中的应用主要是基于PCR的随机扩增多态性DNA遗传多样性分析与物种鉴别探讨的研究[8-12],但在检验检疫中实践应用,缺乏稳定性与准确性。DNA条形码是指生物体内能够代表该物种的,种间存在足够变异,种内相对稳定,易于PCR扩增、适当长度的DNA片段。最早由加拿大生物学家Hebert等[13]研究发现利用线粒体细胞色素C氧化酶亚基Ⅰ(mitochondrial cytochrome c oxidase subunitⅠ,COⅠ)基因一段长度为648 bp的片段,能够在DNA水平上成功的区分物种,提供一种快速、简便、可信的分类方法,并命名为DNA条形码技术。近年来,随着基因条形码技术的发展,除了COⅠ基因DNA序列作为标准条形码应用于物种鉴别[13-15]外,存在物种特异性的线粒体16S rRNA、Cyt b基因、COⅡ基因等的DNA序列,也被作为DNA条形码应用于物种鉴别和遗传多样性分析[16-21],国内有关于鳗鱼线粒体DNA的COⅠ与COⅡ基因序列分析及其系统分类的研究[22-24]报道。国外有人将鳗鱼COⅠ基因序列的DNA条形码与GenBank中鳗鱼DNA序列比对,而判定非法贸易的欧洲鳗物种,但该方法往往存在待测样品与GenBank中多个鳗鱼物种同源率相同,难以准确判断其所属具体物种[25-26]。目前国内外鲜见利用多基因DNA条形码进行鳗鱼物种鉴定的研究报道。本研究在基于16S rRNA基因部分DNA片段建立的DNA条形码(243 bp)鉴定美洲鳗(Anguilla rostrata)、欧洲鳗(A. anguilla)、日本鳗(A. japonica)3 种鳗鱼的基础[27]上,延长16S rRNA基因部分DNA片段为262 bp并筛选除了Cyt b基因与COⅠ基因等多基因的标准DNA条形码,建立了美洲鳗、欧洲鳗、日本鳗、莫桑比克鳗(A. mossambica)、花鳗(A. marmorata)、太平洋双色鳗(A. bicolor pacifica)6 种鳗鱼的多基因DNA条形码物种精准鉴定方法。为维护企业与消费者利益以及正常市场秩序,避免贸易壁垒,提供技术保障,对我国鳗鱼养殖业与加工出口对外贸易具有重要的现实意义。
日本鳗、美洲鳗、欧洲鳗样品,由福建省三明市华晟食品公司提供,莫桑比克鳗、花鳗(A. marmorata)、太平洋双色鳗样品由福州郊区鳗鱼养殖企业提供。用于方法应用实验的待测鳗鱼样品系企业委托实验室留样与鳗鱼养殖企业提供,制作成模拟盲样供实验。
十六烷基三甲基溴化铵 上海博亚生物技术有限公司;2×Taq PCR Master Mix、蛋白酶K、DNA MarkerⅠ 天根生化科技(北京)有限公司;琼脂糖英国Oxoid公司。
Ultrospec 1100 pro核酸蛋白分析仪 瑞典Amersham公司;T-Gradient梯度PCR仪 德国Biometra公司;Power Pac 1000电泳仪 美国Bio-Rad公司;GL212 PRO凝胶成像仪 美国Carestream公司。
1.3.1 引物设计
16S rRNA、Cyt b、COⅠ基因的3 个DNA部分片段PCR扩增通用引物序列[28-30]与自主设计的DNA条形码引物序列(16S rDNA 504、Cyt b 400、COⅠ609)、退火温度与PCR产物长度见表1,由生工生物工程(上海)股份有限公司合成。
表1 通用PCR引物与自主设计的引物序列、退火温度及PCR产物
Table 1 Primer sequences for PCR amplif i cation of 16S rDNA, Cyt b,and COⅠ genes, annealing temperatures and PCR product size

1.3.2 DNA的提取及浓度、纯度测定
参照文献[27]方法进行。1.5 mL离心管中加入约50 mg样品粉末或鲜样100 mg,加200 μL TE溶液(pH 8.0),旋涡混匀;加入400 μL裂解液,旋涡混匀;加600 μL Tri-饱和酚-氯仿-异戊醇溶液(25∶24∶1,V/V),剧烈振荡,12 000×g离心10 min;取上清液,加入等体积的氯仿-异戊醇溶液(24∶1,V/V),剧烈振荡,12 000×g离心10 min;取上清液,加0.8 倍体积异丙醇,12 000×g离心10 min;取沉淀,加入400 μL NaCl溶液(1 mol/L)于65 ℃温浴中溶解,再加入5 μL蛋白酶K(20 mg/mL),5 μL Rnase A(10 μg/mL),剧烈振荡30 s以上,37 ℃温浴1 h,期间定期晃动。加等体积的水饱和酚-氯仿-异戊醇溶液(25∶24∶1,V/V),振荡,12 000 r/min离心15 min,重复该步骤1~2 次,直到蛋白质去除干净为止。取上清液,加2 倍体积的无水乙醇,轻微晃动,12 000 r/min离心10 min。取沉淀,70%乙醇溶液清洗2 次,干燥,加100 μL TE(pH 8.0)溶解。每个样品设2 个重复。
样品DNA用核酸蛋白分析仪测定波长260 nm和280 nm处的OD,分别计算DNA纯度与质量浓度,计算公式如下:DNA纯度=OD260nm/OD280nm,比值在1.7~2.0之间较为理想;双链DNA质量浓度/(μg/mL)=50×OD260nm值。
1.3.3 PCR反应热循环参数的设置与PCR反应体系
PCR扩增条件:94 ℃预变性3 min。94 ℃变性30 s,50~62 ℃(各对引物相应温度见表1)退火时间30 s,72 ℃延伸60 s,40 个循环;72 ℃延伸10 min。4 ℃保存。反应体系为:10×Taq buffer 2.5 μL,Taq酶1.0 U,模板DNA 200 ng,MgCl23.5 mmol/L,dNTPs 300 μmol/L,上游引物0.5 μmol/L,下游引物0.5 μmol/L,用ddH2O将总体积补至25 μL。
1.3.4 PCR扩增产物的电泳检测
用0.5×TBE电泳缓冲液制备质量分数2%的琼脂糖凝胶。将8~10 μL的PCR扩增产物分别与1~2 μL的6×加样缓冲液(电泳前沿指示剂)混合后,在凝胶板上的孔中点样。用分子质量大小适当的DNA Marker做分子质量标记。选择合适的电压(80~90 V),电泳时间为50~60 min。用凝胶成像仪观察、拍照、记录与分析。
1.3.5 PCR扩增产物的DNA测序与分析
PCR扩增产物委托铂尚(Biosune)生物技术(上海)有限公司进行双向测序,各基因片段序拷贝在word格式文档中,序列首尾各用相应的8 个碱基小片段作为查找的引子,截取特异性目的片段,作为各鳗鱼物种标准DNA条形码,供比对鉴定。用DNAMAN V6(中文版)进行DNA序列整理,以*.seq格式输出,然后进行同源性比对与同源树构建,比对结果与同源树以*.emf格式输出。
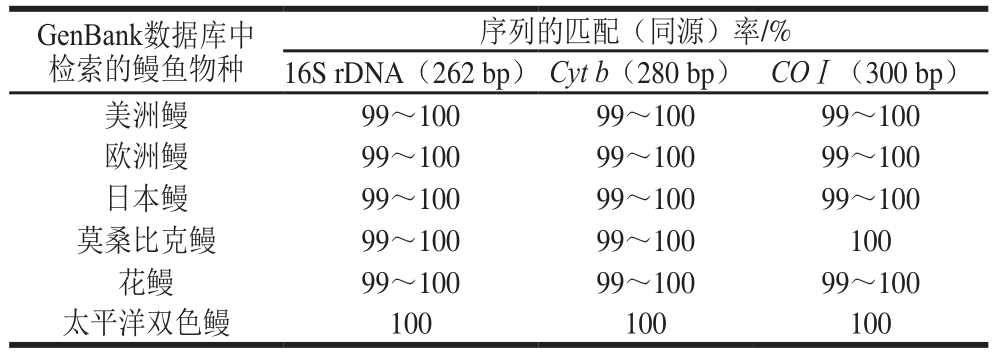
采用3 对通用引物(表1)对6 种鳗鱼的3 个基因(16S rRNA、Cyt b、COⅠ)部分DNA进行PCR扩增,PCR产物双向测序,6 种鳗鱼各获得3 条16S rDNA、Cyt b、COⅠ基因部分DNA序列,片段长度分别为638~643、464~466、705~707 bp,在GenBank数据库中BLAST比对,分别与数据库中的美洲鳗、欧洲鳗、日本鳗、莫桑比克鳗、花鳗、太平洋双色鳗的相应基因序列的同源率均为99%,序列ID号见表2。基于各基因的6 条序列的同源性比对,Cyt b基因部分图见图1(16S rRNA与COⅠ基因图略),排除序列中存在碱基缺失部分与不具差异的区间,选取各物种序列同源的片段(图1,下划黑线部分)用于设计新引物(16S rDNA 504、Cyt b 400、COⅠ609,表1)。

图1 6 种鳗鱼Cyt b基因部分DNA片段(464~466 bp)序列比对
Fig. 1 Sequence (464–466 bp) alignment of Cyt b gene fragments among 6 eel species
表2 6 种鳗鱼的16S rDNA、Cyt b、COⅠ基因通用引物PCR扩增产物序列在GenBank中对应的同源率最高的DNA序列号
Table 2 GenBank DNA sequence accession numbers with the highest homology to PCR amplif i ed products with universal primers of the 16S rDNA, Cyt b and COⅠ genes of six eel species


图
2 6 种鳗鱼16S rRNA基因(A)、Cyt b基因(B)和COⅠ基因(C)的3 条DNA片段(504~507、400、609 bp)PCR产物电泳图
Fig. 2 PCR amplif i ed results of three DNA fragments (504–507, 400, and 609 bp) of 16S rRNA (A), Cyt b (B) and COⅠ(C) gene in 6 species of eel
应用自主设计的3 对引物16S rDNA 504、Cyt b 400、COⅠ609(表1)对6 种鳗鱼进行PCR扩增与测序,如图2所示,从各序列中筛选、截取6 种鳗鱼间碱基序列同源性低、物种特异性强、不存在碱基缺失或插入的片段,作为6 种鳗鱼的16S rRNA、Cyt b 、COⅠ3个基因标准DNA条形码,片段长度分别为262、280、300 bp,如图3所示,各物种序列相同的首尾下划线部分8 个碱基为截取目的片段的引子,用于在word文档中“编辑-查找”(参照文献[27])。6 个鳗鱼物种各3 个基因共18 条DNA条形码碱基序列在GenBank数据库中检索比对(BLAST)的结果(按同源率高低排序显示出100 条),如表3所示。除太平洋双色鳗外,日本鳗、美洲鳗、欧洲鳗、莫桑比克鳗、花鳗的DNA序列匹配(同源)率除了个别情况,均为99%~100%。在16S rDNA条形码碱基序列比对中,欧洲鳗样品的DNA条形码在GenBank数据库中比对与一个A. rostrata(KJ564271.1)片段同源率为100%,可能是序列提供者提交时弄错种名。样品莫桑比克鳗的Cyt b(280 bp)DNA条形码比对中,出现与一个印度洋双色鳗(A. bicolor bicolor)(AB021780.1)片段同源率是100%,也可能是序列提供者提交了错误的种名。另外,在供试样品太平洋双色鳗的3 个基因DNA条形码与GenBank数据库比对中发现,供试样品与印度洋双色鳗物种亲缘关系非常接近,16S rDNA条形码碱基序列比对中,同源率为100%的,排序前面100 条片段中,前6 个片段是太平洋双色鳗,接在后面的是82 条印度洋双色鳗,同源率为99%的12 条也都是印度洋双色鳗;在Cyt b(280 bp)DNA条形码比对中,同源率为100%的16 个片段都是太平洋双色鳗,同源率99%的57 条中,太平洋双色鳗占23 条,印度洋双色鳗占34 条,余下同源率98%的27 条也均为印度洋双色鳗;在样品太平洋双色鳗的COⅠ(300 bp)DNA条形码序列与GenBank数据库比对中发现,同源率为100%的只有1 条片段(A. bicolor pacifica,AP007237.3),其他99 条均是同源率为98%的印度洋双色鳗。综合上述分析结果,本研究归纳出6 个鳗鱼物种的3 个基因标准DNA条形码序列应用于鳗鱼物种鉴别实践的同源率判断指标如表4所示。例如,某盲样3 个基因的序列与美洲鳗标准DNA条形码同源率均为99%~100%,可判定该样品为美洲鳗,如果与莫桑比克鳗(A. mossambica)标准DNA条形码16S rDNA(262 bp)与Cyt b(280 bp)的同源率均为99%~100%,与COⅠ(300 bp)的同源率为100%,样品可判定为莫桑比克鳗;某盲样3 个基因的序列与太平洋双色鳗(A.bicolor pacif i ca)标准DNA条形码同源率均为100%,可判定该样品为太平洋双色鳗。


图3 6 种鳗鱼的16S rRNA基因(A)、Cyt b基因(B)和COⅠ基因(C)的3 条DNA条形码(262、280、300 bp)标准序列
Fig. 3 Three standard DNA barcode sequences (262, 280, and 300 bp)based on 16S rRNA (A), Cyt b (B) and COⅠ(C) gene fragments in 6 species of eel
表3 6 个鳗鱼物种的3 个基因共18 条DNA条形码碱基序列在GenBank数据库中比对的结果
Table 3 Alignment of 18 DNA barcode sequences from 3 genes in 6 eel species with the GenBank database
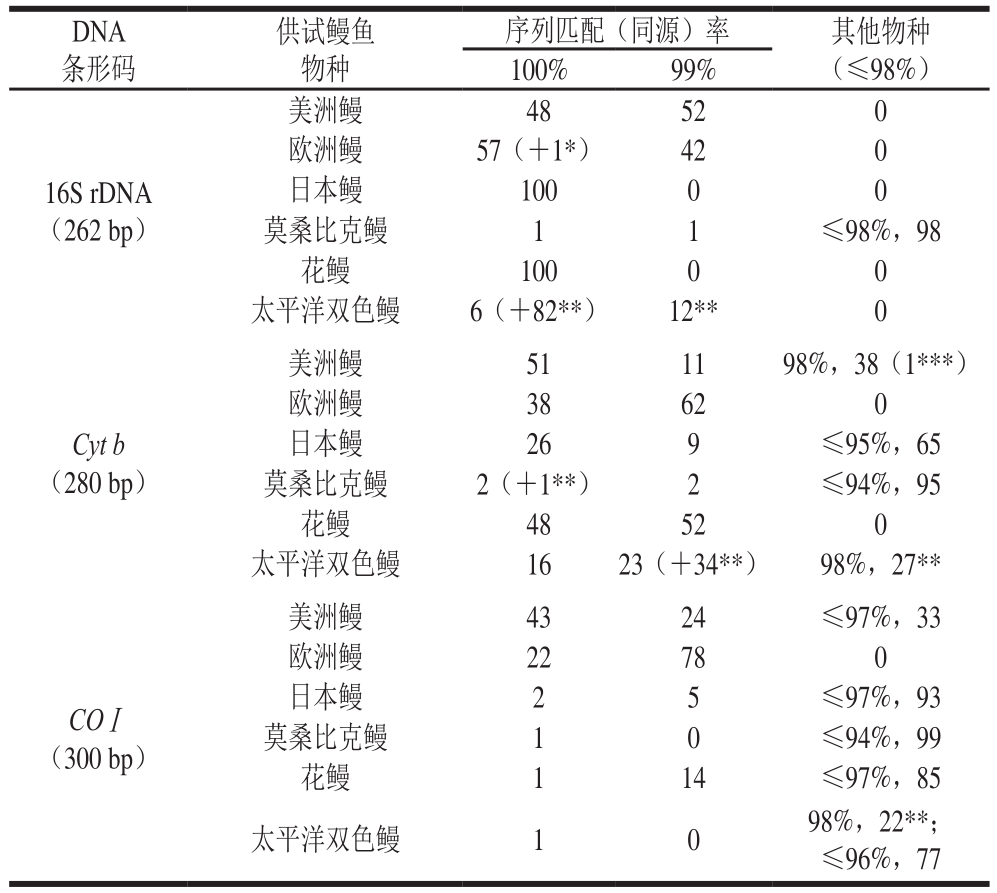
注:*.16S rDNA(262 bp)的欧洲鳗中有1 条美洲鳗(A. rostrata)(KJ564271.1);**.印度洋双色鳗(A. bicolor bicolor);***.在Cyt b(280 bp)中美洲鳗(A. rostrata)有1 条同源率是98%。
表4 6 个鳗鱼物种的18 条标准DNA条形码序列应用判断指标
Table 4 Homology of 18 standard DNA barcode sequences for identif i cation of 6 eels species
2.3.1 DNA条形码鉴定方法的建立
样品制作、DNA提取、DNA质量检测、PCR扩增与测序均按照1.2.2~1.2.5节进行。以鳗鱼DNA为模板,采用表1中自主设计的3 对引物,进行16S rRNA、Cyt b、COⅠ编码基因的部分DNA片段序列的PCR扩增,PCR产物经DNA测序,大小分别为504~507、400、609 bp,在word页面上用图3中下划线部分8 个碱基作为引子从各片段中截取出具有6 种鳗鱼物种特异性强的、碱基数分别为262、280、300 bp的3 条DNA片段序列,作为6 种鳗鱼物种的标准DNA条形码。用同样方法,截取待检鳗鱼样品的3 个基因DNA片段,借助DNAMAN V6软件,待检鳗鱼样品的3 个基因DNA片段与相应的标准DNA条形码进行序列同源性比对,3 条DNA条形码比对结果互相验证,与某物种同源率符合表4中的指标,即可判别待测样品为某物种,从而精准鉴定待检鳗鱼样品的所属物种。如果不符合表4中的指标,可以在GenBank数据库中检索比对(BLAST)。
2.3.2 DNA条形码鉴定方法在鳗鱼物种鉴定中的应用
表5 DNA条形码技术在30 个鳗鱼样品物种鉴定中的应用
Table 5 Application of DNA barcoding method in species identif i cation of 30 eel samples
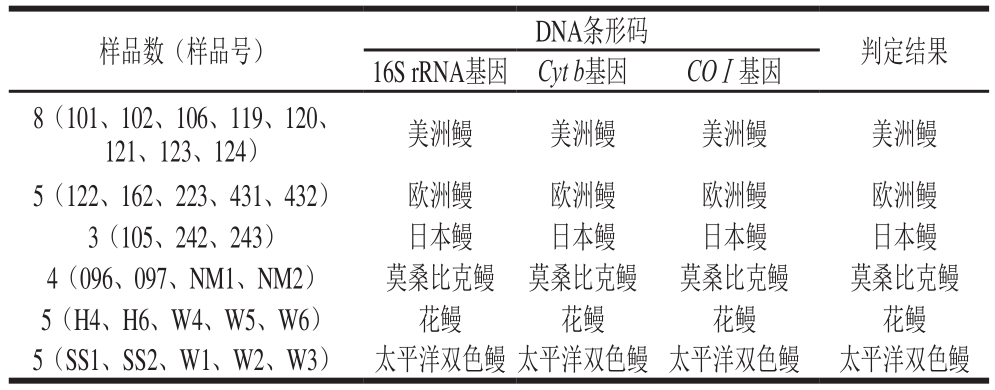
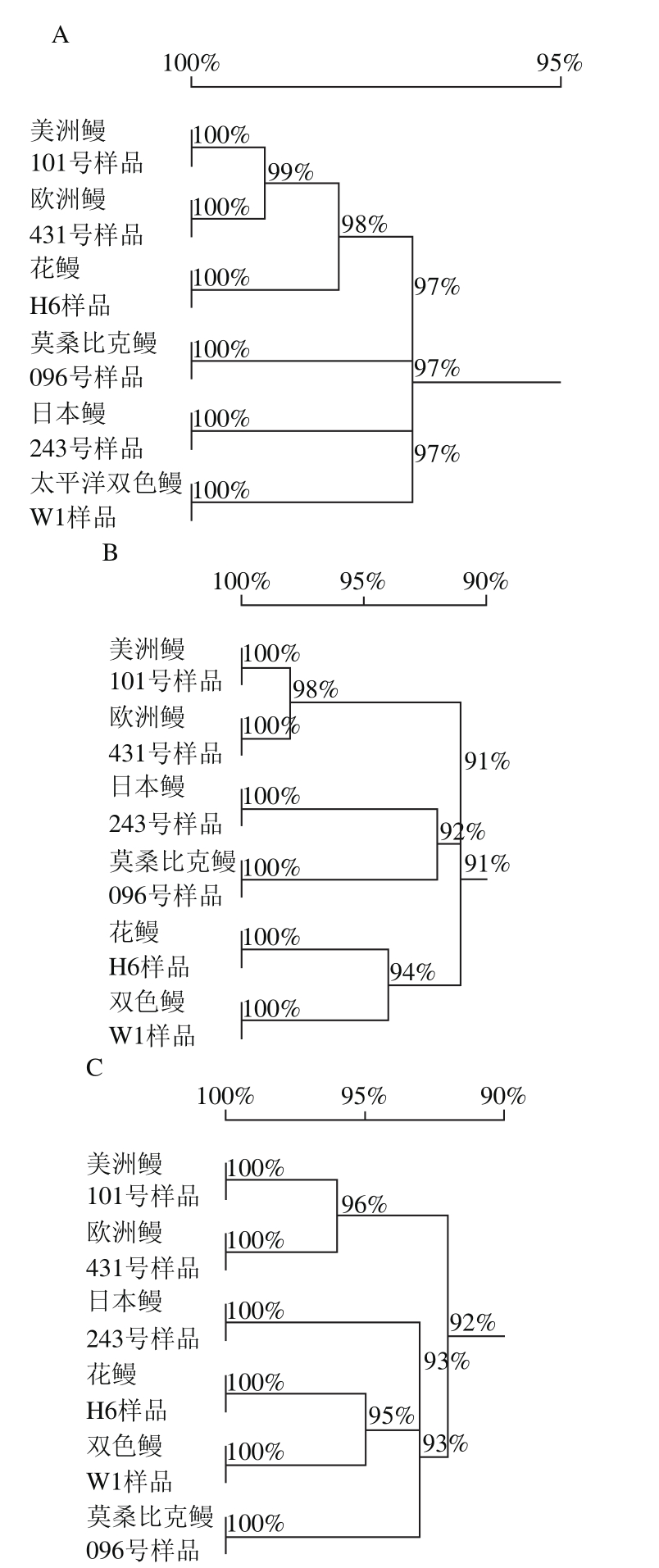
图4 6 种待测样品16S rRNA基因(A)、Cyt b基因(B)和COⅠ基因(C)的3 条DNA条形码与6 种鳗鱼对应基因标准DNA条形码(262、280、300 bp)的比对同源树
Fig. 4 Homology tree based on three DNA barcode sequences (262,280, and 300 bp) from 16S rRNA (A), Cyt b (B) and COⅠ (C) gene fragments among 6 samples and 6 known species of eel
如表5所示,各样品3 个基因的DNA序列与相应DNA标准条形码的同源率均为100%(图4,其他样品图略)。其中,8 个样品判定为美洲鳗、5 个样品为欧洲鳗,3 个样品为日本鳗,4 个样品为莫桑比克鳗,5 个样品为花鳗,5 个样品为太平洋双色鳗。
Hebert等[13]最早提出DNA条形码的定义,以COⅠ基因部分DNA序列作为物种分类指标,种内遗传距离一定要小于种间距离,种间遗传距离在2%以上,且是种内的10 倍以上。COⅠ、COⅡ基因序列应用于鳗鱼系统发育与分类的研究,发现种内个体间也存在遗传距离[21-25],虽然比种间距离小,但在其他物种存在种内与种间距离重叠的现象[21],即有时种内距离大于种间。由于种间界限不够明显,存在模糊地带,因此,DNA条形码选择,关系到物种间界限明了与否,能否准确判断待检样品所属具体物种。Vandamme[25]与Stein[26]等应用COⅠ基因序列鉴别非法贸易的濒危物种欧洲鳗,结果因存在待测样品同时与GenBank数据库中2 个以上物种序列同源率相同的问题,即种间界限不明显,无法做出判定,只能借助其他数据库(如BOLD(The Barcode of Life Data System))收录的DNA序列加以比对,做出综合判定,主要原因是所用的DNA片段太长,碱基数太多,序列同源性比对时,难以将物种间的同源率距离拉开。最理想的物种鉴定DNA条形码指标,应该是物种间碱基序列差异大、同源率低的DNA序列。可见,作为物种特异指标的条形码DNA片段选择至关重要。
随着基因条形码技术的发展,除了COⅠ基因DNA序列作为标准条形码[11-13]外,作为重要条形码的线粒体16S rRNA、Cyt b等基因的DNA序列,近年也越来越多地被应用于物种鉴别和遗传多样性分析[14-20]。本研究在应用基于16S rRNA基因部分DNA片段建立的DNA条形码(2 4 3 b p)鉴定美洲鳗、欧洲鳗、日本鳗3 种鳗鱼的基础上[27],先采用3 对通用引物对6 种鳗鱼的3 个基因(16S rRNA、Cyt b、COⅠ)部分D N A片段进行P C R扩增、测序,6 种鳗鱼各获得3 条16S rDNA(638~643 bp)、Cyt b(464~466 bp)、COⅠ(705~707 bp)基因的DNA片段。但由于通用引物适用于许多物种,往往不同物种,在PCR产物靶DNA片段首尾两端引物位置,PCR扩增常常碱基配对不好,影响PCR产物的测序结果,重复性不好,不宜作为条形码序列,因此从通用引物的PCR产物序列中选出稳定的、各供试物种同源性相同的区域,设计了3 对新引物,对6 种鳗鱼的16S rRNA、Cyt b、COⅠ基因进行PCR扩增,其产物大小分别为504~507、400、609 bp,再从各片段中截取出具有6 种鳗鱼物种特异性强的、碱基数分别为262、280、300 bp的3 条DNA片段,作为6 种鳗鱼物种3 个基因的标准DNA条形码,应用DNAMAN V6软件进行同源性分析,建立了鳗鱼物种的多基因条形码检测方法。本研究之所以要用3 个基因的标准DNA条形码鉴定物种,是因为3 个基因的条形码组合使用,可以砌高物种间的界限“围墙”,能更清晰区分物种,也为有效区分尚未收集到的鳗鱼物种与已知供试6 个物种,提供基因数据“围墙”,避免可能存在种间界限模糊的问题。
在3 个基因16S rRNA(262 bp)、Cyt b(280 bp)与COⅠ(300 bp)各6 条标准条形码在GenBank数据库中比对的基础上,建立了应用于鳗鱼物种鉴别实践的同源率判断指标(表4)。待测鳗鱼样品获得的3 条条形码与本方法建立的3 条标准条形码之间的同源率符合表4的判定指标时,就能判定其所属物种,不存在待测样品同时与GenBank中多个物种DNA序列同源率相同而无法进行物种判定的问题。本方法可能还存在缺陷,应用者如有检测结果不符合表4的各项指标,可以在GenBank数据库中加以比对验证,再作判断。
本研究建立的方法在30 个待检鳗鱼模拟盲样的物种鉴定中应用,结果表明:各样品中获得的3个基因的DNA片段与相应基因的标准DNA条形码进行序列同源性比对,同源率均为100%,获得的物种判别结果互相吻合,从而精准判别鳗鱼样品所属的物种,与预期结果完全一致,表明该方法稳定、精准、易于操作,可应用于6 种鳗鱼的物种鉴定,值得推广。目前还只收集到6 个物种,将收集更多的鳗鱼物种样品进行进一步研究,建立适合于更多鳗鱼物种的检测方法。
参考文献:
[1] The IUCN red list of threatened species. Anguilla anguilla[DB/OL].http://www.iucnredlist.org.
[2] JACOBY D M P, CASSELMAN J M, CROOK V, et al. Synergistic patterns of threat and the challenges facing global anguillid eel conservation[J]. Global Ecology & Conservation, 2015, 4: 321-333.DOI:10.1016/j.gecco.2015.07.009CrossRef.
[3] 张舒亚, 金丽琴, 蒋剑琼, 等. 食品中鱼源性成分PCR的检测方法[J].食品与发酵工业, 2010, 36(1): 142-145.
[4] 陈文炳, 赵晨, 邵碧英, 等. PCR方法检测河豚鱼的引物筛选及反应体系的优化[J]. 食品科学, 2010, 31(20): 376-381.
[5] 陈文炳, 林少华, 邵碧英, 等. 河豚鱼Cyt b基因部分DNA序列分析与应用[J]. 食品科学, 2012, 33(20): 227-232.
[6] 陈双雅, 陈文炳, 张津, 等. 应用PCR-RFLP和芯片生物分析系统鉴别河豚鱼品种[J]. 食品科学, 2012, 33(22): 200-202.
[7] 曲良苗, 陈文炳, 缪婷玉, 等. 限制性内切酶酶切确证河豚鱼成分PCR检测结果[J]. 食品科学, 2014, 35(8): 169-173. DOI:10.7506/spkx1002-6630-201408034.
[8] 杨弘, 王希道, 吴婷婷, 等. 用RAPD技术研究3 种鳗的种质鉴定[J]. 中国水产科学, 2002, 9(3): 269-272. DOI:10.3321/j.issn:1005-8737.2002.03.018.
[9] 胡庚东, 尤洋, 陈家长. 澳洲鳗、日本鳗和欧洲鳗的RAPD初步比较分析[J]. 浙江海洋学院学报(自然科学版), 2006, 25(3): 254-257.DOI:10.3969/j.issn.1008-830X.2006.03.004.
[10] 张新艳, 樊海平, 范斯敏, 等. 应用RAPD技术对6 种玻璃鳗的种质鉴定和遗传多样性分析[J]. 江西农业大学学报, 2010, 32(4): 683-688.DOI:10.3969/j.issn.1000-2286.2010.04.010.
[11] 刘光明, 史千玉, 曹敏杰, 等. 利用PCR和限制性酶切技术鉴别3 种鳗鱼[J]. 集美大学学报(自然科学版), 2011, 16(3): 178-181.DOI:10.3969/j.issn.1007-7405.2011.03.004.
[12] KESZKA S, PANNICZ R, KEMPTER J. Eel species identification by polymerase chain reaction followed by reaction fragment length polymorphism (PCR-RFLP)[J]. Medycyna Wet, 2009, 65(5): 315-318.
[13] HEBERT P D N, CYWINSKA A, BALI S L, et a1. Biological identifications through DNA barcodes[J]. Proceedings of Royal Society of London, 2003, 270: 313-321. DOI:10.1098/rspb.2002.2218.
[14] 彭居俐, 王绪祯, 王丁, 等. 基于线粒体COⅠ基因序列的DNA条形码在鲤科鲌属鱼类物种鉴定中的应用[J]. 水生生物学报, 2009,33(2): 271-276.
[15] 李新光, 王璐, 赵峰, 等. DNA条形码技术在鱼肉及其制品鉴别中的应用[J]. 食品科学, 2013, 34(18): 337-342. DOI:10.7506/spkx1002-6630-201318069.
[16] 邵爱华, 朱江, 陈葵, 等. 暗纹东方豚线粒体COⅡ及两侧tRNA基因的克隆和序列分析[J]. 动物学杂志, 2005, 40(6): 1-8. DOI:10.3969/j.issn.0250-3263.2005.06.001.
[17] 邵爱华, 朱江, 陈葵, 等. 暗纹东方豚线粒体COⅠ及其侧翼tRNA基因的克隆与序列分析[J]. 遗传, 2006, 28(8): 963-971. DOI:10.3321/j.issn:0253-9772.2006.08.011.
[18] 毕潇潇, 高天翔, 肖永双, 等. 4 种鳕鱼线粒体16S rRNA、COⅠ和Cyt b基因片段序列的比较研究[J]. 南方水产科学, 2009, 5(3): 46-52.DOI:10.3969/j.issn.1673-2227.2009.03.008.
[19] 陈文炳, 翁国柱, 陈融斌, 等. 河豚鱼16S rRNA基因部分DNA序列分析及应用探讨[J]. 食品科学, 2015, 36(21): 140-144. DOI:10.7506/spkx1002-6630-201521027.
[20] 马梅, 李薇, 龚玲, 等. 基于COⅠ和16S rRNA基因的地龙药材及其混淆品的DNA条形码鉴定[J]. 中药新药与临床药理, 2014, 25(2):5954-598. DOI:10.3969/j.issn.1003-9783.2014.05.018.
[21] 李海涛, 张保学, 高阳, 等. DNA条形码技术在海洋贝类鉴定中的实践: 以大亚湾生态监控区为例[J]. 生物多样性, 2015, 23(3): 299-305.DOI:10.17520/biods.2014261.
[22] 吴宁, 黎中宝, 林小云, 等. 6 种鳗鲡(Anguilla)线粒体DNA COⅠ序列的比较研究[J]. 海洋与湖沼, 2010, 41(6): 930-934.
[23] 田鑫江, 吴宁, 黎中宝, 等. 6 种鳗鲡(Anguilla)线粒体DNA COⅡ基因序列分析及其系统发育[J]. 海洋与湖沼, 2011, 42(2): 279-283.DOI:10.11693/hyhz201102018018.
[24] 龚小玲, 岳丽佳, 崔忠凯, 等. 鳗鲡属6 种鱼线粒体COⅠ和COⅡ基因序列分析和分类的有效性[J]. 上海海洋大学学报, 2013, 22(4):524-530.
[25] VANDAMME S G, GRIFFITHS A M, TAYLOR S, et al. Sushi barcoding in the UK: another kettle of fi sh[J]. Peerj, 2016, 4: e1891.DOI:10.7717/peerj.1891.
[26] STEIN F M, WONG J C Y, SHENG V, et al. First genetic evidence of illegal trade in endangered European eel (Anguilla anguilla) from Europe to Asia[J]. Conservation Genetics Resources, 2016, 8(4): 1-5.DOI:10.1007/s12686-016-0576-1.
[27] 陈文炳, 缪婷玉, 彭娟, 等. 基于16S rRNA基因DNA条形码鉴定美洲鳗、欧洲鳗、日本鳗[J]. 食品科学, 2017, 38(4): 283-289.DOI:10.7506/spkx1002-6630-201704046.
[28] INOUE J G, MIYA M, TSUKAMOTO K, et al. Amitogenomic perspective on the basal teleostean phylogeny: resolving higher-level relationships with longer DNA sequences[J]. Molecular Phylogenetic Evolution, 2001, 20(2): 275-285. DOI:10.1006/mpev.2001.0970.
[29] WOLF C, BURGENER M, HUBNER P, et al. PCR-RFLP analysis of mithochondrial DNA: differentiation of fish species[J]. LWTFood Science and Technology, 2000, 33(2): 144-150. DOI:10.1006/fstl.2000.0630.
[30] WARD R D, ZEMLAK T S, INNES B H, et al. DNA barcoding Australia’s fish species[J]. Philosophical Transactions of the Royal Society of London, 2005, 360(1462): 1847-1857. DOI:10.1098/rstb.2005.1716.
Identif i cation of Six Eel Species Using Polygenic DNA Barcoding
CHEN Wenbing, SHAO Biying , MIAO Tingyu, PENG Juan, CHEN Bin, ZHANG Zhideng, JIANG Shuxun
(Fujian Provincial Key Laboratory of Inspection and Quarantine Technology Research, Inspection and Quarantine Technology Center,Fujian Entry-Exit Inspection and Quarantine Bureau, Fuzhou 350003, China)
Abstract:This paper develops an accurate method to identify six eel species including Anguilla rostrata, A. anguilla, A.japonica, A. mossambica, A. marmorata, and A. bicolor pacif i ca using polygenic DNA barcoding. Three pairs of universal primers were used to amplify partial fragments of the 16S rRNA, Cyt b and COⅠgenes of the eel species with the total genomic DNA from eel as a template and the amplif i ed products were sequenced. As a result, 16S rDNA (638–643 bp), Cyt b(464–466 bp) and COⅠ(705–707 bp) gene fragments were obtained for each species. Homologous sequences of these genes were found. Three new primer pairs were designed to amplify 638–643, 400 and 609 bp fragments of the 16S rRNA,Cyt b and COⅠ genes by PCR, respectively. The specif i c 16S rRNA, Cyt b and COⅠfragments of 262, 280 and 300 bp were selected as standard DNA barcodes for the identification of eel species. Homology analysis was performed using DNAMAN software (version 6) and the homology for species discrimination was developed by aligning the DNA barcodes with the GenBank database. Analysis of 30 samples from six eel species by the developed method showed that the species were consistently identif i ed using the DNA barcodes. In conclusion, the method is stable, precise and easy to operate and can be applied to the identif i cation of six species of eels.
Keywords:16S rRNA; Cyt b; COⅠ; polygenic DNA barcode; identif i cation of eel species
DOI:10.7506/spkx1002-6630-201802026
中图分类号:S917.4;Q78
文献标志码:A
文章编号:1002-6630(2018)02-0163-07
引文格式:陈文炳, 邵碧英, 缪婷玉, 等. 多基因DNA条形码鉴定6 个鳗鱼物种[J]. 食品科学, 2018, 39(2): 163-169.
DOI:10.7506/spkx1002-6630-201802026. http://www.spkx.net.cn
CHEN Wenbing, SHAO Biying, MIAO Tingyu, et al. Identif i cation of six eel species using polygenic DNA barcoding[J]. Food Science,2018, 39(2): 163-169. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201802026. http://www.spkx.net.cn
收稿日期:2017-06-02
基金项目:福建省科技计划农业引导性项目(2015N0016)
第一作者简介:陈文炳(1962—),男,研究员,博士,研究方向为农产品、食品分子生物学检测技术。E-mail:621213wbc@163.com